一种双特异性嵌合抗原受体分子及其在肿瘤治疗上的应用的制作方法
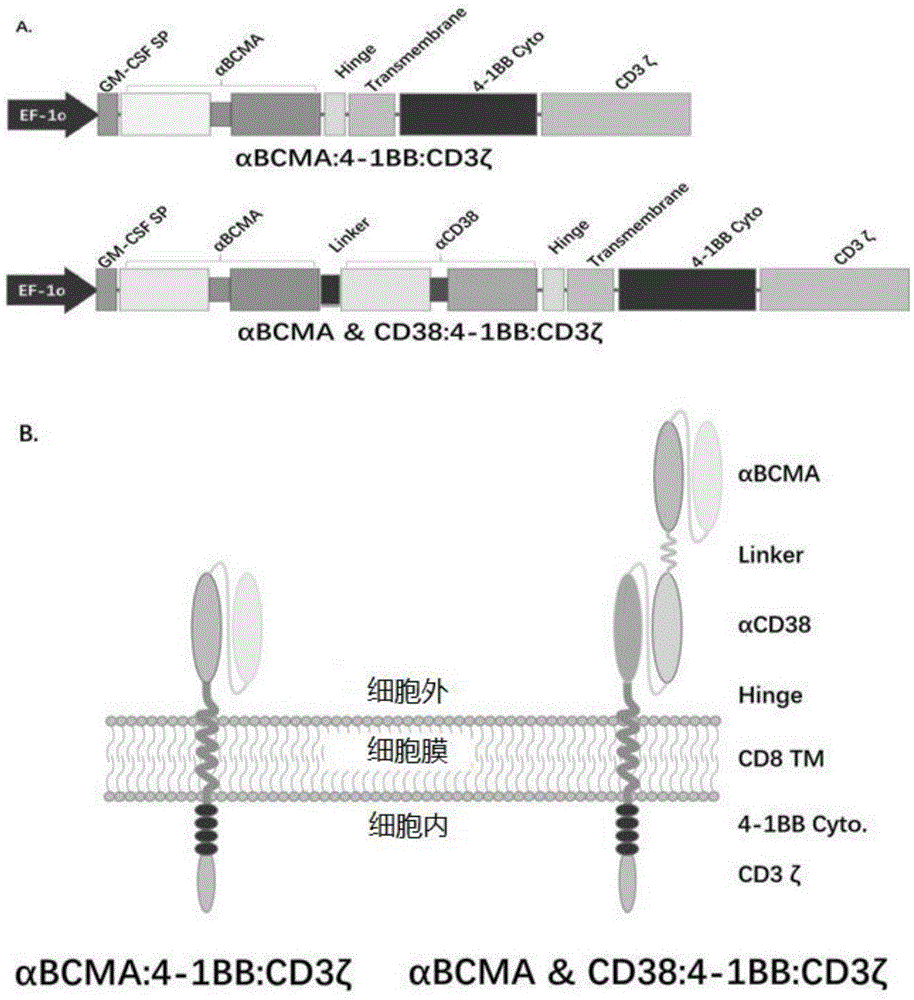
本发明涉及细胞免疫治疗领域,具体涉及一种双特异性嵌合抗原受体分子及其在肿瘤治疗上的应用。
背景技术:
:随着肿瘤免疫学理论和临床技术的发展,嵌合抗原受体t细胞疗法(chimericantigenreceptort-cellimmunotherapy,car-t)成为目前最有发展前景的肿瘤免疫疗法之一。嵌合抗原受体car由一个肿瘤相关抗原结合区、胞外铰链区、跨膜区域以及胞内信号转导区组成。car-t细胞疗法通过外源基因转染技术,把识别肿瘤相关抗原的单链抗体(singlechainfragmentvariable,scfv)和t细胞活化序列的融合蛋白表达到t细胞表面,使可以特异识别肿瘤相关抗原的scfv通过跨膜区与t细胞胞内的活化增殖信号域偶联。表达car的t细胞以抗原依赖、但非mhc限制的方式结合肿瘤抗原,启动并活化特异性杀伤肿瘤反应。car-t细胞的有效激活均严重依赖识别肿瘤相关抗原的抗体的特异性以及抗原结合的亲和力高低等性质。在目前car-t细胞胞内信号转导区的设计已经趋于成熟的现状下,抗原结合区的设计成为新型car-t技术开发的重点和关键,而其中的重点是防止脱靶问题。car-t淋巴细胞临床应用的首要风险是脱靶效应,可导致针对正常组织或细胞的免疫反应。由于目前已知的肿瘤特异性抗原较少或缺乏肿瘤细胞特异性的抗原,大多数car针对的是重要组织不表达或表达量极少的肿瘤相关抗原。因此,如何提高car-t淋巴细胞的靶向性是目前临床应用面临的首要问题。car-t细胞的激活及产生对靶细胞的有效杀伤依赖于识别并结合肿瘤相关抗原的抗体的亲和力。表达在t细胞膜上的car通过最前端的scfv抗原识别区特异性的结合肿瘤细胞膜表面的肿瘤抗原,使得肿瘤细胞和car-t细胞发生物理性的接触,car结构产生形变并将t细胞激活信号杀伤信号传递到t细胞内。在此期间car-t细胞表面形成类似于神经突触样的免疫突触结构包裹部分肿瘤细胞,免疫突触形成的大小、强度等与car受体对肿瘤相关抗原的亲和力直接相关,car对肿瘤相关抗原的亲和力越大,则形成的免疫突触越大。car-t细胞与肿瘤细胞形成免疫突触,同时car结构的构象变化也引起t细胞的激活信号和杀伤效应信号的传导,诱导t细胞释放穿孔素、颗粒酶以及其他效应因子的转录、表达以及分泌。针对car结构中不同近膜区、不同肿瘤抗原靶点以及不同共刺激信号区的结构域功能的关联分析表明,抗原结合区对肿瘤相关抗原的亲和力直接影响car结构的构象转变能力及向t细胞内传递信号的能力,即二者的亲和力越大,传导到car-t细胞的t细胞激活和杀伤效应信号就越强。另外,从抗体对抗原的结合的动力学平衡关系来考虑,抗体对抗原的亲和力越高,在单位时间内二者发生解离的概率越低,宏观上解释即说明car-t细胞一旦识别并结合带有正确肿瘤相关抗原的肿瘤细胞,不易发生解离使肿瘤细胞逃逸。综上所述,对具有较高特异性的肿瘤抗原或者如cd19、cd20等仅仅表达在成熟b淋巴细胞上的抗原,以较高的亲和力的抗体为基础构建的car-t细胞可以获得高效率的杀伤及优异的临床治疗效果。对于car-t技术和产品的开发者来说,如何增强car-t细胞在患者体内的持续性是关系到car-t的长期治疗效果的关键之一。car-t的研究和开发历史主要是围绕靶点开发和增强car-t的杀伤活性为主,从一代的car-t到现如今的四代car-t的技术变迁体现在了car-t的胞内信号区和共刺激分子的变化。最早的car只用cd3ζ信号链的酪氨酸序列作为共刺激信号来激活t细胞,虽然可以激活car-t细胞并使其靶向地清除细胞,可惜无法促进细胞的持续增殖和促进il-2的分泌,导致t细胞在体内很快就凋亡,其持久性成为第一代car-t细胞在临床应用上的一大障碍。为了提高活性,第二代car-t细胞在信号1的基础上加上了一个共刺激信号cd28或cd137,作为“信号2”。信号2不仅促进t细胞的分裂,il-2的合成与表达和抗凋亡蛋白bcl-xl的分泌,还能够使其抵消肿瘤细胞微环境带来的不利影响,但不影响其抗原特异性。不同研究都表明搭载了信号2的car-t细胞与第一代相比,展现出了更优越的显疗效和持续性。而第三代的car-t,则是在信号1和信号2的基础上再加上一个共刺激信号分子,使其活性获得了进一步提高。除了在胞内信号区增加共刺激信号分子来提高细胞活性以外,研究者也利用其他方法来巧妙地改造car,使改造后的t细胞能在体内发挥最大的治疗效果。与肿瘤细胞的斗争中,car-t的免疫攻击常常由于肿瘤产生的免疫抑制信号而削弱。这些信号包括抑制性细胞因子il-4,il-10和肿瘤生长因子β(tgf-β)等,这些因子可由肿瘤微环境中的细胞或基质组分产生。moleculartherapy的一篇报道中,研究人员在靶向前列腺干细胞抗原(psca,一种在前列腺癌细胞中高表达,但是在正常细胞中不表达的蛋白)的car-t细胞上进一步改造,将il-4受体胞外段与il-7受体胞内段结合使这些car-t细胞产生了一种新的反转细胞因子受体(icr),研究表明,在一种产生il-4的胰腺癌细胞模型,这些表达icr的t细胞在il-4的刺激下增殖能力反而升高了。纵观从一代car-t到四代car-t的结构与临床的疗效可以发现,car-t细胞通过识别和结合肿瘤细胞的抗原,传到激活、杀伤和增殖信号到t细胞内部,期间car-t细胞行使杀伤肿瘤细胞的功能,之后大部分细胞发生功能耗竭而凋亡,只有极少部分的car-t细胞可以转变成记忆性的car-t细胞,这一部分记忆性的t细胞与患者的长期疗效直接相关。因此,在car结构的设计与治疗过程中,能促进长期记忆性t细胞形成的结构和机制有助于患者获得长期受益,例如,采用cd137的共刺激信号,使得car-t细胞在收到肿瘤抗原刺激发生增殖及杀伤时的强度更为缓和,延缓car-t细胞发生功能耗竭的时间以及促进更多的记忆性t细胞的产生。更为有效的策略是在设计car结构时,人为的设计针对特异或非特异抗原的低亲和力的car结构,持续给予car-t细胞以极弱的信号刺激,促进car-t细胞的存活、增殖以及向记忆性car-t细胞的分化。car结构中来源于鼠源单克隆抗体scfv序列在回输给肿瘤患者之后会引起严重的人抗鼠抗体反应(hama),严重制约了鼠源抗体来源的治疗性药物的安全性和临床疗效。由于历史原因,全球开发car-t产品如cd19car-t的企业大多采用鼠源的fmc63单抗株,鼠源抗体会诱导人体产生排斥反应,严重缩短了car-t细胞在患者体内的持续性,最终会影响临床疗效及较快的复发。例如诺华已上市的car-t产品kymriah以fmc63位抗原识别去,治疗后6个月内即有10%的患者复发,12个月后有45%左右的患者发生复发,car-t细胞引入的鼠源成分诱导的免疫排斥即是其中一个重要原因。采用基因工程手段对鼠源抗体进行人源化改造或者直接筛选人员抗体库获得人源抗体,再构建人源car-t,理论上有助于减少或避免患者免疫系统对car-t细胞的免疫排斥,维持car-t细胞在患者体内长期存在,提升长期治疗效果。多发性骨髓瘤(multiplemyeloma,mm)是骨髓和外周血中恶性浆细胞克隆性增殖引起的疾病,导致骨髓造血抑制和溶骨性症状。尽管应用传统药物治疗和造血干细胞移植以及靶向性药物(如化疗药物、蛋白酶体抑制剂类药物以及免疫调节类药物)的治疗可以获得十分不错的临床效果,大部分的患者在经过一定阶段的治疗后或发生药物耐受或复发进展。现阶段,mm仍然属于一类不能获得临床治愈的恶性疾病,其中一个重要原因就是随着疾病进展而出现的免疫缺陷和免疫耐受,因此免疫疗法在未来极有可能成为临床治愈mm的一种方法之一。天然免疫系统中,mm患者nk细胞的细胞毒性和免疫调节功能都受到削弱,肿瘤相关性巨噬细胞被激活,并且分泌大量促炎细胞因子如tnf-α、il-6等促进mm细胞生长。mm患者中的树突细胞吞噬细菌和递呈抗原能力下降,不能有效的利用肿瘤抗原刺激和活化t细胞以发挥抗肿瘤作用。获得性免疫系统中,mm患者的t细胞和b细胞的功能都被削弱,mm细胞和mm来源的骨髓基质细胞促使treg/th17平衡向th17漂移,而th17细胞具有免疫抑制功能并能促进肿瘤生长。无论是先天免疫系统,或者是后天获得新免疫系统都对mm发生免疫耐受,因此仅仅依赖患者自身的免疫调节不能有效对抗mm的发生和发展,及时在外界药物干预的mm细胞生长的条件下,免疫系统的调节作用依然不足以完全控制肿瘤的进展,因此现行的治疗策略中必须考虑强化患者自身免疫系统的作用和功能。car基因修饰的自体t细胞将肿瘤抗原特异性识别抗体或结构域与刺激t细胞激活增殖的共刺激信号进行重组融合,使其能在体外和体内环境中模拟细胞毒性t细胞或效应t细胞对肿瘤细胞的杀伤作用。最为重要的一点是这种基因工程修饰的car-t细胞在行使功能是具有mhc非依赖性的肿瘤抗原识别、和杀伤能力,且在特异抗原的刺激下这一类细胞能发生特异性的增殖。当这一部分car-t细胞回输患者后,在行使特异杀伤功能的同时,其中部分细胞或可以转化成为特异性的记忆性t细胞并在患者体内长期存活,当再次有肿瘤抗原刺激这一类记忆性的t细胞时,这部分细胞会发生快速增值并消灭新生的肿瘤细胞,如此患者可以获得长时间的临床缓解甚至治愈。近年来,靶向b淋巴细胞来源的恶性血液肿瘤的car-t疗法获得了极大的临床成功,例如靶向cd19、cd20、cd22等靶点的car-t可以使临床上复发难治性的急性b淋巴细胞白血病获得90%的完全缓解率,在b淋巴细胞来源的复发难治性淋巴瘤的治疗上也获得了50%的完全缓解率,充分说明car-t治疗在恶性肿瘤的治疗上大有可为。现阶段,国内外多家研究机构和药物开发企业的靶向mm特异性抗原的嵌合抗原受体t细胞陆续完成临床前开发并进入临床研究并取得了里程碑式的治疗效果。在2017年美国临床肿瘤学会年会的报告中,来自中国的南京传奇生物治疗公司报道了一款靶向cd269(b-cellmaturationantigen,bcma)的lcar-b38m-car-t疗法,有35名复发或耐药性多发性骨髓瘤患者参与此临床研究,该疗法的客观缓解率达到了100%。在最早接受治疗的19名患者里,14名达到了严格的完全缓解(scr),剩下5名部分缓解,其中有5名经治超过1年的患者依旧处于scr期;来自美国蓝鸟公司的car-t疗法bb2121也是针对cd269蛋白。接受3种不同剂量bb2121治疗的15名患者全部出现了客观反应,其中有4名患者达到了完全反应。还有3名患者接受了第四种较低的bb2121剂量治疗,结果全部不治。如果将这3名患者也计入其中,bb2121的整体反应率为89%。这些临床研究取得的成功使得mm的治疗进入一个全新的免疫治疗时代,未来靶向mm特异抗原的car-t治疗手段极有希望成为治愈mm的一种方案。bcma(bcellmaturationantigen,cd269)微量表达于成熟的b细胞和浆细胞,在mm中表达量大幅上调,是临床上诊断mm发生发展的一个可靠指标。同时,bcma的蛋白质仅仅表达在成熟b细胞或浆细胞中,在记忆性的b细胞和幼稚性b细胞及其他组织中并无bcma蛋白的表达,因此可以说bcma是一个非常理想的免疫治疗如car-t或者抗体药物治疗的靶点。针对bcma的adc药物(gsk2857916)和双靶向(t细胞和肿瘤细胞)抗体药物(bi836909)现正处于一期临床和临床前研发过程中。针对bcma的car-t在宾夕法尼亚大学,美国国立癌症研究所和bluebirdbio公司的bcmacar-t产品都已进入一期临床试验,国内的南京传奇生物技术有限公司的针对bcma的car-t产品已进入一期临床试验研究阶段。上述研究机构或者药品开发企业的bcmacar-t产品都是特异性靶向mm细胞膜上的bcma蛋白,但是mm细胞膜上表达的bcma蛋白在生理条件下可以被蛋白酶切割后进入血液循环,尤其是在mm病人血清中可溶性bcma蛋白水平会升高,且升高的水平与肿瘤恶性程度呈正相关。血清中游离的bcma可以结合靶向bcma的car-t细胞,直接引起car-t细胞的功能耗竭,或可降低靶向bcma的car-t细胞的抗肿瘤效果,因此迫切需要改进或者联合靶向bcma的car-t技术,以期到达更好的临床治疗效果。cd38是一个定位于膜上的糖蛋白,催化环腺苷二磷酸核糖(cadpr,cyclicadp-ribose)的合成和降解。cd38分子的表达与分布相当广泛,无细胞系限制性,在定向髓系和淋巴细胞系祖细胞表达水平很高,在nk细胞、t细胞、b细胞等也有一定的表达。与正常浆细胞相比,骨髓瘤细胞表面cd38的表达明显增加,细胞表面的cd38活化后促进细胞底物的磷酸化激活nf-κb信号通路,而nf-κb信号通路与mm细胞的耐药密切相关,这些证据说明cd38是一个潜在的多发性骨髓瘤治疗靶点。2015年fda加速批准了针对cd38的单克隆抗体药物darzalex(daratumumab)用于mm的治疗。daratumumab具有广谱杀伤活性,靶向mm细胞表面高表达的跨膜胞外酶cd38分子,可通过多种机制诱导肿瘤细胞的快速凋亡,延长病人的存活期,并且没有严重的髓细胞生长抑制作用。以cd38为靶点的car-t细胞(cd38car-t细胞)在体外特异性杀伤mm细胞系细胞和原代mm细胞,但是迄今还没有cd38car-t的临床数据报道。此外,传统的b细胞标志如cd19不被传统的mm免疫治疗所看好,但是去年upenn和novartis在nejm上报道的一个cd19car-t成功治疗mm的病例似乎为这一类抗原带来了新的希望。其文章指出,mm肿瘤细胞中一组抗药性强,增殖能力强(具有肿瘤干细胞特性的)的细胞克隆虽然cd19表达量低,但的确是cd19阳性的。被报道的这个病例中,尽管99.5%的恶性增生的浆细胞上都缺乏cd19的表达,该患者依旧获得完全治愈。由于患者接受了自体造血干细胞移植,car-t细胞治疗后的成效不排除asct的作用。如果cd19car-t在未来的mm临床实验中,在更多的病人体内证明了其疗效,将是mm治疗的一个重大突破。另外,诸如细胞膜表面糖蛋白(cs1或signalinglymphocyticactivationmolecule7,slam7)、免疫球蛋白轻链、lewisy抗原、食管鳞状细胞癌抗原(newyorkesophageal-1,ny-eso-1)、cd44的异构体6(cd44v6)、cd138等肿瘤抗原在mm的细胞系或患者样品中也有很高的检出率,未来靶向这些位点的抗体药物或car-t产品对mm的治疗也将具有积极的作用。技术实现要素:有鉴于此,本发明为了解决上述问题而提供了一种双特异性嵌合抗原受体修饰的t淋巴细胞。为实现上述目的,本发明通过如下技术方案予以实现:本发明的第一方面提供了一种嵌合抗原受体,所述嵌合抗原受体由信号肽,两个特异性抗原结合片段,膜外间隔区,跨膜区,胞内共刺激信号区组成,所述特异性抗原结合片段识别和结合的第一个抗原为cd19,cd20,cd22,cd33,cd269,cd138,cd79a,cd79b,cd23,ror1,cd30,b细胞表面抗体轻链,cd44,cd123,lewisy,cd7或cd46中的一种;特异性抗原结合片段识别和结合的第二个抗原为cd38,两个特异性抗原结合片段由一个连接肽连接。优选的,所述嵌合抗原受体,由细胞膜定位信号肽、cd269特异性抗原结合片段、连接肽、cd38特异性抗原结合片段、膜外间隔区、跨膜区、膜内信号转导区和共刺激结构域依次串联连接而成。更加优选的,所述的cd269抗原特异性结合片段scfv包括框架区和互补决定区cdr1-3,所述cd269抗原特异性结合片段scfv的氨基酸序列是序列表中seqidno:1-seqidno:90所列的氨基酸序列。所述的cd38抗原特异性结合片段scfv包括框架区和互补决定区cdr1-3,所述cd38抗原特异性结合片段scfv的氨基酸序列是序列表中seqidno:91-seqidno:180所列的氨基酸序列。进一步优选的,本发明筛选了不同亲和力的靶向cd269、cd38的人源单克隆抗体,使靶向cd38的单链抗体对靶向蛋白具有较低亲和力,靶向cd269的单链抗体对靶向蛋白具有高亲和力,具体的,所述cd269抗原特异性结合片段结合cd269的亲和力常数kd<5.4x10-8m。所述cd38抗原特异性结合片段结合cd38的亲和力常数kd介于5.2x10-7m和4.2x10-5m之间。在本发明中,特定亲和力的搭配组合使低亲和力具有快速结合快速释放抗原的能力,主要是能够增强bcma识别区抓取抗原的能力,避免低抗原密度的条件下亲和力不足,低亲和力抗原本身不具备杀伤效应。更加优选的,所述连接肽为:(ggggs)n或(eaaak)n,其中1≤n<4。两种分别代表柔性连接肽和刚性连接肽,研究发现刚性连接肽具有更好的杀伤效果,原因在于两个位点都结合,刚性连接肽带来的结构形变更大,引起的t细胞特异杀伤更强。作为进一步的优选,连接肽为eaaak。更加优选的,所述嵌合抗原受体的细胞膜定位信号肽选自以下细胞膜定位蛋白的膜定位信号肽:cd4,cd8,g-csfr,gm-csfr;为了在t细胞中更好的表达膜蛋白,选择的都是在t细胞中表达量比较高的蛋白的信号肽,作为进一步的优选,所述细胞膜定位信号肽包括cd8,gm-csfr的膜定位信号肽。更加优选的,所述嵌合抗原受体的膜外间隔区选自以下蛋白质分子的膜外结构域:cd4,cd8,cd28,cd137,cd27,pd-1,ox40,tlr4,icam-1,icos(cd278),nkp80(klrf1),nkp44,nkp30,nkp46。本发明优选t细胞的天然蛋白的跨膜区。更加优选的,所述嵌合抗原受体的跨膜区为选自以下蛋白质分子的跨膜结构域:cd4,cd8,cd28,cd137,cd27,pd-1,il2rβ,il2rγ,il7rα,nkg2d,nkg2c,igg4,igg1。本发明优选t细胞的天然蛋白的跨膜区,cd38靶点在car结构内侧,可以选用胞外区较短的结构域,结构紧凑,在结合抗原之后,引起的t细胞形变更大,杀伤反应更强。更加优选的,所述嵌合抗原受体的膜内信号转导区选自以下蛋白质分子的膜内结构域:cd2,cd3ζ,cd7,cd27,cd28,cd137,cd134,lck,tnfr-1,tnfr-2,fas,nkg-2d,dap10,dap12,b7-h3,tlr2,tlr4,il7r或其任意组合;优选地膜内信号转导区包括由cd137的胞内结构域与cd3ζ的胞内结构域的组合,对car-t细胞具有温和的刺激,具有更好的持续增殖能力。更加优选的,所述嵌合抗原受体包含:(1)序列表seqidno:1-seqidno180中所列的任一轻链可变区的氨基酸序列;或(2)具有序列表seqidno:1-seqidno180中提供的任一轻链可变区的氨基酸序列的至少一个修饰但不超过30个修饰的氨基酸序列;或(3)与序列表seqidno:1-seqidno180中提供的任一轻链可变区的氨基酸序列具有90-99%同一性的氨基酸序列。或者,更加优选的,所述嵌合抗原受体包含:(1)序列表seqidno:1-seqidno180中所列的任一重链可变区的氨基酸序列;或(2)具有序列表seqidno:1-seqidno180中提供的任一重链可变区的氨基酸序列的至少一个修饰但不超过30个修饰的氨基酸序列;或(3)与序列表seqidno:1-seqidno180中提供的任一重链可变区的氨基酸序列具有90-99%同一性的氨基酸序列。或者,更加优选的,所述嵌合抗原受体具有表4中所述的不同的构建体所描述的氨基酸序列。所述嵌合抗原受体的氨基酸序列由附表1中所述的核苷酸密码子编码。本发明还提供了上述双嵌合抗原受体修饰的t淋巴细胞的制备方法,制备过程简述如下:(1)合成car结构序列,构建慢病毒表达载体;(2)质粒提取制备慢病毒包装载体;(3)包装载体转染hek-293t细胞;(4)慢病毒收集与纯化;(5)t细胞分离、激活、病毒转导及细胞扩增。本发明第二方面提供了一种多肽,所述多肽编码上述的双靶向特异性嵌合抗原受体。本发明第三方面提供了一种基因,所述基因编码上述的双靶向特异性嵌合抗原受体。本发明第四方面提供了一种基因工程改造的病毒,所述病毒在宿主细胞中可以表达上述的双靶向特异性嵌合抗原受体。本发明第五方面提供了一种基因工程改造的效应细胞,表达上述的双特异性嵌合抗原受体的多肽序列,所述的效应细胞选自t淋巴细胞,nk细胞以及造血干细胞、多能干细胞、胚胎干细胞、诱导型多能干细胞或由上述干细胞诱导培养分化而成的t淋巴细胞和nk细胞;优选地选用自体的或同种异体的t淋巴细胞。优选的,所述基因工程改造的效应细胞中,上述的双特异性嵌合抗原受体表达在细胞膜上,且可以和对应的抗原发生特异性结合。本发明第六方面提供了上述嵌合抗原受体在抗肿瘤药物、抗自身免疫性疾病药物或抗病毒感染疾病药物中的应用。优选的,上述嵌合抗原受体应用在抗血液系统恶性肿瘤的药物中。相比于现有技术,本发明所述制备方法具有以下优点:本发明提供的双特异性嵌合抗原受体修饰的t淋巴细胞可以特异性识别和杀伤同时表达cd269以及cd38抗原的肿瘤细胞,具有非常强的特异性。本发明构建低亲和力嵌合抗原受体和高亲和力嵌合抗原受体,分别识别两种肿瘤相关抗原,将它转染至t淋巴细胞中,修饰后的t淋巴细胞可以同时识别带有两种肿瘤相关抗原并被有效激活,增强了car-t细胞杀伤肿瘤的靶向性以及car-t细胞在患者体内的持续性,防止患者在接受car-t治疗后的短期内再次复发。附图说明图1bcma&cd38嵌合抗原受体结构图2bcmascfv对bcma重组抗原的结合分析图3bcma单抗的筛选图4bcma重组抗体的表达与纯化图5facs分析重组抗体对k562-bcma细胞表面bcma的结合活性图6cd38单抗的筛选图7cd38scfv对cd38重组抗原的结合分析图8car重组慢病毒的构架流程图9bcma-fc重组蛋白检测t细胞中car结构的表达图10bm38car-t细胞体外杀伤活性分析图11bm38car-t回输荷瘤小鼠的生存期分析图12荷瘤小鼠的外周血及骨髓流式分析附表说明表1氨基酸密码子表表2重组bcma单抗结合rbcma的亲和力表3重组cd38单抗结合rcd38的亲和力表4不同car构建体的设计表5qpcrmix总管ⅰ内组分表6qpcrmix总管ⅱ内组分表7重组慢病毒的滴度检测表8bcma-fc重组慢病毒感染jurkar细胞的滴度表9bm38car-t体外杀伤效率分析具体实施方式下面结合附图和实施方式对本发明做进一步的详细说明。以下实施例仅是范例性的,仅用以对本发明的技术方案做更进一步的详细说明,本领域的普通技术人员应当理解,在不偏离本发明技术方案的精神和范围内,对技术方案进行的修改或者替换均应涵盖在本发明的权利要求保护范围内。实施例中未注明具体条件的实验方法,通常按照常规条件或按照生产厂商所建议的条件。本发明提供了特异性靶向bcma和靶向cd38的人源单链抗体,在以下的实施方案中,本发明的抗体源自特定的重链和轻链种系序列,和/或包含特定结构特征,如包含特定氨基酸序列的cdr区和框架区。本发明提供筛选抗体、制备抗体的方法以及通过流式细胞术对上述抗体的结合特性的验证、car结构的构建以及携带相应car分子的car-t细胞的制备及应用。在本发明的多个实施例中提供了至少一种嵌合抗原受体,其结构及布局如图1所示。嵌合抗原受体通过氨基端到羧基端顺次拼接所述的靶向cd269的特异性抗原结合片段、连接肽、靶向cd38的特异性抗原结合片段、cd8α近膜区及跨膜区、4-1bb胞内结构域和cd3ζ链胞内结构域,所得到的嵌合抗原受体car的结构为cd269(scfv)-cd38(scfv)-cd8α-cd3ζ,且研究发现双靶点的前后顺序对所得到的嵌合抗原受体car有较大影响。通过基因工程手段将嵌合抗原受体表达在t淋巴细胞膜上,则t细胞可同时特异性识别和结合靶细胞表面的cd269和cd38分子胞外结构域。其中靶向cd38的单链抗体对靶蛋白cd38分子具有略低亲和力,kd值介于5.2x10-7m和4.2x10-5m之间。实施例1.人bcma稳定表达细胞系的构建1.1质粒载体的构建本实施例使用的载体系统属于第三代自灭活慢病毒载体系统,该系统共有三个质粒即编码蛋白gag/pol、编码rev蛋白的包装质粒pspax2;编码vsv-g蛋白的包膜质粒pmd2.g及基于空载体pcdh-cmv-mcs-ef1-gfp-t2a-puro(购自addgene公司)的编码人bcma胞外区域和跨膜区域的重组质粒pcdh-cmv-hucd19-ef1-gfp-t2a-puro。根据genbank登录号nm_001192.2提供的人bcma序列,使用基于pcr搭桥的基因合成方法,合成包含信号肽、人bcma胞外区域、跨膜区域、胞内区域,通过引物对seqidno:187(hubcma-f):5>atgttgcagatggctgggcag<3seqidno:188(hubcma-f):5>tacctagcagaaattgatttc<3进行pcr扩增,扩增条件为预变性:94℃,4min;变性:94℃,30s;退火:58℃,30s;延伸:68℃,80s;30个循环。获得的片段理论大小为1716bp,扩增产物经琼脂糖电泳确认与理论大小一致。其中在开放阅读框的上下游引入xhoi和bamhi酶切位点。上述获得的目的基因由xhoi和bamhi双酶切,连入同样双酶切的pcdh-cmv-mcs-ef1-gfp-t2a-puro载体中,构建成功的慢病毒载体pcdh-cmv-hubcma-ef1-gfp-t2a-puro,经xhoi和bamhi酶切鉴定及序列测定正确后进行慢病毒包装。1.2质粒转染293t细胞包装慢病毒以6×106的密度接种培养至第6~10代的293t细胞(atcc:crl-11268)于10cm培养皿中,37℃,5%co2培养过夜准备用于转染。培养基为含10%无噬菌体胎牛血清(杭州四季青)的dmem(thermofisher公司),次日,在转染前约2小时更换培养液为无血清dmem。转染的步骤如下:1)将5μg目的基因质粒pcdh-cmv-hubcma-ef1-gfp-t2a-puro,分别与7.5μg包装质粒pspax2和2.5μg包膜质粒pmd2.g,溶入500μlmillq水中,混匀;2)逐滴加入62μl2.5mcacl2(sigma公司),以1200rpm/minvortex混匀;3)最后逐滴加入500μl2×hbs(280mmnacl,10mmkcl,1.5mmna2hpo4,12mm葡萄糖,50mmhepes(sigma公司),ph7.05,0.22μm过滤除菌),1200rpm/min振荡混匀10s,4)立即逐滴加入培养皿中,轻轻摇匀,37℃,5%co2,培养4~6h后,更换为含10%胎牛血清的dmem。在转染48h或72h后,离心除去细胞碎片,然后用0.45μm滤器(millipore公司)过滤收集病毒。1.3重组慢病毒感染白血病细胞hela或k562将上述收集的病毒液经浓缩滴定后,分别感染铺于6孔板中的hela细胞或k562细胞。感染三天后收集细胞,取部分细胞使用流式细胞术检测细胞表面bcma的表达。其余的细胞扩大培养后冻存一部分,另一部分传代于6孔板中,加入2ug/ml的puromycin(购买自sigma)抗生素,筛选1-2周的时间,显微镜下观察,至视野中全部细胞均表达gfp或用流式检测bcma的表达,与阴性细胞比较稳定转染的细胞应呈现gfp和bcma表达阳性。期间冻存部分细胞用于后续的流式细胞术检测及杀伤实验,后续培养期间将抗生素的用量减半维持相应基因的过表达。实施例2结合人bcma的特异性单链抗体(scfv)的筛选和验证2.1基于噬菌体展示的抗人bcma特异性单链抗体的筛选利用单链抗体噬菌体展示技术,从定向改造的人源单链抗体噬菌体文库中筛选特异性结合人bcma的单链抗体序列。为达到此目的,在400ml2×yt/氨苄青霉素培养基中接种噬菌体展示全人源单链抗体天然库的甘油菌(自建文库),使细胞密度达到od600=0.1,在37℃和200rpm条件下振荡培养直至细胞密度达到od600=0.5。用1012pfu的m13ko7辅助噬菌体(购买自thermofisher公司)感染,在30℃和50rpm条件下培养30分钟。加入50mg/l卡那霉素后在37℃和200rpm条件下振荡培养30分钟后,通过离心(15分钟,1600×g,4℃)分离沉淀,重悬于400ml2×yt/氨苄青霉素/卡那霉素培养基,在37℃和200rpm条件下振荡培养16小时。最后通过离心(20分钟,5000×g,4℃)分离沉淀并丢弃,上清用0.45μm规格滤膜过滤后,加入1/4体积20%(w/v)peg8000、2.5mnacl溶液并在冰浴中孵育1小时沉淀噬菌体颗粒。随后离心(20分钟,8000×g,4℃),弃上清,将噬菌体沉淀重悬于25ml预冷pbs溶液(137mmnacl,2.7mmkcl,8mmna2hpo4,2mmkh2po4)中,离心(5分钟,20000×g,4℃)。向上清液加入1/4体积20%(w/v)peg8000、2.5mnacl溶液,并冰浴30分钟再次沉淀噬菌体颗粒。离心沉淀(30分钟,20000×g,4℃),再次将噬菌体沉淀重悬于2ml预冷pbs中,在冰上保持30分钟并离心(30分钟,17000×g,4℃)。上清液与含4%(w/v)bsa的pbs溶液以1:1混合,置于旋转混合器上,室温下保温30分钟,然后直接用于筛选。利用上述噬菌体抗体库,针对生物素标记的人fc-bcma重组蛋白(购自acrobiosystems公司)实施了3轮定向筛选,筛选方案如下:将噬菌体抗体库与生物素标记的重组人bcma抗原,在室温下孵育2小时,然后与经2%bsa溶液封闭过的链霉亲和素标记的磁珠(购自thermofisher公司)在室温下孵育30分钟。随后用pbst(含0.1%吐温-20)缓冲液洗涤磁珠,除去非特异性结合或结合能力较弱的噬菌体。结合能力强的噬菌体,则用甘氨酸-盐酸缓冲液(ph2.2)从磁珠上洗脱下来,用tris中和液(ph9.1)中和后,用于感染处于对数生长期的大肠杆菌er2738,并被用于下一轮筛选。在3轮筛选中,磁珠的用量分别为50μl、20μl和10μl,生物素标记的人fc-bcma抗原浓度分别为100nm、10nm和1nm,pbst的洗涤次数分别为10次、15次和20次。1.2人bcma特异性结合单链抗体的鉴定从第三轮筛选所得的克隆中随机挑选单克隆,并用单噬菌体elisa(酶联免疫吸附实验)分析其与人fc-bcma的结合能力。为此目的,每个单菌落接种300μl2×yt/氨苄青霉素培养基(含2%葡萄糖)于96孔深孔培养板,并在37℃和250rpm下振荡培养16小时。用20μl培养物接种到500μl2×yt/氨苄青霉素培养基(含0.1%葡萄糖),在37℃和250rpm下振荡培养1.5小时。准备辅助噬菌体溶液,取75μl的m13ko7(滴度为3×1012pfu/ml)混入到15ml2×yt培养基中,50μl/孔加到培养板中。在37℃和150rpm条件培养30分钟,然后加入准备好的卡那霉素溶液50μl/孔(取180μl的50mg/ml卡那霉素,加入到15ml2×yt培养基),在37℃和250rpm下振荡培养16小时。最后离心沉淀细胞(30分钟,5000×g,4℃),将上清转移到新的96孔深孔培养板。为进行单噬菌体elisa,在96孔medisorpelisa板(购自thermofisher)上分别使用100ng/孔人fc-bcma重组抗原以及阴性对照蛋白fc(100μl/孔),在4℃包被过夜。每个孔用含2%bsa的pbst溶液封闭。随后用pbst清洗孔三次并拍净。然后加入100μl/孔上面制备的每种噬菌体溶液到板上各孔中。37℃保温2小时后,用pbst洗涤三次。为了检测结合的噬菌体,将抗m13抗体过氧化物歧化酶偶联物(购自武汉三鹰)以1:5000稀释于pbst中,取100μl加到每个孔中。37℃保温1小时后用pbst漂洗三次,然后用pbs漂洗三次。最后吸取50μltmb底物加入到孔中,并在室温下显色10分钟,随后加入每孔50μl的2mh2so4终止显色反应。用酶联免疫检测仪(bio-rad)在450nm测量消光值。根据消光值判定为阳性的孔,提取噬菌体质粒进行scfv序列的测序验证,分析获得单链抗体的氨基酸序列如序列表所示,本发明中所述单链抗体在elisa实验中对人fc-bcma结合信号极强,对fc重组蛋白无结合或极弱的结合,所述单链抗体对不同浓度的bcma抗原的结合曲线如图2所示。为了初步确定筛选得到的表达scfv的噬菌体是否可以结合bcma的天然抗原,用离心浓缩的噬菌体对bcma-k562细胞进行染色和流式分析。通过流式细胞仪(cytoflex,beckman)分析上述浓缩的噬菌体各自与细胞表面的人bcma抗原的结合能力。具体方法如下:1)取对数生长期的细胞raji细胞、k562-bcma和k562分别接种到t25细胞培养瓶中,接种细胞密度约为5x105个/ml,37℃孵箱过夜培养。2)轻微晃动培养皿内的培养液,200g×5min离心收集细胞。以2~3×106/ml的浓度重悬于1%含小牛血清的磷酸盐缓冲液(nbspbs)中,按100ul/管的量加入流式专用管中。3)200g×5min离心,弃上清。4)两个实验组分别加入待测噬菌体及阳性抗体,另设一个对照组为不加抗体的pbs空白对照。各噬菌体的终浓度均为20μg/ml,每管加入100ul。冰浴,45分钟。5)每管加入2ml1%nbspbs,以200g×5min离心,共二遍。6)弃上清,加入1:100稀释的羊抗人抗体-fitc(武汉三鹰生物),每管加入100ul。冰浴,45分钟。7)每管加入2ml1%nbspbs,以200g×5min离心,共二遍。8)弃上清,重悬于300μl1%nbspbs中,流式细胞仪检测。9)应用流式细胞仪数据分析软件cytoexpert2.0分析数据。流式细胞分析结果表明,和同型对照igg相比,靶向bcma单链抗体染色的bcma-k562细胞呈现的荧光峰值具有显著差异,在人bcma表达阴性的k562细胞上无明显差别,表明上述筛选获得的结合bcma的噬菌体可以特异识别人bcma胞外区,典型的流式分析的结果如图3所示。实施例3、抗bcma的抗体的制备和活性分析3.1对选定的单链抗体进行轻链和重链真核表达载体的构建,转染hek293f诱导重组表达并纯化。将实施例1中所获得的单链抗体序列中的轻链和重链分别构建至单克隆抗体表达质粒pcmv-v5-fc中,测序确认序列无误后大量制备该质粒。将上述轻链和重链的表达质粒以适当比例混合后转染生长良好的hek-293f细胞,37℃,5%co2,125rpm摇床连续培养7天,4000rpm离心10min,去除沉淀,收集上清,并用0.45μm滤膜过滤,将处理好的样品以proteina(购自ge公司)亲和柱进行亲和纯化,最终获得纯化的带有鼠iggfc区的bcma重组抗体,聚丙烯酰氨凝胶电泳鉴定结果如图4所示。图4为bcma重组抗体的表达检测图(其中,泳道1:bm-4g12-pcdna3.4原液;泳道2:bm-4g12-pcdna3.4穿透;泳道3:bm-4g12-pcdna3.4洗脱峰;泳道4:bm-9b5-pcdna3.4原液;泳道5:bm-9b5-pcdna3.4穿透;泳道6:bm-9b5-pcdna3.4洗脱峰;泳道7:bm-6e7-pcdna3.4原液;泳道8:bm-6e7-pcdna3.4穿透;泳道9:bm-6e7-pcdna3.4洗脱峰。)3.2elisa分析重组抗体对人bcma抗原的结合活性通过浓度梯度elisa实验,测定筛选到的抗体对抗原人bcma的结合活性。为此目的,用0.1mnahco3(ph9.6)包被液稀释抗原人bcma-his,每孔包被100ng,50μl/孔,4℃包被过夜,并用含2%bsa和0.01%(v/v)的tween-20的pbst封闭液于室温下封闭2小时。然后用pbst漂洗平板三次并去除干净。随后,向每个孔板加入100μl含一系列浓度(起始浓度10nm,进行3倍梯度稀释,直至稀释到1:729)的各抗体蛋白的pbst溶液,每个样品的测定使用平行三孔分析。37℃保温2小时后,用pbst漂洗三次,随后加入1:20000稀释的带辣根过氧化物酶标记的羊抗人抗体(购自武汉三鹰)100μl/孔,37℃反应1小时。为了检测,用pbst漂洗孔三次,然后用pbs漂洗三次,最后加入tmb显示15分钟,用每孔50μl的2mh2so4终止显色反应,用酶联免疫检测仪(bio-rad)在450nm测量消光值。用graphpad软件评估所得到的吸光强度值,计算抗体的结合强度。为此目的,每种情况下测量的消光值对相应的抗体浓度作图,并用下面的非线性回归对所得曲线进行拟合,依据公式a0/(a0-a)=1+kd/a(其中a0为无抗原存在时抗体的od值,a为加入了质量摩尔浓度为a的抗原后的od值,kd为亲和力常数的倒数)计算表观亲和力,表2所示,实施例1中所筛选得到的抗人bcma单链抗体经重组表达成完整抗体之后对bcma-his抗原的结合活性介于54nm至0.91nm之间。实施例4.人bcma定表达细胞系的构建4.1质粒载体的构建本实施例使用的载体系统属于第三代自灭活慢病毒载体系统,该系统共有三个质粒即编码蛋白gag/pol、编码rev蛋白的包装质粒pspax2;编码vsv-g蛋白的包膜质粒pmd2.g及基于空载体pcdh-cmv-mcs-ef1-gfp-puro(购自addgene)的编码人bcma胞外区域和跨膜区域的重组质粒pcdh-cmv-hubcma-ef1-gfp-puro。根据genbank登录号nm_001192.2提供的人bcma序列,使用基于pcr搭桥的基因合成方法,合成包含信号肽、人bcma胞外区域、跨膜区域、胞内区域,通过引物hubcma-f(seqidno189):5’-tgtgatcatgttgcagat-3’hubcma-r(seqidno190):5’-tacctagcagaaattgat-3’进行pcr扩增,扩增条件为预变性:94℃,4min;变性:94℃,30s;退火:58℃,30s;延伸:68℃,80s;30个循环。获得的片段理论大小为1716bp,扩增产物经琼脂糖电泳确认与理论大小一致。其中在开放阅读框的上下游引入xhoi和bamhi酶切位点。上述获得的目的基因由xhoi和bamhi双酶切,连入同样双酶切的pcdh-cmv-mcs-ef1-gfp-puro载体中,构建成功的慢病毒载体pcdh-cmv-hubcma-ef1-gfp-puro,经xhoi和bamhi酶切鉴定及序列测定正确后进行慢病毒包装。4.2质粒转染293t细胞包装慢病毒以6×106的密度接种培养至第6~10代的293t细胞(atcc:crl-11268)于10cm培养皿中,37℃,5%co2培养过夜准备用于转染。培养基为含10%无噬菌体胎牛血清(杭州四季青)的dmem(thermofisher公司),次日,在转染前约2小时更换培养液为无血清dmem。转染的步骤如下:1)将5μg目的基因质粒pcdh-cmv-hubcma-ef1-gfp-t2a-puro,分别与7.5μg包装质粒pspax2和2.5μg包膜质粒pmd2.g,溶入500μlmillq水中,混匀;2)逐滴加入62μl2.5mcacl2(sigma公司),以1200rpm/minvortex混匀,3)最后逐滴加入500μl2×hbs(280mmnacl,10mmkcl,1.5mmna2hpo4,12mm葡萄糖,50mmhepes(sigma公司),ph7.05,0.22μm过滤除菌),1200rpm/min振荡混匀10s,4)立即逐滴加入培养皿中,轻轻摇匀,37℃,5%co2,培养4~6h后,更换为含10%胎牛血清的dmem。5)在转染48h或72h后,离心除去细胞碎片,然后用0.45μm滤器(millipore公司)过滤收集病毒。4.3重组慢病毒感染白血病细胞hela或k562将上述收集的病毒液经浓缩滴定后,分别感染铺于6孔板中的hela细胞或k562细胞。感染三天后收集细胞,取部分混合克隆,使用流式细胞术及靶向bcma的荧光标记抗体检测细胞表面bcma的表达情况,流式细胞术的方法与步骤同实施例5。其余的细胞扩大培养后冻存一部分,另一部分传代于6孔板中,加入2ug/ml的puromycin(购买自sigma)抗生素,筛选1-2周的时间,显微镜下观察,至视野中全部细胞均表达gfp为止,此时冻存部分细胞用于后续的流式细胞术检测及杀伤实验。例如,使用实施例2中表达纯化的重组抗bcma的单克隆抗体以及相应的荧光标记二抗,采用流式细胞分析的方法检测上诉构建的稳定表达bcma的k562和hela细胞系,结果图下图5所示,重组表达的抗体可以显著的区分过表达bcma的k562或hela细胞。实施例5靶向cd38结合的单链抗体的筛选及分析为了获得靶向结合cd38的单克隆抗体序列,本文采用与实施例2中相同的方法构建了cd38的人源噬菌体文库,通过筛选获得了10株对人的cd38具有特异性结合能力的单链抗体,通过流式检测确认结合k562细胞膜表面的cd38分子,如图6所示。通过重组表达的cd38分子胞外区抗原以及elisa(方法同实施例3中的elisa方法)测定,本实施例中所筛选得到的靶向结合cd38的重组抗体对cd38的结合曲线如图7所示,其中靶向cd38的抗体的表观亲和力位于4.2x10-5m至5.2x10-7m之间,亲和力数据见表3。实施例6靶向bcma、cd38的嵌合抗原受体构建靶向bcma和cd38的双特异性嵌合抗原受体的序列如表4所示,通过组合其中一条靶向bcma的单链抗体及一条靶向cd38的单链抗体,两条单链抗体之间以连接肽连接,第二条单链抗体后连接跨膜区及共刺激信号分子。本实施例以一株高亲和力的bcma单克隆抗体分别与三株不同亲和力的cd38的单克隆抗体组合成形成双抗原识别区为例,对双靶向嵌合抗原受体的构建、重组慢病毒的包装、car-t细胞转导制备以及肿瘤细胞杀伤能力测试方法进行说明。在每种组合中,两段scfv分别以不同的先后顺序和刚性串联的方式连接,共形成了6种双抗原识别区(bm38-07,bm38-06,bm38-05,38bm-05,38bm-06,38bm-07),描述了不同组合方式所获得特别的肿瘤杀伤能力及避免靶向非肿瘤细胞的杀伤行为。本实施例所用的实验材料及仪器设备如下:慢病毒骨架质粒plvx-ef1由本公司自行构建并经全长测序验证无误后保存,慢病毒包装质粒prsv.rev(rev表达质粒),pmdlg/p.rre(gag/pol表达质粒),pvsv-g(vsv糖蛋白表达质粒)购买自addgene,hek293t细胞,jurkat细胞购买自中国典型培养物保藏中心,lentix-293t细胞购买自clontech,bcma-k562细胞由本公司自行构建;人新鲜外周血由健康志愿者提供;0.22μm-0.8μmpes滤器购自pall公司;200目细胞筛网、10cm,15cm细胞培养皿、1l培养袋、24孔、6孔培养板、10层细胞培养工厂购自corning公司;d-pbs、0.4%台盼蓝购自thermo公司,聚乙烯亚胺(polyethylenimine,pei)购自polysciences公司;opti-mem、pen-srep、hepes、fbs、aim-v、rpmi1640、dmem、trypsin、lipofectamine3000购自thermo公司;biotinylatedproteinl购自genescrip公司;ldh检测试剂盒购自promega公司;ficoll淋巴细胞分离液购自ge公司;20%人血白蛋白注射液购自cslbehring公司;ril-2,ril-7,,ril-15,ril-21购自cytocares公司;cd3单克隆抗体,cd28单克隆抗体,cd3/cd28磁珠以及cd4/cd8磁珠购自miltenyi公司;cd4-fitc、cd8-apc购自biolegend公司,phycoerythrin(pe)-conjugatedstreptavidin购自bdbioscience公司;proteinlmagneticbeads购自biovision公司;car构建体bm38-07,bm38-06,bm38-05,38bm-05,38bm-06,38bm-07的编码dna序列及氨基酸组合设计参见表4,序列由武汉金开瑞生物工程有限公司合成,合成的寡核苷酸序列由限制性内切酶酶切或同源重组的方式装入载体plvx-ef-1中,所得重组质粒经测序确认无误后保存。实施例7双靶向嵌合抗原受体重组慢病毒制备溶液配置:dmem完全培养基:取出2-8℃环境中保存的dmem预制培养基,加入10%(v/v)的无噬菌体胎牛血清,上下颠倒混匀后置2-8℃保存备用;1xpbs溶液:0.5mcacl2溶液:1g/l的pei溶液:称取1g的pei粉末,用900mlmilli-qgrade超纯水溶解水溶解,水浴加热至60-80℃并不断搅拌,校准ph计后加hcl调ph至2.0左右,盖上烧杯连续搅拌3h直至粉末完全溶解,冷却至室温后再加naoh调节ph至6.9-7.1,转移至容量瓶中并加水定容至1l,0.22μmpes针头滤器过滤后分装,-20℃长期保存,短期内使用即可存放在2-8℃冰柜中;2xhbs溶液:0.25%(m/v)trypsin溶液:称量trypsin2.50g,edta0.20g置于1000ml烧杯中,加入900ml1xpbs溶解,溶解完成后,使用1000ml量筒定容至1000ml,0.22μm过滤pes一次性针头式过滤器过滤除菌,分装保存到50ml离心管中,长期使储存可保存至-20℃冰箱,短期保存可放置在2-8℃冰箱;1mnaoh溶液1.5mnacl溶液1mnacl溶液0.15mnacl溶液:。1mtris-hcl(ph6-8)溶液250mmtris-hcl(ph6-8)溶液:25mmtris-hcl(ph6-8)溶液:本发明所述构建重组慢病毒的流程如如图8所示。1.重组慢病毒骨架质粒的构建如上图所示,将全合成的基于bcma、cd38双靶点的嵌合抗原受体(bm38-05,bm38-06,bm38-07,38bm-05,38bm-06,38bm-07)序列通过双酶切后再连接的方法构建至慢病毒骨架质粒plvx-ef1的启动子ef1下游的多克隆位点mcs中,分别得到重组慢病毒骨架质粒plvx-ef1-bm38-05,plvx-ef1-bm38-06,plvx-ef1-bm38-07,plvx-ef1-38bm-05,plvx-ef1-38bm-06,plvx-ef1-38bm-07,所用的酶切位点为5’-bamhⅰ,3’-xhoⅰ。构建完成的重组骨架质粒经测序验证无误后大量制备用于重组慢病毒的包装。2.重组慢病毒骨架质粒、包装质粒的大量制备针对上述构建的慢病毒骨架质粒及其它包装质粒,建立标准菌种库之后活化菌种并大量培养,收集菌体,采用天根生物质粒大量提取试剂盒(endofreemaxiplasmidkit)及质粒提取试剂盒指南方法进行上述质粒的大量提取制备,用于后续的细胞转染级重组病毒包装。3.lentix-293t细胞培养1)从液氮罐中取出冻存的lentix-293t细胞,迅速转移到37℃水浴中,1~2min后转移到生物安全柜中;预先在15ml离心管中加入9ml预冷的含10%fbs的完全培养基,用1ml移液器将冻存管中的细胞液缓慢转移至15ml离心管中,1000g离心10分钟后弃上清,沉淀用3ml完全培养基重悬后转移至10cm培养皿中,补足含10%fbs的完全培养基至8ml/10cm培养皿,24h后显微镜观察细胞,细胞汇合度达到70%左右时再次进行传代;2)选择细胞状态良好、无污染的lentix-293t细胞,每5个培养皿为一组,将细胞胰酶消化后,用电动移液器吸取8ml完全培养基,向每个消化后的培养皿中加2ml避免培养皿变干,使用10ml移液器将所有细胞吹打成单细胞悬液,转移到t75培养基瓶中;3)将上述5个培养皿中的剩余细胞转移到培养基瓶中,并用培养基再润洗一遍培养皿后一并转入培养瓶中;4)盖紧培养基瓶盖,上下颠倒或用电动移液器充分混匀细胞悬液,取适量细胞悬液稀释后进行计数,根据计数结果将细胞悬液均匀分配到20个10cm培养皿中,确保每个培养皿的细胞密度约为4×106个/10ml,前后及左右呈十字形晃动数次,使细胞充分铺开,然后放入饱和湿度及5%(v/v)co2培养箱中培养;5)检查所传代细胞,当细胞汇合度达到70-80%,细胞贴壁良好且轮廓饱满,在细胞培养皿底部均匀分布时为细胞换液,将培养基替换为8ml预热的新鲜完全培养基。4.lentix-293t细胞转染6)按照1:1(v/v)的比例配制dna/pei溶液,以下所述操作均应在生物安全柜中严格按照无菌操作的标准来进行操作。每皿lentix-293t细胞转染质粒量按照下列比例使用:重组慢病毒骨架质粒(20μg),prsv-rev(15μg),pmdl-rre(10μg),pvsv-g(7.5μg)。取一个新的5ml离心管,加入上述用量的包装质粒后补充dmem至1.0ml后盖上盖子,充分混匀;7)按照pei转染试剂盒上操作进行转染8)72小时后,再次将同一培养皿中的病毒上清液收集到一起,此时收集的上清里包含了重组慢病毒lv-bm38-07,lv-bm38-06,lv-bm38-05,lv-38bm-05,lv-38bm-06,lv-38bm-07。5.离子交换色谱法纯化重组慢病毒1)将收集的上清液使用真空泵,经0.22μm-0.8μm的pes滤器抽滤,除去杂质及细胞碎片;2)按1:1-1:10的比例往上清中加入1.5mnacl250mmtris-hcl(ph6-8);3)将2个离子交换柱串联放置,用4ml1mnaoh、4ml1mnacl、5ml0.15mnacl25mmtris-hcl(ph6-8)溶液依次过柱;4)将步骤2中获得的溶液通过蠕动泵以1-10ml/min的速度给离子交换柱上样;5)全部上清液过柱后,使用10ml0.15mnacl25mmtris-hcl(ph6-8)溶液清洗一遍;6)根据上样量使用1-5ml1.5mnacl25mmtris-hcl(ph6-8)进行洗脱,收集洗脱液;7)将洗脱液分成50μl一管,冻存到-80℃冰箱,进行长期保存。6.重组慢病毒滴度测定1)取24孔板接种293t细胞。每孔细胞为5×104个,所加培养基体积为500ul,不同种类的细胞生长速度有所差异,进行病毒感染时的细胞融合率为40%-60%;2)准备3个无菌ep管,在每个管中加入90ul的新鲜完全培养基(高糖dmem+10%fbs)接种细胞24小时后,取两个孔的细胞用血球计数板计数,确定感染时细胞的实际数目,记为n;3)取待测定的病毒原液10ul加入到第一个管中,轻轻混匀后,取10ul加入到第二个管中,然后依次操作直到最后一管;在每管中加入410ul完全培养基(高糖dmem+10%fbs),终体积为500ul;4)感染开始后20小时,除去培养上清,更换为500μl完全培养基(高糖dmem+10%fbs),5%co2继续培养48小时;5)72小时后,观察荧光表达情况,正常情况下,荧光细胞数随稀释倍数增加而相应减少,并拍照;6)用0.2ml0.25%胰酶-edta溶液消化细胞,在37℃放置1分钟。用培养基吹洗整个细胞面,离心收集细胞。按照dneasy试剂盒的说明抽提基因组dna。每个样品管中加入200μl洗脱液洗下dna并定量;7)准备目的dna检测qpcrmix总管ⅰ(qpcr引物序列为seqidno.191——seqidno.196):ef1α-f:5-tatcgatgctccggtgcccgtcagt-3(seqidno.191)ef1α-r:5-tcacgacacctgaaatggaaga-3(seqidno.192)wpre-qpcr-f:5-tccgggactttcgcttt-3(seqidno.193)wpre-qpcr-r:5-cagaatccaggtggcaaca-3(seqidno.194)actin-qpcr-f:5-catgtacgttgctatccaggc-3(seqidno.195)actin-qpcr-r:5-tccttaatgtcacgcacgat-3(seqidno.196)qpcrmix总管ⅰ内组分如表5所示。表5中n=numberofreactions.例如:总反应数为40,将1ml2×taqmanuniversalpcrmastermix,4μlforwardprimer,4μlreverseprimer,4μlprobe和788μlh2o混和,震荡后放在冰上。准备内参dna检测qpcrmix管ⅱ(qpcr引物序列为seqidno.194,seqidno.195):actin-qpcr-f:5-catgtacgttgctatccaggc-3(seqidno.194)actin-qpcr-r:5-tccttaatgtcacgcacgat-3(seqidno.195)qpcrmix总管ⅱ内组分如表6所示。表6中n=numberofreactions.例如:总反应数为40,将1ml2×taqmanuniversalpcrmastermix,100μl10×rnasepprimer/probemix和700μlh2o混和,震荡后放在冰上。在预冷的96孔pcr板上完成pcr体系建立。从总管ⅰ中各取45μl加入到a-d各行的孔中,从总管ⅱ中各取45μl加入到e-g各行的孔中。分别取5μl质粒标准品和待测样品基因组dna加入到a-d行中,每个样品重复1次。另留1个孔加入5μl的水做为无模板对照(no-templatecontrol)。分别取5μl基因组标准品和待测样品基因组dna加入到e-g行中,每个样品重复1次。另留1个孔加入5μl的水做为无模板对照(no-templatecontrol)。所使用定量pcr仪为rochelc96定量系统。循环条件设定为:94℃3分钟,然后是94℃15秒,60℃1分钟的40个循环最后72℃3分钟结束程序。数据分析:测得的dna样品中整合的慢病毒载体拷贝数用基因组数加以标定,得到每基因组整合的病毒拷贝数。滴度(integrationunitsperml,iuml-1)的计算公式如下:iu/ml=(c×n×d×1000)/v其中:c=平均每基因组整合的病毒拷贝数n=感染时细胞的数目(约为1×105)d=病毒载体的稀释倍数v=加入的稀释病毒的体积数包含car基因的重组慢病毒lv-bm38-07,lv-bm38-06,lv-bm38-05,lv-38bm-05,lv-38bm-06,lv-38bm-07的滴度测定结果如表7所示。7.重组慢病毒jurkat细胞感染滴度测定使用离子交换色谱纯化得到的重组慢病毒溶液,通过转导jurkat细胞及car表达或标记基因的表达来评估测定病毒滴度。jurkat细胞在第1天用病毒上清液的3倍梯度稀释系列转导,起始浓度为1:300。在第5天用bcma-fc抗原(acrobiosystem)评价car表达。根据下式计算病毒滴度:(%car+)×(#jurkat细胞)/(病毒量(ml))×(稀释度)从1至20%car阳性的线性范围内的稀释点计算平均病毒滴度。bcma-fc重组慢病毒感染jurkar细胞的滴度如表8所示。实施例8t细胞的分离培养及慢病毒转导t细胞抽取50ml健康志愿者的新鲜外周血,通过常规方法获得人外周血单个核细胞(pbmc)。用适量的macs缓冲液(按1.5ml/107个pbmc)洗涤细胞一次,800r/min离心10min沉淀细胞,弃上清。用适量的macs缓冲液重悬细胞(按80μl/107个pbmc)后,加入适量的anti-human-cd3免疫磁珠(按20μl/107个pbmc)混匀,4℃孵育15min。再加入适量的macs缓冲液洗涤细胞(按1.5ml/107个pbmc),800r/min离心10min沉淀细胞,弃上清,用500μlmacs缓冲液重悬细胞。将ms分离柱放入minimacs分离器上,用500μlmacs缓冲液洗涤分离柱一次。加入细胞悬液于ms分离柱内,先流出来的细胞是未被磁珠标记的cd3-细胞。用500μlmacs缓冲液洗涤分离柱三次,从minimacs分离器上取下ms分离柱,放到15ml离心管上。加入1mlmacs缓冲液至分离柱上,迅速将滞留的细胞洗脱下来,洗脱液即为分离的cd3+t淋巴细胞。加入适量的macs缓冲液,充分混匀后计数。1000r/min离心10min,弃上清。用含10%fbs的rpmi1640培养基重悬,调整细胞浓度至1×106/ml于6孔板中。将培养板置于37℃、5%co2培养箱中培养。按照人t淋巴细胞cd3/cd28免疫激活磁珠说明书激活t淋巴细胞。将分离出的cd3+t淋巴细胞以每孔1×106个铺于24孔板上。每孔加入25μl已预洗的磁珠,加入重组人il-2(购自上海华新生物高技术有限公司)使终浓度为30u/ml。将24孔板置于37℃、5%co2培养箱中培养。每2-3d更换含有重组人il-2的培养基。根据细胞生长密度进行传代。当cd3+t淋巴细胞生长状态良好,密度为2x106个/ml左右时可进行重组慢病毒感染,感染的moi依据重组慢病毒感染jurkat细胞的活性滴度计算,一般不超过5。将polybrene加入24孔板中至终浓度为4μg/ml,同时加入慢病毒悬液lv-bm38-07,lv-bm38-06,lv-bm38-05,lv-38bm-05,lv-38bm-06,lv-38bm-07,6h后补充新鲜培养液或全换液并继续培养。实施例9bm38-05,bm38-06,bm38-07car-t细胞的检测感染的t细胞在培养第6天时通过流式细胞术检测嵌合抗原受体表达。首先,感染后的car-t细胞与生物素标记的人fc-bcma蛋白37℃孵育30min,d-pbs洗2次,然后加入pe-标记的streptavidin在37℃孵育30min。d-pbs洗3次后流式细胞仪检测阳性细胞比率。以未感染的t淋巴细胞和只加了pe-标记的streptavidin染色的t细胞作为阴性对照,鉴定表达嵌合抗原受体的病毒感染t细胞及其阳性率水平,结果如图9所示。另外,也可以通过在构建car表达载体的时候同时表达gfp等标记蛋白或标签,通过荧光成像或facs分析确认表达标记蛋白如gfp的t细胞的阳性率,对照fc-bcma重组抗原检测的t细胞表达car基因的阳性水平,可以确认携带car基因的慢病毒感染的t细胞可以成功的将car结构展示到t细胞的细胞膜上,同时两种检测方法获得的阳性率结果无显著差异,说明通过慢病毒转导的方法能够获得稳定表达嵌合抗原受体受体的t细胞。实施例10、表达嵌合抗原受体的t淋巴细胞的体外毒性效果实验在体外适宜的细胞培养体系中,混合带有特异性肿瘤抗原的靶细胞和t淋巴细胞或car-t细胞,一段时间后即可观察到t细胞或car-t细胞对靶细胞实施了识别(聚集)和杀伤(肿瘤细胞数量减少)行为。例如本实验混合过表达bcma的k562细胞和普通扩增培养的t细胞或bm38-05,bm38-06,bm38-07car-t。细胞本实验所用的细胞为k562(k562细胞本身不表达bcma蛋白)和转染了bcma的k562细胞(k562-bcma)作为靶细胞,效应细胞为前述制备的car-t细胞,效靶比视情况分别为2.5,12.5:1和25:1,靶细胞数量为10000个/孔,根据不同效靶比添加适当数量的效应细胞。所有细胞均培养于5%co2,37℃的细胞培养箱内。实验开始时,先接种10000个/孔的k562-bcma细胞至6孔板中,培养12h后根据效靶比添加适当数量的t细胞或car-t细胞,继续在5%co2,37℃培养箱中培养4h后,取出培养板在倒置显微镜下观察成像并计数计算杀伤效率,结果如表9所示,bm38-05,bm38-06,bm38-07对bcma阳性的k562细胞具有显著的杀伤效应,而38bm-05、38bm-06、38bm-07则具有显著一致的极弱的杀伤效应。实施例11、表达嵌合抗原受体的t淋巴细胞的体外毒性效果实验car-t细胞杀伤肿瘤细胞时,t细胞首先会和携带肿瘤抗原的肿瘤细胞进行识别查证,然后形成免疫突触,进而释放杀伤因子完成t细胞的免疫监视及杀伤功能,因此这个过程是一个持续的过程。传统的检测t细胞杀伤肿瘤细胞的方法往往是取一个时间点进行终点检测,其获得的结果往往误差较大,也不能真实的反映出t细胞杀伤肿瘤细胞的完整过程。为了突破这一类检测手段的技术瓶颈的限制,实时无标记细胞分析技术(realtimecellularanalysis,rtca)基于细胞在带有微电极的培养板中贴壁后产生的电阻抗值的检测,可以实现实时、无标记、持续动态的监测小分子化合物、抗体药物、t细胞等引起的细胞毒性效应。基于这种电阻抗的实时细胞杀伤分析技术,研究者能获得很高灵敏度的量化数据,有助于研究和揭示抗肿瘤化合物或细胞的具体作用机制,同时也能显著的降低实验的成本和提高结果的准确度。本实验所用的细胞为人宫颈癌细胞系hela和转染了bcma的hela细胞(bcma-hela)作为靶细胞,效应细胞为前述制备的bm38-06car-t细胞,效靶比视情况分别为2.5,12.5:1和25:1,靶细胞数量为10000个/孔,根据不同效靶比添加适当数量的效应细胞。所有细胞均培养于5%co2,37℃的细胞培养箱内。rtca技术持续动态的监测靶细胞的贴壁、伸展以及繁殖过程,使用细胞指数cellindex表示靶细胞的生长状态,或可等同于靶细胞的数量。整个实验过程除了添加效应细胞时,应保证培养板及检测仪器处于稳定的培养环境中,避免cellindex数据出现较大的波动。为了获得时间依赖的细胞效应特征曲线,在16孔的e-plate的培养孔中加入90ul的培养基,测得背景基线后在加入100ul的细胞悬液,混匀后放置于co2培养箱中的检测台上,并持续获得反应细胞生长状态的电阻抗值cellindex。18h后待靶细胞完全贴壁生长一段时间后加入等体积的效应细胞悬液,然后再放入培养箱中的检测台上,继续收集反应细胞生长状态的电阻抗值cellindex数据。48h后结束整个实验,导出采集的数据并作图,结果如图10所示。实施例12体内car-t细胞的抗肿瘤作用(动物实验生存曲线)为了确证在体外具有良好杀伤肿瘤细胞能力的car-t细胞在荷瘤小鼠模型中对肿瘤细胞的杀伤能力,本实施例通过尾静脉注射k562-bcma细胞和对照k562细胞建立小鼠模型,一周后再次通过尾静脉注射不同数量的bcmacar-t、bm38car-t以及对照t细胞,通过观察模型小鼠的生存时间及在确定的时间点处死小鼠,采集小鼠的外周血及骨髓组织样本,通过流式检测其中的car-t细胞及肿瘤细胞数量,以此来评价在荷瘤小鼠模型中不同的car-t细胞的肿瘤杀伤能力。实验过程如下:如前所述k562-bcma细胞在含有10%热灭活胎牛血清的rpmi1640培养基中生长,从北京百奥赛图基因生物科技公司购买小鼠(nod-prkdcscidil2rgtm1/bcgen),无菌条件下饲养1周后通过尾静脉注射5x10^5个/100μl的k562-bcma细胞建立荷瘤小鼠模型。在肿瘤细胞植入后7-8天给小鼠回输5x106的对照t细胞或car-t细胞。将t细胞或car-t细胞在37℃水浴中部分解冻,然后通过向含有细胞的管中加入1ml冷的无菌pbs完全解冻。将解冻的细胞转移至15mlfalcon管中,并用pbs调整至最终体积为10ml。将细胞以1000rpm洗涤两次,每次10分钟,然后在血细胞计数器上计数。cart细胞针对car转导进行归一化,使得每组具有相同百分比的car+t细胞。然后将5x106个细胞的总数以50x106个细胞/ml冷pbs的浓度重悬,并保持在冰上直到小鼠给药。通过尾静脉用100μlcart细胞静脉内注射小鼠,每只小鼠5x106个t细胞的剂量。每组5至7只小鼠用100μlpbs单独(pbs),未转导的t细胞(mock),bcmacar-t细胞或bm38car-t细胞处理,t细胞均由相同的健康人体志愿者供给。完成car-t注射后,每天检测实验动物的生存状态,记录体重变化。如若在监测到小鼠生存状态极差,如无法进食、瘫痪等症状时判定小鼠出现死亡事件,此时处死小鼠后采集外周血及骨髓样本,用流式细胞仪分析外周血及骨髓中肿瘤细胞(k562-bcma,gfp阳性)和car-t细胞(humancd45+car+)的细胞数量。记录小鼠死亡事件发生的时间,统计分析各组小鼠的生存时间曲线。结果如下图11所示,结果显示回输bm38car-t细胞的荷瘤小鼠具有更长的生存期。针对上述荷瘤小鼠的外周血及骨髓的流式分析显示,回输bm38car-t细胞的荷瘤小鼠的外周血和骨髓中具有更少的k562-bcma细胞,同时却具有更多的car-t细胞,如图12所示。尽管已描述了本发明的优选实施例,但本领域内的技术人员一旦得知了基本创造性概念,则可对这些实施例作出另外的变更和修改。所以,所附权利要求意欲解释为包括优选实施例以及落入本发明范围的所有变更和修改。显然,本领域的技术人员可以对本发明进行各种改动和变型而不脱离本发明的精神和范围。这样,倘若本发明的这些修改和变型属于本发明权利要求及其等同技术的范围之内,则本发明也意图包含这些改动和变型在内。本发明涉及到的附表如下:表1氨基酸密码子表名称英文名称三字母缩写单字母缩写核苷酸密码子甘氨酸glycineglygggu,ggc,gga,ggg丙氨酸alaninealaagcu,gcc,gca,gcg结氨酸valinevalvguu,guc,gua,gug亮氨酸leucineleulcuu,cuc,cua,cug,uua,uug异亮氨酸isoleucineileiauu,auc,aua脯氨酸prolinepropccu,cca,ccg,ccc苯丙氨酸phenylalaninephefuuu,uuc酪氨酸tyrosinetyryuau,uac色氨酸tryptophantrpwugg丝氨酸serinesersucu,uca,ucc,ucg,agu,agc苏氨酸threoninethrtacu,acc,acg,aca半胱氨酸cystinecyscugu,ugc蛋氨酸methioninemetmaug天冬氨酸asparagineasnnaau,aac谷氨酰胺glutarnineglnqcaa,cag天冬酰胺asparticacidaspdgau,gac谷氨酸glutamicacidgluegaa,gag赖氨酸lysinelyskaaa,aag精氨酸arginineargrcgu,cgc,cgg,cga,aga,agg组氨酸histidinehishcau,cac终止密码---uaa,uag,uga表2重组bcma单抗结合fc-bcma的亲和力antibodyka(1/ms)kd(1/s)kd(m)bm-3e23.5e+053.6e-056.9e-09bm-6g34.8e+063.6e-045.6e-09bm-7a95.2e+051.6e-027.3e-09bm-4g126.2e+041.5e-054.2e-09bm-6e77.3e+044.4e-049.1e-10bm-9b56.2e+065.8e-046.5e-09bm-4c113.8e+053.2e-056.9e-08bm-3g72.9e+056.1e-067.2e-09bm-6f55.4e+047.6e-055.4e-08bm-5c82.9e+052.9e-068.2e-09表3重组cd38单抗结合rcd38的亲和力antibodyka(1/ms)kd(1/s)kd(m)38-4a67.2e+064.4e-042.5e-0638-4b76.3e+065.6e-054.2e-0538-5a48.5e+058.2e-033.6e-0638-6c91.9e+069.1e-065.2e-0738-6d122.4e+066.3e-055.1e-0638-3f105.3e+045.7e-057.1e-0638-4f96.7e+065.9e-055.6e-0538-6b106.5e+056.2e-068.6e-0638-7a125.9e+046.8e-049.8e-0638-4b54.8e+064.7e-057.2e-05表4不同car构建体的设计表表5qpcrmix总管ⅰ内组分表2×taqmanmastermix25μl×nforwardprimer(100pmol/ml)0.1μl×nreverseprimer(100pmol/ml)0.1μl×nprobe(100pmol/ml)0.1μl×nh2o19.7μl×n表6qpcrmix总管ⅱ内组分表2×taqmanmastermix25μl×n10×rnasepprimer/probemix2.5μl×nh2o17.5μl×n表7重组慢病毒的滴度检测结果表lentivirustitelv-bm38-079.3e+10lv-bm38-061.2e+11lv-bm38-054.5e+11lv-38bm-078.9e+10lv-38bm-066.7e+10lv-38bm-052.4e+10表8bcma-fc重组慢病毒感染jurkar细胞的滴度表lentivirus感染率tite(tu/ml)lv-bm38-0762.5%1.25e+08lv-bm38-0674.8%1.50e+08lv-bm38-0568.4%1.37e+08lv-38bm-0756.3%1.13e+08lv-38bm-0645.8%0.92e+08lv-38bm-0567.6%1.35e+08表9bm38car-t细胞体外杀伤效率分析表序列表<110>生研医药科技(武汉)有限公司<120>一种双特异性嵌合抗原受体分子及其在肿瘤治疗上的应用<141>2018-09-30<160>196<170>siposequencelisting1.0<210>1<211>7<212>prt<213>artificialsequence<400>1glyphethrpheserglytyr15<210>2<211>6<212>prt<213>artificialsequence<400>2asnproaspglyserser15<210>3<211>7<212>prt<213>artificialsequence<400>3asptyrtyrglypheaspile15<210>4<211>11<212>prt<213>artificialsequence<400>4glnglyaspserleuargthrtyrhisalaser1510<210>5<211>7<212>prt<213>artificialsequence<400>5glylysaspasnargproser15<210>6<211>11<212>prt<213>artificialsequence<400>6tyrserargaspserserglyasnhispheval1510<210>7<211>116<212>prt<213>artificialsequence<400>7glnvalasnleuarggluserglyglyglyleuvalglnproglygly151015serleuargleusercysalaalaserglyphethrpheserglytyr202530trpmethistrpvalargglnalaproglygluglyleuvalserval354045serargileasnproaspglyserserthriletyralaaspserval505560lysglyargphethrileserargaspasnalalysasnthrleutyr65707580leuglnmetasnserleulysthrgluaspthralavaltyrtyrcys859095serargasptyrtyrglypheaspiletrpglyglnglythrmetval100105110thrvalserser115<210>8<211>107<212>prt<213>artificialsequence<400>8gluleuthrglnaspproalavalservalalavalglyglnthrval151015argleuthrcysglnglyaspserleuargthrtyrhisalasertrp202530tyrglnglnargproglyglnalaproleuleuvalphepheglylys354045aspasnargproserglyileproaspargpheserglyserserser505560glyasnthralaserleuthrilethrglyalaglnalagluaspglu65707580alaasptyrtyrcystyrserargaspserserglyasnhispheval859095pheglythrglythrlysvalthrvalleugly100105<210>9<211>238<212>prt<213>artificialsequence<400>9glnvalasnleuarggluserglyglyglyleuvalglnproglygly151015serleuargleusercysalaalaserglyphethrpheserglytyr202530trpmethistrpvalargglnalaproglygluglyleuvalserval354045serargileasnproaspglyserserthriletyralaaspserval505560lysglyargphethrileserargaspasnalalysasnthrleutyr65707580leuglnmetasnserleulysthrgluaspthralavaltyrtyrcys859095serargasptyrtyrglypheaspiletrpglyglnglythrmetval100105110thrvalserserglyglyglyglyserglyglyglyglyserglygly115120125glyglysergluleuthrglnaspproalavalservalalavalgly130135140glnthrvalargleuthrcysglnglyaspserleuargthrtyrhis145150155160alasertrptyrglnglnargproglyglnalaproleuleuvalphe165170175pheglylysaspasnargproserglyileproaspargphesergly180185190serserserglyasnthralaserleuthrilethrglyalaglnala195200205gluaspglualaasptyrtyrcystyrserargaspserserglyasn210215220hisphevalpheglythrglythrlysvalthrvalleugly225230235<210>10<211>7<212>prt<213>artificialsequence<400>10glyphethrphesersertyr15<210>11<211>6<212>prt<213>artificialsequence<400>11serglyserglyglyser15<210>12<211>15<212>prt<213>artificialsequence<400>12glypheleuargargaspglytyrasnthrasnalapheaspval151015<210>13<211>11<212>prt<213>artificialsequence<400>13argalaserglnserileserhistyrleuala1510<210>14<211>7<212>prt<213>artificialsequence<400>14aspthrserlysargalathr15<210>15<211>8<212>prt<213>artificialsequence<400>15glnglnargseriletrpargthr15<210>16<211>124<212>prt<213>artificialsequence<400>16glnvalasnleuarggluserglyglyglyleuvalglnproglygly151015serleuargleusercysalaalaserglyphethrphesersertyr202530alametsertrpvalargglnalaproglylysglyleuglutrpval354045seralaileserglyserglyglyserthrtyrtyralaaspserval505560lysglyargphethrileserargaspasnserlysasnthrleutyr65707580leuglnvalasnserleuargvalgluaspthralavaltyrtyrcys859095alaargglypheleuargargaspglytyrasnthrasnalapheasp100105110valtrpglyglnglythrmetvalthrvalserser115120<210>17<211>107<212>prt<213>artificialsequence<400>17aspvalvalmetthrglnserproalathrleuservalserprogly151015gluargalathrleusercysargalaserglnserileserhistyr202530leualatrptyrglnglnlysproglyglnalaproargleuleuile354045tyraspthrserlysargalathrglyileproalaargphesergly505560serglyserglythraspphethrleuthrileserserleuglupro65707580aspaspphealathrtyrtyrcysglnglnargseriletrpargthr859095pheglyglnglythrlysvalgluilelysarg100105<210>18<211>246<212>prt<213>artificialsequence<400>18glnvalasnleuarggluserglyglyglyleuvalglnproglygly151015serleuargleusercysalaalaserglyphethrphesersertyr202530alametsertrpvalargglnalaproglylysglyleuglutrpval354045seralaileserglyserglyglyserthrtyrtyralaaspserval505560lysglyargphethrileserargaspasnserlysasnthrleutyr65707580leuglnvalasnserleuargvalgluaspthralavaltyrtyrcys859095alaargglypheleuargargaspglytyrasnthrasnalapheasp100105110valtrpglyglnglythrmetvalthrvalserserglyglyglygly115120125serglyglyglyglyserglyglyglyglyseraspvalvalmetthr130135140glnserproalathrleuservalserproglygluargalathrleu145150155160sercysargalaserglnserileserhistyrleualatrptyrgln165170175glnlysproglyglnalaproargleuleuiletyraspthrserlys180185190argalathrglyileproalaargpheserglyserglyserglythr195200205aspphethrleuthrileserserleugluproaspaspphealathr210215220tyrtyrcysglnglnargseriletrpargthrpheglyglnglythr225230235240lysvalgluilelysarg245<210>19<211>9<212>prt<213>artificialsequence<400>19glyglyserileserserserglytyr15<210>20<211>5<212>prt<213>artificialsequence<400>20asnhisserglyser15<210>21<211>9<212>prt<213>artificialsequence<400>21seralatyrtrpthrgluargasptyr15<210>22<211>11<212>prt<213>artificialsequence<400>22argalaserglnserleuargserasnleuala1510<210>23<211>7<212>prt<213>artificialsequence<400>23aspalaserargargalathr15<210>24<211>9<212>prt<213>artificialsequence<400>24glnglnserglyglylysproserthr15<210>25<211>119<212>prt<213>artificialsequence<400>25glnvalglnleuglngluserglyalaglyleuleulysproserglu151015thrleuserleuthrcysthrvalserglyglyserileserserser202530glytyrtyrtrpsertrpileargglnproproglylysglyleuglu354045trpileglygluileasnhisserglyserthrasntyrasnproser505560leulysserargvalthrileservalaspthrserlysasnglnphe65707580serleulysleuserservalthralaalaaspthralavaltyrtyr859095cysalaargseralatyrtrpthrgluargasptyrtrpglyglngly100105110thrleuvalthrvalserser115<210>26<211>120<212>prt<213>artificialsequence<400>26ilethrsertyrserilehistyrthrlysleuserlysilevalleu151015thrglnserproglythrleuserleuserproglygluargalathr202530leutyrcysargalaserglnserleuargserasnleualatrptyr354045glnglnlysproglyglnalaproargleuleuiletyraspalaser505560argargalathrglyileproaspargpheserglyserglysergly65707580thraspphethrleuthrileserserleuglnprogluasppheala859095thrtyrtyrcysglnglnserglyglylysproserthrpheglygln100105110glythrlysvalgluilelysarg115120<210>27<211>254<212>prt<213>artificialsequence<400>27glnvalglnleuglngluserglyalaglyleuleulysproserglu151015thrleuserleuthrcysthrvalserglyglyserileserserser202530glytyrtyrtrpsertrpileargglnproproglylysglyleuglu354045trpileglygluileasnhisserglyserthrasntyrasnproser505560leulysserargvalthrileservalaspthrserlysasnglnphe65707580serleulysleuserservalthralaalaaspthralavaltyrtyr859095cysalaargseralatyrtrpthrgluargasptyrtrpglyglngly100105110thrleuvalthrvalserserglyglyglyglyserglyglyglygly115120125serglyglyglyglyserilethrsertyrserilehistyrthrlys130135140leuserlysilevalleuthrglnserproglythrleuserleuser145150155160proglygluargalathrleutyrcysargalaserglnserleuarg165170175serasnleualatrptyrglnglnlysproglyglnalaproargleu180185190leuiletyraspalaserargargalathrglyileproaspargphe195200205serglyserglyserglythraspphethrleuthrileserserleu210215220glnprogluaspphealathrtyrtyrcysglnglnserglyglylys225230235240proserthrpheglyglnglythrlysvalgluilelysarg245250<210>28<211>7<212>prt<213>artificialsequence<400>28glyphethrpheserasnphe15<210>29<211>6<212>prt<213>artificialsequence<400>29thrthrglyglyglyasp15<210>30<211>12<212>prt<213>artificialsequence<400>30hisglytyrtyraspglytyrhisleupheasptyr1510<210>31<211>11<212>prt<213>artificialsequence<400>31argalaasnglnglyileserasnasnleuasn1510<210>32<211>7<212>prt<213>artificialsequence<400>32tyrthrserasnleuglnser15<210>33<211>9<212>prt<213>artificialsequence<400>33glnglnphethrserleuprotyrthr15<210>34<211>121<212>prt<213>artificialsequence<400>34gluvalglnleuvalgluserglyglyglyleuvalglnproglygly151015serleuargleusercysalaalaserglyphethrpheserasnphe202530aspmetalatrpvalargglnalaproglylysglyleuvaltrpval354045serserilethrthrglyglyglyaspthrtyrtyralaaspserval505560lysglyargphethrileserargaspasnalalysserthrleutyr65707580leuglnmetaspserleuargsergluaspthralavaltyrtyrcys859095valarghisglytyrtyraspglytyrhisleupheasptyrtrpgly100105110glnglythrleuvalthrvalserser115120<210>35<211>107<212>prt<213>artificialsequence<400>35aspileglnmetthrglnserproserserleuseralaservalgly151015aspargvalthrilethrcysargalaasnglnglyileserasnasn202530leuasntrptyrglnglnlysproglylysalaprolysproleuile354045tyrtyrthrserasnleuglnserglyvalproserargphesergly505560serglyserglythrasptyrthrleuthrileserserleuglnpro65707580gluaspphealathrtyrtyrcysglnglnphethrserleuprotyr859095thrpheglyglnglythrlysleugluilelys100105<210>36<211>243<212>prt<213>artificialsequence<400>36gluvalglnleuvalgluserglyglyglyleuvalglnproglygly151015serleuargleusercysalaalaserglyphethrpheserasnphe202530aspmetalatrpvalargglnalaproglylysglyleuvaltrpval354045serserilethrthrglyglyglyaspthrtyrtyralaaspserval505560lysglyargphethrileserargaspasnalalysserthrleutyr65707580leuglnmetaspserleuargsergluaspthralavaltyrtyrcys859095valarghisglytyrtyraspglytyrhisleupheasptyrtrpgly100105110glnglythrleuvalthrvalserserglyglyglyglyserglygly115120125glyglyserglyglyglyglyseraspileglnmetthrglnserpro130135140serserleuseralaservalglyaspargvalthrilethrcysarg145150155160alaasnglnglyileserasnasnleuasntrptyrglnglnlyspro165170175glylysalaprolysproleuiletyrtyrthrserasnleuglnser180185190glyvalproserargpheserglyserglyserglythrasptyrthr195200205leuthrileserserleuglnprogluaspphealathrtyrtyrcys210215220glnglnphethrserleuprotyrthrpheglyglnglythrlysleu225230235240gluilelys<210>37<211>7<212>prt<213>artificialsequence<400>37glypheasnpheasnasptyr15<210>38<211>6<212>prt<213>artificialsequence<400>38trphisaspglysergln15<210>39<211>10<212>prt<213>artificialsequence<400>39proalaleuleugluvalilepheaspasp1510<210>40<211>13<212>prt<213>artificialsequence<400>40argserserglnserleuleuaspseraspaspglyasn1510<210>41<211>7<212>prt<213>artificialsequence<400>41thrleusertyrargalaser15<210>42<211>9<212>prt<213>artificialsequence<400>42metglnglyleuglnasnproilethr15<210>43<211>119<212>prt<213>artificialsequence<400>43glnvalglnleuvalglnserglyglyglyvalvalglnproglyarg151015serleuargleusercysthralaserglypheasnpheasnasptyr202530glymettyrtrpvalargglnalaproglylysglyleuglutrpval354045alaasniletrphisaspglyserglnarghistyralaalaserval505560glnglyargphethrthrserargaspasnserargasnthrvaltyr65707580leuglnmetasnserleuargalagluaspthralaleutyrtyrcys859095valargproalaleuleugluvalilepheaspasptrpglyglngly100105110thrleuvalthrvalserser115<210>44<211>114<212>prt<213>artificialsequence<400>44aspileglnmetthrglnserproleuserleuprovalthrprogly151015gluproalaserilesercysargserserglnserleuleuaspser202530aspaspglyasnthrtyrleuasptrptyrleuglnlysproglygln354045serproglnleuleuiletyrthrleusertyrargalaserglyval505560proaspargpheserglyserglyserglythraspphethrleulys65707580ileserargvalglualagluaspvalglyiletyrtyrcysmetgln859095glyleuglnasnproilethrpheglyglnglythrargleugluile100105110lysarg<210>45<211>248<212>prt<213>artificialsequence<400>45glnvalglnleuvalglnserglyglyglyvalvalglnproglyarg151015serleuargleusercysthralaserglypheasnpheasnasptyr202530glymettyrtrpvalargglnalaproglylysglyleuglutrpval354045alaasniletrphisaspglyserglnarghistyralaalaserval505560glnglyargphethrthrserargaspasnserargasnthrvaltyr65707580leuglnmetasnserleuargalagluaspthralaleutyrtyrcys859095valargproalaleuleugluvalilepheaspasptrpglyglngly100105110thrleuvalthrvalserserglyglyglyglyserglyglyglygly115120125serglyglyglyglyseraspileglnmetthrglnserproleuser130135140leuprovalthrproglygluproalaserilesercysargserser145150155160glnserleuleuaspseraspaspglyasnthrtyrleuasptrptyr165170175leuglnlysproglyglnserproglnleuleuiletyrthrleuser180185190tyrargalaserglyvalproaspargpheserglyserglysergly195200205thraspphethrleulysileserargvalglualagluaspvalgly210215220iletyrtyrcysmetglnglyleuglnasnproilethrpheglygln225230235240glythrargleugluilelysarg245<210>46<211>7<212>prt<213>artificialsequence<400>46glyleuthrphesersertyr15<210>47<211>6<212>prt<213>artificialsequence<400>47servalthrglyglythr15<210>48<211>9<212>prt<213>artificialsequence<400>48metlysglyglnleuvalglyserphe15<210>49<211>11<212>prt<213>artificialsequence<400>49argalaserglnserileserthrtyrleuasn1510<210>50<211>7<212>prt<213>artificialsequence<400>50glyalaserserleuglnarg15<210>51<211>9<212>prt<213>artificialsequence<400>51glnglntyraspasnleuproleuthr15<210>52<211>120<212>prt<213>artificialsequence<400>52glnvalglnleuglngluserglyglyglyleuvalglnproglygly151015serleuargleusercysalaalaserglyleuthrphesersertyr202530alametglytrpvalargglnalaproglylysglyleuglutrpval354045serserileservalthrglyglythrthrtyrhisalaalaserval505560argglyargphethrileserargaspasnserlysasnthrleutyr65707580leuglnmetasnserleuargalaaspaspthralailetyrtyrcys859095vallysmetlysglyglnleuvalglyserpheasptyrtrpglygln100105110glythrleuvalthrvalserser115120<210>53<211>108<212>prt<213>artificialsequence<400>53gluilevalleuthrglnserproserserleuseralaservalgly151015aspargvalthrilethrcysargalaserglnserileserthrtyr202530leuasntrptyrglnglnlysproglylysalaproasnleuleuile354045tyrglyalaserserleuglnargglyvalproserargphesergly505560serglyserglythrgluphethrleuthrileserserleuglnpro65707580gluaspilealathrtyrtyrcysglnglntyraspasnleuproleu859095thrpheglyglyglythrlysleugluilelysarg100105<210>54<211>243<212>prt<213>artificialsequence<400>54glnvalglnleuglngluserglyglyglyleuvalglnproglygly151015serleuargleusercysalaalaserglyleuthrphesersertyr202530alametglytrpvalargglnalaproglylysglyleuglutrpval354045serserileservalthrglyglythrthrtyrhisalaalaserval505560argglyargphethrileserargaspasnserlysasnthrleutyr65707580leuglnmetasnserleuargalaaspaspthralailetyrtyrcys859095vallysmetlysglyglnleuvalglyserpheasptyrtrpglygln100105110glythrleuvalthrvalserserglyglyglyglyserglyglygly115120125glyserglyglyglyglysergluilevalleuthrglnserproser130135140serleuseralaservalglyaspargvalthrilethrcysargala145150155160serglnserileserthrtyrleuasntrptyrglnglnlysprogly165170175lysalaproasnleuleuiletyrglyalaserserleuglnarggly180185190valproserargpheserglyserglyserglythrgluphethrleu195200205thrileserserleuglnprogluaspilealathrtyrtyrcysgln210215220glntyraspasnleuproleuthrpheglyglyglythrlysleuglu225230235240ilelysarg<210>55<211>7<212>prt<213>artificialsequence<400>55glytyrserpheserthrtyr15<210>56<211>6<212>prt<213>artificialsequence<400>56sertyraspglyserasn15<210>57<211>8<212>prt<213>artificialsequence<400>57hislysgluileasnpheasptyr15<210>58<211>11<212>prt<213>artificialsequence<400>58argalaserglnserileserserargleuser1510<210>59<211>7<212>prt<213>artificialsequence<400>59seralaserserleuglnthr15<210>60<211>9<212>prt<213>artificialsequence<400>60glnglnsertyrthrthrproargthr15<210>61<211>117<212>prt<213>artificialsequence<400>61glnvalglnleuglngluserglyglyglyvalvalglnproglyarg151015serleuargleusercysalaalaserglytyrserpheserthrtyr202530alailehistrpvalargglnalaproglylysglyleuglutrpval354045alavalilesertyraspglyserasnlysasntyralaaspserval505560lysglyargphethrileserargaspasnserlysserthrleutyr65707580leuaspmetasnserleuargalagluaspthralavaltyrtyrcys859095alathrhislysgluileasnpheasptyrtrpglyglnglythrleu100105110valthrvalserser115<210>62<211>108<212>prt<213>artificialsequence<400>62aspvalvalmetthrglnserproalaserleuseralaservalgly151015aspargvalthrilethrcysargalaserglnserileserserarg202530leusertrptyrglnglnlysproglylysalaprolysleuleuile354045tyrseralaserserleuglnthrglyvalproserargphesergly505560glyglyserargthraspphethrleuthrileserasnleuglnpro65707580gluaspphealaprotyrtyrcysglnglnsertyrthrthrproarg859095thrpheglyglnglythrlysvalaspilelysarg100105<210>63<211>240<212>prt<213>artificialsequence<400>63glnvalglnleuglngluserglyglyglyvalvalglnproglyarg151015serleuargleusercysalaalaserglytyrserpheserthrtyr202530alailehistrpvalargglnalaproglylysglyleuglutrpval354045alavalilesertyraspglyserasnlysasntyralaaspserval505560lysglyargphethrileserargaspasnserlysserthrleutyr65707580leuaspmetasnserleuargalagluaspthralavaltyrtyrcys859095alathrhislysgluileasnpheasptyrtrpglyglnglythrleu100105110valthrvalserserglyglyglyglyserglyglyglyglysergly115120125glyglyglyseraspvalvalmetthrglnserproalaserleuser130135140alaservalglyaspargvalthrilethrcysargalaserglnser145150155160ileserserargleusertrptyrglnglnlysproglylysalapro165170175lysleuleuiletyrseralaserserleuglnthrglyvalproser180185190argpheserglyglyglyserargthraspphethrleuthrileser195200205asnleuglnprogluaspphealaprotyrtyrcysglnglnsertyr210215220thrthrproargthrpheglyglnglythrlysvalaspilelysarg225230235240<210>64<211>7<212>prt<213>artificialsequence<400>64glyphethrpheseralatyr15<210>65<211>6<212>prt<213>artificialsequence<400>65seralaserglythrser15<210>66<211>10<212>prt<213>artificialsequence<400>66glyglyalatyrargthralaleuaspser1510<210>67<211>12<212>prt<213>artificialsequence<400>67argalaserglnservalsersersertyrleuala1510<210>68<211>7<212>prt<213>artificialsequence<400>68alaalaserasnargalathr15<210>69<211>8<212>prt<213>artificialsequence<400>69glnglncysserthrproargthr15<210>70<211>119<212>prt<213>artificialsequence<400>70glnvalasnleuarggluserglyglyglyvalvalglnproglyarg151015serleuargleusercysalaalaserglyphethrpheseralatyr202530glymetsertrpvalargglnalaproglylysglyleuglutrpval354045alavalthrseralaserglythrserthrphehisalaaspserval505560lysglyargphethrileserargaspasnalalysthrthrvalphe65707580leuglnleuasnasnleuargaspaspaspthralavaltyrtyrcys859095alaargglyglyalatyrargthralaleuaspsertrpglyhisgly100105110thrleuvalthrvalserser115<210>71<211>108<212>prt<213>artificialsequence<400>71gluilevalleuthrglnserproglythrleuserleuserprogly151015gluargalathrleusercysargalaserglnservalserserser202530tyrleualatrptyrglnglnlysproglyglnalaproargleuleu354045iletyralaalaserasnargalathrglyileproaspargpheser505560glyasnglyserglythraspphethrleuthrvalserargleuglu65707580progluaspphealavaltyrtyrcysglnglncysserthrproarg859095thrpheglyglnglythrlysleugluilelysarg100105<210>72<211>242<212>prt<213>artificialsequence<400>72glnvalasnleuarggluserglyglyglyvalvalglnproglyarg151015serleuargleusercysalaalaserglyphethrpheseralatyr202530glymetsertrpvalargglnalaproglylysglyleuglutrpval354045alavalthrseralaserglythrserthrphehisalaaspserval505560lysglyargphethrileserargaspasnalalysthrthrvalphe65707580leuglnleuasnasnleuargaspaspaspthralavaltyrtyrcys859095alaargglyglyalatyrargthralaleuaspsertrpglyhisgly100105110thrleuvalthrvalserserglyglyglyglyserglyglyglygly115120125serglyglyglyglysergluilevalleuthrglnserproglythr130135140leuserleuserproglygluargalathrleusercysargalaser145150155160glnservalsersersertyrleualatrptyrglnglnlysprogly165170175glnalaproargleuleuiletyralaalaserasnargalathrgly180185190ileproaspargpheserglyasnglyserglythraspphethrleu195200205thrvalserargleugluprogluaspphealavaltyrtyrcysgln210215220glncysserthrproargthrpheglyglnglythrlysleugluile225230235240lysarg<210>73<211>7<212>prt<213>artificialsequence<400>73glytyrthrpheasnthrtyr15<210>74<211>6<212>prt<213>artificialsequence<400>74aspproasnaspserser15<210>75<211>12<212>prt<213>artificialsequence<400>75glyprolystrpgluleuhisasntyrpheasptyr1510<210>76<211>11<212>prt<213>artificialsequence<400>76glnglyaspthrvalargasnhispheproser1510<210>77<211>7<212>prt<213>artificialsequence<400>77glylysasnasnargproser15<210>78<211>12<212>prt<213>artificialsequence<400>78asnserargaspthrargglyasnglnmetglyleu1510<210>79<211>121<212>prt<213>artificialsequence<400>79glyvalglnleuvalgluserglyalaglualalyslysproglyglu151015serleuargilesercysglnileserglytyrthrpheasnthrtyr202530trpilesertrpleuargglnmetproglylysglyproglutrpmet354045glyargileaspproasnaspserserthrasptyrserproserphe505560glnglyhisilethrileserthraspasnserileargthralatyr65707580leuglntrpserserleuargthrseraspthralaleutyrtyrcys859095alalysglyprolystrpgluleuhisasntyrpheasptyrtrpgly100105110glnglythrleuvalthrvalserser115120<210>80<211>108<212>prt<213>artificialsequence<400>80gluleuthrglnaspproalavalservalalaleuglyglnthrval151015argilethrcysglnglyaspthrvalargasnhispheprosertrp202530tyrglnglnlysproglyglnalaprolysvalvalleutyrglylys354045asnasnargproserglyvalproaspargpheserglyserasnser505560glyasnthralaserleuileilethrglyalaglnalagluaspglu65707580alaasptyrtyrcysasnserargaspthrargglyasnglnmetgly859095leupheglythrglythrlysvalthrvalleugly100105<210>81<211>246<212>prt<213>artificialsequence<400>81glyvalglnleuvalgluserglyalaglualalyslysproglyglu151015serleuargilesercysglnileserglytyrthrpheasnthrtyr202530trpilesertrpleuargglnmetproglylysglyproglutrpmet354045glyargileaspproasnaspserserthrasptyrserproserphe505560glnglyhisilethrileserthraspasnserileargthralatyr65707580leuglntrpserserleuargthrseraspthralaleutyrtyrcys859095alalysglyprolystrpgluleuhisasntyrpheasptyrtrpgly100105110glnglythrleuvalthrvalserserglyglyglyglyserglygly115120125glyglyserglyglyglyglysersersergluleuthrglnasppro130135140alavalservalalaleuglyglnthrvalargilethrcysglngly145150155160aspthrvalargasnhispheprosertrptyrglnglnlysprogly165170175glnalaprolysvalvalleutyrglylysasnasnargprosergly180185190valproaspargpheserglyserasnserglyasnthralaserleu195200205ileilethrglyalaglnalagluaspglualaasptyrtyrcysasn210215220serargaspthrargglyasnglnmetglyleupheglythrglythr225230235240lysvalthrvalleugly245<210>82<211>7<212>prt<213>artificialsequence<400>82glyphethrpheseralatyr15<210>83<211>5<212>prt<213>artificialsequence<400>83seralaserglythr15<210>84<211>10<212>prt<213>artificialsequence<400>84glyglyalatyrargthralaleuaspser1510<210>85<211>12<212>prt<213>artificialsequence<400>85argalaserglnservalsersersertyrleuala1510<210>86<211>7<212>prt<213>artificialsequence<400>86alaalaserasnargalathr15<210>87<211>8<212>prt<213>artificialsequence<400>87glnglncysserthrproargthr15<210>88<211>119<212>prt<213>artificialsequence<400>88glnvalasnleuarggluserglyglyglyvalvalglnproglyarg151015serleuargleusercysalaalaserglyphethrpheseralatyr202530glymetsertrpvalargglnalaproglylysglyleuglutrpval354045alavalthrseralaserglythrserthrphehisalaaspserval505560lysglyargphethrileserargaspasnalalysthrthrvalphe65707580leuglnleuasnasnleuargaspaspaspthralavaltyrtyrcys859095alaargglyglyalatyrargthralaleuaspsertrpglyhisgly100105110thrleuvalthrvalserser115<210>89<211>108<212>prt<213>artificialsequence<400>89gluilevalleuthrglnserproglythrleuserleuserprogly151015gluargalathrleusercysargalaserglnservalserserser202530tyrleualatrptyrglnglnlysproglyglnalaproargleuleu354045iletyralaalaserasnargalathrglyileproaspargpheser505560glyasnglyserglythraspphethrleuthrvalserargleuglu65707580progluaspphealavaltyrtyrcysglnglncysserthrproarg859095thrpheglyglnglythrlysleugluilelysarg100105<210>90<211>242<212>prt<213>artificialsequence<400>90glnvalasnleuarggluserglyglyglyvalvalglnproglyarg151015serleuargleusercysalaalaserglyphethrpheseralatyr202530glymetsertrpvalargglnalaproglylysglyleuglutrpval354045alavalthrseralaserglythrserthrphehisalaaspserval505560lysglyargphethrileserargaspasnalalysthrthrvalphe65707580leuglnleuasnasnleuargaspaspaspthralavaltyrtyrcys859095alaargglyglyalatyrargthralaleuaspsertrpglyhisgly100105110thrleuvalthrvalserserglyglyglyglyserglyglyglygly115120125serglyglyglyglysergluilevalleuthrglnserproglythr130135140leuserleuserproglygluargalathrleusercysargalaser145150155160glnservalsersersertyrleualatrptyrglnglnlysprogly165170175glnalaproargleuleuiletyralaalaserasnargalathrgly180185190ileproaspargpheserglyasnglyserglythraspphethrleu195200205thrvalserargleugluprogluaspphealavaltyrtyrcysgln210215220glncysserthrproargthrpheglyglnglythrlysleugluile225230235240lysarg<210>91<211>7<212>prt<213>artificialsequence<400>91glyglythrphesersertyr15<210>92<211>6<212>prt<213>artificialsequence<400>92ileproileleuglyile15<210>93<211>8<212>prt<213>artificialsequence<400>93glntyrserserserpheasppro15<210>94<211>11<212>prt<213>artificialsequence<400>94argalaserargserileasnlystrpleuala1510<210>95<211>7<212>prt<213>artificialsequence<400>95seralaserthrleugluser15<210>96<211>8<212>prt<213>artificialsequence<400>96glnglntyrhisasptyrprothr15<210>97<211>117<212>prt<213>artificialsequence<400>97glnvalglnleuleuglnseralaalagluvallyslysproglyser151015servallysvalsercyslysalaserglyglythrphesersertyr202530thrilesertrpvalargglnalaproglyglnglyleuglutrpmet354045glyargileileproileleuglyilealaasntyralaglnlysphe505560glnglyargvalthrilethralaasplysserthrserthralatyr65707580metgluleuserserleuargsergluaspthralavaltyrtyrcys859095alaargglntyrserserserpheaspprotrpglyglnglythrleu100105110valthrvalserser115<210>98<211>107<212>prt<213>artificialsequence<400>98aspilevalmetthrglnserproserthrleuseralaservalgly151015aspargvalthrilethrcysargalaserargserileasnlystrp202530leualatrptyrglnhislysproglylysvalprolysleuleuile354045pheseralaserthrleugluserglyvalproserargphesergly505560serglyserglythrgluphethrleuthrileasnserleuglnpro65707580aspaspphealathrphetyrcysglnglntyrhisasptyrprothr859095pheglyglnglythrlysvalgluilelysarg100105<210>99<211>239<212>prt<213>artificialsequence<400>99glnvalglnleuleuglnseralaalagluvallyslysproglyser151015servallysvalsercyslysalaserglyglythrphesersertyr202530thrilesertrpvalargglnalaproglyglnglyleuglutrpmet354045glyargileileproileleuglyilealaasntyralaglnlysphe505560glnglyargvalthrilethralaasplysserthrserthralatyr65707580metgluleuserserleuargsergluaspthralavaltyrtyrcys859095alaargglntyrserserserpheaspprotrpglyglnglythrleu100105110valthrvalserserglyglyglyglyserglyglyglyglysergly115120125glyglyglyseraspilevalmetthrglnserproserthrleuser130135140alaservalglyaspargvalthrilethrcysargalaserargser145150155160ileasnlystrpleualatrptyrglnhislysproglylysvalpro165170175lysleuleuilepheseralaserthrleugluserglyvalproser180185190argpheserglyserglyserglythrgluphethrleuthrileasn195200205serleuglnproaspaspphealathrphetyrcysglnglntyrhis210215220asptyrprothrpheglyglnglythrlysvalgluilelysarg225230235<210>100<211>7<212>prt<213>artificialsequence<400>100glytyrthrphealasertyr15<210>101<211>6<212>prt<213>artificialsequence<400>101thrproserserglyasn15<210>102<211>15<212>prt<213>artificialsequence<400>102glyproglnserglytyrthrtyrtyrtyrtyrglyleuaspval151015<210>103<211>11<212>prt<213>artificialsequence<400>103argalaserglnserileserthrtrpleuala1510<210>104<211>7<212>prt<213>artificialsequence<400>104lysalaserserleugluser15<210>105<211>10<212>prt<213>artificialsequence<400>105glnglntyrasnsertyrserprogluthr1510<210>106<211>124<212>prt<213>artificialsequence<400>106glnvalglnleuvalglnserglyalagluvallyslysproglyala151015servallysvalsercysthralaserglytyrthrphealasertyr202530aspileasntrpvalargglnalathrglyglnglyleuglutrpmet354045glytrpmetthrproserserglyasnthrglytyralaglnlysphe505560glnglyargvalthrmetthrargasnthrserileserthralatyr65707580metgluvalserserleuargsergluaspthralavaltyrtyrcys859095alaargglyproglnserglytyrthrtyrtyrtyrtyrglyleuasp100105110valtrpglyglnglythrmetvalthrvalserser115120<210>107<211>109<212>prt<213>artificialsequence<400>107gluilevalleuthrglnserproserthrleuseralaservalgly151015aspargvalthrilethrcysargalaserglnserileserthrtrp202530leualatrptyrlysglnlysproglylysalaprogluleuleuile354045tyrlysalaserserleugluserglyvalproserargphesergly505560serglyserglythrgluphethrleuthrileserserleuglnpro65707580aspaspphealathrtyrtyrcysglnglntyrasnsertyrserpro859095gluthrpheglyglnglythrlysvalaspilelysarg100105<210>108<211>248<212>prt<213>artificialsequence<400>108glnvalglnleuvalglnserglyalagluvallyslysproglyala151015servallysvalsercysthralaserglytyrthrphealasertyr202530aspileasntrpvalargglnalathrglyglnglyleuglutrpmet354045glytrpmetthrproserserglyasnthrglytyralaglnlysphe505560glnglyargvalthrmetthrargasnthrserileserthralatyr65707580metgluvalserserleuargsergluaspthralavaltyrtyrcys859095alaargglyproglnserglytyrthrtyrtyrtyrtyrglyleuasp100105110valtrpglyglnglythrmetvalthrvalserserglyglyglygly115120125serglyglyglyglyserglyglyglyglysergluilevalleuthr130135140glnserproserthrleuseralaservalglyaspargvalthrile145150155160thrcysargalaserglnserileserthrtrpleualatrptyrlys165170175glnlysproglylysalaprogluleuleuiletyrlysalaserser180185190leugluserglyvalproserargpheserglyserglyserglythr195200205gluphethrleuthrileserserleuglnproaspaspphealathr210215220tyrtyrcysglnglntyrasnsertyrserprogluthrpheglygln225230235240glythrlysvalaspilelysarg245<210>109<211>5<212>prt<213>artificialsequence<400>109glytyrserphethr15<210>110<211>5<212>prt<213>artificialsequence<400>110tyrproglyasppro15<210>111<211>13<212>prt<213>artificialsequence<400>111servalserthrvalvalthrproglyglypheaspser1510<210>112<211>13<212>prt<213>artificialsequence<400>112thrargserserglyserilealaserasntyrvalhis1510<210>113<211>7<212>prt<213>artificialsequence<400>113gluaspasnhisargproser15<210>114<211>9<212>prt<213>artificialsequence<400>114glnsertyraspserthrthrtrpphe15<210>115<211>122<212>prt<213>artificialsequence<400>115glnvalglnleuglnglnserglyalagluvallyslysproglyglu151015serleulysilesercyslysalaserglytyrserphethrsertyr202530trpileglytrpvalargglnmetproglylysglyleuglutrpmet354045glymetiletyrproglyaspprogluileargtyrserproserphe505560glnglyglnvalserileseralaasplysservalasnthralatyr65707580leuglntrpserserleulysalaseraspthralamettyrtyrcys859095metargservalserthrvalvalthrproglyglypheaspsertrp100105110glyglnglythrleuvalthrvalserser115120<210>116<211>111<212>prt<213>artificialsequence<400>116asnphemetleuthrglnprohisservalsergluserproglylys151015thrvalthrilethrcysthrargserserglyserilealaserasn202530tyrvalhistrpcysglnglnargproglyseralaprothrthrval354045ilephegluaspasnhisargproserglyvalproaspargpheser505560glyserileaspargserserasnseralaserleuthrilesergly65707580leuglnalagluaspglualaasptyrtyrcysglnsertyraspser859095thrthrtrpphepheglythrglythrglnleuthrvalleuser100105110<210>117<211>248<212>prt<213>artificialsequence<400>117glnvalglnleuglnglnserglyalagluvallyslysproglyglu151015serleulysilesercyslysalaserglytyrserphethrsertyr202530trpileglytrpvalargglnmetproglylysglyleuglutrpmet354045glymetiletyrproglyaspprogluileargtyrserproserphe505560glnglyglnvalserileseralaasplysservalasnthralatyr65707580leuglntrpserserleulysalaseraspthralamettyrtyrcys859095metargservalserthrvalvalthrproglyglypheaspsertrp100105110glyglnglythrleuvalthrvalserserglyglyglyglysergly115120125glyglyglyserglyglyglyglyserasnphemetleuthrglnpro130135140hisservalsergluserproglylysthrvalthrilethrcysthr145150155160argserserglyserilealaserasntyrvalhistrpcysglngln165170175argproglyseralaprothrthrvalilephegluaspasnhisarg180185190proserglyvalproaspargpheserglyserileaspargserser195200205asnseralaserleuthrileserglyleuglnalagluaspgluala210215220asptyrtyrcysglnsertyraspserthrthrtrpphepheglythr225230235240glythrglnleuthrvalleuser245<210>118<211>7<212>prt<213>artificialsequence<400>118glyphethrphesersertyr15<210>119<211>6<212>prt<213>artificialsequence<400>119serthrglyglyserthr15<210>120<211>11<212>prt<213>artificialsequence<400>120ileserglyphetyrphetyrglymetaspval1510<210>121<211>11<212>prt<213>artificialsequence<400>121glnglyaspserleuargiletyrtyrprogly1510<210>122<211>7<212>prt<213>artificialsequence<400>122glylysasnmetargproser15<210>123<211>11<212>prt<213>artificialsequence<400>123asnserargaspserserglylysargvalleu1510<210>124<211>120<212>prt<213>artificialsequence<400>124glnvalglnleuvalglnserglyglyaspleuvalglnproglygly151015serleuargleusercysalaalaproglyphethrphesersertyr202530glumetasntrpvalargglnalaproglylysglyleuglutrpval354045sertyrileserthrglyglyserthriletyrtyralaaspserval505560lysglyargphethrileserargaspasnalaargaspserleutyr65707580leuglumetasnserleuargalagluaspthralavaltyrtyrcys859095alaargileserglyphetyrphetyrglymetaspvaltrpglygln100105110glythrmetvalthrvalserser115120<210>125<211>107<212>prt<213>artificialsequence<400>125gluleuthrglnaspproalavalservalalaleuglyglnthrval151015argilethrcysglnglyaspserleuargiletyrtyrproglytrp202530tyrglnglnlysproglyglnalaproileleuvaliletyrglylys354045asnmetargproserglyvalproaspargpheserglyserargser505560glyasnthralaserleuthrilethrglyserargalagluaspglu65707580alaasptyrtyrcysasnserargaspserserglylysargvalleu859095pheglyglyglythrlysleuthrvalleugly100105<210>126<211>244<212>prt<213>artificialsequence<400>126glnvalglnleuvalglnserglyglyaspleuvalglnproglygly151015serleuargleusercysalaalaproglyphethrphesersertyr202530glumetasntrpvalargglnalaproglylysglyleuglutrpval354045sertyrileserthrglyglyserthriletyrtyralaaspserval505560lysglyargphethrileserargaspasnalaargaspserleutyr65707580leuglumetasnserleuargalagluaspthralavaltyrtyrcys859095alaargileserglyphetyrphetyrglymetaspvaltrpglygln100105110glythrmetvalthrvalserserglyglyglyglyserglyglygly115120125glyserglyglyglyglysersersergluleuthrglnaspproala130135140valservalalaleuglyglnthrvalargilethrcysglnglyasp145150155160serleuargiletyrtyrproglytrptyrglnglnlysproglygln165170175alaproileleuvaliletyrglylysasnmetargproserglyval180185190proaspargpheserglyserargserglyasnthralaserleuthr195200205ilethrglyserargalagluaspglualaasptyrtyrcysasnser210215220argaspserserglylysargvalleupheglyglyglythrlysleu225230235240thrvalleugly<210>127<211>8<212>prt<213>artificialsequence<400>127glyglyserileserglyglyasp15<210>128<211>5<212>prt<213>artificialsequence<400>128tyrhisthrglygly15<210>129<211>12<212>prt<213>artificialsequence<400>129alaproaspaspthrserproglyglyleuasptyr1510<210>130<211>11<212>prt<213>artificialsequence<400>130argalaproglnaspileargasnserleuala1510<210>131<211>7<212>prt<213>artificialsequence<400>131alaalaserserleuglnser15<210>132<211>9<212>prt<213>artificialsequence<400>132glnglntyrglyaspserproleuthr15<210>133<211>121<212>prt<213>artificialsequence<400>133glnvalglnleuglngluserglyproglyleuvallysprosergly151015thrleuserleuthrcysalavalserglyglyserileserglygly202530asptrptrpsertrpvalargglnproproglylysglyleuglutrp354045ileglygluiletyrhisthrglyglythrasntyrasnproserleu505560lysargargvalthrileservalaspthrserargasnglnpheser65707580leuglnleuthrservalthralaalaaspthralavaltyrtyrcys859095metthralaproaspaspthrserproglyglyleuasptyrtrpgly100105110glnglythrleuvalthrvalserser115120<210>134<211>108<212>prt<213>artificialsequence<400>134aspilevalmetthrglnserproserserleuseralaservalgly151015aspargvalthrilethrcysargalaproglnaspileargasnser202530leualatrptyrglnglnlysproglylysalaprolysleuleuile354045seralaalaserserleuglnserglyvalproserargphesergly505560serglyserglythraspphethrleuthrileserargleuglupro65707580gluaspphealavaltyrtyrcysglnglntyrglyaspserproleu859095thrvalglyglyglythrlysvalgluilelysarg100105<210>135<211>244<212>prt<213>artificialsequence<400>135glnvalglnleuglngluserglyproglyleuvallysprosergly151015thrleuserleuthrcysalavalserglyglyserileserglygly202530asptrptrpsertrpvalargglnproproglylysglyleuglutrp354045ileglygluiletyrhisthrglyglythrasntyrasnproserleu505560lysargargvalthrileservalaspthrserargasnglnpheser65707580leuglnleuthrservalthralaalaaspthralavaltyrtyrcys859095metthralaproaspaspthrserproglyglyleuasptyrtrpgly100105110glnglythrleuvalthrvalserserglyglyglyglyserglygly115120125glyglyserglyglyglyglyseraspilevalmetthrglnserpro130135140serserleuseralaservalglyaspargvalthrilethrcysarg145150155160alaproglnaspileargasnserleualatrptyrglnglnlyspro165170175glylysalaprolysleuleuileseralaalaserserleuglnser180185190glyvalproserargpheserglyserglyserglythraspphethr195200205leuthrileserargleugluprogluaspphealavaltyrtyrcys210215220glnglntyrglyaspserproleuthrvalglyglyglythrlysval225230235240gluilelysarg<210>136<211>5<212>prt<213>artificialsequence<400>136glytyrserphethr15<210>137<211>6<212>prt<213>artificialsequence<400>137tyrproglyaspserasp15<210>138<211>18<212>prt<213>artificialsequence<400>138argleuserileargargglnmetasntrpglyproglyaspalaphe151015aspleu<210>139<211>11<212>prt<213>artificialsequence<400>139argalaserglnservalserlyspheleuasn1510<210>140<211>7<212>prt<213>artificialsequence<400>140aspvalserthrleuglnthr15<210>141<211>9<212>prt<213>artificialsequence<400>141glnglnsertyrserthrproleuthr15<210>142<211>127<212>prt<213>artificialsequence<400>142glnvalglnleuglnglnserglyalagluvallyslysproglyglu151015serleulysilesercyslysglyserglytyrserphethrsertyr202530trpileglytrpvalargglnmetproglylysglyleuglutrpmet354045glyileiletyrproglyaspseraspthrargtyrserproserphe505560glnglyglnvalthrileseralaasplysserileserthralatyr65707580leuglntrpsercysleulysalaseraspthralamettyrtyrcys859095alaargargleuserileargargglnmetasntrpglyproglyasp100105110alapheaspleutrpglyglnglythrmetvalthrvalserser115120125<210>143<211>108<212>prt<213>artificialsequence<400>143gluilevalleuthrglnserproserserleuseralaservalgly151015aspargvalthrilethrcysargalaserglnservalserlysphe202530leuasntrptyrglnleulysproglylysalaprolysleuleuile354045tyraspvalserthrleuglnthrglyvalproserargphethrgly505560serargserglythraspphethrleuthrileasnserleuglnpro65707580gluaspphealathrtyrphecysglnglnsertyrserthrproleu859095thrpheglyglyglythrlysleugluilelysarg100105<210>144<211>250<212>prt<213>artificialsequence<400>144glnvalglnleuglnglnserglyalagluvallyslysproglyglu151015serleulysilesercyslysglyserglytyrserphethrsertyr202530trpileglytrpvalargglnmetproglylysglyleuglutrpmet354045glyileiletyrproglyaspseraspthrargtyrserproserphe505560glnglyglnvalthrileseralaasplysserileserthralatyr65707580leuglntrpsercysleulysalaseraspthralamettyrtyrcys859095alaargargleuserileargargglnmetasntrpglyproglyasp100105110alapheaspleutrpglyglnglythrmetvalthrvalsersergly115120125glyglyglyserglyglyglyglyserglyglyglyglysergluile130135140valleuthrglnserproserserleuseralaservalglyasparg145150155160valthrilethrcysargalaserglnservalserlyspheleuasn165170175trptyrglnleulysproglylysalaprolysleuleuiletyrasp180185190valserthrleuglnthrglyvalproserargphethrglyserarg195200205serglythraspphethrleuthrileasnserleuglnprogluasp210215220phealathrtyrphecysglnglnsertyrserthrproleuthrphe225230235240glyglyglythrlysleugluilelysarg245250<210>145<211>7<212>prt<213>artificialsequence<400>145glyglyserileserglytyr15<210>146<211>5<212>prt<213>artificialsequence<400>146hisaspthrglygly15<210>147<211>9<212>prt<213>artificialsequence<400>147glyiletyrglyalatyrpheaspser15<210>148<211>11<212>prt<213>artificialsequence<400>148argthrserglnserileasnargtyrleuala1510<210>149<211>7<212>prt<213>artificialsequence<400>149argalaserserargalathr15<210>150<211>8<212>prt<213>artificialsequence<400>150glnglntyrserasptrpprothr15<210>151<211>117<212>prt<213>artificialsequence<400>151glnvalglnleuglngluserglyproglyleuvallysproserglu151015thrleuserleuthrcysthrleuserglyglyserileserglytyr202530tyrtrpsertrpileargglnproproglylysglyleuglutrpile354045alatyrilehisaspthrglyglyileglutyrtyrproserleulys505560thrargleuthrileserseraspalaserargasnglnpheserleu65707580lysleuserservalthralaalaaspthralavaltyrtyrcysala859095argglyiletyrglyalatyrpheaspsertrpglyglnglythrleu100105110valthrvalserser115<210>159<211>107<212>prt<213>artificialsequence<400>159gluilevalleuthrglnserproalathrleuserleuserprogly151015gluargalathrleusercysargthrserglnserileasnargtyr202530leualatrptyrglnglnlysleuglyglnalaproargleuleuile354045tyrargalaserserargalathrglyileproaspargphesergly505560serglyserglythraspphethrleuthrileserargleuglupro65707580gluasppheglyvaltyrtyrcysglnglntyrserasptrpprothr859095pheglyglyglythrlysvalgluilelysarg100105<210>152<211>239<212>prt<213>artificialsequence<400>152glnvalglnleuglngluserglyproglyleuvallysproserglu151015thrleuserleuthrcysthrleuserglyglyserileserglytyr202530tyrtrpsertrpileargglnproproglylysglyleuglutrpile354045alatyrilehisaspthrglyglyileglutyrtyrproserleulys505560thrargleuthrileserseraspalaserargasnglnpheserleu65707580lysleuserservalthralaalaaspthralavaltyrtyrcysala859095argglyiletyrglyalatyrpheaspsertrpglyglnglythrleu100105110valthrvalserserglyglyglyglyserglyglyglyglysergly115120125glyglyglysergluilevalleuthrglnserproalathrleuser130135140leuserproglygluargalathrleusercysargthrserglnser145150155160ileasnargtyrleualatrptyrglnglnlysleuglyglnalapro165170175argleuleuiletyrargalaserserargalathrglyileproasp180185190argpheserglyserglyserglythraspphethrleuthrileser195200205argleugluprogluasppheglyvaltyrtyrcysglnglntyrser210215220asptrpprothrpheglyglyglythrlysvalgluilelysarg225230235<210>153<211>7<212>prt<213>artificialsequence<400>153glyglythrphesersertyr15<210>154<211>6<212>prt<213>artificialsequence<400>154ileileargpheleugly15<210>155<211>12<212>prt<213>artificialsequence<400>155gluproglygluargaspproaspalavalaspile1510<210>156<211>7<212>prt<213>artificialsequence<400>156glyglythrphesersertyr15<210>157<211>6<212>prt<213>artificialsequence<400>157ileileargpheleugly15<210>158<211>12<212>prt<213>artificialsequence<400>158gluproglygluargaspproaspalavalaspile1510<210>160<211>124<212>prt<213>artificialsequence<400>160glnvalglnleuvalglnserglyalagluvallyslysproglyser151015servallysvalsercyslysalapheglyglythrphesersertyr202530alailesertrpvalargglnalaproglyglnglyleuglutrpmet354045glyargileileargpheleuglyilealaasntyralaglnlysphe505560glnglyargvalthrleuilealaasplysserthrasnthralatyr65707580metgluleuserserleuargsergluaspthralavaltyrtyrcys859095alaglygluproglygluproglygluargaspproaspalavalasp100105110iletrpglyglnglythrmetvalthrvalserser115120<210>161<211>107<212>prt<213>artificialsequence<400>161aspileglnmetthrglnserproserserleuseralaservalgly151015aspargvalthrilethrcysargalaserglnglyileserasntyr202530leualatrppheglnglnlysproglylysalaprolysserleuile354045tyralaalaserserleuglnserglyvalproserargphesergly505560serglyserglythraspphethrleuthrileserserleuglnpro65707580gluaspphealathrtyrtyrcysglnglntyrasnsertyrprotyr859095thrpheglyglnglythrlysleugluilelys100105<210>162<211>246<212>prt<213>artificialsequence<400>162glnvalglnleuvalglnserglyalagluvallyslysproglyser151015servallysvalsercyslysalapheglyglythrphesersertyr202530alailesertrpvalargglnalaproglyglnglyleuglutrpmet354045glyargileileargpheleuglyilealaasntyralaglnlysphe505560glnglyargvalthrleuilealaasplysserthrasnthralatyr65707580metgluleuserserleuargsergluaspthralavaltyrtyrcys859095alaglygluproglygluproglygluargaspproaspalavalasp100105110iletrpglyglnglythrmetvalthrvalserserglyglyglygly115120125serglyglyglyglyserglyglyglyglyseraspileglnmetthr130135140glnserproserserleuseralaservalglyaspargvalthrile145150155160thrcysargalaserglnglyileserasntyrleualatrpphegln165170175glnlysproglylysalaprolysserleuiletyralaalaserser180185190leuglnserglyvalproserargpheserglyserglyserglythr195200205aspphethrleuthrileserserleuglnprogluaspphealathr210215220tyrtyrcysglnglntyrasnsertyrprotyrthrpheglyglngly225230235240thrlysleugluilelys245<210>163<211>7<212>prt<213>artificialsequence<400>163glyglyserileserserleu15<210>164<211>5<212>prt<213>artificialsequence<400>164argtyrserglylys15<210>165<211>11<212>prt<213>artificialsequence<400>165aspserglyglyglytyrasntrppheasppro1510<210>166<211>11<212>prt<213>artificialsequence<400>166argalaserglnserileserthrtyrleuasn1510<210>167<211>7<212>prt<213>artificialsequence<400>167alaalaserserleuglngly15<210>168<211>9<212>prt<213>artificialsequence<400>168glnglnsertyrasnalaproargthr15<210>169<211>119<212>prt<213>artificialsequence<400>169glnvalglnleuglnglnserglyproglyleuvallysproserglu151015thrleuserleuthrcysthrvalserglyglyserileserserleu202530glntrpasntrpileargglnproleuglylysglyleuglutrpile354045glyphevalargtyrserglylysasnsertyrasnproserleugly505560serargvalthrmetserleuaspmetserlysasnglnpheserleu65707580asnleuserserleuthralaalaaspthralavaltyrtyrcysala859095argaspserglyglyglytyrasntrppheaspprotrpglyglngly100105110thrleuvalthrvalserser115<210>170<211>108<212>prt<213>artificialsequence<400>170gluilevalleuthrglnserproasppheglnservalthrprolys151015glulysvalthrilethrcysargalaserglnserileserthrtyr202530leuasntrptyrglnglnlysproglylysalaprolysileleuile354045seralaalaserserleuglnglyglyvalproserargphesergly505560serglyserglythraspphethrleuileileserasnleuglnpro65707580gluaspphealasertyrtyrcysglnglnsertyrasnalaproarg859095thrpheglyglnglythrlysvalgluilelysarg100105<210>171<211>242<212>prt<213>artificialsequence<400>171glnvalglnleuglnglnserglyproglyleuvallysproserglu151015thrleuserleuthrcysthrvalserglyglyserileserserleu202530glntrpasntrpileargglnproleuglylysglyleuglutrpile354045glyphevalargtyrserglylysasnsertyrasnproserleugly505560serargvalthrmetserleuaspmetserlysasnglnpheserleu65707580asnleuserserleuthralaalaaspthralavaltyrtyrcysala859095argaspserglyglyglytyrasntrppheaspprotrpglyglngly100105110thrleuvalthrvalserserglyglyglyglyserglyglyglygly115120125serglyglyglyglysergluilevalleuthrglnserproaspphe130135140glnservalthrprolysglulysvalthrilethrcysargalaser145150155160glnserileserthrtyrleuasntrptyrglnglnlysproglylys165170175alaprolysileleuileseralaalaserserleuglnglyglyval180185190proserargpheserglyserglyserglythraspphethrleuile195200205ileserasnleuglnprogluaspphealasertyrtyrcysglngln210215220sertyrasnalaproargthrpheglyglnglythrlysvalgluile225230235240lysarg<210>172<211>7<212>prt<213>artificialsequence<400>172glyphethrpheserasntyr15<210>173<211>6<212>prt<213>artificialsequence<400>173serglyserglyileasn15<210>174<211>6<212>prt<213>artificialsequence<400>174hisglyglyglyserphe15<210>175<211>11<212>prt<213>artificialsequence<400>175argalaserglnasnmetasnsertyrleuasn1510<210>176<211>7<212>prt<213>artificialsequence<400>176alaalaserserleuglnser15<210>177<211>9<212>prt<213>artificialsequence<400>177glnglnalaasnserpheprotyrthr15<210>178<211>115<212>prt<213>artificialsequence<400>178glnvalglnleuglnglnserglyglyglyleuvalglnproglygly151015serleuargleusercysalaalaserglyphethrpheserasntyr202530alaileiletrpvalargglnalaproglylysglyleuglutrpval354045servalileserglyserglyileasnthrtyrtyralaaspserval505560lysglyargphethrileserargaspasnserlysasnthrvaltyr65707580leuglnleuasnserleuargalagluaspthralailetyrtyrcys859095valglyhisglyglyglyserphetrpglyglnglythrleuvalthr100105110valserser115<210>179<211>108<212>prt<213>artificialsequence<400>179gluilevalleuthrglnserproserserleuseralaservalgly151015aspargvalthrilethrcysargalaserglnasnmetasnsertyr202530leuasntrptyrglnglnlysproglylysserprolysvalleuile354045tyralaalaserserleuglnserglyvalproserargphesergly505560serglyserglythraspphethrleuthrileserserleuglnpro65707580gluaspphealathrtyrtyrcysglnglnalaasnserpheprotyr859095thrpheglyproglythrlysvalgluilelysarg100105<210>180<211>238<212>prt<213>artificialsequence<400>180glnvalglnleuglnglnserglyglyglyleuvalglnproglygly151015serleuargleusercysalaalaserglyphethrpheserasntyr202530alaileiletrpvalargglnalaproglylysglyleuglutrpval354045servalileserglyserglyileasnthrtyrtyralaaspserval505560lysglyargphethrileserargaspasnserlysasnthrvaltyr65707580leuglnleuasnserleuargalagluaspthralailetyrtyrcys859095valglyhisglyglyglyserphetrpglyglnglythrleuvalthr100105110valserserglyglyglyglyserglyglyglyglyserglyglygly115120125glysergluilevalleuthrglnserproserserleuseralaser130135140valglyaspargvalthrilethrcysargalaserglnasnmetasn145150155160sertyrleuasntrptyrglnglnlysproglylysserprolysval165170175leuiletyralaalaserserleuglnserglyvalproserargphe180185190serglyserglyserglythraspphethrleuthrileserserleu195200205glnprogluaspphealathrtyrtyrcysglnglnalaasnserphe210215220protyrthrpheglyproglythrlysvalgluilelysarg225230235<210>181<211>22<212>prt<213>artificialsequence<400>181metleuleuleuvalthrserleuleuleucysgluleuprohispro151015alapheleuleuilepro20<210>182<211>45<212>prt<213>artificialsequence<400>182thrthrthrproalaproargproprothrproalaprothrileala151015serglnproleuserleuargproglualacysargproalaalagly202530glyalavalhisthrargglyleuaspphealacysasp354045<210>183<211>24<212>prt<213>artificialsequence<400>183iletyriletrpalaproleualaglythrcysglyvalleuleuleu151015serleuvalilethrleutyrcys20<210>184<211>42<212>prt<213>artificialsequence<400>184lysargglyarglyslysleuleutyrilephelysglnprophemet151015argprovalglnthrthrglnglugluaspglycyssercysargphe202530proglugluglugluglyglycysgluleu3540<210>185<211>68<212>prt<213>artificialsequence<400>185phetrpvalleuvalvalvalglyglyvalleualacystyrserleu151015leuvalthrvalalapheileilephetrpvalargserlysargser202530argglyglyhisserasptyrmetasnmetthrproargargprogly354045prothrarglyshistyrglnprotyralaproproargasppheala505560alatyrargser65<210>186<211>112<212>prt<213>artificialsequence<400>186argvallyspheserargseralaaspalaproalatyrglnglngly151015glnasnglnleutyrasngluleuasnleuglyargarggluglutyr202530aspvalleuasplysargargglyargaspproglumetglyglylys354045proargarglysasnproglngluglyleutyrasngluleuglnlys505560asplysmetalaglualatyrsergluileglymetlysglygluarg65707580argargglylysglyhisaspglyleutyrglnglyleuserthrala859095thrlysaspthrtyraspalaleuhismetglnalaleuproproarg100105110<210>187<211>21<212>dna<213>artificialsequence<400>187atgttgcagatggctgggcag21<210>188<211>21<212>dna<213>artificialsequence<400>188tacctagcagaaattgatttc21<210>189<211>18<212>dna<213>artificialsequence<400>189tgtgatcatgttgcagat18<210>190<211>18<212>dna<213>artificialsequence<400>190tacctagcagaaattgat18<210>191<211>25<212>dna<213>artificialsequence<400>191tatcgatgctccggtgcccgtcagt25<210>192<211>22<212>dna<213>artificialsequence<400>192tcacgacacctgaaatggaaga22<210>193<211>17<212>dna<213>artificialsequence<400>193tccgggactttcgcttt17<210>194<211>19<212>dna<213>artificialsequence<400>194cagaatccaggtggcaaca19<210>195<211>21<212>dna<213>artificialsequence<400>195catgtacgttgctatccaggc21<210>196<211>20<212>dna<213>artificialsequence<400>196tccttaatgtcacgcacgat20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1