一种TEM8抗体及其应用的制作方法
本发明属于基因工程抗体技术领域,具体的涉及一种tem8抗体及其应用。
背景技术:
肿瘤内皮标记物(tem8)是肿瘤内皮标记物(tumorendothelialmarkers,tems)中的一种,是2000年{bradst.croix}等利用基因表达系列分析技术对正常内皮细胞及癌症内皮细胞的基因进行研究中发现的。tem8由564个氨基酸残基组成的具有完整跨膜螺旋的i型跨膜蛋白,胞外有大约300个氨基酸,包括一个约200个氨基酸组成的vwa结构域和近膜的一个功能未知的ig样结构域。后来研究确定了tem8的三个剪接变体v1,v2和v3。该变体分别由564,368和333个氨基酸残基组成。其中v1和v2是具有单个跨膜螺旋的i型整合膜蛋白。由于拼接的差异性,v2与v1在前364个氨基酸序列相同,但v2的最后4个残基是唯一的。v3的胞外部分与v1和v2的胞外部分相同,但其在羧基末端拥有13个独特的残基。{kennetha.bradley}等人使用cdna文库进行tem8表达,确定v2变体为炭疽毒素受体,除v3外,v1也是一种功能炭疽毒素受体。
tem8不仅是功能炭疽毒素受体,而且tem8胞质结构域可以将细胞外基质分子连接到肌动蛋白细胞骨架从而调整其在胞外的结构,tem8还可以与胶原蛋白i和胶原蛋白vi等胶原蛋白结合,从而促进内皮细胞的迁移。在研究肿瘤血管形成相关基因的表达时,发现tem8在人的各种肿瘤血管细胞表面或者肿瘤细胞表面的表达量均有上调。另外tem8还具有高度保守性,小鼠与人类的tem8的氨基酸存在98%的同源性,且在小鼠肿瘤血管中也存在tem8过表达的现象。在敲除tem8的小鼠模型中发现,小鼠发育性的血管生成未受影响而肿瘤血管生成却受到了明显抑制。起初发现tem8表达于结肠癌内皮细胞中,而在周围正常组织中表达微乎其微,随后在结肠癌组织的血管中也检测到tem8蛋白,后来tem8还被发现在许多不同类型的人类肿瘤(包括膀胱癌、结肠癌、食管癌和肺癌等)的血管内皮细胞中表达增加,而在正常组织的血管中没有表达或表达很少。研究还表明tem8促进肿瘤血管生成而不参与生理性血管生成。
基于早期的机理研究,tem8开始被应用于抑制肿瘤药物的研发中。研究表明,可溶tem8-fc融合蛋白对多种人类异种移植肿瘤具有有效的杀伤活性(hai-fengduanetal.jnatlcancerinst2007;99:1551-5)。而针对tem8的dna疫苗已经被证明减缓了体内肿瘤的生长。另外,tem8单克隆抗体一直是tem8靶点抑制肿瘤药物研究的重点。在tem8抗体的研究过程中发现无论是市售的tem8鼠源性抗体还是miyoungyang等研究出的sb系列的抗体,都不能识别tem8表达在细胞表面的某些形态,这是由于微丝骨架可以调节细胞膜表面的tem8结构形态。
{amitchaudhary}等人利用噬菌体展示技术(amitchaudharyetal.cancercell21,212–226.2012),得到了5个独立的fab片段,分别命名为l1、l2、l3、l5和id2,这5个抗体均能识别tem8的各种形态。考虑到fab在体内半衰期短的问题,于是将l2的fab片段连接到恒定区段构成完整lgg抗体。在对l2的研究过程中发现l2能够在不对生理血管生成造成影响的情况下抑制肿瘤生长,并且对于包括黑素瘤,结肠癌和肺癌等癌症均有抑制效果,对其他组织并无明显毒性。另外,l2还被开发成car-t药物,并在动物模型实验中显示出很强的抑制肿瘤生长的活性。
以上的研究表明tem8可以成为治疗肿瘤的新靶点。虽然对tem8的研究已经十多年,也取得了一定的研究成果,但是想要将这些成果转化为生产上市的药品还需要经过很多努力。
技术实现要素:
本发明为了解决上述背景技术中的技术问题,提供一种能够转化为生产上市的tem8及其应用。
11.本发明解决上述技术问题的技术方案如下:一种分离的单克隆抗体或其抗原结合部分,所述分离的单克隆抗体或其抗原结合部分与tem8特异性结合,所述分离的单克隆抗体或其抗原结合部分包括第一组cdr区、第二组cdr区或第三组cdr区中的一组;
其中,所述第一组cdr区为氨基酸序列如seqidno:1所示的vh互补决定区1(vhcdr1)序列、氨基酸序列如seqidno:2所示的vh互补决定区2(vhcdr2)序列、氨基酸序列如seqidno:3所示的vh互补决定区3(vhcdr3)序列、氨基酸序列如seqidno:4所示的vl互补决定区1(vlcdr1)序列、氨基酸序列如seqidno:5所示的vl互补决定区2(vlcdr2)序列和氨基酸序列如seqidno:6所示的vl互补决定区3(vlcdr3)序列;
其中,所述第二组cdr区为氨基酸序列如seqidno:1所示的vh互补决定区1(vhcdr1)序列、氨基酸序列如seqidno:2所示的vh互补决定区2(vhcdr2)序列、氨基酸序列如seqidno:7所示的vh互补决定区3(vhcdr3)序列、氨基酸序列如seqidno:4所示的vl互补决定区1(vlcdr1)序列、氨基酸序列如seqidno:5所示的vl互补决定区2(vlcdr2)序列和氨基酸序列如seqidno:6所示的vl互补决定区3(vlcdr3)序列;
其中,所述第三组cdr区为氨基酸序列如seqidno:1所示的vh互补决定区1(vhcdr1)序列、氨基酸序列如seqidno:2所示的vh互补决定区2(vhcdr2)序列、氨基酸序列如seqidno:8所示的vh互补决定区3(vhcdr3)序列、氨基酸序列如seqidno:4所示的vl互补决定区1(vlcdr1)序列、氨基酸序列如seqidno:5所示的vl互补决定区2(vlcdr2)序列和氨基酸序列如seqidno:6所示的vl互补决定区3(vlcdr3)序列。
本发明的有益效果是:本发明的特异性结合tem8的分离单克隆抗体或其抗原展现出下述性质:
1.与重组人tem8蛋白结合;
2.与tem8过表达细胞特异性结合;
3.刺激tem8过表达细胞对抗体的内吞;
4.抑制裸小鼠移植瘤的生长。
在上述技术方案的基础上,本发明还可以做如下改进。
进一步,所述分离的单克隆抗体或其抗原结合部分还包括第一重链可变区和第一轻链可变区;
其中,所述第一重链可变区来自氨基酸序列如seqidno:9的重链可变区或氨基酸序列如seqidno:11的重链可变区或氨基酸序列如seqidno:12的重链可变区中的一种;
所述第一轻链可变区的氨基酸序列如seqidno:10的轻链可变区。
进一步,所述分离的单克隆抗体或其抗原结合部分还包括第二重链可变区和第二轻链可变区;
所述第二重链可变区与所述第一重链可变区和所述第二轻链可变区与所述第一轻链可变区均具有90%~100%的整体序列同一性。
采用上述进一步方案的有益效果是,具有90~100%的整体氨基酸序列同一性是指指序列同一性为91%或92%或93%或94%或95%或96%或97%或98%或99%或100%。
进一步,所述分离的单克隆抗体或其抗原结合部分还包括一条重链和一条轻链,所述重链的氨基酸序列如seqidno:19所示,所述轻链的氨基酸序列如seqidno:18所示。
本发明还提供一种抗体药物偶联物,包括上述所述的单克隆抗体或其抗原结合部分。
本发明的有益效果是,本发明的抗体药物偶联物展现出下述性质:
1.与重组人tem8蛋白结合;
2.与tem8过表达细胞特异性结合;
3.直接杀伤tem8过表达的细胞或肿瘤细胞;
4.抑制裸小鼠移植瘤的生长。
在上述技术方案的基础上,本发明还可以做如下改进。
进一步,所述抗体药物偶联物还包括接头和细胞毒剂,其结构形式为抗体-(接头-细胞毒剂)。
采用上述进一步方案的有益效果是,本发明所述tem8抗体药物偶联物分子式如下,ab表示tem8抗体,括号内是接头和细胞毒剂分子式:
进一步,所述接头为vc,所述vc为胞内蛋白酶切割的肽酰接头。
进一步,所述细胞毒剂为单甲基澳瑞他汀e,所述单甲基澳瑞他汀e简称mmae,所述mmae的分子式如下,其中波浪线表示共价连接至所述抗体:
本发明还提供一种上述所述的抗体药物偶联物在治疗疾病领域的应用,其特征在于,所述抗体药物偶联物用于治疗癌症或感染性疾病药物中的一种或两种;
所述癌症包括肺癌、结肠癌、黑色素瘤、肾癌、膀胱癌、乳腺癌、肝癌、淋巴瘤、恶性血液病、胃癌、宫颈癌、前列腺癌、卵巢癌和胰腺癌;
所述感染性疾病包括炭疽杆菌,枯草芽胞杆菌、蜡样芽孢杆菌和多粘杆菌。
本发明的有益效果是,所述的癌症为包括但不限于肺癌、结肠癌、黑色素瘤、肾癌、膀胱癌、乳腺癌、肝癌、淋巴瘤、恶性血液病、胃癌、宫颈癌、前列腺癌、卵巢癌、胰腺癌;所述感染性疾病包括但不限于炭疽杆菌,枯草芽胞杆菌、蜡样芽孢杆菌、多粘杆菌等。本发明tem8抗体的重链和轻链,以及tem8抗体或其抗体药物偶联物还可以用于与tem8相关的科学研究,如发育生物学、细胞生物学、代谢、结构生物学、功能基因组学等多个领域的科学研究、或肿瘤、自身免疫性疾病等医学和药学的应用研究。
本发明还提供了一种药物组合物,包括上述所述的分离的单克隆抗体或其抗原结合部分和上述所述的抗体药物偶联物中的一种或两种。
本发明的有益效果是,本发明的药物组合物能够有效的治疗癌症和感染性疾病。本发明所述tem8抗体可以为单链抗体、双链抗体、嵌合抗体、人源化抗体、以及前述抗体的衍生物、功能等同物和同源物,也包括抗体片段和含有抗原结合结构域的任何多肽。
进一步,所述药物组合物中还含有药学上可接受的载体和/或稀释剂。
本发明还可以是一种试剂、试剂盒或芯片,包含前述的tem8抗体或其抗体药物偶联物.
本发明的抗体可用基因工程技术制得,因为编码本发明人源化抗体的dna序列可用本领域技术人员熟知的常规手段,如根据本发明公开的氨基酸序列人工合成或用pcr法扩增得到,因而也可用重组dna方法,可用本领域熟知的各种方法将该序列连入合适的表达载体中。
本发明中,术语“抗体”包括完整抗体及其任何抗原结合片段(即“抗原结合部分”)或单链。“抗体”是指包含通过二硫键互相连接在一起的至少两条重(h)链和两条轻(l)链的糖蛋白,或其抗原结合部分。每条重链由重链可变区(vh)和重链恒定区组成。重链恒定区由三个结构域ch1,ch2和ch3组成。每条轻链由轻链可变区(vl)和轻链恒定区组成.链恒定区由一个结构域cl组成。vh和vl区可进一步分为高变区,称为互补决定区(cdr),cdr散布在被称为构架区(fr)的更加保守的区域中。每个vh和vl均由三个cdr和四个fr组成,它们从氨基端向羧基端以如下顺序排列:fr1-cdr1-fr2-cdr2-fr3-cdr3-fr4。重链和轻链的可变区含有可与抗原相互作用的结合结构域。抗体的恒定区可以介导免疫球蛋白与宿主组织或因子的结合,该宿主组织或因子包括免疫系统的各种细胞(例如效应细胞)和经典补体系统的第一成分(c1q)。构架区和互补决定区的范围已被精确定义(参见"sequencesofproteinsofimmunologicalinterest,"e.kabat等,u.s.departmentofhealthandhumanservices,1991)。此处所讨论的所有抗体氨基酸序列的排序都参照kabat系统。术语“抗体”不受任何特定的产生抗体的方法限制,例如,其包括,特别地,重组抗体、单克隆抗体和多克隆抗体。
本发明中,术语“抗原结合部分”是指保留与抗原特异性结合的能力的抗体的一个或多个片段。已证明抗体的抗原结合功能可由全长抗体的片段来行使。术语“抗原结合部分”中所包括的结合片段的例子包括:(1)fab片段,即由vl、vh、cl和ch1结构域组成的单价片段;(2)f(ab’)2片段,即包含在铰链区处通过二硫键连接的两个fab片段的双价片段;(3)由vh和ch1结构域组成的fd片段;(4)由抗体单臂的vl和vh结构域组成的fv片段;(5)由vh结构域组成的dab片段(ward等(1989)nature341:544-546);和(6)分离的互补决定区(cdr)。
本发明中,术语“同种型”是指由重链恒定区基因编码的抗体类别(例如igm或igg1)。
术语“人源化抗体”是指其中来源于另外一种哺乳动物种如小鼠的种系的cdr序列被移植到人构架序列上的抗体。在人构架序列内也可以进行其它的修饰。
术语“嵌合抗体”是其重链和轻链基因经过构建的抗体,特别是利用基因工程改造的属于不同物种的抗体可变区和恒定区基因。例如,可以将鼠单克隆抗体基因的可变区片段连接到人抗体恒定区片段如γ1和γ3。当然,嵌合抗体的基因来源也可以使用其它哺乳动物物种。
本发明中“kd”是指解离常数,它是由kd和ka的比值获得(即kd/ka),并且表示为摩尔浓度(m).抗体的kd值可用本领域建立的方法测定。测定kd的一种优选方法是使用表面等离子共振,优选使用生物传感器系统,如biacore系统。
抗体药物偶联物(adc),是指偶联细胞毒剂(如化疗剂,药物、生长抑制剂、毒素(如细菌、真菌、植物或动物来源的酶活性毒素或其片段)或放射性同位素(即放射性偶联物))的抗体。
“细胞毒剂”指抑制或阻止细胞的表达活性、细胞功能,和/或导致细胞破坏的物质。该术语旨在包括放射性同位素、化疗剂和毒素,如细菌、真菌、植物或动物来源的小分子毒素或酶活性毒素,包括其片段和/或变体。细胞毒剂的示例包括但不限于:澳瑞他汀(例如澳瑞他汀e、澳瑞他汀f、mmae和mmaf)、金霉素、美登木素(maytansinoid)、蓖麻毒蛋白、蓖麻毒蛋白a链、考布他汀(combrestatin)、多卡米星、尾海兔素、多柔比星、柔红霉素、紫杉酚、顺铂、cc1065、溴化乙锭、丝裂霉素、依托泊苷、替尼泊苷、长春新碱、长春花碱、秋水仙碱、二羟基蒽二酮、放线菌素、白喉毒素、假单胞菌外毒素(pe)a、pe40、相思豆毒蛋白、相思豆毒蛋白a链、蒴莲根毒素a链、á八叠球菌、白树毒素、迈托毒素、限曲菌素、酚霉素、依诺霉素、麻疯树毒蛋白、抗巴豆毒素、卡奇霉素、肥皂草(sapaonariaofficinalis)抑制剂和糖皮质激素以及其它的化疗剂和放射性同位素例如at211、i131、i125和包括lu177在内的lu的放射性同位素。抗体也可与抗癌前药的活化酶偶联,该酶能将前药转变成其活性形式。
“接头”是指可用于连接药物单元和抗体单元以形成抗体-药物偶联化合物的双功能化合物。包括但不限于胞内蛋白酶切割的肽酰接头(例如val-cit接头或phe-lys接头)、ph敏感型接头(腙、缩氨基脲、缩氨基硫脲等)、二硫键接头、丙二酸接头、马来酰亚胺苯甲酰接头等。
附图说明
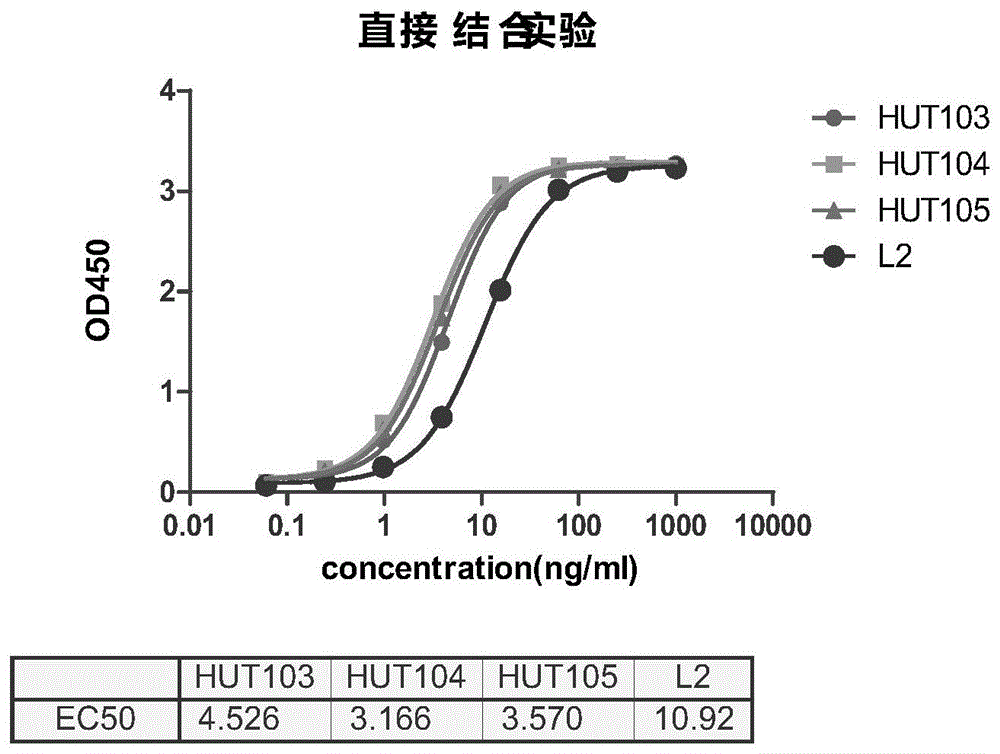
图1为本发明实施例2中tem8抗体与tem8重组蛋白结合活性的检测结果图;
图2为本发明实施例2中tem8抗体与tem8过表达细胞结合活性的检测结果图;
图3为本发明实施例3中tem8抗体的细胞内吞活性的hut104内吞效果检测结果图;
图4为本发明实施例3中tem8抗体的细胞内吞活性的l2内吞效果检测结果图;
图5为本发明实施例4中sds-page电泳分析抗体与mmae偶联效果的实验结果图;
图6为本发明实施例5中tem8抗体药物偶联物与tem8重组蛋白结合活性的检测结果;
图7为本发明实施例5中tem8抗体药物偶联物与tem8过表达细胞结合活性的检测结果;
图8为本发明实施例6的tem8抗体及其药物偶联物抑制肿瘤细胞生长的检测结果;
图9为本发明实施例7的tem8抗体药物偶联物作用于细胞周期的检测结果;
图10为本发明实施例8的tem8抗体及其药物偶联物治疗体内肿瘤的药效结果。
具体实施方式
以下结合附图对本发明的原理和特征进行描述,所举实例只用于解释本发明,并非用于限定本发明的范围。
实施例1、
本实施例通过杂交瘤细胞技术制备鼠源特异性结合tem8单克隆抗体,有关实验方案参见文献(edharlow,davidlane.antibody:alaboratorymanual.1988),具体按照以下步骤进行:
1.免疫原准备:合成tem8全长基因。并构建到pcdna3.1(invitrogen)表达载体,将tem8表达载体转染进入cho-k1(atccno.ccl-60)细胞株,经过g418(gibco)加压筛选,获得tem8稳定过表达细胞株;
2.动物免疫:分别在第1天,第14天和第35天,将1×107个tem8稳定表达细胞通过腹腔注射,对balb/c小鼠进行免疫,在第56天用1×106个细胞对balb/c小鼠静脉注射进行最终免疫,4天后取脾脏进行融合;
3.杂交瘤融合:将鼠脾细胞与sp2/0细胞(atccno.crl-1581)按数量比5:1比例融合,在96孔板(corning)中以hat(gbico)培养基培养,然后进行杂交瘤细胞筛选,所得细胞克隆进行鉴定筛选;
4.杂交瘤筛选:
将重组tem8蛋白(义翘神州)固定化于96孔酶联免疫吸附板(costar)上,加入克隆表达上清孵育1h后,用pbst洗涤3次,使用羊抗鼠igg抗体-hrp(jacksonimmuno)鉴定具有tem8结合活性细胞克隆上清,从而获得可与tem8直接结合的阳性克隆;
随后将上述中的阳性克隆转移入24孔板(corning)培养,以获得更多的表达产物,将tem8稳定过表达细胞株接种于96孔板(corning)上过夜培养,加入阳性克隆表达上清共同孵育1h,再用pbs洗涤3次,使用eu标记的羊抗鼠igg(perkinelmer)进行检测,从而鉴别出能够结合细胞表面tem8的阳性克隆;
5.提取tem8鼠源抗体的抗体基因:将所筛选的阳性克隆的杂交瘤细胞裂解,提取rna后逆转录得cdna,以此cdna为模板,采用pcr方法分别扩增出鼠igg抗体的轻链和重链可变区核酸序列,对重链可变区、轻链可变区进行测序,其编码的重链可变区序列如seqidno.15所示、轻链可变区如seqidno.16所示。
实施例2、
人源化和亲和力成熟实验
本实施例是将实施例1中获取的鼠源特异性结合tem8单克隆抗体进行人源化,具体按照以下步骤进行:
1.人源化设计
分析鼠源抗体序列,与imgt的人胚系(germline)基因比对,最终确定igkv3-15*01为轻链fr1和fr2的人源化提供框架序列,igkv3-11*01和igkj4*01分别为轻链fr3和fr4的人源化提供框架序列,确定ighv4-59*06,ighv1-3*01,ighv6-1*02和ighj6*01分别为重链fr1,fr2,fr3和fr4的人源化提供框架序列,通过cdr-grafting,将重链和轻链的cdr并置到构架序列,同时,对重链构架区上的关键位点进行回复突变设计,包括q1e,g27d和p71r,构建人源化抗体,称为hut101,其重链可变区氨基酸序列如seqidno.13所示,轻链可变区如seqidno.10所示。
2.亲和力成熟
对hut101抗体进行亲和力成熟筛选,基因合成hut101抗体的scfv片段,dna序列如seqidno.21所示,氨基酸如seqidno.14所示,并将scfv连接到pcomb3噬菌体质粒中,设计引物序列,将重链cdr3的氨基酸dssf所在的引物位置设计为nns,使之编码任意的氨基酸,以hut101抗体scfv为模板,pcr扩增scfv抗体库,将scfv的抗体库,通过sfii酶切位点,构建到pcomb3噬菌体质粒中,构建抗体库,然后通过噬菌体展示进行高亲和力抗体筛选,具体步骤如下:
①通过电转化,将含scfv的抗体库的噬菌体质粒转化到tg1中,经过37度,250rpm,反应1h的恢复后,将helperphage加入到菌液中,另加入ampicillin,37度,250rpm,反应1h,接着在2500rpmx5min条件离心去掉上清液,剩下的用2xyt-ak吹悬菌泥,37度,250rpm过夜培养;②包被抗原:用包被缓冲液稀释tem8蛋白,混匀加入到免疫管中,4度包被保存10h;③重组噬菌体收集:上述过夜培养菌液,2500rpmx5min离心,收集上清10ml,加入2ml的peg/nacl,混匀放置冰上1h,10000g×20min离心,去上清,用pbs溶解噬菌体库;④封闭:免疫管用pbs洗两次,加入封闭液,室温1h;另外,取等体积封闭液与噬菌体库混合,室温封闭10-15min;⑤孵育噬菌体库:免疫管用pbs洗2次,加入封闭好的噬菌体库,37度培养箱2-3h;⑥洗脱:取100ultg1菌液(前一天接种)到10ml2xyt中,37度,250rpm培养到a600值0.4-0.5。用pbst洗涤免疫管8次,再用pbs洗2次,加入5ml对数期生长的菌液,37度,250rpm,1h;⑦稀释上述菌液10倍和100倍分别取100ul涂平板。⑧下-轮筛选:取200ulhelperphage加入到5ml洗脱后的菌液中,同时加入5ulampicillin,37度,250rpm,1h,2500rpmx5min离心去上清,用10ml的2xyt-ak吹悬菌泥,37度,250rpm过夜培养,重复步骤①-⑦。
经过三轮筛选,挑选单克隆抗体,通过噬菌体elisa方法,检测phage活性,具体如下:包被tem8蛋白,0.5ug/ml,4度过夜;pbst洗两次,加入50ulphage上清和50ul1%bsa,37℃,1h;pbst洗三次,加入100ul1:10000稀释的anti-m13-ab-hrp(义翘神州),在37℃下反应1h;④pbst洗三次,加入100ul预热的tmb,室温下处理10min;加入50ul的1mol的h2so4中止反应,od450检测吸光值。
挑选阳性单克隆抗体,通过噬菌体与tem8过表达细胞结合实验,检测单克隆抗体的细胞结合活性,具体如下:用胰酶消化tem8过表达细胞,200gx5min,用质量分数为1%的bsa重悬细胞,室温,1h;封闭phage:50ul的phage上清和50ul的质量分数为1%的bsa,室温,1h;细胞铺板,100ul/孔,2500rpmx5min;弃上清,用封闭好的phage重悬细胞,100ul/well,室温,1h;细胞pbs洗两次,加入100ul1:10000稀释的anti-m13-ab-hrp,室温,1h;细胞pbst洗两次,加入100ul预热的tmb,室温,10min;加入50ul的1mol的h2so4中止反应,od450检测吸光值。
挑选细胞结合阳性的克隆进行测序分析,共获得4个序列不同的抗体,分别命名为hut102,hut103,hut104和hut105.其中hut103的重链可变区氨基酸序列如seqidno.9所示,轻链可变区如seqidno.10所示,hut104的重链可变区氨基酸序列如seqidno.11所示,轻链可变区如seqidno.10所示,hut105的重链可变区氨基酸序列如seqidno.12所示,轻链可变区如seqidno.10所示。
3.亲和力成熟抗体表达载体构建及抗体制备
构建hut103,hut104和hut105的全长抗体:通过pcr法,将抗体的重链可变区dna片段和重链igg1的恒定区连接,获得全长重链,将轻链可变区dna片段分别与和轻链k链的恒定区连接,获得全长轻链。hut103的重链氨基酸序列和轻链氨基酸序列如seqidno.17和seqidno.18所示,hut103的重链全长dna序列和轻链dna序列如seqidno.22和seqidno.23所示;hut104的重链氨基酸序列和轻链氨基酸序列如seqidno.19和seqidno.18所示,hut104的重链全长dna序列和轻链dna序列如seqidno.24和seqidno.23所示;hut105的重链氨基酸序列和轻链氨基酸序列如seqidno.20和seqidno.18所示,hut105的重链全长dna序列和轻链dna序列如seqidno.25和seqidno.23所示。全长重链和轻链dna片段,均包含信号肽,分别克隆入pcdna3.1(invitrogen)质粒。
将hut103,hut104和hut105表达质粒瞬转293c18细胞株(atccno.crl-10852),用proteina(ge)纯化上清,获取纯化抗体。通过elisa方法,检测hut103,hut104,hut105以及对照抗体l2(序列来自于专利wo2012/172495a1,通过基因合成和瞬转293细胞表达获得)和tem8重组蛋白与tem8过表细胞的结合活性。
4.重组tem8蛋白结合活性测定
将tem8蛋白包被于96孔板(costar)上,4度过夜,1%bsa封闭,37度孵育1小时,然后,纯化抗体以1ug/ml起始,4倍稀释,8浓度,100ul/孔,37度孵育1小时,pbs洗两次;接着加入鼠抗人igg抗体-hrp(jacksonimmuno),37度孵育1小时;最后,pbst洗三次,加入100ul预热的tmb,室温10min;加入50ul1mh2so4中止反应,od450检测吸光值。结果如图1所示,所筛选的单克隆抗体hut103,hut104与hut105和tem8蛋白的直接结合ec50分别为4.526ng/ml,3.166ng/ml3.57ng/ml,均优于对照抗体l2,其ec50为10.92ng/ml.
5.与tem8过表达细胞结合活性测定
将tem8过表达细胞铺板于黑色96孔板,培养过夜,质量分数为1%的bsa封闭,37度,孵育1小时,抗体2ug/ml起始,4倍稀释,8浓度,100ul/孔,25度孵育1小时,pbs洗一次,然后,加入anti-human-igg-eu(perkinelmer),50ng/ml,每孔100ul,25度反应0.5小时,pbs洗两次,最后,加入荧光增强液,激发光337nm/发射光620nm读数。结果显示如图2所示,hut103,hut104与hut105和tem8过表达细胞结合ec50分别为130.2ng/ml,105.5ng/ml和113ng/ml。均优于对照抗体l2(ec50为428.6ng/ml).
实施例3、
tem8抗体的内吞活性实验
为了验证tem8抗体与tem8过表细胞结合后,能在tem8介导下被细胞内吞。按照荧光标记试剂盒(thermo,alexafluor488proteinlabelingkit)说明书对hut104和对照抗体l2进行绿色荧光标记,然后,与过表达tem8细胞反应,30分钟后,去除上清,pbs清洗细胞。最后,通过荧光显微镜观察绿色荧光在细胞的分布情况。hut104内吞效果结果如图3所示,l2内吞效果结果如图4所示。经过pbs清洗细胞后,绿色荧光分布细胞内部,说明tem8抗体已经被tem8细胞内吞。
实施例4、
tem8抗体药物偶联物的制备
用适量浓度的还原剂将tem8抗体hut104还原,暴露出抗体hut104链间的巯基,然后加入vcmmae(包含vc接头和细胞毒素mmae)进行反应,通过hut104上巯基与接头上的马来酰亚胺基团发生加成反应,从而将抗体和细胞毒素mmae偶联在一起。将偶联后的混合物进行脱盐处理,除去残留的小分子毒素,然后利用sds-page电泳进行分析。结果如图5所示,hut104与细胞毒素mmae偶联后,hut104-adc的分子量略大于未标记hut104抗体,说明偶联成功。
实施例5、
抗体药物偶联物与tem8的结合活性
通过elisa实验,验证tem8抗体偶联药物后与tem8蛋白的结合活性,方法同实施例2。结果如图6所示,偶联药物并没有影响抗体与tem8蛋白的结合活性,hut104-adc与hut104与tem8蛋白的结合ec50分别为2.73ng/ml和1.764ng/ml。
通过转染tem8表达质粒到hct116细胞,构建tem8过表达的hct116/tem8细胞。检测hut104-adc与hct116/tem8细胞的结合活性。cell-basedelisa结果如图7所示,hut104-adc与hct116/tem8结合活性,与hut104的活性相当,ec50值分别为153.2ng/ml和128.2ng/ml。
实施例6、
tem8抗体药物偶联物对肿瘤细胞的抑制作用
胰酶消化处于对数生长期的hct116、hct116/tem8,分别以7000个/孔铺96孔板,于37℃的条件下充满体积分数为5%的co2恒温培养箱中孵育1h。用细胞培养基稀释hut104-adc和hut104,40ug/ml作为起始浓度,6倍稀释,共8个梯度。加入等体积的稀释样品到细胞板内,混匀后置于37℃的条件下质量分数为5%的co2恒温培养箱中孵育42h。加入100ul
实施例7、
tem8抗体药物偶联物对细胞周期的影响
取hut104-adc和hut104分别加入到hct116/tem8细胞,置于37℃条件下体积分数为5%的co2恒温培养箱中孵育24h。胰酶消化细胞,培养基终止消化,1200rpm离心5min,弃上清,并用预冷pbs洗涤2次,最用250ul预冷pbs重悬细胞,使细胞分散。将细胞逐滴缓慢滴入750ul预冷乙醇中,轻轻吹打混匀,4℃放置过夜。然后1200rpm离心5min,弃上清,加入预冷pbs洗涤细胞,离心弃上清,200ul预冷pbs重悬。加入50ug/mlrnase溶液,37℃水浴30min以去除rna。1200rpm离心5min,弃上清,用1×assaybuffer吹匀,加入5ulpi染料,避光冰浴30min,吹匀后上流式细胞仪检测,结果用flowjo进行分析。结果如图9所示,hut104-adc处理后的细胞,由于在毒素细胞内的释放,从而将细胞抑制在g2/m期,使细胞无法正常分裂。
实施例8、
体内动物实验
采用hct116/tem8细胞皮下balb/c裸小鼠模型,检查tem8抗体及tem8抗体药物偶联物对肿瘤生长的体内效果。收集对数生长期的hct116/tem8细胞,去除培养液并用pbs洗两次后接种到balb/c裸小鼠,接种量为2*10^6/100ul/只(不添加基质胶)。待6天后肿瘤长出,将肿瘤体积达到标准的小鼠随机分为4组,分别为对照组(pbs),阳性药物(l2inpbs),hut104组(hut104inpbs)和hut104-adc组(hut104-adcinpbs),分组后第二天开始给药,给药周期为q2dx8,给药方式为i.p,给药剂量为3mg/kg。给药当日测量小鼠体重和肿瘤体积。每次给药时用游标卡尺测量小鼠肿瘤大小,体积计算公式为v=(l×w^2)/2,其中l为肿瘤最长直径,w为垂直于最长直径的短径。给药第8次后,实验终止,对全部小鼠执行安乐死。结果如图10所示,hut104-adc抑制小鼠体内肿瘤生长的活性显著优于其它组,hut104组和阳性对照l2组也显示出一定抑制肿瘤生长的活性。最终数据表明,hut104-adc组的肿瘤体积只有117.3±111.2mm3,明显小于对照组的1036.4±278.1mm3、hut104组的727.5±298.3mm3和l2组的665.5±112.58mm3,说明adc的具有强大的抑制肿瘤的活性。
序列表
seqidno:1sdywn
seqidno:2yisysgntytnpslks
seqidno:3yktitsvldv
seqidno:4rasqnirdflh
seqidno:5yasqsis
seqidno:6qnghsfpyt
seqidno:7yktitsafdv
seqidno:8yktitsmfdv
seqidno:9
evqlqesgpglvkpsetlsltctvtgdsissdywnwvrqapgqrlewmgyisysgntytnpslksritinrdtsknqfslqlnsvtpedtavyycaryktitsvldvwgqgttvtvss
seqidno:10
eivmtqspatlsvspgeratlscrasqnirdflhwyqqkpgqaprlliyyasqsisgiparfsgsgsgtdftltisslepedfavyycqnghsfpytfgggtkveik
seqidno:11
evqlqesgpglvkpsetlsltctvtgdsissdywnwvrqapgqrlewmgyisysgntytnpslksritinrdtsknqfslqlnsvtpedtavyycaryktitsafdvwgqgttvtvss
seqidno:12
evqlqesgpglvkpsetlsltctvtgdsissdywnwvrqapgqrlewmgyisysgntytnpslksritinrdtsknqfslqlnsvtpedtavyycaryktitsmfdvwgqgttvtvss
seqidno:13
evqlqesgpglvkpsetlsltctvtgdsissdywnwvrqapgqrlewmgyisysgntytnpslksritinrdtsknqfslqlnsvtpedtavyycaryktidssfdvwgqgttvtvss
seqidno:14
evqlqesgpglvkpsetlsltctvtgdsissdywnwvrqapgqrlewmgyisysgntytnpslksritinrdtsknqfslqlnsvtpedtavyycaryktidssfdvwgqgttvtvssggggsggggsggggseivmtqspatlsvspgeratlscrasqnirdflhwyqqkpgqaprlliyyasqsisgiparfsgsgsgtdftltisslepedfavyycqnghsfpytfgggtkveikr
seqidno:15
evqlqesgpslvkpsqtlsltcsvtgdsitsdywnwirkfpgnkleymgyisysgntytnpslksrisitrdtsknqyylqlnsmttedtatyycaryktidssfdvwgagttvtvss
seqidno:16
divmtqspatlsvtpgdrvslscrasqnirdflhwyqqkshesprllikyasqsisgipsrfsgsgsgsdfslninsvepedvgvyycqnghsfpytfgggtkleik
seqidno:17
evqlqesgpglvkpsetlsltctvtgdsissdywnwvrqapgqrlewmgyisysgntytnpslksritinrdtsknqfslqlnsvtpedtavyycaryktitsvldvwgqgttvtvssastkgpsvfplapsskstsggtaalgclvkdyfpepvtvswnsgaltsgvhtfpavlqssglyslssvvtvpssslgtqtyicnvnhkpsntkvdkkvepkscdkthtcppcpapellggpsvflfppkpkdtlmisrtpevtcvvvdvshedpevkfnwyvdgvevhnaktkpreeqynstyrvvsvltvlhqdwlngkeykckvsnkalpapiektiskakgqprepqvytlppsreemtknqvsltclvkgfypsdiavewesngqpennykttppvldsdgsfflyskltvdksrwqqgnvfscsvmhealhnhytqkslslspgk-
seqidno:18
eivmtqspatlsvspgeratlscrasqnirdflhwyqqkpgqaprlliyyasqsisgiparfsgsgsgtdftltisslepedfavyycqnghsfpytfgggtkveikrtvaapsvfifppsdeqlksgtasvvcllnnfypreakvqwkvdnalqsgnsqesvteqdskdstyslsstltlskadyekhkvyacevthqglsspvtksfnrgec-
seqidno:19
evqlqesgpglvkpsetlsltctvtgdsissdywnwvrqapgqrlewmgyisysgntytnpslksritinrdtsknqfslqlnsvtpedtavyycaryktitsafdvwgqgttvtvssastkgpsvfplapsskstsggtaalgclvkdyfpepvtvswnsgaltsgvhtfpavlqssglyslssvvtvpssslgtqtyicnvnhkpsntkvdkkvepkscdkthtcppcpapellggpsvflfppkpkdtlmisrtpevtcvvvdvshedpevkfnwyvdgvevhnaktkpreeqynstyrvvsvltvlhqdwlngkeykckvsnkalpapiektiskakgqprepqvytlppsreemtknqvsltclvkgfypsdiavewesngqpennykttppvldsdgsfflyskltvdksrwqqgnvfscsvmhealhnhytqkslslspgk-
seqidno:20
evqlqesgpglvkpsetlsltctvtgdsissdywnwvrqapgqrlewmgyisysgntytnpslksritinrdtsknqfslqlnsvtpedtavyycaryktitsmfdvwgqgttvtvssastkgpsvfplapsskstsggtaalgclvkdyfpepvtvswnsgaltsgvhtfpavlqssglyslssvvtvpssslgtqtyicnvnhkpsntkvdkkvepkscdkthtcppcpapellggpsvflfppkpkdtlmisrtpevtcvvvdvshedpevkfnwyvdgvevhnaktkpreeqynstyrvvsvltvlhqdwlngkeykckvsnkalpapiektiskakgqprepqvytlppsreemtknqvsltclvkgfypsdiavewesngqpennykttppvldsdgsfflyskltvdksrwqqgnvfscsvmhealhnhytqkslslspgk-
seqidno:21
gaggtgcaactgcaggagtccggacctggcctggtgaagccctccgagaccctgagcctgacctgcaccgtgacaggcgactccatttccagcgattactggaactgggtgaggcaggctcctggacagaggctcgagtggatgggctacatctcctactccggcaatacctacaccaacccctccctgaagagccggatcaccatcaacagggacacctccaagaaccagttcagcctgcagctgaactccgtgacccccgaggacaccgccgtgtactactgcgccaggtacaagaccatcgacagcagcttcgacgtgtggggccagggcaccaccgtgacagtgtcctccggaggcggcggaagcggcggcggcggctccggaggcggcggcagcgaaattgtgatgacccagagccctgctaccctgagcgtgtcccctggagagcgggctaccctgtcctgtagggccagccagaacatcagggacttcctgcactggtatcaacagaagcctggacaggcccccaggctgctgatctactatgccagccagagcatctccggcattcccgctaggttctccggcagcggctccggcacagacttcaccctgaccatctccagcctggagcccgaagacttcgccgtgtattactgccagaacggccactcctttccctataccttcggcggcggcaccaaggtggagatcaagagg
seqidno:22
gaggtgcaactgcaggagtccggacctggcctggtgaagccctccgagaccctgagcctgacctgcaccgtgacaggcgactccatttccagcgattactggaactgggtgaggcaggctcctggacagaggctcgagtggatgggctacatctcctactccggcaatacctacaccaacccctccctgaagagccggatcaccatcaacagggacacctccaagaaccagttcagcctgcagctgaactccgtgacccccgaggacaccgccgtgtactactgcgccaggtacaagaccatcacctcggtgttggacgtgtggggccagggcaccaccgtgacagtgtcctccgccagcaccaagggaccatccgtgttcccactggctccaagctctaaatccactagcggaggcaccgcagccctgggatgtctggtgaaggattacttcccagagcccgtcacagtgtcatggaactccggggctctgacctctggtgtccacacatttccagcagtgctgcagagttcaggcctgtactccctgtccagcgtggtcacagtgccctctagttcactgggaactcagacctatatctgcaacgtgaatcacaagccatccaatactaaagtcgacaagaaagtggagcccaagagctgtgataaaacacatacttgccccccttgtcctgcaccagaactgctgggaggaccatccgtgttcctgtttccacccaagcctaaagacactctgatgatttctcgaacacccgaggtcacttgcgtggtcgtggacgtgtcccacgaggatcctgaagtcaagtttaactggtacgtggatggagtcgaagtgcataatgctaagacaaaacctagagaggaacagtacaacagtacatatagagtcgtgtcagtcctgactgtgctgcatcaggactggctgaacgggaaggagtataagtgcaaagtgtccaataaggctctgcccgcacctatcgagaaaactattagcaaggctaaaggccagcctagggaaccacaggtgtacaccctgcctccatctcgggaggaaatgactaagaaccaggtcagtctgacctgtctggtgaaaggcttctatccttccgacatcgcagtggagtgggaaagcaatggacagccagagaacaattacaagaccacaccccctgtgctggacagcgatgggtctttctttctgtatagtaagctgaccgtggataaatcacggtggcagcagggtaatgtcttttcttgtagtgtgatgcacgaagccctgcacaaccattacactcagaaatccctgtcactgtcccctggaaagtga
seqidno:23
gaaattgtgatgacccagagccctgctaccctgagcgtgtcccctggagagcgggctaccctgtcctgtagggccagccagaacatcagggacttcctgcactggtatcaacagaagcctggacaggcccccaggctgctgatctactatgccagccagagcatctccggcattcccgctaggttctccggcagcggctccggcacagacttcaccctgaccatctccagcctggagcccgaagacttcgccgtgtattactgccagaacggccactcctttccctataccttcggcggcggcaccaaggtggagatcaagaggactgtggctgcaccatctgtcttcatcttcccgccatctgatgagcagttgaaatctggaactgcctctgttgtgtgcctgctgaataacttctatcccagagaggccaaagtacagtggaaggtggataacgccctccaatcgggtaactcccaggagagtgtcacagagcaggacagcaaggacagcacctacagcctcagcagcaccctgacgctgagcaaagcagactacgagaaacacaaagtctacgcctgcgaagtcacccatcagggcctgagctcgcccgtcacaaagagcttcaacaggggagagtgttaa
seqidno:24
gaggtgcaactgcaggagtccggacctggcctggtgaagccctccgagaccctgagcctgacctgcaccgtgacaggcgactccatttccagcgattactggaactgggtgaggcaggctcctggacagaggctcgagtggatgggctacatctcctactccggcaatacctacaccaacccctccctgaagagccggatcaccatcaacagggacacctccaagaaccagttcagcctgcagctgaactccgtgacccccgaggacaccgccgtgtactactgcgccaggtacaagaccatcacctccgccttcgacgtgtggggccagggcaccaccgtgacagtgtcctccgccagcaccaagggaccatccgtgttcccactggctccaagctctaaatccactagcggaggcaccgcagccctgggatgtctggtgaaggattacttcccagagcccgtcacagtgtcatggaactccggggctctgacctctggtgtccacacatttccagcagtgctgcagagttcaggcctgtactccctgtccagcgtggtcacagtgccctctagttcactgggaactcagacctatatctgcaacgtgaatcacaagccatccaatactaaagtcgacaagaaagtggagcccaagagctgtgataaaacacatacttgccccccttgtcctgcaccagaactgctgggaggaccatccgtgttcctgtttccacccaagcctaaagacactctgatgatttctcgaacacccgaggtcacttgcgtggtcgtggacgtgtcccacgaggatcctgaagtcaagtttaactggtacgtggatggagtcgaagtgcataatgctaagacaaaacctagagaggaacagtacaacagtacatatagagtcgtgtcagtcctgactgtgctgcatcaggactggctgaacgggaaggagtataagtgcaaagtgtccaataaggctctgcccgcacctatcgagaaaactattagcaaggctaaaggccagcctagggaaccacaggtgtacaccctgcctccatctcgggaggaaatgactaagaaccaggtcagtctgacctgtctggtgaaaggcttctatccttccgacatcgcagtggagtgggaaagcaatggacagccagagaacaattacaagaccacaccccctgtgctggacagcgatgggtctttctttctgtatagtaagctgaccgtggataaatcacggtggcagcagggtaatgtcttttcttgtagtgtgatgcacgaagccctgcacaaccattacactcagaaatccctgtcactgtcccctggaaagtga
seqidno:25
gaggtgcaactgcaggagtccggacctggcctggtgaagccctccgagaccctgagcctgacctgcaccgtgacaggcgactccatttccagcgattactggaactgggtgaggcaggctcctggacagaggctcgagtggatgggctacatctcctactccggcaatacctacaccaacccctccctgaagagccggatcaccatcaacagggacacctccaagaaccagttcagcctgcagctgaactccgtgacccccgaggacaccgccgtgtactactgcgccaggtacaagaccatcacgagcatgttcgacgtgtggggccagggcaccaccgtgacagtgtcctccgccagcaccaagggaccatccgtgttcccactggctccaagctctaaatccactagcggaggcaccgcagccctgggatgtctggtgaaggattacttcccagagcccgtcacagtgtcatggaactccggggctctgacctctggtgtccacacatttccagcagtgctgcagagttcaggcctgtactccctgtccagcgtggtcacagtgccctctagttcactgggaactcagacctatatctgcaacgtgaatcacaagccatccaatactaaagtcgacaagaaagtggagcccaagagctgtgataaaacacatacttgccccccttgtcctgcaccagaactgctgggaggaccatccgtgttcctgtttccacccaagcctaaagacactctgatgatttctcgaacacccgaggtcacttgcgtggtcgtggacgtgtcccacgaggatcctgaagtcaagtttaactggtacgtggatggagtcgaagtgcataatgctaagacaaaacctagagaggaacagtacaacagtacatatagagtcgtgtcagtcctgactgtgctgcatcaggactggctgaacgggaaggagtataagtgcaaagtgtccaataaggctctgcccgcacctatcgagaaaactattagcaaggctaaaggccagcctagggaaccacaggtgtacaccctgcctccatctcgggaggaaatgactaagaaccaggtcagtctgacctgtctggtgaaaggcttctatccttccgacatcgcagtggagtgggaaagcaatggacagccagagaacaattacaagaccacaccccctgtgctggacagcgatgggtctttctttctgtatagtaagctgaccgtggataaatcacggtggcagcagggtaatgtcttttcttgtagtgtgatgcacgaagccctgcacaaccattacactcagaaatccctgtcactgtcccctggaaagtga
以上所述仅为本发明的较佳实施例,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
序列表
<110>江苏诺迈博生物医药科技有限公司
<120>tem8抗体及其应用
<141>2019-05-17
<160>25
<170>siposequencelisting1.0
<210>1
<211>5
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>1
serasptyrtrpasn
15
<210>2
<211>16
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>2
tyrilesertyrserglyasnthrtyrthrasnproserleulysser
151015
<210>3
<211>10
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>3
tyrlysthrilethrservalleuaspval
1510
<210>4
<211>11
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>4
argalaserglnasnileargasppheleuhis
1510
<210>5
<211>7
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>5
tyralaserglnserileser
15
<210>6
<211>9
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>6
glnasnglyhisserpheprotyrthr
15
<210>7
<211>10
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>7
tyrlysthrilethrseralapheaspval
1510
<210>8
<211>10
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>8
tyrlysthrilethrsermetpheaspval
1510
<210>9
<211>118
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>9
gluvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuthrcysthrvalthrglyaspserileserserasp
202530
tyrtrpasntrpvalargglnalaproglyglnargleuglutrpmet
354045
glytyrilesertyrserglyasnthrtyrthrasnproserleulys
505560
serargilethrileasnargaspthrserlysasnglnpheserleu
65707580
glnleuasnservalthrprogluaspthralavaltyrtyrcysala
859095
argtyrlysthrilethrservalleuaspvaltrpglyglnglythr
100105110
thrvalthrvalserser
115
<210>10
<211>107
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>10
gluilevalmetthrglnserproalathrleuservalserprogly
151015
gluargalathrleusercysargalaserglnasnileargaspphe
202530
leuhistrptyrglnglnlysproglyglnalaproargleuleuile
354045
tyrtyralaserglnserileserglyileproalaargphesergly
505560
serglyserglythraspphethrleuthrileserserleuglupro
65707580
gluaspphealavaltyrtyrcysglnasnglyhisserpheprotyr
859095
thrpheglyglyglythrlysvalgluilelys
100105
<210>11
<211>118
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>11
gluvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuthrcysthrvalthrglyaspserileserserasp
202530
tyrtrpasntrpvalargglnalaproglyglnargleuglutrpmet
354045
glytyrilesertyrserglyasnthrtyrthrasnproserleulys
505560
serargilethrileasnargaspthrserlysasnglnpheserleu
65707580
glnleuasnservalthrprogluaspthralavaltyrtyrcysala
859095
argtyrlysthrilethrseralapheaspvaltrpglyglnglythr
100105110
thrvalthrvalserser
115
<210>12
<211>118
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>12
gluvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuthrcysthrvalthrglyaspserileserserasp
202530
tyrtrpasntrpvalargglnalaproglyglnargleuglutrpmet
354045
glytyrilesertyrserglyasnthrtyrthrasnproserleulys
505560
serargilethrileasnargaspthrserlysasnglnpheserleu
65707580
glnleuasnservalthrprogluaspthralavaltyrtyrcysala
859095
argtyrlysthrilethrsermetpheaspvaltrpglyglnglythr
100105110
thrvalthrvalserser
115
<210>13
<211>118
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>13
gluvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuthrcysthrvalthrglyaspserileserserasp
202530
tyrtrpasntrpvalargglnalaproglyglnargleuglutrpmet
354045
glytyrilesertyrserglyasnthrtyrthrasnproserleulys
505560
serargilethrileasnargaspthrserlysasnglnpheserleu
65707580
glnleuasnservalthrprogluaspthralavaltyrtyrcysala
859095
argtyrlysthrileaspserserpheaspvaltrpglyglnglythr
100105110
thrvalthrvalserser
115
<210>14
<211>241
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>14
gluvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuthrcysthrvalthrglyaspserileserserasp
202530
tyrtrpasntrpvalargglnalaproglyglnargleuglutrpmet
354045
glytyrilesertyrserglyasnthrtyrthrasnproserleulys
505560
serargilethrileasnargaspthrserlysasnglnpheserleu
65707580
glnleuasnservalthrprogluaspthralavaltyrtyrcysala
859095
argtyrlysthrileaspserserpheaspvaltrpglyglnglythr
100105110
thrvalthrvalserserglyglyglyglyserglyglyglyglyser
115120125
glyglyglyglysergluilevalmetthrglnserproalathrleu
130135140
servalserproglygluargalathrleusercysargalasergln
145150155160
asnileargasppheleuhistrptyrglnglnlysproglyglnala
165170175
proargleuleuiletyrtyralaserglnserileserglyilepro
180185190
alaargpheserglyserglyserglythraspphethrleuthrile
195200205
serserleugluprogluaspphealavaltyrtyrcysglnasngly
210215220
hisserpheprotyrthrpheglyglyglythrlysvalgluilelys
225230235240
arg
<210>15
<211>118
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>15
gluvalglnleuglngluserglyproserleuvallysprosergln
151015
thrleuserleuthrcysservalthrglyaspserilethrserasp
202530
tyrtrpasntrpilearglyspheproglyasnlysleuglutyrmet
354045
glytyrilesertyrserglyasnthrtyrthrasnproserleulys
505560
serargileserilethrargaspthrserlysasnglntyrtyrleu
65707580
glnleuasnsermetthrthrgluaspthralathrtyrtyrcysala
859095
argtyrlysthrileaspserserpheaspvaltrpglyalaglythr
100105110
thrvalthrvalserser
115
<210>16
<211>107
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>16
aspilevalmetthrglnserproalathrleuservalthrprogly
151015
aspargvalserleusercysargalaserglnasnileargaspphe
202530
leuhistrptyrglnglnlysserhisgluserproargleuleuile
354045
lystyralaserglnserileserglyileproserargphesergly
505560
serglyserglyserasppheserleuasnileasnservalglupro
65707580
gluaspvalglyvaltyrtyrcysglnasnglyhisserpheprotyr
859095
thrpheglyglyglythrlysleugluilelys
100105
<210>17
<211>448
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>17
gluvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuthrcysthrvalthrglyaspserileserserasp
202530
tyrtrpasntrpvalargglnalaproglyglnargleuglutrpmet
354045
glytyrilesertyrserglyasnthrtyrthrasnproserleulys
505560
serargilethrileasnargaspthrserlysasnglnpheserleu
65707580
glnleuasnservalthrprogluaspthralavaltyrtyrcysala
859095
argtyrlysthrilethrservalleuaspvaltrpglyglnglythr
100105110
thrvalthrvalserseralaserthrlysglyproservalphepro
115120125
leualaproserserlysserthrserglyglythralaalaleugly
130135140
cysleuvallysasptyrpheprogluprovalthrvalsertrpasn
145150155160
serglyalaleuthrserglyvalhisthrpheproalavalleugln
165170175
serserglyleutyrserleuserservalvalthrvalproserser
180185190
serleuglythrglnthrtyrilecysasnvalasnhislysproser
195200205
asnthrlysvalasplyslysvalgluprolyssercysasplysthr
210215220
histhrcysproprocysproalaprogluleuleuglyglyproser
225230235240
valpheleupheproprolysprolysaspthrleumetileserarg
245250255
thrprogluvalthrcysvalvalvalaspvalserhisgluasppro
260265270
gluvallyspheasntrptyrvalaspglyvalgluvalhisasnala
275280285
lysthrlysproargglugluglntyrasnserthrtyrargvalval
290295300
servalleuthrvalleuhisglnasptrpleuasnglylysglutyr
305310315320
lyscyslysvalserasnlysalaleuproalaproileglulysthr
325330335
ileserlysalalysglyglnproarggluproglnvaltyrthrleu
340345350
proproserarggluglumetthrlysasnglnvalserleuthrcys
355360365
leuvallysglyphetyrproseraspilealavalglutrpgluser
370375380
asnglyglnprogluasnasntyrlysthrthrproprovalleuasp
385390395400
seraspglyserphepheleutyrserlysleuthrvalasplysser
405410415
argtrpglnglnglyasnvalphesercysservalmethisgluala
420425430
leuhisasnhistyrthrglnlysserleuserleuserproglylys
435440445
<210>18
<211>214
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>18
gluilevalmetthrglnserproalathrleuservalserprogly
151015
gluargalathrleusercysargalaserglnasnileargaspphe
202530
leuhistrptyrglnglnlysproglyglnalaproargleuleuile
354045
tyrtyralaserglnserileserglyileproalaargphesergly
505560
serglyserglythraspphethrleuthrileserserleuglupro
65707580
gluaspphealavaltyrtyrcysglnasnglyhisserpheprotyr
859095
thrpheglyglyglythrlysvalgluilelysargthrvalalaala
100105110
proservalpheilepheproproseraspgluglnleulyssergly
115120125
thralaservalvalcysleuleuasnasnphetyrproarggluala
130135140
lysvalglntrplysvalaspasnalaleuglnserglyasnsergln
145150155160
gluservalthrgluglnaspserlysaspserthrtyrserleuser
165170175
serthrleuthrleuserlysalaasptyrglulyshislysvaltyr
180185190
alacysgluvalthrhisglnglyleuserserprovalthrlysser
195200205
pheasnargglyglucys
210
<210>19
<211>448
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>19
gluvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuthrcysthrvalthrglyaspserileserserasp
202530
tyrtrpasntrpvalargglnalaproglyglnargleuglutrpmet
354045
glytyrilesertyrserglyasnthrtyrthrasnproserleulys
505560
serargilethrileasnargaspthrserlysasnglnpheserleu
65707580
glnleuasnservalthrprogluaspthralavaltyrtyrcysala
859095
argtyrlysthrilethrseralapheaspvaltrpglyglnglythr
100105110
thrvalthrvalserseralaserthrlysglyproservalphepro
115120125
leualaproserserlysserthrserglyglythralaalaleugly
130135140
cysleuvallysasptyrpheprogluprovalthrvalsertrpasn
145150155160
serglyalaleuthrserglyvalhisthrpheproalavalleugln
165170175
serserglyleutyrserleuserservalvalthrvalproserser
180185190
serleuglythrglnthrtyrilecysasnvalasnhislysproser
195200205
asnthrlysvalasplyslysvalgluprolyssercysasplysthr
210215220
histhrcysproprocysproalaprogluleuleuglyglyproser
225230235240
valpheleupheproprolysprolysaspthrleumetileserarg
245250255
thrprogluvalthrcysvalvalvalaspvalserhisgluasppro
260265270
gluvallyspheasntrptyrvalaspglyvalgluvalhisasnala
275280285
lysthrlysproargglugluglntyrasnserthrtyrargvalval
290295300
servalleuthrvalleuhisglnasptrpleuasnglylysglutyr
305310315320
lyscyslysvalserasnlysalaleuproalaproileglulysthr
325330335
ileserlysalalysglyglnproarggluproglnvaltyrthrleu
340345350
proproserarggluglumetthrlysasnglnvalserleuthrcys
355360365
leuvallysglyphetyrproseraspilealavalglutrpgluser
370375380
asnglyglnprogluasnasntyrlysthrthrproprovalleuasp
385390395400
seraspglyserphepheleutyrserlysleuthrvalasplysser
405410415
argtrpglnglnglyasnvalphesercysservalmethisgluala
420425430
leuhisasnhistyrthrglnlysserleuserleuserproglylys
435440445
<210>20
<211>448
<212>prt
<213>2ambystomalateralexambystomajeffersonianum
<400>20
gluvalglnleuglngluserglyproglyleuvallysproserglu
151015
thrleuserleuthrcysthrvalthrglyaspserileserserasp
202530
tyrtrpasntrpvalargglnalaproglyglnargleuglutrpmet
354045
glytyrilesertyrserglyasnthrtyrthrasnproserleulys
505560
serargilethrileasnargaspthrserlysasnglnpheserleu
65707580
glnleuasnservalthrprogluaspthralavaltyrtyrcysala
859095
argtyrlysthrilethrsermetpheaspvaltrpglyglnglythr
100105110
thrvalthrvalserseralaserthrlysglyproservalphepro
115120125
leualaproserserlysserthrserglyglythralaalaleugly
130135140
cysleuvallysasptyrpheprogluprovalthrvalsertrpasn
145150155160
serglyalaleuthrserglyvalhisthrpheproalavalleugln
165170175
serserglyleutyrserleuserservalvalthrvalproserser
180185190
serleuglythrglnthrtyrilecysasnvalasnhislysproser
195200205
asnthrlysvalasplyslysvalgluprolyssercysasplysthr
210215220
histhrcysproprocysproalaprogluleuleuglyglyproser
225230235240
valpheleupheproprolysprolysaspthrleumetileserarg
245250255
thrprogluvalthrcysvalvalvalaspvalserhisgluasppro
260265270
gluvallyspheasntrptyrvalaspglyvalgluvalhisasnala
275280285
lysthrlysproargglugluglntyrasnserthrtyrargvalval
290295300
servalleuthrvalleuhisglnasptrpleuasnglylysglutyr
305310315320
lyscyslysvalserasnlysalaleuproalaproileglulysthr
325330335
ileserlysalalysglyglnproarggluproglnvaltyrthrleu
340345350
proproserarggluglumetthrlysasnglnvalserleuthrcys
355360365
leuvallysglyphetyrproseraspilealavalglutrpgluser
370375380
asnglyglnprogluasnasntyrlysthrthrproprovalleuasp
385390395400
seraspglyserphepheleutyrserlysleuthrvalasplysser
405410415
argtrpglnglnglyasnvalphesercysservalmethisgluala
420425430
leuhisasnhistyrthrglnlysserleuserleuserproglylys
435440445
<210>21
<211>723
<212>dna
<213>2ambystomalateralexambystomajeffersonianum
<400>21
gaggtgcaactgcaggagtccggacctggcctggtgaagccctccgagaccctgagcctg60
acctgcaccgtgacaggcgactccatttccagcgattactggaactgggtgaggcaggct120
cctggacagaggctcgagtggatgggctacatctcctactccggcaatacctacaccaac180
ccctccctgaagagccggatcaccatcaacagggacacctccaagaaccagttcagcctg240
cagctgaactccgtgacccccgaggacaccgccgtgtactactgcgccaggtacaagacc300
atcgacagcagcttcgacgtgtggggccagggcaccaccgtgacagtgtcctccggaggc360
ggcggaagcggcggcggcggctccggaggcggcggcagcgaaattgtgatgacccagagc420
cctgctaccctgagcgtgtcccctggagagcgggctaccctgtcctgtagggccagccag480
aacatcagggacttcctgcactggtatcaacagaagcctggacaggcccccaggctgctg540
atctactatgccagccagagcatctccggcattcccgctaggttctccggcagcggctcc600
ggcacagacttcaccctgaccatctccagcctggagcccgaagacttcgccgtgtattac660
tgccagaacggccactcctttccctataccttcggcggcggcaccaaggtggagatcaag720
agg723
<210>22
<211>1347
<212>dna
<213>2ambystomalateralexambystomajeffersonianum
<400>22
gaggtgcaactgcaggagtccggacctggcctggtgaagccctccgagaccctgagcctg60
acctgcaccgtgacaggcgactccatttccagcgattactggaactgggtgaggcaggct120
cctggacagaggctcgagtggatgggctacatctcctactccggcaatacctacaccaac180
ccctccctgaagagccggatcaccatcaacagggacacctccaagaaccagttcagcctg240
cagctgaactccgtgacccccgaggacaccgccgtgtactactgcgccaggtacaagacc300
atcacctcggtgttggacgtgtggggccagggcaccaccgtgacagtgtcctccgccagc360
accaagggaccatccgtgttcccactggctccaagctctaaatccactagcggaggcacc420
gcagccctgggatgtctggtgaaggattacttcccagagcccgtcacagtgtcatggaac480
tccggggctctgacctctggtgtccacacatttccagcagtgctgcagagttcaggcctg540
tactccctgtccagcgtggtcacagtgccctctagttcactgggaactcagacctatatc600
tgcaacgtgaatcacaagccatccaatactaaagtcgacaagaaagtggagcccaagagc660
tgtgataaaacacatacttgccccccttgtcctgcaccagaactgctgggaggaccatcc720
gtgttcctgtttccacccaagcctaaagacactctgatgatttctcgaacacccgaggtc780
acttgcgtggtcgtggacgtgtcccacgaggatcctgaagtcaagtttaactggtacgtg840
gatggagtcgaagtgcataatgctaagacaaaacctagagaggaacagtacaacagtaca900
tatagagtcgtgtcagtcctgactgtgctgcatcaggactggctgaacgggaaggagtat960
aagtgcaaagtgtccaataaggctctgcccgcacctatcgagaaaactattagcaaggct1020
aaaggccagcctagggaaccacaggtgtacaccctgcctccatctcgggaggaaatgact1080
aagaaccaggtcagtctgacctgtctggtgaaaggcttctatccttccgacatcgcagtg1140
gagtgggaaagcaatggacagccagagaacaattacaagaccacaccccctgtgctggac1200
agcgatgggtctttctttctgtatagtaagctgaccgtggataaatcacggtggcagcag1260
ggtaatgtcttttcttgtagtgtgatgcacgaagccctgcacaaccattacactcagaaa1320
tccctgtcactgtcccctggaaagtga1347
<210>23
<211>645
<212>dna
<213>2ambystomalateralexambystomajeffersonianum
<400>23
gaaattgtgatgacccagagccctgctaccctgagcgtgtcccctggagagcgggctacc60
ctgtcctgtagggccagccagaacatcagggacttcctgcactggtatcaacagaagcct120
ggacaggcccccaggctgctgatctactatgccagccagagcatctccggcattcccgct180
aggttctccggcagcggctccggcacagacttcaccctgaccatctccagcctggagccc240
gaagacttcgccgtgtattactgccagaacggccactcctttccctataccttcggcggc300
ggcaccaaggtggagatcaagaggactgtggctgcaccatctgtcttcatcttcccgcca360
tctgatgagcagttgaaatctggaactgcctctgttgtgtgcctgctgaataacttctat420
cccagagaggccaaagtacagtggaaggtggataacgccctccaatcgggtaactcccag480
gagagtgtcacagagcaggacagcaaggacagcacctacagcctcagcagcaccctgacg540
ctgagcaaagcagactacgagaaacacaaagtctacgcctgcgaagtcacccatcagggc600
ctgagctcgcccgtcacaaagagcttcaacaggggagagtgttaa645
<210>24
<211>1347
<212>dna
<213>2ambystomalateralexambystomajeffersonianum
<400>24
gaggtgcaactgcaggagtccggacctggcctggtgaagccctccgagaccctgagcctg60
acctgcaccgtgacaggcgactccatttccagcgattactggaactgggtgaggcaggct120
cctggacagaggctcgagtggatgggctacatctcctactccggcaatacctacaccaac180
ccctccctgaagagccggatcaccatcaacagggacacctccaagaaccagttcagcctg240
cagctgaactccgtgacccccgaggacaccgccgtgtactactgcgccaggtacaagacc300
atcacctccgccttcgacgtgtggggccagggcaccaccgtgacagtgtcctccgccagc360
accaagggaccatccgtgttcccactggctccaagctctaaatccactagcggaggcacc420
gcagccctgggatgtctggtgaaggattacttcccagagcccgtcacagtgtcatggaac480
tccggggctctgacctctggtgtccacacatttccagcagtgctgcagagttcaggcctg540
tactccctgtccagcgtggtcacagtgccctctagttcactgggaactcagacctatatc600
tgcaacgtgaatcacaagccatccaatactaaagtcgacaagaaagtggagcccaagagc660
tgtgataaaacacatacttgccccccttgtcctgcaccagaactgctgggaggaccatcc720
gtgttcctgtttccacccaagcctaaagacactctgatgatttctcgaacacccgaggtc780
acttgcgtggtcgtggacgtgtcccacgaggatcctgaagtcaagtttaactggtacgtg840
gatggagtcgaagtgcataatgctaagacaaaacctagagaggaacagtacaacagtaca900
tatagagtcgtgtcagtcctgactgtgctgcatcaggactggctgaacgggaaggagtat960
aagtgcaaagtgtccaataaggctctgcccgcacctatcgagaaaactattagcaaggct1020
aaaggccagcctagggaaccacaggtgtacaccctgcctccatctcgggaggaaatgact1080
aagaaccaggtcagtctgacctgtctggtgaaaggcttctatccttccgacatcgcagtg1140
gagtgggaaagcaatggacagccagagaacaattacaagaccacaccccctgtgctggac1200
agcgatgggtctttctttctgtatagtaagctgaccgtggataaatcacggtggcagcag1260
ggtaatgtcttttcttgtagtgtgatgcacgaagccctgcacaaccattacactcagaaa1320
tccctgtcactgtcccctggaaagtga1347
<210>25
<211>1347
<212>dna
<213>2ambystomalateralexambystomajeffersonianum
<400>25
gaggtgcaactgcaggagtccggacctggcctggtgaagccctccgagaccctgagcctg60
acctgcaccgtgacaggcgactccatttccagcgattactggaactgggtgaggcaggct120
cctggacagaggctcgagtggatgggctacatctcctactccggcaatacctacaccaac180
ccctccctgaagagccggatcaccatcaacagggacacctccaagaaccagttcagcctg240
cagctgaactccgtgacccccgaggacaccgccgtgtactactgcgccaggtacaagacc300
atcacgagcatgttcgacgtgtggggccagggcaccaccgtgacagtgtcctccgccagc360
accaagggaccatccgtgttcccactggctccaagctctaaatccactagcggaggcacc420
gcagccctgggatgtctggtgaaggattacttcccagagcccgtcacagtgtcatggaac480
tccggggctctgacctctggtgtccacacatttccagcagtgctgcagagttcaggcctg540
tactccctgtccagcgtggtcacagtgccctctagttcactgggaactcagacctatatc600
tgcaacgtgaatcacaagccatccaatactaaagtcgacaagaaagtggagcccaagagc660
tgtgataaaacacatacttgccccccttgtcctgcaccagaactgctgggaggaccatcc720
gtgttcctgtttccacccaagcctaaagacactctgatgatttctcgaacacccgaggtc780
acttgcgtggtcgtggacgtgtcccacgaggatcctgaagtcaagtttaactggtacgtg840
gatggagtcgaagtgcataatgctaagacaaaacctagagaggaacagtacaacagtaca900
tatagagtcgtgtcagtcctgactgtgctgcatcaggactggctgaacgggaaggagtat960
aagtgcaaagtgtccaataaggctctgcccgcacctatcgagaaaactattagcaaggct1020
aaaggccagcctagggaaccacaggtgtacaccctgcctccatctcgggaggaaatgact1080
aagaaccaggtcagtctgacctgtctggtgaaaggcttctatccttccgacatcgcagtg1140
gagtgggaaagcaatggacagccagagaacaattacaagaccacaccccctgtgctggac1200
agcgatgggtctttctttctgtatagtaagctgaccgtggataaatcacggtggcagcag1260
ggtaatgtcttttcttgtagtgtgatgcacgaagccctgcacaaccattacactcagaaa1320
tccctgtcactgtcccctggaaagtga1347
- 还没有人留言评论。精彩留言会获得点赞!