应用枯草芽孢杆菌群体响应调控系统合成MK-7的方法与流程
本发明涉及应用枯草芽孢杆菌群体响应调控系统合成mk-7的方法,属于遗传工程领域。
背景技术:
七烯甲萘醌(mk-7)是一类重要的脂溶性维生素,作为γ-谷氨酸羧化酶的辅因子,在调节钙的分配、促进骨骼发育逆转骨质疏松以及在保护血管,防止动脉粥样硬化和心血管疾病反应中起到重要作用。由于七烯甲萘醌对人体具有更好的亲和性和更长的半衰期,得到人们更多的关注。
枯草芽孢杆菌(bacillussubtilis)常被广泛用在食品酶制剂及重要营养化学品的生产中,其产品被fda认证为“generallyregardedassafe”(gras)安全级别。因此,运用代谢工程手段构建重组枯草芽孢杆菌是高效合成mk-7的有效途径。然而,mk-7的合成途径十分复杂,导致重要前体磷酸烯醇式丙酮酸pep大部分流向三羧酸tca循环,而使得分支酸的合成受到限制,另外,异戊二烯焦磷酸也是细胞壁合成的重要物质,如果敲除则会抑制细胞的生长;mep途径中间产物1-羟基-2-甲基-2-丁烯基4-二磷酸酯(hmbpp)和二甲基烯丙基二磷酸酯(dmapp)均对细胞有毒性。如何调整枯草芽孢杆菌代谢流供给,增加mk-7的合成是一个值得深入探讨的问题。
九州大学hanai.t教授课题组采用来自费氏弧菌(vibriofischeri)的luxr群体响应调控系统,在大肠杆菌中实现了碳流从中心代谢途径到异丙醇途径的自动切换。麻省理工学院prather.k教授课题组利用来自玉米细菌性枯萎病菌(pantoeastewartii)的esa系统,将esar的结合位点整合至大肠杆菌果糖磷酸激酶(pfka)基因的启动子区,抑制了pfka基因的转录,产物肌醇的产量提高了5.5倍。然而这些研究均使用异源的群体响应系统进行调控,细胞代谢负担加重,系统稳定性较弱,而若利用细胞自身的群体响应系统对代谢网络进行动态调控,则不会对细胞的代谢造成额外负担。在七烯甲萘醌的研究中还未有使用群体响应动态调控的研究报道。
技术实现要素:
为了解决上述存在的技术问题,本发明构建了phr60-rap60-spo0a的群体响应动态调控系统,并改造pspoiia和pabrb启动子,获得受spo0a-p调控转录活性不同的启动子库,并将获得的启动子动态调控七烯甲萘醌合成途径中关键基因heps/t、isph、pyk和upps,最终得到的枯草芽孢杆菌重组菌bs19、bs20摇瓶发酵生产mk-7的产量分别高达170mg/l、360mg/l,分别是枯草芽孢杆菌(bacillussubtilis)168的17.89、37.89倍,且重组菌bs20在发酵罐中发酵3天,mk-7的产量可达230mg/l。
本发明的第一个目的是提供一种应用枯草芽孢杆菌群体响应调控系统合成mk-7的方法,所述方法包括在枯草芽孢杆菌的染色体上进行了如下改造:
(1)将组氨酸激酶kina的基因进行截短突变并且进行了组成型表达,同时敲除组氨酸激酶kinb的基因;
(2)将含有phag启动子的rap60基因整合到染色体上,并在染色体上rap60基因下游整合两个拷贝的信号分子phr60的表达框;
(3)使用pabrb(cs-1)启动子替换丙酮酸激酶基因和十一碳烯基焦磷酸合成酶基因的启动子;所述pabrb(cs-1)启动子的序列如seqidno.14所示。
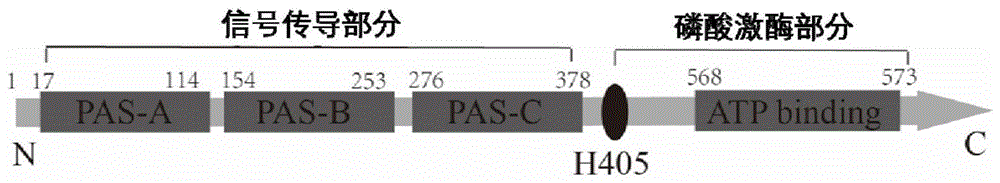
在一种实施方式中,步骤(1)中所述组氨酸激酶kina的genebankid为939230;所述截短突变是指敲除组氨酸激酶kina基因的信号传导部分的pas-a区域,敲除后的组氨酸激酶kina基因kina-δpas-a的序列如seqidno.9所示;所述组氨酸激酶kinb的genebankid为937167。
在一种实施方式中,步骤(2)中所述phag启动子序列如seqidno.16所示;所述转录因子rap60的genebankid为1115983;所述信号分子phr60的表达框的序列如seqidno.17所示。
在一种实施方式中,步骤(3)中所述丙酮酸激酶基因pyk的genebankid为936596;所述十一碳烯基焦磷酸合成酶基因upps的genebankid为939640。
在一种实施方式中,所述枯草芽孢杆菌为枯草芽孢杆菌(bacillussubtilis)168。
在一种实施方式中,所述枯草芽孢杆菌为bacillussubtilis168p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbrp43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxsp43-fnipmena-mena::ganapmena-mena::thrcpmena-mena::daca,其是在枯草芽孢杆菌(bacillussubtilis)168的基础上,用启动子p43替换menf、menb基因的天然启动子;用phbs启动子替换mene基因的天然启动子;用含有p43启动子的entc基因替换dhbb基因;用phbs启动子替换tkt基因的天然启动子;用含有p43启动子的ppsa基因整合到枯草芽孢杆菌染色体上,并敲除ptsg基因;将arogfbr基因与启动子phbs融合后整合到染色体上;利用p43启动子替换arok基因的天然启动子;利用phbs启动子替换ispa基因的天然启动子;利用p43启动子替换heps/t基因的天然启动子;将kdpg基因与启动子phbs融合后整合到染色体上;用p43启动子替换dxr基因、dxs基因、fni基因的天然启动子;在枯草芽孢杆菌基因组的gana、thrc、daca位点整合表达mena的开放阅读框,将最后得到的菌株命名为bs17。
在一种实施方式中,所述menf的genebankid为937190;所述menb的genebankid为937195;所述phbs启动子的序列如seqidno.5所示;所述mene的genebankid为937132;所述entc基因来源于e.colik12,genebankid为945511;所述dhbb的genebankid为936582;所述tkt基因的genebankid为937377;所述ppsa来源于e.colik12,其genebankid为946209;所述ptsg的genebankid为939255;所述arogfbr基因的序列如seqidno.6所示;所述aroa的genebankid为937853;所述arok的genebankid为938343;所述heps/t,genebankid为938998;所述kdpg的genebankid为33073472;所述dxr的genebankid为939636;所述dxs的genebankid为938609;所述fni的genebankid为938985;所述mena的开放阅读框的序列如seqidno.8所示;所述gana的genebankid为936313;所述thrc的genebankid为936660;所述daca的genebankid为940000。
在一种实施方式中,所述方法还包括使用pspoiia(cs-1,3)启动子表达七聚二烯丙基二磷酸合成酶基因和4-羟基-3-甲基丁-2-烯基二磷酸还原酶基因;所述pspoiia(cs-1,3)启动子的序列如seqidno.12所示。
在一种实施方式中,所述七聚二烯丙基二磷酸合成酶基因heps/t的genebankid为938998;所述4-羟基-3-甲基丁-2-烯基二磷酸还原酶基因isph的genebankid为937900。
本发明的第二个目的是提供一种生产七烯甲萘醌的重组菌,其特征在于,在枯草芽孢杆菌的染色体上进行了如下改造:
(1)将组氨酸激酶kina的基因进行截短突变并且进行了组成型表达,同时敲除组氨酸激酶kinb的基因;
(2)将含有phag启动子的rap60基因整合到染色体上,并在染色体上rap60基因下游整合两个拷贝的信号分子phr60的表达框;
(3)使用pabrb(cs-1)启动子替换丙酮酸激酶基因和十一碳烯基焦磷酸合成酶基因的启动子;所述pabrb(cs-1)启动子的序列如seqidno.14所示。
在一种实施方式中,所述重组菌在bs17的基础上还做了如下改造:将组氨酸激酶kina的基因进行截短突变并且进行了组成型表达,敲除组氨酸激酶kinb的基因;将含有phag启动子的rap60基因整合到染色体上,并在染色体上rap60基因下游整合两个拷贝的信号分子phr60的表达框;最后构建得到菌株bacillussubtilis168,p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbrp43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxsp43-fnipmena-mena::ganapmena-mena::thrcpmena-mena::dacapveg-kina-δpas-aδkinbphag-rap60(pnative-phr60)2::hag,将其命名为bs18。
在一种实施方式中,所述重组菌在bs18的基础上还做了如下改造:使用pabrb(cs-1)启动子替换丙酮酸激酶(pyk,genebankid:936596)基因和十一碳烯基焦磷酸合成酶(upps,genebankid:939640)基因的启动子,最后得到菌株bacillussubtilis168,p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbrp43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxsp43-fnipmena-mena::ganapmena-mena::thrcpmena-mena::dacapveg-kina-δpas-aδkinbphag-rap60(pnative-phr60)2::hagpabrb(cs-1)-pyk::pykpabrb(cs-1)-upps::upps,将其命名为bs19。
在一种实施方式中,所述重组菌在bs19的基础上还做了如下改造:使用pspoiia(cs-1,3)启动子表达七聚二烯丙基二磷酸合成酶(heps/t,genebankid:938998)基因和4-羟基-3-甲基丁-2-烯基二磷酸还原酶(isph,genebankid:937900)基因,最后得到菌株bacillussubtilis168,p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbrp43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxsp43-fnipmena-mena::ganapmena-mena::thrcpmena-mena::dacapveg-kina-δpas-aδkinbphag-rap60(pnative-phr60)2::hagpabrb(cs-1)-pyk::pykpabrb(cs-1)-upps::uppspspoiia(cs-1,3)-isph::isphpspoiia(cs-1,3)-heps/t::heps/t,将其命名为bs20。
本发明的第三个目的是提供一种生产七烯甲萘醌的方法,所述方法是应用所述的重组菌进行发酵生产。
本发明的有益效果:
(1)通过对磷酸激酶kina截短突变,并用组成型启动子pveg表达,使细胞能够在培养基营养丰富的情况下产生芽孢信号。
(2)应用本发明方法得到受phr60-rap60群体响应调控的spo0a系统,通过对mk-7合成途径的动态调控,实现了mk-7的高效合成。
(3)不需要额外诱导剂,实现了目标产物高效合成与细胞生长之间的动态平衡。
(4)应用本发明获得的菌株在添加蔗糖的情况下,mk-7的产量明显提高。
(5)最终得到的枯草芽孢杆菌重组菌bs19、bs20摇瓶发酵生产mk-7的产量分别为170mg/l、360mg/l,是枯草芽孢杆菌(bacillussubtilis)168的17.89、37.89倍。且重组菌bs20在发酵罐中发酵3天,mk-7的产量可达230mg/l。
生物材料
本发明的枯草芽孢杆菌(bacillussubtilis)168购自美国典型微生物保藏中心,保藏编号为atccno.27370。
表1菌株基因型
表2序列表
附图说明
图1:磷酸激酶kina结构示意图与截短突变。
图2:信号分子phr60-rap60不同比例对spo0a调控能力的影响;pspoiia-gfp:菌株bs168-pspoiia-gfp;rap60:菌株bs17-2-rap60-gfp;phr60-rap60:菌株bs17-2-phr60-rap60-gfp;2*phr60-rap60:菌株bs17-2-(phr60)*2-rap60-gfp。
图3:spo0a结合位点个数与序列变化对启动子调控能力的变化。
图4:细胞浓度对spo0a调控能力的影响。
图5:phr60-rap60-spo0a动态调控mk-7合成途径。
图6:关键基因的转录水平。
图7:重组菌摇瓶发酵生产mk-7。
图8:重组菌发酵罐发酵生产mk-7。
具体实施方式
mk-7的萃取和hplc检测:在发酵液中加入4倍体积异丙醇和正已烷混合物(1:2v/v),涡旋振荡提取30min后,滤出提取液,8000r/min离心15min。收集上清液,此时mk-7溶解于该相中,置于-80℃冰箱中冷冻,使脂类物质结晶除去,收集滤液,hplc检测mk-7的含量。
hplc检测mk-7产量:采用agilentzorbaxeclipsexdb-c18分离柱(5μm,250×4.6mm),检测的温度在40℃,流动相使用甲醇:二氯甲烷(9:1,v/v),流速为1ml/min,检测波长254nm,进样量10μl。
实施例1:重组菌bs17的构建
(1)重组菌bs1的构建
通过组成型启动子p43替换枯草芽孢杆菌染色体上menf的天然启动子来增强甲基萘醌异分支酸合成酶(menf,genebankid:937190)基因的表达。使用无标记的遗传修饰策略,参见文章(yan,x.,yu,h.-j.,hong,q.,li,s.p.,2008.cre/loxsystemandpcr-basedgenomeengineeringinbacillussubtilis.applenvironmicrob.74,5556-5562),具体构建过程如下:
(a)基因的克隆
①以枯草芽孢杆菌168基因组为模板,采用引物menfup.for和menfup.rev扩增得到menf基因的上游同源臂序列menfup(序列如seqidno.1所示)。
②人工合成含有博莱霉素基因的lox71-zeo-lox66盒(序列如seqidno.2所示)。
③以枯草芽孢杆菌168基因组为模板,采用引物p43.for和p43.rev扩增得到p43启动子序列(序列如seqidno.3所示)。
④以枯草芽孢杆菌168基因组为模板,采用引物menf.for和menf.rev扩增得到menf基因片段(序列如seqidno.4所示)。
(b)融合片段的获得
将步骤(a)中获得的四个片段menfup、lox71-zeo-lox66盒、p43启动子序列、menf基因片段进行重叠延伸pcr,pcr条件:98℃,5min预变性,然后98℃,变性10s,55℃,退火5s,72℃,延伸2min,总共30个循环,切胶回收大小正确的片段,得到融合基因片段menfup-lox71-zeo-lox66-p43-menf。
(c)同源重组
将步骤(b)中得到的融合片段转化到野生型菌株bacillussubtilis168感受态细胞中。由于融合片段中存在menf的上游序列与枯草芽孢杆菌168染色体上的menf的上游序列基因同源,融合片段中存在menf基因与枯草芽孢杆菌168染色体上的menf基因同源,通过同源重组,融合片段中的博来霉素抗性基因zeo和p43启动子替换枯草芽孢杆菌168染色体上menf基因天然启动子。具体步骤为:
①将步骤(b)中构建好的融合片段电转化枯草芽孢杆菌168的感受态细胞,融合片段添加量为100-300ng,电转化条件:电压2.5kv,电击时间5ms,37℃复苏5h涂布终浓度为10μg/ml的博来霉素抗性的lb平板,37℃厌氧培养48h。博来霉素抗性呈阳性的为转化成功的枯草芽孢杆菌。
②挑选平板上长出的单菌落,利用引物bs1yz.for和bs1yz.rev进行菌落pcr验证,替换后扩增得到的片段长度为1350bp。并进行测序,测序正确的即为lox71-zeo-lox66-p43融合基因成功替换枯草芽孢杆菌168染色体上的menf基因天然启动子。最后通过cre/lox重组系统,敲除博来霉素抗性基因zeo,验证正确后得到菌株bacillussubtilis168,p43-menf,将其命名为bs1。
表3引物序列表
(2)重组菌bs2的构建
在步骤(1)得到的菌株bs1的基础上,采用与步骤(1)类似的方法,利用p43启动子替换枯草芽孢杆菌168染色体上二羟基萘甲酸合成酶(menb,genebankid:937195)基因的天然启动子,验证正确后得到菌株bacillussubtilis168,p43-menfp43-menb,将其命名为bs2。
(3)重组菌bs3的构建
在步骤(2)得到的菌株bs2的基础上,采用与步骤(1)类似的方法,利用phbs启动子(序列如seqidno.5所示)替换枯草芽孢杆菌中o-琥珀酰苯甲酸-coa连接酶(mene,genebankid:937132)基因的天然启动子,验证正确后得到菌株bacillussubtilis168,p43-menfp43-menbphbs-mene,将其命名为bs3。
(4)重组菌bs4的构建
在步骤(3)得到的菌株bs3的基础上,用含有p43启动子的来源于e.colik12的异分支酸合酶(entc,genebankid:945511)基因替换枯草芽孢杆菌染色体上的异分支酸(dhbb,genebankid:936582)基因。具体构建过程如下:
(a)融合片段的获得
以枯草芽孢杆菌168基因组为模板,分别扩增得到dhbb基因的上游同源臂序列dhbbup、dhbb下游同源臂序列dhbbdown、p43启动子序列;人工合成lox71-zeo-lox66盒序列(序列如seqidno.2所示);以e.colik12基因组为模板,扩增得到entc基因序列,然后将五个片段dhbbup、lox71-zeo-lox66盒、p43启动子序列、entc基因序列、dhbbdown基因片段进行重叠延伸pcr,得到融合基因片段dhbbup-lox71-zeo-lox66-p43-entc-dhbbdown。
(b)同源重组
将步骤(a)中得到的融合片段转化到bs3感受态细胞中,然后通过cre/lox重组系统,敲除菌株中博来霉素抗性基因zeo,得到菌株bacillussubtilis168,p43-menfp43-menbphbs-menep43-entcδdhbb,将其命名为bs4。
(5)重组菌bs5的构建
在步骤(4)得到的菌株bs4的基础上,采用与步骤(1)类似的方法,利用phbs启动子替换枯草芽孢杆菌染色体上转酮酶(tkt,genebankid:937377)基因的天然启动子,验证正确后得到菌株bacillussubtilis168,p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tkt,将其命名为bs5。
(6)重组菌bs6的构建
在步骤(5)得到的菌株bs5的基础上,将含有p43启动子的来源于e.colik12的磷酸烯醇式丙酮酸合酶(ppsa,genebankid:946209)基因整合到枯草芽孢杆菌染色体上的多糖脱乙酰酶c(yjea,genebankid:936440)基因和yjfa(genebankid:939830)基因之间,并敲除染色体上的葡萄糖磷酸转运酶(ptsg,genebankid:939255)基因。具体构建过程为:
(a)融合片段的获得
以e.colik12基因组为模板,扩增得到ppsa基因,以枯草芽孢杆菌168基因组为模板,分别扩增得到yjea基因和yjfa基因、p43启动子序列;人工合成lox71-zeo-lox66盒序列(序列如seqidno.2所示)。然后将五个片段yjea、lox71-zeo-lox66盒、p43、ppsa、yjfa进行重叠延伸pcr,得到融合基因片段yjea-lox71-zeo-lox66-p43-ppsa-yjfa。
以枯草芽孢杆菌168的基因组为模板分别扩增ptsg上游同源臂ptsgup和下游同源臂ptsgdown,将ptsgup、lox71-zeo-lox66、ptsgdown进行重叠延伸pcr,得到融合基因片段ptsgup-lox71-zeo-lox66-ptsgdown。
(b)同源重组
将步骤(a)中得到的融合片段yjea-lox71-zeo-lox66-p43-ppsa-yjfa和ptsgup-lox71-zeo-lox66-ptsgdown均转化到bs5感受态细胞中,然后通过cre/lox重组系统,敲除菌株中博来霉素抗性基因zeo,验证正确后得到菌株bacillussubtilis168p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsg,将其命名为bs6。
(7)重组菌bs7的构建
在步骤(6)得到的菌株bs6的基础上,将人工合成的arogfbr基因(序列如seqidno.6所示)与启动子phbs(序列如seqidno.5所示)融合后整合到枯草芽孢杆菌染色体上应激蛋白(ytxj,genebankid:937308)基因和3-脱氧-d-阿拉伯糖-庚磺酸盐7-磷酸合成酶酶(aroa,genebankid:937853)基因之间,具体构建过程为:
(a)融合片段的获得
人工合成arogfbr基因(序列如seqidno.6所示);以枯草芽孢杆菌168基因组为模板,分别扩增得到ytxj基因和aroa基因、phbs启动子序列。然后将五个片段ytxj、lox71-zeo-lox66、phbs、arogfbr、aroa进行重叠延伸pcr,得到融合基因片段ytxj-lox71-zeo-lox66-phbs-arogfbr-aroa。
(b)同源重组
将步骤(a)中得到的融合片段转化到bs6感受态细胞中,然后通过cre/lox重组系统,敲除菌株中博来霉素抗性基因zeo,验证正确后得到菌株bacillussubtilis168p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbr,将其命名为bs7。
(8)重组菌bs8的构建
在步骤(7)得到的菌株bs7的基础上,采用与步骤(1)类似的方法,利用p43启动子替换枯草芽孢杆菌染色体上莽草酸激酶(arok,genebankid:938343)基因的天然启动子,验证正确后得到菌株bacillussubtilis168p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbrp43-arok,将其命名为bs8。
(9)重组菌bs9的构建
在步骤(8)得到的菌株bs8的基础上,采用与步骤(1)类似的方法,利用phbs启动子替换枯草芽孢杆菌染色体上法尼基二磷酸合酶(ispa,genebankid:938652)基因的天然启动子,验证正确后得到菌株bacillussubtilis168p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbrp43-arokphbs-ispa,将其命名为bs9。
(10)重组菌bs10的构建
在步骤(9)得到的菌株bs9的基础上,采用与步骤(1)类似的方法,利用p43启动子替换枯草芽孢杆菌染色体上七聚二烯丙基二磷酸合成酶(heps/t,genebankid:938998)基因的天然启动子,验证正确后得到菌株bacillussubtilis168p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbrp43-arokphbs-ispap43-heps/t,将其命名为bs10。
(11)重组菌bs11的构建
在步骤(10)得到的菌株bs10的基础上,将来源于运动发酵单胞菌(zymomonasmobilis)中的2-脱氢-3-脱氧-磷酸葡萄糖酸醛缩酶(kdpg,genebankid:33073472)基因与启动子phbs融合后整合到枯草芽孢杆菌染色体上尿激酶(yclg,genebankid:938292)基因和芽孢萌发响应蛋白(gerka,genebankid:938285)基因之间,具体构建过程为:
(a)融合片段的获得
以运动发酵单胞菌(zymomonasmobilis)基因组为模板,合成kdpg基因;以枯草芽孢杆菌168基因组为模板,分别扩增得到yclg基因、phbs启动子序列和gerka基因;人工合成lox71-zeo-lox66盒序列(序列如seqidno.2所示)。然后将五个片段yclg、lox71-zeo-lox66盒、phbs启动子序列、kdpg、gerka进行重叠延伸pcr,得到融合基因片段yclg-lox71-zeo-lox66-phbs-kdpg-gerka
(b)同源重组
将步骤(a)中得到的融合片段转化到bs10感受态细胞中,然后通过cre/lox重组系统,敲除菌株中博来霉素抗性基因zeo,验证正确后得到菌株bacillussubtilis168p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbr::lox72p43-arokphbs-ispap43-heps/tphbs-kdpg,将其命名为bs11。
(12)重组菌bs12的构建
在步骤(11)得到的菌株bs11的基础上,采用与步骤(1)类似的方法,利用p43启动子替换枯草芽孢杆菌染色体上1-脱氧-d-木酮糖-5-磷酸还原异构酶酶(dxr,genebankid:939636)基因的天然启动子,验证正确后得到菌株bacillussubtilis168p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbrp43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxr,将其命名为bs12。
(13)重组菌bs13的构建
在步骤(12)得到的菌株bs12的基础上,采用与步骤(1)类似的方法,利用p43启动子替换枯草芽孢杆菌168中1-脱氧木酮糖-5-磷酸合成酶(dxs,genebankid:938609)基因的天然启动子,验证正确后得到菌株bacillussubtilis168p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbrp43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxs,将其命名为bs13。
(14)重组菌bs14的构建
在步骤(13)得到的菌株bs13的基础上,采用与步骤(1)类似的方法,利用p43启动子替换枯草芽孢杆菌染色体上异戊烯焦磷酸异构酶酶(fni,genebankid:938985)中基因的天然启动子,验证正确后得到菌株bacillussubtilis168p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbrp43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxsp43-fni,将其命名为bs14。
(15)重组菌bs15的构建
在步骤(14)得到的菌株bs14的基础上,在枯草芽孢杆菌基因组gana位点插入mena的阅读框。gana(genebankid:936313)表达阿拉伯半乳聚糖i型寡聚体外切水解酶。具体构建过程为:
(a)基因的克隆
①以枯草芽孢杆菌168基因组为模板,采用引物mena-up::gana.for和mena-up::gana.rev扩增得到gana基因的上游同源臂序列ganaup。
②人工合成含有博莱霉素基因的lox71-zeo-lox66盒(序列如seqidno.2所示)。
③以枯草芽孢杆菌(bacillussubtilis)168基因组为模板,利用引物mena-orf.for和mena-orf.rev扩增1,4-二羟基-2-萘甲酸七戊烯基转移酶(mena)的开放阅读框,将扩增得到的基因片段进行琼脂糖凝胶检测大小为1537bp,切胶回收大小正确的片段,获得核苷酸序列如seqidno.8的mena阅读框序列pmena-mena。
④以枯草芽孢杆菌168基因组为模板,采用引物mena-down::gana.for和mena-down:gana.rev扩增得到gana基因的下游同源臂序列ganadown。
(b)融合片段的获得
将步骤(a)中获得的四个片段ganaup、lox71-zeo-lox66盒、pmena-mena、ganadown基因片段进行重叠延伸pcr,得到融合基因片段ganaup-lox71-zeo-lox66-pmena-mena-ganadown。
(c)同源重组
将步骤(b)中得到的融合片段转化到bs14感受态细胞中,并涂布于博来霉素抗性的lb平板,挑选平板上长出的单菌落,进行菌落pcr验证并进行测序。最后通过cre/lox重组系统,敲除菌株中博来霉素抗性基因zeo,最后得到菌株bacillussubtilis168p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbr::lox72p43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxsp43-fnipmena-mena::gana,将其命名为bs15。
表4引物序列表
(16)重组菌bs16的构建
在步骤(15)得到的菌株bs15的基础上,采用与步骤(15)类似的方法,在枯草芽孢杆菌基因组thrc位点插入mena的阅读框。thrc(genebankid:936660)翻译表达苏氨酸合成酶。验证正确后得到菌株bacillussubtilis168p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbr::lox72p43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxsp43-fnipmena-mena::ganapmena-mena::thrc,将其命名为bs16。
(17)重组菌bs17的构建
在步骤(16)得到的菌株bs16的基础上,采用与步骤(15)类似的方法,在枯草芽孢杆菌基因组daca位点插入mena的阅读框。daca(genebankid:940000)表达丙氨酸羧肽酶。验证正确后得到菌株bacillussubtilis168p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbr::lox72p43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxsp43-fnipmena-mena::ganapmena-mena::thrcpmena-mena::daca,将其命名为bs17。
实施例2:组氨酸激酶kina的截短突变以及利用组成型启动子表达
组氨酸激酶kina、kinb只有在碳饥饿的情况下,自身才能磷酸化,然后使得dna结合蛋白spo0a磷酸化从而被激活,磷酸化的spo0a与启动子结合,从而上调或下调相应的基因表达,发挥调控基因表达的能力。但是目的产物的高效合成,需要有效碳源的持续供应。因此,通过截短组氨酸激酶kina,并利用组成型启动子pveg表达截短的磷酸激酶kina,截短pas-adomain的突变方法如图1所示,使组氨酸磷酸激酶kina能够持续的表达。
(1)融合片段的获得
以枯草芽孢杆菌168基因组为模板,分别扩增得到组氨酸激酶(kina,genebankid:939230)的上游同源臂序列kinaup、截短后的组氨酸激酶基因片段kina-δpas-a(序列如seqidno.9所示)和pveg启动子序列(序列如seqidno.10所示),人工合成lox71-zeo-lox66盒序列(序列如seqidno.2所示),然后将四个片段kinaup、lox71-zeo-lox66盒、pveg启动子序列、kina-δpas-a基因片段进行重叠延伸pcr,得到融合基因片段kinaup-lox71-zeo-lox66-pveg-kina-δpas-a。
②同源重组
将步骤①中得到的融合片段转化到实施例1中得到的bs17感受态细胞中,并涂布于博来霉素抗性的lb平板,挑选平板上长出的单菌落,进行菌落pcr验证并进行测序。最后通过cre/lox重组系统,敲除菌株中博来霉素抗性基因zeo,得到菌株bacillussubtilis168,p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbr::lox72p43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxsp43-fnipmena-mena::ganapmena-mena::thrcpmena-mena::dacapveg-kina-δpas-a,将其命名为bs17-1。
实施例3:组氨酸激酶kinb的敲除
为了减少其他组氨酸磷酸激酶对调控系统的影响,敲除另一个主要的磷酸激酶kinb(kinb,genebankid:937167),以保证调控系统的单一性。在实施例2中得到的重组菌bs17-1的基础上,敲除枯草芽孢杆菌染色体上的磷酸激酶基因kinb。
(1)融合片段的获得
以枯草芽孢杆菌168基因组为模板,分别扩增得到kinbup、kinbdown、人工合成lox71-zeo-lox66盒序列,然后将三个片段kinbup、lox71-zeo-lox66盒、kinbdown进行重叠延伸pcr,得到融合基因片段kinbup-lox71-zeo-lox66-kinbdown。
②同源重组
将步骤①中得到的融合片段转化到bs17-1受态细胞中,并涂布于博来霉素抗性的lb平板,挑选平板上长出的单菌落,进行菌落pcr验证并进行测序。最后通过cre/lox重组系统,敲除菌株中博来霉素抗性基因zeo,最后得到菌株bacillussubtilis168,p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbr::lox72p43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxsp43-fnipmena-mena::ganapmena-mena::thrcpmena-mena::dacapveg-kina-δpas-aδkinb,将其命名为bs17-2。
实施例4:phr60-rap60-spo0a的群体响应动态调控系统的构建
spo0a的磷酸化水平受到了组氨酸激酶kina的调控,在实施例3得到的菌株bs17-2中,将kina进行了截短突变并且进行了组成型表达同时敲除了另一个主要的磷酸激酶kinb,这使spo0a能够在营养丰富的情况下得到磷酸化修饰,从而发挥功能。但是,枯草芽孢杆菌中存在的phr-rap群体响应调控系统仅仅能够控制spo0a-p的磷酸化水平,并不能调控kina的磷酸化程度,这使得kina在细胞生长前期就具有了磷酸化的能力,这不利于七烯甲萘醌的动态调控合成,解决方式是kina的磷酸化在细胞生长前期能够受到抑制,随着细胞的生长能够解除。据相关报道(doi:10.1111/mmi.12939)phr60-rap60不仅能够抑制spo0a的磷酸化水平,而且能够抑制kina的磷酸化水平。所以在细胞中引入phr60-rap60群体响应系统。并通过实验确定引入的phr60和rap60的比例关系,具体过程如下:
(1)重组载体pht01-pspoiia-gfp的构建
为了验证phr60-rap60对spo0a-p磷酸化的影响,使用受spo0a-p调控的pspoiia启动子融合绿色荧光蛋白(gfp),通过荧光值的变化表征spo0a-p的调控能力。
①用引物pspoiia.for和pspoiia.rev扩增启动子pspoiia(序列如seqidno.11所示),利用引物gfp.for和gfp.rev扩增得到绿色荧光蛋白gfp基因(序列如seqidno.15)所示,通过gibson多片段组装技术将俩个片段连接到pht01质粒上,最终获得重组载体pht01-pspoiia-gfp。
表5引物序列表
(2)在实施例3得到的重组菌bs17-2的基础上,在枯草芽孢杆菌染色体上插入异源转录因子rap60(genebankid:1115983)。
①融合片段的获得
以枯草芽孢杆菌168基因组为模板,分别扩增得到rap60基因的上游同源臂序列rap60up、rap60基因片段和phag启动子序列(序列如seqidno.16所示),人工合成lox71-zeo-lox66盒序列(序列如seqidno.2所示),然后将四个片段rap60up、lox71-zeo-lox66盒、phag启动子序列、rap60基因片段进行重叠延伸pcr,得到融合基因片段rap60up-lox71-zeo-lox66-phag-rap60。
②同源重组
将步骤①中得到的融合片段转化到bs17-2感受态细胞中,并涂布于博来霉素抗性的lb平板,挑选平板上长出的单菌落,进行菌落pcr验证并进行测序。最后通过cre/lox重组系统,敲除菌株中博来霉素抗性基因zeo,最后得到菌株bacillussubtilis168,p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbr::lox72p43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxsp43-fnipmena-mena::lacapmena-mena::thrcpmena-mena::dacapveg-kina-δpas-aδkinbphag-rap60,将其命名为bs17-2-rap60。
(3)在步骤(2)得到的重组菌bs17-2-rap60的基础上,在枯草芽孢杆菌染色体rap60基因下游,分别不插入、插入一个拷贝、插入两个拷贝的信号分子phr60的表达框(序列如seqidno.17所示),phr60的genebankid为1115984,得到三株菌bs17-2-rap60,bs17-2-phr60-rap60,bs17-2-(phr60)*2-rap60。
(4)将(1)中构建得到的重组质粒pht01-pspoiia-gfp分别转化野生型bacillussubtilis168和步骤(3)中得到的三株菌后,得到四株菌,分别命名为bs168-pspoiia-gfp,bs17-2-rap60-pspoiia-gfp,bs17-2-phr60-rap60-pspoiia-gfp,bs17-2-(phr60)*2-rap60-pspoiia-gfp。
(5)gfp荧光值的测定
将步骤(4)中构建的4株菌使用lb培养基培养,每个小时使用96孔板通过酶标仪检测荧光值的变化,荧光蛋白激发波长:395nm,发射波长509nm。结果如图2所示,仅表达rap60的菌株,荧光值很弱说明spo0a不能激活启动子pspoiia的表达,表达两个phr60的细胞,荧光强度明显高于表达一个拷贝的细胞,说明信号分子浓度越高激活spo0a磷酸化程度越高,越能够激活启动子的表达。因此,将菌株bs17-2-(phr60)*2-rap60作为接下来实验的出发菌株,将其命名为bs18,其基因型为bacillussubtilis168,p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbrp43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxsp43-fnipmena-mena::ganapmena-mena::thrcpmena-mena::dacapveg-kina-δpas-aδkinbphag-rap60(pnative-phr60)2::hag,用于后续的实验。
实施例5:spo0a-p调控基因表达与细胞浓度之间的关系
由于本发明基于细胞浓度与基因表达强度之间的关系,为了确定细胞浓度对spo0a-p的影响,检测构建好的群体响应phr60-rap60-spo0a与细胞生长之间的关系,在pht01质粒上使用pspoiia和pabrb启动子表达gfp蛋白,分别构建得到重组质粒pht01-pspoiia-gfp和pht01-pabrb-gfp,然后将重组质粒转入到实施例4中得到的菌株bs18中,检测随着细胞生长荧光值的变化,结果如图4,从图中可以看出当细胞生长到1.433左右时,启动子的转录能力受到spo0a-p的调控。
实施例6:突变启动子的获得
(1)上调启动子的获得
为了获得受phr60-rap60调控能力不同的启动子,将受spo0a-p上调的启动子pspoiia(序列如seqidno.11所示)上的四个spo0a结合位点分别进行突变,获得3个不同转录能力的启动子pspoiia(cs-3)、pspoiia(oa-3)、pspoiia(cs-1,3),将这些启动子融合gfp基因(序列如seqidno.15所示),使用荧光值的变化表征不同启动子的转录能力。
结果表明:突变后的pspoiia(cs-1,3)启动子(序列如seqidno.12所示)的转录能力比原始提高约7倍,因此将启动子pspoiia(cs-1,3),用于增强七烯甲萘醌合成途径中的关键基因表达。
(2)下调启动子的获得
将受spo0a-p下调的启动子pabrb(序列如seqidno.13所示)上的spo0a-p结合位点进行保守序列突变,获得3个不同转录能力的启动子pabrb(oa-2)、pabrb(cs-1)、pabrb(cs-2),并将这些启动子融合gfp基因(序列如seqidno.15所示),使用荧光值的变化表征不同启动子的转录能力。
结果表明:如图3所示,pabrb(cs-1)(序列如seqidno.14所示)启动子的转录能力仅有原来的18%,因此将启动子pabrb(cs-1),用于抑制七烯甲萘醌竞争途径关键基因的表达。
实施例7:重组菌bs19的构建
为了增加mk-7的合成,在重组菌bs18的基础上使用pabrb(cs-1)启动子原位替换丙酮酸激酶(pyk,genebankid:936596)基因和十一碳烯基焦磷酸合成酶酶(upps,genebankid:939640)基因的启动子获得重组菌株bs19,随着细胞的生长逐渐限制碳流量向竞争途径的流量。最终相关基因的转录水平如图6所示。具体构建过程如下:
(1)基因的克隆
①以枯草芽孢杆菌168基因组为模板,分别扩增得到pyk基因的上游同源臂序列pykup、pyk基因片段,人工合成pabrb(cs-1)启动子序列(序列如seqidno.14所示)、lox71-zeo-lox66盒序列(序列如seqidno.2所示),然后将四个片段pykup、lox71-zeo-lox66盒、pabrb(cs-1)启动子序列、pyk基因片段进行重叠延伸pcr,得到融合基因片段pykup-lox71-zeo-lox66-pabrb(cs-1)-pyk。
②以枯草芽孢杆菌168基因组为模板,分别扩增得到upps基因的上游同源臂序列uppsup、upps基因片段,人工合成pabrb(cs-1)启动子序列(序列如seqidno.14所示)、lox71-zeo-lox66盒序列(序列如seqidno.2所示),然后将四个片段uppsup、lox71-zeo-lox66盒、pabrb(cs-1)启动子序列、upps基因片段进行重叠延伸pcr,得到融合基因片段uppsup-lox71-zeo-lox66-pabrb(cs-1)-upps。
(2)同源重组
将步骤(1)中得到的融合片段转化到bs18感受态细胞中,并涂布于博来霉素抗性的lb平板,挑选平板上长出的单菌落,进行菌落pcr验证并进行测序。最后通过cre/lox重组系统,敲除菌株中博来霉素抗性基因zeo,最后得到菌株bacillussubtilis168,p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbrp43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxsp43-fnipmena-mena::lacapmena-mena::thrcpmena-mena::dacapveg-kina-δpas-aδkinbphag-rap60(pnative-phr60)2::hagpabrb(cs-1)-pyk::pykpabrb(cs-1)-upps::upps,将其命名为bs19。
实施例8:重组菌bs20的构建
采用与实施例7类似的方法,在实施例7得到的重组菌bs19的基础上使用实施例6中得到的pspoiia(cs-1,3)启动子表达七聚二烯丙基二磷酸合成酶(heps/t,genebankid:938998)基因和4-羟基-3-甲基丁-2-烯基二磷酸还原酶(isph,genebankid:937900)基因,获得重组菌株bs20,并随着细胞的生长,使碳通量流向mk-7的合成途径。最终相关基因的转录水平如图6所示。
(1)融合片段的获得
以枯草芽孢杆菌168基因组为模板,分别扩增得到heps/t基因的上游同源臂序列heps/tup、heps/t基因片段、lox71-zeo-lox66盒序列(序列如seqidno.2所示),然后将四个片段heps/tup、lox71-zeo-lox66盒、pspoiia(cs-1,3)启动子序列、heps/t基因片段进行重叠延伸pcr,得到融合基因片段heps/tup-lox71-zeo-lox66-pspoiia(cs-1,3)-heps/t。
以枯草芽孢杆菌168基因组为模板,分别扩增得到isph基因的上游同源臂序列isphup、isph基因片段,lox71-zeo-lox66盒序列(序列如seqidno.2所示),然后将四个片段isphup、lox71-zeo-lox66盒、pspoiia(cs-1,3)启动子序列、isph基因片段进行重叠延伸pcr,得到融合基因片段isphup-lox71-zeo-lox66-pspoiia(cs-1,3)-isph。
(2)同源重组
将步骤(1)中得到的融合片段转化到bs19感受态细胞中,并涂布于博来霉素抗性的lb平板,挑选平板上长出的单菌落,进行菌落pcr验证并进行测序。最后通过cre/lox重组系统,敲除菌株中博来霉素抗性基因zeo,最后得到菌株bacillussubtilis168,p43-menfp43-menbphbs-menep43-entcδdhbbphbs-tktp43-ppsaδptsgphbs-arogfbrp43-arokphbs-ispap43-heps/tphbs-kdpgp43-dxrp43-dxsp43-fnipmena-mena::ganapmena-mena::thrcpmena-mena::dacapveg-kina-δpas-aδkinbphag-rap60(pnative-phr60)2::hagpabrb(cs-1)-pyk::pykpabrb(cs-1)-upps::uppspspoiia(cs-1,3)-isph::isphpspoiia(cs-1,3)-heps/t::heps/t,将其命名为bs20。
实施例9:菌株的发酵
种子培养基配方(质量百分比):胰蛋白胨1%,酵母提取物0.5%,氯化钠1%。
摇瓶发酵培养基配方(质量百分比):大豆蛋白胨3-8%,葡萄糖3-8%,蔗糖3-8%,kh2po30.06%。
发酵罐发酵培养基配方(质量百分比):大豆蛋白胨3-8%,葡萄糖3-8%,蔗糖3-8%,kh2po30.06%。
(1)种子液制备:
将实施例1、7、8中构建得到的重组菌bs1~bs17、bs19、bs20分别接种到种子培养基中,在37℃、220rpm下培养12h得到枯草芽孢杆菌种子液。
(2)摇瓶发酵培养
将步骤(1)中得到的种子液以5%的接种量转入250ml摇瓶中,装液量为15ml。发酵条件40℃,220rpm培养6天,发酵结果如图7所示,bs19在发酵6天之后产量达到170mg/l,bs20在发酵6天之后产量达到360mg/l,其mk-7产量是枯草芽孢杆菌(bacillussubtilis)168的37.89倍。
(3)发酵培养
将步骤(1)中得到的种子液以15%的接种量转入发酵培养基,使用15-l发酵罐对重组菌株bs20进行发酵,发酵条件40℃,罐压0.05mpa,通气量10vvm,搅拌500rpm,通过流加葡萄糖的方式控制葡萄糖的浓度在5g/l。发酵结果如图8所示,产量达到230mg/l,且发酵后期发酵液的ph偏碱。
表6重组菌株发酵生产mk-7
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
sequencelisting
<110>江南大学
<120>应用枯草芽孢杆菌群体响应调控系统合成mk-7的方法
<160>37
<170>patentinversion3.3
<210>1
<211>846
<212>dna
<213>bacillussubtilis168
<400>1
gcattcatcgtcatatcatcatagctgagcggagggttttccatatcctgcggcgtttgt60
gagttcccatatttttcgacatcctgataggaatcctcactgtcatatggcgctctgatt120
tcttcatcatcatcaaattcaaattgcccgaatggtgtttcttcttcaatcggacggtct180
ttggaaaccacgtcctgtgacgaatactccgcgagggttgtggcagtcggaagcgcttca240
agtcgttcgaaagggatttcctttccgctgacttcacaaatgccatacgtaccgttttct300
atcgccttcaatgaatgctcaatgtcccgaaggtgctctctctcatgcaagtctagagcg360
atgtctttctcacgctcgtaaagttctgtcgcctgatcgccgggatggttgtcgtatgcc420
gaaagctcaccccacgaatcataaggaaaggctgagttaagctgaaaatgatcattgtct480
ttgaaacggtttaagatatcttttttcgtttgttccagttcattttttaaatgctgaagc540
tgttctttcgtaagcaatgtgatcgcctcgtttctctgtgatgcatacctttagtatgaa600
cagatcgcctgagaactttcataaatggcgggtggaggaatataggaggttttcctttta660
tggtaagcggatacaacctttgctatcagtggagaaagaaatttaagctttgtttctttt720
tcatttctgaaattaggtttataataggtaaggcaggccatttggactgcatgatctgtg780
tttgacacaaaggagacacaggtgtatggtattaaaagcattttaaggattatggaggaa840
tgtctc846
<210>2
<211>588
<212>dna
<213>人工合成
<400>2
gagcggataacaatttcacacaggaaacagctatgaccatgattacgaattcgagctcgg60
tacccggggatcctctagagataccgttcgtatagcatacattatacgaagttatcttga120
tatggctttttatatgtgttactctacatacagaaaggaggaactaaacatggccaagtt180
gaccagtgccgttccggtgctcaccgcgcgcgacgtcgccggagcggtcgagttctggac240
cgaccggctcgggttctcccgggacttcgtggaggacgacttcgccggtgtggtccggga300
cgacgtgaccctgttcatcagcgcggtccaggaccaggtggtgccggacaacaccctggc360
ctgggtgtgggtgcgcggcctggacgagctgtacgccgagtggtcggaggtcgtgtccac420
gaacttccgggacgcctccgggccggccatgaccgagatcggcgagcagccgtgggggcg480
ggagttcgccctgcgcgacccggccggcaactgcgtgcacttcgtggccgaggagcagga540
ctgaataacttcgtatagcatacattatacgaacggtaaatcgtcgac588
<210>3
<211>300
<212>dna
<213>bacillussubtilis168
<400>3
tgataggtggtatgttttcgcttgaacttttaaatacagccattgaacatacggttgatt60
taataactgacaaacatcaccctcttgctaaagcggccaaggacgctgccgccggggctg120
tttgcgtttttgccgtgatttcgtgtatcattggtttacttatttttttgccaaagctgt180
aatggctgaaaattcttacatttattttacatttttagaaatgggcgtgaaaaaaagcgc240
gcgattatgtaaaatataaagtgatagcggtaccattataggtaagagaggaatgtacac300
<210>4
<211>847
<212>dna
<213>bacillussubtilis168
<400>4
atggtgacaacggtgcagcgtacgttccgaaaggaagttctacatgcattacataaagcc60
aaagaagtcaaccatgctgtcttaataagctattcgagacaaatcgagtctcttgaccct120
ctatcatttttcaattacggagcaaaaaaatatacaggcaatcgatttttttggtcagat180
cctgaaagtgaattgacaatagtcggtcttggcaaagaagcggttttccagacaaatcaa240
aaaaacagcgagcggtatcgtgaggtttttgaacaatgggagcgctttaaaaagacggct300
tttcatatttatgaagaagaaaagctgcagcattctgcagtgggacctgtgttattcgga360
ggattttcttttgacccttgcgaagaaagaggttcacaatgggaccatttctcggaaggg420
gatttctttgtgcctgcgcttatgctgacgatgactgctgaaggcccgttcttaacagtt480
aacagatgggtaagcggaggagaagacgcagaagctgttttagaaggcttaaaagctttt540
gcggcggaatttatggttcccgatttcaagcaagaagatcaggctgtgattgcagcagcc600
gaagagctggataaggatgattggctgaaagcaatcgaaacggccacaagccaaattaaa660
gagaaacaatatgataaggttgttcttgcccgagagctgctgctcacgtttgacggtcca720
atccaaattgaaccggtgcttaaaacgcttctggacgatcagcagacaagctatgttttt780
gcaattgaacaagaaggcaaaacctttgtcggcgcgtctccggaaagactgatcaaaaga840
gacggcg847
<210>5
<211>328
<212>dna
<213>bacillussubtilis168
<400>5
cttaataatggaaaaggatcaaggaataggatgaaaaaaggaaaaaaaggaatattcgtt60
cggtaaatcaccttaaatccttgacgagcaagggattgacgctttaaaatgcttgatatg120
gctttttatatgtgttactctacatacagaaattcttcactttgttggacaaacattcct180
cagagtgcagtttttcttaaaaagccgtttaattgtctttctcttacttgctctcatttt240
tttctgagacaggtttagaatcagactgaactgtgaagaaatgataataaacgaactgaa300
tgtatccttttgggaggaggtgaaaggc328
<210>6
<211>1053
<212>dna
<213>人工合成
<400>6
atgaattatcagaacgacgatttacgcatcaaagaaatcaaagagttacttcctcctgtc60
gcattgctggaaaaattccccgctactgaaaatgccgcgaatacggttgcccatgcccga120
aaagcgatccataagatcctgaaaggtaatgatgatcgcctgttggttgtgattggccca180
tgctcaattcatgatcctgtcgcggcaaaagagtatgccactcgcttgctggcgctgcgt240
gaagagctgaaagatgagctggaaatcgtaatgcgcgtctattttgaaaagccgcgtacc300
acggtgggctggaaagggctgattaacgatccgcatatggataatagcttccagatcaac360
gacggtctgcgtatagcccgtaaattgctgcttgatattaacgacagcggtctgccagcg420
gcaggtgagtttctcgatatgatcaccctgcaatatctcgctgacctgatgagctggggc480
gcaattggcgcacgtaccaccgaatcgcaggtgcaccgcgaactggcatcagggctttct540
tgtccggtcggcttcaaaaatggcaccgacggtacgattaaagtggctatcgatgccatt600
aatgccgccggtgcgccgcactgcttcctgtccgtaacgaaatgggggcattcggcgatt660
gtgaataccagcggtaacggcgattgccatatcattctgcgcggcggtaaagagcctaac720
tacagcgcgaagcacgttgctgaagtgaaagaagggctgaacaaagcaggcctgccagca780
caggtgatgatcgatttcagccatgctaactcgtccaaacaattcaaaaagcagatggat840
gtttgtgctgacgtttgccagcagattgccggtggcgaaaaggccattattggcgtgatg900
gtggaaagccatctggtggaaggcaatcagagcctcgagagcggggagccgctggcctac960
ggtaagagcatcaccgatgcctgcatcggctgggaagataccgatgctctgttacgtcaa1020
ctggcgaatgcagtaaaagcgcgtcgcgggtaa1053
<210>7
<211>936
<212>dna
<213>bacillussubtilis168
<400>7
atgaaccaaacaaataagggtgagggtcagacagcgccgcaaaaagaaagcatggggcag60
atcctttggcagttaacccgtcctcatacgttaaccgcatcgtttgtgcctgtgctgctc120
ggaaccgttttggcgatgttttatgtgaaggttgatctgctgctgtttttggctatgctg180
ttttcttgcctatggatccagatcgcgacgaacttatttaatgaatactatgattttaaa240
cgcggattagatacagcagaatcagtcggaatcggaggggccattgtacgccacggaatg300
aagcctaaaacgattttgcaattagctcttgcctcatacgggattgccattttgctcggt360
gtctatatttgtgccagcagcagctggtggcttgcgctgatcggccttgtcggcatggcg420
atcggctacctgtatacaggcgggccgctgccgattgcgtacacgccgttcggtgaatta480
ttctcaggcatttgcatgggttcggtgtttgtgctgatttcgtttttcattcagacagat540
aagatcaatatgcagagcattttgatttccatcccgattgcgattcttgtcggcgcgatt600
aacctgtcgaacaacattcgtgatattgaagaggacaaaaaaggcggacgcaaaacattg660
gcgattttgatggggcataagggagctgttactctgttagctgcgtcgtttgccgtcgct720
tatatctgggttgtcggcttggttattaccggtgccgcaagcccatggctgtttgtcgtc780
tttttgagcgtgcctaagccggttcaggcagtgaagggcttcgtccagaacgaaatgccg840
atgaatatgattgtcgcaatgaaatcaacagcccaaacaaatacatttttcggattcctg900
ctctcgatcggattattgatcagctatttccgataa936
<210>8
<211>1537
<212>dna
<213>bacillussubtilis168
<400>8
cttgacagcgggtttttcatacagctgtttcattaattgcagcatatcaaaaattccctt60
cccgttttttcgacaatcacatgattatttgacgatcagatgctcaaaaagtttcatttt120
ttccctaatctccctcaaattaatatatcggcgtatgtttttgaaaatgaatctttatac180
ctgcaagggcaagaatggaatgtttcgagctgattcataataaaggaaaagatacctgga240
gtcaaaagatctttttaacttttaaaaagtgtaatgtacttaaaaaaatttttctgtacc300
tgttcgttcaagtgaaagcagaaacctaatgaaatcagggcgagaagcgtataataagaa360
agaaaacgccttcttgcggtatcttgtgaaataagggagaagtcattatgatagataagg420
aagcaaaaggagttttttattaagctgccggctgaaaaaaagtaagccttttaatagaaa480
ggaagaggaagatgaaccaaacaaataagggtgagggtcagacagcgccgcaaaaagaaa540
gcatggggcagatcctttggcagttaacccgtcctcatacgttaaccgcatcgtttgtgc600
ctgtgctgctcggaaccgttttggcgatgttttatgtgaaggttgatctgctgctgtttt660
tggctatgctgttttcttgcctatggatccagatcgcgacgaacttatttaatgaatact720
atgattttaaacgcggattagatacagcagaatcagtcggaatcggaggggccattgtac780
gccacggaatgaagcctaaaacgattttgcaattagctcttgcctcatacgggattgcca840
ttttgctcggtgtctatatttgtgccagcagcagctggtggcttgcgctgatcggccttg900
tcggcatggcgatcggctacctgtatacaggcgggccgctgccgattgcgtacacgccgt960
tcggtgaattattctcaggcatttgcatgggttcggtgtttgtgctgatttcgtttttca1020
ttcagacagataagatcaatatgcagagcattttgatttccatcccgattgcgattcttg1080
tcggcgcgattaacctgtcgaacaacattcgtgatattgaagaggacaaaaaaggcggac1140
gcaaaacattggcgattttgatggggcataagggagctgttactctgttagctgcgtcgt1200
ttgccgtcgcttatatctgggttgtcggcttggttattaccggtgccgcaagcccatggc1260
tgtttgtcgtctttttgagcgtgcctaagccggttcaggcagtgaagggcttcgtccaga1320
acgaaatgccgatgaatatgattgtcgcaatgaaatcaacagcccaaacaaatacatttt1380
tcggattcctgctctcgatcggattattgatcagctatttccgataataaaaaagaccgc1440
tcgtttcatgcggtctttttttgttacaatcgaccgcattttgtaaaaaaattcatagaa1500
ccttgcagcagacagggacgtctagtacatggacagc1537
<210>9
<211>1490
<212>dna
<213>人工合成
<400>9
tggaacaggatgaagaagaaacaggccatcaatccctaaactgcgaaaaacatgaaatcg60
aacctgcaagcccggaatcgactacatatataacggatgattatgaacggttggttgaaa120
atctcccgagtccgctatgcatcagtgtcaaaggcaagatcgtctatgtaaacagcgcga180
tgctttcaatgctgggagccaaaagcaaggatgctattattggtaaatcgtcctatgaat240
ttattgaagaagaatatcatgatatcgtgaaaaacaggattatacgaatgcaaaaaggaa300
tggaagtcggaatgattgaacagacgtggaaaaggcttgatggcacacctgttcatttag360
aagtgaaagcatccccgaccgtctacaaaaaccagcaggctgagctgctgctgctgatcg420
atatctcttcaaggaaaaaattccaaaccatcctgcaaaaaagccgtgaacgatatcagc480
tgctgattcaaaattccattgataccattgcggtgattcacaatggaaaatgggtattta540
tgaatgaatcgggaatttccctgtttgaagcggctacatatgaggatttaattggcaaaa600
acatatacgatcagctgcatccttgcgatcacgaggatgtaaaagagagaatccaaaaca660
ttgccgagcaaaaaacagaatctgaaattgtcaagcaatcctggttcacctttcagaaca720
gggtcatctatacggagatggtctgcattccgacgaccttttttggtgaagcggccgtcc780
aggtcattcttcgggacatctcagagagaaaacaaacagaagaattgatgctgaaatcgg840
aaaaattatcaatcgcagggcagctcgcggcgggaatcgcccatgagatccgcaaccctc900
ttacagcgatcaaaggatttttacagctgatgaaaccgacaatggaaggcaacgaacatt960
actttgatattgtgttttctgaactcagccgtatcgaattaatactcagtgaactgctca1020
tgctggcgaaacctcagcaaaatgctgtcaaagaatatttgaacttgaaaaaattaattg1080
gtgaggtttcagccctgttagaaacgcaggcgaatttaaatggcatttttatcagaacaa1140
gttatgaaaaagacagcatttatataaacggggatcaaaaccaattaaagcaggtattca1200
ttaatttaatcaaaaatgcagttgaatcaatgcctgatgggggaacagtagacattatca1260
taaccgaagatgagcattctgttcatgttactgtcaaagacgaaggggaaggtatacctg1320
aaaaggtactaaaccggattggagagccatttttaacaacaaaagaaaaaggtacggggc1380
ttggattaatggtgacatttaatatcattgaaaaccatcagggagttatacatgtggaca1440
gccatcctgaaaaaggcacagcgtttaaaatttcatttccaaaaaaataa1490
<210>10
<211>320
<212>dna
<213>bacillussubtilis168
<400>10
tatgggaagtgctccgtaatacgctgacaagagagaaagggcttggaggtattgaaacaa60
gaggagttctgagaattggtatgccttataagtccaattaacagttgaaaacctgcatag120
gagagctatgcgggttttttattttacataatgatacataatttaccgaaacttgcggaa180
cataattgaggaatcatagaattttgtcaaaataattttattgacaacgtcttattaacg240
ttgatataatttaaattttatttgacaaaaatgggctcgtgttgtacaataaatgtagtg300
aaaggaggtgaaatgtacac320
<210>11
<211>574
<212>dna
<213>bacillussubtilis168
<400>11
ctggtgtagacggcgtaaaaacaggctatacaggcgaagcgaaatattgtctgactgctt60
cggctaaaaaaggaaacatgcgggccatagcggttgtattcggagcgagcacgcctaaag120
aaagaaacgcgcaagtgacaaaaatgcttgacttcgcctttagccaatatgaaacgcatc180
ctttatataaacgaaatcaaacagtagcaaaagtaaaggtcaaaaaagggaaacaaaaat240
ttatcgaactcactacatctgagccgatttcaatattgacgaaaaaaggcgaggatatga300
acgatgtgaaaaaagaaatcaagatgaaggacaatattagtgctccgattcaaaaaggcc360
aagagcttggcactcttgttctgaaaaaggatggagaagtactcgctgaaagtcctgttg420
ctgcaaaagaagatatgaagaaagccgggtttatcacattcttaaagcggacgatgggag480
actggacaaaatttaagtaattatgaccactagtttgtgaaggaattcattcatcgaaac540
actcattatccgatcatatcaaggaggaatgagc574
<210>12
<211>603
<212>dna
<213>人工合成
<400>12
ctggtgtagacggcgtaaaaacaggctatacaggcgaagcgaaatattgtctgactgctt60
cggctaaaaaaggaaacatgcgggccatagcggttgtattcggagcgagcacgcctaaag120
aaagaaacgcgcaagtgacaaaaatgcttgacttcgcctttagccaatatgaaacgcatc180
ctttatataaacgaaatcaaacagtagcaaaagtaaaggtcaaaaaagggaaacaaaaat240
ttatcgaactcactacatctgagccgatttcaatattgacgaaaaaaggcgaggatatga300
acgatgtgaaaaaagaaatcaagatgaaggacaatattagtgctccgattcaaaaaggcc360
aagagcttggcactcttgttctgaaaaaggatggagaagtactcgctgaaagtcctgttg420
ctgcaaaagaagatatgaagaaagccgggtttatcacattcttaaagcggacgatgggag480
actggacaaaatttaagtaattatttgtcgaaaatgaccactagttttgtcacggtgaag540
gaattcattctttgtcgaaaaatcgaaacactcattatccgatcatatcaaggaggaatg600
agc603
<210>13
<211>580
<212>dna
<213>bacillussubtilis168
<400>13
catctcctgcgactgtcgtatatgcatggccgatatgtaattttccgctcggataataaa60
tcggtgttgtaatgtaaaatgtattgttttcttgcggcatcttgaaacctcctaatcaga120
atgtcaaaaagctcccgcccaaaggacgagagcggcgataagtgcaaatcatatatgaat180
attatttatatttatcctttatctatgataccatcttagcctttaagatcaagaatatat240
acatacgccctgaaaaagaataattaaaattaaaaatctttaatgaaaataaaaaaagtc300
tgttcgtgtcgaattttgtcgtatataatcaaatgaaataaaaaactttggttcgaaagt360
cttgatttaaaaggatttttagtaggataatagctttattaaatatttataaaatgctgt420
tatttcggtagtttccaagacattactgactataagaactaattcttacaatcaatagta480
aacaaaatgattgacgattattggaaaccttgttatgctatgaaggtaaggattttaaga540
aaaatataatttaaacaaataagtatctcttgggaggaga580
<210>14
<211>601
<212>dna
<213>人工合成
<400>14
catctcctgcgactgtcgtatatgcatggccgatatgtaattttccgctcggataataaa60
tcggtgttgtaatgtaaaatgtattgttttcttgcggcatcttgaaacctcctaatcaga120
atgtcaaaaagctcccgcccaaaggacgagagcggcgataagtgcaaatcatatatgaat180
attatttatatttatcctttatctatgataccatcttagcctttaagatcaagaatatat240
acatacgccctgaaaaagaataattaaaattaaaaatctttaatgaaaataaaaaaagtc300
tgttcgtgtcgaattttgtcgtatataatcaaatgaaataaaaaactttggttcgaaagt360
cttgatttaaaaggatttttagtaggataatagctttattaaatatttataaaatgctgt420
tatttcggtagtttccaagacattactgactataagaactaattcttacaatcaatagta480
aacaaaatgattgacgattattggaaaccttgttatgctatgaaggtaaggattttttgt540
cgaaaataatttgtcgaaagaaaaatataatttaaacaaataagtatctcttgggaggag600
a601
<210>15
<211>717
<212>dna
<213>人工合成
<400>15
atgggtaagggagaagaacttttcactggagttgtcccaattcttgttgaattagatggt60
gatgttaatgggcacaaattttctgtcagtggagagggtgaaggtgatgcaacatacgga120
aaacttacccttaaatttatttgcactactggaaagctgcctgttccttggccaacactt180
gtcactactcttacttatggtgttcaatgcttttcaagatacccagatcatatgaagcgg240
cacgacttcttcaagagcgccatgcctgagggatacgtgcaggagaggaccatcttcttc300
aaggacgacgggaactacaagacacgtgctgaagtcaagtttgagggagacaccctcgtc360
aacagaatcgagcttaagggaatcgatttcaaggaggacggaaacatcctcggccacaag420
ttggaatacaactacaactcccacaacgtatacatcatggcagacaaacaaaagaatgga480
atcaaagttaacttcaaaattagacacaacattgaagatggaagcgttcaactagcagac540
cattatcaacaaaatactccaattggcgatggccctgtccttttaccagacaaccattac600
ctgtccacacaatctgccctttcgaaagatcccaacgaaaagagagaccacatggtcctt660
cttgagtttgtaacagctgctgggattacacatggcatggatgaactgtacaaataa717
<210>16
<211>564
<212>dna
<213>人工合成
<400>16
aactgctgaacttttggatatcgataatattcaagacgtagaagtcatgacaatattgac60
tatggcagagccatttgaaaagtctactgcgaatttattggctcccattattgtgaatcg120
caagaacatgatggctaagcaagtcgttttacacgactcctcatatacgacaaagcatcc180
gattggaggagaatcatgctagttttatcgcggaaaataaacgaagcgattcaaataggt240
gctgatattgaagtaaaagtgattgcggttgaaggggatcaagtgaagcttggaattgac300
gccccaaagcatattgatattcacaggaaagaaatttacttgaccattcaggaagaaaat360
aaccgtgcagcagcgttatccagcgatgtgatctccgcattatcctcacaaaaaaagtga420
ggatttttttatttttgtattaacaaaatcagagacaatccgatattaatgatgtagccg480
ggaggaggcgcaaaagactcagccagttacaaaataagggcacaaggacgtgccttaaca540
acatattcagggaggaacaaaaca564
<210>17
<211>396
<212>dna
<213>bacillussubtilis168
<400>17
gctttttcggaaaagcaatggaacaggcggaagaatttaacgattctctgtttcaagatc60
tgcttaacgttctgaaagccctgtttatcgaaacaggctcaagacagaaagtgatgaacg120
ctcttgaagccttacgcacaggccaaggatacccgtactttgaagaacttgcactgatcg180
ctgccgaattttatacaatggataaacgcatggaagattcaatctacttttacaacgaaa240
tggtttgtgcgcaaagacagatccaacgcggagattttctgtatgaagtttaaaggctta300
ttttctgctgtcctgattgtttcactgcttgtgggcgcaggatactcatttgttcatcat360
gatgaagtttcagtggcaagcagaaatgcgacataa396
<210>18
<211>30
<212>dna
<213>人工合成
<400>18
gcattcatcgtcatatcatcatagctgagc30
<210>19
<211>59
<212>dna
<213>人工合成
<400>19
tgtgaaattgttatccgctcgagacattcctccataatccttaaaatgcttttaatacc59
<210>20
<211>27
<212>dna
<213>人工合成
<400>20
tgataggtggtatgttttcgcttgaac27
<210>21
<211>28
<212>dna
<213>人工合成
<400>21
gtgtacattcctctcttacctataatgg28
<210>22
<211>53
<212>dna
<213>人工合成
<400>22
tttaaggattatggaggaatgtctcgagcggataacaatttcacacaggaaac53
<210>23
<211>30
<212>dna
<213>人工合成
<400>23
aatcatacctaccacaatatcatgctcaag30
<210>24
<211>22
<212>dna
<213>人工合成
<400>24
ttccatatcctgcggcgtttgt22
<210>25
<211>25
<212>dna
<213>人工合成
<400>25
acgaagttattcagtcctgctcctc25
<210>26
<211>25
<212>dna
<213>人工合成
<400>26
cttgacagcgggtttttcatacagc25
<210>27
<211>23
<212>dna
<213>人工合成
<400>27
gctgtccatgtactagacgtccc23
<210>28
<211>19
<212>dna
<213>人工合成
<400>28
actggcttggacggtggtt19
<210>29
<211>47
<212>dna
<213>人工合成
<400>29
gtatgaaaaacccgctgtcaagtcctccttgttctcttagcccttcg47
<210>30
<211>39
<212>dna
<213>人工合成
<400>30
ctgggaaaaccctggcggctgatgctccgctcgatatgg39
<210>31
<211>20
<212>dna
<213>人工合成
<400>31
cgccaatcgctgacctgaag20
<210>32
<211>24
<212>dna
<213>人工合成
<400>32
ctggtgtagacggcgtaaaaacag24
<210>33
<211>32
<212>dna
<213>人工合成
<400>33
gctcattcctccttgatatgatcggataatga32
<210>34
<211>41
<212>dna
<213>人工合成
<400>34
tatcaaggaggaatgagcatgggtaagggagaagaactttt41
<210>35
<211>42
<212>dna
<213>人工合成
<400>35
ccaagcttctgcagttatttgtacagttcatccatgccatgt42
<210>36
<211>36
<212>dna
<213>人工合成
<400>36
ctgtacaaataactgcagaagcttggcgtaatcatg36
<210>37
<211>44
<212>dna
<213>人工合成
<400>37
gccgtctacaccagttgctcaaaaaaatctcggtcagatgttac44
- 还没有人留言评论。精彩留言会获得点赞!