基于石榴全基因组序列开发的SSR核心引物组及其应用的制作方法
本发明涉及分子生物
技术领域:
,具体来说是利用石榴全基因组序列开发ssr核心引物组及在石榴品种鉴定、指纹图谱构建及遗传多样性评价与系统发育研究中的应用。
背景技术:
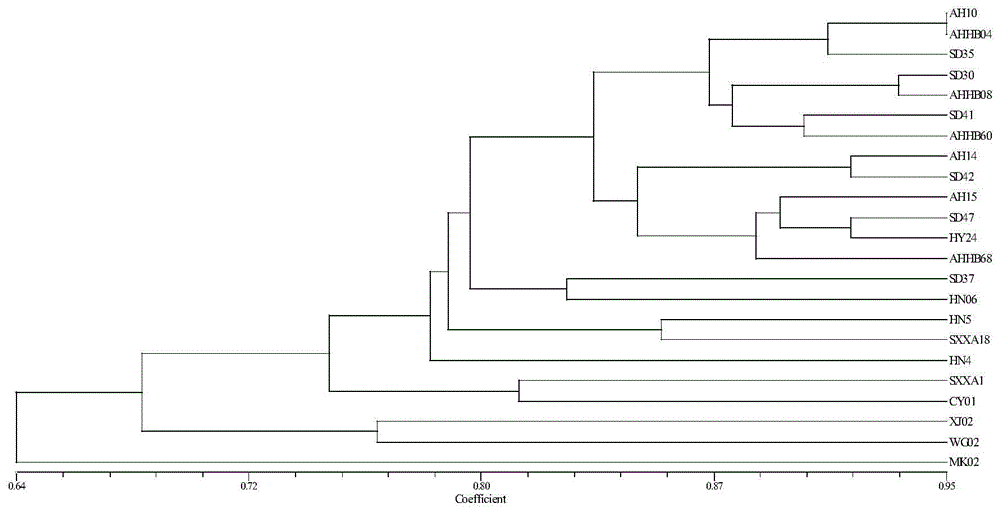
:微卫星(simplesequencerepeats,ssr)标记,是指以1~6个核苷酸为单位,广泛存在于基因组中的简单重复序列。ssr具有分布广、共显性遗传、多态性高、检测方便且稳定的特点,被广泛应用于品种鉴定、遗传多样性评价以及系统发育关系研究、遗传图谱构建及qtls定位等诸多研究领域。前期,ssr标记的开发主要基于基因组dna文库或者富含特异微卫星的文库,该方法工作量大且得到的标记数量比较有限;近年来,随着全基因组测序工作的完成,基于全基因组序列开发ssr标记,可获得的标记数量丰富,覆盖整个基因组,且在基因组上分布均匀,被广泛应用于多种已测序植物中。石榴是最早被人类认识可以食用的果树,原产于波斯地区(今伊朗)一带,公元前5世纪开始被人类驯化。据记载,石榴在张骞出使西域时传入我国,在我国已有2000多年的栽培的历史。石榴遗传资源丰富,据《中国果树志(石榴卷)》记载,我国现有石榴资源300多份,根据风味可分为甜石榴和酸石榴,根据籽粒硬度可分为硬籽石榴和软籽石榴,根据其利用部位可分为观赏石榴和食用石榴,根据颜色分可为红皮、白皮、黄皮以及粉皮石榴等。由于石榴独特的营养、保健、观赏以及显著的经济和生态效益,石榴的栽培范围逐渐扩大,地方间引种频繁;但由此也带来了石榴品种间混杂严重,给石榴生产者和育种者带来了新的挑战。基于石榴栽培历史悠久、资源丰富、品种交流频繁的现状,开展石榴品种鉴定、指纹图谱构建、遗传多样性评价及系统发育关系研究均有重要的现实意义,然而该工作的有效开展有赖于多态性高、重复性好、标记稳定的分子标记开发。基于此,根据已有的石榴基因组序列,在全基因组上开发ssr标记核心引物具有重要的价值。技术实现要素:本发明的目的是在于提供利用全基因组数据快速开发ssr标记,挑选引物组,这些引物具有扩增稳定、电泳条带清晰、多态性丰富的优点,可有效用于石榴品种鉴定、dna指纹图谱构建及遗传多样性评价与系统发育研究等领域。本发明第一方面提供了一种基于石榴全基因组序列开发的ssr核心引物组,该引物组包括11对引物:pg080、pg130、pg139、pg152、pg153、pg140、pg098、pg070、pg077、pg090、pg093,其核苷酸序列依次如seqidno.1-22所示。本发明第二方面提供了上述基于石榴全基因组序列开发的ssr核心引物组的开发方法,包括如下步骤:(1)石榴基因组dna提取:采用ctab法提取dna,加入50μl0.1m的te缓冲液,溶解过夜,-20℃保存备用;(2)石榴全基因组数据从ddbj/ena/genbank数据库下载,序列号mtkt00000000;利用misa软件在全基因组范围内挖掘不同重复单元ssr位点,ssr查找的标准为二核苷酸、三核苷酸、四核苷酸、五核苷酸及六核苷酸的重复次数分别为11、8、6、5、4;(3)ssr引物设计上述获得的ssr位点,在每个染色体上随机选择5对,利用重复序列两端的保守侧翼序列,用primer3.0设计引物,引物设计的条件是:pcr产物长度为100~350bp;退火温度tm为50~70℃,保证两引物间tm值相差不超过4℃;gc%含量为40~65%;引物长度为18~28bp;为了保证引物的特异性,用于设计引物的保守侧翼序列与微卫星位点至少间隔20~23个碱基;利用该方法,成功设计出45对引物;(4)引物筛选采用合成的45对引物,通过对6个不同产地有代表性的石榴品种的基因组dna进行扩增,pcr扩增采用20μl的反应体系,含1.0ngdna,0.4μm正向引物,0.4μm反向引物,4mmmgcl2,400μmdntps,1.0utaq-dna酶,加ddh20至总体积20μl;pcr采用touchdown反应,反应条件如下:5min,95℃;接下来11个循环,每个循环后退火温度下降0.8℃{30s,95℃;30s,65℃;50s,72℃};22个循环{30s,95℃;30s,55℃;50s,72℃};最后72℃延伸8min;pcr产物采取毛细管电泳确定条带大小;根据扩增结果,筛选可稳定扩增、带型清晰且多态性丰富的引物共11对。本发明第三方面提供了上述基于石榴全基因组序列开发的ssr核心引物组在石榴品种鉴定方面的应用。上述品种鉴定是采用11对引物进行荧光毛细管电泳检测,根据毛细管电泳的检测结果,品种间差异位点数≥3为不同品种,品种间差异位点数<3为近似品种。本发明第四方面提供了上述基于石榴全基因组序列开发的ssr核心引物组在石榴dna指纹图谱构建方面的应用。本发明第五方面提供了遗传多样性评价与系统发育研究石榴遗传多样性评价与系统发育研究方面的应用。本发明的有益效果:1.本发明新开发了一批石榴ssr核心引物,解决了石榴基因组ssr标记相对缺乏的问题。2.本发明建立了石榴全基因组ssr核心引物组的开发方法,相对于其它的石榴标记引物开发方法,本方法具有方法简单、成本低廉、开发效率高等特点,且开发的标记数量丰富,能覆盖整个基因组,有着重要的实用价值。3.本方明开发的11对ssr核心引物具有多态性高,重复性好、标记稳定、带型清晰易判读等优点,可应用于石榴品种鉴定、dna指纹图谱构建及遗传多样性评价与系统发育研究等方面,为石榴分子辅助育种提供了新的工具,具有良好的应用前景。附图说明图1为23个石榴品种聚类图。具体实施方式下面结合实施例和附图对本发明做更进一步地解释。下列实施例仅用于说明本发明,但并不用来限定本发明的实施范围。1.石榴基因组dna提取(1)选取不同产区的石榴品种资源23份23份石榴品种的名称及来源见表1。表123份石榴品种信息(2)ctab法提取基因组dna称取0.2~0.3g新鲜的叶片,加入液氮迅速研磨成粉状,将粉末转入2.0ml离心管中;加入1.0ml65℃预热的3×ctab提取液65℃水浴1h;室温下12000rpm离心10min;吸取上清液于2.0ml离心管中;加入于上清液等体积的苯酚/氯仿/异戊醇(25:24:1,v/v/v),轻轻颠倒混匀成乳浊液;12000r/min离心8min,取上清液,加入等体积的氯仿/异戊醇(24:1,v/v),混匀后12000r/min离心8min;取上清液加入等体积冷冻的异丙醇和10μl3m醋酸钠,置于-20℃条件下沉淀半个小时;12000r/min离心8min,小心弃上清液,dna留在离心管中;加入75%的乙醇洗2次,再用无水乙醇洗一次,离心后弃无水乙醇;室温下晾干,加入50μlte缓冲液(0.1m),溶解过夜,20℃保存备用。2.石榴基因组ssr引物的开发(1)石榴全基因组数据从ddbj/ena/genbank数据库下载,序列号mtkt00000000。利用misa(microsatellitesidentificationtool,http://pgrc.ipk-gatersleben.de/misa)软件在全基因组范围内挖掘不同重复单元ssr位点,ssr查找的标准为二核苷酸、三核苷酸、四核苷酸、五核苷酸及六核苷酸的重复次数分别为11、8、6、5、4。(2)ssr引物设计上述获得的ssr位点,在每个染色体上随机选择5对,利用重复序列两端的保守侧翼序列,用primer3.0设计引物,引物设计的条件是:pcr产物长度为100~350bp;退火温度(tm)为50~70℃,保证两引物间tm值相差不超过4℃;gc%含量为40~65%;引物长度为18~28bp;引物5’端最好是g/c,3’端避免出现a。为了保证引物的特异性,用于设计引物的保守侧翼序列与微卫星位点至少间隔20~23个碱基。利用该方法,成功设计出45对引物,引物由生工生物工程(上海)股份有限公司合成。(3)引物筛选采用合成的45对引物,对6个不同产地有代表性的石榴品种(ahhb04、sxxa1、cy01、xj02、hn4、sd47)的基因组dna进行扩增,根据扩增结果,筛选可稳定扩增、带型清晰且多态性丰富的引物,共11对(表2)。表211对引物序列(4)利用11对引物对23份石榴品种进行pcr扩增及毛细管电泳pcr扩增采用20μl的反应体系,含1.0ngdna,0.4μm正向引物,0.4μm反向引物,4mmmgcl2,400μmdntps,1.0utaq-dna酶,加ddh20至总体积20μl。pcr采用touchdown反应,反应条件如下:5min,95℃;接下来11个循环,每个循环后退火温度下降0.8℃{30s,95℃;30s,65℃;50s,72℃};22个循环{30s,95℃;30s,55℃;50s,72℃};最后72℃延伸8min。pcr产物用毛细管电泳确定片断大小。毛细管电泳按照以下步骤进行,将6-fam或hex荧光标记的pcr产物用超纯水稀释30倍,吸取1μl产物加入到dna分析仪专用深孔板孔中。板中各孔分别加入0.1μlliz500分子量内标和8.9μl去离子甲酰胺。95℃变性5min,于4℃冷却10min。瞬时离心10s后置放到dna分析仪上(abi3730xl)进行自动荧光检测。(5)结果记录及分析纯合位点的等位变异大小数据记录为x/x,其中x为该位点等位变异的大小;杂合位点的等位变异数据记录为x/y,其中x、y为该位点上两个不同的等位变异,小片段在前,大片段在后。构建的23份石榴资源的指纹图谱见表3。表323份石榴品种的指纹图谱数据samplepg098pg070pg130pg090pg152pg153pg139pg077pg093pg140pg080ah10213/229224/224158/158222/226142/154157/157158/162153/159164/170154/154157/183ah14213/229224/224158/158222/226154/154157/157162/162153/153164/166154/154157/183ah15229/229224/224158/158222/222154/154157/157158/162153/161158/166154/156157/157ahhb04213/213224/224158/158222/222142/154157/157158/162153/159164/170154/154157/157ahhb08229/229224/224158/158222/226142/142163/163162/162159/159164/170154/154157/183ahhb60213/229224/224158/158226/226142/142163/163158/158153/153164/164154/154157/183ahhb68229/229202/224158/158222/226154/154157/157162/162153/161156/170154/154157/157sd35213/229224/224158/158222/222142/154163/163158/162153/153170/170154/154157/157sd30229/229224/224158/158222/226142/154163/163158/158157/159164/170154/154157/183sd41229/229224/224158/158222/226142/142157/163158/162153/153164/166154/154183/183sd42213/229224/224158/158222/226154/154157/157160/162137/153158/164154/156157/183sd47229/229202/224158/158222/226142/154157/157158/158153/153158/158154/154157/157sd37229/229202/224158/158226/226142/154157/163158/162137/159156/164154/156153/157hn5229/231224/224158/158222/226142/154157/157158/160153/159166/166154/156157/157hn06227/229202/202150/158226/226142/154157/157154/158137/137156/164154/154157/157hn4217/217202/224150/158222/226142/154157/157154/158153/161164/164154/154189/189sxxa18231/231224/224158/158222/222142/142157/157160/160159/161166/166154/156161/183sxxa1215/229222/224158/158226/226142/142157/157154/154137/161164/164154/154183/183cy01229/229224/224158/158224/224142/142157/157154/154137/159160/160148/148140/140xj02233/233202/202150/150226/226142/154157/157154/154137/159164/164154/154157/157wg02227/229224/224148/156226/226142/154157/157158/158137/159162/164156/156153/153mk02213/213202/224158/158224/224142/142157/157154/154159/159158/158154/154153/153hy24229/229202/202158/158222/222154/154157/157158/162153/153158/170154/154157/157品种间差异位点数≥3为不同品种;品种间差异位点数〈3为近似品种,比较23份石榴材料的指纹数据,发现任意两个材料间的差异位点数都大于3,说明11对核心引物可以有效鉴别这些石榴资源。利用ntsys-pcv2.10e软件计算品种间的遗传相似性系数,用upgma法进行聚类分析,绘制树状图(图1)。序列表<110>安徽省农业科学院园艺研究所<120>基于石榴全基因组序列开发的ssr核心引物组及其应用<160>22<170>siposequencelisting1.0<210>1<211>24<212>dna<213>pg080的正向引物(人工序列)<400>1ctgactgttgcagagagtaggctg24<210>2<211>24<212>dna<213>pg080的反向引物(人工序列)<400>2aggaggtgaaacaacgaatagctg24<210>3<211>24<212>dna<213>pg130的正向引物(人工序列)<400>3ctcatatggcgattctctgtcctt24<210>4<211>24<212>dna<213>pg130的反向引物(人工序列)<400>4aagttcgataaattgcactggtgg24<210>5<211>22<212>dna<213>pg139的正向引物(人工序列)<400>5gtttccttccctcaacccaaaa22<210>6<211>24<212>dna<213>pg139的反向引物(人工序列)<400>6agtgggattttaccaagtcgaaca24<210>7<211>21<212>dna<213>pg152的正向引物(人工序列)<400>7catcagaatcgtccccttgtg21<210>8<211>24<212>dna<213>pg152的反向引物(人工序列)<400>8cagagagaagaagagagaccgagc24<210>9<211>24<212>dna<213>pg153的正向引物(人工序列)<400>9gtgtttgatgctcccatttcattt24<210>10<211>24<212>dna<213>pg153的反向引物(人工序列)<400>10gccttcaacggtctttcttcttct24<210>11<211>24<212>dna<213>pg140的正向引物(人工序列)<400>11caagaaagtgtgtgagcgattgat24<210>12<211>24<212>dna<213>pg140的反向引物(人工序列)<400>12ccaaaccttacccctctctctctc24<210>13<211>24<212>dna<213>pg098的正向引物(人工序列)<400>13tgccttcttaaggacttcaccaac24<210>14<211>24<212>dna<213>pg098的反向引物(人工序列)<400>14ctaacctcatgcacttgtcatcca24<210>15<211>24<212>dna<213>pg070的正向引物(人工序列)<400>15cacctctgcttcagcaaacaaata24<210>16<211>24<212>dna<213>pg070的反向引物(人工序列)<400>16caactcaacacaatatccaaccca24<210>17<211>24<212>dna<213>pg077的正向引物(人工序列)<400>17gtcagtctcctccttcttcaatgg24<210>18<211>23<212>dna<213>pg077的反向引物(人工序列)<400>18agacgaagcacctgagaaggaat23<210>19<211>28<212>dna<213>pg090的正向引物(人工序列)<400>19attcttttatactaaccaaaatttgcga28<210>20<211>22<212>dna<213>pg090的反向引物(人工序列)<400>20atgtcatgagaggacccacaaa22<210>21<211>24<212>dna<213>pg093的正向引物(人工序列)<400>21cgtcaataggacgtccctgagata24<210>22<211>24<212>dna<213>pg093的反向引物(人工序列)<400>22gatgacgtggcagagtaagagagc24当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1