用于肝癌检测的组合标志物及其应用的制作方法
本发明属于生物医学领域,具体涉及用于肝癌检测的组合标志物及其应用。
背景技术:
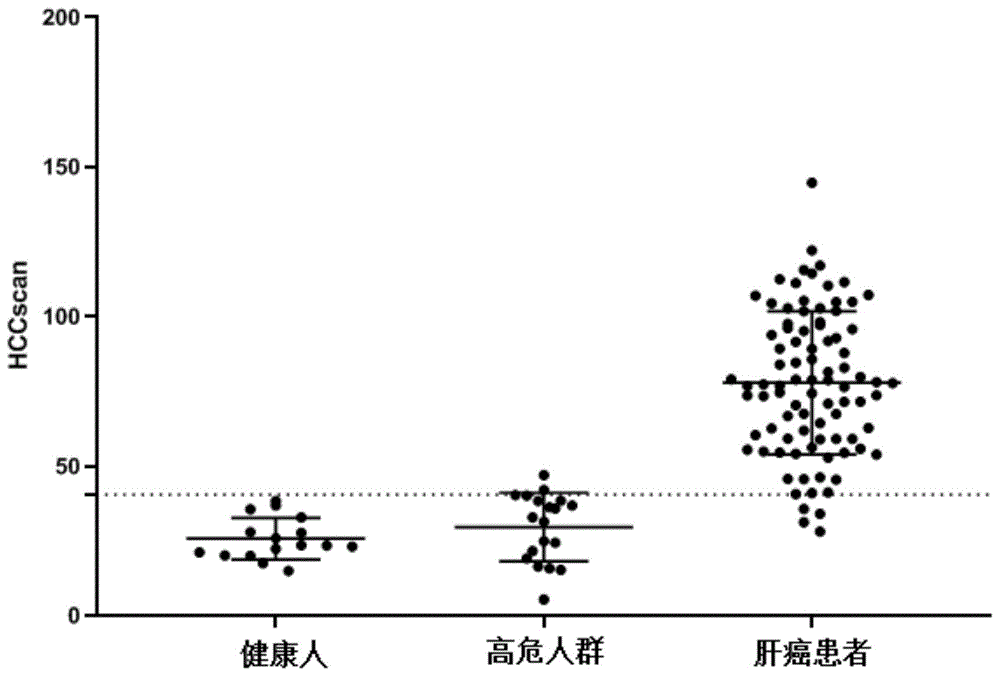
:目前,肝癌的诊疗仍然面临着重大问题,例如早期诊断困难、复发转移率高、放化疗耐受和缺少有效治疗靶点等。其实对于任何癌种,早筛始终是抗击癌症的关键性一步。肝癌早筛的常规监测筛查指标主要包括甲胎蛋白(afp)和肝脏超声检查(us)。综合国内外的报道和临床实际,afp诊断敏感性在60%左右,漏诊率为40%,特异性在80%左右,误诊率为20%,这也是美国肝病研究学会(aasld)指南不再将afp作为筛查指标的一个重要原因。超声检查对于大于20mm的结节,诊断符合率尚为84%,但对于肝内5-20mm的结节,超声的敏感性就只有33%。因此,迫切需要研发新的肝癌早筛生物标记物。通过液体活检开发癌症早筛的技术最早在美国兴起,包括ctdna、ctc和外泌体。相比传统的组织切片检测,其侵入性和副作用都要小得多,因此为癌症早筛带来了新的希望。其中ctdna是患者血液中游离的来自肿瘤的dna,属于高敏感性、高特异性的肿瘤标志物。其相当于肿瘤细胞释放到血液中的“身份指纹”,因其携带有与原发肿瘤组织相一致的基因背景,因此理论上可以利用ctdna对肝癌进行早筛。dna甲基化是一种重要的表观遗传学标记信息,也是最早发现的dna修饰途径之一。在肝癌发生过程中,dna甲基化改变是一个早期事件,包括肿瘤抑制基因甲基化水平升高或者原癌基因甲基化降低,且该变化早于驱动基因的突变。因此,甲基化模式的改变被认为是最先能检测到的与肿瘤发生密切相关的恶化指标,为肝癌早筛提供了一个筛查窗。技术实现要素:本发明的目的是如何进行肝癌检测。本发明首先保护一种用于肝癌检测的组合标志物,包括ak055957、bdh1、c6orf223、dab2ip、f12、grasp、lrrc4、nfix、oplah、ppfia1、psd4和tbx15的甲基化程度。所述用于肝癌检测的组合标志物,具体可由ak055957、bdh1、c6orf223、dab2ip、f12、grasp、lrrc4、nfix、oplah、ppfia1、psd4和tbx15的甲基化程度组成。上述任一所述组合标志物在制备肝癌检测试剂盒中的应用也属于本发明的保护范围。本发明还保护一种肝癌检测试剂盒,可包括上述任一所述组合标志物的检测试剂。上述任一所述组合标志物的检测试剂可包括用于检测所述组合标志物中各个基因甲基化水平的引物和/或探针。上述任一所述组合标志物的检测试剂具体可由用于检测所述组合标志物中各个基因甲基化水平的引物和/或探针组成。上述任一所述用于检测组合标志物中各个基因甲基化水平的引物分别可为seqidno:1所示的引物ak055957-f、seqidno:3所示的引物ak055957-r、seqidno:4所示的引物bdh1-f、seqidno:6所示的引物bdh1-r、seqidno:7所示的引物c6orf223-f、seqidno:9所示的引物c6orf223-r、seqidno:10所示的引物dab2ip-f、seqidno:12所示的引物dab2ip-r、seqidno:13所示的引物f12-f、seqidno:15所示的引物f12-r、seqidno:16所示的引物grasp-f、seqidno:18所示的引物grasp-r、seqidno:19所示的引物lrrc4-f、seqidno:21所示的引物lrrc4-r、seqidno:22所示的引物nfix-f、seqidno:24所示的引物nfix-r、seqidno:25所示的引物oplah-f、seqidno:27所示的引物oplah-r、seqidno:28所示的引物ppfia1-f、seqidno:30所示的引物ppfia1-r、seqidno:31所示的引物psd4-f、seqidno:33所示的引物psd4-r、seqidno:34所示的引物tbx15-f和seqidno:36所示的引物tbx15-r。上述任一所述用于检测组合标志物中各个基因甲基化水平探针分别可为seqidno:2所示的探针ak055957-p、seqidno:5所示的探针bdh1-p、seqidno:8所示的探针c6orf223-p、seqidno:11所示的探针dab2ip-p、seqidno:14所示的探针f12-p、seqidno:17所示的探针grasp-p、seqidno:20所示的探针lrrc4-p、seqidno:23所示的探针nfix-p、seqidno:26所示的探针oplah-p、seqidno:29所示的探针ppfia1-p、seqidno:32所示的探针psd4-p和seqidno:35所示的探针tbx15-p。上述任一所述试剂盒还可包括内参基因检测试剂。具体的,所述内参基因可为gapdh基因。所述内参基因检测试剂可包括检测内参基因的引物和/或探针。所述内参基因检测试剂具体可由检测内参基因的引物和/或探针组成。上述任一所述检测内参基因的引物分别可为seqidno:37所示的引物gapdh-f和seqidno:39所示的引物gapdh-r。上述任一所述检测内参基因的探针分别可为seqidno:38所示的探针gapdh-p。上述任一所述肝癌检测试剂盒具体可由上述任一所述组合标志物的检测试剂和上述任一所述内参基因检测试剂组成。上述任一所述探针一端均具有荧光标记,另一端均具有荧光猝灭标记。所述荧光标记可为fam或vic标记。所述荧光猝灭标记可为bhq1标记。上述任一所述试剂盒还可包括dna的重亚硫酸转化试剂。上述任一所述试剂盒还可包括预扩增体系,所述预扩增体系用于dna重亚硫酸转化后的扩增,以提高dna含量。上述任一所述试剂盒还可包括数据处理系统;所述数据处理系统将组合标志物中各个基因的甲基化水平转化为待测者的hccscore,用于判断待测者是否为肝癌患者;所述待测者的hccscore的计算方法为:hccscore=lak055957+lbdh1+lc6orf223+ldab2ip+lf12+lgrasp+llrrc4+lnfix+loplah+lppfia1+lpsd4+ltbx15;各基因的l值为:如果(10-某基因的δct)>0,则该基因的l值=10-δct;如果(10-某基因的δct)≤0,则该基因的l值为0;所述δct值为:某基因的ct值-内参基因的ct值;所述判断的方法为:如果待测者的hccscore≥阈值,则待测者为肝癌患者或疑似为肝癌患者;如果待测者的hccscore<阈值,则待测者为非肝癌患者或疑似为非肝癌患者;所述阈值通过受试者工作特征曲线确定,具体方法为:构建训练集,训练集由n例肝癌患者和m例非肝癌者组成,n和m均为自然数;获得训练集中所有患者的hccscore;根据敏感性和特异性制作受试者工作特征曲线(roc曲线),所述阈值为roc曲线上的最优值,即受试者工作特征曲线上(1-特异性)越小且敏感性越大的值。上述任一所述试剂盒还可包括dna纯化试剂。上述任一所述肝癌检测试剂盒具体可由上述任一所述组合标志物的检测试剂、上述任一所述内参基因检测试剂、所述dna的重亚硫酸转化试剂、所述预扩增体系、所述数据处理系统和dna纯化试剂组成。上述任一所述试剂盒的检测样本为基因组dna或血浆cfdna。所述基因组dna可为肝脏组织的基因组dna。上述任一所述dna的重亚硫酸转化试剂具体可为ezdnamethylation-lightningtmkit(zymo公司)。上述任一所述预扩增体系可包括由上述任一所述用于检测组合标志物中各个基因甲基化水平的引物组成的引物mix。所述引物mix中,各个引物的浓度均相同。所述引物的浓度具体可为10μm。上述任一所述预扩增体系还可包括onetaq热启动2×预混液(thermofisher公司)。上述任一所述预扩增体系具体可由所述引物mix组成。上述任一所述预扩增体系具体可由所述引物mix和onetaq热启动2×预混液组成。上述任一所述dna纯化试剂具体可为dnaclean&concentratortm试剂盒(zymo公司)。所述标志物的甲基化程度包括是否为甲基化的状态。采用上述任一所述试剂盒判断待测肝脏组织的基因组dna或血浆cfdna来源于肝癌患者还是非肝癌患者的方法(本质即判断待测者是否患有肝癌)具体可如下:1、取待测肝脏组织的基因组dna或血浆cfdna,进行重亚硫酸转化,得到重亚硫酸转化的dna。如果待测肝脏组织的基因组dna低于60ng或待测样本为血浆cfdna,完成重亚硫酸转化后,还可以进行预扩增,得到预扩增产物。预扩增的方法为:以重亚硫酸转化的dna为模板,采用上述任一所述用于检测组合标志物中各个基因甲基化水平的引物组成的引物mix进行预扩增,得到预扩增产物。2、取重亚硫酸转化的dna(如果待测肝脏组织的基因组dna低于60ng或待测样本为血浆cfdna,则取预扩增产物),采用dna纯化试剂进行纯化。3、以完成步骤2的dna为模板,采用上述任一所述组合标志物的检测试剂和上述任一所述内参基因检测试剂进行实时荧光定量pcr反应,获得组合标志物中各个基因的甲基化的水平。荧光定量pcr检测时的体系是单个目的基因和内参gapdh的引物探针混合,形成一个双重pcr体系,每个目的基因都检测一次。目的基因即组合标志物中基因。4、完成步骤3后,获得各个基因的ct值,如果扩增曲线不为“s”型或ct值为空白,则ct值记为45;之后分别计算各个基因的δct值,δct目的基因=ct目的基因-ctgapdh;对δct目的基因进行归一化处理,获得待测者目的基因的归一值l(记为l目的基因):如果(10-δct目的基因)>0时,l目的基因=10-δct目的基因;如果(10-δct目的基因)≤0时,l目的基因=0。目的基因即组合标志物中基因。5、计算评估分数(记为hccscore),计算公式如下:hccscore=lak055957+lbdh1+lc6orf223+ldab2ip+lf12+lgrasp+llrrc4+lnfix+loplah+lppfia1+lpsd4+ltbx15。6、将步骤5得到的hccscore和阈值进行比较,判断来源于肝癌患者还是非肝癌患者。判断标准如下:如果hccscore≥阈值,则待测肝脏组织的基因组dna或血浆cfdna来源于肝癌患者,如果hccscore<阈值,则待测肝脏组织的基因组dna或血浆cfdna来源于非肝癌患者。在本发明的一个实施例中,训练集为35例非肝癌(16例健康人+19例高危人群)和89例肝癌患者,检测样本为血浆cfdna,根据hccscore制作roc曲线,阈值为40.5。上述任一所述肝癌检测可为肝癌早筛。上述任一所述肝癌检测可为肝癌诊断或辅助诊断。所述tbx15的geneid为6913。所述ak055957的genbank为ak055957.1。所述bdh1的geneid为622。所述c6orf223的geneid为221416。所述dab2ip的geneid为153090。所述ppfia1的geneid为8500)。所述lrrc4的geneid为64101)。所述nfix的geneid为4784)。所述psd4的geneid为23550。所述grasp的geneid为160622。所述oplah的geneid为26873。所述f12的geneid为2161。所述gapdh的geneid为2597。本发明具有以下优点:通过检测肝脏组织的基因组dna或血浆cfdna中的组合标志物中各个基因的甲基化水平,进行肝癌检测,其所携带的分子遗传学信息来自原发肿瘤,具有高度敏感性和特异性;本发明为无创性检测,仅凭少量外周血标本即可完成检测,不会引起肝癌播散;可实时、动态反应肝癌筛查和身体状况,避免反复有创的组织活检;可全面反映体内肝癌细胞的分子遗传信息的全貌,避免了瘤内异质性对检测结果的影响;通过预扩增方式,可以对1管血的cfdna做多基因的甲基化检测。预扩增后增加了纯化的步骤,这一步极大的提高的检测的敏感性。实时荧光定量pcr检测相对于二代测序来说,简单方便、成本低廉。实验证明,采用本发明提供的试剂盒可以判断待测肝脏组织的基因组dna或血浆cfdna来源于肝癌患者还是非肝癌患者,具有较高的准确性。本发明具有重要的应用价值。附图说明图1为实施例1中的124例待测者hccscore的分析结果。图2为实施例1中的roc曲线分析。具体实施方式以下的实施例便于更好地理解本发明,但并不限定本发明。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。以下实施例中的定量试验,均设置三次重复实验,结果取平均值。高效游离dna富集分离试剂盒为深圳市南科征途有限公司的产品。ezdnamethylation-lightningtmkit为zymo公司的产品。dnaclean&concentratortm试剂盒为zymo公司的产品。dnaminikit试剂盒为qiagen公司的产品。受试者工作特征曲线(receiveroperatingcharacteristiccurve,简称roc曲线):是根据一系列不同的二分类方式(分界值或决定阈),以敏感性(真阳性率)为纵坐标,1-特异性(假阳性率)为横坐标绘制的曲线。roc曲线下面积是重要的试验准确度指标,roc曲线下面积越大,试验的诊断价值越大。实施例1、肝癌检测的方法的建立一、获得用于肝癌检测的标志物本发明的发明人结合多种数据库以及综合临床信息,通过大规模筛选,设计探针富集相关基因的甲基化位点,再通过建库测序,对比肝癌确诊前3-6个月的cfdna样本与非肝癌患者的cfdna样本中甲基化水平的差异,获得用于肝癌检测的标志物。该标志物由tbx15(geneid:6913)、ak055957(genbank:ak055957.1)、bdh1(geneid:622)、c6orf223(geneid:221416)、dab2ip(geneid:153090)、ppfia1(geneid:8500)、lrrc4(geneid:64101)、nfix(geneid:4784)、psd4(geneid:23550)、grasp(geneid:160622)、oplah(geneid:26873)和f12(geneid:2161)组成。根据上述各个基因的核苷酸序列,分别设计并人工合成表1中所示的引物和探针。表1注:gapdh(geneid:2597)基因为内参基因;引物名称中含有“f”为表示上游引物,含有“r”为表示下游引物,含有“p”为表示探针,fam表示fam标记,vic表示vic标记,bhq1表示荧光猝灭标记。二、采用标志物进行肝癌检测124例待测者包括89例早期肝癌患者(编号为g1-g89),16例健康人(编号为j1-j16)和19例“携带乙肝病毒、患有脂肪肝或肝硬化的患者”(简称高危人群)(编号为x1-x19)。89例早期肝癌患者组成肝癌组。16例健康人组成健康组。19例高危人群组成高危组。健康组和高危组组成非肝癌组。每个待测者进行如下实验:1、将表1中ak055957基因、bdh1基因、c6orf223基因、dab2ip基因、f12基因、grasp基因、lrrc4基因、nfix基因、oplah基因、ppfia1基因、psd4基因和tbx15基因的上游引物和下游引物分别用水分别稀释至10μm,然后等摩尔混合,得到引物mix。2、取待测者的外周血,采用高效游离dna富集分离试剂盒提取纯化,得到待测者血浆cfdna。3、取待测者血浆cfdna,使用ezdnamethylation-lightningtmkit进行重亚硫酸转化,然后纯化,得到待测者转化的cfdna。4、配制表2所示的反应体系,然后按照表3所示的反应程序进行预扩增,得到预扩增产物。表2注:onetaq热启动2×预混液为thermofisher公司的产品。表35、取预扩增产物,使用dnaclean&concentratortm试剂盒纯化,最后用20μldnaelutionbuffer(zymo公司的产品)洗脱,得到洗脱液。6、将ak055957-f、ak055957-p、ak055957-r、gapdh-f、gapdh-r和gapdh-p分别用水稀释,得到相应的稀释液。配制表4所示的反应体系,然后按照表5所示的反应程序进行实时荧光定量pcr反应(60℃时收集fam和vic荧光),获得ak055957基因的ct值(记为ctak055957)和gapdh基因的ct值(记为ctgapdh);如果扩增曲线不为“s”型或ct值为空白,则ct值记为45。进一步计算ak055957基因的δct值(记为δctak055957),δctak055957=ctak055957-ctgapdh。对δctak055957进行归一化处理,获得待测者ak055957基因的归一值l(记为lak055957):如果(10-δctak055957)>0时,lak055957=10-δctak055957;如果(10-δctak055957)≤0时,lak055957=0。表4试剂体积(μl)lunauniversalprobeqpcrmastermix10ak055957-f(10μm)0.5ak055957-r(10μm)0.5ak055957-p(10μm)0.3gapdh-f(10μm)0.5gapdh-r(10μm)0.5gapdh-p(10μm)0.3洗脱液2.0ddh2o5.4注:lunauniversalprobeqpcrmastermix为neb公司的产品。表5温度(℃)时间循环数95℃1min1cycle95℃15s45cycles60℃30s按照上述方法,将ak055957-f分别替换为bdh1-f、c6orf223-f、dab2ip-f、f12-f、grasp-f、lrrc4-f、nfix-f、oplah-f、ppfia1-f、psd4-f和tbx15-f,将ak055957-p分别替换为bdh1-p、c6orf223-p、dab2ip-p、f12-p、grasp-p、lrrc4-p、nfix-p、oplah-p、ppfia1-p、psd4-p和tbx15-p,将ak055957-r分别替换为bdh1-r、c6orf223-r、dab2ip-r、f12-r、grasp-r、lrrc4-r、nfix-r、oplah-r、ppfia1-r、psd4-r和tbx15-r,其它步骤均不变,依次得到lbdh1、lc6orf223、ldab2ip、lf12、lgrasp、llrrc4、lnfix、loplah、lppfia1、lpsd4和ltbx15。即参考lak055957的计算方法,将ak055957基因的上游引物替换为目的基因的上游引物,ak055957基因的下游引物替换为目的基因的下游引物,ak055957基因的探针替换为目的基因的探针,获得目的基因的归一值l。7、计算评估分数(记为hccscore),计算公式如下:hccscore=lak055957+lbdh1+lc6orf223+ldab2ip+lf12+lgrasp+llrrc4+lnfix+loplah+lppfia1+lpsd4+ltbx15。124例待测者的hccscore见表6和图1。结果表明,与健康组相比,肝癌组的hccscore显著增加,健康组和高危组的hccscore无显著差异。表68、阈值的获得:用spss16.0软件对步骤7的结果进行roc曲线分析(图2)。roc曲线下的面积是0.98。roc曲线上(1-特异性)越小且敏感性越大的值即为阈值。roc曲线的阈值为40.5;这一节点上,显示出95.5%的敏感性和94.3%的特异性。9、将步骤7得到的hccscore和阈值进行比较,区分肝癌患者或非肝癌患者。区分标准如下:如果待测者的hccscore≥40.5,则待测者为肝癌患者或疑似为肝癌患者;如果待测者的hccscore<40.5,则待测者为非肝癌患者或疑似为非肝癌患者。本实施例提供的方法通过对血浆cfdna的检测就可以鉴定待测者为肝癌患者还是非肝癌患者,且不受乙肝病毒携带、脂肪肝或肝硬化等病情的影响。实施例2、准确性实验选取11例肝癌组织样本和12例健康人的白细胞样本,对其hccscore进行测定,样本名称和类型依次见表7中第1列和第2列。每个待测样本进行如下实验:1、同实施例1步骤二中1。2、采用dnaminikit提取待测样本的基因组dna,获得待测样本的基因组dna(od260/280在1.8-2.1之间)。3、取待测样本的基因组dna,使用ezdnamethylation-lightningtmkit进行重亚硫酸转化,然后纯化,得到待测样本转化的dna。4、将实施例1步骤二中4的“待测者转化的cfdna”替换为待测样本转化的dna,其它步骤均不变。5、同实施例1步骤二中5。6、同实施例1步骤二中6。7、同实施例1步骤二中7。23个待测样本的hccscore见表7中第3列。8、同实施例1步骤二中9,判断样本来源于肝癌患者还是非肝癌患者。23个待测样本的区分结果见表7中第4列。结果表明(表7中第5列),采用实施例1建立的方法可以判断待测样本来源于肝癌患者还是非肝癌患者,具有较高的准确性。表7待测样本名称待测样本类型hccscore判断结果一致性wbc21白细胞3.60阴性一致wbc35白细胞8.33阴性一致wbc38白细胞6.87阴性一致wbc54白细胞5.82阴性一致wbc62白细胞5.23阴性一致wbc64白细胞5.37阴性一致wbc100白细胞7.74阴性一致wbc120白细胞6.36阴性一致wbc01白细胞5.43阴性一致wbc02白细胞6.80阴性一致wbc03白细胞6.61阴性一致wbc04白细胞5.51阴性一致t40肝癌组织84.88阳性一致t41肝癌组织97.14阳性一致t42肝癌组织108.29阳性一致t43肝癌组织105.96阳性一致t45肝癌组织115.86阳性一致t46肝癌组织89.91阳性一致t47肝癌组织101.02阳性一致t48肝癌组织94.28阳性一致t62肝癌组织101.70阳性一致t63肝癌组织108.45阳性一致t66肝癌组织111.20阳性一致注:阳性为来源于肝癌患者,阴性为来源于非肝癌患者。<110>中国医学科学院肿瘤医院北京泛生子基因科技有限公司<120>用于肝癌检测的组合标志物及其应用<160>39<170>patentinversion3.5<210>1<211>27<212>dna<213>artificialsequence<400>1gtggaatttagtagcgttgattgagtc27<210>2<211>22<212>dna<213>artificialsequence<400>2aagtgtcggaacgtattatttg22<210>3<211>26<212>dna<213>artificialsequence<400>3caaacgacgctctaaaatttaactcc26<210>4<211>20<212>dna<213>artificialsequence<400>4ttcgggattggttagggtgg20<210>5<211>17<212>dna<213>artificialsequence<400>5ttaggcggcgtattcgc17<210>6<211>22<212>dna<213>artificialsequence<400>6caaccaaaacccccttatcctc22<210>7<211>24<212>dna<213>artificialsequence<400>7aggcgtagacgtagttttttagga24<210>8<211>25<212>dna<213>artificialsequence<400>8tatttgtttcgggtttttcggcgcg25<210>9<211>17<212>dna<213>artificialsequence<400>9ccttcccaccacctccc17<210>10<211>27<212>dna<213>artificialsequence<400>10gtttaagtggttagggtattgttaggc27<210>11<211>20<212>dna<213>artificialsequence<400>11ttagtcggcgttttagtcga20<210>12<211>26<212>dna<213>artificialsequence<400>12tcgataataccgataaaaacgaacgc26<210>13<211>24<212>dna<213>artificialsequence<400>13ggtcgttagtatttagtagggggc24<210>14<211>25<212>dna<213>artificialsequence<400>14atgaggttgtcggcgtagaaattgt25<210>15<211>21<212>dna<213>artificialsequence<400>15ctacatcgccgcgctatacta21<210>16<211>27<212>dna<213>artificialsequence<400>16cgtagtcgttatttttgggtttttagc27<210>17<211>26<212>dna<213>artificialsequence<400>17tcctccaacgccgcgtacaactcgtc26<210>18<211>26<212>dna<213>artificialsequence<400>18gcgcgatacaactcgacaaaataata26<210>19<211>24<212>dna<213>artificialsequence<400>19cgtggttgtcgtttgtaatatcgc24<210>20<211>21<212>dna<213>artificialsequence<400>20cgcccaccgtctcctcctcgc21<210>21<211>19<212>dna<213>artificialsequence<400>21aacaacgaaacccctacgc19<210>22<211>20<212>dna<213>artificialsequence<400>22atgatgcgcgtagggaattc20<210>23<211>18<212>dna<213>artificialsequence<400>23atcgttcgggcgtatttg18<210>24<211>18<212>dna<213>artificialsequence<400>24ccctccaaaccttcgctc18<210>25<211>18<212>dna<213>artificialsequence<400>25ttcgttgtgttgggcgtc18<210>26<211>25<212>dna<213>artificialsequence<400>26ttcgcgttttggcgttcgtttgtcg25<210>27<211>24<212>dna<213>artificialsequence<400>27cgataaaaaaaacgaaacaacgcc24<210>28<211>21<212>dna<213>artificialsequence<400>28tacgaagagatggaaacgttc21<210>29<211>19<212>dna<213>artificialsequence<400>29ttacgtcgtaaacggttag19<210>30<211>29<212>dna<213>artificialsequence<400>30cgcgctaaaaaaaatacaaactataaaac29<210>31<211>24<212>dna<213>artificialsequence<400>31gtttgtttagtgaatgtggttgtg24<210>32<211>24<212>dna<213>artificialsequence<400>32acggttggagttttcgattttcgt24<210>33<211>30<212>dna<213>artificialsequence<400>33gcttaactaaaaaaccctaaaaaaacgacg30<210>34<211>21<212>dna<213>artificialsequence<400>34ttggcgttgtttttaggagtc21<210>35<211>22<212>dna<213>artificialsequence<400>35tcgttgaaagttagcgcgtatt22<210>36<211>22<212>dna<213>artificialsequence<400>36ccaacttctctactaaaacccg22<210>37<211>22<212>dna<213>artificialsequence<400>37tgggtggttattgtgaaaagtt22<210>38<211>28<212>dna<213>artificialsequence<400>38cctttcctactaaaacccaaaaccaaac28<210>39<211>22<212>dna<213>artificialsequence<400>39ctccaatccctaaccctacctt22当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1