一种高效稳定的纤维素酶突变体的制作方法
本发明属于基因工程领域,具体的说,涉及一种高效稳定的纤维素酶突变体。
背景技术:
大额牛终年散养,放牧在大山上,没有相应的科学饲养措施,能以竹子等其它反刍家畜所不能利用的饲料为主要食物,对竹子干物质消化率可达61%,大额牛是高效纤维分解酶筛选和发掘的珍贵种质资源之一。大额牛的瘤胃内存在大量未知的纤维降解菌,且这些纤维降解菌可能携带着丰富的功能新基因,能够分泌表达高效的纤维降解酶。筛选高效分解粗纤维的基因和酶并对其进行定向改造,如果能提高大额牛瘤胃中纤维素酶的活性或改善原有纤维素酶的特性,其结果不仅对大额牛的人工饲养及生物资源的开发具有重要的意义,而且对提高其他反刍动物利用粗饲料的能力和效率具有更重大的经济意义。
技术实现要素:
本发明目的在于提供了一种高效稳定的纤维素酶突变体。本发明采用易错pcr对来源于大额牛瘤胃cmc-1基因进行了体外定向改造,获得了突变酶,其酶活、热稳定性和ph稳定性均明显提高,具有一定的开发价值。
为实现上述目的,本发明是通过如下技术方案实现的:
所述的高效稳定的纤维素酶突变体ep-15,其氨基酸序列如seqidno.3所示。
所述的高效稳定的纤维素酶突变体ep-15编码基因,用于编码所述的高效稳定的纤维素酶突变体ep-15。所述的高效稳定的纤维素酶突变体ep-15编码基因,其核苷酸序列如seqidno.4所示。
本发明还涉及包含所述的高效稳定的纤维素酶突变体ep-15编码基因的重组表达载体和包含所述的高效稳定的纤维素酶突变体ep-15编码基因的表达菌株。
本发明还提供了一种纤维素酶突变体ep-3,其氨基酸序列如seqidno.5所示。
所述的纤维素酶突变体ep-3编码基因,用于编码所述的纤维素酶突变体ep-3。所述的纤维素酶突变体ep-3编码基因,其核苷酸序列如seqidno.6所示。
本发明还涉及包含所述的纤维素酶突变体ep-3编码基因的重组表达载体和包含所述的纤维素酶突变体ep-3编码基因的表达菌株。
本发明还提供了一种纤维素酶突变体ep-6,其氨基酸序列如seqidno.7所示。
所述的纤维素酶突变体ep-6编码基因,用于编码所述的纤维素酶突变体ep-6。所述的纤维素酶突变体ep-6编码基因,其核苷酸序列如seqidno.8所示。
本发明还涉及包含所述的纤维素酶突变体ep-6编码基因的重组表达载体和包含所述的纤维素酶突变体ep-6编码基因的表达菌株。
本发明还提供了一种纤维素酶突变体ep-7,其氨基酸序列如seqidno.9所示。
所述的纤维素酶突变体ep-7编码基因,用于编码所述的纤维素酶突变体ep-7。所述的纤维素酶突变体ep-7编码基因,其核苷酸序列如seqidno.10所示。
本发明还涉及包含所述的纤维素酶突变体ep-7编码基因的重组表达载体和包含所述的纤维素酶突变体ep-7编码基因的表达菌株。
本发明的有益效果:
本发明通过构建了大额牛瘤胃纤维素酶cmc-1基因突变文库,筛选到四个突变基因ep-3、ep-6、ep-7、ep-15,这些突变基因在e.colibl21中能表达出纤维素酶活性。ep-3、ep-6、ep-15突变酶的最适温度由突变前(cmc-1)的50℃降到45℃,更接近瘤胃的生理温度(40℃)。ep-3、ep-7和ep-15较未突变酶cmc-1具有更强的热稳定性和ph稳定性,酶活力有1~2倍提高;ep-15酶活力在45℃或ph5.0时较cmc-1酶约有2倍提高。ep-15是四个突变酶中酶活最大,热稳定性和ph稳定性最强的突变酶,具有一定的开发价值,不仅对大额牛的人工饲养及生物资源的开发具有重要的意义,而且对提高其他反刍动物利用粗饲料的能力和效率具有更重大的经济意义。
附图说明
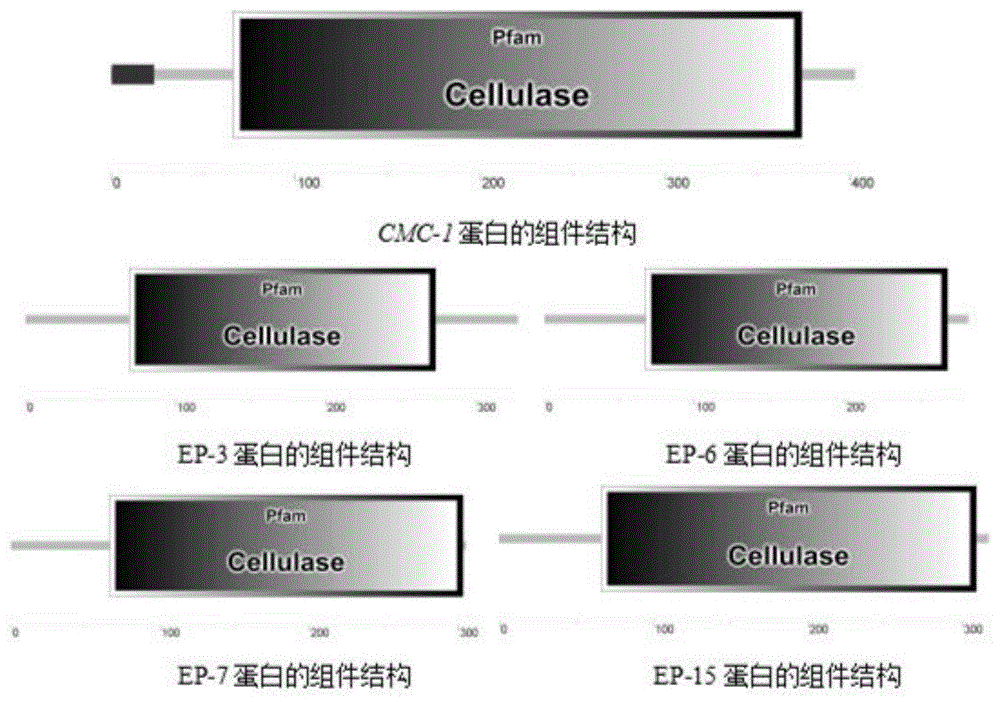
图1是蛋白的组件结构;
图2是cmc-1及其四个突变体的基因dna序列比对;
图3是葡萄糖浓度标准曲线;
图4是纤维素酶反应最适温度;
图5是40℃纤维素酶反应的热稳定性;
图6是45℃纤维素酶反应的热稳定性;
图7是50℃纤维素酶反应的热稳定性;
图8是纤维素酶反应最适ph;
图9是ph4.5纤维素酶反应的ph稳定性;
图10是ph5.0纤维素酶反应的ph稳定性;
图11是ph5.5纤维素酶反应的ph稳定性。
具体实施方式
申请人从大额牛瘤胃fosmid文库中获得的纤维素酶基因cmc-1(其氨基酸序列为seqidno.1,编码核苷酸序列为seqidno.2),通过原核表达及酶学性质分析发现cmc-1酶最适温度是50℃,最适ph为5.0。为了使cmc-1酶反应更接近瘤胃环境的生理温度(40℃)或提高纤维素酶的活力,申请人采用易错pcr对cmc-1基因进行了体外定向改造,并对改造后的突变体进行了序列分析、重组表达和酶学性质分析;从而选择出活性更高、稳定性更好以及温度或ph更适宜瘤胃内环境的纤维素酶。
【实施例1】纤维素酶突变体的获得
1.1突变文库的构建
采用ep-pcr对纤维素酶基因cmc-1进行随机突变,先酶切突变基因再连接转化进行突变文库的构建,转化至bmtop10感受态细胞中,扩大突变体文库容量。
1.1.1纤维素酶cmc-1基因易错pcr扩增
通过设计带有酶切位点(ecorⅰ、smaⅰ)的上下游引物(见表1)。
表1cmc-1引物扩增序列
注:下划线为酶切识别位点。
根据即用型易错pcr试剂盒(instanterror-pronepcrkit)自主设计如表2中的ep-pcr条件,以cmc-1-ecorⅰ-fp和cmc-1-smaⅰ-rp为引物进行ep-pcr,以体系各组分正常pcr作为对照。
表2易错pcr反应体系及其对照
反应程序:95℃5min;95℃30s,54℃40s,72℃90s,24cycles;72℃5min;4℃hold。0.8%琼脂糖凝胶电泳查看pcr结果。
1.1.2构建突变文库
采用即用型易错pcr试剂盒(instanterror-pronepcrkit)自主设计突变效果较好ep-pcr条件对纤维素酶基因cmc-1进行随机突变,随后切胶回收ep-pcr后的产物,双酶切(ecorⅰ、smaⅰ)ep-pcr后的产物,回收纯化酶切产物,得到cmc-1-ep-pcr-ecorⅰ-smaⅰ和载体pgex-6p-1-ecorⅰ-smaⅰ;使用t4dna连接酶再连接到载体上,进行构建突变文库,然后转化至bmtop10感受态细胞中,涂lb固体培养基平板(含0.1%体积amp),37℃过夜培养后,此平板内的所有菌落所含的突变基因即为cmc-1突变文库。
表3pgex-6p-1与cmc-1-ep-pcrecorⅰ酶切体系
反应条件:37℃保温2-4h。酶切完后放入65℃水浴中20min使酶失活。
表4pgex-6p-1-ecorⅰ和cmc-1-ep-pcr-ecorⅰsmaⅰ酶切体系
反应条件:25℃水浴保温2-4h。酶切完后放入65℃水浴中20min使酶失活。
表5t4连接体系
反应条件:16℃温育16h。连接完后放入65℃水浴中20min使酶失活。初步获得重组载体质粒pgex-6p-1—cmc-1-ep-pcr。(注:pgex-6p-1—cmc-1-ep-pcr不是代表一个突变体的重组质粒而是代表所有突变体的重组质粒)
1.2突变文库的转化及突变体的筛选
从突变文库中挑取单菌落编号,并进行点lb固体培养基平板(含0.1%体积am)、含cmc-na底物的lb固体培养基平板(含0.1%体积amp),采用刚果红染色平板法对突变文库进行活性较强或稳定性较强纤维素酶cmc-1突变体的初步筛选。经过刚果红染色法初步筛选得到的透明圈较大的一些菌落,随机选择几个进行菌落pcr,并且摇菌提取质粒后进行质粒pcr。最后选择效果较好的,送硕擎公司测序,通过基因序列分析确定突变率,由序列查看碱基的突变。
1.2.1突变文库的转化
转化方法大致如下:
将t4dna连接酶连接的产物pgex-6p-1—cmc-1-ep-pcr(或者突变文库、质粒)转化到感受态细胞bmtop10中,然后涂布于lb固体培养基平板(含相应抗生素如amp),37℃培养箱中过夜培养。转化方法如下:
(1)从-80℃冰箱中取出所需要的感受态细胞若干ml(100ml/管),置于冰浴中,放在无菌操作台上。如需分装可将融化细胞悬液分装到无菌预冷的离心管中,置于冰浴中。本实验用50μl感受态细胞做为实验用量。
(2)向融化的感受态细胞悬液中加入目的dna,本次实验中加入7μlt4dna连接酶连接的产物pgex-6p-1—cmc-1或者质粒。
(3)小心吹打混匀或者轻旋混匀,冰浴静置30min。
(4)42℃水浴热激90秒,然后快速转入冰浴中冷却2min,勿摇动。
(5)加入500μl无菌lb液体培养基(不含抗生素),37℃,250rpm,摇床振荡培养45min。
(6)取适量涂布lb固体培养基平板(含抗生素amp0.1%,v/v),37℃培养箱中过夜培养。本实验涂布平板的量分别是100μl和200μl。
1.2.2突变体的筛选
将t4dna连接酶连接的产物pgex-6p-1—cmc-1-ep-pcr转化后,37℃孵育平板至克隆产生,挑取单菌落编号,并进行点固体lb平板(含0.1%体积amp)、含cmc-na底物的固体lb平板(含0.1%体积amp),液体lb培养基摇菌并提取质粒。
以四种方法筛选验证重组质粒pgex-6p-1—cmc-1-ep-pcr的存在和自主性:a.对进行编号的菌落进行培养后,以cmc-1-ecorⅰ-fp和cmc-1-smaⅰ-rp为引物,进行菌落pcr;b.对进行编号的菌落进行摇菌培养后提取质粒,以cmc-1-ecorⅰ-fp和cmc-1-smaⅰ-rp为引物,提取的质粒为模板进行pcr;c.对进行编号的菌点含羧甲基纤维素钠cmc-na底物的lb固体培养基平板(含0.1%vamp),刚果红平板染色筛选验证纤维素酶的存在;d.对进行编号的菌摇菌培养后,送入公司测序,测序确定突变率,由序列查看碱基的突变,筛选突变体。
表6菌落pcr反应体系
反应程序:95℃5min;95℃30s,54℃40s,72℃90s,32cycles;72℃5min;4℃hold。0.8%琼脂糖凝胶电泳查看pcr结果。
表7质粒pcr反应体系
反应程序:95℃5min;95℃30s,54℃40s,72℃90s,32cycles;72℃5min;4℃hold。0.8%琼脂糖凝胶电泳查看pcr结果。
【实施例2】纤维素酶突变体的原核表达
2.1纤维素酶cmc-1突变体导入表达菌bl21
提取纤维素酶突变体的质粒dna并且转入到感受态细胞bl21中(转化方法同前),筛选含纤维素酶突变体的质粒dna的菌株,挑菌接种到cmc为底物的lb(含0.1%vamp)平板上,37℃过夜培养,次日用刚果红染色法确定表达菌bl21是否有纤维素酶活性。
2.2粗酶液的制备
工程菌的培养:将含有纤维素酶活(突变体)特性的表达菌bl21接种于10ml含0.1%vamp的2×yt培养基中37℃(或者16℃低温诱导表达),250rpm摇菌,当菌液od600达到0.6时加入终浓度为0.1mm的iptg,继续诱导培养10-14h。
收集菌体:将菌液于2ml离心管中,4℃,10000g,离心1min得菌体,可重复收集,直至10ml菌液全部收集完。用1×pbs洗菌体2次,离心,得菌体,置于冰上。
菌体破碎:将得到的菌体用1ml1×pbs悬置摇匀,冰上超声破碎,超声3s,停顿7s,超声30min,得全菌液。
粗酶液收集:将破碎的菌体4℃,12000g,离心10min,得上清和沉淀,上清即为粗酶液,使用时将上清过0.22μm滤膜。得到的粗酶液于4℃或者-20℃中保存备用。
【实施例3】纤维素酶突变体的序列分析
将不同的纤维素酶进行同源性比对,比对结果见表8。
表8序列同源性比对结果
由上表可得cmc-1酶和四个突变体的编码氨基酸序列与genbank数据库的部分纤维素酶的氨基酸序列同源性比对结果显示:cmc-1和ep-3、ep-6、ep-7、ep-15最大同源性依次分别为68%、64%、68%、80%、76%。
cmc-1突变体的蛋白组件分析图见图1,蛋白序列分析结果见表9。
表9纤维素酶基因的蛋白序列分析
通过对ep-3、ep-6、ep-7、ep-15突变体和cmc-1酶的序列分析,得到四个突变体和cmc-1酶都属于糖苷水解酶家族5(gh5),但四个突变体的编码aa、理论分子量、等电点、纤维素酶结构功能域都与cmc-1酶有很大差异。
cmc-1突变体的基因序列分析,分析结果见下图2。
纤维素酶基因cmc-1序列长度为1239bp,测序得到ep-3序列长度为1033bp,ep-6序列长度为1024bp,ep-7序列长度为952bp,ep-15序列长度为952bp。对各序列进行比对分析,结果如图2。具体碱基变化见表10。由图2和表10可知,ep-3、ep-6、ep-7、ep-15相对于cmc-1都发生了序列的变化。其中ep-3发生了8处碱基位置的变化,ep-6也发生了8处碱基位置突变,且与ep-3的突变位置不同;ep-7发生了一处碱基的缺失,ep-15发生了两处碱基的变化,缺失和突变。由于有碱基的插入或者缺失,使得表达产物的氨基酸变化也较大;故可推测这些cmc-1突变体与原来的突变前的cmc-1酶有一定的区别,从而导致酶活力的增强或者稳定性的提高。
表10四个突变体碱基的变化
【实施例4】纤维素酶突变体的酶学特性
4.1葡萄糖标准曲线
以葡萄糖浓度为横坐标,光吸收值od540为纵坐标,绘制标准曲线(如下图3)。相关系数为0.9925。
4.2纤维素酶cmc-1突变体的活性及稳定性
4.2.1温度对纤维素酶cmc-1突变体活性的影响
测cmc-1酶及四个突变体酶在25℃~70℃条件下的酶活力,结果如下图4。cmc-1的最适温度为50℃,此温度条件下的cmc-1酶活最大,以此为对照(设为100%),计算四个突变体在各自最适温度条件下的最大相对酶活。由图4可知,ep-3、ep-6、ep-15的最适温度均为45℃;且ep-15在45℃时的酶活比cmc-1的最大酶活提高了约2倍,ep-3的酶活提高了近1.5倍;即使在50℃条件下,ep-15和ep-3的相对酶活较cmc-1提高了约1.4倍。ep-7的最适温度仍为50℃,但其酶活较cmc-1酶提高了近1.4倍。此外,cmc-1酶及四个突变体酶在35℃以下及60℃以上酶活较低。
4.2.2纤维素酶活力
cmc-1酶及四个突变体酶在各自最适温度及ph5.0条件下的酶活力见表11。纤维素酶活力最高的是ep-15,为334.82u/ml;其它突变体酶活力高低依次为ep-3(259.23u/ml)、ep-7(247.76u/ml)和ep-6(182.97u/ml),而cmc-1为178.92u/ml。四个突变体酶活力较cmc-1酶约有1~2倍提高。
表11纤维素酶活力
4.2.3温度对纤维素酶cmc-1突变体稳定性的影响
40℃条件下,cmc-1酶及四个突变体酶的热稳定性见图5,随着处理时间的延长,各种酶活力呈下降趋势。处理30min后,除了ep-6酶外,其它四种酶的酶活都保持在80%以上;处理120min后,除了ep-6酶外,ep-7和ep-15酶活在70%以上,ep-3和cmc-1酶活力仍能保持在60%以上;说明这四种酶在120min钟内40℃条件下都具有很好的热稳定性。
45℃条件下,cmc-1酶及四个突变体酶的热稳定性见图6,随着处理时间的延长,各种酶活力呈下降趋势。处理30min后,除了ep-3、ep-6酶活在50%以下外,其他各种酶的活力都保持在50%以上。处理120min后,只有ep-15酶活仍接近70%。,其他各种酶活力已下降到50%以下,说明ep-15酶在45℃条件下的热稳定性高于其它酶。
50℃条件下,cmc-1酶及四个突变体酶的热稳定性见图7,随着处理时间的延长,各种酶活力呈下降趋势。处理120min后,唯有ep-15酶活在50%以上,而其他三个突变体及cmc-1酶活都降到50%以下。由此可见,较高的温度对纤维素酶的热稳定性影响较大,但ep-15酶在50℃条件下依然能保持较高的热稳定性。
4.2.4ph值对纤维素酶cmc-1突变体活性的影响
测ph在3.0~9.0条件下cmc-1酶及四个突变体酶的活力,结果如下图8。cmc-1酶最适ph为5.0,以cmc-1酶最适ph的酶活为对照(设定为100%),计算四种突变体在各自最适ph下的相对酶活。ep-15与cmc-1的最适ph一样仍为5.0,ep-6和ep-7的最适ph升高到5.5,而ep-3的最适ph则下降到4.5。ep-15、ep-7、ep-3和ep-6在各自最适ph时的酶活比cmc-1酶活分别提高了近2.8倍、2.6倍、1.2倍和1.3倍。
4.2.5ph值对纤维素酶cmc-1突变体稳定性的影响
ph4.5条件下,cmc-1酶及四个突变体酶的稳定性见图9,随着处理时间的延长,各种酶活力呈下降趋势。处理30min后,除了ep-6的酶活下降到40%以下外,其它的各种酶活都下降到40%以上。处理120min后,只有ep-7和ep-15的酶活下降到50%以上,而cmc-1及ep-6的酶活却下降到20%以下;由此可见ph4.5对纤维素酶cmc-1和ep-6的稳定性影响较大,特别是ep-6酶活力的稳定性影响最大,酶将近失活。
ph5.0条件下,cmc-1酶及四个突变体酶的稳定性见图10,随着处理时间的延长,各种酶活力呈下降趋势。处理30min后的各种酶活力多保持在80%以上,说明在30min钟内五种酶在ph5.0条件下的稳定性较好。处理120min后,各酶剩余酶活都高于60%,其中ep-7和ep-15酶剩余酶活达到了70%以上。结果说明这五种纤维素酶在ph5.0条件下具有一定的稳定性,且ep-15酶稳定性最高。
ph5.5条件下,不同纤维素酶活的cmc-1酶及四个突变体酶的稳定性见图11,随着处理时间的延长,各种酶活力呈下降趋势。处理30min后的各种酶活残余率大多在80%以上,其中ep-7酶活残余最高,保持在90%以上,说明在30min钟内五种酶在ph5.5条件下的稳定性较好。处理120min后,除了cmc-1酶活残余率为50%左右,其它四种突变酶活残余率都高于60%。由此可见该5个种纤维素酶在ph5.5条件下都具有较强的稳定性。
本发明构建了大额牛瘤胃纤维素酶cmc-1基因突变文库,筛选得到四个突变基因ep-3、ep-6、ep-7、ep-15,这些突变基因在e.colibl21中能表达出纤维素酶活性。
(1)ep-3、ep-6、ep-15突变酶的最适温度由突变前(cmc-1)的50℃降到45℃,更接近瘤胃的生理温度(40℃)。
(2)ep-15与cmc-1的最适ph一样仍为5.0,ep-6和ep-7的最适ph升高到5.5,而ep-3的最适ph则下降到4.5。
(3)cmc-1酶及四个突变体酶在各自最适温度及ph5.0条件下,纤维素酶活力最高的是ep-15,为334.82u/ml;其它突变体酶活力高低依次为ep-3(259.23u/ml)、ep-7(247.76u/ml)和ep-6(182.97u/ml),而cmc-1为178.92u/ml。四个突变体酶活力较cmc-1酶约有1~2倍提高。
(4)将四个突变体与cmc-1在40℃、45℃、50℃分别放置2h及与底物(1%cmc-na)作用30min后观察酶活稳定性,结果显示ep-15和ep-7在40℃条件下的酶活仍能保持在70%以上,高于cmc-1酶(68%);ep-15在45℃和50℃条件下酶活分别保持在70%和50%以上,而另三个突变体以及cmc-1的酶活却都降到50%以下。
(5)ep-3、ep-7和ep-15在ph5.0或ph5.5放置2h后及与底物(1%cmc-na)作用30min观察ph稳定性,结果显示三个突变体在ph5.0或ph5.5具有较强的ph稳定性,且酶活都高于cmc-1酶;尤其是在ph5.5,三个突变体的酶活仍能保持在70%以上,而cmc-1酶活却下降到50%左右。
最后说明的是,以上优选实施例仅用于说明本发明的技术方案而非限制,尽管通过上述优选实施例已经对本发明进行了详细的描述,但本领域技术人员应当理解,可以在形式上和细节上对其作出各种各样的改变,而不偏离本发明权利要求书所限定的范围。
sequencelisting
<110>云南农业大学
<120>一种高效稳定的纤维素酶突变体
<130>20191206
<160>10
<170>patentinversion3.5
<210>1
<211>413
<212>prt
<213>prevotella
<400>1
valiletyrasnarggluasnaspthrthrmetargleuleuserile
151015
serleualaileileileserleualacyscysglyservalgluser
202530
serasnileglugluprolysprovalaspasnalaalaprothrala
354045
lysglntrpasnglygluilethralaglytrpasnleuglyasngln
505560
pheglucysseralaproglyglngluhistyrserleuaspilegly
65707580
metproalaasnserileasnalagluthralatrpglyasnprolys
859095
valthrlysasnmetilelysalavallysalaalaglypheasnala
100105110
ileargileproileargtrpglncyshisilethrasnproglnala
115120125
metservalasplysalatrpmetalaargvallysglnvalvalasp
130135140
trpcysleugluleuaspmetlysvalileileasnthrhishisglu
145150155160
glntrpleugluserargproleuasnargtyrlysglugluasncys
165170175
glnlysleualaleuleutrppheasnilealasergluphealaasp
180185190
tyrasptyrargleualaphealaglythrasngluvalhisglulys
195200205
aspasntrpglylysprothralagluasnleugluvalglnasnala
210215220
tyrasnglnthrphevalaspilevalargalathrglyglyasnasn
225230235240
alalysarghisleuleuvalglnthrtyrvalcysasnprogluphe
245250255
glyleuserasnglyasppheileileprothraspilegluglyasn
260265270
glyasnlystyrmetservalgluilehistyrtyrasnprotrpasp
275280285
tyralaglygluglylystyrtyrtyrtrpglyglualatyrsergln
290295300
tyrglygluileserproserlysglualaaspmetileasnleuphe
305310315320
aspargvalalaargthrtrpglyasplysglyleuglyilevalile
325330335
glyglutrpglyvalthrasphistyrlysglyasnglnthraspleu
340345350
methisalaasnmetthrtyrtyrcyslysthrtyrvalthrgluala
355360365
arglysargglyphealathrpheiletrpaspasnasnserphegly
370375380
asnglymetglulystyrglyilepheasparglysasnasnmetlys
385390395400
vallysthrprotrpileleuaspglyilephealalys
405410
<210>2
<211>1239
<212>dna
<213>prevotellaruminicola
<400>2
gtgatatataatcgcgaaaacgacactactatgagacttctttcaatttcactcgcaatc60
atcatttcgctggcctgctgtggctcagtagagagcagcaacattgaagagccaaaaccg120
gtggacaatgctgcgccaaccgcaaagcaatggaacggcgagataacagccggctggaat180
ctgggcaaccagtttgaatgctcggcgccaggacaagagcattacagtctggacataggc240
atgccggctaactccatcaacgccgagaccgcatggggaaatcccaaagtaacaaaaaac300
atgatcaaggctgtgaaagcggccggtttcaatgccatccgcattcccattcgatggcag360
tgccacatcaccaaccctcaagccatgagcgttgataaggcttggatggcccgcgtcaag420
caggtggttgactggtgtcttgaacttgacatgaaggtcataatcaacacccaccatgag480
caatggcttgaatcacgtccccttaatcgctacaaagaggagaattgccaaaagctcgca540
ttgttgtggtttaacatcgcaagcgagtttgccgattacgactatcgtcttgcctttgcc600
ggcaccaacgaggttcacgagaaagataactggggcaagccaaccgccgagaatcttgag660
gttcaaaatgcctacaaccagacttttgttgacatcgttcgtgccacaggtggcaacaat720
gccaagcgtcacctgctcgttcagacctatgtgtgtaacccagagtttggtctcagcaac780
ggcgacttcataatccccaccgacatcgagggcaacggcaacaaatacatgagcgtggaa840
atccactactacaatccttgggattatgccggtgaagggaaatactattactggggtgag900
gcctacagccagtatggcgaaatctcgccaagcaaggaggccgacatgataaatctcttc960
gaccgtgtggcaagaacatggggcgacaaaggcttgggaatcgtgataggcgaatggggc1020
gttaccgaccactacaaaggcaatcaaactgacttgatgcacgccaacatgacctactac1080
tgcaagacctacgtcaccgaggcgcgcaagcgcggttttgctaccttcatttgggataac1140
aacagttttggcaacggtatggagaaatatggcattttcgaccgcaagaacaacatgaag1200
gtcaagactccctggattttagatggcatcttcgcaaaa1239
<210>3
<211>317
<212>prt
<213>prevotellaruminicola
<400>3
leuasnargthrglyvalleupheglnglyproleuglyserproglu
151015
pheglyservalgluserserasnileglugluprolysprovalasp
202530
asnalaalaprothralalysglntrpasnglygluilethralagly
354045
trpasnleuglyasnglnpheglucysseralaproglyglngluhis
505560
tyrserleuaspileglymetproalaasnserileasnalagluthr
65707580
alatrpglyasnprolysvalthrlysasnmetilelysalavallys
859095
alaalaglypheasnalaileargileproileargtrpglncyshis
100105110
ilethrasnproglnalametservalasplysalatrpmetalaarg
115120125
vallysglnvalvalasptrpcysleugluleuaspmetlysvalile
130135140
ileasnthrhishisgluglntrpleugluserargproleuasnarg
145150155160
tyrlysglugluasncysglnlysleualaleuleutrppheasnile
165170175
alasergluphealaasptyrasptyrargleualaphealaglythr
180185190
asngluvalhisglulysaspasntrpglylysprothralagluasn
195200205
leugluvalglnasnalatyrasnglnthrphevalaspilevalarg
210215220
alathrglyglyasnasnalalysarghisleuleuvalglnthrtyr
225230235240
valcysasnproglupheglyleuserasnglyasppheileilepro
245250255
thraspilegluglyasnglyasnlystyrmetservalgluilehis
260265270
tyrtyrasnprotrpasptyralaglygluglylystyrtyrtyrtrp
275280285
glyglualatyrserglntyrglygluileserproserlysgluala
290295300
aspmetileasnleupheaspargglygluargasngly
305310315
<210>4
<211>952
<212>dna
<213>prevotellaruminicola
<400>4
tctcaatcggactggggttctgttccaggggcccctgggatccccggaattcggctcagt60
agagagcagcaacattgaagagccaaaaccggtggacaatgctgcgccaaccgcaaagca120
atggaacggcgagataacagccggctggaatctgggcaaccagtttgaatgctcggcgcc180
aggacaagagcattacagtctggacataggcatgccggctaactccatcaacgccgagac240
cgcatggggaaatcccaaagtaacaaaaaacatgatcaaggctgtgaaagcggccggttt300
caatgccatccgcattcccatccgatggcagtgccacatcaccaaccctcaagccatgag360
cgttgataaggcttggatggcccgcgtcaagcaggtggttgactggtgtcttgaacttga420
catgaaggtcataatcaacacccaccatgagcaatggcttgaatcacgtccccttaatcg480
ctacaaagaggagaattgccaaaagctcgcattgttgtggtttaacatcgcaagcgagtt540
tgccgattacgactatcgtcttgcctttgccggcaccaacgaggttcacgagaaagataa600
ctggggcaagccaaccgccgagaatcttgaggttcaaaatgcctacaaccagacttttgt660
tgacatcgttcgtgccacaggtggcaacaatgccaagcgtcacctgctcgttcagaccta720
tgtgtgtaacccagagtttggtctcagcaacggcgacttcataatccccaccgacatcga780
gggcaacggcaacaaatacatgagcgtggaaatccactactacaatccttgggattatgc840
cggggaagggaaatactattactggggtgaggcctacagccagtatggcgaaatctcgcc900
aagcaaggaggccgacatgataaatctcttcgaccggggggaaagaaatggg952
<210>5
<211>328
<212>prt
<213>prevotellaruminicola
<400>5
alaglualaleuasnargthrglyvalleupheglnglyproleugly
151015
serproglupheglyservalgluserserasnileglugluprolys
202530
provalaspasnalaalaprothralalysglntrpasnglygluile
354045
thralaglytrpasnleuglyasnglnpheglucysseralaprogly
505560
glngluhistyrserleuaspileglymetproalaasnserileasn
65707580
alagluthralatrpglyasnprolysvalthrlysasnmetilelys
859095
alavallysalaalaglypheasnalaileargileproileargtrp
100105110
glncyshisilethrasnproglnalametservalasplysalatrp
115120125
metalaargvallysglnvalvalasptrpcysleugluleuaspmet
130135140
lysvalileileasnthrhishisgluglntrpleugluserargpro
145150155160
leuasnargtyrlysglugluasncysglnlysleualaleuleutrp
165170175
pheasnilealasergluphealaasptyrasptyrargleualaphe
180185190
alaglythrasngluvalhisglulysaspasntrpglylysprothr
195200205
alagluasnleugluvalglnasnalatyrasnglnthrphevalasp
210215220
ilevalargalathrglyglyasnasnalalysarghisleuleuval
225230235240
glnthrtyrvalcysasnproargvaltrpserglnglnargargleu
245250255
hisasnprohisarghisargglyglnargglnglnleuhisgluala
260265270
trplysserthrthrthrileleuglyileilealaglygluglyasn
275280285
thrileasntrpglyglualatyrserglntyrglygluileserpro
290295300
serlysglualaaspmetileilephepheaspargglyglnglyhis
305310315320
glyalathrlysalatrpgluser
325
<210>6
<211>1033
<212>dna
<213>prevotellaruminicola
<400>6
ggcggaggctctcaatcggactggagttctgttccaggggcccctgggatccccggaatt60
cggctcagtagagagcagcaacattgaagagccaaaaccggtggacaatgctgcgccaac120
cgcaaagcaatggaacggcgagataacagccggctggaatctgggcaaccagtttgaatg180
ctcggcgccaggacaagagcattacagtctggacataggcatgccggctaactccatcaa240
cgccgagaccgcatggggaaatcccaaagtaacaaaaaacatgatcaaggctgtgaaagc300
ggccggtttcaatgccatccgcattcccattcgatggcagtgccacatcaccaaccctca360
agccatgagcgttgataaggcttggatggcccgcgtcaagcaggtggttgactggtgtct420
tgaacttgacatgaaggtcataatcaacacccaccatgagcaatggcttgaatcacgtcc480
ccttaatcgctacaaagaggagaattgccaaaagctcgcattgttgtggtttaacatcgc540
aagcgagtttgccgattacgactatcgtcttgcctttgccggcaccaacgaggttcacga600
gaaagataactggggcaagccaaccgccgagaatcttgaggttcaaaatgcctacaacca660
gacttttgttgacatcgttcgtgccacaggtggcaacaatgccaagcgtcacctgctcgt720
tcagacctatgtgtgtaacccaagagtttggtctcagcaacggcgacttcataatcccca780
ccgacatcgagggcaacggcaacaattacatgaagcgtggaaatccactactacaatcct840
tgggattattgccggtgaagggaatactattaactggggtgaggcctacagccagtatgg900
cgaaatctcgccaagcaaggaggccgacatgataatcttcttcgaccgggggcaaggaca960
tggggcgacaaaggcttgggaatcgtgaaagggcgaatggggcgttaccgacactacaag1020
gggatcaaactga1033
<210>7
<211>282
<212>prt
<213>prevotellaruminicola
<400>7
serleuasnargthrglyvalleupheglnglyproleuglyserpro
151015
glupheglyservalgluserserasnileglugluprolysproval
202530
aspasnalaalaprothralalysglntrpasnglygluilethrala
354045
glytrpasnleuglyasnglnpheglucysseralaproglyglnglu
505560
histyrserleuaspileglymetproalaasnserileasnalaglu
65707580
thralatrpglyasnprolysvalthrlysasnmetilelysalaval
859095
lysalaalaglypheasnalaileargileproileargtrpglncys
100105110
hisilethrasnproglnalametservalasplysalatrpmetala
115120125
argvallysglnvalvalasptrpcysleugluleuaspmetlysval
130135140
ileileasnthrhishisgluglntrpleugluserargproleuasn
145150155160
argtyrlysglugluasncysglnlysleualaleuleutrppheasn
165170175
ilealasergluphealaasptyrasptyrargleualaphealagly
180185190
thrasngluvalhisglulysaspasntrpglylysprothralaglu
195200205
asnleugluvalglnasnalatyrasnglnthrphevalaspileval
210215220
argalathrglyglyasnasnalalysarghisleuleuvalglnthr
225230235240
tyrvalcysasnproglupheglyleuserasnglyasppheileile
245250255
prothraspilegluglyasnglyasnlystyrmetlysargglyasn
260265270
proleuleuglnserleuglyleucysarg
275280
<210>8
<211>1024
<212>dna
<213>prevotellaruminicola
<400>8
tctctcaatcggactggagttctgttccaggggcccctgggatccccggaattcggctca60
gtagagagcagcaacattgaagagccaaaaccggtggacaatgctgcgccaaccgcaaag120
caatggaacggcgagataacagccggctggaatctgggcaaccagtttgaatgctcggcg180
ccaggacaagagcattacagtctggacataggcatgccggctaactccatcaacgccgag240
accgcatggggaaatcccaaagtaacaaaaaacatgatcaaggctgtgaaagcggccggt300
ttcaatgccatccgcattcccattcgatggcagtgccacatcaccaaccctcaagccatg360
agcgttgataaggcttggatggcccgcgtcaagcaggtggttgactggtgtcttgaactt420
gacatgaaggtcataatcaacacccaccatgagcaatggcttgaatcacgtccccttaat480
cgctacaaagaggagaattgccaaaagctcgcattgttgtggtttaacatcgcaagcgag540
tttgccgattacgactatcgtcttgcctttgccggcaccaacgaggttcacgagaaagat600
aactggggcaagccaaccgccgagaatcttgaggttcaaaatgcctacaaccagactttt660
gttgacatcgttcgtgccacaggtggcaacaatgccaagcgtcacctgctcgttcagacc720
tatgtgtgtaacccagagtttggtctcagcaacggcgacttcataatccccaccgacatc780
gagggcaacggcaacaaatacatgaagcgtggaaatccactactacaatccttgggatta840
tgccggtgaagggaatactatttactggggtgaggcctacagccagtatggcgaatctcg900
ccaagcaggaggccggatgataatctcttcgacgggggcaggacatgggggacaaaggtt960
ggaatcgtgataggcgaatggggcgtaccgacactacaaggcatcaactgattgatgccg1020
cact1024
<210>9
<211>305
<212>prt
<213>prevotellaruminicola
<400>9
serglntrpthrvalvalleupheglnglyproleuglyserproglu
151015
pheglyservalgluserserasnileglugluprolysprovalasp
202530
asnalaalaprothralalysglntrpasnglygluilethralagly
354045
trpasnleuglyasnglnpheglucysseralaproglyglngluhis
505560
tyrserleuaspileglymetproalaasnserileasnalagluthr
65707580
alatrpglyasnprolysvalthrlysasnmetilelysalavallys
859095
alaalaglypheasnalaileargileproileargtrpglncyshis
100105110
ilethrasnproglnalametservalasplysalatrpmetalaarg
115120125
vallysglnvalvalasptrpcysleugluleuaspmetlysvalile
130135140
ileasnthrhishisgluglntrpleugluserargproleuasnarg
145150155160
tyrlysglugluasncysglnlysleualaleuleutrppheasnile
165170175
alasergluphealaasptyrasptyrargleualaphealaglythr
180185190
asngluvalhisglulysaspasntrpglylysprothralagluasn
195200205
leugluvalglnasnalatyrasnglnthrphevalaspilevalarg
210215220
alathrglyglyasnasnalalysarghisleuleuvalglnthrtyr
225230235240
valcysasnproglupheglyleuserasnglyasppheileilepro
245250255
thraspilegluglyasnglyasnlystyrmetservalgluilehis
260265270
tyrtyrasnprotrpasptyralaglygluglyasnthrilethrgly
275280285
valargprothralasermetalalysserargglnalaargargpro
290295300
thr
305
<210>10
<211>997
<212>dna
<213>prevotellaruminicola
<400>10
tgtcgttccgacgcgcgaggcagatcgtcagtcagtcacgatgcggccgctcgagtcgac60
ccgggtaattggtcgtttcgcattgcttattaatattagaagggagagaatctatatatg120
gggttattttgcgaagatgccatctaaaatccagggagtcttgaccttcatgttgttctt180
gcggtcgaaaatgccatatttctccataccgttgccaaaactgttgttatcccaaatgaa240
ggtagcaaaaccgcgcttgcgcgcctcggtgacgtaggtcttgcagtagtaggtcatgtt300
ggcgtgcatcaagtcagtttgattgcctttgtagtggtcggtaacgccccattcgcctat360
cacgattcccaagcctttgtcgccccatgttcttgccacacggtcgaagagatttatcat420
gtcggcctccttgcttggcgagatttcgccatactggctgtaggcctcaccccagtaata480
gtatttcccttcaccggcataatcccaaggattgtagtagtggatttccacgctcatgta540
tttgttgccgttgccctcgatgtcggtggggattatgaagtcgccgttgctgagaccaaa600
ctctgggttacacacataggtctgaacgagcaggtgacgcttggcattgttgccacctgt660
ggcacgaacgatgtcaacaaaagtctggttgtaggcattttgaacctcaagattctcggc720
ggttggcttgccccagttatctttctcgtgaacctcgttggtgccggcaaaggcaagacg780
atagtcgtaatcggcaaactcgcttgcgatgttaaaccacaacaatgcgagcttttggca840
attctcctctttgtagcgattaaggggacgtgattcaagccattgctcatggtggggggg900
tgataatgaccttcatgtcaagtccagaaaccagtcaaccacctgcttgacggggccatc960
caagcttatcaccgtcatggcttggagggatggggac997
- 还没有人留言评论。精彩留言会获得点赞!