一种用于妊娠期糖尿病肠道菌群检测16SrDNA扩增子建库方法与流程
一种用于妊娠期糖尿病肠道菌群检测16s rdna扩增子建库方法
技术领域
1.本发明属于肠道菌群检测领域,具体涉及妊娠期糖尿病肠道菌群检测16s rdna 扩增子建库方法。
背景技术:
2.illumina测序平台的16s v3
‑
v4区扩增子样本的建库方法目前主要有三种:第一种方法为使用较早且广泛的pcr扩增建库法,即在建库过程中包含pcr扩增步骤。此建库法对携带标签序列的pcr扩增产物进行纯化和定量的方法主要有两种。第一种方法是在pcr扩增后,采用pcr产物回收试剂盒纯化,利用分光光度计定量,然后等摩尔量混合。这种纯化定量的均一化方法无法除去pcr产物中的引物二聚体,影响分光光度计对目标片段的准确定量,使样本间无法真正地等摩尔量混合,而使测序数据的均一性较差,加测样本比例较高,项目周期无法保证。第二种方法是在pcr扩增后,把pcr产物单独切胶回收和qubit定量,然后等摩尔量混合,这种纯化定量的均一化方法,胶回收效率较低,导致pcr产物损失较多,使建库起始量无法保证在正常范围内,进而影响建库成功率。同时操作繁琐,试剂成本较高。另外,在采用上述pcr扩增建库方法对如16s rdna或者its等文库进行构建时,由于16s rdna或者its等在扩增过程中使用的是混合模板,而且这些模板之间序列的相似性很高,所以基因组总dna在16s rdna扩增时容易出现错误,产生一些在环境中原本并不存在的"16s rdna"序列,对文库的构建影响较大,会使我们过高估计环境中微生物的多样性。信息分析表明,这种建库方法使样本间的生物学聚类不合理(有文库趋同的现象),同一样本的技术重复性差。
3.第二种方法为一步扩增法,即通过一步pcr完成建库过程。采用一步扩增建库法对上述样本进行建库时,在上机测序时,不能使用illumina miseq相配套的试剂进行测序,需要自定义测序引物,即单独合成16s v3
‑
v4区上游保守序列作为p5端测序引物,16s v3
‑
v4区下游保守序列引物作为p7端测序引物,16s v3
‑
v4区下游保守序列的反向序列作为index的测序引物,同时需要合成特制的phix文库。上机测序时,只能和相同可变区的扩增子文库混为一个运行通道(run),这样不仅影响排机测序灵活性,而且无法保证测序质量。
4.第三种方法为truseq pcr
‑
free建库法,即完全应用truseq pcr
‑
free试剂盒构建文库。此种建库法可以直接使用illumina miseq相配套的试剂进行测序,且样本间生物学聚类合理,同一样本的重复性好,但是试剂成本较高(truseq dna pcr
‑
free sample preparation kit),需要攻克其技术限制。
5.因此,如何提供一种操作简单、成本低且得到的不同样本间的测序数据均一性好的扩增子文库构建方法成了一个亟待解决的技术问题。
技术实现要素:
6.本发明提供了一种扩增子文库及其构建方法,主要用检测妊娠孕妇肠道菌群的分
布特征,通过孕妇肠道粪便样本进行多样本的目的片段进行pcr扩增,得到多个扩增产物;然后对多个扩增产物进行等量混合,得到混合产物;对混合产物依次进行片段化、末端修复和3’末端加“a”,得到带“a”修复产物;采用pcr
‑
free的接头对带“a”修复产物进行接头连接,得到扩增子文库。
7.通过将扩增产物进行等量混合和使用优化的pcr
‑
free进行接头连接,使该方法避免了pcr产生的偏好性和环境样本中的嵌合体,使不同样本间测序数据均一性好,使后续的聚类分析和技术重复性更合理、准确,能更真实地反应妊娠期糖尿病孕妇肠道菌群物种丰度分布,为妊娠期糖尿病患者提供快速的检测结果。
8.附图说明:图1示出了根据本发明一种典型的实施例中扩增子文库构建的流程示意图;图2示出了根据本发明一种典型的实施例中经pcr扩增后的扩增产物的电泳检测图;图3示出了根据本发明一种典型的实施例中对所构建的扩增子文库进行库检的结果图;图4示出了根据本发明一种典型的实施例中对所构建的扩增子文库结构图;图5示出了采用本发明的方法所构建的扩增子文库所得数据的物种效果图。
9.具体实施方法为实现上述目的,本发明采用如下技术方案:。
10.样品预处理1)将300mg左右的粪便样品放入2ml离心管,加入1ml的pbs(0.1mol/l ph7.4),20μl 20%pvpp,充分振荡混匀2)室温下2000r/min 离心6min,取上清液,沉淀中再加入1ml的pbs混匀,离心,取上清液;3)合并两次的上清液,12000r/min离心6min,收集沉淀。
11.肠道菌群dna提取1)在预处理的样本中依次加入300μl裂解液1(0.15mol/l nacl 0.1mol/l na2edta,ph8.0)100mg/ml溶菌酶100μl 5μl蛋白酶k(10mg/ml),37℃下500r/min振荡30min;2)加入300μl裂解液2(10%sds,0.1mol/l nacl,0.5mol/l tris
‑
hcl,ph8.0),50μl20%pvpp,65℃保温10min,加入750μl tris
‑
饱和酚:氯仿:异戊醇(25:24:1),混合振荡2min,13000r/min离心8min;3)将2.2步骤的上清液转入另一2ml离心管:加入等体积的tris
‑
饱和酚:氯仿:异戊醇 (25:24:1)混合振荡2min,13000r/min离心8min;4)取上清液转入另一2ml离心管:加入1/10体积的3mol/l醋酸钠和2倍体积无水乙醇,
‑
20℃沉淀2h:13000r/min离心15min;5)沉淀用70%冰乙醇洗涤一次,自然风干;6)最后用50μl去离子水溶解,
‑
20℃保存。
12.样本质检1)从步骤1.1提取的dna 1
ꢀµ
l,用qubit3.0检测dna的浓度;
2)吸取1
ꢀµ
l提取的dna,nanodrop200检测dna浓度和吸光值;选择浓度大于10 ng/ul,od260/280=1.8
‑
2.0的dna进行后续文库构建4)吸取2
ꢀµ
l提取的dna,用1%琼脂糖凝胶电泳检测dna的完整性。
13.扩增4.1扩增16s rdna的 v3
‑
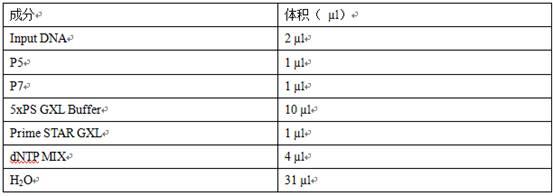
v41)样品混匀离心按下列表格配置体系本步骤在使用一步扩增法,使用的正向引物为341f:actcctacgggaggcagcag反向引物为806r:ggactachvgggtwtctaat样品混匀,短暂离心,跑胶观察文库的片段长度图2文库结构示意图如图3pcr程序设置如下。
14.文库纯化1) 磁珠平衡至室温后,涡旋振荡混匀vahts dna clean beads,吸取50
ꢀµ
l vahts dna clean beads 到上述cdna扩增反应体系中,使用移液器混匀10次以上保证整个体系均匀;2) 室温孵育8 min,使cdna结合到磁珠上。将反应管置于磁力架上分离磁珠和液体,待溶液澄清(约5 min)后,小心吸取上清,加入27
ꢀµ
l vahts dna clean beads 。使用移液器混匀10次以上保证整个体系均匀。室温孵育8 min,使cdna结合到磁珠上;3)将反应管置于磁力架上分离磁珠和液体,待溶液澄清(约5 min)后,吸取上清67
µ
l并加入40
ꢀµ
l vahts dna clean beads。使用移液器混匀10次以上保证整个体系均匀。室温孵育8 min,使cdna结合到磁珠上;
4)将反应管置于磁力架上分离磁珠和液体,待溶液澄清(约5 min)后,吸去上清,保持pcr管始终处在磁力架上,加入200
ꢀµ
l新鲜配制的80%乙醇,室温孵育30 sec,小心移除上清;5)重复步骤4,共漂洗两次;6)保持pcr管始终置于磁力架中,开盖空气干燥磁珠5
ꢀ‑ꢀ
10 min至无乙醇残留;7)磁珠晾干后,将pcr管从磁力架上取下,加入17
ꢀµ
l elution buffer覆盖磁珠,使用移液器吹打混匀磁珠。室温孵育2 min。如果磁珠干燥开裂,适当延长孵育时间;8)将pcr管短暂离心收集后置于磁力架中,分离磁珠和液体直到溶液澄清(约5 min);9)小心吸取15
ꢀµ
l上清转移到新的低吸附ep管中,
‑
80
ꢀ°
c 保存。
15.文库库检1)用4
µ
l样品进行文库检测检测结果如下表1和图4表1 7测序在illumina miseq平台上进行双端300bp 测序 8数据分析1)将测序得到的raw reads拼接、去除barcode和primer,得到raw tags;2)根据碱基质量过滤,得到clean tags;3)最后用usearch61方法参考gold数据库去除嵌合体,得到effective tags;4)对effective tags 进行数据库比对和聚类,生成 otu(operational taxonomic units);如下表2表2
ꢀ
5)利用生成的 otu 进行 物种分类和统计,以获得样品的丰度信息图5根据不同妊娠糖尿病患者中肠道微生物的物种分布。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1