一种病原耐药基因的存在及表达情况的共检测方法与流程
本发明属于微生物检测技术领域,具体涉及一种病原耐药基因的存在及表达情况的共检测方法,尤其涉及一种基于二代测序平台共检测耐药基因的存在及表达情况的方法。
背景技术:
抗菌药物(抗生素)通过杀灭细菌发挥治疗感染的作用,包括β-内酰胺类、氨基糖苷类、四环素类、氟喹诺酮类、叶酸途径抑制剂类、多肽类和大环内酯类等,在治疗时需根据不同的感染性疾病进行合理选择。细菌作为一类广泛存在的生物体,可以通过多种形式获得对抗菌药物的抵抗作用,逃避被杀灭的危险,这种抵抗作用被称为“细菌耐药”(drugresistance),获得耐药能力的细菌就被称为“耐药菌”。耐药菌对人类健康构成重大威胁,其常出现在医疗机构内,医院科室中内、外科均以大肠埃希菌(escherichiacoli)检出率最高,而icu最常见病原菌是鲍曼不动杆菌(acinetobacterbaumannii),且icu耐药率普遍高于非icu。同时,越来越多的同一菌株携带多种耐药基因,这也进一步警示人们要合理地使用抗菌药物。
目前,临床常用的针对耐药菌的耐药基因检测方法主要是两种:1、药敏鉴定,该方法虽然是耐药基因检测的金标准,但是存在需要培养,鉴定周期长,灵敏度不高等缺点;2、荧光pcr,相较于药敏鉴定,虽然不依赖培养,提高了灵敏度,但也存在通量不高限制,一次只能检测几个耐药位点等问题。
宏基因组测序技术(mngs)作为一种无差别测序方法,也可以应用于临床病原抗生素耐药基因检测和分析。但是,目前mngs在临床方面的反馈效果并不理想,主要原因包括基因碎片化过于严重导致漏检或者检测到的片段临床意义不明确等问题。
靶向测序(targetsequencing)是一种对感兴趣的基因或区域进行特异富集并测序的方法,包括多重扩增测序法和探针捕获测序法两种方式。使用靶向探针捕获抗生素耐药基因或位点的高通量测序法来检测耐药基因已成为一个有潜力的方法。该方法和多重扩增测序法相比有以下几个优势:1、捕获探针的设计难度低于多重扩增;2、探针捕获的覆盖区域大,可以一次性捕获检测更多的耐药相关基因;3、部分耐药基因存在序列相似度高的特点,特别是归属于同一耐药基因家族的不同成员,如果使用多重扩增测序法难度较大。
目前为止,尚未开发出一种既能检测耐药基因片段是否存在又能够同时检测耐药基因转录表达情况的测序方法。
技术实现要素:
针对现有技术存在的不足,本发明的目的在于提供一种病原耐药基因的存在及表达情况的共检测方法。所述方法通过单次实验测序即可得到耐药相关基因在dna和rna两个层面的测序数据,既能够检测耐药基因片段是否存在、又能够同时检测耐药基因转录表达情况。
为达此目的,本发明采用以下技术方案:
第一方面,本发明提供一种用于检测病原耐药基因的存在及表达情况的反转录随机引物,所述反转录随机引物包括5~10个随机碱基和2~5个固定碱基,所述固定碱基包括act。
由于rna需要进行反转录步骤操作生成cdna,所以,在测序序列含有固定碱基标记的序列,其原始序列均为rna。本发明中,使用固定碱基act进行标记,可以防止其与其他扩增得到的测序序列混淆,区分度较高,反转录结束后就可以与核酸中的dna合并打断建库和测序。
本发明中,所述反转录随机引物中随机碱基的个数可以为5个、6个、7个、8个、9个或者10个,固定碱基的个数可以为2个、3个、4个或者5个。
同时,本发明中,固定碱基的选择不考虑三者完全相同的碱基,如aaa,ccc等,其会影响引物整体的gc含量,也会影响引物的碱基复杂度;且本发明中,选择的固定碱基为act,其反转录后共检测的效果最好。
本发明中,所述固定碱基可以直接位于5'末端,或者与5'末端间隔若干个随机碱基;其中,优选为间隔1个随机碱基。
优选地,所述反转录随机引物自5'端起依次包括1~2个随机碱基、3个固定碱基和5~8个随机碱基。
优选地,所述反转录随机引物的序列如seqidno.1所示,具体为:
5'-nactnnnnnn-3'。
随机引物中,n表示随机碱基a、t、g或c中的任意一个。
第二方面,本发明还包括如第一方面所述的反转录随机引物在实时荧光定量pcr检测、宏基因组测序或捕获测序中的应用。
第三方面,本发明提供一种病原耐药基因的存在及表达情况的共检测方法,所述共检测方法包括如下步骤:
(1)提取样本中的核酸;
(2)对步骤(1)所得核酸进行片段化处理;
(3)使用第一方面所述的反转录随机引物和逆转录酶对步骤(2)中所得片段化核酸进行反转录,再对片段化的核酸进行末端修复和加a尾,而后连接测序接头,纯化;
(4)对步骤(3)所得纯化后的产物进行pcr文库扩增并加index,纯化得到dna文库;
(5)对所得dna文库进行宏基因组测序,得到病原耐药基因的存在情况和表达情况;
或者,将所得dna文库和捕获探针混合进行文库杂交反应,纯化捕获片段,再对所述纯化后的捕获片段进行二代高通量测序,获得病原耐药基因的存在情况和表达情况。
需要注意的是,本发明中所述“样本”或“样品”是指需要进行检测分析的物质,其可以来自于个体(如人或动物,更具体地如全血、血浆、唾液、脑髓液、关节液、痰液、肺泡灌洗液、尿液、粪便、腹胸腹水或组织等),也可以是其它来源的,例如一些经处理或未经处理的实验室材料,或人工合成的测试品。应理解,对“样本”或“样品”的检测并非仅涉及诊断目的,还可涉及其它非诊断目的。
作为本发明优选的技术方案,步骤(5)所述捕获探针检测的基因包括vana、kpc、ndm-1、imp、imi、vim、ind、sme-1、embb、etha、gyra、katg、ahpc、pand、pnca、murm、rpob、rpsa、ctx-m、rpsl、rrs、tetm、meca、mecb、mecc、cau-1、fez-1、cpha、sfh-1、oxa-2、mcr-1、acc或mox中的任意一种或至少两种的组合。
本发明中上述检测基因对应耐受的药物包括碳青霉烯类、头孢菌素类、甲氧西林类、β-内酰胺类、氟醛诺酮类、多肽类、氨基糖苷类、大环内酯类、四环素类、多粘菌素类或结核相关药物等。
作为本发明优选的技术方案,步骤(5)所述捕获探针还包含对照探针,所述对照探针用于检测人内源基因,包括gapdh、atm、rad51或β-actin中的任意一种或至少两种的组合。
优选地,步骤(5)所述捕获探针带有生物素标记,长度为80~120bp,例如80bp、85bp、90bp、95bp、100bp、105bp、110bp、115bp或120bp等。
本发明中,所述捕获在目标基因的外显子上均匀分布;同时,本发明还对设计的所有探针序列进行比对去除冗余探针,形成bed文件。
此外,由于捕获测序在方法学上的优势,本发明可以在上述基因的基础上进一步增加捕获基因或者扩大区域范围,进而得到更多的测序信息。
作为本发明优选的技术方案,步骤(5)所述二代高通量测序的数据分析方法包括:
使用fastp对低质量的数据进行过滤,使用bwa比对耐药基因数据库,使用samtools进行去重并实现dna和rna数据的分类。
本发明中,所述数据分析流程可以按照如下流程进行:
1)下机数据质控:使用fastp(https://github.com/opengene/fastp)对低质量的数据进行过滤;
2)序列比对:使用bwa(http://bio-bwa.sourceforge.net/)比对耐药基因数据库;
3)数据处理:数据使用samtools(http://samtools.sourceforge.net/)和gencore(https://github.com/opengene/gencore)进行dna和rna数据sort和去除重复(duplicate)。
本发明中,所述共检测方法可以按照如下步骤进行:
(1)样本提取:对样本中的dna和/或rna进行提取,所述样本包括全血、血浆、唾液、脑髓液、关节液、痰液、肺泡灌洗液、尿液、粪便、腹胸腹水和组织等,也可包括经处理或未经处理的实验室材料或人工合成的测试品等;
(2)片段化方式可以是超声法或酶切法等;例如,本发明中可使用bioruptorpico打断仪进行超声打断;
打断条件:4℃条件下,设置超声30s,间隔30s,4~6个循环,取出样品,振荡混匀,短暂离心,重复上述打断步骤8~10次;
(3)cdna第一条链合成:使用反转录随机引物和逆转录酶对片段化的核酸进行反转录;
再对片段化的核酸进行末端修复和加碱基a,然后两端加上测序接头,并用磁珠纯化以除去未连接的接头序列;
(4)pcr文库扩增加index区分不同样本,纯化后并进行文库质控;构建好的文库通过qubit3.0和agilent2100进行浓度检测和长度分布检测来进行质量评价;
随后,可以使用mngs直接进行测序,例如利用illuminanextseq550平台进行pe75测序,即可得到病原耐药基因的存在情况和表达情况。
若需要进行序列捕获则进行后续步骤;
(5)将dna文库和探针进行文库杂交反应,杂交时间为0.5~5h(例如0.5h、1h、1.5h、2h、2.5h、3h、3.5h、4h、4.5h或5h等),杂交后再pcr然后纯化捕获片段;
(6)将捕获的目标dna文库进行二代高通量测序,对低质量的数据进行过滤,比对耐药基因数据库去除重复并结合人参照基因的测序深度,实现dna和rna数据的分类,进而得到病原耐药基因的存在情况和表达情况。
第四方面,本发明还提供一种如第三方面所述的共检测方法在非疾病的诊断和治疗目的的、病原微生物的鉴定和/或检测中的应用。
本发明所述的数值范围不仅包括上述列举的点值,还包括没有列举出的上述数值范围之间的任意的点值,限于篇幅及出于简明的考虑,本发明不再穷尽列举所述范围包括的具体点值。
与现有技术相比,本发明的有益效果为:
(1)本发明提供一种反转录随机引物,其在5'端增加固定碱基序列标记,能够并不影响反转录的进行,同时还能标记反转录后的rna,达到区分耐药基因的dna和表达的rna的目的;该结构为rt-qpcr和宏基因组测序中dna和rna的区分提供了实验基础;
(2)本发明提供的共检测方法,配合反转录随机引物,无论是通过mngs还是捕获测序,都可以通过单次实验测序得到耐药相关基因在dna和rna两个层面的测序数据,并根据所得数据进行分析;且结合本发明中实施例所得的数据可知,在多数情况下耐药基因dna存在与否和药敏相关性较低,所以判断耐药基因是否转录和表达对指导临床用药更有意义;
(3)由于部分耐药基因存在序列相似度高的特点,特别是归属于同一耐药基因家族的不同成员,如果使用多重扩增测序法难度较大,但设计探针捕获可以有效避开;本发明还结合快速杂交,在保证质量的前提下,尽可能缩短监测时间,满足临床需求;同时,鉴于捕获测序的方法学优势,本发明还可以在所述的耐药基因基础上进一步增加捕获基因或扩大区域范围,得到更多测序信息,因此,本发明提供的共检测方法在病原微生物的鉴定或检测中具有广阔的应用前景。
附图说明
图1为实施例3中捕获的目的文库的质控结果图。
图2为实施例3中下机测序数据的分析处理流程图。
具体实施方式
下面结合附图并通过具体实施方式来进一步说明本发明的技术方案,但下述的实例仅仅是本发明的简易例子,并不代表或限制本发明的权利保护范围,本发明的保护范围以权利要求书为准。
以下实施例中,若无特殊说明,所用试剂及耗材均购自本领域常规试剂厂商;若无特殊说明,所用实验方法和技术手段均为本领域常规的方法和手段。
实施例1
本实施例中使用反转录随机引物进行rt-qpcr检测,以验证反转录随机引物能够正常使用完成反转录步骤。
(1)以三份正常人新鲜血液作为待测样本;
(2)rna提取:使用qiagenqiaamprnabloodminikit提取三份样本的全血rna,其操作步骤参考试剂盒的使用说明书;
(3)cdna第一条链合成:反转录中使用的反转录酶为invitrogen公司的superscriptiv逆转录酶,具体反应体系如下表1所示:
其中,随机引物中n表示随机碱基a、t、g或c中的任意一个,随机引物的长度10bp。
在冰上配制体系,混合均匀后短暂离心,然后在23℃,10min;55℃,10min;80℃,10min进行反转录;
(4)qpcr扩增:针对人同源重组蛋白rad51基因设计引物和探针,引物探针列表如下表2所示,每个样本三个重复,一共9个复孔。
以上一步的反转录产物为模板在abi7900上进行定量荧光pcr,反应体系和条件如下表3和表4所示:
(5)结果分析:
样本1的平均ct值(meanct)为27.8,样本2的平均ct值(meanct)为28.1,样本3的平均ct值(meanct)为27.9。
由实验结果可知,检测得到的ct值正常且不同样本的结果接近,证明本发明设计的随机引物可以正常使用完成反转录实验。
实施例2
本实施例中以实施例1中提供的反转录随机引物配合mngs检测rna病毒及其耐药基因。
(1)样本选择:以5例明确rna病毒阳性的肺泡灌洗液为样本;
(2)核酸提取和片段化:本实施例使用广州赛哲生物科技股份有限公司的dna和rna共提取试剂盒(粤穗械备20200771)进行核酸提取,
核酸片段化:取上一步处理的核酸,置于打断管中,使用bioruptorpico打断仪进行打断;
打断条件:4℃条件下,设置超声30s,间隔30s,5个循环,取出样品,振荡混匀,短暂离心,重复上述打断步骤10次。
(3)反转录:cdna第一条链合成参考实施例1中所述的步骤;
(4)末端修复:采用kapahyper文库构建试剂盒,其中反应体系如下表5所示:
在冰上配制上述反应体系,混合均匀后短暂离心,然后在20℃反应30min;65℃反应30min;
(5)接头连接:按照下表6配制体系;
混合均匀,短暂离心后20℃反应15min,4℃保存;
(6)连接产物纯化:提前半小时将agencourtampurexp磁珠放在室温下平衡,使用0.6×磁珠对连接产物进行纯化,用22μl无核酶水溶解,取20μl到下一步反应中;
(7)文库pcr扩增:在冰上配制如下表7所示的反应体系;
将配制好的体系混合均匀,短暂离心,然后运行如下表8所示的反应程序;
(8)文库纯化:使用1.0×ampurexp磁珠对pcr产物进行纯化,用22μl无核酶水溶解,吸取20μl纯化后的文库到干净的1.5ml离心管中。
(9)质控和测序:构建好的文库通过qubit3.0和agilent2100进行浓度检测和长度分布检测来进行质量评价;纯化的文库利用illuminanextseq550平台进行pe75测序。
(10)测序结果分析:所得结果如下表9所示,由表可知,mngs宏基因组检测和临床结果一致,rna病毒确诊病例中有耐药基因检出但多数不表达。
实施例3
本实施例中以实施例1中提供的反转录随机引物配合捕获测序检测肺泡灌洗液样本中的耐药基因及其表达情况。
(1)样本选择:以4例抗生素耐受的肺泡灌洗液作为待测样本;
(2)杂交捕获前步骤同实施例2;
(3)杂交捕获测序:在1.5mllobind离心管中将4个文库(500ng/文库)混合成一个pool,充分拨弹混匀后离心将混合物收集到管底,然后用封口膜封住离心管口,并在封口膜上刺几个小孔,最后用抽真空浓缩仪抽干;
按照表10加入各组分到离心管底,吹吸、混匀后全部转移到200μlpcr管中,95℃变性10min;
加入2μl探针mix后涡旋大约3s混合均匀(最后体积21μl),65℃经过0.5h快速杂交捕获;同时将dynabeadsm270链酶亲和素t1磁珠从4℃冰箱取出,室温平衡30min;
所述探针序列的序列由表11中seqidno.5~37所示;
完成杂交反应后用t1磁珠吸附捕获文库并纯化。
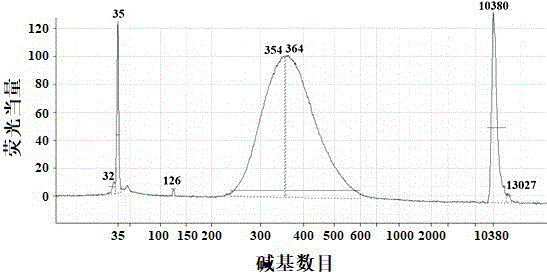
将捕获的目的文库用通用引物进行8个循环扩增,用1.0×ampurexp磁珠纯化,质控结果如图1所示。
如图1所示,所得文库的片段长度集中在300~500bp,所得文库质量较为稳定。
(4)测序数据分析处理:
具体流程如图2所示:
s1)测序下机reads:纯化的文库利用illuminaminiseq平台进行pe75测序;
s2)低质量序列过滤:将测序下机reads使用fastp(https://github.com/opengene/fastp)对低质量的数据进行过滤;
s3)捕获基因序列比对,该步骤包括:
s4)使用bwa(http://bio-bwa.sourceforge.net/)比对耐药基因数据库,去除重复的耐药基因序列;和s5)结合人参照内源基因(即人源基因gapdh、atm、rad51和β-actin)的测序深度;
s6)区分dna和rnareads,即进行dna和rna数据分类。
所得结果如下表12所示,其中包括总映射率(totalmappingrate,%)、目标上限率(targetcaprate,%)、20×覆盖率(20×coverage,%)、存在的dna片段、表达的rna和临床表现的耐药种类。
由表可知,待测样本中测得的dna片段明显多于rna片段,说明在耐药菌中可能含有多种耐药基因,但是多数的耐药基因并未表达;而且,通过上述方法测得的表达的rna,其对应的耐药基因基本与药敏试验结果一致。
综上所述,本发明提供的共检测方法,可以通过单次实验测序得到耐药相关dna和rna两个层面的测序数据进行分析,且从结果可知,多数情况下耐药基因dna的存在与否和药敏相关性较低,需要依据其表达情况即rna来判断其药敏类型,因此,本发明所述的共检测方法对指导临床用药具有重要的意义。
申请人声明,以上所述仅为本发明的具体实施方式,但本发明的保护范围并不局限于此,所属技术领域的技术人员应该明了,任何属于本技术领域的技术人员在本发明揭露的技术范围内,可轻易想到的变化或替换,均落在本发明的保护范围和公开范围之内。
序列表
<110>广州赛哲生物科技股份有限公司
<120>一种病原耐药基因的存在及表达情况的共检测方法
<130>20210618
<160>37
<170>siposequencelisting1.0
<210>1
<211>10
<212>dna
<213>人工合成()
<220>
<221>misc_feature
<222>(1)..(1)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(5)..(10)
<223>nisa,c,g,ort
<400>1
nactnnnnnn10
<210>2
<211>18
<212>dna
<213>人工合成()
<400>2
gaattagtccttacgcaa18
<210>3
<211>21
<212>dna
<213>人工合成()
<400>3
ttggcgggaattctgaaagcc21
<210>4
<211>17
<212>dna
<213>人工合成()
<400>4
cgtgaccctgggcgaga17
<210>5
<211>94
<212>dna
<213>人工合成()
<400>5
atacgaaggtgaacatcatagacacgccaggacatatggatttctttatatcgttcatta60
tcagttttagatggggcaattctactgatttctg94
<210>6
<211>91
<212>dna
<213>人工合成()
<400>6
ttcttcaccacccagggacgggtttatcgggccaaggcctacgacttgcccgaggcctcc60
cggacggcgcgcgggcagcacgtggccaacc91
<210>7
<211>100
<212>dna
<213>人工合成()
<400>7
tctcttgccactttcgcgcatgcgcaagaaggcacgctagaacgttctgactggaggaag60
tttttcagcgaatttcaagccaaaggcacgatagttgtgg100
<210>8
<211>97
<212>dna
<213>人工合成()
<400>8
gtttagctttgtcggcatttaatgctcatgctaacaaaagtgatgctgcggcaaaacaaa60
taaaaaaattagaggaagactttgatgggaggattgg97
<210>9
<211>102
<212>dna
<213>人工合成()
<400>9
ctatggtaatgctcttgcacgagcaaataaagaatatgtccctgcatcaacatttaagat60
gctaaatgctttaatcgggctagaaaatcataaagcaacaac102
<210>10
<211>106
<212>dna
<213>人工合成()
<400>10
tgtactctcgccggataaaaaaatgcacggattacttgttaaaaagaaccatgaatatga60
aatcaaccatgttgatgtagcattttcagctttgcatggcaagtca106
<210>11
<211>100
<212>dna
<213>人工合成()
<400>11
gtggaaagcgctttagcggtgtgggatgagcccgcggcctatcagcttgttggtggggtg60
acggcctaccaaggcgacgacgggtagccggcctgagagg100
<210>12
<211>103
<212>dna
<213>人工合成()
<400>12
gattaattcgatgcaacgcgaagaaccttacctgggtttgacatgcacaggacgcgtcta60
gagataggcgttcccttgtggcctgtgtgcaggtggtgcatgg103
<210>13
<211>106
<212>dna
<213>人工合成()
<400>13
aatagtgatcttgaggctaatgttcccatgattaaagcaagtataaaaaaactaggtttt60
aaattcagtgatactaaaattttgctgattagccatgctcattttg106
<210>14
<211>102
<212>dna
<213>人工合成()
<400>14
ccatactctccccggataggaaaacgcatgggctgcttgtcatgaaagaaagcgaatacg60
aaacacggcgtattgatgtggctttcccggttttgcatggca102
<210>15
<211>101
<212>dna
<213>人工合成()
<400>15
tcgggctgtgaggtcgggtgtgcggtcatggggaacgaggatgatttgattgtcggcgaa60
gtggatcaaatccggctgagccacggtatcttccgcatcca101
<210>16
<211>100
<212>dna
<213>人工合成()
<400>16
accgcatcgtcatggatcgggaggtcgtcaagctgggcggcatcgcgttcaccgcccatt60
tcatgcccgggcacacgccggggagcaccgcctggacctg100
<210>17
<211>97
<212>dna
<213>人工合成()
<400>17
cgcgcacaccgatcacgccggccctgtggccgagatcaagcgccgcacgggggcgcaggt60
ggtcgtgaacgccgaaaccgcagtgctgctggcccga97
<210>18
<211>101
<212>dna
<213>人工合成()
<400>18
gatgaacatcggccagattttggagacccacctgggttggtgtgcccacagcggctggaa60
ggtcgacgccgccaagggggttccggactgggccgccaggc101
<210>19
<211>104
<212>dna
<213>人工合成()
<400>19
ggacatgacgtacgcggctccactgttcgtcaccgccgagttcatcaacaacaacaccgg60
tgagatcaagagtcagacggtgttcatgggtgacttcccgatga104
<210>20
<211>102
<212>dna
<213>人工合成()
<400>20
cacagaaagcgcctgagtaagcgtttgctgctgcccatgttgctgtgtttattggctcaa60
caaacgcaggctgtggcagctgagcagaccaaggtcagtgac102
<210>21
<211>104
<212>dna
<213>人工合成()
<400>21
gatgaaaccagtcgttttagctgggacggaaaggttcgcgaaattgccgtctggaacagg60
gaccagagttttcgcaccgcaatgaagtactctgtggtgcctgt104
<210>22
<211>104
<212>dna
<213>人工合成()
<400>22
ataagtattggggcttgtgctgagcatagtatgagtcgagcaaaaacaagtacaattcca60
caagtgaataactcaatcatcgatcagaatgttcaagcgctttt104
<210>23
<211>96
<212>dna
<213>人工合成()
<400>23
ccatgcaagcatctacagtgcctgtatatcaagaattggcacgtcgtattggtccaagct60
taatgcaaagtgaattgcaacgtattggttatggca96
<210>24
<211>102
<212>dna
<213>人工合成()
<400>24
tcaagatactgaacacgctgatctcgctcgaagaaaaagccatctctggaaaagacgatg60
tgctgaaatgggacgggcatatttacgattttccagattgga102
<210>25
<211>104
<212>dna
<213>人工合成()
<400>25
caagatactgaacacgctgatctcgctcgaagaaaaagccatctctggaaaagacgatgt60
gctgaaatgggacgggcatatttacgattttccagattggaatc104
<210>26
<211>104
<212>dna
<213>人工合成()
<400>26
tgcctcgacgctactgatggtatcttttagttcattcgaaacctcagcgcaatccccact60
gttaaaagagcaaattgaatccatagtcattggaaaaaaagcca104
<210>27
<211>101
<212>dna
<213>人工合成()
<400>27
tagccgtcaacgacataggctctagcgaggactttctcgccgcaatagacaaaacgatca60
agtacttcaacgatggcgacatcgtcgaaggcaccatcgtc101
<210>28
<211>100
<212>dna
<213>人工合成()
<400>28
agggtgtcgtgtcctcgatcgtcaacttcggcgcgttcgtcgatctcggcggtgtggacg60
gtctggtgcatgtctccgagctatcgtggaagcacatcga100
<210>29
<211>105
<212>dna
<213>人工合成()
<400>29
agcagagtctttgccagatttaaaaattgaaaagcttgatgaaggcgtttatgttcatac60
ttcgtttgaagaagttaacgggtggggcgttgttcctaaacatgg105
<210>30
<211>106
<212>dna
<213>人工合成()
<400>30
ttggcgaagtaagaatgcagaaataccttaaaaaattttcctatggcaaccagaatatca60
gtggtggcattgacaaattctggttggaaggccagcttagaatttc106
<210>31
<211>105
<212>dna
<213>人工合成()
<400>31
gaaaaccagctaatagtaaaagaggctttggtaacggaggcggcacctgaatatctagtg60
cattcaaaaactggtttttctggtgtgggaactgagtcaaatcct105
<210>32
<211>107
<212>dna
<213>人工合成()
<400>32
ctctttgatgttccatgggaaaaaatacagtaccaaagcctcatggataccattaaaaaa60
cgtcataatttaccggttgtagcggtatttgccacacactcccatga107
<210>33
<211>105
<212>dna
<213>人工合成()
<400>33
gaatttcggccacggagcaaatcagctttttaagaaagctgtatcacaataagttacacg60
tatcggagcgcagccagcgtattgtcaaacaagccatgctgaccg105
<210>34
<211>105
<212>dna
<213>人工合成()
<400>34
cgatgaaatattcagttgtgcctgtttatcaagaatttgcccgccaaattggcgaggcac60
gtatgagcaagatgctacatgctttcgattatggtaatgaggaca105
<210>35
<211>106
<212>dna
<213>人工合成()
<400>35
tcgatggtggtattcgcatttcggctacccagcaaatcgcttttttacgcaagctgtatc60
acaacaagctgcacgtttctgagcgtagtcagcgcatcgtgaaaca106
<210>36
<211>106
<212>dna
<213>人工合成()
<400>36
ccgcgatgaagtactcagttgtgcctgtttatcaagaatttgcccgccaaattggtgagg60
cacgtatgagtaaaatgctgcacgccttcgattatggcaatgagga106
<210>37
<211>105
<212>dna
<213>人工合成()
<400>37
gcatctttgtctacttccccgaccagcaggtgctctatggcaactgcatcctcaaggaga60
agctgggcaacctgagctttgccgatgtgaaggcctatccgcgca105
- 还没有人留言评论。精彩留言会获得点赞!