一种药物-疾病关系识别方法、系统和装置与流程
本发明涉及计算机辅助药物设计领域,尤其是一种药物-疾病关系识别方法、系统和装置。
背景技术:
新药研发一直是一个费时、费力、高投入、高风险的过程。充分挖掘已有药物的新的治疗效果,对药物进行重定位已经成为新药研发中降低风险和投入、提高成功率的一种方法。目前,美国食品药品监督管理局批准上市的药物已经超过2000种,医学一体化语言系统数据库收集的疾病已经超过25000种。这些药物和疾病组成数以亿计的药物-疾病关系对。如何从这些关系对中发现具有潜在治疗关系的药物-疾病关系,是药物重定位以及新药研究的关键。由于药物-疾病关系对数量巨大等原因,采用传统的医学、生物学或者化学等实验方法不能在合理的时间内逐一研究这些药物-疾病关系对。因此,为了分析并且整合已有的药物和疾病的相关信息、识别潜在的药物-疾病关系对,本发明采用深度学习等技术研究药物-疾病关系对。
技术实现要素:
为了解决上述技术问题,本发明的第一目的在于提供一种药物-疾病关系识别方法,第二目的在于提供一种药物-疾病关系识别系统,第三目的在于提供一种药物-疾病关系识别装置。
本发明所采取的第一技术方案是:
一种药物-疾病关系识别方法,包括以下步骤:
获取待识别药物-疾病关系对所对应的药物-疾病关系二维矩阵和/或灰度图;
将获得的药物-疾病关系二维矩阵和/或灰度图输入到卷积神经网络中进行处理,从而输出药物-疾病关系识别结果。
进一步地,所述将药物-疾病关系二维矩阵和/或灰度图输入到卷积神经网络中进行处理这一步骤之前,设有建立卷积神经网络的步骤,所述建立卷积神经网络的步骤具体包括:
获取药物-疾病数据库中的药物-疾病关系对所对应的药物-疾病关系二维矩阵、灰度图和药物-疾病关系值,利用所得的药物-疾病关系二维矩阵和灰度图构建输入数据正样本,利用所得药物-疾病关系值构建输出数据正样本;
获取药物-疾病数据库外的药物-疾病关系对所对应的药物-疾病关系二维矩阵、灰度图和药物-疾病关系值,利用所得药物-疾病关系二维矩阵和灰度图构建输入数据负样本,利用所得药物-疾病关系值构建输出数据负样本;
抽选输入数据正样本和输入数据负样本从而分别构建训练输入数据集和测试输入数据集;抽选输出数据正样本和输出数据负样本从而分别构建训练输出数据集和测试输出数据集;
用训练输入数据集以及训练输出数据集训练卷积神经网络,用测试输入数据集以及测试输出数据集测试卷积神经网络;
将训练和测试结束后得到的卷积神经网络作为所需建立的卷积神经网络。
进一步地,所述药物-疾病数据库外的药物-疾病关系对,其通过以下步骤来获取得到:
随机选择药物-疾病数据库中药物-疾病关系对的药物和疾病,匹配成新的药物-疾病关系对;
判断新的药物-疾病关系对是否已存在于药物-疾病数据库中,若是,重新进行上一步骤,反之,将新的药物-疾病关系对作为所需获取的药物-疾病数据库外药物-疾病关系对。
进一步地,所述药物-疾病关系对所对应的药物-疾病关系二维矩阵和灰度图,其通过以下步骤来获取得到:
获取药物-疾病关系对中药物的药物分子结构信息,根据药物分子结构信息得到分子指纹描述符;
获取药物-疾病关系对中疾病的疾病信息,根据疾病信息得到疾病症状向量;
根据分子指纹描述符和疾病症状向量,从而构建药物-疾病关系二维矩阵和灰度图。
进一步地,所述根据分子指纹描述符和疾病症状向量,从而构建药物-疾病关系二维矩阵和灰度图这一步骤中,使用以下公式构建药物-疾病关系二维矩阵:
式中,
di表示第i个药物分子,dj表示第j个疾病;
di(m)表示第i个药物分子的指纹描述符中的第m个元素;
dj(n)表示第j个疾病的疾病症状向量中的第n个元素。
进一步地,所述根据分子指纹描述符和疾病症状向量,从而构建药物-疾病关系二维矩阵和灰度图这一步骤中,使用以下方法构建灰度图:
利用matlab软件中的imwrite命令对药物-疾病关系二维矩阵进行处理,从而得到对应的灰度图。
进一步地,所述建立卷积神经网络的步骤之前,设有将药物-疾病数据库去冗余的步骤,所述将药物-疾病数据库去冗余的步骤具体包括:
在药物-疾病数据库中获取两个药物-疾病关系对;
计算两个药物-疾病关系对中两个药物分子的相似性;
计算两个药物-疾病关系对中两个疾病的相似性;
根据两个药物分子的相似性以及两个疾病的相似性,计算两个药物-疾病关系对的相似性;
判断两个药物-疾病关系对的相似性是否大于预设的阈值,若是,随机选择其中一个药物-疾病关系对并将其从药物-疾病数据库中删除,然后返回所述在药物-疾病数据库中获取两个药物-疾病关系对这一步骤,反之,直接返回所述在药物-疾病数据库中获取两个药物-疾病关系对这一步骤;直至使药物-疾病数据库中任意两个药物-疾病关系对的相似性均小于预设的阈值为止。
进一步地,所述两个药物分子的相似性,其计算公式如下所示:
式中,
j(d1,d2)表示两个药物分子的相似性;
∩和∪分别表示几何理论中的交集和并集操作;
所述两个疾病的相似性,其计算公式如下所示:
式中,
cos(d1,d2)表示第一疾病d1和第二疾病d2的相似性;
所述两个药物-疾病关系对的相似性,其计算公式如下所示:
式中,
s(d1-d1,d2-d2)表示两个药物-疾病关系对的相似性;
d1-d1表示第一药物分子d1和第一疾病d1组成的第一药物-疾病关系对;
d2-d2表示第二药物分子d2和第二疾病d2组成的第二药物-疾病关系对。
本发明所采取的第二技术方案是:
一种药物-疾病关系识别系统,其包括:
获取模块,用于获取待识别药物-疾病关系对对应的药物-疾病关系二维矩阵和/或灰度图;
处理模块,用于将获得的药物-疾病关系二维矩阵和/或灰度图输入到卷积神经网络中进行处理,从而输出药物-疾病关系识别结果。
一种药物-疾病关系识别系统,其包括:
获取模块,用于获取待识别药物-疾病关系对对应的药物-疾病关系二维矩阵和/或灰度图;
处理模块,用于将获得的药物-疾病关系二维矩阵和/或灰度图输入到卷积神经网络中进行处理,从而输出药物-疾病关系识别结果。
本发明所采取的第三技术方案是:
一种药物-疾病关系识别装置,其包括:
存储器,用于存储至少一个程序;
处理器,用于加载所述至少一个程序以执行权利要求1-8任一项所述一种药物-疾病关系识别方法。
本发明的第一有益效果是:通过本发明方法,考虑了药物分子的结构、疾病的症状以及两者之间的关系,用卷积神经网络对待识别药物-疾病关系对对应的药物-疾病关系二维矩阵和/或灰度图进行处理,从而对药物-疾病关系进行识别研究,充分利用了卷积神经网络运算速度快、识别率高的优点,能够快速准确地识别药物-疾病关系对是否具有治疗关系,即待识别药物-疾病关系对中的药物能否治疗对应的疾病,从而识别潜在的药物-疾病相互作用,开展先导化合物识别和药物重定位研究。
进一步通过本发明方法,还可以根据数据库中已知的药物-疾病对应治疗关系,构建正样本和负样本,从而得到合适的训练集和测试集,用来训练和测试卷积神经网络,利用卷积神经网络的学习功能,使得训练和测试后的卷积神经网络具备药物-疾病关系识别功能。进一步通过本发明方法,还可以根据药物-疾病关系对得到其对应的药物-疾病关系二维矩阵和/或灰度图,使其与卷积神经网络的输入数据格式要求匹配,从而利用已知关系的药物-疾病关系对卷积神经网络进行训练,或用卷积神经网络识别待识别药物-疾病关系对。进一步通过本发明方法,还可以对药物-疾病数据库进行去冗余处理,提高卷积神经网络的训练和测试效率,对药物和疾病的相似性具有鲁棒性,具有简单、快速和准确等优点。
本发明的第二有益效果是:通过本发明系统,考虑了药物分子的结构、疾病的症状以及两者之间的关系,用卷积神经网络对待识别药物-疾病关系对对应的药物-疾病关系二维矩阵和/或灰度图进行处理,从而对药物-疾病关系进行识别研究,充分利用了卷积神经网络运算速度快、识别率高的优点,能够快速准确地识别药物-疾病关系对是否具有治疗关系,即待识别药物-疾病关系对中的药物能否治疗对应的疾病,从而识别潜在的药物-疾病相互作用,开展先导化合物识别和药物重定位研究。
本发明的第三有益效果是:通过本发明装置,考虑了药物分子的结构、疾病的症状以及两者之间的关系,用卷积神经网络对待识别药物-疾病关系对对应的药物-疾病关系二维矩阵和/或灰度图进行处理,从而对药物-疾病关系进行识别研究,充分利用了卷积神经网络运算速度快、识别率高的优点,能够快速准确地识别药物-疾病关系对是否具有治疗关系,即待识别药物-疾病关系对中的药物能否治疗对应的疾病,从而识别潜在的药物-疾病相互作用,开展先导化合物识别和药物重定位研究。
附图说明
图1是本发明药物-疾病关系识别方法的流程图;
图2是获取药物-疾病关系二维矩阵和灰度图的流程图;
图3是本发明药物-疾病关系识别系统的结构图;
图4是本发明药物-疾病关系识别装置的结构图。
具体实施方式
实施例1
本发明公开的一种药物-疾病关系识别方法,如图1所示,包括以下步骤:
获取待识别药物-疾病关系对所对应的药物-疾病关系二维矩阵和/或灰度图;
将获得的药物-疾病关系二维矩阵和/或灰度图输入到卷积神经网络中进行处理,从而输出药物-疾病关系识别结果。
卷积神经网络可以对图像或图像对应的矩阵进行处理,从而输出处理结果。本发明方法中,为了识别某种药物d能否治疗疾病d,将药物d与疾病d视作或匹配成一个药物-疾病关系对d-d,获取药物-疾病关系对d-d的药物-疾病关系二维矩阵和/或灰度图,并且将药物-疾病关系二维矩阵和/或灰度图输入到卷积神经网络中进行处理,从而输出药物-疾病关系识别结果。卷积神经网络输出的输出药物-疾病关系识别结果,按照卷积神经网络所受的训练,可以是图像、矩阵、数值或其他形式的数据,其表示药物d能够治疗疾病d或者药物d不能治疗疾病d,还可表示药物d对疾病d的治疗效果定量评价值或其他识别结果。需要指出的是,将药物d与疾病d匹配成一个药物-疾病关系对d-d可以是出于处理上的方便而在观念上将其视为一个整体,并不限制于对药物d与疾病d做任何实质处理使其连结成一个结合体。本发明中,对“药物”和“药物分子”两个概念不做严格区分。
实施例2
以下结合本实施例的优选内容对本发明做进一步地阐述。本实施例的药物-疾病关系识别方法可以通过实施例3中的药物-疾病关系识别系统以及实施例4中的药物-疾病关系识别装置实现。
一种药物-疾病关系识别方法,包括以下步骤:
获取待识别药物-疾病关系对对应的药物-疾病关系二维矩阵和/或灰度图;
将获得的药物-疾病关系二维矩阵和/或灰度图输入到卷积神经网络中进行处理,从而输出药物-疾病关系识别结果。
卷积神经网络可以对图像或图像对应的矩阵进行处理,从而输出处理结果。
进一步作为优选的实施方式,所述将药物-疾病关系二维矩阵和/或灰度图输入到卷积神经网络中进行处理这一步骤之前,设有建立卷积神经网络的步骤,建立卷积神经网络的步骤具体包括:
获取药物-疾病数据库中药物-疾病关系对对应的药物-疾病关系二维矩阵、灰度图和药物-疾病关系值,用所得药物-疾病关系二维矩阵和灰度图构建输入数据正样本,用所得药物-疾病关系值构建输出数据正样本;
获取药物-疾病数据库外药物-疾病关系对对应的药物-疾病关系二维矩阵、灰度图和药物-疾病关系值,用所得药物-疾病关系二维矩阵和灰度图构建输入数据负样本,用所得药物-疾病关系值构建输出数据负样本;
抽选输入数据正样本和输入数据负样本从而分别构建训练输入数据集和测试输入数据集;抽选输出数据正样本和输出数据负样本从而分别构建训练输出数据集和测试输出数据集;
用训练输入数据集以及训练输出数据集训练卷积神经网络,用测试输入数据集以及测试输出数据集测试卷积神经网络;
将训练和测试结束后得到的卷积神经网络作为所需建立的卷积神经网络。
上述方法中所用的药物-疾病数据库储存了已知的药物-疾病对应关系,即何种药物能够治疗何种疾病。药物-疾病数据库中药物-疾病关系对是指根据药物-疾病数据库中已知的药物-疾病对应关系配对成的药物-疾病关系对,每一对药物-疾病关系对中的药物已知都能够治疗对应的疾病,且“每一对药物-疾病关系对中的药物已知都能够治疗对应的疾病”这一事实也定量表示为药物-疾病关系值。因此,药物-疾病数据库中药物-疾病关系对对应的药物-疾病关系二维矩阵和灰度图,可以作为用来训练和测试卷积神经网络的输入数据正样本,对应的药物-疾病关系值,可以作为用来训练和测试卷积神经网络的输出数据正样本。
为了构建用来训练和测试卷积神经网络的输入数据负样本和输出数据负样本,需要用到上述药物-疾病数据库不存在的药物-疾病关系对,即药物-疾病数据库外药物-疾病关系对,其每一对药物-疾病关系对中的药物已知不能够或未知能不能治疗对应的疾病,且“每一对药物-疾病关系对中的药物已知不能够或未知能不能治疗对应的疾病”这一事实也定量表示为药物-疾病关系值。因此,药物-疾病数据库外药物-疾病关系对对应的药物-疾病关系二维矩阵和灰度图,可以作为用来训练和测试卷积神经网络的输入数据负样本,对应的药物-疾病关系值,可以作为用来训练和测试卷积神经网络的输出数据负样本。
进一步作为优选的实施方式,上述方法用到的药物-疾病数据库为ctd数据库,即comparativetoxicogenomicsdatabase。在应用上述方法时,优选地并不直接使用原始ctd数据库,而是使用通过以下方法筛选后的ctd数据库:
删除标记为“marker/mechanism”的药物与疾病关系数据,废除标记为“drugcombination”的药物与疾病关系数据。根据药物在mesh数据库中的id号,获取在pubchemcid数据库中的id号和smiles格式信息,然后去掉没有对应药物smiles格式信息的药物与疾病关系数据。
进一步作为优选的实施方式,通过以下步骤获取所述药物-疾病数据库外药物-疾病关系对:
随机选择药物-疾病数据库中药物-疾病关系对的药物和疾病,匹配成新的药物-疾病关系对;
判断新的药物-疾病关系对是否已存在于药物-疾病数据库中,若是,重新进行上一步骤,反之,将新的药物-疾病关系对作为所需匹配的药物-疾病数据库外药物-疾病关系对。
为了得到药物-疾病数据库中不存在的药物-疾病关系对,优选地在药物-疾病数据库中随机选择一个药物,然后在药物-疾病数据库中随机选择一个疾病,如果这个药物和这个疾病匹配成的新的药物-疾病关系对已在药物-疾病数据库存在,则重新选择并匹配,否则,由于药物-疾病数据库中并不存在这个新的药物-疾病关系对,可以认为这个新的药物-疾病关系对中的药物已知不能够或未知能不能治疗对应的疾病,从而作为所需匹配的药物-疾病数据库外药物-疾病关系对。多次使用本方法,便可以得到足够多的药物-疾病数据库外药物-疾病关系对,从而得到足够大的负样本。
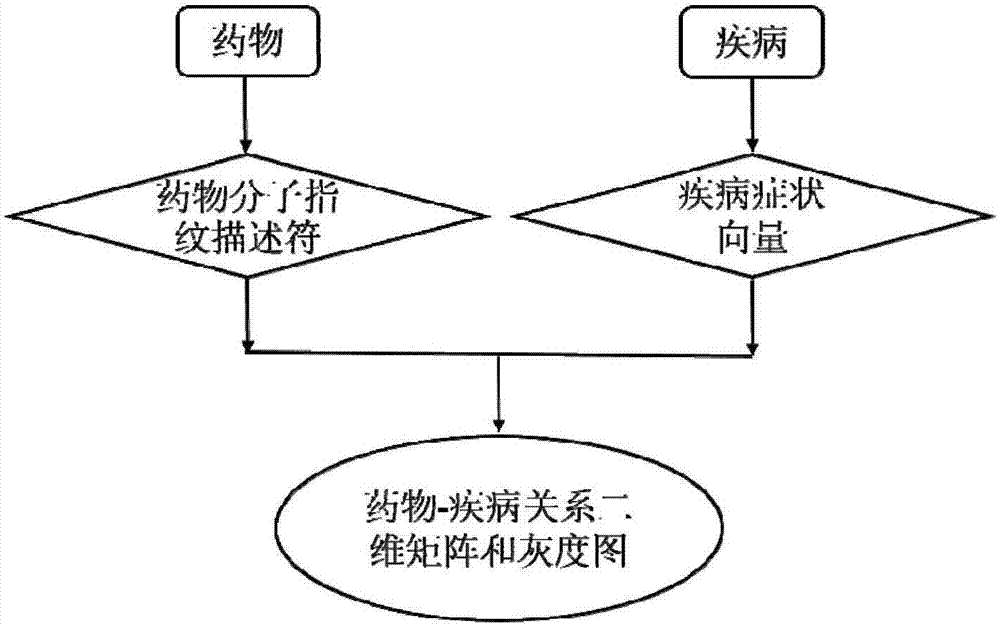
本发明方法中,无论是用药物-疾病数据库中/外药物-疾病关系对对卷积神经网络进行训练和测试,还是使用已经完成训练和测试的卷积神经网络对未知的药物-疾病关系对进行识别,都需要先得到药物-疾病关系对对应的药物-疾病关系二维矩阵和灰度图,如图2所示,通过下列方法可以获得任一个药物-疾病关系对对应的药物-疾病关系二维矩阵和灰度图。
进一步作为优选的实施方式,所述药物-疾病关系对所对应的药物-疾病关系二维矩阵和灰度图,其通过以下步骤来获取得到:
获取药物-疾病关系对中药物的药物分子结构信息,根据药物分子结构信息得到分子指纹描述符;
获取药物-疾病关系对中疾病的疾病信息,根据疾病信息得到疾病症状向量;
根据分子指纹描述符和疾病症状向量,从而构建药物-疾病关系二维矩阵和灰度图。
进一步作为优选的实施方式,根据药物在mesh数据库中的id号,获取对应药物分子的smiles格式信息,smiles格式信息是一种药物分子结构信息。根据药物分子的smiles格式信息,使用padel-descriptor软件包进行计算,从而得到药物分子的pubchem分子指纹描述符,该描述符是一个881维的二进制特征向量,其各元素表示药物分子含有特定分子结构或者官能团的信息,其中,元素取值为“1”表示此药物分子具有某种分子结构或者官能团,元素取值为“0”表示此药物分子没有某种分子结构或者官能团。
进一步作为优选的实施方式,药物-疾病关系对中疾病的疾病信息,具体指此种疾病对应的症状信息,具体为此种疾病包括某些症状,以及不包括某些症状。基于参考文献(zhoux,menchej,barabasial,sharmaa.humansymptoms-diseasenetwork.nat.commun.,2014,5:4212)公开的方法,可以得到药物-疾病关系对中每一种疾病对应的症状信息,从而构建疾病症状向量。所得到的疾病症状向量是322维的二进制特征向量,其各元素表示疾病具有特定症状的信息,其中,元素取值为“1”表示此疾病具有某种症状,元素取值为“0”表示此疾病没有某种症状。
进一步作为优选的实施方式,所述根据分子指纹描述符和疾病症状向量,从而构建药物-疾病关系二维矩阵和灰度图这一步骤中,使用以下公式构建药物-疾病关系二维矩阵:
式中,
di表示第i个药物分子,dj表示第j个疾病;
di(m)表示第i个药物分子的指纹描述符中的第m个元素;
dj(n)表示第j个疾病的疾病症状向量中的第n个元素。
上述给出了药物di和疾病dj匹配成的药物-疾病关系对di-dj对应的药物-疾病关系二维矩阵
进一步作为优选的实施方式,使用以下方法构建灰度图:
利用matlab软件中的imwrite命令对药物-疾病关系二维矩阵进行处理,从而得到对应的灰度图。
matlab是一款矩阵处理软件,使用其imwrite命令对已得到的药物-疾病关系二维矩阵进行处理,便能得到对应的灰度图。
实施例3
本实施例中,将应用实施例2说明的方法来测试卷积神经网络模型性能并给出实验结果。
进一步作为一种优选的实施方式,应用实施例2说明的方法时所用的卷积神经网络模型具体参数如下:其包括1个输入层、3个卷积层(每一层分别包含32,64和128个大小为5×5的卷积核,每一个卷积层后面紧跟一个修正线性单元激活函数和一个2×2大小的下采样层)、4个全连接层(每一层均包含500个神经元,每一层后连接一个dropout层,并且在训练过程中每次50%的神经元不更新连接权重)、1个全连接层(仅包含两个神经元)、1个softmax层和1个分类层的深度卷积神经网络模型。采用带有动量的随机梯度下降算法优化连接权重,动量设置为0.9、迭代次数为50、批尺寸为64、初始学习率为0.01、每迭代10次学习率下降为原来的10%。
进一步作为一种优选的实施方式,用于训练和测试卷积神经网络的数据集大小,由以下方法确定:
在构建得到的输入数据正样本、输出数据正样本、输入数据负样本和输出数据负样本中,随机选取输入数据正样本总数的75%部分、输入数据负样本的75%部分用来构建训练输入数据集;输入数据正样本和输入数据负样本各自剩余的25%部分用来构建测试输入数据集;随机选取输出数据正样本总数的75%部分、输出数据负样本的75%部分用来构建训练输出数据集;输出数据正样本和输出数据负样本各自剩余的25%部分用来构建测试输出数据集。
采用预测总精度,敏感性、特异性、马氏相关系数和受试者工作特征曲线面积评估模型预测能力。以下训练输入数据集和训练输出数据集统称训练集,测试输入数据集和测试输出数据集统称测试集。
(1)基于pubchem分子指纹描述符的训练集和测试集结果如表1所示。对于训练集,当前发明方法获得了89.90%的预测总精度、88.96%的敏感性、90.85%的特异性、0.7982的马氏相关系数和0.9637的受试者工作特征曲线面积。对于测试集,当前发明方法获得了86.51%的预测总精度、86.23%的敏感性、86.79%的特异性、0.7302的马氏相关系数和0.9360的受试者工作特征曲线面积。训练集和测试集的预测总精度、敏感性和特异性均大于85%,马氏相关系数均高于0.7,受试者工作特征曲线面积均高于0.9,表明了当前发明方法的有效性。
表1.基于各种分子指纹描述符的训练集和测试集预测结果
(2)上述(1)的实验中,药物的分子指纹描述符为由smiles结构信息计算得到的pubchem分子指纹描述符。除了pubchem分子指纹描述符,本发明方法针对药物分子的smiles结构信息,也计算了autopair2d、estate、finger、graphonly、maccs和substructure等六类分子指纹描述符。基于这些分子指纹描述符,结合疾病症状向量,构建了新的药物-疾病关系二维矩阵和灰度图。采用深度卷积神经网络构建了模型预测潜在药物-疾病关系,训练集和测试集结果如表1所示。
对于训练集,estate分子指纹描述符获得了最差的预测结果,总预测精度、敏感性、特异性、马氏相关系数和受试者工作特征曲线面积仅83.51%、80.22%、86.79%、0.6716和0.9106。对于测试集,estate分子指纹描述符获得的总预测精度、敏感性、特异性、马氏相关系数和受试者工作特征曲线面积仅83.04%、80.07%、86.00%、0.6619和0.9041。
对于训练集,autopair2d、finger、maccs和substructure分子指纹描述符获得的预测总精度均介于88%和89%之间、敏感性介于86%和88%之间、特异性介于90%左右,马氏相关系数介于0.77和0.78之间,受试者工作特征曲线面积介于0.95和0.96之间。对于测试集,这四类分子指纹描述符获得的预测总精度介于85%和86%之间、敏感性位于84%和86%之间、特异性位于86%和87%间接、马氏相关系数位于0.66和0.72之间、受试者工作特征曲线面积位于0.92和0.94之间。
对于训练集,虽然graphonly分子指纹描述符获得了较高的预测总精度89.28%、敏感性88.55%、特异性90.02%、马氏相关系数0.7858和受试者工作特征曲线面积0.9606,但是这些结果均低于pubchem分子指纹描述符获得的结果。
同样的,对于测试集,虽然graphonly分子指纹描述符获得了总预测精度86.09%、敏感性85.75%、特异性86.43%、马氏相关系数0.7218和受试者工作特征曲线面积0.9330,这些结果也低于pubchem描述符结果。
所有这些结果均表明,pubchem分子指纹描述符能够充分地表征药物分子结构和官能团信息,是最优的分子指纹描述符。
(3)本发明方法中,输入正样本和输出数据正样本(统称正样本)来源于药物-疾病数据库中药物-疾病关系对,输入负样本和输出数据负样本(统称负样本)来源于药物-疾病数据库外药物-疾病关系对。而本发明公开了一个优选的根据药物-疾病数据库中药物-疾病关系对随机匹配出药物-疾病数据库外药物-疾病关系对的方法,由于是随机匹配的,因此每一次匹配得到的药物-疾病数据库外药物-疾病关系对或其组合都不一样,因此每一次根据其得到的负样本组合也就不一样。重复负样本的产生这一过程10次,并且每一次均保证正样本和负样本的数目比例为1:1。然后按照75%和25%的比例,随机划分训练集和测试集,实验结果如表2所示。
表2.10次训练集和测试集统计平均结果
对于训练集,平均预测总精度、敏感性、特异性、马氏相关系数和受试者工作特征曲线面积分别是90.02%、89.15%、90.88%、0.8004和0.9648。这些结果与表1中,pubchem分子指纹描述符获得的结果非常接近。例如,预测总精度、敏感性、特异性、马氏相关系数和受试者工作特征曲线面积仅相差0.12%、0.19%、0.03%、0.0022和0.0011。另外,10次训练集预测结果的相对标准偏差均小于1%,表明,基于10次随机产生负样本的训练集预测结果重现性好。
对于测试集,平均预测总精度、敏感性、特异性、马氏相关系数和受试者工作特征曲线面积分别是86.70%、86.56%、86.84%、0.7340和0.9364。这些结果比表1中pubchem分子指纹描述符结果仅相差0.19%、0.33%、0.05%、0.0038和0.0004。另外,10次测试集预测结果的相对标准偏差均小于1%,表明,基于10次随机产生负样本的测试集预测结果重现性好。
这些结果表明,基于收集的药物-疾病关系正样本数据,采用随机匹配药物和疾病的策略产生负样本构建数据集,10次随机产生负样本的训练集和测试集的预测结果重现性好。因此,本发明构建的随机产生负样本方法是合理的,并具有较好的鲁棒性。
卷积神经网络在训练过程中具有学习功能,能够在用训练输入数据集和训练输出数据集对其的训练过程中自动调整网络参数,以学习从训练输入数据集到训练输出数据集的映射关系。在本发明中,卷积神经网络的训练输入数据集和训练输出数据集来源于药物-疾病数据库中的药物-疾病关系对,以及由药物-疾病数据库中的药物-疾病关系对衍生的药物-疾病数据库外的药物-疾病关系对,因此,药物-疾病数据库中的药物-疾病关系对将影响训练输入数据集和训练输出数据集的内容,进而影响卷积神经网络的学习。药物-疾病数据库中有可能存在多个相似的药物-疾病关系对,通常用多个相似的药物-疾病关系对得到的训练输入数据集和训练输出数据集去对卷积神经网络进行训练,其训练效果对比只用其中一个药物-疾病关系对去进行训练不会有太大的提升,反而因大量使用计算资源而导致训练效率降低,这表明对于卷积神经网络的训练而言,药物-疾病数据库是存在冗余度的,如果将药物-疾病数据库中的多个相似的药物-疾病关系对只保留其中一个而将其他从数据库中剔除,那么就可以降低药物-疾病数据库中的药物-疾病关系对两两之间的相似性,从而将药物-疾病数据库去冗余,即减小药物-疾病数据库的冗余度,通过本发明方法从去冗余后的药物-疾病数据库中获取训练集和测试集对卷积神经网络进行训练和测试,从而提高卷积神经网络的训练和测试效率。
进一步作为一种优选的实施方式,在建立卷积神经网络这一步骤之前,设有将药物-疾病数据库去冗余的步骤,将药物-疾病数据库去冗余的步骤具体包括:
在药物-疾病数据库中获取两个药物-疾病关系对;
计算两个药物-疾病关系对中两个药物分子的相似性;
计算两个药物-疾病关系对中两个疾病的相似性;
根据两个药物分子的相似性以及两个疾病的相似性,计算两个药物-疾病关系对的相似性;
判断两个药物-疾病关系对的相似性是否大于预设的阈值,若是,随机选择其中一个药物-疾病关系对并将其从药物-疾病数据库中删除,然后返回所述在药物-疾病数据库中获取两个药物-疾病关系对这一步骤,反之,直接返回所述在药物-疾病数据库中获取两个药物-疾病关系对这一步骤;直至使药物-疾病数据库中任意两个药物-疾病关系对的相似性均小于预设的阈值为止。
进一步作为一种优选的实施方式,使用下式计算两个药物分子的相似性:
式中,
∩和∪分别表示几何理论中的交集和并集操作;
使用下式计算两个疾病的相似性:
式中,
使用下式计算两个药物-疾病关系对的相似性:
式中,
d1-d1表示第一药物分子d1和第一疾病d1组成的第一药物-疾病关系对;
d2-d2表示第二药物分子d2和第二疾病d2组成的第二药物-疾病关系对。
上述方法计算所得的j(d1,d2)为jaccard相似性系数,表示第一药物分子d1和第二药物分子d2的相似性大小;cos(d1,d2)为余弦夹角值,表示第一疾病d1和第二疾病d2的相似性大小,根据j(d1,d2)与cos(d1,d2)计算所得的s(d1-d1,d2-d2)表示第一药物-疾病关系对d1-d1和第二药物-疾病关系对d2-d2的相似性大小。
对于任何d1-d1和d2-d2,s(d1-d1,d2-d2)的值总是介于0和1之间,如果等于1,表示这两个药物-疾病关系对是完全相同的,如果等于0,表示这两个药物-疾病关系对完全不相同。
进一步作为优选的实施方式,如果预设一个阈值t,并且对于药物-疾病数据库中所有相似性大小大于t的任意两个药物-疾病关系对,随机保留一个,另一个从药物-疾病数据库中剔除,反复操作遍历药物-疾病数据库中所有任意两个药物-疾病关系对,那么就可以保证药物-疾病数据库最后保留的所有药物-疾病关系对任意两个的相似性大小均不超过t,实现药物-疾病数据库的去冗余。
分别设定阈值为0.5、0.6、0.7、0.8和0.9,分别用上述方法将药物-疾病数据库去冗余后,再用本发明方法,获得训练输入数据集、训练输出数据集(统称训练集)和测试输入数据集、测试输出数据集(统称测试集),建立卷积神经网络,卷积神经网络基于不同阈值训练集和测试集的预测结果如表3所示。
表3.基于不同阈值时的训练集和测试集结果
对于训练集,当阈值从0.9减小到0.5时,预测总精度仅从89.37%减小为86.65%、敏感性仅从88.22%变为84.56%、特异性从90.51%减小为88.74%、马氏相关系数从0.7876降低为0.7339、受试者工作特征曲线面积从0.9582减小到0.9403。预测总精度、敏感性和特异性值的减小幅度均小于5%,马氏相关系数和受试者工作特征曲线面积值的降低幅度也小于0.05。
对于测试集,当阈值从0.9减小到0.5时,预测总精度仅从84.88%减小为80.33%、敏感性仅从84.10%变为78.78%、特异性从85.67%减小为81.88%、马氏相关系数从0.6979降低为0.6071、受试者工作特征曲线面积从0.9216减小到0.8766。预测总精度、敏感性和特异性值的减小幅度均小于6%,马氏相关系数和受试者工作特征曲线面积值的降低幅度也小于0.09。
这些结果表明,随着阈值的减小,训练集和测试集性能仅稍微有所降低,本发明方法对药物-疾病关系对的相似性具有鲁棒性,也表明了本发明对药物-疾病数据库的去冗余方法可以在卷积神经网络训练效果仅受很小的影响下实现药物-疾病数据库的去冗余。
实施例4
本发明公开的一种药物-疾病关系识别系统,如图3所示,包括以下模块:
获取模块,用于获取待识别药物-疾病关系对对应的药物-疾病关系二维矩阵和/或灰度图;
处理模块,用于将获得的药物-疾病关系二维矩阵和/或灰度图输入到卷积神经网络中进行处理,从而输出药物-疾病关系识别结果。
这种药物-疾病关系识别系统可以用于实现本发明药物-疾病关系识别方法,包括实施例1-3所涉及的方法。
实施例5
本发明公开的一种药物-疾病关系识别装置,如图4所示,其包括:
存储器,用于存储至少一个程序;
处理器,用于加载所述至少一个程序以执行本发明药物-疾病关系识别方法,包括实施例1-3所涉及的方法。
以上是对本发明的实施进行了具体说明,但本发明创造并不限于所述实施例,熟悉本领域的技术人员在不违背本发明精神的前提下还可做出种种的等同变形或替换,这些等同的变形或替换均包含在本申请权利要求所限定的范围内。
- 还没有人留言评论。精彩留言会获得点赞!