一种双抗体组合物及在制备COVID-19治疗药物中的应用的制作方法
本发明公开了针对sars-cov-2的抗体组合物,属于微生物学和免疫学领域。
背景技术:
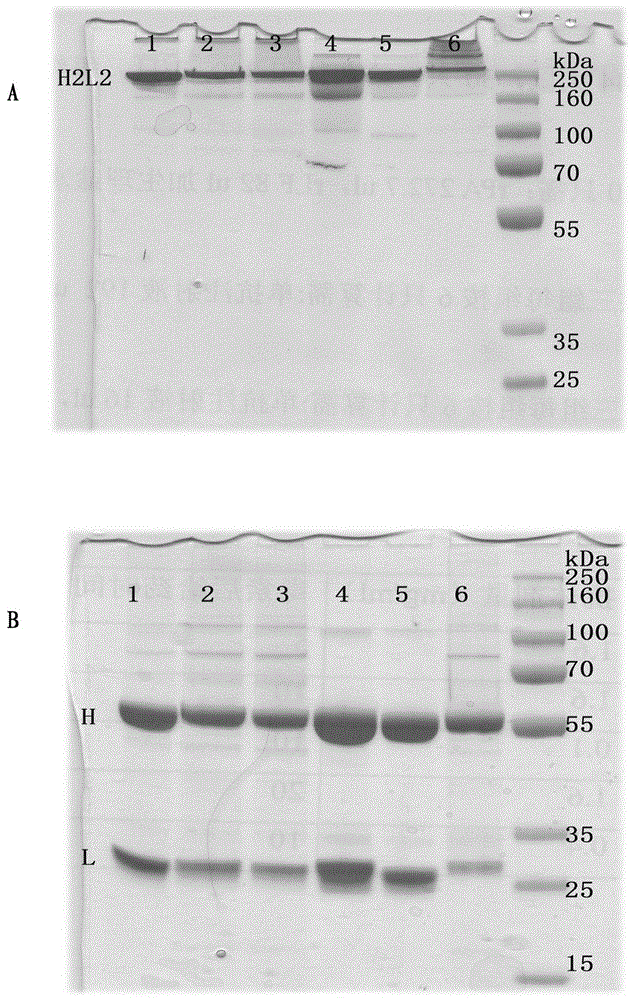
:sars-cov-2属冠状病毒科(coro-naviridae)冠状病毒属(coronavirus),是一类有包膜的单股正链rna病毒。结构蛋白s可特异性与宿主细胞受体结合,是病毒入侵宿主易感细胞的关键蛋白。不同冠状病毒可利用不同的细胞受体来完成入侵,如sars-cov的受体是血管紧张素转化酶2(ace2),而mers-cov的受体是氨基肽酶4(dpp4,也称cd26)。研究证明,氨肽酶n不是sars-cov-2的受体,而ace2可作为sars-cov-2的受体。s蛋白可被宿主蛋白酶裂解为s1与s2两个亚单位,其中s1亚单位位于包膜刺突的顶端,介导病毒与宿主细胞表面的病毒受体结合,是决定病毒入侵易感细胞的关键蛋白;而s2亚单位则形成刺突的茎部,介导病毒包膜与宿主细胞膜融合,促使病毒进入宿主细胞。sars-cov-2的s蛋白为一单次跨膜蛋白,n端大部分氨基酸位于膜外(胞外区),c末端约60个氨基酸组成跨膜区和膜内区。s蛋白胞外区含有1213个氨基酸(yp_009724390.1),分别有s1和s2(yp_009724390.1,ser686-pro1213,528个氨基酸)组成。s1含有685个氨基酸,其中n端1-15个氨基酸为信号肽,成熟的s1含有670个氨基酸,中间部分(arg319-phe541))为受体结合区rbd(receptorbindingresion,yp_009724390.1),s1的n端称为ntd区。血管紧张素转化酶2(angiotensiniconvertingenzyme2,ace2),被证明是新型冠状病毒(severeacuterespiratorysyndromecoronavirus2,sars-cov-2)重要受体。阻断ace2结合sars-cov的s蛋白的抗体可保护小鼠免受sars-cov病毒感染。目前已有研究发现,sars-cov-2的rbd序列(包括直接接触ace2的受体结合基序(receptor-bindingmotif),rbm)与sars-cov的序列相似,sars-cov-2rbm中的几个关键残基与人ace2有良好的相互作用,虽然sars-cov-2rbm中的其他几个关键残基与ace2结合不理想,但这些证据仍表明ace2为致使人类细胞感染sars-cov-2病毒的受体。ace2mrna在支气管和肺实质以及心脏、肾脏和胃肠道中高效表达;这也与新型冠状病毒sars-cov-2引起呼吸系统、消化道反应为主的症状高度吻合。目前还没有专门用于预防感染sars-cov-2所致的冠状病毒病2019(coronavirusdisease2019,covid-19)的疫苗和治疗covid-19的特效药物。一般采用非特异性治疗,预防严重的并发症,降低重症发病率和病死率,提高治愈率。研发covid-19的疫苗和特异治疗药物成为全球应急科研攻关的重要任务。sars-cov-2的s刺突蛋白在病毒对宿主细胞的吸附和侵入过程中发挥着重要作用,而通过阻断病毒对宿主细胞的吸附和穿入是特异性预防sars-cov-2感染的重要途径。研发针对s蛋白的特异性单抗被认为是covid-19的特效药,被寄予厚望。单抗用于传染病治疗,最近比较经典的是埃博拉治疗药物研发,加拿大学者研发的zmapp(嵌合抗体2g4、4g7和13c6组合而成)。2g4和4g7的中和表位交叉,都作用于zaire型gp蛋白的柄部,单独使用不具有中和活性,13c6结合于gp蛋白的头部,单独使用不具有中和活性。临床实验使用zmapp治疗的36个病人中有8人死亡(死亡率22%),而对照组35个病人中有13人死亡(死亡率37%),zmapp展现出了保护效果。2018年11月,who首次多药随机对照试验,用于评估治疗埃博拉的4种药物zmapp、mab114、regn-eb3和remdesvir的安全性和有效性。使用regn-eb3和mab114的患者病死率分别为27%和34%,而使用zmapp和remdesvir的患者死亡率分别为49%和53%。regn-eb3和来自美国国家过敏和传染病研究所(niaid)的mab114显示出“显著的存活率”。相比,再生元研发的单抗组合物在临床试验中效果更好。提示组合物也应该是具有高效中和活性的单抗组合,可以发挥协同提高疗效。regn-cov2是再生元生物公司开发的regn10933中和单抗和regn10987中和抗体的混合物,这两种抗体以非竞争性方式与sars-cov-2病毒刺突蛋白的受体结合区结合。目前,regn-cov-2治疗covid-19住院和非住院患者的评估也已经进入到ii/iii期研究阶段。单抗应用于病毒治疗的潜在风险是病毒逃逸突变株的产生,解决途径是采用“鸡尾酒”疗法,同时给予针对不同识别区域或者不同表位的单抗组合物,增强抗病毒效应,同时最大限度克服病毒逃逸突变株。中国发明专利申请cn111303280a公开了一种靶向新型冠状病毒s蛋白n端结构域的抗体4a8,通过识别表位分析发现,单抗4a8识别的表位位于s1非rbd区。该抗体对于新冠病毒s蛋白具有极高的亲和力,平衡解离常数(kd)为1.00nm,在对veroe6细胞模型感染sars-cov-2的保护效果实验中,显示其ic50达到0.58ug/ml。4a8抗体的获得为中和性抗体治疗covid-19提供了应用前景,也为“鸡尾酒”疗法治疗covid-19提供了应用可能。本发明的目的就是提供一种具有独特结合表位的抗sars-cov-2单克隆抗体,所述表位与现有技术已有的抗sars-cov-2单克隆抗体明显不同,以期能够与现有技术抗体形成双抗体组合物,所述双抗体组合物能够充分发挥抗体“鸡尾酒”疗法的协同功效,提高阻断新型冠状病毒入侵感染的中和活性,进而提供所述双抗体组合物在制备covid-19治疗药物中的应用。技术实现要素:基于上述发明目的,本发明首先提供了一种双抗体组合物,所述双抗体组合物含有特异性结合于sars-cov-2的s蛋白受体结合区的单克隆抗体和特异性结合于sars-cov-2的s蛋白非受体结合区的抗体,所述特异性结合于sars-cov-2的s蛋白受体结合区的单克隆抗体的重链可变区的cdr1、cdr2和cdr3区的氨基酸序列分别如seqidno:1第26-33、51-58、97-111位氨基酸序列所示;轻链可变区的cdr1、cdr2和cdr3区的氨基酸序列分别如seqidno:5第27-35、52-54、91-100位氨基酸序列所示。在一个优选的实施方案中,所述特异性结合于sars-cov-2的s蛋白受体结合区的单克隆抗体重链可变区的氨基酸序列如seqidno:1所示,轻链可变区的氨基酸序列如seqidno:5所示,在本申请中具有该重链可变区和轻链可变区的抗体被命名为“4b7”。在一个更为优选的实施方案中,所述特异性结合于sars-cov-2的s蛋白非受体结合区的抗体特异性结合于sars-cov-2的s蛋白的n端结构域。更为优选地,所述特异性结合于sars-cov-2的s蛋白的n端结构域的抗体的重链可变区的cdr1、cdr2和cdr3区的氨基酸序列分别如seqidno:11第26-33、51-58、97-117位氨基酸序列所示;轻链可变区的cdr1、cdr2和cdr3区的氨基酸序列分别如seqidno:12第27-37、55-57、94-102位氨基酸序列所示。再为优选地,所述特异性结合于sars-cov-2的s蛋白的n端结构域的抗体的重链可变区的氨基酸序列如seqidno:11所示,轻链可变区的氨基酸序列如seqidno:12所示,在本申请中具有该重链可变区和轻链可变区的抗体被命名为“4a8”。尤为优选地,所述特异性结合于sars-cov-2的s蛋白受体结合区的单克隆抗体和所述特异性结合于sars-cov-2的s蛋白非受体结合区的抗体的重链恒定区的氨基酸序列均如seqidno:3所示,轻链恒定区的氨基酸序列均如seqidno:7或seqidno:9所示。在一个优选的实施方案中,所述特异性结合于sars-cov-2的s蛋白受体结合区的单克隆抗体和所述特异性结合于sars-cov-2的s蛋白非受体结合区的抗体的配伍比例为1-10:10-1。在一个更为优选的实施方案中,所述特异性结合于sars-cov-2的s蛋白受体结合区的单克隆抗体和所述特异性结合于sars-cov-2的s蛋白非受体结合区的抗体的配伍比例为1-2:2-1。在一个尤为优选的实施方案中,所述特异性结合于sars-cov-2的s蛋白受体结合区的单克隆抗体和所述特异性结合于sars-cov-2的s蛋白非受体结合区的抗体的配伍比例为2:3或4:3。其次,本发明提供了上述的抗体组合物的制备方法,所述方法包括将特异性结合于sars-cov-2的s蛋白受体结合区的单克隆抗体和所述特异性结合于sars-cov-2的s蛋白非受体结合区的抗体混合的步骤。在一个优选的实施方案中,所述特异性结合于sars-cov-2的s蛋白受体结合区的单克隆抗体和所述特异性结合于sars-cov-2的s蛋白非受体结合区的抗体按照1-2:2-1的配伍比例混合。在一个更为优选的实施方案中,所述特异性结合于sars-cov-2的s蛋白受体结合区的单克隆抗体和所述特异性结合于sars-cov-2的s蛋白非受体结合区的抗体按照2:3或4:3的配伍比例混合。最后,本发明提供了上述的抗体组合物在制备covid-19治疗和/或预防药物中的应用。本发明提供了一种双抗体组合物,所述双抗体组合物含有特异性结合于sars-cov-2的s蛋白受体结合区的单克隆抗体和特异性结合于sars-cov-2的s蛋白非受体结合区的抗体,所述双抗体组合物的一个具体的实施方案就是靶向新型冠状病毒s蛋白n端结构域的单抗4a8和靶向新型冠状病毒s蛋白受体结合区的单抗4b7形成的组合物,两种抗体单独使用均具有高效中和活性,分别为0.57和0.96nm;该组合物相比较原先的单抗,其在阻断新型冠状病毒入侵感染中中和活性显著提高,分别提高到4a8的3.8倍和4b7的6.4倍,具有显著的协同效应。所述两种抗体协同产生的高效的中和活性显示含有该两种抗体的组合物可以应用于“鸡尾酒”方法治疗和/或预防covid-19;而且,本发明提供的抗体组合物能够通过稳定的工程菌株以及工业化制药方法进行规模化制备,具有巨大的产业化前景。附图说明图1.亲和层析纯化后的单克隆抗体的sds-page检测图谱;图2.elisa检测纯化单抗4b7与s蛋白、s1蛋白、s2蛋白、rbd蛋白的结合活性随浓度变化的曲线图;图3.biacoret200测定单抗4b7与s蛋白、s1蛋白、rbd蛋白的亲和力结果输出图;图4.抗体4b7存在时抗体4a8的结合抑制曲线图;图5.抗体4a8存在时抗体4b7的结合抑制曲线图;图6.单抗4b7在假病毒细胞模型上的ec50测定曲线图;图7.单抗4a8和单抗4b7的协同效应曲线。具体实施方式下面结合具体实施例来进一步描述本发明,本发明的优点和特点将会随着描述而更为清楚。但这些实施例仅是范例性的,并不对本发明的权利要求所限定的保护范围构成任何限制。实施例1.人源抗sars-cov-2单克隆抗体的筛选和制备人源抗sars-cov-2单克隆抗体的筛选和制备参照中国发明专利申请cn111303280a公开的方法,本申请通过引用将cn111303280a公开的内容作为本申请说明书的一部分。在本申请之前的研究中,cn111303280a筛选获得了一株抗新型冠状病毒刺突蛋白s的单克隆抗体4a8。通过识别表位分析发现,单抗4a8识别的表位位于s1非rbd区,该抗体对于新冠病毒s蛋白具有极高的亲和力,平衡解离常数(kd)为1.00nm,在对veroe6细胞模型感染sars-cov-2的保护效果实验中,显示其ec50达到0.58ug/ml。本申请通过上述筛选方法,筛选到了一组抗sars-cov-2单克隆抗体。通过对筛选抗体的sds-page检测分析,结果如图1所示,其中图1的a为非还原sds-page检测结果,图1的b为还原sds-page检测结果,泳道1-3为单抗3h3,泳道4为4b7,泳道5为4c12,泳道6为4e1电泳结果。在还原电泳中,预期重链和轻链的分子量分别为50kda和25kda,泳道m为分子量标记。在非还原电泳中,预期全分子单抗的分子量为150kda,符合预期。对单抗4b7序列的分析结果如下:重链可变区的氨基酸序列如seqidno:1所示,重链可变区的cdr1、cdr2和cdr3区的氨基酸序列分别如seqidno:1第26-33、51-58、97-111位氨基酸序列所示,编码重链可变区的多核苷酸序列由seqidno:2所示,重链恒定区的氨基酸序列如seqidno:3所示,编码重链恒定区的多核苷酸序列由seqidno:4所示;轻链可变区的氨基酸序列如seqidno:5所示,轻链可变区的cdr1、cdr2和cdr3区的氨基酸序列分别如seqidno:5第27-35、52-54、91-100位氨基酸序列所示,编码轻链可变区的多核苷酸序列由seqidno:6所示,轻链恒定区的氨基酸序列如seqidno:7或9所示,编码轻链恒定区的多核苷酸序列由seqidno:8或10所示。实施例2人源抗sars-cov-2单克隆抗体4b7与s、s1、s2和rbd的结合活性分析2.1包被:取重组的s抗原、s1抗原、rbd抗原和s2抗原用包被液稀释至浓度2μg/ml,包被酶标板,每孔100μl,4℃包被过夜。2.2封闭:每孔加入300μlpbst洗液,洗涤3次×3min/次;拍净孔内液体,加入2%bsa,200μl/孔,37℃封闭1h。2.3样品孵育:每孔加入300μlpbst洗液,洗涤3次×3min/次;拍净孔内液体,加入pbs稀释后的纯化单抗,首孔9ug/ml,3倍系列稀释100μl/孔,37℃孵育1h。2.4二抗孵育:洗涤,同上;加入hrp羊抗人fc二抗(1:20000稀释),100μl/孔,37℃孵育1h。2.5显色:洗涤,同上;每孔加入100μltmb单组分显色液,37℃显色5min后每孔加50μl终止液终止反应,使用酶标仪检测450nm处的吸光值。使用graphpad非线性回归,四参数拟合绘制标准曲线,并根据标准曲线及稀释倍数计算单抗的ec50浓度。2.6结果:见图2和表1。图2中,圆标曲线表示与s蛋白的检测结果,显示特异结合呈现剂量反应关系;方标曲线表示与s1抗原的检测结果,显示特异结合呈现剂量反应关系;正三角标曲线表示与s2抗原的检测结果,显示不结合;倒三角标曲线显示与rbd的检测结果,显示特异结合呈现剂量反应关系。分析结果证明,单抗4b7识别的表位位于s蛋白,s1区,更具体的是位于rbd区。表1中,单抗4b7与s、s1、rbd均具有高结合活性,说明单抗是rbd特异的。表1.单抗4b7与s、s1、s2和rbd的结合活性抗原ss1s2rbdec50(ug/ml)0.0021560.0018990.27580.001808cn111303280a公开的4a8和本申请鉴定的4b7在于新型冠状病毒刺突蛋白s的结合方法具有不同的结合区。单抗4a8与s、s1特异结合,不与rbd结合,单抗4b7与s、s1和rbd均特异结合,上面结果推测二者空间上可能不具有竞争抑制关系。实施例3.biacoret200测定单抗与s、s1、rbd抗原的亲和力测定的基本原理是基于一种表面等离子共振(spr)的物理光学现象的生物传感分析技术。不必使用荧光标记和同位素标记,从而保持了生物分子的天然活性。当入射光以临界角入射到两种不同透明介质的界面时将产生全反射,且反射光强度在各个角度上都应相同,但若在介质表面镀上一层金属薄膜后,由于入射光可以引起金属中自由电子的共振,从而导致反射光在一定的角度内大大减弱,其中使反射光完全消失的角度称为共振角。共振角会随金属薄膜表面通过的液相的折射率的改变而改变,折射率的变化(ru)又和结合在金属表面的大分子质量成正比。因此,bia技术可以通过对反应全过程中各种分子反射光的吸收获得初始的数据,并经相关处理获得结果-传感图。实验步骤:(1)换液:抗体0429-4b7换液到hepes缓冲液中,nano测定浓度为1.67mg/ml。抗原缓冲液中由于含有tris,影响包被,需要换液到pbs中,浓度介于0.8-1mg/ml。(2)预富集,确定包被缓冲液ph:s蛋白采用ph4.0的甘氨酸缓冲液,s1和rbd蛋白采用ph5.0的缓冲液。(3)配体固定:使用cm5芯片、氨基偶联试剂盒进行操作,分别将s、s1、rbd蛋白包被在芯片的2、3、4通道,1通道作为参比通道。包被结果:s(437ru)s1(107ru)rbd(41ru)。(4)抗体稀释:最高浓度为100nm,对倍稀释,10个稀释度。最终结果采用25nm、12.5nm、6.25nm、3.125nm、1.5625nm稀释度的曲线进行拟合。(5)结合时间100s,解离时间900s。拟合方法采用1:1model拟合。分析及结果:通过软件拟合计算得到4b7与s蛋白、s1和rbd的亲和常数,见图3和表2。说明单抗与s、s1和rbd均具有高的亲和力。表2.单抗4b7与s、s1和rbd抗原的亲和力检测结果kd(m)ka(1/ms)kd(1/s)4b7和ncovs4.533e-109.355e+54.240e-44b7和ncovs12.170e-101.609e+63.492e-44b7和ncovrbd3.866e-101.101e+64.256e-4实施例4.bli方法4a8和4b7非竞争结合s蛋白进一步采用薄膜干涉技术测定抗体4a8和4b7是否存在竞争结合,分别检测4a8存在时是否影响4b7与s蛋白的结合或者4b7存在时是否影响4a8与s蛋白的结合。使用薄膜干涉技术测定抗体4a8和4b7是否竞争结合新冠病毒s蛋白。将生物素标记的s蛋白以10μg/ml包被在生物传染器上,包被至约1nm,而后在缓冲液中校正120s。将3根生物传感器分别浸入35μg/ml的竞争抗体或缓冲液中300s,随后将3根传感器浸入分析抗体或缓冲液中,浸入缓冲液的传感器将用于扣除系统偏移误差。竞争百分率=不存在竞争抗体时分析抗体的结合最大值/存在竞争抗体时结合抗体的结合最大值*100%。抑制率>70%表明两种抗体不存在竞争,抑制率<70%且>30%表明两种抗体存在微弱竞争,抑制率<30%表明两种抗体存在竞争。实验结果如图4、图5和表3所示,图4中,2根生物传感器首先分别与两种竞争抗体4a8或4b7结合,随后2根生物传感器均浸入分析抗体4a8中,在竞争抗体4a8存在的情况下,分析抗体4a8与s蛋白的结合受到完全抑制,竞争率为3.2%。在竞争抗体4b7存在的情况下,4a8的结合抑制率为74.7%,说明4a8的结合未受4b7的抑制。图5中,2根生物传感器首先分别与两种竞争抗体4a8或4b7结合,随后2根生物传感器均浸入分析抗体4b7中,结果显示,在竞争抗体4b7存在的情况下,分析抗体4b7结合受到完全抑制,竞争率为0.2%。在竞争抗体4a8存在的情况下,结合抑制率为88.0%,说明4b7与s蛋白的结合同样未受到4a8的抑制。综上,单抗4a8与4b7不存在与s蛋白的竞争结合。表3.4a8或4b7对对方抗体与s蛋白的结合抑制率实施例5sars-cov-2假病毒细胞模型上中和活性分析5.1假病毒包装:将编码全长sars-cov-2s蛋白的基因(genbankid:qhd43416.1)插入pdc316载体,得到质粒pdc316-sars-cov-2-s。将ace2基因稳定转染入hek293细胞,构建稳定表达人ace2的细胞ace2-293t细胞。将7.0×106ace2-293t细胞接种在10cm细胞培养皿中,并在37℃,5%co2下生长过夜。用lipofectamine3000转染试剂(invitrogen)将pdc316-sars-cov-2-s和hiv骨架载体pnl4-3.luc.r-e-共转染到ace2-293t细胞中。6小时后更换培养基。转染后48小时收集含有hiv假型病毒和s蛋白的上清液,并通过0.45μm过滤器过滤。然后将上清液等分并保存在-80℃。5.2稀释抗体:4b7(20200525批,原始浓度8.35mg/ml,稀释到1.67mg/ml)将抗体用dmem培养基稀释到需要的浓度,首孔浓度为6μg/ml(100μl体系),首孔100μl,吸取50μl加入到下一孔,对倍稀释。每个梯度设置两个复孔。5.3稀释假病毒:将假病毒与dmem培养基按照3:7的比例混合,混匀后每孔加入50μl,轻轻混匀。37℃孵育1h。5.4将ace2-293t细胞稀释到2×105cells/ml,每孔加入100μl,37℃、5%co2培养48h。5.5检测luciferase读值,使用briteliteplusreportergeneassaysystem(perkinelmer,6066769)。每孔吸弃100μl培养基,加入100μlbriteliteplus显色底物,避光孵育2min,用枪头吹吸3次后吸取150μl转移到白色96孔板,使用tecanspark10m进行读数。5.6统计分析:没有病毒和抗体的细胞用作空白对照,没有抗体的细胞用作病毒对照。中和百分比计算为(样品信号-空白对照信号)/(病毒对照信号-空白对照信号)×100%。使用graphpadprism7进行统计分析。结果:见图6(图6横坐标表示对数浓度,纵坐标表示相对阴性对照组的保护率%)。本发明公开的单抗4b7在假病毒模型上ec50是0.052ug/ml(0.35nm)。实施例6sars-cov-2感染时细胞模型上各抗体中和活性分析sars-cov-2病毒进入宿主细胞是病毒感染的第一步,抑制病毒的进入就可有效阻断病毒感染;sars-cov-2病毒包膜刺突蛋白(spikeprotein,s)是介导病毒吸附并进入靶细胞的关键蛋白。s蛋白胞外区包括s1和s2,s1介导病毒与细胞表面受体的结合,而s2促进膜融合过程。s1蛋白又包括n端结构域(nterminalregion,ntd)和受体结合区(receptorbindgingdomain,rbd)。因为rbd在病毒入侵中发挥重要作用,是中和单抗的重要靶标。除rbd区外,ntd和s2区也有中和单抗报道。研发针对rbd不同识别表位或者针对不同区的单抗组合,可能避免病毒逃逸突变。本研究中的抗体组合物包括针对ntd的单抗4a8(4a8识别抗原表位的进一步晶体解析研究见sciencechietal.,science10.1126/science.abc6952(2020)。本申请通过引证,将该文献作为本申请说明书公开的一部分)和针对rbd的单抗4b7。假病毒模型可以很好的评价4b7,而不适用4a8的活性评价。又因为相比较假病毒活性评价方法,真病毒评价模型更可靠,因此本研究直接选择使用真病毒评价模型。评价方法描述如下:6.1将veroe6细胞用0.25%的胰酶消化后,用培养基(dmem+10%fbs)稀释至3×105cells/ml浓度,接种到96孔细胞培养板中,接种体积100μl/孔,置37℃5%co2细胞培养箱培养过夜。6.2实验当天,将纯化单抗用培养基dmem+2%fbs自初始浓度(4b7单抗初始浓度92.78ug/ml,3倍系列稀释,加入96孔培养板,体积120μl/孔;随即每孔加入120μlcovid-19病毒悬液(用dmem+2%fbs稀释病毒,加入100tcld50/孔),充分混匀,置细胞培养箱孵育1h。6.3弃去96孔板中细胞培养上清,每孔加入200μl共孵育后的病毒-抗体混合悬液;另设置存活对照(不加病毒和抗体)和死亡对照(只加病毒),置37℃5%co2细胞培养箱继续培养72h。6.472h后弃去细胞培养上清,加入50μl结晶紫染色液室温染色30min,弃去染液,加入200μl/孔纯水,重复洗涤6次。6.5弃尽洗液,用吸水纸拍干板孔中水份,加入100μl脱色液充分溶解,以od620为参考,用酶标仪测od570值;使用od570为纵坐标(样品od570-只加病毒对照od570),抗体浓度(ug/ml)为横坐标,用graphpadprism5拟合曲线,计算抗体ec50值。6.6结果及讨论:单独单抗的中和活性见表4,4a8和4b7具有高中和活性,ace2-fc和3h3具有中等中和活性,l12、4c12和4e1具有弱中和活性。表4.单抗单独作用时的细胞水平中和活性比较4a84b7l124c12ace2-fc3h34e1识别表位ntdrbdntdrbdrbds2s2ec50(ug/ml)0.570.9636.5838.524.537.4830.97单独和联合应用,优选高中和活性单抗,进一步考察高中和活性单抗4a8和4b7与其他单抗的协同作用(见表5),1:1摩尔比的单抗组合进行实验,实验结果显示4a8和4b7具有明显的协同效应,图7为4a8和4b7的协同效应曲线。实验结果显示,4a8和4b7组成使用的ec50明显小于4a8和4b7单独使用,4a8与其他单抗的协同作用不明显;包括与针对s蛋白其他表位的单抗联合应用,如4a8(识别s蛋白的ntd)与4c12(识别rbd),4a8与3h3(识别s蛋白的s2)均不产生协同效用;同样,4b7与4a8具有明显的协同,与其他单抗的协同不明显。表5.1:1摩尔比的单抗组合细胞水平的中和活性(ec50:ug/ml)比较(一)4a84b7l124c12ace2-fc3h34e14a80.570.150.560.680.572.21.14b70.150.961.021.091.790.781.4为了筛选到能够产生协同效应的针对不同表位的单抗组合,又进行下述实验。l12单抗识别s蛋白的ntd,4c12识别s蛋白rbd,考察它们与不同单抗的协同效应,结果见表6。l12和4c12联合使用不具有协同效应。与其他单抗也不具有明显的协同效应。实验证明并不是针对不同表位的单抗联合使用就能产生协同效应。表6.1:1摩尔比的单抗组合细胞水平的中和活性(ec50:ug/ml)比较(二)4a84b7l124c12ace2-fc3h34e1l120.561.0236.5838.5210.0423.64-4c120.681.0938.5220.4513.87--为了进一步优化摩尔比,采用不同摩尔比的4a8和4b7的混合物进行中和活性检测,结果(见表7)显示不同摩尔比组别之间具有一定的差异,在4a8和4b7摩尔比为2:3和3:4时,中和活性(ec50:ug/ml)达到最优的0.08,但是在4a8和4b7摩尔比为1-2:2-1的范围内,中和活性的差异并不显著,均显著高于4a8或4b7的单独应用,以及4a8或4b7与除及4a8和4b7之外的抗体的组合应用。考虑到抗体药物在实际应用中的有效浓度可以保持在一个较广泛的范围,本领域技术人员能够合理预见,4a8和4b7的摩尔比在一个更大的范围例如1-4:4-1,甚至更大的范围例如1-10:10-1仍然可以发挥充分的协同效应。又由于考虑到应用的简单、方便性,实际应用中推荐4a8和4b7摩尔比为1:1的组合。表7.不同摩尔比的4a8和4b7的细胞水平的中和活性比较1:12:33:23:44:3ec500.130.080.220.130.08序列表<110>中国人民解放军军事科学院军事医学研究院<120>一种双抗体组合物及在制备covid-19治疗药物中的应用<160>12<170>siposequencelisting1.0<210>1<211>122<212>prt<213>homosapiens<400>1gluvalglnleuvalgluserglyglyglyleuvalglnproglygly151015serleuargleusercysalaalaserglypheprophesersertyr202530alametsertrpvalargglnalaproglylysglyleuglutrpval354045seralailethrglyserglyglyserthrtyrtyralaaspserval505560lysglyargphethrileserargaspasnserlysasnthrleutyr65707580leuhismetasnserleuargalagluaspthralavaltyrtyrcys859095alalysaspasptyrglyasptyrvalleuglyalapheaspiletrp100105110glyglnglythrthrvalthrvalserser115120<210>2<211>366<212>dna<213>homosapiens<400>2gaggtgcagctggtggagtctgggggaggcttggtacagcctggggggtccctgagactc60tcctgtgcagcctctggattcccctttagcagctatgccatgagctgggtccgccaggct120ccagggaaggggctggagtgggtctcagctattactggtagtggtggtagcacatactac180gcagactccgtgaagggccggttcaccatctccagagacaattccaagaacacgctctat240ctgcatatgaacagcctgagagccgaggacacggccgtatattactgtgcgaaggatgac300tacggtgactacgtcttgggtgcttttgatatctggggccaagggacaacggtcaccgtc360tcctca366<210>3<211>330<212>prt<213>homosapiens<400>3alaserthrlysglyproservalpheproleualaproserserlys151015serthrserglyglythralaalaleuglycysleuvallysasptyr202530pheprogluprovalthrvalsertrpasnserglyalaleuthrser354045glyvalhisthrpheproalavalleuglnserserglyleutyrser505560leuserservalvalthrvalproserserserleuglythrglnthr65707580tyrilecysasnvalasnhislysproserasnthrlysvalasplys859095lysvalgluprolyssercysasplysthrhisthrcysproprocys100105110proalaprogluleuleuglyglyproservalpheleuphepropro115120125lysprolysaspthrleumetileserargthrprogluvalthrcys130135140valvalvalaspvalserhisgluaspprogluvallyspheasntrp145150155160tyrvalaspglyvalgluvalhisasnalalysthrlysproargglu165170175gluglntyrasnserthrtyrargvalvalservalleuthrvalleu180185190hisglnasptrpleuasnglylysglutyrlyscyslysvalserasn195200205lysalaleuproalaproileglulysthrileserlysalalysgly210215220glnproarggluproglnvaltyrthrleuproproserargaspglu225230235240leuthrlysasnglnvalserleuthrcysleuvallysglyphetyr245250255proseraspilealavalglutrpgluserasnglyglnprogluasn260265270asntyrlysthrthrproprovalleuaspseraspglyserphephe275280285leutyrserlysleuthrvalasplysserargtrpglnglnglyasn290295300valphesercysservalmethisglualaleuhisasnhistyrthr305310315320glnlysserleuserleuserproglylys325330<210>4<211>993<212>dna<213>homosapiens<400>4gctagcaccaagggcccatcggtcttccccctggcaccctcctccaagagcacctctggg60ggcacagcggccctgggctgcctggtcaaggactacttccccgaaccggtgacggtgtcg120tggaactcaggcgccctgaccagcggcgtgcacaccttcccggctgtcctacagtcctca180ggactctactccctcagcagcgtggtgaccgtgccctccagcagcttgggcacccagacc240tacatctgcaacgtgaatcacaagcccagcaacaccaaggtggacaagaaagttgagccc300aaatcttgtgacaaaactcacacatgcccaccgtgcccagcacctgaactcctgggggga360ccgtcagtcttcctcttccccccaaaacccaaggacaccctcatgatctcccggacccct420gaggtcacatgcgtggtggtggacgtgagccacgaagaccctgaggtcaagttcaactgg480tacgtggacggcgtggaggtgcataatgccaagacaaagccgcgggaggagcagtacaac540agcacgtaccgtgtggtcagcgtcctcaccgtcctgcaccaggactggctgaatggcaag600gagtacaagtgcaaggtctccaacaaagccctcccagcccccatcgagaaaaccatctcc660aaagccaaagggcagccccgagaaccacaggtgtacaccctgcctccatctcgggatgag720ctgaccaagaaccaggtcagcctgacctgcctggtcaaaggcttctatcccagcgacatc780gccgtggagtgggagagcaatgggcagccggagaacaactacaagaccacgcctcccgtg840ctggactccgacggctccttcttcctctatagcaagctcaccgtggacaagagcaggtgg900cagcaggggaacgtcttctcatgctccgtgatgcatgaggctctgcacaaccactacacg960cagaagagcctctccctgtctccgggtaaatga993<210>5<211>110<212>prt<213>homosapiens<400>5glnseralaleuthrglnproalaservalserglyserproglygln151015serilethrilesercysthrglythrserseraspvalglythrtyr202530lysphevalsertrptyrglnglnhisproglylysalaprolysleu354045metiletyrgluglyserlysargproserglyvalserasnargphe505560serglyserlysserglyasnthralaserleuthrileserglyleu65707580glnalagluaspglualaasptyrtyrcyscyssertyralaglyser859095serthrleuvalpheglyglyglythrlysleuthrvalleu100105110<210>6<211>330<212>dna<213>homosapiens<400>6cagtctgccctgactcagcctgcctccgtgtctgggtctcctggacagtcgatcaccatc60tcctgcactggaaccagcagtgatgttgggacttataagtttgtctcctggtaccaacag120cacccaggcaaagcccccaaactcatgatttatgagggcagtaagcggccctcaggggtt180tctaatcgcttctctggctccaagtctggcaacacggcctccctgacaatctctgggctc240caggctgaggacgaggctgattattactgctgctcatatgcaggtagtagcactttggtg300ttcggcggagggaccaagctgaccgtccta330<210>7<211>107<212>prt<213>homosapiens<400>7argthrvalalaalaproservalpheilepheproproseraspglu151015glnleulysserglythralaservalvalcysleuleuasnasnphe202530tyrproargglualalysvalglntrplysvalaspasnalaleugln354045serglyasnserglngluservalthrgluglnaspserlysaspser505560thrtyrserleuserserthrleuthrleuserlysalaasptyrglu65707580lyshislysvaltyralacysgluvalthrhisglnglyleuserser859095provalthrlysserpheasnargglyglucys100105<210>8<211>324<212>dna<213>homosapiens<400>8cggaccgtggcggcgccatctgtcttcatcttcccgccatctgatgagcagttgaaatct60ggtaccgctagcgttgtgtgcctgctgaataacttctatcccagagaggccaaagtacag120tggaaggtggataacgccctccaatcgggtaactcccaggagagtgtcacagagcaggac180agcaaggacagcacctacagcctcagcagcaccctgacgctgagcaaagcagactacgag240aaacacaaagtctacgcctgcgaagtcacccatcagggcctgagctcgcccgtcacaaag300agcttcaacaggggagagtgttag324<210>9<211>106<212>prt<213>homosapiens<400>9glyglnprolysalaalaproservalthrleupheproproserser151015glugluleuglnalaasnlysalathrleuvalcysleuileserasp202530phetyrproglyalavalthrvalalatrplysalaaspserserpro354045vallysalaglyvalgluthrthrthrproserlysglnserasnasn505560lystyralaalasersertyrleuserleuthrprogluglntrplys65707580serhislyssertyrsercysglnvalthrhisgluglyserthrval859095glulysthrvalalaprothrglucysser100105<210>10<211>321<212>dna<213>homosapiens<400>10ggtcagcccaaggctgccccctcggtcactctgttcccaccctcgagtgaggagcttcaa60gccaacaaggccacactggtgtgtctcataagtgacttctacccgggagccgtgacagtg120gcctggaaggcagatagcagccccgtcaaggcgggagtggagaccaccacaccctccaaa180caaagcaacaacaagtacgcggccagcagctacctgagcctgacgcctgagcagtggaag240tcccacaaaagctacagctgccaggtcacgcatgaagggagcaccgtggagaagacagtg300gcccctacagaatgttcataa321<210>11<211>128<212>prt<213>homosapiens<400>11gluvalglnleuvalgluserglyalagluvallyslysproglyala151015servallysvalsercyslysvalserglytyrthrleuthrgluleu202530sermethistrpvalargglnalaproglylysglyleuglutrpmet354045glyglypheaspprogluaspglygluthrmettyralaglnlysphe505560glnglyargvalthrmetthrgluaspthrserthraspthralatyr65707580metgluleuserserleuargsergluaspthralavaltyrtyrcys859095alathrserthralavalalaglythrproaspleupheasptyrtyr100105110tyrglymetaspvaltrpglyglnglythrthrvalthrvalserser115120125<210>12<211>112<212>prt<213>homosapiens<400>12gluilevalmetthrglnserproleuserserprovalthrleugly151015glnproalaserilesercysargserserglnserleuvalhisser202530aspglyasnthrtyrleusertrpleuglnglnargproglyglnpro354045proargleuleuiletyrlysileserasnargpheserglyvalpro505560aspargpheserglyserglyalaglythraspphethrleulysile65707580serargvalglualagluaspvalglyvaltyrtyrcysthrglnala859095thrglnpheprotyrthrpheglyglnglythrlysvalaspilelys100105110当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1