基于CRISPR/Cas9技术的单子叶植物基因敲除载体及其应用的制作方法
本发明属于生物技术领域,具体涉及基于CRISPR/Cas9技术的单子叶植物基因敲除载体及其应用。
背景技术:
II型CRISPR/Cas9系统是来自链球菌属的一种获得性免疫系统,能够抵御外源基因的入侵。经过人工修饰,这种系统在动植物中得到了广泛应用。它主要由CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)和特异的Cas9蛋白组成。CRISPR是一成簇的规律间隔的短回文DNA重复序列,它由一系列短的高度保守的正向重复序列和长度相似的序列间隔排列组成。Cas9蛋白是一种由1409个氨基酸组成的多结构域蛋白,含有两个核酸酶结构域RuvC-like和HNH。CRISPR/Cas9系统于2013年被Seung等人(Seung Woo Cho et al.,2013)首次成功运用于真核细胞的基因组编辑。基于CRISPR/Cas9的基因组编辑体系包括2部分:sgRNA及酶Cas9。这种系统能够特异的识别毗邻PAM(NGG)基序的靶序列,并在其上游3nt处进行切割,同时RuvC-like结构域在PAM区上游3~8nt处进行切割,形成DSB(double strains break双链断裂),随后机体通过自身的修复机制如同源重组、非同源重组来修复损伤的DNA,进而产生Indel(包括缺失和插入)的现象。
目前应用于植物上的CRISPR/Cas9载体构建体系并不多,有基于基因枪转化的载体构建体系和基于农杆菌转化的载体构建体系。在目前普遍使用的植物农杆菌转化方面,基于CRISPR/Cas9技术的植物基因敲除载体体系构建过程比较繁琐,需要多步才能将各个部分整合到双元载体上。已报道的CRISPR/Cas9体系,有的体系是借助限制性内切酶BbsI先把sgRNA插入pATU6-26SK/pOsU6SK载体,再通过限制性内切酶KpnI和SalI酶切得到AtU6-sgRNA片段或通过KpnI和HindIII双酶切获得OsU6-sgRNA片段,同时将酶切过的SalI-Cas9-EcoRI或HindIII-Cas9-EcoRI片段与已用KpnI和EcoRI酶切的线性化的质粒pCAMBIA1300连接构建双元载体,这种系统在构建过程中比较复杂而且周期长(ZY Feng,JK Zhu et al.,2013);另外有利用合成的方法直接合成启动子和sgRNA单元,再构建到双元载体上,这种体系成本高,周期长(Wenzhi Jiang et al.,2013)。而在基因枪转化方面中,已报道的体系都是将pU6-gRNA或pU3-gRNA载体与Cas9载体共同包裹在金粉中打入植物组织中,这种费用比较高。以上这些体系,要完成一个植物基因CRISPR/Cas9敲除载体的构建,要么成本高昂,要么过程繁琐,且不能满足高通量植物基因CRISPR/Cas9敲除载体构建的需求。
还有一种体系是将启动子、sgRNA scaffold和Cas9片段整合到一个双元载体,并选用Bsa I作为插入位点,但是这种体系的缺陷在于,在两个Bsa I之间存在SpR(spectinomycin resistance gene壮观霉素抗性基因)片段,在构建过程中需要Bsa I酶切回收线性化载体,再连接target sites(靶位)(HL Xing,L Dong et al.,2014)。
而本发明是将水稻OsU3启动子及sgRNA元件与35S启动子启动的Cas9元件直接整合在双元载体pCAMBIA1300上,并在sgRAN scaffold元件中选用Aar I作为靶序列的插入位点,且两个Aar I只间隔7bp碱基,只需要通过酶切纯化获取线性化载体,再一步连接的方法插入target oligos,从而实现简便、快速和高效的构建CRISPR/Cas9基因敲除载体。
技术实现要素:
本发明的一个目的在于提供一种基于CRISPR/Cas9技术的单子叶植物基因敲除载体pCAMBIA1300-OsU3-2x35s-Cas9,该载体具有以下特征:
(1)骨架载体为双元载体pCAMBIA1300;
(2)在所述双元载体上,同时连入了sgRNA元件和Cas9元件;
(3)所述sgRNA元件由sgRNA启动子、sgRNA-scaffold及靶点插入酶切位点组成;
(4)所述sgRNA启动子为OsU3启动子;
(5)所述靶点插入酶切位点为Aar I,酶切位点信息为:
Aar I:CTTGGCTCGCAGGTGAACACAACACCTGCACAC(SEQ ID NO:1)
(6)所述Cas9元件为2x35S-Cas9-ter,2x35S为串联的2个35S启动子,ter为终止子(terminator),Cas9为编码基因Cas9的全长CDS序列并且两端加有核定位的信号NLS(Nuclear localization signal),N端加有Flag标签。
本发明是将sgRNA结构单元与Cas9基因共同整合到骨架载体pCAMBIA1300上的敲除载体pCAMBIA1300-OsU3-2x35s-Cas9,同时sgRNA结构单元中有Aar I的插入位点,其序列如下:
Aar I:CTTGGCTCGCAGGTGAACACAACACCTGCACAC(SEQ ID NO:1)
本发明使用的启动子是OsU3启动子,在植物中启动sgRNA scaffold结构的启动子属于Pol III promoters,包括U3和U6的启动子,而OsU3是单子叶植物中启动效率较高的启动子;同时选用的限制性酶的插入位点也是一类特征的酶—Type IIs限制性内切酶,可选用的酶有Aar I、BspQ I、Bbs I/Bpi l、BsmB I/Esp3 I、BfuA I/Be I和Bsa I/Eco31 I,而在本发明中选用Aar I的插入位点(其他位点在pCAMBIA1300载体上都存在)。
本发明使用pCAMBIA1300作为骨架载体,将2x35s-Cas9-ter(2x35s:2个串联在一起的35S启动子;cas9:cas9酶的CDS序列;ter:终止子序列)及sgRNA元件(单子叶植物使用水稻的OsU3启动子启动)连入骨架载体;同时将Aar I的酶切位点连入sgRNA元件的靶位点插入处(sgRNA启动子与sgRNA scaffold之间),构建出适用于单子叶植物基因敲除载体pCAMBIA1300-OsU3(Aar I)-2x35s-Cas9。
2x35s-Cas9-ter的序列(SEQ ID NO:2):
CGGTATCGATAAGCTTGCATGCCTGCAGGTCAACATGGTGGAGCACGACACACTTGTCTACTCCAAAAATATCAAAGATACAGTCTCAGAAGACCAAAGGGCAATTGAGACTTTTCAACAAAGGGTAATATCCGGAAACCTCCTCGGATTCCATTGCCCAGCTATCTGTCACTTTATTGTGAAGATAGTGGAAAAGGAAGGTGGCTCCTACAAATGCCATCATTGCGATAAAGGAAAGGCCATCGTTGAAGATGCCTCTGCCGACAGTGGTCCCAAAGATGGACCCCCACCCACGAGGAGCATCGTGGAAAAAGAAGACGTTCCAACCACGTCTTCAAAGCAAGTGGATTGATGTGATAACATGGTGGAGCACGACACACTTGTCTACTCCAAAAATATCAAAGATACAGTCTCAGAAGACCAAAGGGCAATTGAGACTTTTCAACAAAGGGTGATATCCGGAAACCTCCTCGGATTCCATTGCCCAGCTATCTGTCACTTTATTGTGAAGATAGTGGAAAAGGAAGGTGGCTCCTACAAATGCCATCATTGCGATAAAGGAAAGGCCATCGTTGAAGATGCCTCTGCCGACAGTGGTCCCAAAGATGGACCCCCACCCACGAGGAGCATCGTGGAAAAAGAAGACGTTCCAACCACGTCTTCAAAGCAAGTGGATTGATGTGATATCTCCACTGACGTAAGGGATGACGCACAATCCCACTATCCTTCGCAAGACCCTTCCTCTATATAAGGAAGTTCATTTCATTTGGAGAGGACCTCGACCTCAACACAACATATACAAAACAAACGAATCTCAAGCAATCAAGCATTCTACTTCTATTGCAGCAATTTAAATCATTTCTTTTAAAGCAAAAGCAATTTTCTGAAAATTTTCACCATTTACGAACGATACTCGAGATGGACTATAAGGACCACGACGGAGACTACAAGGATCATGATATTGATTACAAAGACGATGACGATAAGATGGCCCCAAAGAAGAAGCGGAAGGTCGGTATCCACGGAGTCCCAGCAGCCGACAAGAAGTACAGCATCGGCCTGGACATCGGCACCAACTCTGTGGGCTGGGCCGTGATCACCGACGAGTACAAGGTGCCCAGCAAGAAATTCAAGGTGCTGGGCAACACCGACCGGCACAGCATCAAGAAGAACCTGATCGGAGCCCTGCTGTTCGACAGCGGCGAAACAGCCGAGGCCACCCGGCTGAAGAGAACCGCCAGAAGAAGATACACCAGACGGAAGAACCGGATCTGCTATCTGCAAGAGATCTTCAGCAACGAGATGGCCAAGGTGGACGACAGCTTCTTCCACAGACTGGAAGAGTCCTTCCTGGTGGAAGAGGATAAGAAGCACGAGCGGCACCCCATCTTCGGCAACATCGTGGACGAGGTGGCCTACCACGAGAAGTACCCCACCATCTACCACCTGAGAAAGAAACTGGTGGACAGCACCGACAAGGCCGACCTGCGGCTGATCTATCTGGCCCTGGCCCACATGATCAAGTTCCGGGGCCACTTCCTGATCGAGGGCGACCTGAACCCCGACAACAGCGACGTGGACAAGCTGTTCATCCAGCTGGTGCAGACCTACAACCAGCTGTTCGAGGAAAACCCCATCAACGCCAGCGGCGTGGACGCCAAGGCCATCCTGTCTGCCAGACTGAGCAAGAGCAGACGGCTGGAAAATCTGATCGCCCAGCTGCCCGGCGAGAAGAAGAATGGCCTGTTCGGAAACCTGATTGCCCTGAGCCTGGGCCTGACCCCCAACTTCAAGAGCAACTTCGACCTGGCCGAGGATGCCAAACTGCAGCTGAGCAAGGACACCTACGACGACGACCTGGACAACCTGCTGGCCCAGATCGGCGACCAGTACGCCGACCTGTTTCTGGCCGCCAAGAACCTGTCCGACGCCATCCTGCTGAGCGACATCCTGAGAGTGAACACCGAGATCACCAAGGCCCCCCTGAGCGCCTCTATGATCAAGAGATACGACGAGCACCACCAGGACCTGACCCTGCTGAAAGCTCTCGTGCGGCAGCAGCTGCCTGAGAAGTACAAAGAGATTTTCTTCGACCAGAGCAAGAACGGCTACGCCGGCTACATTGACGGCGGAGCCAGCCAGGAAGAGTTCTACAAGTTCATCAAGCCCATCCTGGAAAAGATGGACGGCACCGAGGAACTGCTCGTGAAGCTGAACAGAGAGGACCTGCTGCGGAAGCAGCGGACCTTCGACAACGGCAGCATCCCCCACCAGATCCACCTGGGAGAGCTGCACGCCATTCTGCGGCGGCAGGAAGATTTTTACCCATTCCTGAAGGACAACCGGGAAAAGATCGAGAAGATCCTGACCTTCCGCATCCCCTACTACGTGGGCCCTCTGGCCAGGGGAAACAGCAGATTCGCCTGGATGACCAGAAAGAGCGAGGAAACCATCACCCCCTGGAACTTCGAGGAAGTGGTGGACAAGGGCGCTTCCGCCCAGAGCTTCATCGAGCGGATGACCAACTTCGATAAGAACCTGCCCAACGAGAAGGTGCTGCCCAAGCACAGCCTGCTGTACGAGTACTTCACCGTGTATAACGAGCTGACCAAAGTGAAATACGTGACCGAGGGAATGAGAAAGCCCGCCTTCCTGAGCGGCGAGCAGAAAAAGGCCATCGTGGACCTGCTGTTCAAGACCAACCGGAAAGTGACCGTGAAGCAGCTGAAAGAGGACTACTTCAAGAAAATCGAGTGCTTCGACTCCGTGGAAATCTCCGGCGTGGAAGATCGGTTCAACGCCTCCCTGGGCACATACCACGATCTGCTGAAAATTATCAAGGACAAGGACTTCCTGGACAATGAGGAAAACGAGGACATTCTGGAAGATATCGTGCTGACCCTGACACTGTTTGAGGACAGAGAGATGATCGAGGAACGGCTGAAAACCTATGCCCACCTGTTCGACGACAAAGTGATGAAGCAGCTGAAGCGGCGGAGATACACCGGCTGGGGCAGGCTGAGCCGGAAGCTGATCAACGGCATCCGGGACAAGCAGTCCGGCAAGACAATCCTGGATTTCCTGAAGTCCGACGGCTTCGCCAACAGAAACTTCATGCAGCTGATCCACGACGACAGCCTGACCTTTAAAGAGGACATCCAGAAAGCCCAGGTGTCCGGCCAGGGCGATAGCCTGCACGAGCACATTGCCAATCTGGCCGGCAGCCCCGCCATTAAGAAGGGCATCCTGCAGACAGTGAAGGTGGTGGACGAGCTCGTGAAAGTGATGGGCCGGCACAAGCCCGAGAACATCGTGATCGAAATGGCCAGAGAGAACCAGACCACCCAGAAGGGACAGAAGAACAGCCGCGAGAGAATGAAGCGGATCGAAGAGGGCATCAAAGAGCTGGGCAGCCAGATCCTGAAAGAACACCCCGTGGAAAACACCCAGCTGCAGAACGAGAAGCTGTACCTGTACTACCTGCAGAATGGGCGGGATATGTACGTGGACCAGGAACTGGACATCAACCGGCTGTCCGACTACGATGTGGACCATATCGTGCCTCAGAGCTTTCTGAAGGACGACTCCATCGACAACAAGGTGCTGACCAGAAGCGACAAGAACCGGGGCAAGAGCGACAACGTGCCCTCCGAAGAGGTCGTGAAGAAGATGAAGAACTACTGGCGGCAGCTGCTGAACGCCAAGCTGATTACCCAGAGAAAGTTCGACAATCTGACCAAGGCCGAGAGAGGCGGCCTGAGCGAACTGGATAAGGCCGGCTTCATCAAGAGACAGCTGGTGGAAACCCGGCAGATCACAAAGCACGTGGCACAGATCCTGGACTCCCGGATGAACACTAAGTACGACGAGAATGACAAGCTGATCCGGGAAGTGAAAGTGATCACCCTGAAGTCCAAGCTGGTGTCCGATTTCCGGAAGGATTTCCAGTTTTACAAAGTGCGCGAGATCAACAACTACCACCACGCCCACGACGCCTACCTGAACGCCGTCGTGGGAACCGCCCTGATCAAAAAGTACCCTAAGCTGGAAAGCGAGTTCGTGTACGGCGACTACAAGGTGTACGACGTGCGGAAGATGATCGCCAAGAGCGAGCAGGAAATCGGCAAGGCTACCGCCAAGTACTTCTTCTACAGCAACATCATGAACTTTTTCAAGACCGAGATTACCCTGGCCAACGGCGAGATCCGGAAGCGGCCTCTGATCGAGACAAACGGCGAAACCGGGGAGATCGTGTGGGATAAGGGCCGGGATTTTGCCACCGTGCGGAAAGTGCTGAGCATGCCCCAAGTGAATATCGTGAAAAAGACCGAGGTGCAGACAGGCGGCTTCAGCAAAGAGTCTATCCTGCCCAAGAGGAACAGCGATAAGCTGATCGCCAGAAAGAAGGACTGGGACCCTAAGAAGTACGGCGGCTTCGACAGCCCCACCGTGGCCTATTCTGTGCTGGTGGTGGCCAAAGTGGAAAAGGGCAAGTCCAAGAAACTGAAGAGTGTGAAAGAGCTGCTGGGGATCACCATCATGGAAAGAAGCAGCTTCGAGAAGAATCCCATCGACTTTCTGGAAGCCAAGGGCTACAAAGAAGTGAAAAAGGACCTGATCATCAAGCTGCCTAAGTACTCCCTGTTCGAGCTGGAAAACGGCCGGAAGAGAATGCTGGCCTCTGCCGGCGAACTGCAGAAGGGAAACGAACTGGCCCTGCCCTCCAAATATGTGAACTTCCTGTACCTGGCCAGCCACTATGAGAAGCTGAAGGGCTCCCCCGAGGATAATGAGCAGAAACAGCTGTTTGTGGAACAGCACAAGCACTACCTGGACGAGATCATCGAGCAGATCAGCGAGTTCTCCAAGAGAGTGATCCTGGCCGACGCTAATCTGGACAAAGTGCTGTCCGCCTACAACAAGCACCGGGATAAGCCCATCAGAGAGCAGGCCGAGAATATCATCCACCTGTTTACCCTGACCAATCTGGGAGCCCCTGCCGCCTTCAAGTACTTTGACACCACCATCGACCGGAAGAGGTACACCAGCACCAAAGAGGTGCTGGACGCCACCCTGATCCACCAGAGCATCACCGGCCTGTACGAGACACGGATCGACCTGTCTCAGCTGGGAGGCGACAAAAGGCCGGCGGCCACGAAAAAGGCCGGCCAGGCAAAAAAGAAAAAGTAAGGATCCTGATTGATCGATAGAGCTCGAATTTCCCCGATCGTTCAAACATTTGGCAATAAAGTTTCTTAAGATTGAATCCTGTTGCCGGTCTTGCGATGATTATCATATAATTTCTGTTGAATTACGTTAAGCATGTAATAATTAACATGTAATGCATGACGTTATTTATGAGATGGGTTTTTATGATTAGAGTCCCGCAATTATACATTTAATACGCGATAGAAAACAAAATATAGCGCGCAAACTAGGATAAATTATCGCGCGCGGTGTCATCTATGTTACTAGATCGG
sgRNA scaffold的序列为(SEQ ID NO:3):
GTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTTTTTTT
本发明基于CRISPR/Cas9技术的植物基因敲除载体能够应用到植物靶基因的敲除中,同时也能够以试剂盒的形式运用到植物基因编辑中。
本发明的有益效果在于:
1、由Aar I插入位点组成的sgRNA-OsU3结构序列与2x35s-Cas9-ter序列共同构建的CRISPR/Cas9植物敲除的双元载体pCAMBIA1300-OsU3-2x35s-Cas9,能够通过一步酶切连接的方法来实现高效、快速构建单子叶植物基因敲除载体的目的。
2、本发明提供的CRISPR/Cas9敲除载体pCAMBIA1300-OsU3-2x35s-Cas9,具有打靶效率高、脱靶效率极低的特点,本发明载体的打靶效率高于10%,优选达到60%,特别适合应用于植物靶基因的敲除或者以试剂盒的形式运用到植物基因编辑中。
附图说明
图1为单子叶基因敲除载体结构图—pCAMBIA1300-OsU3(Aar I)-Cas9。
图2为敲除基因OsPDS的水稻表型及检测的酶切和测序结果图。
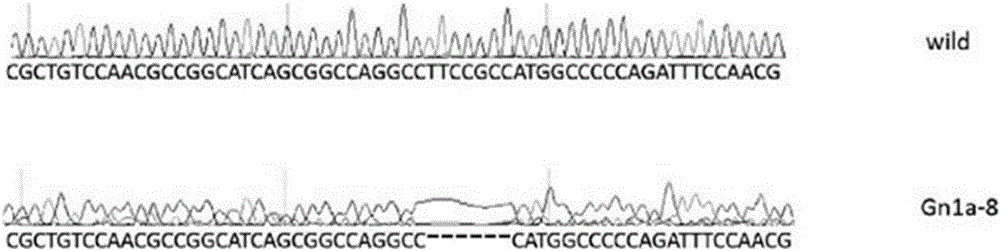
图3为敲除基因Gn1a的测序结果和测序峰图
具体实施方案:
一.单子叶基因敲除载体构建:
1.设计含有Hind III酶切序列的Hind III-OsU3-F/R引物并扩增质粒pOsU3-gRNA中的OsU3(Aar I插入位点)(中科院遗传与发育研究所高彩霞实验室馈赠)片段,
Hind III-OsU3-F:5’-CCCAAGCTTAGGAATCTTTAAACATACGAAC-3’(SEQ ID NO:5);
Hind III-OsU3-R:5’-CAAGCTTGATCCTCTAGAGATTATGTGG-3’(SEQ ID NO:6)
PCR反应条件95℃,30s;56℃,30s;72℃,40s;35cycles。
2.胶回收OsU3片段,连入T载体PMD19-T进行T/A克隆,并挑选阳性克隆送测序,所测得的OsU3(Aar I)-sgRNA结构序列(画下划线的为Aar I的识别序列)为(SEQ ID NO:4):
AAGGAATCTTTAAACATACGAACAGATCACTTAAAGTTCTTCTGAAGCAACTTAAAGTTATCAGGCATGCATGGATCTTGGAGGAATCAGATGTGCAGTCAGGGACCATAGCACAAGACAGGCGTCTTCTACTGGTGCTACCAGCAAATGCTGGAAGCCGGGAACACTGGGTACGTCGGAAACCACGTGATGTGAAGAAGTAAGATAAACTGTAGGAGAAAAGCATTTCGTAGTGGGCCATGAAGCCTTTCAGGACATGTATTGCAGTATGGGCCGGCCCATTACGCAATTGGACGACAACAAAGACTAGTATTAGTACCACCTCGGCTATCCACATAGATCAAAGCTGATTTAAAAGAGTTGTGCAGATGATCCGTGGCAGCTCGCAGGTGAACACAACACCTGCACACGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTTTTTCCACATAATCTCTAGAGGATC
3.酶切获得Hind III-OsU3片段:直接用Hind III限制性内切酶从pMD19-T质粒上酶切获得Hind III-OsU3片段。并与已用Hind III酶切线性化的pCAMBIA1300进行T4连接,构建pCAMBIA1300-OsU3质粒,构建成功后将质粒送去测序,其中测序引物OsU3-F的序列为:CCCAAGCTTAGGAATCTTTAAACATACGAAC(SEQ ID NO:5)。
4.从2X35s-Cas9质粒(中科院上海生命科学研究院朱健康实验室馈赠)上用EcoR I和Sal I酶酶切得到2X35s-Cas9-ter片段,并进行胶回收。同时用EcoR I和Sal I酶切pCAMBIA1300-OsU3质粒,获得线性化质粒。
5.将2X35s-Cas9-ter片段连入pCAMBIA1300-OsU3线性化质粒中,经转化、挑克隆、酶切检测和测序,获得正确的pCAMBIA300-OsU3(Aar I)-Cas9质粒。
实施例1:水稻白化基因的敲除与验证
一、水稻白化基因OsPDS敲除载体的构建
1.根据水稻白化基因OsPDS序列和sgRNA设计工具设计特意的靶序列,序列如下:
OsPDS-oligo1:GGCAGTTGGTCTTTGCTCCTGCAG(SEQ ID NO:7)
OsPDS-oligo2:AAACCTGCAGGAGCAAAGACCAAC(SEQ ID NO:8)
2.将设计的target oligos按如下条件进行退化磷酸化过程:
在PCR管中配制反应体系:
在PCR仪上按以下程序完成反应过程:
3.用Aar I酶酶切pCAMBIA1300-OsU3(Aar I)-Cas9质粒,获取线性化载体,并用CIP酶做去磷酸化处理,以免自连。
4.将退火磷酸化产物稀释100倍,取2μl与线性化载体进行T4连接:
在PCR管中配制连接体系:
16℃连接0.5~1h,连接产物转入大肠杆菌DH5a感受态细胞中并筛选阳性克隆2~4个送去测序。成功插入靶位点的测序结果为:
GGCAGTTGGTCTTTGCTCCTGCAGGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTTTTTTT
表明水稻白化基因OsPDS的CRIPSR/Cas9敲除载体构建成功。
5.将其敲除载体转至农杆菌中,并完成整个组培过程,获得转基因阳性幼苗,做检测验证。
6.酶切试验和目的片段PCR产物的测序结果都表明13%的单株中产生了缺失现象,同时植株中出现白化苗表型,见附图2。
实施例2:水稻Gn1a基因的敲除与验证
1.根据水稻基因Gn1a序列和sgRNA设计工具设计特意的靶序列,序列如下:
Gn1a-2-oligo1:GGCAGCGGCCAGGCCTTCCGCCA(SEQ ID NO:9)
Gn1a-2-oligo2:AAACTGGCGGAAGGCCTGGCCGC(SEQ ID NO:10)
2.按照实施例1中的步骤成功构建Gn1a的敲除载体。
3.将其敲除载体转至农杆菌中,并完成整个组培过程,获得转基因阳性幼苗,做检测验证。
4.Gn1a转基因阳性植株的检测:利用Gn1a-F/R引物
Gn1a-F:5’-GATTGATTGATTGATAATGAAGC-3’(SEQ ID NO:11);
Gn1a-R:5’-CCTATACCTTAATTACCTC-3’(SEQ ID NO:12)
进行目的片段PCR扩增,并将PCR产物进行测序,PCR反应条件为95℃,30s;55℃,30s;72℃,40s;35cycles。
5.目的片段的PCR产物测序结果:在14株转基因水稻中有5株存在突变现象,即突变频率为35%,见附图3。
从这些实施例可知,本发明所构建的单子叶敲除载体pCAMBIA1300(Aar I)-OsU3-Cas9具有简便、快速、高效的优点,特别是具有较高的打靶效率和极低的脱靶率。
所属领域的普通技术人员应当理解:以上任何实施例的讨论仅为示例性的,并非旨在暗示本公开的范围(包括权利要求)被限于这些例子;在本发明的思路下,以上实施例或者不同实施例中的技术特征之间也可以进行组合,步骤可以以任意顺序实现,并存在如上所述的本发明的不同方面的许多其它变化,为了简明它们没有在细节中提供。因此,凡在本发明的精神和原则之内,所做的任何省略、修改、等同替换、改进等,均应包含在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!
- 基于CRISPR/Cas9技术的单子叶植物基因敲除载体及其应用的制作方法与工艺
- 一种T1R2基因过表达慢病毒载体、慢病毒及其构建方法与流程
- 一种Gα基因过表达慢病毒载体、慢病毒及其构建方法与流程
- 弱毒力CMV载体表达抗性基因增强植物抗重金属性能的应用的制作方法与工艺
- 猪ApoE基因敲除载体及其构建方法与应用与流程
- 基于CRISPR/Cas9技术的单子叶植物基因敲除载体及其应用的制作方法与工艺
- 敲除衣藻内源基因和表达外源基因的载体构建方法及应用与流程
- 一种能够快速构建植物基因定点敲除载体的新反应体系的制造方法与工艺
- 介导整合的HIV‑1前病毒基因敲除的锌指核酸内切酶及其制法和应用的制造方法与工艺
- 病毒载体、细胞及构建体的制造方法与工艺