一种利用烟草高效表达IL‑37各亚型成熟蛋白的方法与流程
本发明涉及基因工程和蛋白质表达领域,具体涉及一种利用烟草高效表达IL-37各亚型成熟蛋白的方法。
背景技术:
人白介素-37(IL-37) 是细胞因子IL-1族中新添加的一成员,于2000年发现。与IL-1族中的其它成员相反,IL-37属于抗炎症细胞因子具有广泛的抗炎功效,在限制固有炎症反应(innate inflammation)和抑制获得性免疫(acquired immunity)反应两个方面都起着关键的作用。体外实验研究发现,抑制人细胞内IL-37的正常表达会增加由IL-1,Toll样受体(Toll-link receptor,TRL)和尿酸结晶誘发的炎性细胞因子的表达和释放,包括TNF-α,IL-1α,IL-1ß,IL-6 和G-CSF 。尽管到目前为止在小鼠身上还没有发现与人相对应的 IL-37 基因,但人IL-37 在小鼠体内具有活性。用构建的表达IL-37转基因小鼠(IL37-tg)研究其对不同炎症疾病的影响,结果发现,IL-37-tg 小鼠展现出对多种炎症相关疾病的抵抗力,如内毒素血症,结肠炎,脊髓损伤,代谢综合症,心肌缺血等。此外,注射重组IL-37蛋白质到非IL-37转基因的正常小鼠,可同样地保护由内毒素血症,代谢综合症如肥胖引起的II 型糖尿病,急性肺部损伤,脊髓损伤,心肌梗死,哮喘和肝脏缺血等疾病对动物所造成的损伤。还有,很多人类炎症疾病的严重程度和IL-37 在体内的表达量相关联。例如,炎症性肠疾病患者的结肠组织中有IL-37 表达,但健康个体的相应组织中不存在。类风湿关节炎患者体内IL-37水平升高,某些患者的IL-37基因编码区出现突变,他们的下肢关节疾病得分相对无突变的患者要低,暗示这种突变增强了IL-37抗炎症反应的功能。检测感染HIV-1患者的外周血液发现,其IL-37的稳台态(steady state)mRNA浓度相对未感染者要高4倍。这些结果提示炎症会增加机体内IL-37表达,而IL-37表达量的增加有助于对疾病症状的控制。一般认为,IL-37抑制炎症反应的分子机制是通过和IL-18竟争与IL-18受体(IL-18 receptor) 的结合。IL-37结合到IL-18Rα上后,复合体会捕捉到孤儿誘捕受体(orphan decoy)IL-1R8 与之结合,随后IL1R8 的TLRb 结合区吸引MyD88 (myeloid differentiation factor 88)与之结合. 这一相互间的作用能激活细胞内的信号通路用于阻断炎症的发生,包括对MAPKS和NF-KB的抑制,mTOR 信号通路的抑制以及对抗炎症通路STAT3,Mer and PTEN的激活。同时,因为MyD88分子被困,它参与IL-1和IL-18信号通路的程度也相应降低。IL-37 对抗原特异性获得性免疫的抑制主要是通过耐受树突状细胞的产生。 因此,IL-37不仅可能提供一种治疗诸多炎症疾病的新的有效方法,而且还有可能用于炎性疾病的早期检测和诊断。目前只有大肠杆菌表达生产小量重组人IL-37作试剂用,远不能满足作为新型检测,诊断和治疗疾病手段的需求,迫切需要开发另外一种表达系统能够廉价大批量生产高活性的重组IL-37蛋白质。
植物作为一种生物反应器生产具有医药用途或其他具有商业价值的外源蛋白已展现出聚大的优势和竟争力,具有广阔的市场前景。植物作为重组蛋白生产系统有很多优点:(1) 低成本、易大规模商业化生产。植物能进行光合作用,仅需要来自土壤的矿物质和水分便可在适宜的条件下获得大量人们所需的转基因产物。扩大生产规模容易,只需要很少量的再投入。这与微生物和动物细胞表达系统形成明显的对照。动物细胞培养需要使用昂贵的培养基。扩大生产规模需要大量新的投资包括购买昂贵的新设备(如发效罐等);(2) 由于动物和人病毒无法跨界感染植物细胞,植物表达生产的药物蛋白受人或动物病毒感染的机会很小,因此,與动物细胞生产的药物蛋白比較,植物生产的药物蛋白临床使用更安全;(3) 作为高等真核细胞生物,植物细胞和动物细胞一样可以对新合成的蛋白质进行翻译后加工修饰,如糖基化(glycosylation)、磷酸化( phosphorylation) and亚基组装(subunit assembly),因此表达产物具有与高等动物细胞一样的免疫原性和生物活性。细菌作为生物反应器时,不能对真核生物的蛋白进行有效的翻译后加工,仅适合表达结构简单非糖化的蛋白质,而且本身可能是人类病原物。烟草(Nicotiana tabacum) 作为表达平台具有其他植物生产体系不可比拟的优点。首先,自1989年第一次用烟草表达抗体[3]以来,烟草已经成为生产重组蛋白的主要平台;其次,烟草是叶子植物产量高,且叶片可溶性蛋白含量高,作为重组蛋白生产平台可提高外源蛋白在植物中的表达量;再次,烟草的遗传转化技术非常成熟,重组蛋白在烟草中的表达可选择不同的方式,比如细胞核表达(转基因植物)、叶绿体表达和瞬时表达;最后,烟草既不是人类食物,也不是饲料作物,因此降低了转基因材料污染食物链或饲料链的风险。
IL-37基因位于人类第二号染色体上,其基因表达产物共有五个剪切亚型 (isoforms a to e) 。 目前的研究主要集中在IL-37b,对于其他亚型的功能还不太了解。不过因IL-37a和IL-37d的结构与IL-37b相似,即它们的编码区都包含第四个外显子,可以编码β-三叶草结构。此结构被认为是IL-1家族细胞因子的特征性结构. 所以推测和IL-37b 一样,IL-37a和IL-37d具有生物学功能。而IL-37c和IL-37e缺乏第四个外显子,不能编码β-三叶草结构,推测它们可能没有生物学功能或与IL-37b的功能有所不同。 IL-37在多种组织中表达,包括血液单核细胞,组织聚吞噬细胞,滑膜细胞,扁桃体B细胞,浆细胞,肿瘤细胞以及皮肤,肾脏和肠道的上皮细胞。IL-37 剪切亚型的分布也有一定的组织特异性。例如,IL-3a主要在大脑中表达,IL-37b主要存在于肾脏,心脏特异性表达IL-37c,而IL-37d和IL-37e则主要在骨髓和睾丸中表达。IL-37的五个亚型都是先合成蛋白前体,切除信号肽后产生成熟蛋白。但它们的信号肽都非经典序列。信号肽的切割主要由半胱天冬酶-1(caspase-1)完成。不过最新的研究认为IL-37信号肽的完全切割需要别的酶参与其中。在IL-37的五个亚型中,IL-37b蛋白链最长,由218个氨基酸组成,含一个45个氨基酸组成的N端信号肽。虽然IL-37b前体和IL-37b 成熟蛋白形式都发现有活性,但IL-37b成熟蛋白的活性比IL-37b前体蛋白的活性高出20-30倍。IL-37成熟蛋白也可以进入细胞核与核DNA结合从而影响基因转录,而其前体蛋白则不能。人IL-37天然以二聚体形式(dimer)存在,但二聚体的形成不是以共价键(二硫鍵)方式连接。到目前为此,在植物细胞中还没有发现编码caspase-1酶的基因,尽管某些植物蛋白质序列表现出与动物caspases 酶一定的相似性。这意味着当用植物细胞表达IL-37基因时,获得的表达产物是IL-37前体而非成熟蛋白,因植物细胞无法识别位于IL-37前体的N末端信号肽序列並加以剪切。
目前,一种可能的方法是只把编码成熟IL-37的基因在植物细胞内表达,但此方法具有明显的缺点那就是目的蛋白的低表达量和低活性,因为新合成的蛋白由于缺少信号肽介导转运易引起错误折叠不仅使之失去活性而且易被植物细胞细胞质中 的蛋白酶降解 。
另一种可能的方法是用植物蛋白信号肽替换IL-37基因的原信号肽序列使新合成的目的蛋白分子能实现跨膜转运并在此过程中通过切掉嫁接的植物蛋白信号肽产生成熟的具有天然N端的目的蛋白. 但这需要测试许多不同的植物蛋白的信号肽才有可能找到合适的信号肽,因为不同信号肽有不同的作用对蛋白质表达量有显著影响。此外,即便用植物蛋白信号肽作替换,以前的发现表明由此法表达的外源基因,蛋白质终产物中有相当一部分是植物信号肽未被除掉的前体蛋白。
技术实现要素:
本发明的目的在于克服现有技术的缺点,提供一种利用利用TEV 蛋白酶在体内和体外高特异性和高活性的双重特征,通过设计和合成与TEV 蛋白酶在构架内连接的IL-37融合基因烟草高效表达IL-37各亚型成熟蛋白的方法。
本发明的目的通过以下技术方案来实现:一种利用烟草高效表达IL-37各亚型成熟蛋白的方法,包括以下步骤:
S1. 设计IL-37-TEV融合基因:
包括在信号肽和IL-37成熟蛋白各亚型编码序列之间插入TEV蛋白酶及其酶切位点编码序列获得融合基因、在IL-37成熟蛋白各亚型的天然信号肽和TEV蛋白酶序列间添加一段柔性连接肽,所述IL-37-TEV融合基因的基本结构顺序为:自身原始信号肽-柔性连接肽-TEV蛋白酶-TEV酶识别位点-成熟蛋白各亚型IL-37编码序列;
所述IL-37成熟蛋白各亚型为IL-37a、IL-37b、IL-37c、IL-37d和IL-37e,其中, IL-37a-TEV融合基因的核苷酸序列如SEQ ID NO.1所示,IL-37b-TEV融合基因的核苷酸序列如SEQ ID NO.2所示,IL-37c-TEV融合基因的核苷酸序列如SEQ ID NO.3所示,IL-37d-TEV融合基因的核苷酸序列如SEQ ID NO.4所示,IL-37e-TEV融合基因的核苷酸序列如SEQ ID NO.5所示;其IL-37a-TEV融合基因编码的氨基酸序列如SEQ ID NO.6所示,IL-37b-TEV融合基因编码的氨基酸序列如SEQ ID NO.7所示,IL-37c-TEV融合基因编码的氨基酸序列如SEQ ID NO.8所示,IL-37d-TEV融合基因编码的氨基酸序列如SEQ ID NO.9所示,IL-37e-TEV融合基因编码的氨基酸序列如SEQ ID NO.10所示;所述柔性连接肽序列如SEQ ID NO.11所示;
S2. 构建重组表达载体:
将步骤S1设计的IL-37-TEV融合基因连接到过渡载体PRTL-2-Gus,经酶切分离后再克隆到植物表达双元空载体的T-DNA ,获得IL-37成熟蛋白各亚型的重组表达载体;
S3.表达:
将步骤S2构建的重组表达载体导入到烟草中进行表达。
进一步地,所述IL-37成熟蛋白各亚型为IL-37a、IL-37b、IL-37c、IL-37d和IL-37e。
进一步地,所述重组表达载体由植物组成型E35S启动子及带有5’端非翻译的TEV前导序列驱动的基因进行的表达,以Kanamycin 抗性的NPTII基因作为筛选标记。
进一步地,将步骤S2构建的重组表达载体导入到烟草细胞中获得IL-37的转基因烟草植株进行稳定表达IL-37各亚型成熟蛋白。
进一步地,将步骤S2构建的重组表达载体注射到烟草中瞬时表达IL-37各亚型成熟蛋白。
本发明中通过常用的农杆菌介导的叶片遗传转化方法将重组表达载体质粒转入烟草细胞中,经愈伤细胞诱导,分化再生和kanamycin抗性筛选获得稳定表达IL-37的转基因烟草植株。
将重组表达载体质粒与含有可抑制基因沉默的番茄丛矮病毒(tomato bushy stunt virus,TBSV)基因P19的载体质粒用农杆菌叶片注射法共注射到烟草叶片中实现IL-37 成熟蛋白在烟草中的瞬时表达。
本发明具有以下优点:
在利用植物作为生物器表达IL-37时,由于植物细胞不含有半胱天冬酶-1,因此不能够识别和剪切IL-37前体的信号肽序列而产生成熟目的蛋白。本发明公布了一种利用烟草高效表达IL-37各亚型成熟蛋白的方法,首先利用烟草的蚀纹病毒(TEV)蛋白酶在体内和体外高特异性和高活性的双重特点,设计在信号肽和IL-37成熟蛋白编码序列之间插入 TEV蛋白酶及其识别位点编码序列使之形成融合基因。由此,由植物细胞表达产生的单链多肽融合蛋白在细胞内通过TEV酶的活性进行自我特异性切割从而产生具有天然结构的IL-37成熟蛋白。此外在信号肽和TEV酶之间添加一段柔性的连接肽序列以保证作为融合单位的TEV酶能相对独立的折叠从而最大程度地保持其酶的活性功能。其后,构建了含合成的,密码子优化的融合基因的表达载体,以植物组成型E35S 启动子驱动融合基因在植物细胞内的表达;利用脓杆菌介导的叶片转化方法将载体导入叶细胞,经愈伤细胞诱导、分解再生和卡钠霉素抗性筛选获得转基因烟草植株。将重组表达载体质粒与含有可抑制基因沉默的番茄丛矮病毒基因P19的载体质粒用农杆菌叶片注射法共注射到烟草叶片中实现IL-37 成熟蛋白在烟草中的瞬时表达。由于T-DNA 插入植物细胞染色体DNA的随机性,及其由此对外原蛋白质表达带来的影响,因此每一重组表达载体转化烟草后获得的独立转基因植株的筛选不少于25株。利用Western blot等方法,对获得的转基因烟草植株进行检查分析。结果显示,所构建的表达载体正确地表达IL-37成熟蛋白,并以二聚体形式存在,这与自然的人IL-37蛋白的形式完全一致。最后,对C-端含有Hisx6 标鉴的IL-37通过His蛋白纯化试剂盒的方法从转基因烟草植株叶中分离。纯化的Hisx6 标鉴IL-37成熟蛋白与小鼠肾上皮细胞培养。结果表明植物表达的IL-37成熟蛋白能有效地抑制由细菌脂多醣诱发的炎症子的分泌,如TNF-a。因此植物表达的IL-37 具有明显的活性,为进一步利用植物生物反应器大规模生产重组IL-37奠定了基础。
为同时实施IL-37 成熟蛋白在烟草中的瞬时表达,分别将重组表达载体质粒与含有可抑制基因沉默的番茄丛矮病毒基因P19的表达载体混合通过农杆菌叶片注射法共注射到本氏烟草叶片中,侵染后1-6d内采收叶片。利用Western blot等方法对侵染的烟草叶片进行检查分析。结果显示,所构建的表达载体同样能够在烟草叶片中瞬时表达IL-37成熟蛋白,所得产品对治疗诸多炎症疾病和炎症疾病的早期检测和诊断具有重要应用价值。
附图说明
图1为IL-37-TEV融合基因结构示意图;
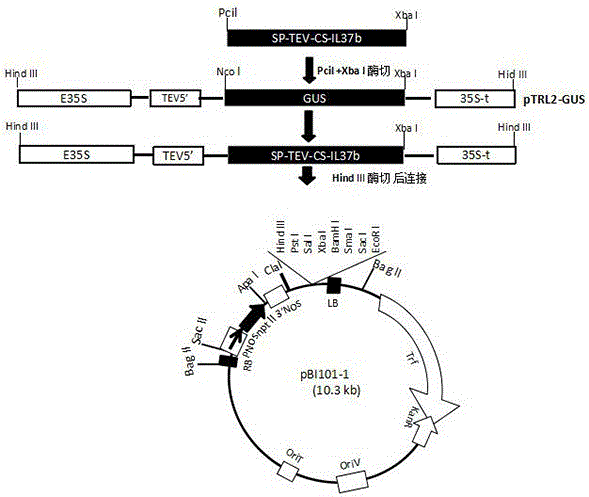
图2为植物表达载体质粒构建流程图;
图3为 pBI-SP-TEV-CS-IL-37b转基因烟草总可溶性蛋白免疫印迹法 检测IL-37b 成熟蛋白的表达,图中,A为使用 anti-IL-37 抗体; B为使用 anti-TEV 抗体;
图4为pBI-SP-TEV-CS-IL-37bHis6转基因烟草总可溶性蛋白免疫印迹法检测IL-37bHis6成熟蛋白的表达,图中,A为使用 anti-IL-37 抗体;B为使用 anti-TEV 37 抗体;
图5为注射pBI-SP-TEV-CS-IL-37b表达载体质粒+ P19 的烟草在不同时间表达IL-37b蛋白的Western blot 检测,使用 anti-IL-37 抗体;
图6为 Western blot检测经Ni柱亲和层析法获得的纯化IL-37b蛋白,图中, A为使用 anti-IL-37 抗体; B为使用 anti-TEV 抗体;
图7为烟草表达的IL-37成熟蛋白对抑制细胞释放炎症因子的活性分析,其中,所使用的细胞为小鼠肾脏表皮细胞;Med 指细胞培养基中含有1ug/ml 的LPS但不含IL-37蛋白作对照用,IL-37 (STAND) 为标准蛋白。
具体实施方式
下面结合附图及实施例对本发明做进一步的描述,本发明的保护范围不局限于以下所述。
本发明公开了一种利用烟草高效表达IL-37各亚型成熟蛋白的方法,利用烟草蚀纹病毒蛋白酶(tobacco etch virus protease, TEV protease) 的高特异性和高活性的双重特点,在信号肽和成熟IL-37蛋白编码序列之间插入TEV蛋白酶及其酶切位点编码序列形成IL-37-TEV 融合基因;根据烟草密码子的偏好性,在保证氨基酸序列不变的情况下,对融合基因序列进行优化。并委托加拿大多伦多的Bio Basic 公司合成;将构建的IL-37-TEV 融合基因的5'端上游与常用的启动子包括其他调节区域(如增强子) 连接, 并在3'端下游与转录终止子连接;将构建的重组表达载体通过转化整合到烟草细胞染色体上,培育高效稳定表达IL-37 的转基因烟草植株以及通过注射构建的重组表达载体到烟草叶瞬时表达目的蛋白。烟草蚀纹病毒蛋白酶 (TEV protease) 是Nla蛋白酶的27 kDa催化域的简称。TEV蛋白酶为高度位点特异性半胱氨酸蛋白酶,该酶可识别7个氨基酸识别位点(谷氨酸-天冬酰胺-亮氨酸-酪氨酸-苯丙氨酸-谷氨酰胺-甘氨酸)并切断谷氨酰胺与甘氨酸之间的肽键。由于其高活性、高特异性的双重特点,TEV蛋白酶为融合蛋白质裂解的有效试剂。其上游裂解产物含6个多增加的氨基酸,而下游裂解产物只含有1个多增加的甘氨酸。Shin et al (Protein Sci. 2005; 14, 936–941) 报道了细菌表达的含TEV 蛋白酶的融合蛋白通过其酶活性能在细胞内实施特异性自我切割释放出具有天然结构(native) 的活性融合伴侣(fusion partner) 。Marcos and Beachy (J Gen Virol. 1997;78, 1771–1778) 报道外源TEV 蛋白酶在植物细胞内有生物活性。这些最初結果表明通过构建与TEV 蛋白酶连接的融合基因可达到表达具有天然结构的重组目的蛋白。
实施例1:设计和合成Sp-TEV-CS-IL-37b,Sp-TEV-CS-37a,Sp-TEV-CS-IL-37d,Sp-TEV-CS-IL-37c和 Sp-TEV-CS-IL-37e 融合基因
首先在NCBI网站的GenBank 中获得IL-37各蛋白亚型的cDNA序列,编码为Gen Bank accession NO.NM_014439.3 for IL37b;Gen Bank accession NO.NM_173205.1 for IL-37a;Gen Bank accession NO.NM_173204.1 for IL-37c;Gen Bank accession NO.NM_173202.1 for IL-37d and Gen Bank accession NO.NM_173203.1 for IL-37e。同样地,从Gen Bank 中获得编码TEV蛋白酶的核酸序列,编码为Gen Bank accession NO.NP_062908.1。
设计IL-37-TEV 融合基因。
如图 1所示,融合基因的基本结构顺序是:自身原始信号肽-柔性连接肽(GGGGSx3)-TEV蛋白酶-TEV酶识别位点-成熟IL-37编码序列。
根据烟草密码子的偏好性对设计的TEV-IL-37融合基因在不改变氨基酸的前提下进行优化,优化后的融合基因由人工合成,合成时在5'端添加PcI1酶切位点,在3'端增加Xba I酶切位点。本实施例的融合基因由加拿大多伦多Bio Basics 公司合成并克隆到质粒PUC57。合成后的融合基因的核苷酸序列如SEQ ID NO: 1、2、3、4、5 所示,其编码的氨基酸序列如SEQ ID NO: 6、7、8、9、10 所示。
实施例2:重组表达载体pBI-Sp-TEV-CS-IL-37b,pBI-Sp-TEV-CS-IL-37a, pBI-Sp-TEV-CS-IL-37d,pBI-Sp-TEV-CS-IL-37c和 pBI-Sp-TEV-CS-IL-37e的构建
(1) 重组表达载体pBI-Sp-TEV-CS-IL-37b的构建: 构建方法如图 2所示,包括下列主要步骤:
使用Nco I+ Xba I 双酶切过渡载体pRTL2-GUS,回收去除GUS基因的4.2 Kb 的载体片段。质粒pRTL2-GUS 含有E35S启动子 (enhanced 35S promoter)- 5'端非翻译的TEV前导序列-GUS- Nos3’ 终止子的GUS基因表达盒。
使用Pci l 和Xba I酶切所述pUC57-Sp-TEV-CS- IL-37b 质粒,回收融合基因片段 并插入到pRTL2-GUS 经Nco I+ Xba I酶切后回收的载体片段得到过渡表达载体 pRTL2- Sp-TEV-CS- IL-37b。
使用Hind III 酶切所述的过渡表达载体pRTL2- Sp-TEV-CS- IL-37b,回收含融和基因表达盒(E35S-Sp-TEV-CS-IL-37b-Nos3’)DNA片段,并插入到经Hind III酶切的双元载体pBI101-1获得重组表达载体 pBI- Sp-TEV-CS-IL-37b。
由三亲交配法(tri-parental mating) 将构建的表达载体pBI- Sp-TEV-CS-IL-37b转入农杆菌。
(2) 重组表达载体pBI-Sp-TEV-CS-IL-37a 的构建: 其构建方法与载体pBI- Sp-TEV-CS-IL-37b的构建相同。
(3) 重组表达载体pBI-Sp-TEV-CS-IL-37d 的构建: 其构建方法与载体pBI- Sp-TEV-CS-IL-37b的构建相同。
(4) 重组表达载体pBI-Sp-TEV-CS-IL-37c 的构建: 其构建方法与载体pBI- Sp-TEV-CS-IL-37b的构建相同。
(5) 重组表达载体pBI-Sp-TEV-CS-IL-37e 的构建: 其构建方法与载体pBI- Sp-TEV-CS-IL-37b的构建相同。
实施例3: 烟草的稳定转化
将构建的重组表达载体质粒由三亲交配法(tri-parental mating) 分别转入农杆菌LBA4404。
烟草培养。 烟草种子(N. tabacum CV.81V9)用70%酒精消毒后,种植在一个含MS + 8g/L琼脂培养基的植物组织培养盒(magenta vessel)中,培养箱培养。温度25±1°C,湿度70%左右,16小时光照/8小时黑暗循环光照下培养。3-4周后,幼苗长到3-4片真叶时,即可取无菌植物叶作遗传转化材料用。
重组表达载体遗传转化烟草。参照Horsch等 (Science 1985. 227,1229-1231)描述的叶片转化方法引入本发明中的质粒。以重组载体PBI-Sp-TEV-CS-IL-37 b为例,将含有该质粒的农杆菌LBA4404单菌落置于5mL 液体TYC (蛋白胨8g/L,酵母4g/L,氯化钙6.6%) +50mg/L Km +20mg/L Rif培养基中,28℃,180rpm,振荡培养。取 1 mL 菌液置于 50 mL 液体液体TYC +50 mg/L Km +20mg/L Rif培养基中,28℃,180rpm,培养至OD600=0.5~0.6,6000rpm,离心10min,收集菌体,40mL MS 培养液重悬菌体,烟草无菌苗叶片去主叶脉和叶片边缘并切成约1.0cm 小片,在菌液中浸染5min,无菌滤纸吸干菌液,接种于MS+1.0mg/L 6-BA+0.1mg/L NAA 培养基上,25℃,黑暗条件下共培养 3 d 后,转接至选择培养基 (MS+ 6-BA(1 mg/L)+NAA(0.1 mg/L) + Km (100 mg/L) + Cb (500 mg/L)+ 蔗糖(30g/L) + 琼脂 (8g/L)) 上进行芽诱导筛选培养,芽长至约2 cm 时切下,接种于生根培养基(MS+ Km (100 mg/L) + Cb (500 mg/L) +蔗糖(30g/L) + 琼脂 (8g/L))上进行诱导生根培养。生根后的再生苗移植至花盆中,放到(25±0.5)℃ 温室培养。
实施例4:烟草的瞬时转化
烟草培养:本氏烟草种子(N. Benthamiana ),4C°用水浸泡48 h,播种于土壤中,在25± 1℃、16 h/d 光照的培养箱中培养。 约 30 天后待其为五叶或六叶期时即可用于实验。
分别将含有pBI-Sp-TEV-CS-IL-37b,pBI-Sp-TEV-CS-IL-37a, pBI-Sp-TEV-CS-IL-37d,pBI-Sp-TEV-CS-IL-37c和 pBI-Sp-TEV-CS-IL-37e质粒的 LBA4404 农杆菌的单菌落接种到TYC液体培养基+ 50 mg/L Km + 100 mg/L Rif, 28℃下,摇24 至 48h。吸取100ul菌液加入10ml TYC液体中,再培养24 h。收集菌体,4℃下,5000×g 离心 10 min,去除上清液,留菌体。将菌体悬浮在悬浮液 (5 mg/ml d-葡萄糖,500 nM MES,20 nM Na3PO4•12 H2O,1M acetosyringone (乙酰丁香酮)) ,6000rpm,离心10min,收集菌体,悬浮液重悬菌体,测定OD600值。将含有P19 (作用是增加目的蛋白的表达量) 和目的基因的悬浮液根据所定农杆菌的滴度OD=0.5 按1:1比例混合成侵染液。
用去针头的一次性注射器将悬浮有农杆菌的侵染液从烟叶背面注入叶片内。注射完毕后,置于25±1℃人工培养室中培养。注射24 h后收集注射区域烟草叶,每天一次直到第6天。
实施例5: 转基因烟草IL-37蛋白表达的检测
以重组表达载体pBI-Sp-TEV-CS-IL-37b转基因烟草为例。取新鲜转基因烟草叶片 0.5 g,立即放在研钵中加液氮 磨碎,将研磨好的叶片收集于 2mL EP 管中加入 1 mL蛋白提取液(200mM Tris pH8.0,100 mM NaCL,400 mM 蔗糖,10 mM EDTA,0.1mM ß-琉基乙醇,0.05% Tween-20,1 mM苯甲基磺酰氟(PMSF),0.1% aprotitin 和0.1% leupeptin),冰上孵育 20~30min,4℃ 12000 rpm/min离心 20min。收集上清,上清为可溶蛋白提取液。
SDS-PAGE 和 Western 免疫印迹法(Western blot) 分析。取蛋白提取液,加入sample buffer,沸水中煮样10min。蛋白质样品经SDS-PAGE分离后,利用Western免疫印迹方法(Western Blot) 将蛋白质转移到膜上,然后利用抗体进行检测。Western blot 检测结果证明,获得的转基因植株表达IL-37b成熟蛋白,并以期望的二聚体(dimer)形式存在,如图3所示,图中,M 为蛋白分子量marker; WT为非转基因烟草總可溶性蛋白作负对照; lanes b1至b18 为部分转基因植株; 箭头所指为烟草表达的IL-37b 成熟蛋白形式(二聚体); 图左侧的数字为蛋白分子量大小标准。IL-37是非糖蛋白, 不含信号肽的IL-37b成熟蛋白由173个氨基酸构成,分子量约为19 kDa. IL-37b二聚体的分子量与IL-37-TEV融合蛋白在胞内自我剪切后产生的第二个多肽产物 (即信号肽+TEV片段) 的分子量相似,发生两个蛋白片段共迁移为一个宽条带, 如图3A所示。条带中间呈现出的不与IL-37抗体反应的泡状带为SP-TEV多肽。
实施例6:转基因烟草IL-37His6蛋白表达的检测
以重组表达载体pBI-Sp-TEV-CS-IL-37bHis6转基因烟草为例。蛋白提取方法如实施例5的方法提取。
SDS-PAGE 和 Western 免疫印迹法(Western blot) 分析。结果与实施例5所述一致,如图4所示,图中,M 为蛋白分子量marker; WT为非转基因烟草總可溶性蛋白作负对照; lane b’1 to b’18 为部分转基因植株;箭头所指为表达的IL-37bHis6 成熟蛋白形式(二聚体);图左侧的数字为蛋白分子量大小标准。
实施例7:烟草瞬时表达IL-37的检测
以重组表达载体PBI-Sp-TEV-CS-IL-37 b注射烟草瞬时表达为例。使用实施例5所述蛋白质提取方法,从不同时间收集的注射区域烟草中提取蛋白质。
使用实施例5所述的SDS-PAGE和Western blot 方法检测提取的蛋白质。检测结果证明烟草瞬时表达IL-37 b 成熟蛋白,并同样以二聚体(dimer)形式存在,如图5所示,图中,1-6指注射表达载体后的1至6天(DAI)收集的烟草。M 为蛋白分子量marke;IL-37+为IL-37蛋白标准品;WT为非转基因烟草總可溶性蛋白作负对照;箭头所指为烟草瞬时表达的IL-37b 成熟蛋白(二聚体),图左侧的数字为蛋白分子量大小标准。
实施例8:烟草表达的IL-37的纯化和检测
以从转基因烟草纯化IL-37bHis6 为例。使用His 靶向的蛋白纯化试剂盒(His-Trap kit,购自Cedar lane Canada)和根据使用说明书进行纯化。
使用实施例5所述的SDS-PAGE和Western blot 方法检测纯化的蛋白,结果如图6所示, 其中,M为蛋白质分子量marker;TSP为转基因烟草可溶性总蛋白(上柱前);FT为flow- through;W为wash;E1为eluted fraction 1;E2为eluted fraction 2;图 6A箭头所指为IL-37b 成熟蛋白 (二聚体形式) ,当使用抗TEV抗体检查时,E1和E2不与抗体反应,如图6 B,表明纯化的IL-37b蛋白中不含有SP-TEV多肽。图左侧的数字为蛋白分子量大小标准。图6 结果表明纯化的IL-37bHis6中不含有SP-TEV蛋白片段。
实施例9:烟草表达的IL-37成熟蛋白对抑制细胞释放炎症因子的活性分析
无菌取B6小鼠肾脏轻碾,加HANK'S(平衡盐溶液)液冲洗,2000 rpm,5 min,离心弃上清,加1% 胶原溶解酶 V(collagens V,Invitragen),37C°,30min溶解肾组织。加HANK'S液阻止溶解。离心,种植细胞到肾细胞培养液K1+/+(K1+/+ 复合培养液 50:50 DMEM 和Ham’s F12;含有10%小牛血清(FBS),和激素包括5Ig/ml的胰岛素(insulin),34pg/ml三碘甲状腺氨酸(triiodothyronine),5Ig/ml 转铁蛋白(transferrin),1.73ng/ml 亚硒酸钠(sodium selenite),8ng/ml 氢化可的松(hydrocortisone),25ng/ml 上表皮生长因素(epidermal growth factor),100u/ml青霉素(penicillin),100u/ml链霉素(streptomycin )中. 5% CO2,37C° 一定的湿度培养箱培养。5-7天后肾上皮细胞长满到80%。悬浮细胞于K1+/+液中。用0.4% 用台盼蓝 (Trypan blue) 染色,血球计(hemocytometer)计数以0.8x106 细胞浓度种植到96孔培养板。培养24hr后加入100 ul K1-/- (DMEM/Ham’s F12 50/50 Mix),100u/ml青霉素,100u/ml链霉素 和不同浓度纯化的植物表达的IL-37(prIL-37), 每孔最终浓度分别为 0,20,10,5,2,5 0.625 nM). 标准IL-37蛋白作负对照. 培养24hr小时后,弃上清液,加100ul K1-/- 培养液含1ug/ml 脂多糖(lipopolysaccharides)。培养24hr后收集上清液,用TNF-a试剂盒(R&D Canada)测定 TNF-a浓度,如图7所示,结果表明植物表达的IL-37具有高度生物活性。
SEQUENCE LISTING
<110> 马生武、安托尼.迈克尔.耶尼卡、邓绍平、杨洪吉、魏亮
<120> 一种利用烟草高效表达IL-37各亚型成熟蛋白的方法
<130> 2016
<160> 11
<170> PatentIn version 3.5
<210> 1
<211> 1371
<212> DNA
<213> 人工序列
<400> 1
atgtcaggtt gtgatagaag agagacagaa actaagggta aaaacagctt caagaagcga 60
ctaagaggac caaaggttgg tggaggtgga tctggtggag gtgggtcagg agggggggga 120
tctggtgaat ctcttttcaa gggtcctaga gattacaatc caatcagtag tacaatctgt 180
cacttaacca acgaaagcga tggtcataca acaagtctct acggtatcgg atttggtcca 240
ttcattatta ccaacaagca tctcttcaga agaaacaacg gaacactcct cgttcaatca 300
ctgcatggag ttttcaaggt aaagaacaca accaccttgc aacaacattt gatagacgga 360
agagatatga ttataattag gatgccaaag gacttcccgc cttttccaca gaagttgaag 420
tttcgagagc ctcaacgtga agaacgtatt tgccttgtaa ccactaattt tcagacaaag 480
tccatgtcca gcatggtttc agatacttcc tgcacttttc cttcctctga tggaattttc 540
tggaaacact ggattcagac aaaagacggg caatgcgggt ctcctttggt ttcaactagg 600
gatgggttca tagttggcat acattcagca tcaaatttta ctaatactaa taattatttt 660
acttctgtcc ccaaaaattt tatggagctt cttactaatc aggaggctca gcagtgggtc 720
tctggctgga ggttgaatgc tgactctgtt ttgtggggcg gccacaaagt gtttatgtca 780
aaaccagaag aaccatttca acccgtgaaa gaggccactc aattaatgaa tgagcttgtg 840
tattctcaag aaaatttgta ttttcaaggc aagaacttaa acccgaagaa attcagcatt 900
catgaccagg atcacaaagt actggtcctg gactctggga atctcatagc agttccagat 960
aaaaactaca tacgcccaga gatcttcttt gcattagcct catccttgag ctcagcctct 1020
gcggagaaag gaagtccgat tctcctgggg gtctctaaag gggagttttg tctctactgt 1080
gacaaggata aaggacaaag tcatccatcc cttcagctga agaaggagaa actgatgaag 1140
ctggctgccc aaaaggaatc agcacgccgg cccttcatct tttatagggc tcaggtgggc 1200
tcctggaaca tgctggagtc ggcggctcac cccggatggt tcatctgcac ctcctgcaat 1260
tgtaatgagc ctgttggggt gacagataaa tttgagaaca ggaaacacat tgaattttca 1320
tttcaaccag tttgcaaagc tgaaatgagc cccagtgagg tcagcgattg a 1371
<210> 2
<211> 1449
<212> DNA
<213> 人工序列
<400> 2
atgtcctttg tgggggagaa ctcaggagtg aaaatgggct ctgaggactg ggaaaaagat 60
gaaccccagt gctgcttaga agacccggct gtaagccccc tggaaccagg cccaagcctc 120
cccaccatga attttggtgg aggtggatct ggtggaggtg ggtcaggagg ggggggatct 180
ggtgaatctc ttttcaaggg tcctagagat tacaatccaa tcagtagtac aatctgtcac 240
ttaaccaacg aaagcgatgg tcatacaaca agtctctacg gtatcggatt tggtccattc 300
attattacca acaagcatct cttcagaaga aacaacggaa cactcctcgt tcaatcactg 360
catggagttt tcaaggtaaa gaacacaacc accttgcaac aacatttgat agacggaaga 420
gatatgatta taattaggat gccaaaggac ttcccgcctt ttccacagaa gttgaagttt 480
cgagagcctc aacgtgaaga acgtatttgc cttgtaacca ctaattttca gacaaagtcc 540
atgtccagca tggtttcaga tacttcctgc acttttcctt cctctgatgg aattttctgg 600
aaacactgga ttcagacaaa agacgggcaa tgcgggtctc ctttggtttc aactagggat 660
gggttcatag ttggcataca ttcagcatca aattttacta atactaataa ttattttact 720
tctgtcccca aaaattttat ggagcttctt actaatcagg aggctcagca gtgggtctct 780
ggctggaggt tgaatgctga ctctgttttg tggggcggcc acaaagtgtt tatgtcaaaa 840
ccagaagaac catttcaacc cgtgaaagag gccactcaat taatgaatga gcttgtgtat 900
tctcaagaaa atttgtattt tcaaggcgtt cacacaagtc caaaggtgaa gaacttaaac 960
ccgaagaaat tcagcattca tgaccaggat cacaaagtac tggtcctgga ctctgggaat 1020
ctcatagcag ttccagataa aaactacata cgcccagaga tcttctttgc attagcctca 1080
tccttgagct cagcctctgc ggagaaagga agtccgattc tcctgggggt ctctaaaggg 1140
gagttttgtc tctactgtga caaggataaa ggacaaagtc atccatccct tcagctgaag 1200
aaggagaaac tgatgaagct ggctgcccaa aaggaatcag cacgccggcc cttcatcttt 1260
tatagggctc aggtgggctc ctggaacatg ctggagtcgg cggctcaccc cggatggttc 1320
atctgcacct cctgcaattg taatgagcct gttggggtga cagataaatt tgagaacagg 1380
aaacacattg aattttcatt tcaaccagtt tgcaaagctg aaatgagccc cagtgaggtc 1440
agcgattga 1449
<210> 3
<211> 1329
<212> DNA
<213> 人工序列
<400> 3
atgtcctttg tgggggagaa ctcaggagtg aaaatgggct ctgaggactg ggaaaaagat 60
gaaccccagt gctgcttaga agacccggct gtaagccccc tggaaccagg cccaagcctc 120
cccaccatga attttggtgg aggtggatct ggtggaggtg ggtcaggagg ggggggatct 180
ggtgaatctc ttttcaaggg tcctagagat tacaatccaa tcagtagtac aatctgtcac 240
ttaaccaacg aaagcgatgg tcatacaaca agtctctacg gtatcggatt tggtccattc 300
attattacca acaagcatct cttcagaaga aacaacggaa cactcctcgt tcaatcactg 360
catggagttt tcaaggtaaa gaacacaacc accttgcaac aacatttgat agacggaaga 420
gatatgatta taattaggat gccaaaggac ttcccgcctt ttccacagaa gttgaagttt 480
cgagagcctc aacgtgaaga acgtatttgc cttgtaacca ctaattttca gacaaagtcc 540
atgtccagca tggtttcaga tacttcctgc acttttcctt cctctgatgg aattttctgg 600
aaacactgga ttcagacaaa agacgggcaa tgcgggtctc ctttggtttc aactagggat 660
gggttcatag ttggcataca ttcagcatca aattttacta atactaataa ttattttact 720
tctgtcccca aaaattttat ggagcttctt actaatcagg aggctcagca gtgggtctct 780
ggctggaggt tgaatgctga ctctgttttg tggggcggcc acaaagtgtt tatgtcaaaa 840
ccagaagaac catttcaacc cgtgaaagag gccactcaat taatgaatga gcttgtgtat 900
tctcaagaaa atttgtattt tcaaggcgtt cacacagaga tcttctttgc attagcctca 960
tccttgagct cagcctctgc ggagaaagga agtccgattc tcctgggggt ctctaaaggg 1020
gagttttgtc tctactgtga caaggataaa ggacaaagtc atccatccct tcagctgaag 1080
aaggagaaac tgatgaagct ggctgcccaa aaggaatcag cacgccggcc cttcatcttt 1140
tatagggctc aggtgggctc ctggaacatg ctggagtcgg cggctcaccc cggatggttc 1200
atctgcacct cctgcaattg taatgagcct gttggggtga cagataaatt tgagaacagg 1260
aaacacattg aattttcatt tcaaccagtt tgcaaagctg aaatgagccc cagtgaggtc 1320
agcgattga 1329
<210> 4
<211> 1386
<212> DNA
<213> 人工序列
<400> 4
atgtcctttg tgggggagaa ctcaggagtg aaaatgggct ctgaggactg ggaaaaagat 60
ggtggaggtg gatctggtgg aggtgggtca ggaggggggg gatctggtga atctcttttc 120
aagggtccta gagattacaa tccaatcagt agtacaatct gtcacttaac caacgaaagc 180
gatggtcata caacaagtct ctacggtatc ggatttggtc cattcattat taccaacaag 240
catctcttca gaagaaacaa cggaacactc ctcgttcaat cactgcatgg agttttcaag 300
gtaaagaaca caaccacctt gcaacaacat ttgatagacg gaagagatat gattataatt 360
aggatgccaa aggacttccc gccttttcca cagaagttga agtttcgaga gcctcaacgt 420
gaagaacgta tttgccttgt aaccactaat tttcagacaa agtccatgtc cagcatggtt 480
tcagatactt cctgcacttt tccttcctct gatggaattt tctggaaaca ctggattcag 540
acaaaagacg ggcaatgcgg gtctcctttg gtttcaacta gggatgggtt catagttggc 600
atacattcag catcaaattt tactaatact aataattatt ttacttctgt ccccaaaaat 660
tttatggagc ttcttactaa tcaggaggct cagcagtggg tctctggctg gaggttgaat 720
gctgactctg ttttgtgggg cggccacaaa gtgtttatgt caaaaccaga agaaccattt 780
caacccgtga aagaggccac tcaattaatg aatgagcttg tgtattctca agaaaatttg 840
tattttcaag gcgaacccca gtgctgctta gaaggtccaa aggtgaagaa cttaaacccg 900
aagaaattca gcattcatga ccaggatcac aaagtactgg tcctggactc tgggaatctc 960
atagcagttc cagataaaaa ctacatacgc ccagagatct tctttgcatt agcctcatcc 1020
ttgagctcag cctctgcgga gaaaggaagt ccgattctcc tgggggtctc taaaggggag 1080
ttttgtctct actgtgacaa ggataaagga caaagtcatc catcccttca gctgaagaag 1140
gagaaactga tgaagctggc tgcccaaaag gaatcagcac gccggccctt catcttttat 1200
agggctcagg tgggctcctg gaacatgctg gagtcggcgg ctcaccccgg atggttcatc 1260
tgcacctcct gcaattgtaa tgagcctgtt ggggtgacag ataaatttga gaacaggaaa 1320
cacattgaat tttcatttca accagtttgc aaagctgaaa tgagccccag tgaggtcagc 1380
gattga 1386
<210> 5
<211> 1263
<212> DNA
<213> 人工序列
<400> 5
atgtcctttg tgggggagaa ctcaggagtg aaaatgggct ctgaggactg ggaaaaagat 60
ggtggaggtg gatctggtgg aggtgggtca ggaggggggg gatctggtga atctcttttc 120
aagggtccta gagattacaa tccaatcagt agtacaatct gtcacttaac caacgaaagc 180
gatggtcata caacaagtct ctacggtatc ggatttggtc cattcattat taccaacaag 240
catctcttca gaagaaacaa cggaacactc ctcgttcaat cactgcatgg agttttcaag 300
gtaaagaaca caaccacctt gcaacaacat ttgatagacg gaagagatat gattataatt 360
aggatgccaa aggacttccc gccttttcca cagaagttga agtttcgaga gcctcaacgt 420
gaagaacgta tttgccttgt aaccactaat tttcagacaa agtccatgtc cagcatggtt 480
tcagatactt cctgcacttt tccttcctct gatggaattt tctggaaaca ctggattcag 540
acaaaagacg ggcaatgcgg gtctcctttg gtttcaacta gggatgggtt catagttggc 600
atacattcag catcaaattt tactaatact aataattatt ttacttctgt ccccaaaaat 660
tttatggagc ttcttactaa tcaggaggct cagcagtggg tctctggctg gaggttgaat 720
gctgactctg ttttgtgggg cggccacaaa gtgtttatgt caaaaccaga agaaccattt 780
caacccgtga aagaggccac tcaattaatg aatgagcttg tgtattctca agaaaatttg 840
tattttcaag gcgaacccca gtgctgctta gagatcttct ttgcattagc ctcatccttg 900
agctcagcct ctgcggagaa aggaagtccg attctcctgg gggtctctaa aggggagttt 960
tgtctctact gtgacaagga taaaggacaa agtcatccat cccttcagct gaagaaggag 1020
aaactgatga agctggctgc ccaaaaggaa tcagcacgcc ggcccttcat cttttatagg 1080
gctcaggtgg gctcctggaa catgctggag tcggcggctc accccggatg gttcatctgc 1140
acctcctgca attgtaatga gcctgttggg gtgacagata aatttgagaa caggaaacac 1200
attgaatttt catttcaacc agtttgcaaa gctgaaatga gccccagtga ggtcagcgat 1260
tga 1263
<210> 6
<211> 456
<212> PRT
<213> 人工序列
<400> 6
Met Ser Gly Cys Asp Arg Arg Glu Thr Glu Thr Lys Gly Lys Asn Ser
1 5 10 15
Phe Lys Lys Arg Leu Arg Gly Pro Lys Val Gly Gly Gly Gly Ser Gly
20 25 30
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Glu Ser Leu Phe Lys Gly
35 40 45
Pro Arg Asp Tyr Asn Pro Ile Ser Ser Thr Ile Cys His Leu Thr Asn
50 55 60
Glu Ser Asp Gly His Thr Thr Ser Leu Tyr Gly Ile Gly Phe Gly Pro
65 70 75 80
Phe Ile Ile Thr Asn Lys His Leu Phe Arg Arg Asn Asn Gly Thr Leu
85 90 95
Leu Val Gln Ser Leu His Gly Val Phe Lys Val Lys Asn Thr Thr Thr
100 105 110
Leu Gln Gln His Leu Ile Asp Gly Arg Asp Met Ile Ile Ile Arg Met
115 120 125
Pro Lys Asp Phe Pro Pro Phe Pro Gln Lys Leu Lys Phe Arg Glu Pro
130 135 140
Gln Arg Glu Glu Arg Ile Cys Leu Val Thr Thr Asn Phe Gln Thr Lys
145 150 155 160
Ser Met Ser Ser Met Val Ser Asp Thr Ser Cys Thr Phe Pro Ser Ser
165 170 175
Asp Gly Ile Phe Trp Lys His Trp Ile Gln Thr Lys Asp Gly Gln Cys
180 185 190
Gly Ser Pro Leu Val Ser Thr Arg Asp Gly Phe Ile Val Gly Ile His
195 200 205
Ser Ala Ser Asn Phe Thr Asn Thr Asn Asn Tyr Phe Thr Ser Val Pro
210 215 220
Lys Asn Phe Met Glu Leu Leu Thr Asn Gln Glu Ala Gln Gln Trp Val
225 230 235 240
Ser Gly Trp Arg Leu Asn Ala Asp Ser Val Leu Trp Gly Gly His Lys
245 250 255
Val Phe Met Ser Lys Pro Glu Glu Pro Phe Gln Pro Val Lys Glu Ala
260 265 270
Thr Gln Leu Met Asn Glu Leu Val Tyr Ser Gln Glu Asn Leu Tyr Phe
275 280 285
Gln Gly Lys Asn Leu Asn Pro Lys Lys Phe Ser Ile His Asp Gln Asp
290 295 300
His Lys Val Leu Val Leu Asp Ser Gly Asn Leu Ile Ala Val Pro Asp
305 310 315 320
Lys Asn Tyr Ile Arg Pro Glu Ile Phe Phe Ala Leu Ala Ser Ser Leu
325 330 335
Ser Ser Ala Ser Ala Glu Lys Gly Ser Pro Ile Leu Leu Gly Val Ser
340 345 350
Lys Gly Glu Phe Cys Leu Tyr Cys Asp Lys Asp Lys Gly Gln Ser His
355 360 365
Pro Ser Leu Gln Leu Lys Lys Glu Lys Leu Met Lys Leu Ala Ala Gln
370 375 380
Lys Glu Ser Ala Arg Arg Pro Phe Ile Phe Tyr Arg Ala Gln Val Gly
385 390 395 400
Ser Trp Asn Met Leu Glu Ser Ala Ala His Pro Gly Trp Phe Ile Cys
405 410 415
Thr Ser Cys Asn Cys Asn Glu Pro Val Gly Val Thr Asp Lys Phe Glu
420 425 430
Asn Arg Lys His Ile Glu Phe Ser Phe Gln Pro Val Cys Lys Ala Glu
435 440 445
Met Ser Pro Ser Glu Val Ser Asp
450 455
<210> 7
<211> 482
<212> PRT
<213> 人工序列
<400> 7
Met Ser Phe Val Gly Glu Asn Ser Gly Val Lys Met Gly Ser Glu Asp
1 5 10 15
Trp Glu Lys Asp Glu Pro Gln Cys Cys Leu Glu Asp Pro Ala Val Ser
20 25 30
Pro Leu Glu Pro Gly Pro Ser Leu Pro Thr Met Asn Phe Gly Gly Gly
35 40 45
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Glu Ser Leu
50 55 60
Phe Lys Gly Pro Arg Asp Tyr Asn Pro Ile Ser Ser Thr Ile Cys His
65 70 75 80
Leu Thr Asn Glu Ser Asp Gly His Thr Thr Ser Leu Tyr Gly Ile Gly
85 90 95
Phe Gly Pro Phe Ile Ile Thr Asn Lys His Leu Phe Arg Arg Asn Asn
100 105 110
Gly Thr Leu Leu Val Gln Ser Leu His Gly Val Phe Lys Val Lys Asn
115 120 125
Thr Thr Thr Leu Gln Gln His Leu Ile Asp Gly Arg Asp Met Ile Ile
130 135 140
Ile Arg Met Pro Lys Asp Phe Pro Pro Phe Pro Gln Lys Leu Lys Phe
145 150 155 160
Arg Glu Pro Gln Arg Glu Glu Arg Ile Cys Leu Val Thr Thr Asn Phe
165 170 175
Gln Thr Lys Ser Met Ser Ser Met Val Ser Asp Thr Ser Cys Thr Phe
180 185 190
Pro Ser Ser Asp Gly Ile Phe Trp Lys His Trp Ile Gln Thr Lys Asp
195 200 205
Gly Gln Cys Gly Ser Pro Leu Val Ser Thr Arg Asp Gly Phe Ile Val
210 215 220
Gly Ile His Ser Ala Ser Asn Phe Thr Asn Thr Asn Asn Tyr Phe Thr
225 230 235 240
Ser Val Pro Lys Asn Phe Met Glu Leu Leu Thr Asn Gln Glu Ala Gln
245 250 255
Gln Trp Val Ser Gly Trp Arg Leu Asn Ala Asp Ser Val Leu Trp Gly
260 265 270
Gly His Lys Val Phe Met Ser Lys Pro Glu Glu Pro Phe Gln Pro Val
275 280 285
Lys Glu Ala Thr Gln Leu Met Asn Glu Leu Val Tyr Ser Gln Glu Asn
290 295 300
Leu Tyr Phe Gln Gly Val His Thr Ser Pro Lys Val Lys Asn Leu Asn
305 310 315 320
Pro Lys Lys Phe Ser Ile His Asp Gln Asp His Lys Val Leu Val Leu
325 330 335
Asp Ser Gly Asn Leu Ile Ala Val Pro Asp Lys Asn Tyr Ile Arg Pro
340 345 350
Glu Ile Phe Phe Ala Leu Ala Ser Ser Leu Ser Ser Ala Ser Ala Glu
355 360 365
Lys Gly Ser Pro Ile Leu Leu Gly Val Ser Lys Gly Glu Phe Cys Leu
370 375 380
Tyr Cys Asp Lys Asp Lys Gly Gln Ser His Pro Ser Leu Gln Leu Lys
385 390 395 400
Lys Glu Lys Leu Met Lys Leu Ala Ala Gln Lys Glu Ser Ala Arg Arg
405 410 415
Pro Phe Ile Phe Tyr Arg Ala Gln Val Gly Ser Trp Asn Met Leu Glu
420 425 430
Ser Ala Ala His Pro Gly Trp Phe Ile Cys Thr Ser Cys Asn Cys Asn
435 440 445
Glu Pro Val Gly Val Thr Asp Lys Phe Glu Asn Arg Lys His Ile Glu
450 455 460
Phe Ser Phe Gln Pro Val Cys Lys Ala Glu Met Ser Pro Ser Glu Val
465 470 475 480
Ser Asp
<210> 8
<211> 442
<212> PRT
<213> 人工序列
<400> 8
Met Ser Phe Val Gly Glu Asn Ser Gly Val Lys Met Gly Ser Glu Asp
1 5 10 15
Trp Glu Lys Asp Glu Pro Gln Cys Cys Leu Glu Asp Pro Ala Val Ser
20 25 30
Pro Leu Glu Pro Gly Pro Ser Leu Pro Thr Met Asn Phe Gly Gly Gly
35 40 45
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Glu Ser Leu
50 55 60
Phe Lys Gly Pro Arg Asp Tyr Asn Pro Ile Ser Ser Thr Ile Cys His
65 70 75 80
Leu Thr Asn Glu Ser Asp Gly His Thr Thr Ser Leu Tyr Gly Ile Gly
85 90 95
Phe Gly Pro Phe Ile Ile Thr Asn Lys His Leu Phe Arg Arg Asn Asn
100 105 110
Gly Thr Leu Leu Val Gln Ser Leu His Gly Val Phe Lys Val Lys Asn
115 120 125
Thr Thr Thr Leu Gln Gln His Leu Ile Asp Gly Arg Asp Met Ile Ile
130 135 140
Ile Arg Met Pro Lys Asp Phe Pro Pro Phe Pro Gln Lys Leu Lys Phe
145 150 155 160
Arg Glu Pro Gln Arg Glu Glu Arg Ile Cys Leu Val Thr Thr Asn Phe
165 170 175
Gln Thr Lys Ser Met Ser Ser Met Val Ser Asp Thr Ser Cys Thr Phe
180 185 190
Pro Ser Ser Asp Gly Ile Phe Trp Lys His Trp Ile Gln Thr Lys Asp
195 200 205
Gly Gln Cys Gly Ser Pro Leu Val Ser Thr Arg Asp Gly Phe Ile Val
210 215 220
Gly Ile His Ser Ala Ser Asn Phe Thr Asn Thr Asn Asn Tyr Phe Thr
225 230 235 240
Ser Val Pro Lys Asn Phe Met Glu Leu Leu Thr Asn Gln Glu Ala Gln
245 250 255
Gln Trp Val Ser Gly Trp Arg Leu Asn Ala Asp Ser Val Leu Trp Gly
260 265 270
Gly His Lys Val Phe Met Ser Lys Pro Glu Glu Pro Phe Gln Pro Val
275 280 285
Lys Glu Ala Thr Gln Leu Met Asn Glu Leu Val Tyr Ser Gln Glu Asn
290 295 300
Leu Tyr Phe Gln Gly Val His Thr Glu Ile Phe Phe Ala Leu Ala Ser
305 310 315 320
Ser Leu Ser Ser Ala Ser Ala Glu Lys Gly Ser Pro Ile Leu Leu Gly
325 330 335
Val Ser Lys Gly Glu Phe Cys Leu Tyr Cys Asp Lys Asp Lys Gly Gln
340 345 350
Ser His Pro Ser Leu Gln Leu Lys Lys Glu Lys Leu Met Lys Leu Ala
355 360 365
Ala Gln Lys Glu Ser Ala Arg Arg Pro Phe Ile Phe Tyr Arg Ala Gln
370 375 380
Val Gly Ser Trp Asn Met Leu Glu Ser Ala Ala His Pro Gly Trp Phe
385 390 395 400
Ile Cys Thr Ser Cys Asn Cys Asn Glu Pro Val Gly Val Thr Asp Lys
405 410 415
Phe Glu Asn Arg Lys His Ile Glu Phe Ser Phe Gln Pro Val Cys Lys
420 425 430
Ala Glu Met Ser Pro Ser Glu Val Ser Asp
435 440
<210> 9
<211> 461
<212> PRT
<213> 人工序列
<400> 9
Met Ser Phe Val Gly Glu Asn Ser Gly Val Lys Met Gly Ser Glu Asp
1 5 10 15
Trp Glu Lys Asp Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
20 25 30
Gly Gly Ser Gly Glu Ser Leu Phe Lys Gly Pro Arg Asp Tyr Asn Pro
35 40 45
Ile Ser Ser Thr Ile Cys His Leu Thr Asn Glu Ser Asp Gly His Thr
50 55 60
Thr Ser Leu Tyr Gly Ile Gly Phe Gly Pro Phe Ile Ile Thr Asn Lys
65 70 75 80
His Leu Phe Arg Arg Asn Asn Gly Thr Leu Leu Val Gln Ser Leu His
85 90 95
Gly Val Phe Lys Val Lys Asn Thr Thr Thr Leu Gln Gln His Leu Ile
100 105 110
Asp Gly Arg Asp Met Ile Ile Ile Arg Met Pro Lys Asp Phe Pro Pro
115 120 125
Phe Pro Gln Lys Leu Lys Phe Arg Glu Pro Gln Arg Glu Glu Arg Ile
130 135 140
Cys Leu Val Thr Thr Asn Phe Gln Thr Lys Ser Met Ser Ser Met Val
145 150 155 160
Ser Asp Thr Ser Cys Thr Phe Pro Ser Ser Asp Gly Ile Phe Trp Lys
165 170 175
His Trp Ile Gln Thr Lys Asp Gly Gln Cys Gly Ser Pro Leu Val Ser
180 185 190
Thr Arg Asp Gly Phe Ile Val Gly Ile His Ser Ala Ser Asn Phe Thr
195 200 205
Asn Thr Asn Asn Tyr Phe Thr Ser Val Pro Lys Asn Phe Met Glu Leu
210 215 220
Leu Thr Asn Gln Glu Ala Gln Gln Trp Val Ser Gly Trp Arg Leu Asn
225 230 235 240
Ala Asp Ser Val Leu Trp Gly Gly His Lys Val Phe Met Ser Lys Pro
245 250 255
Glu Glu Pro Phe Gln Pro Val Lys Glu Ala Thr Gln Leu Met Asn Glu
260 265 270
Leu Val Tyr Ser Gln Glu Asn Leu Tyr Phe Gln Gly Glu Pro Gln Cys
275 280 285
Cys Leu Glu Gly Pro Lys Val Lys Asn Leu Asn Pro Lys Lys Phe Ser
290 295 300
Ile His Asp Gln Asp His Lys Val Leu Val Leu Asp Ser Gly Asn Leu
305 310 315 320
Ile Ala Val Pro Asp Lys Asn Tyr Ile Arg Pro Glu Ile Phe Phe Ala
325 330 335
Leu Ala Ser Ser Leu Ser Ser Ala Ser Ala Glu Lys Gly Ser Pro Ile
340 345 350
Leu Leu Gly Val Ser Lys Gly Glu Phe Cys Leu Tyr Cys Asp Lys Asp
355 360 365
Lys Gly Gln Ser His Pro Ser Leu Gln Leu Lys Lys Glu Lys Leu Met
370 375 380
Lys Leu Ala Ala Gln Lys Glu Ser Ala Arg Arg Pro Phe Ile Phe Tyr
385 390 395 400
Arg Ala Gln Val Gly Ser Trp Asn Met Leu Glu Ser Ala Ala His Pro
405 410 415
Gly Trp Phe Ile Cys Thr Ser Cys Asn Cys Asn Glu Pro Val Gly Val
420 425 430
Thr Asp Lys Phe Glu Asn Arg Lys His Ile Glu Phe Ser Phe Gln Pro
435 440 445
Val Cys Lys Ala Glu Met Ser Pro Ser Glu Val Ser Asp
450 455 460
<210> 10
<211> 421
<212> PRT
<213> 人工序列
<400> 10
Met Ser Phe Val Gly Glu Asn Ser Gly Val Lys Met Gly Ser Glu Asp
1 5 10 15
Trp Glu Lys Asp Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
20 25 30
Gly Gly Ser Gly Glu Ser Leu Phe Lys Gly Pro Arg Asp Tyr Asn Pro
35 40 45
Ile Ser Ser Thr Ile Cys His Leu Thr Asn Glu Ser Asp Gly His Thr
50 55 60
Thr Ser Leu Tyr Gly Ile Gly Phe Gly Pro Phe Ile Ile Thr Asn Lys
65 70 75 80
His Leu Phe Arg Arg Asn Asn Gly Thr Leu Leu Val Gln Ser Leu His
85 90 95
Gly Val Phe Lys Val Lys Asn Thr Thr Thr Leu Gln Gln His Leu Ile
100 105 110
Asp Gly Arg Asp Met Ile Ile Ile Arg Met Pro Lys Asp Phe Pro Pro
115 120 125
Phe Pro Gln Lys Leu Lys Phe Arg Glu Pro Gln Arg Glu Glu Arg Ile
130 135 140
Cys Leu Val Thr Thr Asn Phe Gln Thr Lys Ser Met Ser Ser Met Val
145 150 155 160
Ser Asp Thr Ser Cys Thr Phe Pro Ser Ser Asp Gly Ile Phe Trp Lys
165 170 175
His Trp Ile Gln Thr Lys Asp Gly Gln Cys Gly Ser Pro Leu Val Ser
180 185 190
Thr Arg Asp Gly Phe Ile Val Gly Ile His Ser Ala Ser Asn Phe Thr
195 200 205
Asn Thr Asn Asn Tyr Phe Thr Ser Val Pro Lys Asn Phe Met Glu Leu
210 215 220
Leu Thr Asn Gln Glu Ala Gln Gln Trp Val Ser Gly Trp Arg Leu Asn
225 230 235 240
Ala Asp Ser Val Leu Trp Gly Gly His Lys Val Phe Met Ser Lys Pro
245 250 255
Glu Glu Pro Phe Gln Pro Val Lys Glu Ala Thr Gln Leu Met Asn Glu
260 265 270
Leu Val Tyr Ser Gln Glu Asn Leu Tyr Phe Gln Gly Glu Pro Gln Cys
275 280 285
Cys Leu Glu Glu Ile Phe Phe Ala Leu Ala Ser Ser Leu Ser Ser Ala
290 295 300
Ser Ala Glu Lys Gly Ser Pro Ile Leu Leu Gly Val Ser Lys Gly Glu
305 310 315 320
Phe Cys Leu Tyr Cys Asp Lys Asp Lys Gly Gln Ser His Pro Ser Leu
325 330 335
Gln Leu Lys Lys Glu Lys Leu Met Lys Leu Ala Ala Gln Lys Glu Ser
340 345 350
Ala Arg Arg Pro Phe Ile Phe Tyr Arg Ala Gln Val Gly Ser Trp Asn
355 360 365
Met Leu Glu Ser Ala Ala His Pro Gly Trp Phe Ile Cys Thr Ser Cys
370 375 380
Asn Cys Asn Glu Pro Val Gly Val Thr Asp Lys Phe Glu Asn Arg Lys
385 390 395 400
His Ile Glu Phe Ser Phe Gln Pro Val Cys Lys Ala Glu Met Ser Pro
405 410 415
Ser Glu Val Ser Asp
420
<210> 11
<211> 15
<212> PRT
<213> 人工序列
<400> 11
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
- 还没有人留言评论。精彩留言会获得点赞!