一种改进的阿尔茨海默病发病风险预测方法与流程
本发明涉及医疗检测领域,特别涉及一种改进的阿尔茨海默病发病风险预测方法。
背景技术:
阿尔茨海默病是一种神经系统退行性疾病,临床上以记忆力减退、认知能力下降等痴呆表现为特征。现代科学认为阿尔茨海默病是基因和环境因素共同作用的结果,其中基因起着主要作用。
目前,阿尔茨海默病患者的比例逐年上升,严重影响着人们的日常生活。近年来,全基因组关联研究和候选基因研究发现了大量阿尔茨海默病易感多态位点。因此,通过阿尔茨海默病个体和正常对照个体的基因型数据建立相应的模型,进而预测个体的阿尔茨海默病发病风险就显的十分重要。
如果测定一个人的基因型数据,利用模型就可以计算出阿尔茨海默病发病风险的大小。若发病风险较高,则需要制定健康的生活、运动及营养平衡方案,从而降低发病风险。
遗传风险评分(Genetic risk score,GRS)作为分析单核苷酸多态性(Single nucleotide polymorphisms,SNP)与复杂疾病临床表型的有效方法。单个SNP对疾病具有微弱效应,该方法整合了若干个SNPs的微弱效应。GRS认为每个风险等位基因对疾病的作用相同,只是把风险等位基因的个数简单相加。实际上,每个风险等位基因对疾病的作用不可能相同,于是诞生了加权的遗传风险评分(wGRS)。
加权的GRS可以表示为:(βi表示第i个SNP的权重,Si表示第i个SNP的风险等位基因的个数,n为SNPs的数量)。该算法认为每个风险等位基因对疾病的影响不同,通过给每个风险等位基因赋予相应的权重来表明SNPs对疾病的影响程度,wGRS较GRS更广泛应用在复杂疾病的预测评估中。
目前研究表明,SNP之间的相互作用对阿尔茨海默病发病有重要影响,而wGRS进行风险预测时忽略了SNP之间的相互作用。
技术实现要素:
本发明的目的是克服上述现有技术中存在的问题,提供一种改进的阿尔茨海默病发病风险预测方法,该方法基于阿尔茨海默病(Alzheimer disease,AD)疾病个体和正常个体的基因型数据,建立一个更准确的阿尔茨海默病风险预测模型,利用该模型及个体的基因型数据预测阿尔茨海默病的发病风险。
本发明的技术方案是:一种改进的阿尔茨海默病发病风险预测方法,包括如下步骤:
(1)获取阿尔茨海默病疾病个体和正常对照个体的基因型数据;
对于阿尔茨海默病,首先对大量阿尔茨海默病病人和正常人的常染色体进行基因测序,得到阿尔茨海默病病人和正常人的原始SNP基因型数据;对原始的SNP基因型数据进行质量控制,剔除最小等位基因频率MAF小于0.02、不满足哈迪-温伯格平衡检验、分型成功比小于75%、及位于连锁不平衡区域的SNP基因型数据;样本所对应的所有SNP的分型成功率需在75%以上,否则,再从SNP基因型数据中剔除不满足样本的基因型缺失比控制的样本;对满足条件的SNP基因型数据保留下来,用于进一步分析;
(2)剔除不满足控制条件的SNP基因型数据后,对保留的SNP基因型数据进行评分;根据SNP基因型数据中含有的高风险等位基因的个数,对于SNP基因型数据进行0,1,2评分,采用0,1,2分来表示相应的SNP基因型数据;
对于SNP基因型数据,规定有两个高风险等位基因的纯合子记为2分,有一个高风险等位基因杂合子记为1分,有两个低风险等位基因的纯合子记为0分;
(3)与阿尔茨海默病关联性水平p<0.05的SNP被认为是与该病显著相关;筛选出与阿尔茨海默病显著相关的SNP以及SNP之间的相互作用对疾病有显著相关的SNP-SNP对;
将患阿尔茨海默病病人用1表示,正常人用0表示;通过单因素logistic回归算法得到校正年龄、性别后与阿尔茨海默病显著相关的SNP,同时利用Lasso多重回归方法得到Bonferroni校正后与阿尔茨海默病显著有关的SNP-SNP对;
(4)得到阿尔茨海默病独立影响的SNP以及SNP之间的相互作用对疾病有独立影响的SNP-SNP对;
比值比OR值表示疾病与暴露之间关联强度的指标,与相对危险度(RR)类似,指暴露者的疾病危险性为非暴露者的倍数;对显著相关的SNP和SNP对进行多因素logistic回归算法分析,得到对阿尔茨海默病独立影响的SNP、SNP-SNP对、相应的比值比OR值、95%置信区间以及logistic回归的常量项α,对每一个SNP和SNP-SNP对的比值比OR值取自然对数,得到每个SNP和SNP-SNP对的权重值β;
(5)利用阿尔茨海默病独立影响的SNP以及SNP-SNP对,建立改进的的wGRS模型;把每个SNP及SNP-SNP对都当作变量S,根据得到的每个SNP和SNP-SNP对的权重值β,于是改进的wGRS模型表示为各变量与自己的权重乘积之和,即其中n为SNP及SNP-SNP对的数量,βi表示第i个变量的权重值,Si表示第i个变量;对阿尔茨海默病独立影响的SNP及SNP-SNP对的OR值取自然对数得到相对应的权重βi;把阿尔茨海默病独立影响的SNP及SNP-SNP对全部纳入wGRS模型,则得到阿尔茨海默病发病风险的模型为logit P(D=1|G)=α+wGRS,其中D=1表示一个人患病,G表示一个人的SNP基因数据,P(D=1|G)为一个人根据自己SNP基因数据计算出可能患阿尔茨海默病的概率,α为logistic回归的常量项;其中n为SNP及SNP-SNP对的数量,βi表示第i个变量的权重值,Si表示第i个变量;
(6)阿尔茨海默病风险预测;
对一个人进行阿尔茨海默病风险预测,只需测定该人的基因型数据,利用步骤(5)中的阿尔茨海默病发病风险的模型就可以计算出该人患阿尔茨海默病的风险大小。
较佳地,步骤(1)中所述的对原始的SNP基因型数据进行质量控制,包括如下具体步骤:
1)对原始的SNP基因型数据,剔除最小等位基因频率MAF小于0.02的SNP;
2)剔除不满足哈迪-温伯格平衡检验的SNP;
3)对于某个SNP在所有样本中的分型成功率需控制在75%以上;剔除不满足SNP分型成功比例控制的SNP;
4)对于基因组范围内关联分析而言,对于一个需要检验的样本。一般情况下,样本所对应的所有SNP的分型成功率需控制在75%以上,对样本的SNP基因型数据质量控制时,从分析数据中剔除不满足样本的基因型缺失比控制的样本;
5)剔除位于连锁不平衡区域的SNP;留下的SNP基因型数据进行下一步分析。
较佳地,步骤(3)具体包括如下步骤:
(3)对SNP基因型数据进行评分完成后,将每个样本的SNP基因型通过0,1,2表示;在进行单因素logistic回归分析时,将单个SNP作为自变量,样本的患病状态0,1作为因变量,同时把年龄、性别当作协变量;则得到该SNP与阿尔茨海默病关联性水平、比值比以及95%置信区间;若SNP与阿尔茨海默病关联性水平p<0.05的SNP被认为是与该病显著相关,则保留下来;
(4)利用Lasso多重回归方法得到Bonferroni校正后与阿尔茨海默病显著有关的SNP-SNP对。
较佳地,步骤(4)具体包括如下步骤:
1)对显著相关的SNP和SNP-SNP对进行多因素logistic回归算法分析时,显著相关的SNP基因型数据通过0,1,2表示,而显著相关的SNP-SNP对通过两个SNP基因型数据的乘积表示,并把每个显著相关的SNP和SNP-SNP对都看作一个变量;通过多因素logistic回归算法,得到每个变量与阿尔茨海默病关联性水平p值、比值比OR值、95%置信区间以及logistic回归的常量项α;认为关联性水平p<0.05的变量是对阿尔茨海默病独立影响的变量;
2)对每一个SNP和SNP-SNP对的比值比OR值取自然对数,得到每个SNP和SNP-SNP对的权重值β,即每个SNP和SNP-SNP对都有对应自己的权重值β。
本发明的有益效果:本发明实施例中,提供一种改进的阿尔茨海默病发病风险预测方法,该方法基于现有的wGRS提出了改进的wGRS方法,计算wGRS时不但考虑了单个SNP的作用,同时也考虑SNP之间的相互作用。该改进的wGRS方法能够对阿尔茨海默病发病风险预测的正确性进一步提高。因此本方法考虑到SNP之间的相互作用对阿尔茨海默病的重要影响,并将SNP之间的相互作用应用到阿尔茨海默病发病风险预测中,进一步提高了阿尔茨海默病发病风险预测的正确率。
附图说明
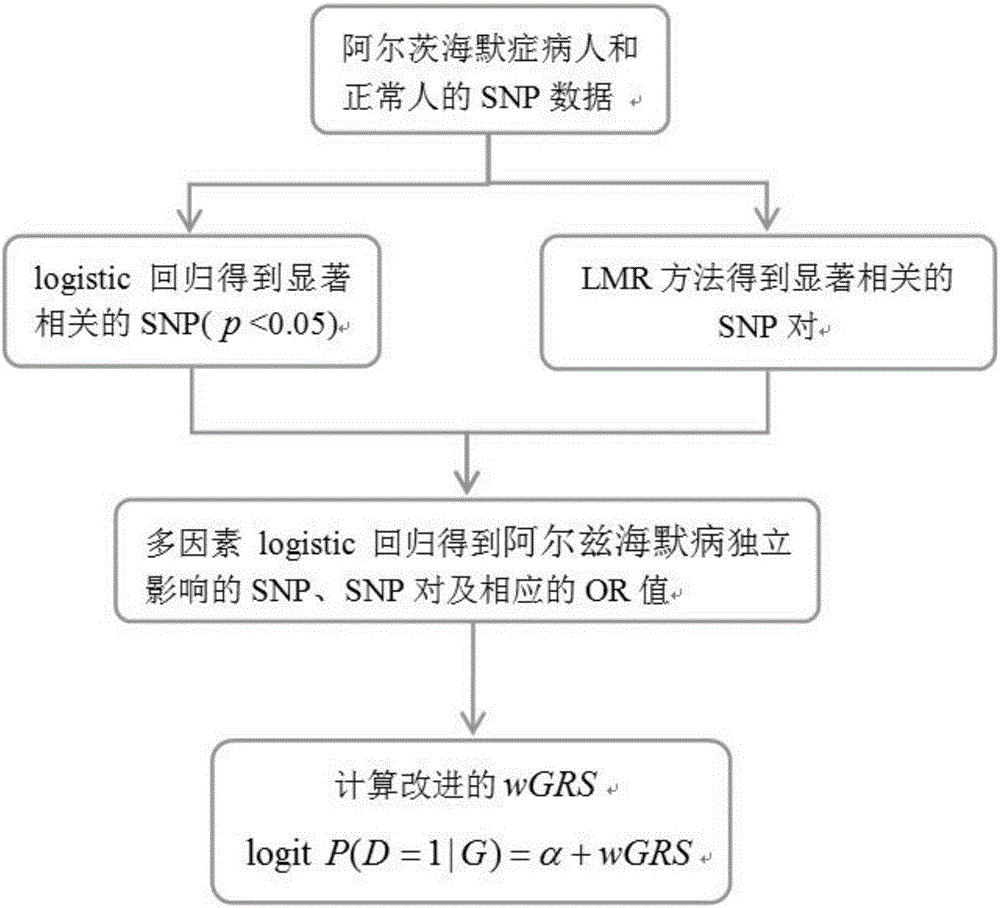
图1为本发明的方法流程图;
图2为对原始样本预测ROC曲线图。
具体实施方式
下面结合附图,对本发明的一个具体实施方式进行详细描述,但应当理解本发明的保护范围并不受具体实施方式的限制。
如图1所示,本发明实施例提供了一种改进的阿尔茨海默病发病风险预测方法,本发明采用基因型数据预测阿尔茨海默病风险时,应用SNP之间的相互作用关系对进行阿尔茨海默病发病风险预测;本发明的目的是利用阿尔茨海默病疾病个体和正常对照个体的基因型数据训练得到一个阿尔茨海默病风险模型,然后利用该模型及待测个体的基因型数据对阿尔茨海默病风险进行预测。本发明方法包括如下步骤:
(1)获取阿尔茨海默病疾病个体和正常对照个体的基因型数据;
对于阿尔茨海默病,首先对大量阿尔茨海默病病人和正常人的常染色体进行基因测序,得到阿尔茨海默病病人和正常人的原始SNP基因型数据;对原始的SNP基因型数据进行质量控制,剔除最小等位基因频率MAF小于0.02、不满足哈迪-温伯格平衡检验、分型成功比小于75%、及位于连锁不平衡区域的SNP基因型数据;样本所对应的所有SNP的分型成功率需在75%以上,否则,再从SNP基因型数据中剔除不满足样本的基因型缺失比控制的样本;对满足条件的SNP基因型数据保留下来,用于进一步分析;
所述的对原始的SNP基因型数据进行质量控制,包括如下具体步骤:
1)在关联研究中,较小的MAF将会使统计效能降低,从而造成假阴性的结果。对原始的SNP基因型数据,剔除最小等位基因频率MAF小于0.02的SNP;
2)在理想状态下,各等位基因的频率和等位基因的基因型频率在遗传中是稳定不变的,即保持着基因平衡。通常哈迪-温伯格平衡检验显著性水平p值取1×10-6。对原始的SNP基因型数据质量控制,剔除不满足哈迪-温伯格平衡检验的SNP;
3)一般情况下,对于某个SNP在所有样本中的分型成功率需控制在75%以上,否则不能通过质量控制;剔除不满足SNP分型成功比例控制的SNP;
4)对于基因组范围内关联分析而言,对于一个需要检验的样本。一般情况下,样本所对应的所有SNP的分型成功率需控制在75%以上,否则不能通过质量控制,对样本的SNP基因型数据质量控制时,从分析数据中剔除不满足样本的基因型缺失比控制的样本;
5)对原始的SNP基因型数据质量控制时,剔除位于连锁不平衡区域的SNP;经过质量控制后,留下的SNP基因型数据进行下一步分析。
(2)剔除不满足控制条件的SNP基因型数据后,对保留的SNP基因型数据进行评分;根据SNP基因型数据中含有的高风险等位基因的个数,对于SNP基因型数据进行0,1,2评分,采用0,1,2分来表示相应的SNP基因型数据;
对于SNP基因型数据,规定有两个高风险等位基因的纯合子记为2分,有一个高风险等位基因杂合子记为1分,有两个低风险等位基因的纯合子记为0分;
(3)与阿尔茨海默病关联性水平p<0.05的SNP被认为是与该病显著相关;筛选出与阿尔茨海默病显著相关的SNP以及SNP之间的相互作用对疾病有显著相关的SNP-SNP对;
将患阿尔茨海默病病人用1表示,正常人用0表示;通过单因素logistic回归算法得到校正年龄、性别后与阿尔茨海默病显著相关的SNP,同时利用Lasso多重回归方法得到Bonferroni校正后与阿尔茨海默病显著有关的SNP-SNP对;
本步骤(3)具体包括:
a)对SNP基因型数据进行评分完成后,将每个样本的SNP基因型通过0,1,2表示;在进行单因素logistic回归分析时,将单个SNP作为自变量,样本的患病状态0,1作为因变量,同时把年龄、性别当作协变量;则得到该SNP与阿尔茨海默病关联性水平、比值比以及95%置信区间;若SNP与阿尔茨海默病关联性水平p<0.05的SNP被认为是与该病显著相关,则保留下来;
b)利用Lasso多重回归方法得到Bonferroni校正后与阿尔茨海默病显著有关的SNP-SNP对。
(4)得到阿尔茨海默病独立影响的SNP以及SNP之间的相互作用对疾病有独立影响的SNP-SNP对;
比值比OR值表示疾病与暴露之间关联强度的指标,与相对危险度(RR)类似,指暴露者的疾病危险性为非暴露者的倍数;对显著相关的SNP和SNP对进行多因素logistic回归算法分析,得到对阿尔茨海默病独立影响的SNP、SNP-SNP对、相应的比值比OR值、95%置信区间以及logistic回归的常量项α,对每一个SNP和SNP-SNP对的比值比OR值取自然对数,得到每个SNP和SNP-SNP对的权重值β;
本步骤(4)具体包括如下步骤:
1)对显著相关的SNP和SNP-SNP对进行多因素logistic回归算法分析时,显著相关的SNP基因型数据通过0,1,2表示,而显著相关的SNP-SNP对通过两个SNP基因型数据的乘积表示,并把每个显著相关的SNP和SNP-SNP对都看作一个变量;通过多因素logistic回归算法,得到每个变量与阿尔茨海默病关联性水平p值、比值比OR值、95%置信区间以及logistic回归的常量项α;认为关联性水平p<0.05的变量是对阿尔茨海默病独立影响的变量;
2)对每一个SNP和SNP-SNP对的比值比OR值取自然对数,得到每个SNP和SNP-SNP对的权重值β,即每个SNP和SNP-SNP对都有对应自己的权重值β。
(5)利用阿尔茨海默病独立影响的SNP以及SNP-SNP对,建立改进的的wGRS模型;把每个SNP及SNP-SNP对都当作变量S,根据得到的每个SNP和SNP-SNP对的权重值β,于是改进的wGRS模型表示为各变量与自己的权重乘积之和,即其中n为SNP及SNP-SNP对的数量,βi表示第i个变量的权重值,Si表示第i个变量;对阿尔茨海默病独立影响的SNP及SNP-SNP对的OR值取自然对数得到相对应的权重βi;把阿尔茨海默病独立影响的SNP及SNP-SNP对全部纳入wGRS模型,则得到阿尔茨海默病发病风险的模型为logit P(D=1|G)=α+wGRS,其中D=1表示一个人患病,G表示一个人的SNP基因数据,P(D=1|G)为一个人根据自己SNP基因数据计算出可能患阿尔茨海默病的概率,α为logistic回归的常量项;其中n为SNP及SNP-SNP对的数量,βi表示第i个变量的权重值,Si表示第i个变量;
(6)阿尔茨海默病风险预测;
对一个人进行阿尔茨海默病风险预测,只需测定该人的基因型数据,利用步骤(5)中的阿尔茨海默病发病风险的模型就可以计算出该人患阿尔茨海默病的风险大小。
本发明从从如下网页:(http://journals.plos.org/plosone/article/asset?unique&id=info:doi/10.1371/journal.pone.0144898.s002)所提供的文献下载中国人群229阿尔茨海默病个体和318正常个体的55个阿尔茨海默病SNP的基因型数据,剔除一个不满足的哈迪-温伯格平衡的SNP。对所有的基因型数据按照高风险等位基因的个数进行0,1,2转换,通过单因素logistic回归分析可以得到与阿尔茨海默病显著相关的SNP。由于基因型数据中没有年龄、性别等信息,所以直接引用原作者校正年龄、性别等信息后13个与阿尔茨海默病显著相关的SNP。详细信息如表1所示:
表1 与AD疾病显著相关的13个SNP
利用LMR方法找出与阿尔茨海默病显著相关的SNP对,结果表明rs6656401-rs3865444、rs28834970-rs6656401、rs28834970-rs3865444与AD显著相关(p<0.05)。
对13个显著相关的SNP、3对SNP进行多因素logistic回归,得到独立影响阿尔茨海默病的SNP和SNP对(p<0.05)及对应的OR值和95%的置信区间(未校正年龄、性别等信息),对OR值取自然对数可以得到相应的权重β。表2是独立影响AD的SNP和SNP对。
表2 独立影响AD的SNP和SNP对
于是,利用独立影响阿尔茨海默病的SNP和SNP对计算改进的wGRS,wGRS=V1*(-0.456)+V2*0.339+V3*(-0.464)+V4*0.374+V5*(-0.754)+V6*0.367+V7*0.667+V8*(-0.308)+V9*(-0.398)+V10*1.664则阿尔茨海默病疾病的模型为logit P(D=1|G)=0.772+wGRS。
为了检验该模型的预测正确率,我们利用改进的wGRS对原始样本(229阿尔茨海默病个体和318正常对照个体)进行预测分析,预测结果如表3:
表3 改进的wGRS对原始样本预测情况表(分类点为0.5)
相应的ROC曲线如图2所示。
ROC曲线的面积为0.721,95%CI为(0.679-0.764)。
如果不考虑SNP之间的相互作用对疾病的影响,直接采用13个显著的SNP,建立wGRS对原始样本进行预测,得到如表4的结果分析:
表4 wGRS对原始样本预测情况表(分类点为0.5)
因此,将阿尔茨海默病显著相关的SNP和SNP对作为影响疾病的因素,通过多因素logistic回归得到独立影响阿尔茨海默病的SNP、SNP对及对应的OR值。采用改进的wGRS对阿尔茨海默病风险预测的正确率为68.7%。只用阿尔茨海默病显著相关的SNP而不考虑SNP之间的相互作用对阿尔茨海默病风险预测的正确率为66.4%。本发明提出改进的wGRS方法充分考虑到SNP之间的相互作用对阿尔茨海默病发病的影响,能够将阿尔茨海默病发病风险预测正确率提高2.3%。如果在进行多因素logistic回归得到独立影响阿尔茨海默病的SNP和SNP对时对年龄、性别等信息进行校正,相信改进的wGRS对阿尔茨海默病风险预测的正确率将会更高。
综上所述,本发明实施例提供的一种改进的阿尔茨海默病发病风险预测方法,该方法基于现有的wGRS提出了改进的wGRS方法,计算wGRS时不但考虑了单个SNP的作用,同时也考虑SNP之间的相互作用。该改进的wGRS方法能够对阿尔茨海默病发病风险预测的正确性进一步提高。因此本方法考虑到SNP之间的相互作用对阿尔茨海默病的重要影响,并将SNP之间的相互作用应用到阿尔茨海默病发病风险预测中,进一步提高了阿尔茨海默病发病风险预测的正确率。
以上公开的仅为本发明的几个具体实施例,但是,本发明实施例并非局限于此,任何本领域的技术人员能思之的变化都应落入本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!