一种稻瘟病抗性基因Pi57及其编码蛋白与应用的制作方法
本发明涉及植物基因工程技术领域,具体涉及稻瘟病抗性基因及其编码蛋白与应用。
背景技术:
作物病害是引起作物产量损失的主要因子。随着世界人口的不断增长,改良作物病害的抗性水平以增加粮食产量、保障粮食安全显得尤为的重要。水稻是世界最重要的粮食作物之一,全球有一半以上的人口以稻米作为主食。由真菌(magnaportheoryzae)引起的稻瘟病是世界水稻生产区为害最为严重的病害之一,全球每年因稻瘟病危害所造成的损失超过70亿美元,发病田块产量造成10-30%的损失,严重的田块甚至绝收。实践证明,选育和推广水稻抗病品种是水稻生产上控制该病害最为经济、有效和环境友好的措施,而抗病基因的发掘、研究与利用是有效开展抗病育种的基础和核心。
通过广泛的遗传分析,目前已从稻属的栽培稻及野生稻中鉴定、定位100多个稻瘟病抗性基因[1]。从1999年第一个稻瘟病抗性基因成功克隆以来,目前至少已克隆了26个稻瘟病抗性基因[1-3]。通过对这些抗病基因编码蛋白的结构发现,在已克隆的这些基因中,除pid-2基因编码一个受体蛋白激酶和pi21基因一个具有重金属结合域及富含脯氨酸结构域的未知蛋白外,其它已克隆的基因均编码具有核苷酸结合位点和亮氨酸重复结构域(nbs-lrr)的蛋白[4],nbs-lrr蛋白又可再分为蛋白n端具有tir结构域或cc结构域的蛋白,即具有tir-nbs-lrr或cc-nbs-lrr结构域的蛋白。进一步研究表明,植物抗病基因编码的nbs-lrr可直接与病原菌无毒基因编码的蛋白可发生直接或间接的互作,进而引起寄主的抗性反应。如在水稻中稻瘟病抗性基因pita编码的蛋白直接与稻瘟病菌无毒基因avrpita编码的蛋白avrpita互作,从而引起抗性反应;而拟南芥中的抗病基因rpm1编码的nbs-lrr蛋白是识别被病原菌修饰的寄主蛋白rin4,进而引起寄主的抗性反应,该类识别即是间接的识别,而非抗病蛋白与病原菌无毒基因的直接互作。抗病基因的不断被克隆,特别是具有不同结构类型抗病基因的克隆,将为进一步深入研究水稻的抗病机理,寻找实现抗病持久性的途径,为实现水稻抗病品种的可持续利用奠定基础。
[1]ashikanis,rafilimy,shabanimofradm,ghasemzadeha,ravanfarsa,latifma(2016)molecularprogressonthemappingandcloningoffunctionalgenesforblastresistanceinrice(oryzasatival.):currentstatusandfutureconsiderations.critrevbiotechnol36:353-367
[2]ballinie,moreljb,drocg,pricea,courtoisb,notteghemjl,tharreaud(2008)agenome-widemeta-analysisofriceblastresistancegenesandquantitativetraitlociprovidesnewinsightsintopartialandcompleteresistance.molplantmicrobeinteract21:859–868
[3]dengy,zhaik,xiez,yangd,zhux,liuj,wangx,qinp,yangy,zhangg,liq,zhangj,wus,milazzoj,maob,wange,xieh,tharreaud,hez.epigeneticregulationofantagonisticreceptorsconfersriceblastresistancewithyieldbalance.science,2017,355:962-965.doi:10.1126/science.aai8898
[4]dangljlandjonesjd.plantpathogensandintegrateddefenceresponsestoinfection.nature,2001,411:826-833
技术实现要素:
为解决现有技术存在的常规杂交育种中由于遗传连锁造成的基因连锁累赘,现有的稻瘟病抗性基因中大部分基因均存在抗谱窄,以及在常规育种中难以获得抗病性好、其它综合农艺性状又优良的新品种的技术问题和克服抗性容易丧失的问题,本发明提供一种稻瘟病抗性基因pi57及其编码蛋白与应用。
本发明的技术方案如下:
1.一种稻瘟病抗性基因pi57,其核苷酸序列如seqidno:2所示。
2.技术方案1所述的稻瘟病抗性基因pi57的编码蛋白,其氨基酸序列如seqidno:3所示。
3.扩增技术方案1所述的稻瘟病抗性基因pi57的引物f57-sali-fw和引物f57-asci-rv,所述引物f57-sali-fw的碱基序列如seqidno:7所示,所述引物f57-asci-rv的碱基序列如seqidno:8所示。
4.技术方案1所述的稻瘟病抗性基因pi57的制备方法:以抗性供体水稻渗入系il-e1454的基因组dna为模板,用技术方案3所述的引物f57-sali-fw和引物f57-asci-rv进行pcr扩增得到稻瘟病抗性基因pi57。
5.技术方案1所述的一种稻瘟病抗性基因pi57在水稻抗稻瘟病分子育种中的应用。
6.根据技术方案5所述的应用,将稻瘟病抗性基因pi57转入到感稻瘟病的水稻中,得到具有稻瘟病抗性的水稻。
与现有技术相比,本发明的创新点和有益效果:
本发明基因pi57对采集自柬埔寨、老挝、缅甸、泰国、越南和中国的322个稻瘟病菌菌株的抗谱平均达到91%以上,具有广谱抗稻瘟病的特点,是抗病育种的优秀抗源。基因pi57来源于长雄野生稻,由于其与栽培稻的基因组差异大,根据已知水稻基因组的参考序列难以获得靶标区域的序列。本发明通过基因组步移(genomewalking)的方法,成功获得水稻渗入系il-e1454中靶标区域的未知dna序列,为后续的克隆奠定了基础。
利用本发明提供的稻瘟病抗性基因pi57转入到感病的水稻品种中,可产生新的抗病水稻品种;同时,克隆抗病基因后,基因的利用可打破传统育种方法中不同物种间基因难以转育的问题,本发明提供的抗稻瘟病基因pi57可用转化技术将其转移到水稻中,通过基因的转化,既可极大地缩短育种所需的周期,同时可避免传统育种技术中存在的靶标基因与其它控制不良性状基因的连锁累赘带来的问题。本发明通过基因转化获得的抗病植株及其后代的种子,可通过常规有性杂交的方式将基因pi57稳定遗传给后代。基因pi57具有广谱抗稻瘟病的特点,利用该基因育成的抗病新品种可大面积进行种植。
序列表中seqidno:1所示的是基因pi57的启动子的碱基序列。
序列表中seqidno:2所示的是基因pi57的核苷酸序列。
序列表中seqidno:3所示的是基因pi57的编码蛋白的氨基酸序列,它包含3个主要的结构域:跨膜结构域、arm(armadillorepeats)重复结构域和富含脯氨酸结构域,其中跨膜结构域为(29-191aa);arm重复结构域(207-811aa);富含脯氨酸结构域(817-839aa)。
序列表中seqidno:4所示的是基因pi57的cdna核苷酸序列。
序列表中seqidno:5所示的是扩增基因pi57的cdna核苷酸序列的引物pi57cdna-fw的碱基序列。
序列表中seqidno:6所示的是扩增基因pi57的cdna核苷酸序列的引物pi57cdna-rv的碱基序列。
序列表中seqidno:7所示的是扩增基因pi57的引物f57-sali-fw的碱基序列。
序列表中seqidno:8所示的是扩增基因pi57的引物f57-asci-rv的碱基序列。
序列表中seqidno:9所示的是扩增潮霉素基因分子鉴定引物hptfw的碱基序列。
序列表中seqidno:10所示的是扩增潮霉素基因分子鉴定引物hptrv的碱基序列。
序列表中seqidno:11所示的是扩增基因pi57分子鉴定引物pi57spfw的碱基序列。
序列表中seqidno:12所示的是扩增基因pi57分子鉴定引物pi57sprv的碱基序列。
附图说明
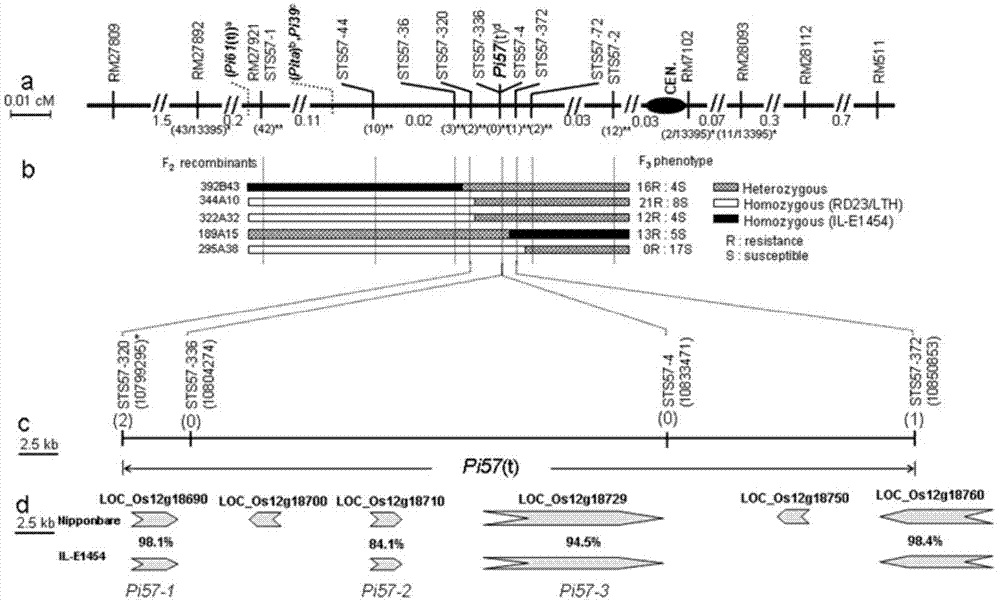
图1:水稻稻瘟病抗性基因pi57的克隆策略图。
图2:水稻转基因植株鉴定电泳图:其中泳道m为分子量标记dl2000,泳道1-17为转化pcambiaasci-pi57-3质粒的不同t0代转基因植株基因组dna为模板进行筛选标记潮霉素基因(hpt)的扩增pcr产物,泳道b为清水对照,泳道n为转化受体品种的dna扩增结果,泳道p为以含有基因pi57的pcambiaasci-pi57-3质粒作阳性对照的扩增结果。
图3:利用稻瘟病菌hn09-1c-7接种转基因阳性转化植株、转基因感病受体品种滇粳优1号和抗稻瘟病供体水稻渗入系il-e1454反应图。第1-第17条叶片是转基因t0代不同植株,djy1是转基因感病受体品种滇粳优1号的叶片,il-e1454是含有基因pi57的抗性供体水稻渗入系il-e1454的叶片。
图4:利用稻瘟病菌hn09-1c-7接种转基因t1代植株的共分离分析图。m为分子量标记dl2000,1为感病受体转化品种滇粳优1号,2为抗稻瘟病基因pi57供体水稻渗入系il-e1454,3-6为转基因pi57的t1代感病植株,7-12为转基因pi57t1代抗病植株。
具体实施方式
根据基因克隆及功能验证的实施策略,对本发明利用附图及实施过程作进一步的阐述如下,但本发明的实施方式不限于此,若未特别指明,实施过程中所采用的技术手段为本领域技术人员所熟知的常规手段。通过实施,完成了基因pi57的分离克隆、功能特征及分子鉴定,本发明中克隆的基因pi57可与适当的遗传转化载体进行组装,通过转基因的策略进行遗传转化,使感病的水稻品种在转基因pi57后获得的转基因材料具有广谱抗稻瘟病的特性。
为实施基因pi57的克隆,本实验室前期开展了如下的实验研究(参见dongetal.,2017.finemappingofpi57(t)conferringbroadspectrumresistanceagainstmagnaportheorzyaeinintrogressionlineil-e1454derivedfromoryzalongistaminata.plosone,12(10):e0186201.https://doi.org/10.1371/journal.pone.o186201):
(1)稻瘟病抗性基因pi57对稻瘟病的抗谱特性
为了明确水稻渗入系il-e1454中基因pi57对稻瘟病的抗性水平,实验室利用来源于中国、柬埔寨、老挝、缅甸、泰国和越南的322个稻瘟病菌菌株接种il-e1454,其中44个来自柬埔寨,30个来自老挝,25个来自缅甸、19个来自泰国、28个来自越南、176个来自中国(表1)。抗谱测定结果表明,基因pi57对中国的菌株抗谱达到90.91%,对柬埔寨的菌株抗谱达到95.45%,对老挝和缅甸的菌株抗谱达到100%,对泰国的菌株抗谱达到83.33%,对越南的菌株抗谱达到78.57%;对6个国家采集的322份菌株的抗谱平均达到91%以上。从这些鉴定的结果可以看出,基因pi57具有广谱抗稻瘟病的特性,可以作为抗病育种的抗源在这些地方进行利用(dongetal.,2017)。
表1稻瘟病抗性基因pi57的抗谱分析
备注:a表示分析的菌株数;b表示特定基因对所测定的稻瘟病菌菌株的抗性频率(%)
(2)抗稻瘟病基因pi57的克隆策略
前期利用来源于il-e1454/rd23和il-e1454/丽江新团黑谷的39445个f2代个体组成的定位群体,本研究室前期完成了抗稻瘟病基因pi57的定位,该基因位于水稻第12染色体短臂近着丝粒区域。利用与基因紧密连锁的标记,构建了以日本晴的基因组为参考序列的基因pi57的电子物理图谱(图1)。然后以日本晴的基因组为参考的设计引物并结合基因组步移(genomewalking)的方法,获得了抗性供体渗入系il-e1454中基因pi57所在区域的序列信息,并开展基因的预测分析(申请人尚未公布il-e1454材料靶标区域的dna序列信息),然后,根据已获得的序列信息,本发明开展了如下的工作:
实施例所涉及的生物材料水稻渗入系il-e1454和稻瘟病菌hn09-1c-7已在非专利文献:dongetal.,2017.finemappingofpi57(t)conferringbroadspectrumresistanceagainstmagnaportheorzyaeinintrogressionlineil-e1454derivedfromoryzalongistaminata.plosone,12(10):e0186201.https://doi.org/10.1371/journal.pone.o186201)公开,申请人有保存,申请人保证自本专利申请日起20年内向公众发放。申请人联系地址:云南省昆明市盘龙区北京路2238号,云南省农业科学院农业环境资源研究所,邮编:650205。
实施例中涉及的pcambia1300asci载体、水稻品种滇粳优1号等各试剂均为市售,稻瘟病菌hn09-1c-7菌株为单位保存。
实施例1通过候选基因分析确定抗稻瘟病基因pi57
一、基因pi57候选基因分析
利用softberry平台(http://www.softberry.com),对课题组获得的il-e1454在靶标区域的序列进行靶标区域基因的注释分析,发现在靶标区域含有3个预测的候选基因(pi57-1(loc_os12g18690),pi57-2(loc_os12g18710)和pi57-3(loc_os12g18729)。根据基因注释的序列信息,设计引物并采用rt-pcr分析表明,3个候选基因仅有pi57-3基因表达,其它的2个基因(pi57-1和pi57-2)均不表达,因而将基因pi57的候选基因确定为pi57-3。
二.基因pi57的全长cdna序列的获得
利用trizol试剂,从水稻渗入系il-e1454的幼叶提取总rna,并用powerscriptiitranscriptase(takara公司)逆转录合成第一链cdna。用引物pi57cdna-fw(其碱基序列如seqidno:5所示)和引物pi57cdna-rv(其碱基序列如seqidno:6所示),通过pcr扩增,获得基因pi57-3的cdna序列即基因pi57的全长cdna序列,基因pi57的cdna序列如seqidno:4所示,为3804bp。其pcr扩增反应条件如下:总反应体系50μl,含有10μl5×primestarbuffer,1μl高保真taq酶primestargxldnapolymerase,4μldntp(2.5μmeach),2μlpi57cdna-fw(10μm),2μlpi57cdna-rv(10μm),2μlcdna模板,并补足双蒸灭菌水至50μl。pcr反应条件为98℃10秒,68℃4分钟,共进行32个循环。
三.基因pi57的序列分析
利用获得的基因pi57的cdna与pi57基因的基因组dna序列比较分析表明,pi57的基因组序列含有3个内含子,其中第一个外显子为第1~1136bp;第二个外显子为第1465~1725bp;第三个外显子为第4393~6319bp;第四个外显子为第6637-7116bp。在cdna序列中,基因pi57的编码区为第1159bp~3726bp,编码区序列长度为2568bps,基因pi57编码一个由855个氨基酸组成的蛋白。
实施例2抗稻瘟病基因pi57的遗传转化互补验证实验
一、基因pi57的扩增及获得
以抗病供体亲本水稻渗入系il-e1454的基因组dna为模板,利用引物f57-sali-fw(其碱基序列如seqidno:7所示)和引物f57-asci-rv(其碱基序列如seqidno:8所示)扩增包含基因pi57自身的启动子(其核苷酸序列如seqidno:1)的序列,获得基因pi57的全长序列。pcr扩增反应条件如下:总反应体系50μl,含有10μl5×primestarbuffer,1μl高保真taq酶primestargxldnapolymerase,4μldntp(2.5μmeach),2μlpi57-sali-fw(10μm),2μlpi57-asci-rv(10μm),il-e1454的基因组dna2μl,补足双蒸灭菌水至50μl。pcr反应条件为98℃10秒,68℃12min,共进行32个循环。基因pi57的核苷酸序列如seqidno:2所示。
实施例3基因pi57的遗传转化
一、基因pi57遗传转化载体的构建及遗传转化
(1)克隆载体及基因pi57扩增产物的制备:利用限制性内切酶sali在两个1.5ml的离心管中分别酶切pcambia1300asci载体和扩增得到的基因pi57产物(按实施例2所述扩增基因pi57):sali酶切缓冲液(neb3.1)2μl、限制性内切酶sali1μl,pcambia1300asci载体5μl或基因pi57片段5μl,并加入双蒸水分别补足20μl反应液;于37℃酶切30min,然后利用回收试剂盒分别进行片段的纯化;纯化后的pcambia1300asci载体和基因pi57产物再分别用限制性内切酶asci进行酶切,两组酶切反应为:酶切缓冲液(cutsmart)2μl、asci1μl、纯化后的pcambia1300asci载体10μl或基因pi57片段10μl,均用双蒸水补足20μl;37℃酶切30min;再次纯化两组酶切反应后的产物分别获得sali和asci双酶切的pcambia1300asci载体和基因pi57的dna,并进行dna浓度的定量。
(2)连接反应及克隆:利用t4连接酶进行目标片段和载体的连接:连接缓冲液(10×t4ligationbuffer)2μl、双酶切后的pcambia1300asci载体1μl(10ng)、双酶切后的基因pi57的dna片段4μl(50ng)、t4连接酶1μl,双蒸水补足20μl;10℃下过夜连接;用无水乙醇沉淀连接产物,并电激转化到大肠杆菌dh10b中,获得阳性克隆(pcambia1300asci-pi57-3)并进行测序分析,基因pi57序列如seqidno:2所示。
(3)水稻愈伤组织的遗传转化及转基因植株的获得
采用农杆菌介导的遗传转化法(hieiy,ohtas,komarit,etal.efficienttransformationofrice(oryzasatival.)mediatedbyagrobacteriumandsequenceanalysisoftheboundariesofthet-dna.theplantjournal,1994,6:271-282)开展基因的遗传转化实验:将获得的pcambia1300asci-pi57克隆转化到农杆菌菌株eha105中,并进一步用农杆菌侵染感稻瘟病的水稻品种滇粳优1号,通过抗性愈伤组织筛选、分化获得转基因植株。
(4)转基因植株的分子鉴定及抗病表型鉴定
获得的转基因植株利用遗传转化载体pcambia1300asci上携带的潮霉素b基因序列引物进行鉴定,通过扩增转基因植株叶片提取的dna进行pcr扩增鉴定(pcr扩增反应体系和反应程序与实施例2相同)。遗传转化共获得17株转基因植株,经鉴定获得的17株t0代转基因植株全部为阳性,电泳图谱见图2,分子鉴定用引物分别为:hptfw(其碱基序列如seqidno:9所示)和hptrv(碱基序列如seqidno:10所示)。
利用对受体品种滇粳优1号致病的稻瘟病菌hn09-1c-7接种17株转基因植株,接种结果及对照的反应如图3所示。第1片-第17片叶子为转基因植株,djy1为转化受体感病品种滇粳优1号的叶片,il-e1454为含抗稻瘟病基因pi57供体水稻渗入系il-e1454的反应。接种t0反应结果发现,17株转基因植株有16株表现抗病。
经过t0代转基因植株繁殖自交后,获得t1代种子,并对获得的t1代群体进行共分离分析实验,对转基因的t1代植株进行分子及表型鉴定分析。分子鉴定用引物pi57spfw(其碱基序列如seqidno:11所示)和pi57sprv(其碱基序列如seqidno:12所示)进行,如图4所示。结果表明,含有转基因pi57的t1代植株表现抗病,而不含转基因pi57的植株表现感病,表现出共分离的特性。表明基因pi57可以通过转化的方法转入到感病品种中,并通过育种的选拔,获得纯合的抗病植株,应用于水稻抗病育种,选育抗稻瘟病的水稻品种,以解决水稻生产上水稻品种被稻瘟病危害的问题。
序列表
<110>云南省农业科学院农业环境资源研究所
<120>一种稻瘟病抗性基因pi57及其编码蛋白与应用
<160>12
<170>siposequencelisting1.0
<210>1
<211>1955
<212>dna
<213>水稻渗入系il-e1454(oryzasativa)
<400>1
ataggagaggaagagagaggctggacagagggagtacttacctgcgcatgccgcatgcac60
gatggggatgagtaccggattttttgtttgttaatatatatgggtccaccgatttaatag120
aaaatcaatggttaaaatatatgggtccaccgatttaacggaaaatcaatagctagatat180
ttattagattttccatgattttctatattttattagagtgccacgtggcgactcaagagt240
gtttataggatgacacatggcggtttaggagcatttgtaggaagtttaatggacttttag300
tatataataatagataatagaagatttattagagtgccacgtggcgactcaagagtgttt360
ataggatgacacatggcggtttaggagcatttgtaggaagtttaatggacttttagtata420
taataatagatagattttgaataagatagcagtaatatagtatatcactatatatacgtt480
attggttcataggtatgcaccattaaattaaatctacaatgtccaaaaataacaagtcat540
attggcgctttgcatggcaatcgagaattattaaaaatcatgaaaaactagtgatatgga600
ttatatcatcatttgaagatgcataaaatttggggtccattttattcagcggttaggaca660
gtacgggaataacctaaagtattcggagtatagggcatgcgattttgtttctctttaatt720
cctatttaaaacaaatggtacatgggtgtttcatactaagtttggttaattagggaacac780
aaatcatactaagcttatatatcagaattcatgcaatgaaagtatacgatataaaaataa840
gaattaaccaaacaaaattatactttaattagtccgtggtgacggaccatttacaaatga900
tccacacctacttattcagatctctttttttttttcaaaaaagagatgtgatgtgaaaaa960
aagaaaggcgcagaagtaaatgtaagtagaatatatatttaaatatatcctaaaatctat1020
aaatacatctatagtataaaagaaattttgtgaccgaaagttttaaaaaataagatatgc1080
taatattcaaaaaagatatacacatagtacaaaatgatagtctaatagtaatcaactcta1140
ttattgcgacacaatattttctcataaatttaagattgcatggttccaagtaatattagt1200
ttagaattcatatgtttcaagggtagatcaaagagtttcataaccaatatactgatataa1260
tgttcaaagcaaccccaaactaattattgacaaaaacaaatttagaagagagtcaaagaa1320
tttcaagaaaaagtaacccacacataaaacatccaagataaaaattcaaacaacatcaaa1380
taaagatgccggcgcatatgcgcgggctacaacttcctagtttcgtaaaaaaagttggac1440
aagatttatccgtagataccgctcttgagcaatggtgtcgggatcgtgattcgtgaaccg1500
tgtggactggggtttgcatcctgttccgtatacaccagatttttttttcctgatgatcac1560
atccccacttgcctgcatcgcttttgtggcctctccatagaatggttaattcacggtaaa1620
aaacgatttttttatgacaccacccttttatttttactggcggccatgacaaaacaccgc1680
ttgtaaaattatacggaaagattcaaagtcacgaacctccagcgaacggcctcttaaaga1740
agccagcaaatagcaatatttttacaagcggacctcttaagaagctactagcgaaaatcc1800
attgtttaccagcagacttcttaagaggccgtgagcaaaaaaaaaccaattttctctgtc1860
ggctgctaagaggtccaccaacaaaaaaaaaaacatcattgcactaaaaaataattgaac1920
acaataaaaaccaccacctaccgtgccaccttgcc1955
<210>2
<211>7116
<212>dna
<213>水稻渗入系il-e1454(oryzasativa)
<220>
<221>exon
<222>(1)..(1136)
<223>第一个外显子
<220>
<221>exon
<222>(1465)..(1725)
<223>第二个外显子
<220>
<221>exon
<222>(4393)..(6319)
<223>第三个外显子
<220>
<221>exon
<222>(6637)..(7116)
<223>第四个外显子
<400>2
accaccacttccctctctcctcctcatcctcatcttcatatctagatagacctcatcttc60
tccagccaccaccgcctccctccccaccggtggtggcggcgggaagacagcggctcggct120
gagcccggcgggtagggcaacgatttagtatgggaggagcagcggcgacttctctacacg180
acggcgttcggaggcggatctagcagcggcgactccatccagcggtggatccgccgcctc240
ctagtcctagctctctctcccccgctcctccctcctccccttcccccgccgccaccaccc300
accccctctccctctctcagaactggtgcgcggggagcggtggaggctgcggcggcgaag360
gctgggggggctcggcagcggcggcgacggtgagaccggtagcagtgatggtggagggga420
cggatccgatggcggtggttctcgtcagccagttcaaggatgacgacagcggcggctgcg480
gcaacgatggacaggctctgggcggctcctccaccctcctcccctctctcatatccaacg540
actcggggtggtaggacgtggccgacgcatccggcggcggagctcgggtatcaatccggc600
atgtttttttttctccaaaatcaaattttcctggtggtcgggaaacggattttcaatttt660
tttttacgctcgaaaatcttttttcgctggcggtcaacgtaacttaaccgcttgtgaaaa720
caagctttaaagtttcgctagtgaagattgttattttcactagccttttgtttgcgggcg780
gctgacagtgccaccagcgaaaaattcttttgctagtagtgattgctagatggtcagcag840
agaacttgtggagtatatgactatcaagtatcaaccaacagtacacttcagcagaaagag900
gatggttctagctctccggtggtttaatattgaaacgtacagcccacccaccaccacccc960
agtcgtccccagctaagcgctcccttgcttgcctcagtagacgagctgccaactagtagc1020
actccagcaagcagcgggtcagcggagtccggagggcgcacgtgcgccgcagcgaggtag1080
cagcgtggccaggcggcggctggctggccagccgggttcggcaagcggcttagcaggtaa1140
ttttctatcacatcgttctaattttaatcaaactactaattttagcatgaactaaacaca1200
cccttggttgcgactttgggagtcgatatataggccttgctcgtagttcgttgattttgg1260
ggttaaactggcgaggaactttacttggataccacctgggagttgatgtgattttagtgt1320
ttagtacagtttagagtttattacctaccggtttcttggtgcctggcgcacactgagcat1380
gcaaatgcagtgagagccagtatcatctaagacggctggacctgaaatattttaccatgt1440
atatatcaaactcgacttgatcaggtgtttagcggaggaaggatcgatgtccggcgccgg1500
acagagccgtggtcatcgccttggattacacattgattcggattggccagaggtcttgtt1560
gatcaatgactatgcggtgttcatggggtacctgtcgatggttgtcaccgggacggggtt1620
ccttgtgctcacttggtccaccgtcatcctcctcggtggattcgtctccatgctatccaa1680
caaggacttctggagtctcacggtgatcacgctcgttcaaacaaggtgagctatctatag1740
ccatctcaaccagacttatatttatttcgcgttcccaacattaaccagttgagccaagca1800
gttggagtttcgcattgctagctagctactagtttactgtcagatctagctttcaacact1860
tcagtgttgcgaactgaatatgctgcaaaacgttgcaactccagagtggctttatatact1920
acattgatatgaaaatattcaagtcccatatttgcaaaatcgactatttttaaaatcaat1980
tgttttctccccctttaaagaagcatgcattttttatatttctctcccctttgacaatga2040
tttcatcaagaattttaaaagagaaaatataaccaataaagcataactatcatcatttca2100
tgattgtcacatgtaataatcacttcttttcttaattttttatttttctgaatttttttt2160
gagagctttgaaatgtacctaaaacaattcatggtcatatgttgcaagacaaatcagata2220
aattcatcaactagttaacaagcatgcattaagtataaacatgaatcaaacatattacaa2280
ttaagctcaaatcaataataaagattaatgatttataatgcaacatatttacaagtacat2340
atgttgaaaaaaaatccctactggattatcactctttctcaaattaacttcatcgcaaaa2400
ataaccaagagaattttccgcaagtattacaaattttggtactcattttgacattgaata2460
taaaatatatatctcaattatttcgagtatgttcttgttgttaagactgtttctaacgat2520
tcggtactcattattttcctcactgcaaatacgtctttatcgtcaagactgtttccaagg2580
attatgtattcatttttttttctctcatcataagcaactttttattctcttgcgacattt2640
ggaacaaacaagaagcaaattgaggaataacaatccaataagcataaatcattcaaactc2700
ggcacttgctacaacattgcatatttacataaatcaagtaaaagaattagtgtcatgcat2760
catctcacttaaaaagcaatatccaagccacatgaaaaggctaaaatgcatacaagctaa2820
catataatatacctcaattattttggaagcatgcacaagaatatactagaggtatatacc2880
aaagttcttcttttctcatttgttctttatttttcagatttagagcaatgcatggatata2940
gacatgaatgcaatgaaaaagaacaaactcccctcaaataaacatccaattctccgcgaa3000
gaaaaatcaagaagaaatcaaaaaaatttgaaaaagacatccaattgccaatgatatcac3060
gaaggttcttatgaattgacataaatccacttgaattaccaaaactaccacttcaaacac3120
tagcatgaaatgctaataaacgaagaagtaaaacaaagttatgtcaactcaaaagtacat3180
ttatgttatcttgaaaagcaatcaccgtctccacattgaacgatcatggggagtatgccg3240
ccaaaatacatacgaacatgaagatgtcgaggccccgtgttggggttagtcataagaaat3300
tttagaatccaacactaagtcacatgacaaatcttaactaattaaaagattcatattttt3360
cctttcatcacctcaattttcgtccgtcccttgcttcgttcgcaagatgcgataactagg3420
actctgcttctttcgttcctcatccgattgtatttaggtagggcaatcggagtgggaaat3480
ccgccgcacggccttgccgggaagaagtaaggaattggggaagagagaagcggagtccta3540
gctatgtaaaccatatgcatgcgatatggaaagcttcaccatatttatgcgatatggaaa3600
ccttcaccatacgcacacaacaccatatgtatgcgatatggaaaccttcacctccacgtg3660
cattgttctctggcctgagcatcctgtatgatatccttgaccatccggacctcaacttct3720
tccaagcctagtcccgatccatcgccagctatactctcatagtcttaagcaacacataca3780
catgaataacaaagaaactccatatccgaatacaagttaacaccaacaatatgactcaca3840
tcagcatacaatagtatcacatacgaaaaccgactcccttctggaatcacccggtctgat3900
cggtgcctcctgcccggtctgacaggcacccaatgctgaccgcattgtttaccatccgaa3960
aatactatccaccgtattttcaacaacacttaccgttgacaattgttcatcaccaaaatt4020
aatcatcatatgattctacaacagtcagatacagtcagatataggcatgttcggagagat4080
gccatactagaattgaaatgtcaaattttggaagagaaaaaaagcaatagagttaataaa4140
aaagaaaaagaaagggctactctctgcattcatcctttcatggcaaaattaattttacac4200
gatcgccaaaagtaaatttgatcactaattaatgtgaaattactttgacacgatggcatg4260
tatcgaatcttattttaccattgtgtgcatccctaacaaaaaaaaaaacgtacttactct4320
ttccactatcgataaatcagtccttttaaccggaaaattcatccttccaaacaaaaaaaa4380
aatctgaaacaggatattcgatgtttttctgaatggaaaagtaagccacattgggtactc4440
attgaagcgcttatgcaaggccgcacgcttcatcgcgttgccccataaccataagaaggt4500
tgggttcaggggtgctgttcgagtgcttgtgttcaccatcgtcttgtgtccgctgttcct4560
gctctacatgtttgggctcttcgtttccccctggatttcgctgtggcgtctaatccagca4620
ggattacggcgtgacggccggagacagcagcagcaaggcacacctgcagcctgcgctggt4680
ggttctctactccctggccctgttccagggcgtcctcttctactacagggccatctctgc4740
ttgggaagaacagaagctagtgaaagatgtggccgacaaatacatgtttgatacagtgtc4800
acgcagttcagtttcggattatttacatgagatcaaggtgggatgtgagaatgacccgtc4860
ctttgccagagggaggaacctgatcacatacgccgtcaagctgatggaatccacatcacc4920
ggatgggtacctttcaggtgcacggattcttgacacactcatcaagtttaatagggatgc4980
tgatgcatcggggagcgaattcccggggcagagtatgcagatttacaatatgattggatc5040
cgcaacctccagtcccatactccacaacttagttcagatgctggattccaagagtgcata5100
tgatggagagatcaggttgcgcgccgcgaggattgtggatcactttgctggtgagatccg5160
tttggacaagatcctacaagggatccggtgtgtatcttccttgcttgaactcgagcagaa5220
attatttcaggattcttctgaagaagaggatgatcaccagatttctgtcaaagaaaagga5280
ttattaccccaaagactatgaacagatgcaacttataggcatgcagatccttttaaacct5340
ctcctatgacaagaacaacttgtccctcatgagcaacacagatgatccggccttgatcaa5400
caagattgtggcacttataacgtccaagggatcacttcacaaaaaagaacataacgaatg5460
gtcccgtatggcagagctcggcgtgaagatactaagccgatttatgcgatttatgtatgg5520
ccctacaaaatcaaacaatattctgtggcatgaaatatcaacaagcagcaaagcaatcgg5580
caccttggagagcattcttgagtgtgaccaatgtgactctgtgctgaagaaacacgccat5640
aaggattctcagaaggatattcatggatacatcttcagctatgggtgaaggagacagaga5700
aaggttcatcggctccttgatggacatgtctcttcacaacagtaatggtgactttcaaaa5760
tctggcaggtgtagatctggcgctcaagaaacaagggttaagcattctcaaagaaatata5820
cttgaatccatcttcgattatggacgaaggagacagagaaaggttcatcggctccctgat5880
ggacatgtttcttgacaacagtaagggcgactttggaaatctgccaggtgaagatctgga5940
cctcaagaaacaagagttaagcattctcaaagaaatatgcatggatccatcttcgtttat6000
gggcgaaagtgacagagaaaaattcatcggcatcctgatggacatgtttcttcacaacag6060
caagggcgacttgtttgaaaaactagcaggtgatgatctggtgcagatatgtagaagaag6120
tggaagcagtgccgccatcatattgaagaaatatggtcatgatattgttaattgtattgc6180
tgatactcgttcaagcgtgtacagcagcatgcacagaaaaattgcagcaaagatcctcaa6240
tcatctgtgtagtccctactccactgacgaggaacatcttcagaatctgaaggaggccat6300
cattgatttgatacccaaggtacgttagtcgctgtcatttatacttcctatgtcaacaaa6360
caatttttttcatgataatgaaatacataactctggcagcctctacattgtcagatgcac6420
gaaaccatagctcattaaatagtttggcaacctctacattcgcagaaatccatgggtaac6480
aaacttgtttaaagcaaaactgaagcacaacttgtatcattaaaatttcatccatgctta6540
ctgaatatccatcagagtagtctccaaaagatgggacactgcttttatatgcatcctatt6600
tatatcggaatgaccgaaagcactgtaatgttgcaggtgctaagagaagcacttggttgg6660
gggttgacaggaaaagagatacaaggagtggcagtttcaggccttgaaggtacccaagat6720
gatgactggaaattgcaggaagcattggcgtccctctgtgcaaccgtgtttaacagaatt6780
gtcagcaaggatgcagatttgactgctcggttcaacaacatcgccgctggtatctgcgac6840
caggcagcgaagcctcgcgtgaccttcgcagacctcatcaaagaagctgtgaaagttcat6900
cgcatggaatttaagaaaccagaaccaccaaaaccacctgccggcccggcactttacgag6960
tttatgcctgcgtactaccccccgccccactttatgtatttgagcgaggaagatcccaac7020
gcgtgttgtatctcttgataactccctaatttgtaatgcaaaccgaggatgaaaaaaata7080
tatatgcctatacggtactgctccatttccaaataa7116
<210>3
<211>855
<212>prt
<213>水稻渗入系il-e1454(oryzasativa)
<220>
<221>transmem
<222>(29)..(191)
<223>跨膜结构域
<220>
<221>domain
<222>(207)..(811)
<223>arm重复结构域
<220>
<221>domain
<222>(817)..(839)
<223>富含脯氨酸结构域
<400>3
metserglyalaglyglnserargglyhisargleuglyleuhisile
151015
aspserasptrpprogluvalleuleuileasnasptyralavalphe
202530
metglytyrleusermetvalvalthrglythrglypheleuvalleu
354045
thrtrpserthrvalileleuleuglyglyphevalsermetleuser
505560
asnlysaspphetrpserleuthrvalilethrleuvalglnthrarg
65707580
ilepheaspvalpheleuasnglylysvalserhisileglytyrser
859095
leulysargleucyslysalaalaargpheilealaleuprohisasn
100105110
hislyslysvalglypheargglyalavalargvalleuvalphethr
115120125
ilevalleucysproleupheleuleutyrmetpheglyleupheval
130135140
serprotrpileserleutrpargleuileglnglnasptyrglyval
145150155160
thralaglyaspserserserlysalahisleuglnproalaleuval
165170175
valleutyrserleualaleupheglnglyvalleuphetyrtyrarg
180185190
alaileseralatrpglugluglnlysleuvallysaspvalalaasp
195200205
lystyrmetpheaspthrvalserargserservalserasptyrleu
210215220
hisgluilelysvalglycysgluasnaspproserphealaarggly
225230235240
argasnleuilethrtyralavallysleumetgluserthrserpro
245250255
aspglytyrleuserglyalaargileleuaspthrleuilelysphe
260265270
asnargaspalaaspalaserglyserglupheproglyglnsermet
275280285
glniletyrasnmetileglyseralathrserserproileleuhis
290295300
asnleuvalglnmetleuaspserlysseralatyraspglygluile
305310315320
argleuargalaalaargilevalasphisphealaglygluilearg
325330335
leuasplysileleuglnglyileargcysvalserserleuleuglu
340345350
leugluglnlysleupheglnaspsersergluglugluaspasphis
355360365
glnileservallysglulysasptyrtyrprolysasptyrglugln
370375380
metglnleuileglymetglnileleuleuasnleusertyrasplys
385390395400
asnasnleuserleumetserasnthraspaspproalaleuileasn
405410415
lysilevalalaleuilethrserlysglyserleuhislyslysglu
420425430
hisasnglutrpserargmetalagluleuglyvallysileleuser
435440445
argphemetargphemettyrglyprothrlysserasnasnileleu
450455460
trphisgluileserthrserserlysalaileglythrleugluser
465470475480
ileleuglucysaspglncysaspservalleulyslyshisalaile
485490495
argileleuargargilephemetaspthrserseralametglyglu
500505510
glyasparggluargpheileglyserleumetaspmetserleuhis
515520525
asnserasnglyasppheglnasnleualaglyvalaspleualaleu
530535540
lyslysglnglyleuserileleulysgluiletyrleuasnproser
545550555560
serilemetaspgluglyasparggluargpheileglyserleumet
565570575
aspmetpheleuaspasnserlysglyasppheglyasnleuprogly
580585590
gluaspleuaspleulyslysglngluleuserileleulysgluile
595600605
cysmetaspproserserphemetglygluseraspargglulysphe
610615620
ileglyileleumetaspmetpheleuhisasnserlysglyaspleu
625630635640
pheglulysleualaglyaspaspleuvalglnilecysargargser
645650655
glyserseralaalaileileleulyslystyrglyhisaspileval
660665670
asncysilealaaspthrargserservaltyrsersermethisarg
675680685
lysilealaalalysileleuasnhisleucysserprotyrserthr
690695700
aspglugluhisleuglnasnleulysglualaileileaspleuile
705710715720
prolysvalleuargglualaleuglytrpglyleuthrglylysglu
725730735
ileglnglyvalalavalserglyleugluglythrglnaspaspasp
740745750
trplysleuglnglualaleualaserleucysalathrvalpheasn
755760765
argilevalserlysaspalaaspleuthralaargpheasnasnile
770775780
alaalaglyilecysaspglnalaalalysproargvalthrpheala
785790795800
aspleuilelysglualavallysvalhisargmetgluphelyslys
805810815
progluproprolysproproalaglyproalaleutyrgluphemet
820825830
proalatyrtyrproproprohisphemettyrleuserglugluasp
835840845
proasnalacyscysileser
850855
<210>4
<211>3804
<212>dna
<213>水稻渗入系il-e1454(oryzasativa)
<220>
<221>5’utr
<222>(1)..(1158)
<223>5’非翻译区
<220>
<221>cds
<222>(1159)..(3726)
<223>基因编码区
<220>
<221>3’utr
<222>(3727)..(3804)
<223>3’非翻译区
<400>4
accaccacttccctctctcctcctcatcctcatcttcatatctagatagacctcatcttc60
tccagccaccaccgcctccctccccaccggtggtggcggcgggaagacagcggctcggct120
gagcccggcgggtagggcaacgatttagtatgggaggagcagcggcgacttctctacacg180
acggcgttcggaggcggatctagcagcggcgactccatccagcggtggatccgccgcctc240
ctagtcctagctctctctcccccgctcctccctcctccccttcccccgccgccaccaccc300
accccctctccctctctcagaactggtgcgcggggagcggtggaggctgcggcggcgaag360
gctgggggggctcggcagcggcggcgacggtgagaccggtagcagtgatggtggagggga420
cggatccgatggcggtggttctcgtcagccagttcaaggatgacgacagcggcggctgcg480
gcaacgatggacaggctctgggcggctcctccaccctcctcccctctctcatatccaacg540
actcggggtggtaggacgtggccgacgcatccggcggcggagctcgggtatcaatccggc600
atgtttttttttctccaaaatcaaattttcctggtggtcgggaaacggattttcaatttt660
tttttacgctcgaaaatcttttttcgctggcggtcaacgtaacttaaccgcttgtgaaaa720
caagctttaaagtttcgctagtgaagattgttattttcactagccttttgtttgcgggcg780
gctgacagtgccaccagcgaaaaattcttttgctagtagtgattgctagatggtcagcag840
agaacttgtggagtatatgactatcaagtatcaaccaacagtacacttcagcagaaagag900
gatggttctagctctccggtggtttaatattgaaacgtacagcccacccaccaccacccc960
agtcgtccccagctaagcgctcccttgcttgcctcagtagacgagctgccaactagtagc1020
actccagcaagcagcgggtcagcggagtccggagggcgcacgtgcgccgcagcgaggtag1080
cagcgtggccaggcggcggctggctggccagccgggttcggcaagcggcttagcaggtgt1140
ttagcggaggaaggatcgatgtccggcgccggacagagccgtggtcatcgc1191
metserglyalaglyglnserargglyhisarg
1510
cttggattacacattgattcggattggccagaggtcttgttgatcaat1239
leuglyleuhisileaspserasptrpprogluvalleuleuileasn
152025
gactatgcggtgttcatggggtacctgtcgatggttgtcaccgggacg1287
asptyralavalphemetglytyrleusermetvalvalthrglythr
303540
gggttccttgtgctcacttggtccaccgtcatcctcctcggtggattc1335
glypheleuvalleuthrtrpserthrvalileleuleuglyglyphe
455055
gtctccatgctatccaacaaggacttctggagtctcacggtgatcacg1383
valsermetleuserasnlysaspphetrpserleuthrvalilethr
60657075
ctcgttcaaacaaggatattcgatgtttttctgaatggaaaagtaagc1431
leuvalglnthrargilepheaspvalpheleuasnglylysvalser
808590
cacattgggtactcattgaagcgcttatgcaaggccgcacgcttcatc1479
hisileglytyrserleulysargleucyslysalaalaargpheile
95100105
gcgttgccccataaccataagaaggttgggttcaggggtgctgttcga1527
alaleuprohisasnhislyslysvalglypheargglyalavalarg
110115120
gtgcttgtgttcaccatcgtcttgtgtccgctgttcctgctctacatg1575
valleuvalphethrilevalleucysproleupheleuleutyrmet
125130135
tttgggctcttcgtttccccctggatttcgctgtggcgtctaatccag1623
pheglyleuphevalserprotrpileserleutrpargleuilegln
140145150155
caggattacggcgtgacggccggagacagcagcagcaaggcacacctg1671
glnasptyrglyvalthralaglyaspserserserlysalahisleu
160165170
cagcctgcgctggtggttctctactccctggccctgttccagggcgtc1719
glnproalaleuvalvalleutyrserleualaleupheglnglyval
175180185
ctcttctactacagggccatctctgcttgggaagaacagaagctagtg1767
leuphetyrtyrargalaileseralatrpglugluglnlysleuval
190195200
aaagatgtggccgacaaatacatgtttgatacagtgtcacgcagttca1815
lysaspvalalaasplystyrmetpheaspthrvalserargserser
205210215
gtttcggattatttacatgagatcaaggtgggatgtgagaatgacccg1863
valserasptyrleuhisgluilelysvalglycysgluasnasppro
220225230235
tcctttgccagagggaggaacctgatcacatacgccgtcaagctgatg1911
serphealaargglyargasnleuilethrtyralavallysleumet
240245250
gaatccacatcaccggatgggtacctttcaggtgcacggattcttgac1959
gluserthrserproaspglytyrleuserglyalaargileleuasp
255260265
acactcatcaagtttaatagggatgctgatgcatcggggagcgaattc2007
thrleuilelyspheasnargaspalaaspalaserglysergluphe
270275280
ccggggcagagtatgcagatttacaatatgattggatccgcaacctcc2055
proglyglnsermetglniletyrasnmetileglyseralathrser
285290295
agtcccatactccacaacttagttcagatgctggattccaagagtgca2103
serproileleuhisasnleuvalglnmetleuaspserlysserala
300305310315
tatgatggagagatcaggttgcgcgccgcgaggattgtggatcacttt2151
tyraspglygluileargleuargalaalaargilevalasphisphe
320325330
gctggtgagatccgtttggacaagatcctacaagggatccggtgtgta2199
alaglygluileargleuasplysileleuglnglyileargcysval
335340345
tcttccttgcttgaactcgagcagaaattatttcaggattcttctgaa2247
serserleuleugluleugluglnlysleupheglnaspserserglu
350355360
gaagaggatgatcaccagatttctgtcaaagaaaaggattattacccc2295
glugluaspasphisglnileservallysglulysasptyrtyrpro
365370375
aaagactatgaacagatgcaacttataggcatgcagatccttttaaac2343
lysasptyrgluglnmetglnleuileglymetglnileleuleuasn
380385390395
ctctcctatgacaagaacaacttgtccctcatgagcaacacagatgat2391
leusertyrasplysasnasnleuserleumetserasnthraspasp
400405410
ccggccttgatcaacaagattgtggcacttataacgtccaagggatca2439
proalaleuileasnlysilevalalaleuilethrserlysglyser
415420425
cttcacaaaaaagaacataacgaatggtcccgtatggcagagctcggc2487
leuhislyslysgluhisasnglutrpserargmetalagluleugly
430435440
gtgaagatactaagccgatttatgcgatttatgtatggccctacaaaa2535
vallysileleuserargphemetargphemettyrglyprothrlys
445450455
tcaaacaatattctgtggcatgaaatatcaacaagcagcaaagcaatc2583
serasnasnileleutrphisgluileserthrserserlysalaile
460465470475
ggcaccttggagagcattcttgagtgtgaccaatgtgactctgtgctg2631
glythrleugluserileleuglucysaspglncysaspservalleu
480485490
aagaaacacgccataaggattctcagaaggatattcatggatacatct2679
lyslyshisalaileargileleuargargilephemetaspthrser
495500505
tcagctatgggtgaaggagacagagaaaggttcatcggctccttgatg2727
seralametglygluglyasparggluargpheileglyserleumet
510515520
gacatgtctcttcacaacagtaatggtgactttcaaaatctggcaggt2775
aspmetserleuhisasnserasnglyasppheglnasnleualagly
525530535
gtagatctggcgctcaagaaacaagggttaagcattctcaaagaaata2823
valaspleualaleulyslysglnglyleuserileleulysgluile
540545550555
tacttgaatccatcttcgattatggacgaaggagacagagaaaggttc2871
tyrleuasnproserserilemetaspgluglyasparggluargphe
560565570
atcggctccctgatggacatgtttcttgacaacagtaagggcgacttt2919
ileglyserleumetaspmetpheleuaspasnserlysglyaspphe
575580585
ggaaatctgccaggtgaagatctggacctcaagaaacaagagttaagc2967
glyasnleuproglygluaspleuaspleulyslysglngluleuser
590595600
attctcaaagaaatatgcatggatccatcttcgtttatgggcgaaagt3015
ileleulysgluilecysmetaspproserserphemetglygluser
605610615
gacagagaaaaattcatcggcatcctgatggacatgtttcttcacaac3063
aspargglulyspheileglyileleumetaspmetpheleuhisasn
620625630635
agcaagggcgacttgtttgaaaaactagcaggtgatgatctggtgcag3111
serlysglyaspleupheglulysleualaglyaspaspleuvalgln
640645650
atatgtagaagaagtggaagcagtgccgccatcatattgaagaaatat3159
ilecysargargserglyserseralaalaileileleulyslystyr
655660665
ggtcatgatattgttaattgtattgctgatactcgttcaagcgtgtac3207
glyhisaspilevalasncysilealaaspthrargserservaltyr
670675680
agcagcatgcacagaaaaattgcagcaaagatcctcaatcatctgtgt3255
sersermethisarglysilealaalalysileleuasnhisleucys
685690695
agtccctactccactgacgaggaacatcttcagaatctgaaggaggcc3303
serprotyrserthraspglugluhisleuglnasnleulysgluala
700705710715
atcattgatttgatacccaaggtgctaagagaagcacttggttggggg3351
ileileaspleuileprolysvalleuargglualaleuglytrpgly
720725730
ttgacaggaaaagagatacaaggagtggcagtttcaggccttgaaggt3399
leuthrglylysgluileglnglyvalalavalserglyleuglugly
735740745
acccaagatgatgactggaaattgcaggaagcattggcgtccctctgt3447
thrglnaspaspasptrplysleuglnglualaleualaserleucys
750755760
gcaaccgtgtttaacagaattgtcagcaaggatgcagatttgactgct3495
alathrvalpheasnargilevalserlysaspalaaspleuthrala
765770775
cggttcaacaacatcgccgctggtatctgcgaccaggcagcgaagcct3543
argpheasnasnilealaalaglyilecysaspglnalaalalyspro
780785790795
cgcgtgaccttcgcagacctcatcaaagaagctgtgaaagttcatcgc3591
argvalthrphealaaspleuilelysglualavallysvalhisarg
800805810
atggaatttaagaaaccagaaccaccaaaaccacctgccggcccggca3639
metgluphelyslysprogluproprolysproproalaglyproala
815820825
ctttacgagtttatgcctgcgtactaccccccgccccactttatgtat3687
leutyrgluphemetproalatyrtyrproproprohisphemettyr
830835840
ttgagcgaggaagatcccaacgcgtgttgtatctcttgataactcccta3736
leuserglugluaspproasnalacyscysileser
845850855
atttgtaatgcaaaccgaggatgaaaaaaatatatatgcctatacggtactgctccattt3796
ccaaataa3804
<210>5
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>5
accaccacttccctctctcctc22
<210>6
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>6
ggaaatggagcagtaccgtataggc25
<210>7
<211>37
<212>dna
<213>人工序列(artificialsequence)
<400>7
acgcgtcgacataggagaggaagagagaggctggaca37
<210>8
<211>37
<212>dna
<213>人工序列(artificialsequence)
<400>8
ttggcgcgccccaaacttgcaggaaactagtagaaac37
<210>9
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>9
acactacatggcgtgatttcat22
<210>10
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>10
tccactatcggcgagtacttct22
<210>11
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>11
agagtgcatatgatggagagat22
<210>12
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>12
tctggtgatcatcctcttcttc22
- 还没有人留言评论。精彩留言会获得点赞!