肺癌相关分子标志物基因突变的检测试剂盒与检测方法与流程
本发明涉及生物检测技术领域,尤其涉及一种肺癌相关分子标志物基因突变的检测试剂盒与检测方法。
背景技术:
肿瘤是人体在各种不良因素的作用下,身体里某个或者某些部位组织中的细胞发生异常增生而形成的新生物,发生异常增生的细胞就是肿瘤细胞。
原发性肺癌是我国发病率及死亡率高居各类恶性肿瘤之首,严重威胁人类健康。全球肺癌的发病率和病死率肺癌患者中约80%-85%为非小细胞肺癌(nscl),约50%的患者确诊时已为晚期,预后差。准确获知肿瘤的生物学信息,从而在基因分型指导下进行个体化治疗对于指导临床用药至关重要。肺癌发病率最为常见的是非小细胞肺腺癌,部分基因的突变会导致肺癌的产生,如在部分患者中能检测到egfr、braf、kars等基因发生突变。其中egfr是一种跨膜受体蛋白,与细胞增殖、转移、凋亡等多种信号转导通路相关。egfrp.l858r突变、外显子19插入或缺失,对egfrtkis药物敏感性增加;而egfr基因p.t790m突变,对第一代egfrtkis药物敏感性降低(厄洛替尼、埃克替尼、吉非替尼);对第三代egfrtkis药物(osimertinib,rociletinib)敏感性增加。因此,对于初期发生p.l858r突变、外显子19插入或缺失突变的患者对于靶向药的疗效观测可以落脚于p.t790m位点是否发生突变,检测egfr基因的突变状态是决定患者是否能够应用egfr-tki治疗的先决条件。
然而目前在现阶段临床可通过定量即时聚合酶链锁反应(quantitativerealtimepolymerasechainreaction,q-pcr)、非放射性原位杂交技术(fluorescenceinsituhybridization,fish)、多重引物pcr(multiplexpcr)等方式对患者进行相关位点的基因突变检测,这些技术存在一次只能检测一个基因或检测成本高等问题,而且无法应用到大规模的临床样本研究,因此,亟需提供一种温度控制简单,且灵敏度、特异性高的特异性片段检测方法。
技术实现要素:
本发明的目的在于克服现有技术的缺陷,提供了一种肺癌相关分子标志物基因突变的检测试剂盒与检测方法,特异性强,灵敏度高,最低检测极限达到10拷贝/ul。
本发明是这样实现的:
本发明的目的之一在于提供一种用于检测肺癌相关分子标志物基因突变的crdna,所述crdna包括:
egfr-t790m突变位点的crdna:其序列如seqidno.6-7中任一所示;
egfr-l858r突变位点的crdna:其序列如seqidno.8-9中任一所示;
braf基因突变位点的crdna:其序列如seqidno.10-11中任一所示;
alk基因突变位点的crdna:其序列如seqidno.12-13中任一所示;
ros1基因突变位点的crdna:其序列如seqidno.14-15中任一所示。
本发明的目的之二在于提供一种用于检测肺癌相关分子标志物基因突变的crrna,所述crrna包括:
egfr-t790m突变位点的crrna:其序列如seqidno.16-17中任一所示;
egfr-l858r突变位点的crrna:其序列如seqidno.18-19中任一所示;
braf基因突变位点的crrna:其序列如seqidno.20-21中任一所示;
alk基因突变位点的crrna:其序列如seqidno.22-23中任一所示;
ros1基因突变位点的crrna:其序列如seqidno.24-25中任一所示。
本发明的目的之三在于提供一种肺癌相关分子标志物基因突变的检测试剂盒,含有所述crdna或者crrna。
优选地,所述试剂盒还包括cpf1蛋白和荧光探针。
所述荧光探针选自如下探针中的一个:
荧光探针1:其序列如seqidno.26所示;
荧光探针2:其序列如seqidno.27所示;
荧光探针3:其序列如seqidno.28所示;
所述荧光探针的序列的5’端均标记有荧光基团,3’端标记均有猝灭基团。
优选地,所述荧光基团包括fam、vic、hex、trt、cy3、cy5、rox、joe和texasred中的一种,所述猝灭基团包括tamra、dabcyl、mgb、bhq-1、bhq-2和bhq-3中的一种。本发明中的荧光探针的序列的5’端标记选用hex基团,3’端标记选用bhq-1基团。
优选地,所述试剂盒还包括dna酶抑制剂。
优选地,所述检测试剂盒为胶体金检测试剂盒或实时荧光定量检测试剂盒。
本发明的目的之四在于提供所述的crrna、以及所述的试剂盒在用于检测肺癌相关分子标志物基因突变上的用途。
本发明的目的之五在于提供一种肺癌相关分子标志物基因突变的检测方法,包括如下步骤:
s1、扩增待测样本的核酸得到扩增产物;
s2、将所述的扩增产物、crrna、cpf1蛋白、seqidno.26-28任一所示的荧光探针和无酶水构成的检测体系进行检测。
优选地,将所述检测体系孵育后加入到胶体金试纸条样品检测区进行检测或者将所述检测体系孵育后荧光检测仪测定荧光值。
作为以上实施方式之一,将扩增产物、cpf1蛋白/crrna复合物溶液,加入荧光探针和无酶水孵育后加入到胶体金试纸条样品检测区。本发明提供了一种基于crispr-cpf1系统以胶体金可视化检测荧光基团,以试纸条的形式呈现检测结果,操作简便,无需仪器,具有很强的适用范围和场所,实用性极强。
作为以上另一种优选方式之一,将扩增产物、cpf1/crrna复合物溶液,加入荧光探针和无酶水孵育后荧光检测仪测定荧光值。可以做成便携式检测仪可以随时随地及时检测,这些应用和方法为后续试剂盒的开发,临床诊断的应用提供了平台,适用于大规模临床样本检测。
与现有技术相比,本发明具有如下优点和效果:
本发明提供的用于检测肺癌相关分子标志物基因突变的crrna、试剂盒及检测方法,设计与人基因突变靶点相关的crrna,运用此crrna结合crispr-cpf1系统进行突变检测的方法,此方法具有成本低、可多次重复检测、方法简单、检测速度快、灵敏(最低检测极限达到10拷贝/ul;)、特异,可在短时间内通过荧光信号的变化检测到含有相应突变的目标核酸:
(1)在对egfr-t790m的检测中,反应进行到20-30分钟时,突变型基因的荧光信号就明显高于野生型基因。该方法可以区分10拷贝突变株与104拷贝的野生株,灵敏度高于普通pcr。
(2)在对egfr-l858r的检测中,反应进行到20-30分钟时,突变型基因的荧光信号就明显高于野生型基因。该方法可以区分10拷贝突变株与104拷贝的野生株,灵敏度高于普通pcr。
(3)在对braf的检测中,反应进行到20-30分钟时,突变型基因的荧光信号就明显高于野生型基因。该方法可以区分10拷贝突变株与104拷贝的野生株,灵敏度高于普通pcr。
(4)在对alk的检测中,反应进行到20-30分钟时,突变型基因的荧光信号就明显高于野生型基因。该方法可以区分10拷贝突变株与104拷贝的野生株,灵敏度高于普通cr。
(5)在对ros1的检测中,反应进行到20-30分钟时,突变型基因的荧光信号就明显高于野生型基因。该方法可以区分10拷贝突变株与104拷贝的野生株,灵敏度高于普通pcr。
附图说明
图1为egfr-t790m的特异性检测结果;
图2为egfr-l858r的特异性检测结果;
图3为braf的特异性检测结果;
图4为alk的特异性检测结果;
图5为ros1的特异性检测结果;
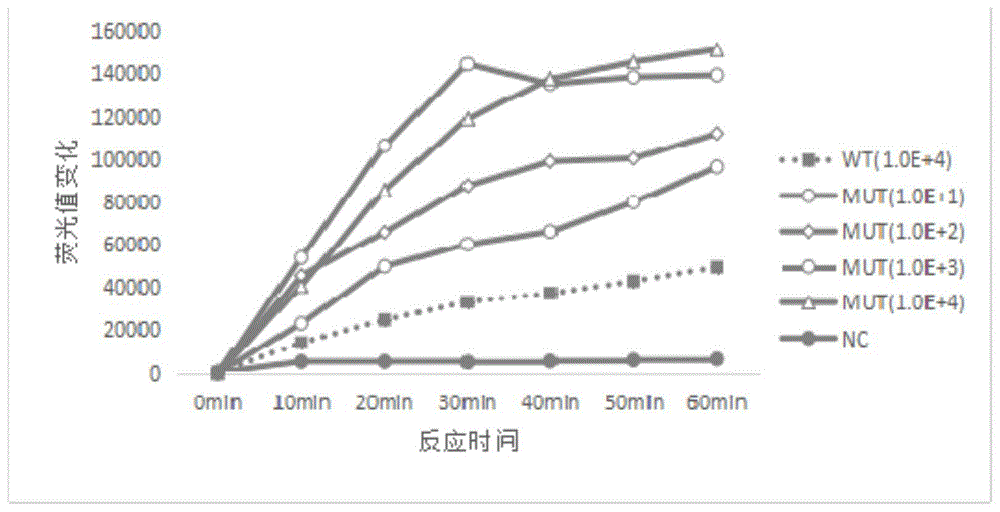
图6为egfr-t790m、egfr-l858r突变的灵敏度检测结果;
图7为braf突变的灵敏度检测结果;
图8为alk突变的灵敏度检测结果;
图9为ros1突变的灵敏度检测结果。
具体实施方式
实施例1靶向基因突变位点crrna的的设计及获得
1、基于crispr-cpf1系统的肺癌检测位点的发现
我们根据最新出具的《nccn指南》,获得肺癌常用突变检测分子标志物基因。根据基因序列及大量临床检测数据确定基因常见突变位点序列,针对这些不同区域设计crrna并构建crispr/cpf1系统进行研究。结果表明,以seqidno.1-5所示区域序列为基于crispr/cpf1系统的肺癌突变检测位点(加粗部分为突变区域),具有很好的检测效果。
表1
2、靶向基因突变位点crrna的设计
(1)靶向基因突变位点crrna设计原则
由于crispr-cpf1系统是一种新型的靶向dna基因编辑系统,其中cpf1与crrna结合形成监测复合物,crrna的向导区域用互补序列识别目标dna,并且cpf1降解目标dna链,所述crrna设计要求:crrna包括蛋白锚定序列和向导序列,序列格式为:5`-与cpf1蛋白结合的锚定序列-crrna向导序列-3`,蛋白锚定序列需要根据cpf1蛋白来确定,使其能够与选定的cpf1蛋白匹配并结合;向导序列则与靶向dna中的片段匹配。crrna向导序列不能离起始密码子(atg)太近;长度为22-24个核苷酸。
2.crdna序列的选择
所述与cpf1蛋白结合的锚定序列seqidno.14所示;所述crrna向导序列根据seqidno.1-5所示序列中突变位点区域设计;最后得到了如下所示的5个突变位点的crdna片段。
egfr-t790m突变位点的crdna:其序列如seqidno.6-7中任一所示;
egfr-l858r突变位点的crdna:其序列如seqidno.8-9中任一所示;
braf基因突变位点的crdna:其序列如seqidno.10-11中任一所示;
alk基因突变位点的crdna:其序列如seqidno.12-13中任一所示;
ros1基因突变位点的crdna:其序列如seqidno.14-15中任一所示。
3、crrna的获得
将所得的5个突变位点的crdna片段分别在t7rna聚合酶(转录反应体系如表2所示)作用下生成rna,回收纯化得到crrna。
表2
所得crrna包括:
egfr-t790m突变位点的crrna:其序列如seqidno.16-17中任一所示;
egfr-l858r突变位点的crrna:其序列如seqidno.18-19中任一所示;
braf基因突变位点的crrna:其序列如seqidno.20-21中任一所示;
alk基因突变位点的crrna:其序列如seqidno.22-23中任一所示;
ros1基因突变位点的crrna:其序列如seqidno.24-25中任一所示。
实施例2肺癌相关分子标志物基因突变的检测试剂盒及检测方法
1、试剂盒的组成
本试剂盒包括肺癌基因突变检测相关的5条crrna(5个突变位点的crrna如实施例1所示获得)或者5个突变位点的crdna(当试剂盒中为crdna时需要操作者先将crdna片段分别在t7rna聚合酶作用下生成rna,回收纯化得到crrna,具体见实施例1)、一条特异性荧光探针(序列见表3,如seqidno.26-28所示的任意一种探针)、cpf1蛋白、无酶水、dna酶抑制剂;
表3
优选地,所述试剂盒还可包括扩增体系,所述扩增体系包括等温扩增引物对,其中egfr-t790m突变位点的等温扩增引物对序列如seqidno.29-30所示;egfr-l858r突变位点的等温扩增引物对序列如seqidno.31-32所示;braf基因突变位点的等温扩增引物对序列如seqidno.33-34所示;alk基因突变位点的等温扩增引物对序列如seqidno.35-36所示;ros1基因突变位点的等温扩增引物对序列如seqidno.37-38所示。
2、肺癌相关分子标志物基因突变的检测方法
(1)取50-100ng待样本的dna,加入等温扩增体系中扩增体系如表4所示;
表4
(2)将所述得到的扩增产物、与所述检测试剂:crrna、cpf1蛋白、荧光探针和无酶水一起制成检测体系,如表5所示。
表5
用100-250nm纯化的cpf1,250-500nmcrrna,1-5μl合成的荧光探针,2μldna酶抑制剂,5-10ul目的dna扩增产物,在检测缓冲液(nebuffer3)中37℃孵育1~3小时。同时设立阴性对照组。将所述检测体系孵育后加入到胶体金试纸条样品检测区进行检测或者将所述检测体系孵育后荧光检测仪测定荧光值。反应结束后统计分析荧光值变化情况。
实施例3突变crrna对野生型和突变型序列的特异性检测
合成野生株和突变株的靶序列,分别利用上述5个突变crrna检测其特异性。将实施例1制备得到的5个突变位点的crrna分别构建crispr-cpf1体系在体外进行切割有效性验证。
用100-250nm纯化的cpf1,250-500nmcrrna,1-5μl合成的荧光探针,2μldna酶抑制剂,不同稀释浓度的目的dna,在检测缓冲液(nebuffer3)中37℃孵育1~3小时。同时设立空白对照。空白对照组为每个实验组相对应的没有加crrna及没有加cpf1的信号组。几组反应混合物同时在便携式检测仪中反应(温度设置为37℃,动力学检测每10min检测一次共1个小时)。使用荧光定量pcr仪检测体系的荧光信号变化。
1、egfr-t790m的特异性检测
我们检测了egfr-t790mcrrna对野生egfr-t790m、突变型egfr-t790m的检测效果,图1为egfr-t790m突变位点的体系荧光值结果。结果显示,空白对照在检测的1h内荧光强度保持在4000-5000范围内不变;加入野生egfr-t790m、突变型egfr-t790m的模板后均出现了荧光信号的升高,其中与egfr-t790mcrrna完全匹配的不同浓度的egfr-t790m突变的模板的信号普遍高于野生型对照组。
2、egfr-l858r的特异性检测
我们检测了egfr-l858rcrrna对野生egfr-l858r、突变型egfr-l858r的检测效果,图2为egfr-l858r突变位点的体系荧光值结果。结果显示,空白对照在检测的1h内荧光强度保持在4000-5000范围内不变;加入野生egfr-l858r、突变型egfr-l858r的模板后均出现了荧光信号的升高,其中与egfr-l858rcrrna完全匹配的egfr-l858r突变模板的信号与野生型模板的信号随着时间的延长普遍高于野生型对照组。
3、braf的特异性检测
我们检测了brafcrrna对野生braf、突变型braf的检测效果,图3为braf突变位点的体系荧光值结果。结果显示,空白对照在检测的1h内荧光强度保持在4000-5000范围内不变;加入野生braf、突变型braf的模板后均出现了荧光信号的升高,其中与brafcrrna完全匹配的braf突变模板的信号与野生型模板的信号随着时间的延长普遍高于野生型对照组。
4、alk的特异性检测
我们检测了alkcrrna对野生alk、突变型alk的检测效果,图4为alk突变位点的体系荧光值结果。结果显示,空白对照在检测的1h内荧光强度保持在4000-5000范围内不变;加入野生alk、突变型alk的模板后均出现了荧光信号的升高,其中与alkcrrna完全匹配的alk突变模板的信号与野生型模板的信号随着时间的延长普遍高于野生型对照组。
5、ros1的特异性检测
我们检测了ros1crrna对野生ros1、突变型ros1的检测效果,图5为ros1突变位点的体系荧光值结果。结果显示,空白对照在检测的1h内荧光强度保持在4000-5000范围内不变;加入野生ros1、突变型ros1的模板后均出现了荧光信号的升高,其中与ros1crrna完全匹配的ros1突变模板的信号与野生型模板的信号随着时间的延长普遍高于野生型对照组。
综上可知,结果均显示靶标crrna检测靶标dna的体系荧光值变化趋势明显高于阴性对照。表明本发明的用于检测肺癌相关分子标志物基因突变的5个crrna可以分别特异性地检测出对应的突变序列。
实施例4突变crrna对野生型和突变型序列的灵敏性试验
1、egfr-t790m、egfr-l858r突变的灵敏度检测
将egfr-t790m、egfr-l858r突变质粒和野生型质粒标准品梯度稀释后作为模板进行pcr扩增,随后用1.0%的琼脂糖凝胶电泳检测,电泳结果如图6所示。阴性对照(nc)无条带,说明扩增过程中无污染;在100-500bp之间分别出现一条与目的条带大小(200bp及306bp)一致的单一条带;模板浓度在104copies/μl以上时,均可观察到电泳条带;而当模板浓度低于为103copies/μl时则观察不到扩增条带,说明利用琼脂糖凝胶电泳检测pcr产物,其灵敏度约能达到103copies/μl左右。
2、braf基因突变的灵敏度检测
将braf突变质粒和野生型质粒标准品梯度稀释后作为模板进行pcr扩增,随后用1.0%的琼脂糖凝胶电泳检测,电泳结果如图7所示。四组模板对应的阴性对照(nc)无条带,说明扩增过程中无污染;在250-500bp之间分别出现一条与目的条带大小(300bp)一致的单一条带;三组模板浓度在105copies/μl以上时,均可观察到清晰的电泳条带;而当模板浓度低于为102copies/μl时观察不到扩增条带,说明利用琼脂糖凝胶电泳检测pcr产物,其灵敏度约能达到102copies/μl左右。
3、alk基因突变的灵敏度检测
将alk突变质粒和野生型质粒标准品梯度稀释后作为模板进行pcr扩增,随后用1.0%的琼脂糖凝胶电泳检测,电泳结果如图8所示。四组模板对应的阴性对照(nc)无条带,说明扩增过程中无污染;在80-120bp之间分别出现一条与目的条带大小(86bp及109bp)一致的单一条带;三组模板浓度在105copies/μl以上时,均可观察到清晰的电泳条带;而当模板浓度低于为102copies/μl时则观察不到扩增条带,说明利用琼脂糖凝胶电泳检测pcr产物,其灵敏度约能达到102copies/μl左右。
4、ros1基因突变的灵敏度检测
将ros1突变质粒和野生型质粒标准品梯度稀释后作为模板进行pcr扩增,随后用1.0%的琼脂糖凝胶电泳检测,电泳结果如图9所示。四组模板对应的阴性对照(nc)无条带,说明扩增过程中无污染;在100-300bp之间分别出现一条与目的条带大小(385bp及346bp)一致的单一条带;三组模板浓度在105copies/μl以上时,均可观察到清晰的电泳条带;而当模板浓度低于为102copies/μl时则观察不到扩增条带,说明利用琼脂糖凝胶电泳检测pcr产物,其灵敏度约能达到102copies/μl左右。
所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包括在本发明的保护范围之内。
序列表
<110>武汉博杰生物医学科技有限公司
<120>肺癌相关分子标志物基因突变的检测试剂盒与检测方法
<160>38
<170>siposequencelisting1.0
<210>1
<211>71
<212>dna
<213>egfr-t790m(突变检测位点)
<400>1
gggcatctgcctcacctccaccgtgcagctcatcacgcagctcatgcccttcggctgcct60
cctggactatg71
<210>2
<211>104
<212>dna
<213>egfr-l858r(突变检测位点)
<400>2
caggaacgtactggtgaaaacacccagcatgtcaagatcacagattttgggctggccaaa60
ctgctgggtgcggaagagaaagaataccatgcagaaggaggcaa104
<210>3
<211>52
<212>dna
<213>braf-v600e(突变检测位点)
<400>3
aataggtgattttggtctagctacagtgaaatctcgatggagtgggtcccat52
<210>4
<211>163
<212>dna
<213>alk-eml-4(突变检测位点)
<400>4
tgtcacccaccccggagccacacctgccactctcgctgatcctctctgtggtgacctctg60
ccctcgtggccgccctggtcctggctttctccggcatcatgattgtgtaccgccggaagc120
accaggagctgcaagccatgcagatggagctgcagagccctga163
<210>5
<211>114
<212>dna
<213>ros1-slc34a2(突变检测位点)
<400>5
gctgcaaataatctagggtttggtgaatatagtggaatcagtgagaatattatattagtt60
ggagatgatttttggataccagaaacaagtttcatacttactattatagttgga114
<210>6
<211>69
<212>dna
<213>人工序列(artificialsequence)
<400>6
acctccaccgtgcagctcatcgtgatctacaagagtagaaattaccctatagtgagtcgt60
attaatttc69
<210>7
<211>67
<212>dna
<213>人工序列(artificialsequence)
<400>7
ctccaccgtgcagctcatcgtgatctacaagagtagaaattaccctatagtgagtcgtat60
taatttc67
<210>8
<211>69
<212>dna
<213>人工序列(artificialsequence)
<400>8
caagatcacagattttgggggggcatctacaagagtagaaattaccctatagtgagtcgt60
attaatttc69
<210>9
<211>67
<212>dna
<213>人工序列(artificialsequence)
<400>9
agatcacagattttgggggggcatctacaagagtagaaattaccctatagtgagtcgtat60
taatttc67
<210>10
<211>69
<212>dna
<213>人工序列(artificialsequence)
<400>10
ggtgattttggtctagctacacagatctacaagagtagaaattaccctatagtgagtcgt60
attaatttc69
<210>11
<211>67
<212>dna
<213>人工序列(artificialsequence)
<400>11
tgattttggtctagctacacagatctacaagagtagaaattaccctatagtgagtcgtat60
taatttc67
<210>12
<211>68
<212>dna
<213>人工序列(artificialsequence)
<400>12
aaaggacctaaagtgtaccgccgatctacaagagtagaaattaccctatagtgagtcgta60
ttaatttc68
<210>13
<211>66
<212>dna
<213>人工序列(artificialsequence)
<400>13
aggacctaaagtgtaccgccgatctacaagagtagaaattaccctatagtgagtcgtatt60
aatttc66
<210>14
<211>66
<212>dna
<213>人工序列(artificialsequence)
<400>14
gctggttggagatgatttttgatctacaagagtagaaattaccctatagtgagtcgtatt60
aatttc66
<210>15
<211>68
<212>dna
<213>人工序列(artificialsequence)
<400>15
cagctggttggagatgatttttgatctacaagagtagaaattaccctatagtgagtcgta60
ttaatttc68
<210>16
<211>44
<212>rna
<213>人工序列(artificialsequence)
<400>16
uaauuucuacucuuguagaucacgaugagcugcacgguggaggu44
<210>17
<211>42
<212>rna
<213>人工序列(artificialsequence)
<400>17
uaauuucuacucuuguagaucacgaugagcugcacgguggag42
<210>18
<211>44
<212>rna
<213>人工序列(artificialsequence)
<400>18
uaauuucuacucuuguagaugcccccccaaaaucugugaucuug44
<210>19
<211>42
<212>rna
<213>人工序列(artificialsequence)
<400>19
uaauuucuacucuuguagaugcccccccaaaaucugugaucu42
<210>20
<211>44
<212>rna
<213>人工序列(artificialsequence)
<400>20
uaauuucuacucuuguagaucuguguagcuagaccaaaaucacc44
<210>21
<211>42
<212>rna
<213>人工序列(artificialsequence)
<400>21
uaauuucuacucuuguagaucuguguagcuagaccaaaauca42
<210>22
<211>43
<212>rna
<213>人工序列(artificialsequence)
<400>22
uaauuucuacucuuguagaucggcgguacacuuuagguccuuu43
<210>23
<211>41
<212>rna
<213>人工序列(artificialsequence)
<400>23
uaauuucuacucuuguagaucggcgguacacuuuagguccu41
<210>24
<211>41
<212>rna
<213>人工序列(artificialsequence)
<400>24
uaauuucuacucuuguagaucaaaaaucaucuccaaccagc41
<210>25
<211>43
<212>rna
<213>人工序列(artificialsequence)
<400>25
uaauuucuacucuuguagaucaaaaaucaucuccaaccagcug43
<210>26
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>26
ctcactacagacgcacgcta20
<210>27
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>27
ctcactacagacgcacgcta20
<210>28
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>28
cacatcagcagcctacagca20
<210>29
<211>31
<212>dna
<213>人工序列(artificialsequence)
<400>29
tccttctggccaccatgcgaagccacactga31
<210>30
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>30
cgatctgcacacaccagttgagcaggtactgg32
<210>31
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>31
tccctcacagcagggtcttctctgtttca29
<210>32
<211>34
<212>dna
<213>人工序列(artificialsequence)
<400>32
ccctggtgtcaggaaaatgctggctgacctaaag34
<210>33
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>33
cacctcagatatatttcttcatgaagacctcac33
<210>34
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>34
cctcaattcttaccatccacaaaatggatc30
<210>35
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>35
ctgcaaataatctagggtttggtgaatatagt32
<210>36
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>36
ccaactataatagtaagtatgaaacttgtttct33
<210>37
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>37
ccacacctgccactctcgctgatcctctc29
<210>38
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>38
ctctgcagctccatctgcatggcttgca28
- 还没有人留言评论。精彩留言会获得点赞!