一种植物表观遗传因子及其编码基因在氮素高效利用中的应用的制作方法
本发明涉及一种植物表观遗传因子cssdg43及其编码基因在氮素高效利用中的应用。
背景技术:
氮素是植物生长所必需的大量元素之一,氮素利用率是影响植物生长速率的重要因素。组蛋白修饰是植物氮素利用效率的重要调控因素,组蛋白修饰酶介导各种组蛋白修饰变化并且调控大量氮素利用效率相关基因的表达,从而提高植物对氮素的利用效率。因此,表观修饰酶及其编码基因在改造植物氮素利用效率方面有着广泛的应用前景。
技术实现要素:
本发明的目的是提供一种植物氮素高效利用相关蛋白cssdg43及其编码基因在植物氮素利用中的应用。
本发明提供的蛋白质,来源于茶树国家级良种福鼎大白,命名为cssdg43,为如下(a)或(b)或(c):
(a)蛋白序列为seqidno:1所示的蛋白质;
(b)与(a)具有95%以上序列相似性且与植物耐旱性相关的蛋白质;
(c)在(a)或(b)的n端或c端连接标签得到的融合蛋白质。
标签具体如表1所示。
表1标签的序列
蛋白质可人工合成,也可先合成其编码基因,再进行生物表达得到。
编码cssdg43的基因,命名为cssdg43基因,也属于本发明的保护范围。
cssdg43基因为如下(1)或(2)所述的dna分子:
(1)编码序列为seqidno:2所示的dna分子;
(2)与(1)具有75%以上序列相似性且编码植物氮素高效利用相关蛋白的dna分子;
含有cssdg43基因的表达盒、重组载体或重组菌都属于本发明的保护范围。
所述重组载体具体可为重组表达载体。可用现有的表达载体构建含有所述基因的重组表达载体。使用所述基因构建重组表达载体时,可在其转录起始核苷酸前加上任何一种增强型、组成型、组织特异型或诱导型启动子,它们可单独使用或与其它的植物启动子结合使用;此外,使用所述基因构建重组表达载体时,还可使用增强子,包括翻译增强子或转录增强子,这些增强子区域可以是atg起始密码子或邻接区域起始密码子等,但必需与编码序列的阅读框相同,以保证整个序列的正确翻译。所述翻译控制信号和起始密码子的来源是广泛的,可以是天然的,也可以是合成的。翻译起始区域可以来自转录起始区域或结构基因。为了便于对转基因植物或转基因微生物进行鉴定及筛选,可对所用表达载体进行加工,如加入在植物或微生物中表达可产生颜色变化的酶或发光化合物的基因、具有抗性的抗生素标记物或是抗化学试剂标记基因等。从转基因安全性考虑,可不加任何选择性标记基因,直接以表型筛选。
本发明还保护cssdg43蛋白的应用,为如下(i)或(ii):
(i)增强植株氮素利用率;
(ii)减弱植株氮素利用率。
本发明还保护一种植物育种方法,包括如下步骤:提高目的植物中cssdg43蛋白的活或含量,从而增强植株氮素利用率。
本发明还保护cssdg43基因在培育氮素利用率增强的转基因植物中的应用。
本发明还保护一种制备转基因植物的方法,包括如下步骤:在植物中导入cssdg43基因,得到氮素利用率增强的转基因植物。
本发明还保护一种植物育种方法,包括如下步骤:降低目的植物中cssdg43蛋白的活性或含量,从而减弱植株氮素利用率。
本发明还保护一种制备转基因植物的方法,包括如下步骤:在植物中导入抑制ssdg43基因表达的物质,得到氮素利用率减弱的转基因植物。
以上任一所述植物为双子叶植物。所述双子叶植物具体为拟南芥和茶树。所述拟南芥具体可为哥伦比亚生态型拟南芥。
本发明提供了一种与植物氮素高效利用相关的蛋白质及其编码基因,将编码基因导入植物后,植物对氮素的利用效率显著升高。本发明对于研究植物氮素利用机制,培育氮素利用率增强或减弱植物具有应用价值,对于植物氮素高效利用育种具有应用前景。
可用现有的植物表达载体构建含有所述基因的重组表达载体。
所述植物表达载体包括双元农杆菌载体和可用于植物微弹轰击的载体等。所述植物表达载体还可包含外源基因的3’端非翻译区域,即包含聚腺苷酸信号和任何其它参与mrna加工或基因表达的dna片段。所述聚腺苷酸信号可引导聚腺苷酸加入到mrna前体的3’端,如农杆菌冠瘿瘤诱导(ti)质粒基因(如胭脂合成酶nos基因)、植物基因(如大豆贮存蛋白基因)3’端转录的非翻译区均具有类似功能。
使用所述基因构建重组植物表达载体时,在其转录起始核苷酸前可加上任何一种增强型启动子或组成型启动子,此外,使用本发明的基因构建植物表达载体时,还可使用增强子,包括翻译增强子或转录增强子,这些增强子区域可以是atg起始密码子或邻接区域起始密码子等,但必需与编码序列的阅读框相同,以保证整个序列的正确翻译。所述翻译控制信号和起始密码子的来源是广泛的,可以是天然的,也可以是合成的。翻译起始区域可以来自转录起始区域或结构基因。
为了便于对转基因植物细胞或植物进行鉴定及筛选,可对所用植物表达载体进行加工,如加入可在植物中表达的编码可产生颜色变化的酶或发光化合物的基因(gus基因、萤光素酶基因等)、具有抗性的抗生素标记物(庆大霉素标记物、卡那霉素标记物等)或是抗化学试剂标记基因(如抗除莠剂基因)等。从转基因植物的安全性考虑,可不加任何选择性标记基因,直接以逆境筛选转化植株.
本发明还提供所述的基因、蛋白、重组表达载体、表达盒、转基因细胞系或重组菌中的至少一种在植物育种中的应用,特别是在培育nai形成正常或粒型变大的植物育种中的应用。
本发明还提供所述的基因、蛋白、重组表达载体、表达盒、转基因细胞系或重组菌中的至少一种在水稻育种中的应用,特别是在培育粒型形成正常或粒型变大的水稻育种中的应用。
本发明所述的氮素利用率异常的植物为氮素利用率变小的植物;所述氮素利用率正常的植物为氮素利用率正常的植物。
附图说明
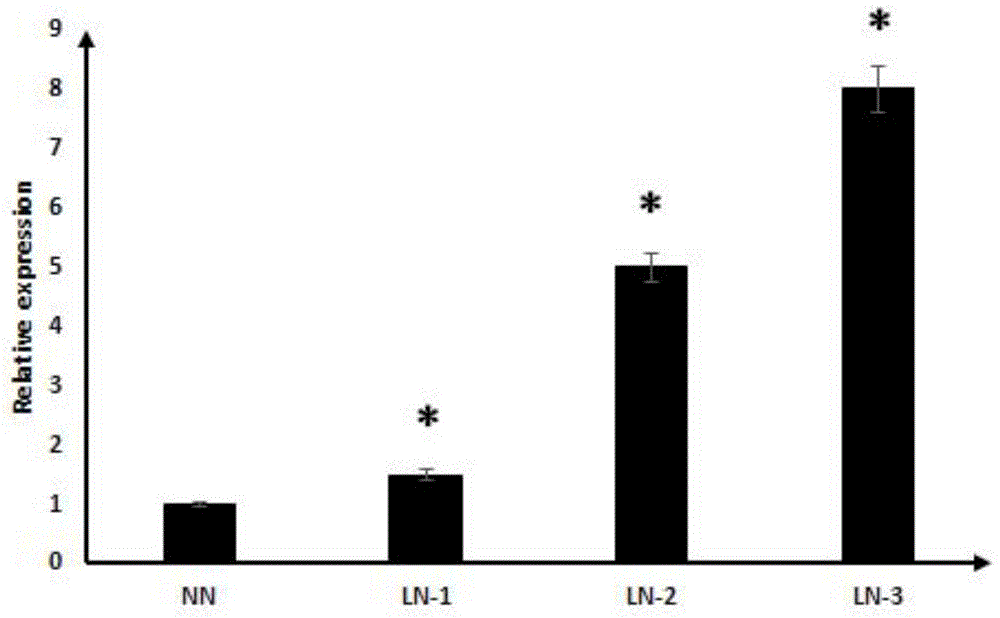
图1为cssdg43基因在正常培养(nn)和低氮(ln-1、ln-2、ln-3的氮素含量分别为正常培养的1/5、1/10、1/20)条件下的相对表达量结果,csgadph为内参基因。
图2为cssdg43蛋白的氨基酸序列。
图3为cssdg43基因的全长cds序列。
图4为纯合转基因株系半定量pcr电泳图(a)和定量pcr结果(b),野生植株(wt)和三个过表达株系(oe1、oe2、oe3)的cssdg43基因表达情况,atgadph为内参基因。
图5为野生植株(wt)和三个过表达株系(oe1、oe2、oe3)正常培养和低氮条件(正常氮素含量的1/20)下的干重统计。
图6为过表达株系(oe1)相对野生植株(wt)的叶片显著上调基因的go途径富集结果。
图7为过表达株系(oe1)相对野生植株(wt)的根部显著上调基因的go途径富集结果。
图8为高亲和性氮转运蛋白基因nrt2.1、nrt2.2、nrt2.5、nrt2.7在过表达株系(oe1、oe2、oe3)和野生植株(wt)根部的相对表达量结果,atgadph为内参基因。
具体实施方式
以下的实施例便于更好地理解本发明,但并不限定本发明。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。以下实施例中的定量试验,均设置三次重复实验,结果取平均值。
哥伦比亚生态型拟南芥又称野生型拟南芥,用wt表示。
从茶树国家级无性系良种“福鼎大白”中发现一个新蛋白,命名为cssdg43蛋白,序列如图2所示。将编码cssdg43蛋白的基因命名为cssdg43基因,开放阅读框如图3所示。
实施例1、cssdg43基因在茶树正常培养和低氮条件下的表达量分析
1、利用正常氮浓度、1/5氮浓度、1/10氮浓度、1/20氮浓度的霍格兰氏营养液培养茶树1个月,每周换一次营养液。
2、提取茶树根部的rna,反转录成cdna,以cdna为模板分析cssdg43基因在低氮条件下的表达量如图1所示。cssdg43基因在茶树低氮条件下表达量显著上调。
实施例2、转基因植物的鉴定
1、利用pcambia1300s载体上带有潮霉素抗性,在鉴定时,将已灭菌的拟南芥种子撒在含有50μg/ml潮霉素的ms培养基上,20℃黑暗处理48h,然后在温度为20℃,光周期为16h/8h(光照/黑暗)条件下培养10-12d,根茎叶长势良好的绿色植株便是转基因成功的阳性苗,将其移入营养土中继续培养。后代全为长势良好的绿色植株的株系为纯合株系。
2、提取拟南芥阳性苗的dna,以dna为模板扩增目的基因,筛选转基因阳性苗。dna提取选用ctab法,保存于-20℃,用于后续pcr反应。
3、提取纯合的t3代转基因拟南芥叶片的总rna,反转录为cdna,使用引物primer-f(tcaatgtcgtcaattaccac)和primer-r(atttcttctcaccatcctcc)检测cssdg43基因的表达量如图4所示。转基因植株中的cssdg43基因表达量显著高于野生植株。
实施例3、野生植株(wt)和三个过表达株系(oe1、oe2、oe3)在正常培养和低氮条件下的干重统计
选取野生植株(wt)和三个过表达株系(oe1、oe2、oe3)的纯合t3代转基因种子,播种于正常培养基上,正常培养14d后,分别转入霍格兰氏和低氮培养液(低氮营养液的氮素含量是霍格兰式营养液的1/20)中培养两周,统计植株干重(见图5)。转基因植株(oe1、oe2、oe3)在低氮条件(ln-1为2周换一次营养液,ln-2为3天换一次营养液)下的干重显著高于野生型植株(wt)。
实施例4、叶片显著上调表达基因(oe1vs.wt)的go途径富集结果。
选取野生植株(wt)和过表达株系(oe1)的纯合t3代转基因种子,播种于培养基上,正常培养14d后,提取叶片rna,逆转录为cdna,构建cdna文库,测序,分析显著上调表达基因(oe1vs.wt)的go途径富集情况(见图6)。光合作用相关途径显著上调。
实施例5、根部显著上调表达基因(oe1vs.wt)的go途径富集结果。
选取野生植株(wt)和过表达株系(oe1)的纯合t3代转基因种子,播种于培养基上,正常培养14d后,提取根部rna,逆转录为cdna,构建cdna文库,测序,分析显著上调表达基因(oe1vs.wt)的go途径富集情况(见图7)。器官发育和营养吸收相关途径显著上调。
实施例6、高亲和性氮转运蛋白基因nrt2.1、nrt2.2、nrt2.5、nrt2.7在过表达株系(oe1、oe2、oe3)和野生植株(wt)根部的相对表达量结果。
选取野生植株(wt)和过表达株系(oe1、oe2、oe3)的纯合t3代转基因种子,播种于培养基上,正常培养14d后,提取根部rna,逆转录为cdna,定量pcr分析高亲和性氮转运蛋白基因nrt2.1、nrt2.2、nrt2.5、nrt2.7在过表达株系(oe1、oe2、oe3)和野生植株(wt)根部的相对表达量如图8所示。过表达株系(oe1、oe2、oe3)的nrt2.1、nrt2.2、nrt2.5、nrt2.7基因表达量显著高于野生型株系(wt)。
上述说明也并不仅限于上述举例,本发明未经描述的技术特征可以通过或采用现有技术实现,在此不再赘述;以上实施例及附图仅用于说明本发明的技术方案并非是对本发明的限制,参照优选的实施方式对本发明进行了详细说明,本技术领域的普通技术人员在本发明的实质范围内所作的任何修改、等同替换、改进等也应属于本发明的权利要求保护范围。
序列表
<110>华中农业大学
中国标准化研究院
<120>一种植物表观遗传因子及其编码基因在氮素高效利用中的应用
<160>2
<170>siposequencelisting1.0
<210>1
<211>1035
<212>prt
<213>茶树(camelliasinensis)
<400>1
metileilelysargasnleulysservalmetproileleulysarg
151015
cysargvalsergluserasnglygluaspaspgluserservalasn
202530
arglyslysarglysvalasnglytyrtyrproleuhisleuleugly
354045
gluvalalavalglyvalileprophethrglytyrglyglyasplys
505560
glypheserglyilealaalaalaalaalatrpcyslysgluvalser
65707580
gluvalgluserlysleulyssergluglulysvalvallysgluleu
859095
asnasnprovalproglualaserserargthrproleuvalargthr
100105110
serargglyargvalglnvalleuproserargpheasnaspserile
115120125
leuaspasntrplyslysaspserlysserservalarggluleuser
130135140
leuaspprogluphethrprotyrlysglulyspheserphelysthr
145150155160
prolysileargglyglnilealaserlysalaargasnglyasplys
165170175
valsertyrglncysargglnleuproproleuleuglyasnglygln
180185190
valvaltyrglnlysserlysasnpheasnvalglylysglnleuser
195200205
serargserthrvalserleuleuhisgluargphevalasnalaglu
210215220
lysserproileglugluproglugluproileaspleuthraspile
225230235240
asnglyleuleuglugluaspglyglulyslyscysglyleutyrgly
245250255
leuasnaspphevalserglyaspilevaltrpalametserglylys
260265270
asntyrproalatrpproalailevalleuaspprolysserglnala
275280285
proglnglnvalleuserpheargvalthrasnalavalcysvalmet
290295300
phepheglytyrserglyasnglythrglnargasptyralatrpval
305310315320
lysargglyargilephepropheileaspphevalaspargphegln
325330335
glyglnthraspleuasnaspserlysproserasnleuargserala
340345350
ilegluglualapheleualagluglnglyphethrglumetleumet
355360365
valgluileasnalaalaalaglyasnleutyrtyrleugluserile
370375380
argglyglyvalglnglualathraspserasnglnglucysasnthr
385390395400
glnlysglnleulyslysserlyshissercysglyilecyslyslys
405410415
ileargasntyrseraspserglythrtrpvalargcysaspglycys
420425430
lysvaltrpleuhisalaalacysasplysvalserproasnpheser
435440445
lysaspleualaalathrasptyrtyrcysproglucysargalalys
450455460
pheasnphegluleuseraspsergluasncysglnprolysvallys
465470475480
asnasnlyslysasnvalleuproasplysvalilevalvalcysser
485490495
glyvalgluglyiletyrpheproserleuhisleuvalvalcyslys
500505510
cysglysercysglythrglulysglnalaleuasnglutrpgluarg
515520525
histhrglyserlysalalysasntrplysthrservalargvallys
530535540
glysermetleuproleugluglntrpmetleuglnilealaglutyr
545550555560
hisalaargthrvalvalservalasncysleulysargproserile
565570575
lysvalarglysglnlysleuleuthrpheleuglnglulystyrglu
580585590
provaltyralalystrpthrthrgluargcysalavalcysargtrp
595600605
valgluasptrpasptyrasnlysileileilecysasnargcysgln
610615620
ilealavalhisglnglucystyrglyalaargasnvallysaspphe
625630635640
thrsertrpvalcysargalacysgluserproaspilelysargglu
645650655
cyscysleucysprovallysglyglyalaleulysprothraspval
660665670
gluserleutrpvalhisvalthrcysalatrppheglnprogluval
675680685
serpheserseraspglulysmetgluproalavalglyileleuarg
690695700
ileproserhisserphevallysilecysvalilecyslysglnile
705710715720
hisglysercysthrglncyscyslyscysserthrtyrtyrhisala
725730735
valcysalaserargalaglytyrargmetgluleuhiscysleuglu
740745750
lysasnglylysglnilethrlysmetvalsertyrcysalatyrhis
755760765
argalaproasnproaspthrvalleuilemetglnthrproarggly
770775780
valpheserthrlysasnleuileglnasplyslysargthrglyser
785790795800
argleuileserserasnargleulysleuglngluproproilemet
805810815
gluileasngluilegluproasnseralaalaargcysargthrphe
820825830
lysargleulysasnlyslysthrglygluglnalaileproileala
835840845
hisargvalmetglyprocyshishisserleuseralaileglnarg
850855860
leuasnalaphearggluileglugluprolysalapheserthrphe
865870875880
arggluargleuhishisleuglnargthrgluasnaspargvalcys
885890895
pheglyargserglyilehisglytrpglyleuphealaargargasn
900905910
ileglngluglyglumetvalleuglutyrargglygluglnvalarg
915920925
argservalalaaspleuargglualaargtyrargleugluglylys
930935940
aspcystyrleuphelysileserglugluvalvalvalaspalathr
945950955960
asplysglyasnilealaargleuileasnhissercysmetproasn
965970975
cystyralaargilemetservalglyhisaspgluserargileval
980985990
leuilealalysthrasnvalseralaglyaspgluleuthrtyrasp
99510001005
tyrleupheaspproaspglucysaspgluphelysvalprocysleu
101010151020
cyslysalaproasncysarglysphemetasn
102510301035
<210>2
<211>3108
<212>dna/rna
<213>茶树(camelliasinensis)
<400>2
atgataatcaagcggaacttgaaatcggtaatgccgattctgaagcgatgcagagtcagc60
gaatcgaatggtgaagacgatgagagttccgtgaataggaagaaacggaaagttaacggc120
tattaccctctgcatcttctcggagaggtcgccgtcggtgtaattccgttcaccggctat180
ggcggcgacaaggggttttccggcatagccgcggcggcggcgtggtgtaaggaggtatca240
gaagtggaatcgaaattgaaaagtgaggagaaggtggtgaaggaattgaacaatccagta300
cccgaggcttcttcgaggactccgcttgtgaggacttctcgagggcgtgttcaggtactt360
ccttctcgattcaatgattcaattcttgataattggaagaaagatagtaaatctagtgtc420
agagaattgagtttggaccctgaattcactccttataaagagaaatttagctttaaaacc480
cctaaaattcgcggacaaatagctagtaaggcgcgtaatggggacaaagttagctatcaa540
tgtcgtcaattaccaccattgttagggaatggacaagttgtgtatcagaagtccaagaat600
tttaatgttgggaagcaattgagttctcgtagtaccgtatcattgctccatgagcgattt660
gtcaacgcagaaaaatcgccaattgaggaaccggaggaacctatagatttgactgacatt720
aatggattgttggaggaggatggtgagaagaaatgtgggttgtatggtctgaatgacttt780
gtttctggcgatatagtttgggcaatgtcagggaagaactatcctgcgtggccagctatt840
gttctcgatcccaaatcgcaagctccccagcaagttctgagttttcgggtcaccaatgct900
gtttgtgtgatgttctttggttactctggaaatggaacacaaagggactatgcttgggtt960
aaacgtggaaggatatttccgttcatagattttgtagacaggtttcagggacagacggac1020
ttgaatgatagcaagcccagcaatcttcggtctgcgatagaagaggcatttttggcagaa1080
caaggttttactgagatgttaatggtggaaattaatgcagcagctgggaatctttattat1140
ttagaatctattcgtggaggagtccaggaagctactgattcaaatcaggaatgcaacact1200
cagaagcagttaaaaaaatcaaaacattcttgtggcatttgcaagaagatcaggaattat1260
tcagacagtggcacttgggtacgctgtgatggttgtaaagtttggctgcatgcagcatgt1320
gacaaagtttcccccaatttttccaaggatttggcagccactgattattactgccctgaa1380
tgccgagcaaagttcaattttgaattatcagattcagaaaactgtcagcctaaagtcaaa1440
aataacaagaagaatgtgctgcctgacaaggttatagttgtctgctcaggtgtggaaggc1500
atatattttccaagccttcatttagttgtttgcaaatgtggatcttgtgggacagaaaaa1560
caggcacttaacgaatgggagcgacatacgggttcaaaagcaaaaaattggaagactagt1620
gtcagagtaaaaggttccatgttgccactggaacaatggatgctgcagatagcagagtat1680
catgcacgtactgttgtttctgttaattgtcttaagcgcccctcaataaaagtgcgaaag1740
cagaagttgcttacttttttgcaagagaaatatgagcctgtttatgccaagtggacaaca1800
gaacggtgtgctgtatgtagatgggttgaagactgggactacaacaaaattattatatgc1860
aacagatgccaaattgctgttcatcaagaatgctatggagcacgaaatgttaaagatttt1920
acttcatgggtttgtcgtgcatgcgagtcacctgatatcaagcgggagtgttgcctctgt1980
cctgtaaaaggaggtgctttgaagcctactgatgtcgagtcattgtgggttcatgttact2040
tgtgcttggtttcaacctgaagtttctttctcaagtgatgaaaagatggagcctgctgtt2100
ggaatcttgagaatcccatcacactcttttgtaaagatctgcgtaatctgcaagcaaatt2160
catggttcctgcacacagtgttgcaagtgttccacttattaccatgcagtatgtgcttca2220
agggcagggtatcgtatggagttgcattgcttggaaaaaaatgggaaacaaattacaaaa2280
atggtttcatattgtgcttatcacagggctccaaatcccgatactgttctaatcatgcag2340
actcctcgaggggttttttccaccaaaaaccttatccaagacaaaaagcgcactggttca2400
aggctaatttcgtcgaacagattgaaacttcaagaacctccaataatggagatcaatgag2460
attgagccaaattctgctgcaagatgtcgaacctttaaaagattgaagaataagaagaca2520
ggtgaacaggccattcccatagcccatcgagtgatgggaccttgccatcattctttaagt2580
gcaatacaaagattgaatgcattcagagaaatagaggagcctaaagcattttctactttc2640
agggaacggttgcaccacttacagagaactgagaatgatcgggtttgctttgggagatct2700
ggaattcatggatggggactctttgcacgtcgaaacatccaagaaggagaaatggttctg2760
gaatatcgtggtgagcaggttaggcgaagtgttgcagatttgagggaggcacgctaccgt2820
ttagaaggcaaagattgctatctgtttaagataagtgaagaagttgtagtagatgccaca2880
gataagggaaacattgcacgcttaatcaaccattcatgtatgcccaactgctatgcaagg2940
atcatgagtgtgggtcatgatgagagtaggattgttctaattgccaagactaacgtatct3000
gctggtgacgagctaacgtatgattacttatttgatccggatgagtgtgacgaattcaaa3060
gtcccttgcttgtgtaaagctccaaactgcagaaaattcatgaattag3108
- 还没有人留言评论。精彩留言会获得点赞!