用于核酸序列的特异性靶向的二聚蛋白质的制作方法
本申请要求2016年4月29日提交的美国临时专利申请号62/329,740的优先权,该文通过引用方式纳入本文。
背景技术:
"cas9"或"cas9核酸酶"指含cas9蛋白的rna引导的核酸酶。酶促活性cas9包含cas9的活性dna切割结构域和向导rna(grna)结合结构域。cas9核酸酶有时也称作casn1核酸酶或crispr(成簇规律间隔短回文重复序列)相关核酸酶。
crispr是适应性免疫系统,其提供针对可动遗传因子(病毒、转座因子和接合型质粒)的保护。crispr簇包含间隔子、与前述可动因子互补的序列,和靶入侵核酸。crispr簇可被转录并加工成crisprrna(crrna)。在ii型crispr系统中,前体crrna的正确加工需要反式编码小rna(tracrrna)、内源性核糖核酸酶3(rnc)和cas9蛋白。tracrrna作为向导用于核糖核酸酶3-协助的前体crrna的加工。随后,cas9/crrna/tracrrna通过内切核苷酸方式切割与间隔子互补的线性或环状dsdna靶标。不与crrna互补的靶链首先通过内切核苷酸方式被切割,随后以外切核苷酸方式由3'-5'修整。自然中,dna结合和切割通常需要蛋白质和两种rna。然而,单向导rna可经工程改造,从而将crrna和tracrrna两者的方面引入单rna物质。参见例如,jinekm.,chylinskik.,fonfarai.,hauerm.,doudnaj.a.,charpentiere.science337:816-821(2012)。cas9识别crispr重复序列中的短基序(pam或前间区序列邻近基序)以协助区分自身和非自身。cas9核酸酶序列和结构已见述于(参见例如,ferretti等,proc.natl.acad.sci.u.s.a.98:4658-4663(2001);mojica等,microbiology155:733–740(2009);deltchevae.等.,nature471:602-607(2011);和jinekm.等.science337:816-821(2012)。cas9同源序列已见述于多个物种,包括但不限于,酿脓链球菌(s.pyogenes)和嗜热链球菌(s.thermophilus)。
不论crispr是用于基因组编辑、基因组靶向还是其它应用,普遍希望crispr系统特异性地靶向感兴趣的核苷酸序列,并避免基因组中的非靶(“脱靶”)序列。例如,wang等,naturebiotechnology33:175–178(2015)显示,脱靶频率可能较高。该文章中,对于wascr-4靶标的全部整合位点的脱靶整合为88%。中靶整合仅为12%。
阿尔古(argonaute,“ago”)蛋白也可用于靶向多核苷酸序列。例如,ago蛋白普遍表达并结合至sirna或mirna,以通过使mrna去稳化或通过翻译抑制来引导转录后基因沉默。ago蛋白一般具有至少四个结构域:n末端结构域、paz结构域、mid结构域和c末端piwi结构域。参见例如,hutvagner,naturereviewsmolecularcellbiology9(1):22–32(2008)和meister,naturereviewsgenetics14:447–459(2013)。
定义
除非另有说明,本文所用的所有科技术语具有本发明所属领域普通技术人员通常所理解的含义。通常,本文所用的命名和下述细胞培养、分子遗传学、有机化学和核酸化学以及杂交中的实验室步骤均为本领域熟知和常用的。使用标准技术进行核酸和肽合成。按照本领域和各种通用参考文献所述的常规方法进行这些技术和步骤(通常参见,sambrook等,《分子克隆:实验室手册》(molecularcloning:alaboratorymanual),第2版(1989)冷泉港实验室出版社(coldspringharborlaboratorypress),纽约冷泉港(coldspringharbor,n.y.),其通过引用纳入本文),将它们引入本文全文中。本文所用的命名以及下述分析化学和有机合成中的实验室步骤均为本领域熟知且常用。
本文所用的“核酸”表示dna、rna、单链、双链、或更高度聚集的杂交基序及其任意化学修饰。修饰包括但不限于,提供引入其它电荷、极化性、氢键、静电相互作用、与核酸配体碱基或核酸配体整体的连接点和作用点的化学基团的那些修饰。这类修饰包括但不限于,肽核酸(pna)、磷酸二酯基团修饰(例如,硫代磷酸酯、甲基膦酸酯)、2'-位糖修饰、5-位嘧啶修饰、8-位嘌呤修饰、环外胺处的修饰、4-硫尿核苷的取代、5-溴或5-碘-尿嘧啶的取代、骨架修饰、甲基化、不常见的碱基配对组合如异碱基(isobase)、异胞苷和异胍(isoguanidine)等。核酸也可包含非天然碱基,如硝基吲哚。修饰还可包括3'和5'修饰,例如用荧光团(例如,量子点)或其它部分加帽。
术语“寡核苷酸”或“多核苷酸”或“核酸”可互换使用以表示对应于核糖核酸(rna)或脱氧核糖核酸(dna)聚合物的单体聚合物或其类似物。这包括核苷酸的聚合物如rna和dna及其经修饰的形式、肽核酸(pna)、锁核酸(lnatm)等。在某些应用中,该核酸可以是包含多种单体类型的聚合物,例如同时包含rna和dna亚基。
术语“多肽”、“肽”和“蛋白质”在本文中互换使用,指氨基酸残基的聚合物。该术语适用于其中一个或多个氨基酸残基是相应天然产生氨基酸的人造化学模拟物的氨基酸聚合物,以及天然产生的氨基酸聚合物和非天然产生的氨基酸聚合物。
术语“氨基酸”指天然产生的和合成的氨基酸,以及作用方式类似于天然产生氨基酸的氨基酸类似物和氨基酸模拟物。天然产生的氨基酸是由遗传密码编码的那些氨基酸,以及随后经修饰的那些氨基酸,例如,羟基脯氨酸、γ-羧基谷氨酸和o-磷酸丝氨酸。氨基酸类似物指与天然产生的氨基酸具有相同基本化学结构的化合物,即碳原子与氢原子、羧基、氨基和r基结合,如高丝氨酸、正亮氨酸、甲硫氨酸亚砜、甲硫氨酸甲基锍。这种类似物具有修饰的r基(如正亮氨酸)或修饰的肽主链,但保留了与天然产生的氨基酸基本相同的化学结构。氨基酸模拟物指结构不同于氨基酸的普通化学结构,但作用方式类似于天然产生氨基酸的化合物。
本文中氨基酸可按iupac-iub生物化学命名委员会推荐的通常已知的三字母符号或单字母符号表述。核苷酸同样可由其普遍接受的单字母代码表述。
“保守修饰变体”可应用于氨基酸和核酸序列。对于特定的核酸序列,保守修饰变体指编码相同或基本相同的氨基酸序列的核酸,或者当核酸不编码氨基酸序列时,指基本相同的序列。由于遗传密码的简并性,大量功能相同的核酸编码任何给定的蛋白质。例如,密码子gca、gcc、gcg和gcu都编码氨基酸丙氨酸。因此,在密码子指定丙氨酸的每个位置上,可将所述密码子改变成所述的任何相应密码子而不改变编码的多肽。这类核酸变异是“沉默变异”,这是保守修饰变异的一种。本文编码多肽的每种核酸序列也描述该核酸的每种可能的沉默变异。本领域技术人员应认识到,可修饰核酸中的各个密码子(除了aug和tgg,aug通常是甲硫氨酸的唯一密码子,tgg通常是色氨酸的唯一密码子),以产生功能相同的分子。因此,在所述的各序列中隐含了编码多肽的各核酸沉默变异。
至于氨基酸序列,本领域技术人员将认识到,在编码序列中改变、加入或删除一个氨基酸或较少百分数氨基酸的某一核酸、肽、多肽或蛋白质序列单独取代、缺失或添加是“保守修饰变体”,其中所述改变导致氨基酸被化学上相似的氨基酸所取代。提供功能上相似氨基酸的保守取代表是本领域熟知的。此类保守修饰的变体是本发明的多态性变体、种间同源物和等位基因的补充且并不排除它们。
术语“编码”指编码一种或多种氨基酸的多核苷酸序列。该术语无需起始或终止密码子。氨基酸序列可编码于多核苷酸序列提供的六种不同的阅读框中任一种中。
术语“启动子”指定位于转录起始上游和/或下游且涉及识别和结合rna聚合酶和其它蛋白质以起始转录的区域或序列。
“载体”指多核苷酸,其独立于宿主染色体时能够在宿主生物体中复制。优选的载体包括质粒且通常具有复制起始点。载体可包括,例如,转录和翻译终止子、转录和翻译起始序列和用于调控特定核酸表达的启动子。本发明所述的任何多核苷酸都可包含在载体内。
如果采用以下序列比较算法之一或通过手工比对和目测检查测量时,当就比较窗口或指定区域上的最大对应性而言,这些序列有特定百分数的核苷酸或氨基酸残基相同(例如在特定区域上至少60%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%相同),则序列彼此间“基本相同”。
对于序列比较,一般将一条序列用作与测试序列比较的参比序列。使用序列比较算法时,测试和参比序列均输入计算机,必要时指定子序列坐标,指定序列算法程序参数。可使用默认的程序参数,或者可指定另外的参数。然后,该序列比较算法基于程序参数计算出测试序列相对于参比序列的序列相同性百分数。
本文所用的比较窗口摂包括参考选自20-600,通常约50-200,更常见约100-150的任一数量的毗连位置的区段,其中对两个序列进行最优比对后,可将序列与具有相同数量毗连位置的参比序列作比较。比对序列的比较方法是本领域熟知的。可进行最优序列比对以作比较,例如,通过smith和waterman的局部同源性算法,adv.appl.math.2:482(1981);通过needleman和wunsch的同源性比对算法,j.mol.biol.48:443(1970);通过pearson和lipman的相似性搜索法,proc.nat’l.acad.sci.usa85:2444(1988);通过计算机执行这些算法(阿克赛勒里公司(accelrys)的威斯康星遗传学软件包(wisconsingeneticssoftwarepackage)中的gap、bestfit、fasta和tfasta),或通过手工比对和目测。
适合确定序列相同性和序列相似性百分数的算法分别是blast和blast2.0算法,分别描述于altschul等(nuc.acidsres.25:3389-402,1977)和altschul等(j.mol.biol.215:403-10,1990)。进行blast分析的软件可从国家生物技术信息中心(nationalcenterforbiotechnologyinformation)公开获得(http://ncbi.nlm.nih.gov/)。此算法包括:首先通过鉴定查询序列中长度为w的短字来鉴定高评分序列对(hsp),与数据库序列中长度相同的字比对时它们能匹配或满足一些正值的阈值评分t。t称为相邻字评分阈值(altschul等,同上)。这些初始相邻字命中(wordhit)用作启动搜索的种子,以便找到含有它们的较长hsp。只要可提高累积比对评分,该字命中在两个方向上沿各序列延伸。出现以下情况时中止字命中在各个方向上的延伸:累积比对评分比其最大获得值降低x;由于一个或多个负评分残基比对的累积,累积评分变为零或零以下;或者达到任一序列的末端。blast算法参数w、t和x决定比对的灵敏度和速度。blast程序使用的默认值为:字长(w)11,blosum62评分矩阵(参见henikoff和henikoff,proc.natl.acad.sci.usa89:10915(1989))比对(b)50,期望值(e)10,m=5,n=-4,以及比较两条链。
blast算法也对两条序列间的相似性进行统计学分析(参见例如,karlin和altschul,proc.nat’l.acad.sci.usa90:5873-5787(1993))。blast算法提供的一种相似性测量是最小概率和(p(n)),它表明两条核苷酸或氨基酸序列之间偶尔发生匹配的概率。例如,如果测试核酸与参比核酸比较时的最小概率和小于约0.2,更优选小于约0.01,最优选小于约0.001,那么认为该核酸与参比序列相似。
术语“操作性连接”指核酸表达控制序列(如启动子,或转录因子结合位点阵列)和第二核酸序列之间的功能连接,其中所述表达控制序列指导所述核酸响应所述第二序列的转录。
“表达盒”是指当导入宿主细胞时,分别导致rna或多肽的转录和/或翻译的核酸构建体。
技术实现要素:
本文提供了一种多肽构建体,其包含第一序列特异性dna结合蛋白,选自cas9蛋白或ago蛋白,连接至第二序列特异性dna结合蛋白。
在一些实施方式中,第一序列特异性dna结合蛋白是cas9蛋白。在一些实施方式中,cas9蛋白是dcas9蛋白。在一些实施方式中,cas9蛋白是核酸酶活性(即“活性”)cas9蛋白。在一些实施方式中,第一dcas9多肽连接至第二cas9多肽。在一些实施方式中,第二cas9多肽第二dcas9多肽。在一些实施方式中,第二cas9多肽是核酸酶活性cas9多肽。cas9蛋白可以是天然产生的,或其保留至少dna结合活性的变体。在一些实施方式中,cas9蛋白基本等同于天然产生的cas9蛋白。
在一些实施方式中,第一序列特异性dna结合蛋白共价连接至第二序列特异性dna结合蛋白。在一些实施方式中,第一序列特异性dna结合蛋白以翻译性融合蛋白形式共价连接至第二序列特异性dna结合蛋白。在一些实施方式中,第一序列特异性dna结合蛋白通过一个或多个二聚化结构域连接至第二序列特异性dna结合蛋白。
在一些实施方式中,第一dcas9多肽结合至第一向导rna,且第二cas9多肽结合至第二向导rna,且第一dcas9多肽通过第一向导rna与第二向导rna的相互作用连接至第二cas9多肽。在一些实施方式中,第一dcas9多肽和第二cas9多肽结合至rna,所述rna包含第一cas9引导序列;第一tracrrna序列;第二cas9引导序列;和第二tracrrna序列,其中:第一dcas多肽结合至第一tracrrna序列,且第二cas9多肽结合至第二tracr序列;或第一dcas多肽结合至第二tracrrna序列,且第二cas9多肽结合至第一tracr序列,从而第一dcas多肽和第二cas多肽通过该rna结合在一起。
在一些实施方式中,第二序列特异性dna结合蛋白包含taledna结合结构域或锌指蛋白。
在一些实施方式中,第一序列特异性dna结合蛋白是ago蛋白。在一些实施方式中,第二序列特异性dna结合蛋白包含cas9蛋白、taledna-结合结构域,或锌指蛋白。
还提供了包含如上文或本文他处所述的多肽构建体的细胞。在一些实施方式中,所述细胞是真核细胞。在一些实施方式中,细胞是人细胞。
还提供了编码如上文或本文他处所述的翻译性融合蛋白的核酸。还提供了包含所述核酸的细胞。在一些实施方式中,所述细胞是真核细胞。在一些实施方式中,细胞是人细胞。
还提供了将多肽构建体引入真核细胞的方法,所述方法包括,将所述多肽构建体引入该细胞。在一些实施方式中,多肽构建体通过电穿孔被引入该细胞。在一些实施方式中,第一序列特异性dna结合蛋白以翻译性融合蛋白形式共价连接至第二序列特异性dna结合蛋白,并且,编码该翻译性融合蛋白的核酸被引入该细胞,其中,所述多肽构建体在该细胞中表达,由此将所述多肽构建体引入该细胞。
还提供从5’到3’包含如下部分的rna:第一cas9引导序列;第一tracrrna序列;第二cas9引导序列;和第二tracrrna序列。
还提供从5’到3’包含如下部分的rna:第一cas9引导序列;第二cas9引导序列;和第一tracrrna序列,后跟第二tracrrna序列;或第二tracrrna序列,后跟第一tracrrna序列。
还提供了一种表达盒,其包含启动子,该启动子操作性地连接至编码如上所述的rna的多核苷酸。还提供一种细胞,其包含所述表达盒。在一些实施方式中,所述细胞是真核细胞。在一些实施方式中,细胞是人细胞。
还提供一种组合物,其包含:如上所述的rna;第一dcas9多肽;和第二cas9多肽。在一些实施方式中,第二cas9多肽是第二dcas9多肽。在一些实施方式中,第二cas9多肽是核酸酶活性cas9多肽。还提供了包含所述组合物的细胞。在一些实施方式中,所述细胞是真核细胞。在一些实施方式中,细胞是人细胞。
还提供了将所述组合物引入细胞的方法。在一些实施方式中,所述方法包括,将所述组合物,或其全部组分(所述rna、第一dcas9多肽和第二cas9多肽),引入所述细胞。在一些实施方式中,所述方法包括,将cas9或dcas9/向导rna/tracrrna复合物(其具有对于靶序列的活性结合活性)引入所述细胞。
附图说明
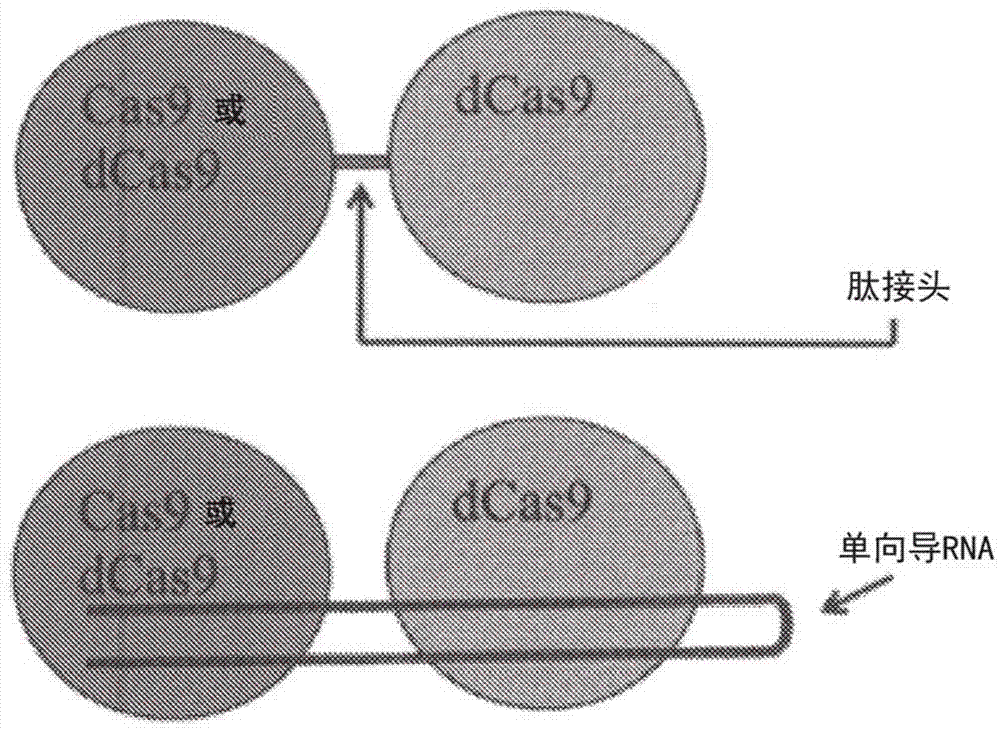
图1示意性地显示将两个cas9多肽连接在一起的两个实施方式。
图2示意性地显示将两个cas9多肽连接在一起的两个替代性的实施方式。
发明详述
绪论
本文提供了一种多肽构建体,其包含第一序列特异性dna结合蛋白(例如,选自cas9或ago蛋白),该第一序列特异性dna结合蛋白连接至第二序列特异性dna结合蛋白。当述及多肽时,所用术语“第一”和“第二”用于简单且更清楚地区分两种多肽序列,且不意在表明顺序。在一些实施方式中,第二序列特异性dna结合蛋白选自cas9多肽、ago多肽、taledna-结合结构域,或锌指。
在一些实施方式中,第一序列特异性dna结合蛋白是cas9蛋白。cas9蛋白可以是经催化死亡的cas9(“dcas9”)多肽或活性cas9多肽。在一些实施方式中,该多肽构建体包含经催化死亡的cas9(“dcas9”)多肽或活性cas9多肽,其连接至第二cas9多肽。第二cas9多肽可按需为活性的或经催化死亡的。
相较于单独的第二cas9多肽,所述的多肽构建体预期具有改善的特异性(降低的“脱靶活性)。当相应的向导rna序列靶向邻近基因组序列从而第一dcas9多肽和第二cas9多肽两者结合紧密靠近的序列时,将获得提高的特异性。所得的双重结合事件(即,第一dcas9多肽和第二cas9多肽两者靶向基因组中它们的相应序列)将大幅降低脱靶结合。
所述的多肽构建体可被应用于cas9或dcas9多肽的任何应用,包括但不限于,基因组编辑(采用活性cas9)或出于其它目的的基因组区域的靶向(例如,采用dcas9)。参见例如,maeder等.,naturemethods10:977–979(2013);gilbert等.,cell154(2):442–451(2013);hu等,nucleicacidsresearch42(7):4375–90(2014)。
第一dcas9多肽可以是缺乏催化活性的任何cas9多肽。dcas9多肽可来自任何物种,例如但不限于,来自酿脓链球菌。用于产生具有失活dna切割结构域的cas9蛋白(或其片段)的方法是已知的。参见例如,jinek等,science337:816-821(2012);qi等,cell.28;152(5):1173-83(2013)。例如,已知cas9的dna切割结构域包括两个亚结构域,hnh核酸酶亚结构域和ruvc1亚结构域。hnh亚结构域切割与grna互补的链,而ruvc1亚结构域切割非互补链。这些亚结构域中的突变可使cas9的核酸酶活性沉默。在一些实施方式中,dcas9多肽在d10和h840残基中含有突变,例如,d10n或d10a和h840a或h840n或h840y,以使蛋白质的核酸酶部分酶促失活的cas9。例如,突变d10a和h840a使酿脓链球菌cas9的核酸酶活性完全失活。示例性的dcas9序列(d10a和h840a)如下:
(seqidno:1)mdkkysiglaigtnsvgwavitdeykvpskkfkvlgntdrhsikknligallfdsgetaeatrlkrtarrrytrrknricylqeifsnemakvddsffhrleesflveedkkherhpifgnivdevayhekyptiyhlrkklvdstdkadlrliylalahmikfrghfliegdlnpdnsdvdklfiqlvqtynqlfeenpinasgvdakailsarlsksrrlenliaqlpgekknglfgnlialslgltpnfksnfdlaedaklqlskdtydddldnllaqigdqyadlflaaknlsdaillsdilrvnteitkaplsasmikrydehhqdltllkalvrqqlpekykeiffdqskngyagyidggasqeefykfikpilekmdgteellvklnredllrkqrtfdngsiphqihlgelhailrrqedfypflkdnrekiekiltfripyyvgplargnsrfawmtrkseetitpwnfeevvdkgasaqsfiermtnfdknlpnekvlpkhsllyeyftvyneltkvkyvtegmrkpaflsgeqkkaivdllfktnrkvtvkqlkedyfkkiecfdsveisgvedrfnaslgtyhdllkiikdkdfldneenediledivltltlfedremieerlktyahlfddkvmkqlkrrrytgwgrlsrklingirdkqsgktildflksdgfanrnfmqlihddsltfkediqkaqvsgqgdslhehianlagspaikkgilqtvkvvdelvkvmgrhkpeniviemarenqttqkgqknsrermkrieegikelgsqilkehpventqlqneklylyylqngrdmyvdqeldinrlsdydvdaivpqsflkddsidnkvltrsdknrgksdnvpseevvkkmknywrqllnaklitqrkfdnltkaergglseldkagfikrqlvetrqitkhvaqildsrmntkydendklirevkvitlksklvsdfrkdfqfykvreinnyhhahdaylnavvgtalikkypklesefvygdykvydvrkmiakseqeigkatakyffysnimnffkteitlangeirkrplietngetgeivwdkgrdfatvrkvlsmpqvnivkktevqtggfskesilpkrnsdkliarkkdwdpkkyggfdsptvaysvlvvakvekgkskklksvkellgitimerssfeknpidfleakgykevkkdliiklpkyslfelengrkrmlasagelqkgnelalpskyvnflylashyeklkgspedneqkqlfveqhkhyldeiieqisefskrviladanldkvlsaynkhrdkpireqaeniihlftltnlgapaafkyfdttidrkrytstkevldatlihqsitglyetridlsqlggd
在一些实施方式中,dcas9等同于或基本(例如,至少70,75,80,85,90,95%)等同于seqidno:1。
如上所示,第二cas9多肽可以是经酶促具有活性或死亡(经酶促失活)的cas9多肽。示例性的dcas9多肽如上所述。示例性的活性cas9多肽可包含,例如,基本(例如,至少70,75,80,85,90,95%)等同于来自以下物种的cas9的序列:酿脓链球菌(streptococcuspyogenes)(ncbi参考序列:nc_017053.1,seqidno:2);溃疡棒状杆菌(corynebacteriumulcerans)(ncbi参考号:nc_015683.1,nc_017317.1);白喉棒状杆菌(corynebacteriumdiphtheria)(ncbi参考号:nc_016782.1,nc_016786.1);栖蚜蝇螺原体(spiroplasmasyrphidicola)(ncbi参考号:nc_021284.1);中间普氏菌(prevotellaintermedia)(ncbi参考号:nc_017861.1);台湾螺原体(spiroplasmataiwanense)(ncbi参考号:nc_021846.1);鱼型链球菌(streptococcusiniae)(ncbi参考号:nc_021314.1);含羞草伯克霍尔德氏菌(belliellabaltica)(ncbi参考号:nc_018010.1);拓奇冷弯菌(psychroflexustorquisi)(ncbi参考号:nc_018721.1);嗜热链球菌(streptococcusthermophilus)(ncbi参考号:yp_820832.1),无害利斯特菌(listeriainnocua)(ncbi参考号:np_472073.1),空肠弯曲杆菌(campylobacterjejuni)(ncbi参考号:yp_002344900.1)或脑膜炎奈瑟氏菌(neisseriameningitidis)(ncbi参考号:yp_002342100.1)。
来自酿脓链球菌的示例性的活性cas9多肽序列为:
(seqidno:2)
mdkkysiglaigtnsvgwavitdeykvpskkfkvlgntdrhsikknligallfdsgetaeatrlkrtarrrytrrknricylqeifsnemakvddsffhrleesflveedkkherhpifgnivdevayhekyptiyhlrkklvdstdkadlrliylalahmikfrghfliegdlnpdnsdvdklfiqlvqtynqlfeenpinasgvdakailsarlsksrrlenliaqlpgekknglfgnlialslgltpnfksnfdlaedaklqlskdtydddldnllaqigdqyadlflaaknlsdaillsdilrvnteitkaplsasmikrydehhqdltllkalvrqqlpekykeiffdqskngyagyidggasqeefykfikpilekmdgteellvklnredllrkqrtfdngsiphqihlgelhailrrqedfypflkdnrekiekiltfripyyvgplargnsrfawmtrkseetitpwnfeevvdkgasaqsfiermtnfdknlpnekvlpkhsllyeyftvyneltkvkyvtegmrkpaflsgeqkkaivdllfktnrkvtvkqlkedyfkkiecfdsveisgvedrfnaslgtyhdllkiikdkdfldneenediledivltltlfedremieerlktyahlfddkvmkqlkrrrytgwgrlsrklingirdkqsgktildflksdgfanrnfmqlihddsltfkediqkaqvsgqgdslhehianlagspaikkgilqtvkvvdelvkvmgrhkpeniviemarenqttqkgqknsrermkrieegikelgsqilkehpventqlqneklylyylqngrdmyvdqeldinrlsdydvdhivpqsflkddsidnkvltrsdknrgksdnvpseevvkkmknywrqllnaklitqrkfdnltkaergglseldkagfikrqlvetrqitkhvaqildsrmntkydendklirevkvitlksklvsdfrkdfqfykvreinnyhhahdaylnavvgtalikkypklesefvygdykvydvrkmiakseqeigkatakyffysnimnffkteitlangeirkrplietngetgeivwdkgrdfatvrkvlsmpqvnivkktevqtggfskesilpkrnsdkliarkkdwdpkkyggfdsptvaysvlvvakvekgkskklksvkellgitimerssfeknpidfleakgykevkkdliiklpkyslfelengrkrmlasagelqkgnelalpskyvnflylashyeklkgspedneqkqlfveqhkhyldeiieqisefskrviladanldkvlsaynkhrdkpireqaeniihlftltnlgapaafkyfdttidrkrytstkevldatlihqsitglyetridlsqlggd
所述多肽构建体包含第一序列特异性dna结合性多肽,其连接至第二序列特异性dna结合性多肽。这两种多肽可按需连接。在一些实施方式中,这两种多肽通过肽键连接(例如,以翻译性融合体形式),其可直接融合或通过肽接头间接融合。参见例如,图1上部。示例性的肽接头序列含有gly、ser、val和thr残基。在一些实施方式中,其它的近中性氨基酸(如ala)也可用于接头序列。可有用地用作接头的氨基酸序列包括以下文献中描述的那些:maratea等(1985)gene40:39-46;murphy等(1986)proc.natl.acad.sci.usa83:8258-8262;美国专利号4,935,233和4,751,180,其各自通过引用全文纳入本文用于所有目的且尤其是所有涉及接头的教导。在一些实施方式中,接头序列的长度通常可以是1至约50个氨基酸,例如长度为3、4、6或10个氨基酸,但在一些实施方式中,长度也可以长达100或200个氨基酸。
融合体中的两种多肽的顺序可变化。因此,在一些实施方式中,第一序列特异性dna结合性多肽羧基末端直接或间接连接至第二序列特异性dna结合性多肽。在一些实施方式中,第二序列特异性dna结合性多肽羧基末端直接或间接连接至第一序列特异性dna结合性多肽。
在一些实施方式中,第一序列特异性dna结合蛋白是cas9(dcas9或活性cas9,例如如上所述),且第二序列特异性结合蛋白ago蛋白、转录活化剂样效应物核酸酶(tale)dna结合结构域,锌指。
示例性的ago多肽包括,例如,mallory,theplantcell22(12):3879-3889(2010)中所述那些。例如,ago蛋白可以是人、拟南芥、酵母或其它ago蛋白,或保留作为序列特异性dna结合蛋白的能力的其基本等同的变体。
来自超嗜热菌(a.aeolicus)(yuan等.mol.cell2005,19:405–419)的示例性的ago蛋白以seqidno:3提供:mgkeallnlyrieyrpkdttftvfkptheiqkeklnkvrwrvflqtglptfrredefwcagkvekdtlyltlsngeivelkrvgeeefrgfqnerecqelfrdfltktkvkdkfisdfykkfrdkitvqgknrkialipevnekvlkseegyfllhldlkfriqpfetlqtllerndfnpkrirvkpigidfvgrvqdvfkakekgeeffrlcxersthksskkaweellknrelrekaflvvlekgytypatilkpvltyenledeernevadivrxepgkrlnliryilrryvkalrdygwyispeeerakgklnfkdtvldakgkntkvitnlrkflelcrpfvkkdvlsveiisvsvykklewrkeeflkelinflknkgiklkikgkslilaqtreeakeklipvinkikdvdlvivfleeypkvdpyksfllydfvkrellkkxipsqvilnrtlknenlkfvllnvaeqvlaktgnipyklkeiegkvdafvgidisritrdgktvnavaftkifnskgelvryyltsypafgekltekaigdvfslleklgfkkgskivvhrdgrlyrdevaafkkygelygyslelleiikrnnprffsnekfikgyfyklsedsvilatynqvyegthqpikvrkvygelpvevlcsqilsltlxnyssfqpiklpatvhysdkitklxlrgiepikkegdixywl。
天然产生的转录活化剂样效应物(tale)是由黄单胞菌(xanthomonas)细菌分泌的蛋白质。tale的dna结合结构域包含高度保守的33-34氨基酸序列,除了第12和第13个氨基酸。这两个位置是高度可变的(重复可变双残基(rvd)),并且显示与特异性核苷酸识别的强相关性。氨基酸序列和dna识别之间的简单关系允许通过选择含有合适rvd的重复区段的组合来构建特异性dna结合结构域。参见例如,美国专利公开号2014/0256798,和美国专利号8,450,471;8,440,431和8,440,432。
来自野油菜黄单胞菌致病变种阿莫瑞斯(xanthomonascampestrispv.armoraciae)的示例性tale蛋白以seqidno:4提供如下:
mqwsgaralealltvagelrgpplqldtgqllkiakrggvtaveavhawrnaltgaplnltpeqvvaiashdggkqaletvqrllpvlcqahgltpqqvvaiashdggkqaletvqrllpvlcqahgltpeqvvaiashdggkqaletvqallpvlcqahgltpeqvvaiasngggkqaletvqrllpvlcqahgltpqqvvaiasngggkqaletvqrllpvlcqahgltpqqvvaiasngggkqaletvqrllpvlcqahgltpqqvvaiasnnggkqaletvqrllpvlcqahgltpqqvvaiasngggkqaletvqrllpvlcqahgltpqqvvaiashdggkqaletvqrllpvlcqahgltpeqvvaiasngggkqaletvqrllpvlcqahgltpeqvvaiashdggkqaletvqrllpvlcqahgltpqqvvaiasngggrpalesivaqlsrpdpalaaltndhlvalaclggrpaldavkkle
锌指是基于zc原子的存在而折叠且以序列特异性方式结合dna的蛋白质。示例性的锌指可按不同结构分类,包括但不限于,例如,cys2his2、gag-节型、高音谱号型(treble-clef)、锌带型(zincribbon)、zn2/cys6锌指蛋白。
示例性的锌指蛋白是zif268,以seqidno:5提供如下:merpyacpvescdrrfsrsdeltrhirihtgqkpfqcricmrnfsrsdhltthirthtgekpfacdicgrkfarsderkrhtkihlrqkd
在其它实施方式中,第一序列特异性dna结合蛋白是ago蛋白。在这些方面中,第二序列特异性dna结合蛋白可以是,例如,taledna-结合结构域,锌指,cas9(dcas9或活性)或第二ago蛋白。
在一些实施方式中,不同于由肽键连接,第一序列特异性dna结合蛋白和第二序列特异性dna结合蛋白通过二聚化结构域连接,即,其中第一序列特异性dna结合蛋白融合至第一二聚化结构域,而第二序列特异性dna结合蛋白连接至第二二聚化结构域,从而第一和第二二聚化结构域通过第一和第二二聚化结构域之间的共价或非共价相互作用连接这两种多肽。参见例如,图2下部。可采用任何方便的二聚化结构域组合。第一和第二二聚化结构域可以是同二聚体形式,从而它们由相同氨基酸序列构成,或异二聚体形式,从而它们由不同氨基酸序列构成。二聚化结构域可变化,其中,感兴趣的结构域包括但不限于:靶生物分子的配体,例如,特异性结合至感兴趣的具体蛋白质的配体(例如,蛋白质:蛋白质相互作用结构域),例如sh2结构域,paz结构域,ring结构域,转录活化剂结构域,dna结合结构域,酶催化结构域,酶调节结构域,酶亚基等。
示例性的二聚化结构域包括,但不限于,以下的蛋白质结构域:idimerize诱导型同二聚体(例如,dmrb)和异二聚体系统(例如,dmra和dmrc)和idimerize反向二聚化系统(例如,dmrd)(clackson等(1998)proc.natl.acad.sci.usa95(18):10437-10442;crabtree,g.r.和schreiber,s.l.(1996)trendsbiochem.sci.21(11):418-422;jin等(2000)nat.genet.26(1):64-66;castellano等(1999)curr.biol.9(7):351-360;crabtree等(1997)embo.j.16(18):5618-5628;muthuswamy等(1999)mol.cell.biol.19(10):6845-6857)。
在一些实施方式中,二聚化结构域是转录活化结构域。感兴趣的转录活化结构域包括,但不限于:h组核受体成员转录活化结构域、甾族/甲状腺激素核受体转录活化结构域、合成或嵌合转录活化结构域、多聚谷氨酰胺转录活化结构域、碱性或酸性氨基酸转录活化结构域、vp16转录活化结构域、gal4转录活化结构域、nf-κb转录活化结构域、bp64转录活化结构域、b42酸性转录活化结构域(b42ad)、p65转录活化结构域(p65ad),或其类似物、组合或修饰形式。
而在其它实施方式中,结合向导rna的第一序列特异性dna结合蛋白(例如,cas9或ago蛋白)和结合向导rna的第二序列特异性dna结合蛋白(例如,cas9或ago蛋白)通过分开的向导rna连接,所述分开的向导rna彼此相互作用(例如,杂交)。参见例如,图2上部。在这些实施方式中,第一序列特异性dna结合蛋白结合至第一向导rna,而第二序列特异性dna结合蛋白结合至第二向导rna,且第一和第二向导rna包括彼此充分相互作用(例如,杂交)的区域,从而第一序列特异性dna结合蛋白多肽和第二序列特异性dna结合蛋白连接在一起。
而在其它实施方式中,结合向导rna的第一序列特异性dna结合蛋白(例如,cas9或ago蛋白)和结合向导rna的第二序列特异性dna结合蛋白(例如,cas9或ago蛋白)通过单个延伸的向导rna连接,所述单个延伸的向导rna由第一序列特异性dna结合蛋白和第二序列特异性dna结合蛋白两者结合。参见例如,图1下部。在一些实施方式中,第一和/或第二序列特异性dna结合蛋白是cas9蛋白(dcas9或活性cas9)。在这些方面中,向导rna将包含第一引导序列和第一反式活化cr(tracr)序列,用于第一dcas9多肽,和第二引导序列和第二tracr序列,用于第二cas9多肽。在一些实施方式中,例如,所述rna将从5’到3’包含如下部分:第一引导序列,第一tracr序列,第二引导序列和第二tracr序列。在一些实施方式中,例如,所述rna将从5’到3’包含如下部分:第一引导序列,第二引导序列,第二tracr序列,和第一tracr序列。所述后者实施方式示于图1下部。
一般而言,引导序列是与靶多核苷酸序列有足够互补性以与靶序列杂交并引导crispr复合物与靶序列的序列特异性结合的任何多核苷酸序列。在一些实施方式中,当使用合适的比对算法进行最佳比对时,引导序列与其相应靶序列之间的互补程度为约或超过约50%、60%、75%、80%、85%、90%、95%、97.5%、99%或更高。在一些实施方式中,引导序列的长度是约或超过约5、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、35、40、45、50、75或更多个核苷酸。在一些实施方式中,引导序列的长度小于约75、50、45、40、35、30、25、20、15、12或更少个核苷酸。可通过任意合适的试验来评价引导序列指导crispr复合物序列特异性结合靶序列的能力。例如,可向具有对应靶序列的宿主细胞提供足以形成crispr复合物的crispr系统的组分(包括待测试的引导序列),例如通过用编码crispr序列的组分的载体进行转染,然后评估靶序列中或靶向靶序列的优先切割。类似地,可通过提供靶序列,crispr复合物的组分,包括待测试的引导序列和与测试引导序列不同的对照引导序列,并比较测试和对照引导序列反应之间在靶序列处的切割速率或结合,来体外评价靶多核苷酸序列的切割。
可选择引导序列来靶向任意靶序列。在一些实施方式中,靶序列是细胞基因组内的序列。示例性的靶序列包括在靶基因组中特有的那些。
在一些实施方式中,选择引导序列以降低引导序列内的二级结构的程度。可通过任意合适的多核苷酸折叠算法来确定二级结构。一些方案基于计算最低吉布斯自由能。一种此类算法的实例是mfold,如zuker和stiegler所述(nucleicacidsres.9(1981),133-148)。另一示例性折叠算法是线上网站服务器rnafold,由维也纳大学的理论化学学院(institutefortheoreticalchemistryattheuniversityofvienna)开发,采用心结构预测算法(参见例如a.r.gruber等,2008,cell106(1):23-24;和pacarr和gmchurch,2009,naturebiotechnology27(12):1151-62)。
模拟tracrrna序列的任何rna序列(即,即由cas9多肽识别的茎环)或以tracrrna形式行使功能的任何rna序列均为“tracr序列”。用于发夹结构的示例性环形成序列长度为4个核苷酸,并且在一些实施方式中,具有序列gaaa。然而,可使用较长或较短的环序列,替代序列也可以。所述序列可包括核苷酸三连体(例如,aaa),和额外核苷酸(例如,c或g)。环形成序列的示例包括caaa和aaag。在一个实施方式中,转录本或转录的多核苷酸序列具有至少两个或更多个发夹。在一些实施方式中,转录本具有2、3、4或5个发夹。在其它实施方式中,转录本具有至多5个发夹。在一些实施方式中,单个转录本还包括转录终止序列,如多聚t序列,例如,具有6个t核苷酸。参见例如,美国专利号8697359。
还提供包含多肽构建体的细胞,所述多肽构建体包含第一序列特异性dna结合蛋白,其连接至第二序列特异性dna结合蛋白。在一些实施方式中,所述细胞包含编码多肽构建体的多核苷酸,所述多肽构建体包含,以翻译性融合体形式直接或间接(即,通过接头)连接至第二序列特异性dna结合蛋白的第一序列特异性dna结合蛋白。示例性的细胞可包括,但不限于,原核细胞(例如,大肠杆菌),动物细胞,真菌细胞或植物细胞。示例性的动物细胞包括但不限于,哺乳动物细胞,例如,小鼠,大鼠,或人细胞。在一些实施方式中,所述多核苷酸还包括控制该多肽构建体的表达的启动子。示例性的启动子可以是组成型、诱导型,或细胞型或组织特异性启动子。
还提供了编码该多肽构建体的核酸(例如,分离的dna或rna)。在一些实施方式中,所述核酸可经密码子优化以供在细胞(包括但不限于上述那些细胞)中表达。在一些实施方式中,所述核酸还包含编码和表达序列,用于一种或多种向导rna的表达。
在一些实施方式中,所述核酸还包含通过翻译方式融合至蛋白质构建体的一个或多个核定位信号序列(nls)。nls的非限制性示例包括源自以下的nls序列:sv40病毒大t-抗原的nls,具有氨基酸序列pkkkrkv(seqidno:6);来自核质蛋白的nls(例如核质蛋白二分体nls,具有序列krpaatkkagqakkkk(seqidno:7));c-mycnls,具有氨基酸序列paakrvkld(seqidno:8)或rqrrnelkrsp(seqidno:9);hrnpaim9nls,具有序列nqssnfgpmkggnfggrssgpyggggqyfakprnqggy(seqidno:10);来自输入蛋白-α的ibb结构域的序列rmrizfknkgkdtaelrrrrvevsvelrkakkdeqilkrrnv(seqidno:11);肌瘤t蛋白的序列vsrkrprp(seqidno:12)和ppkkared(seqidno:13);人p53的序列pqpkkkpl(seqidno:14);小鼠c-abliv的序列salikkkkkmap(seqidno:15);流感病毒ns1的序列drlrr(seqidno:16)和pkqkkrk(seqidno:17);丁型肝炎病毒抗原的序列rklkkkikkl(seqidno:18);小鼠mx蛋白的序列rekkkflkrr(seqidno:19);人多聚(adp-核糖)聚合酶的序列krkgdevdgvdevakkkskk(seqidno:20);和甾族激素受体(人)糖皮质激素的序列rkclqagmnlearktkk(seqidno:21)。
可通过在细胞中表达编码蛋白质构建体的核酸而将该蛋白质构建体引入该细胞,或可将蛋白质构建直接引入该细胞。用核酸转化细胞,以及多种表达盒和表达载体是已知的。公开本将核酸引入细胞的一般方法和重组技术的基础课本包括sambrook和russell,《分子克隆,实验室手册》(molecularcloning,alaboratorymanual)(第3版,2001);kriegler,《基因转移和表达:实验室手册》(genetransferandexpression:alaboratorymanual)(1990);以及《新编分子生物学实验指南》(currentprotocolsinmolecularbiology)(ausubel等编,1994-1999))。
在一些实施方式中,蛋白质构建体,伴随或不伴随对应的向导rna,可被引入细胞。在这些实施方式的一些中,蛋白质构建体包含nls,例如,如上所述。蛋白质构建体向细胞的引入可通过如下方式进行,例如,通过注入,电穿孔,脂质递送(例如,美国专利公开号20150071903),或与多聚精氨酸的混合物(例如,kanwar等.,anticancerdrugs23(5):471-82(2012))。
还提供了rna分子,其包含用于第一dcas多肽的第一引导序列,和用于第二cas9多肽的第二向导rna,例如如上所述。此类rna分子将一般包含两个tracr序列,一个用于第一dcas9多肽以进行结合,和第二tracr序列用于第二cas9多肽以进行结合。在一些实施方式中,例如,所述rna将从5’到3’包含如下部分:第一引导序列,第一tracr序列,第二引导序列和第二tracr序列。在一些实施方式中,例如,所述rna将从5’到3’包含如下部分:第一引导序列,第二引导序列,第二tracr序列,和第一tracr序列。
还提供了编码上述rna分子的表达盒。在一些实施方式中,核酸将包含所述表达盒以及编码上述多肽构建体的表达盒。
在所附的权利要求书中,术语“一个”或“一种”是用来表示“一个(种)或多个(种)”。当术语“包含”及其各种变体例如“包括”和“含有”位于叙述步骤或要素之前的时候,是用来表示添加其它的步骤或要素是任选的,并且是非排它性的。
提供上述公开内容用于说明而非限制本发明的范围。本发明的变化形式对于本领域普通技术人员将是显而易见的并包括在所附权利要求中。通过引用将本文引用的所有发表物、数据库、网络资源、专利、专利申请和登录号全部纳入本文用于所有目的。对于通过引用纳入的此类文件与本说明书中所含公开内容相矛盾的内容,均意于以本说明书为准和/或本说明书优先于任何相矛盾的材料。
序列表
<110>生物辐射实验室股份有限公司(bio-radlaboratories,inc.)
龚小松(gong,xiaosong)
<120>用于核酸序列的特异性靶向的二聚蛋白质
<130>1047380
<150>us62/329,740
<151>2016-04-29
<160>21
<170>patentinversion3.5
<210>1
<211>1368
<212>prt
<213>酿脓链球菌(streptococcuspyogenes)
<400>1
metasplyslystyrserileglyleualaileglythrasnserval
151015
glytrpalavalilethraspglutyrlysvalproserlyslysphe
202530
lysvalleuglyasnthrasparghisserilelyslysasnleuile
354045
glyalaleuleupheaspserglygluthralaglualathrargleu
505560
lysargthralaargargargtyrthrargarglysasnargilecys
65707580
tyrleuglngluilepheserasnglumetalalysvalaspaspser
859095
phephehisargleuglugluserpheleuvalglugluasplyslys
100105110
hisgluarghisproilepheglyasnilevalaspgluvalalatyr
115120125
hisglulystyrprothriletyrhisleuarglyslysleuvalasp
130135140
serthrasplysalaaspleuargleuiletyrleualaleualahis
145150155160
metilelyspheargglyhispheleuilegluglyaspleuasnpro
165170175
aspasnseraspvalasplysleupheileglnleuvalglnthrtyr
180185190
asnglnleupheglugluasnproileasnalaserglyvalaspala
195200205
lysalaileleuseralaargleuserlysserargargleugluasn
210215220
leuilealaglnleuproglyglulyslysasnglyleupheglyasn
225230235240
leuilealaleuserleuglyleuthrproasnphelysserasnphe
245250255
aspleualagluaspalalysleuglnleuserlysaspthrtyrasp
260265270
aspaspleuaspasnleuleualaglnileglyaspglntyralaasp
275280285
leupheleualaalalysasnleuseraspalaileleuleuserasp
290295300
ileleuargvalasnthrgluilethrlysalaproleuseralaser
305310315320
metilelysargtyraspgluhishisglnaspleuthrleuleulys
325330335
alaleuvalargglnglnleuproglulystyrlysgluilephephe
340345350
aspglnserlysasnglytyralaglytyrileaspglyglyalaser
355360365
glnglugluphetyrlyspheilelysproileleuglulysmetasp
370375380
glythrglugluleuleuvallysleuasnarggluaspleuleuarg
385390395400
lysglnargthrpheaspasnglyserileprohisglnilehisleu
405410415
glygluleuhisalaileleuargargglngluaspphetyrprophe
420425430
leulysaspasnargglulysileglulysileleuthrpheargile
435440445
protyrtyrvalglyproleualaargglyasnserargphealatrp
450455460
metthrarglysserglugluthrilethrprotrpasnphegluglu
465470475480
valvalasplysglyalaseralaglnserpheilegluargmetthr
485490495
asnpheasplysasnleuproasnglulysvalleuprolyshisser
500505510
leuleutyrglutyrphethrvaltyrasngluleuthrlysvallys
515520525
tyrvalthrgluglymetarglysproalapheleuserglyglugln
530535540
lyslysalailevalaspleuleuphelysthrasnarglysvalthr
545550555560
vallysglnleulysgluasptyrphelyslysileglucyspheasp
565570575
servalgluileserglyvalgluaspargpheasnalaserleugly
580585590
thrtyrhisaspleuleulysileilelysasplysasppheleuasp
595600605
asnglugluasngluaspileleugluaspilevalleuthrleuthr
610615620
leuphegluaspargglumetileglugluargleulysthrtyrala
625630635640
hisleupheaspasplysvalmetlysglnleulysargargargtyr
645650655
thrglytrpglyargleuserarglysleuileasnglyileargasp
660665670
lysglnserglylysthrileleuasppheleulysseraspglyphe
675680685
alaasnargasnphemetglnleuilehisaspaspserleuthrphe
690695700
lysgluaspileglnlysalaglnvalserglyglnglyaspserleu
705710715720
hisgluhisilealaasnleualaglyserproalailelyslysgly
725730735
ileleuglnthrvallysvalvalaspgluleuvallysvalmetgly
740745750
arghislysprogluasnilevalileglumetalaarggluasngln
755760765
thrthrglnlysglyglnlysasnserarggluargmetlysargile
770775780
glugluglyilelysgluleuglyserglnileleulysgluhispro
785790795800
valgluasnthrglnleuglnasnglulysleutyrleutyrtyrleu
805810815
glnasnglyargaspmettyrvalaspglngluleuaspileasnarg
820825830
leuserasptyraspvalaspalailevalproglnserpheleulys
835840845
aspaspserileaspasnlysvalleuthrargserasplysasnarg
850855860
glylysseraspasnvalproserglugluvalvallyslysmetlys
865870875880
asntyrtrpargglnleuleuasnalalysleuilethrglnarglys
885890895
pheaspasnleuthrlysalagluargglyglyleusergluleuasp
900905910
lysalaglypheilelysargglnleuvalgluthrargglnilethr
915920925
lyshisvalalaglnileleuaspserargmetasnthrlystyrasp
930935940
gluasnasplysleuilearggluvallysvalilethrleulysser
945950955960
lysleuvalseraspphearglysasppheglnphetyrlysvalarg
965970975
gluileasnasntyrhishisalahisaspalatyrleuasnalaval
980985990
valglythralaleuilelyslystyrprolysleuglusergluphe
99510001005
valtyrglyasptyrlysvaltyraspvalarglysmetileala
101010151020
lyssergluglngluileglylysalathralalystyrphephe
102510301035
tyrserasnilemetasnphephelysthrgluilethrleuala
104010451050
asnglygluilearglysargproleuilegluthrasnglyglu
105510601065
thrglygluilevaltrpasplysglyargaspphealathrval
107010751080
arglysvalleusermetproglnvalasnilevallyslysthr
108510901095
gluvalglnthrglyglypheserlysgluserileleuprolys
110011051110
argasnserasplysleuilealaarglyslysasptrpasppro
111511201125
lyslystyrglyglypheaspserprothrvalalatyrserval
113011351140
leuvalvalalalysvalglulysglylysserlyslysleulys
114511501155
servallysgluleuleuglyilethrilemetgluargserser
116011651170
pheglulysasnproileasppheleuglualalysglytyrlys
117511801185
gluvallyslysaspleuileilelysleuprolystyrserleu
119011951200
phegluleugluasnglyarglysargmetleualaseralagly
120512101215
gluleuglnlysglyasngluleualaleuproserlystyrval
122012251230
asnpheleutyrleualaserhistyrglulysleulysglyser
123512401245
progluaspasngluglnlysglnleuphevalgluglnhislys
125012551260
histyrleuaspgluileilegluglnileserglupheserlys
126512701275
argvalileleualaaspalaasnleuasplysvalleuserala
128012851290
tyrasnlyshisargasplysproilearggluglnalagluasn
129513001305
ileilehisleuphethrleuthrasnleuglyalaproalaala
131013151320
phelystyrpheaspthrthrileasparglysargtyrthrser
132513301335
thrlysgluvalleuaspalathrleuilehisglnserilethr
134013451350
glyleutyrgluthrargileaspleuserglnleuglyglyasp
135513601365
<210>2
<211>2736
<212>prt
<213>酿脓链球菌(streptococcuspyogenes)
<400>2
metasplyslystyrserileglyleualaileglythrasnserval
151015
glytrpalavalilethraspglutyrlysvalproserlyslysphe
202530
lysvalleuglyasnthrasparghisserilelyslysasnleuile
354045
glyalaleuleupheaspserglygluthralaglualathrargleu
505560
lysargthralaargargargtyrthrargarglysasnargilecys
65707580
tyrleuglngluilepheserasnglumetalalysvalaspaspser
859095
phephehisargleuglugluserpheleuvalglugluasplyslys
100105110
hisgluarghisproilepheglyasnilevalaspgluvalalatyr
115120125
hisglulystyrprothriletyrhisleuarglyslysleuvalasp
130135140
serthrasplysalaaspleuargleuiletyrleualaleualahis
145150155160
metilelyspheargglyhispheleuilegluglyaspleuasnpro
165170175
aspasnseraspvalasplysleupheileglnleuvalglnthrtyr
180185190
asnglnleupheglugluasnproileasnalaserglyvalaspala
195200205
lysalaileleuseralaargleuserlysserargargleugluasn
210215220
leuilealaglnleuproglyglulyslysasnglyleupheglyasn
225230235240
leuilealaleuserleuglyleuthrproasnphelysserasnphe
245250255
aspleualagluaspalalysleuglnleuserlysaspthrtyrasp
260265270
aspaspleuaspasnleuleualaglnileglyaspglntyralaasp
275280285
leupheleualaalalysasnleuseraspalaileleuleuserasp
290295300
ileleuargvalasnthrgluilethrlysalaproleuseralaser
305310315320
metilelysargtyraspgluhishisglnaspleuthrleuleulys
325330335
alaleuvalargglnglnleuproglulystyrlysgluilephephe
340345350
aspglnserlysasnglytyralaglytyrileaspglyglyalaser
355360365
glnglugluphetyrlyspheilelysproileleuglulysmetasp
370375380
glythrglugluleuleuvallysleuasnarggluaspleuleuarg
385390395400
lysglnargthrpheaspasnglyserileprohisglnilehisleu
405410415
glygluleuhisalaileleuargargglngluaspphetyrprophe
420425430
leulysaspasnargglulysileglulysileleuthrpheargile
435440445
protyrtyrvalglyproleualaargglyasnserargphealatrp
450455460
metthrarglysserglugluthrilethrprotrpasnphegluglu
465470475480
valvalasplysglyalaseralaglnserpheilegluargmetthr
485490495
asnpheasplysasnleuproasnglulysvalleuprolyshisser
500505510
leuleutyrglutyrphethrvaltyrasngluleuthrlysvallys
515520525
tyrvalthrgluglymetarglysproalapheleuserglyglugln
530535540
lyslysalailevalaspleuleuphelysthrasnarglysvalthr
545550555560
vallysglnleulysgluasptyrphelyslysileglucyspheasp
565570575
servalgluileserglyvalgluaspargpheasnalaserleugly
580585590
thrtyrhisaspleuleulysileilelysasplysasppheleuasp
595600605
asnglugluasngluaspileleugluaspilevalleuthrleuthr
610615620
leuphegluaspargglumetileglugluargleulysthrtyrala
625630635640
hisleupheaspasplysvalmetlysglnleulysargargargtyr
645650655
thrglytrpglyargleuserarglysleuileasnglyileargasp
660665670
lysglnserglylysthrileleuasppheleulysseraspglyphe
675680685
alaasnargasnphemetglnleuilehisaspaspserleuthrphe
690695700
lysgluaspileglnlysalaglnvalserglyglnglyaspserleu
705710715720
hisgluhisilealaasnleualaglyserproalailelyslysgly
725730735
ileleuglnthrvallysvalvalaspgluleuvallysvalmetgly
740745750
arghislysprogluasnilevalileglumetalaarggluasngln
755760765
thrthrglnlysglyglnlysasnserarggluargmetlysargile
770775780
glugluglyilelysgluleuglyserglnileleulysgluhispro
785790795800
valgluasnthrglnleuglnasnglulysleutyrleutyrtyrleu
805810815
glnasnglyargaspmettyrvalaspglngluleuaspileasnarg
820825830
leuserasptyraspvalasphisilevalproglnserpheleulys
835840845
aspaspserileaspasnlysvalleuthrargserasplysasnarg
850855860
glylysseraspasnvalproserglugluvalvallyslysmetlys
865870875880
asntyrtrpargglnleuleuasnalalysleuilethrglnarglys
885890895
pheaspasnleuthrlysalagluargglyglyleusergluleuasp
900905910
lysalaglypheilelysargglnleuvalgluthrargglnilethr
915920925
lyshisvalalaglnileleuaspserargmetasnthrlystyrasp
930935940
gluasnasplysleuilearggluvallysvalilethrleulysser
945950955960
lysleuvalseraspphearglysasppheglnphetyrlysvalarg
965970975
gluileasnasntyrhishisalahisaspalatyrleuasnalaval
980985990
valglythralaleuilelyslystyrprolysleuglusergluphe
99510001005
valtyrglyasptyrlysvaltyraspvalarglysmetileala
101010151020
lyssergluglngluileglylysalathralalystyrphephe
102510301035
tyrserasnilemetasnphephelysthrgluilethrleuala
104010451050
asnglygluilearglysargproleuilegluthrasnglyglu
105510601065
thrglygluilevaltrpasplysglyargaspphealathrval
107010751080
arglysvalleusermetproglnvalasnilevallyslysthr
108510901095
gluvalglnthrglyglypheserlysgluserileleuprolys
110011051110
argasnserasplysleuilealaarglyslysasptrpasppro
111511201125
lyslystyrglyglypheaspserprothrvalalatyrserval
113011351140
leuvalvalalalysvalglulysglylysserlyslysleulys
114511501155
servallysgluleuleuglyilethrilemetgluargserser
116011651170
pheglulysasnproileasppheleuglualalysglytyrlys
117511801185
gluvallyslysaspleuileilelysleuprolystyrserleu
119011951200
phegluleugluasnglyarglysargmetleualaseralagly
120512101215
gluleuglnlysglyasngluleualaleuproserlystyrval
122012251230
asnpheleutyrleualaserhistyrglulysleulysglyser
123512401245
progluaspasngluglnlysglnleuphevalgluglnhislys
125012551260
histyrleuaspgluileilegluglnileserglupheserlys
126512701275
argvalileleualaaspalaasnleuasplysvalleuserala
128012851290
tyrasnlyshisargasplysproilearggluglnalagluasn
129513001305
ileilehisleuphethrleuthrasnleuglyalaproalaala
131013151320
phelystyrpheaspthrthrileasparglysargtyrthrser
132513301335
thrlysgluvalleuaspalathrleuilehisglnserilethr
134013451350
glyleutyrgluthrargileaspleuserglnleuglyglyasp
135513601365
metasplyslystyrserileglyleualaileglythrasnser
137013751380
valglytrpalavalilethraspglutyrlysvalproserlys
138513901395
lysphelysvalleuglyasnthrasparghisserilelyslys
140014051410
asnleuileglyalaleuleupheaspserglygluthralaglu
141514201425
alathrargleulysargthralaargargargtyrthrargarg
143014351440
lysasnargilecystyrleuglngluilepheserasnglumet
144514501455
alalysvalaspaspserphephehisargleuglugluserphe
146014651470
leuvalglugluasplyslyshisgluarghisproilephegly
147514801485
asnilevalaspgluvalalatyrhisglulystyrprothrile
149014951500
tyrhisleuarglyslysleuvalaspserthrasplysalaasp
150515101515
leuargleuiletyrleualaleualahismetilelysphearg
152015251530
glyhispheleuilegluglyaspleuasnproaspasnserasp
153515401545
valasplysleupheileglnleuvalglnthrtyrasnglnleu
155015551560
pheglugluasnproileasnalaserglyvalaspalalysala
156515701575
ileleuseralaargleuserlysserargargleugluasnleu
158015851590
ilealaglnleuproglyglulyslysasnglyleupheglyasn
159516001605
leuilealaleuserleuglyleuthrproasnphelysserasn
161016151620
pheaspleualagluaspalalysleuglnleuserlysaspthr
162516301635
tyraspaspaspleuaspasnleuleualaglnileglyaspgln
164016451650
tyralaaspleupheleualaalalysasnleuseraspalaile
165516601665
leuleuseraspileleuargvalasnthrgluilethrlysala
167016751680
proleuseralasermetilelysargtyraspgluhishisgln
168516901695
aspleuthrleuleulysalaleuvalargglnglnleuproglu
170017051710
lystyrlysgluilephepheaspglnserlysasnglytyrala
171517201725
glytyrileaspglyglyalaserglnglugluphetyrlysphe
173017351740
ilelysproileleuglulysmetaspglythrglugluleuleu
174517501755
vallysleuasnarggluaspleuleuarglysglnargthrphe
176017651770
aspasnglyserileprohisglnilehisleuglygluleuhis
177517801785
alaileleuargargglngluaspphetyrpropheleulysasp
179017951800
asnargglulysileglulysileleuthrpheargileprotyr
180518101815
tyrvalglyproleualaargglyasnserargphealatrpmet
182018251830
thrarglysserglugluthrilethrprotrpasnphegluglu
183518401845
valvalasplysglyalaseralaglnserpheilegluargmet
185018551860
thrasnpheasplysasnleuproasnglulysvalleuprolys
186518701875
hisserleuleutyrglutyrphethrvaltyrasngluleuthr
188018851890
lysvallystyrvalthrgluglymetarglysproalapheleu
189519001905
serglygluglnlyslysalailevalaspleuleuphelysthr
191019151920
asnarglysvalthrvallysglnleulysgluasptyrphelys
192519301935
lysileglucyspheaspservalgluileserglyvalgluasp
194019451950
argpheasnalaserleuglythrtyrhisaspleuleulysile
195519601965
ilelysasplysasppheleuaspasnglugluasngluaspile
197019751980
leugluaspilevalleuthrleuthrleuphegluaspargglu
198519901995
metileglugluargleulysthrtyralahisleupheaspasp
200020052010
lysvalmetlysglnleulysargargargtyrthrglytrpgly
201520202025
argleuserarglysleuileasnglyileargasplysglnser
203020352040
glylysthrileleuasppheleulysseraspglyphealaasn
204520502055
argasnphemetglnleuilehisaspaspserleuthrphelys
206020652070
gluaspileglnlysalaglnvalserglyglnglyaspserleu
207520802085
hisgluhisilealaasnleualaglyserproalailelyslys
209020952100
glyileleuglnthrvallysvalvalaspgluleuvallysval
210521102115
metglyarghislysprogluasnilevalileglumetalaarg
212021252130
gluasnglnthrthrglnlysglyglnlysasnserarggluarg
213521402145
metlysargileglugluglyilelysgluleuglyserglnile
215021552160
leulysgluhisprovalgluasnthrglnleuglnasnglulys
216521702175
leutyrleutyrtyrleuglnasnglyargaspmettyrvalasp
218021852190
glngluleuaspileasnargleuserasptyraspvalasphis
219522002205
ilevalproglnserpheleulysaspaspserileaspasnlys
221022152220
valleuthrargserasplysasnargglylysseraspasnval
222522302235
proserglugluvalvallyslysmetlysasntyrtrparggln
224022452250
leuleuasnalalysleuilethrglnarglyspheaspasnleu
225522602265
thrlysalagluargglyglyleusergluleuasplysalagly
227022752280
pheilelysargglnleuvalgluthrargglnilethrlyshis
228522902295
valalaglnileleuaspserargmetasnthrlystyraspglu
230023052310
asnasplysleuilearggluvallysvalilethrleulysser
231523202325
lysleuvalseraspphearglysasppheglnphetyrlysval
233023352340
arggluileasnasntyrhishisalahisaspalatyrleuasn
234523502355
alavalvalglythralaleuilelyslystyrprolysleuglu
236023652370
sergluphevaltyrglyasptyrlysvaltyraspvalarglys
237523802385
metilealalyssergluglngluileglylysalathralalys
239023952400
tyrphephetyrserasnilemetasnphephelysthrgluile
240524102415
thrleualaasnglygluilearglysargproleuilegluthr
242024252430
asnglygluthrglygluilevaltrpasplysglyargaspphe
243524402445
alathrvalarglysvalleusermetproglnvalasnileval
245024552460
lyslysthrgluvalglnthrglyglypheserlysgluserile
246524702475
leuprolysargasnserasplysleuilealaarglyslysasp
248024852490
trpaspprolyslystyrglyglypheaspserprothrvalala
249525002505
tyrservalleuvalvalalalysvalglulysglylysserlys
251025152520
lysleulysservallysgluleuleuglyilethrilemetglu
252525302535
argserserpheglulysasnproileasppheleuglualalys
254025452550
glytyrlysgluvallyslysaspleuileilelysleuprolys
255525602565
tyrserleuphegluleugluasnglyarglysargmetleuala
257025752580
seralaglygluleuglnlysglyasngluleualaleuproser
258525902595
lystyrvalasnpheleutyrleualaserhistyrglulysleu
260026052610
lysglyserprogluaspasngluglnlysglnleuphevalglu
261526202625
glnhislyshistyrleuaspgluileilegluglnileserglu
263026352640
pheserlysargvalileleualaaspalaasnleuasplysval
264526502655
leuseralatyrasnlyshisargasplysproileargglugln
266026652670
alagluasnileilehisleuphethrleuthrasnleuglyala
267526802685
proalaalaphelystyrpheaspthrthrileasparglysarg
269026952700
tyrthrserthrlysgluvalleuaspalathrleuilehisgln
270527102715
serilethrglyleutyrgluthrargileaspleuserglnleu
272027252730
glyglyasp
2735
<210>3
<211>706
<212>prt
<213>嗜火液菌(aquifexpyrophilus)
<220>
<221>misc_feature
<222>(214)..(214)
<223>xaa可为任何天然产生的氨基酸
<220>
<221>misc_feature
<222>(278)..(278)
<223>xaa可为任何天然产生的氨基酸
<220>
<221>misc_feature
<222>(450)..(450)
<223>xaa可为任何天然产生的氨基酸
<220>
<221>misc_feature
<222>(665)..(665)
<223>xaa可为任何天然产生的氨基酸
<220>
<221>misc_feature
<222>(689)..(689)
<223>xaa可为任何天然产生的氨基酸
<220>
<221>misc_feature
<222>(703)..(703)
<223>xaa可为任何天然产生的氨基酸
<400>3
metglylysglualaleuleuasnleutyrargileglutyrargpro
151015
lysaspthrthrphethrvalphelysprothrhisgluileglnlys
202530
glulysleuasnlysvalargtrpargvalpheleuglnthrglyleu
354045
prothrpheargarggluaspgluphetrpcysalaglylysvalglu
505560
lysaspthrleutyrleuthrleuserasnglygluilevalgluleu
65707580
lysargvalglyglugluglupheargglypheglnasngluargglu
859095
cysglngluleupheargasppheleuthrlysthrlysvallysasp
100105110
lyspheileseraspphetyrlyslyspheargasplysilethrval
115120125
glnglylysasnarglysilealaleuileprogluvalasnglulys
130135140
valleulysserglugluglytyrpheleuleuhisleuaspleulys
145150155160
pheargileglnprophegluthrleuglnthrleuleugluargasn
165170175
asppheasnprolysargileargvallysproileglyileaspphe
180185190
valglyargvalglnaspvalphelysalalysglulysglygluglu
195200205
phepheargleucysxaagluargserthrhislysserserlyslys
210215220
alatrpglugluleuleulysasnarggluleuargglulysalaphe
225230235240
leuvalvalleuglulysglytyrthrtyrproalathrileleulys
245250255
provalleuthrtyrgluasnleugluaspglugluargasngluval
260265270
alaaspilevalargxaagluproglylysargleuasnleuilearg
275280285
tyrileleuargargtyrvallysalaleuargasptyrglytrptyr
290295300
ileserprogluglugluargalalysglylysleuasnphelysasp
305310315320
thrvalleuaspalalysglylysasnthrlysvalilethrasnleu
325330335
arglyspheleugluleucysargprophevallyslysaspvalleu
340345350
servalgluileileservalservaltyrlyslysleuglutrparg
355360365
lysgluglupheleulysgluleuileasnpheleulysasnlysgly
370375380
ilelysleulysilelysglylysserleuileleualaglnthrarg
385390395400
gluglualalysglulysleuileprovalileasnlysilelysasp
405410415
valaspleuvalilevalpheleugluglutyrprolysvalasppro
420425430
tyrlysserpheleuleutyraspphevallysarggluleuleulys
435440445
lysxaaileproserglnvalileleuasnargthrleulysasnglu
450455460
asnleulysphevalleuleuasnvalalagluglnvalleualalys
465470475480
thrglyasnileprotyrlysleulysgluilegluglylysvalasp
485490495
alaphevalglyileaspileserargilethrargaspglylysthr
500505510
valasnalavalalaphethrlysilepheasnserlysglygluleu
515520525
valargtyrtyrleuthrsertyrproalapheglyglulysleuthr
530535540
glulysalaileglyaspvalpheserleuleuglulysleuglyphe
545550555560
lyslysglyserlysilevalvalhisargaspglyargleutyrarg
565570575
aspgluvalalaalaphelyslystyrglygluleutyrglytyrser
580585590
leugluleuleugluileilelysargasnasnproargphepheser
595600605
asnglulyspheilelysglytyrphetyrlysleusergluaspser
610615620
valileleualathrtyrasnglnvaltyrgluglythrhisglnpro
625630635640
ilelysvalarglysvaltyrglygluleuprovalgluvalleucys
645650655
serglnileleuserleuthrleuxaaasntyrserserpheglnpro
660665670
ilelysleuproalathrvalhistyrserasplysilethrlysleu
675680685
xaaleuargglyilegluproilelyslysgluglyaspilexaatyr
690695700
trpleu
705
<210>4
<211>493
<212>prt
<213>野油菜黄单胞菌致病变种阿莫瑞斯(xanthomonascampestrispv.armoraciae)
<400>4
metglntrpserglyalaargalaleuglualaleuleuthrvalala
151015
glygluleuargglyproproleuglnleuaspthrglyglnleuleu
202530
lysilealalysargglyglyvalthralavalglualavalhisala
354045
trpargasnalaleuthrglyalaproleuasnleuthrproglugln
505560
valvalalailealaserhisaspglyglylysglnalaleugluthr
65707580
valglnargleuleuprovalleucysglnalahisglyleuthrpro
859095
glnglnvalvalalailealaserhisaspglyglylysglnalaleu
100105110
gluthrvalglnargleuleuprovalleucysglnalahisglyleu
115120125
thrprogluglnvalvalalailealaserhisaspglyglylysgln
130135140
alaleugluthrvalglnalaleuleuprovalleucysglnalahis
145150155160
glyleuthrprogluglnvalvalalailealaserasnglyglygly
165170175
lysglnalaleugluthrvalglnargleuleuprovalleucysgln
180185190
alahisglyleuthrproglnglnvalvalalailealaserasngly
195200205
glyglylysglnalaleugluthrvalglnargleuleuprovalleu
210215220
cysglnalahisglyleuthrproglnglnvalvalalailealaser
225230235240
asnglyglyglylysglnalaleugluthrvalglnargleuleupro
245250255
valleucysglnalahisglyleuthrproglnglnvalvalalaile
260265270
alaserasnasnglyglylysglnalaleugluthrvalglnargleu
275280285
leuprovalleucysglnalahisglyleuthrproglnglnvalval
290295300
alailealaserasnglyglyglylysglnalaleugluthrvalgln
305310315320
argleuleuprovalleucysglnalahisglyleuthrproglngln
325330335
valvalalailealaserhisaspglyglylysglnalaleugluthr
340345350
valglnargleuleuprovalleucysglnalahisglyleuthrpro
355360365
gluglnvalvalalailealaserasnglyglyglylysglnalaleu
370375380
gluthrvalglnargleuleuprovalleucysglnalahisglyleu
385390395400
thrprogluglnvalvalalailealaserhisaspglyglylysgln
405410415
alaleugluthrvalglnargleuleuprovalleucysglnalahis
420425430
glyleuthrproglnglnvalvalalailealaserasnglyglygly
435440445
argproalaleugluserilevalalaglnleuserargproasppro
450455460
alaleualaalaleuthrasnasphisleuvalalaleualacysleu
465470475480
glyglyargproalaleuaspalavallyslysleuglu
485490
<210>5
<211>90
<212>prt
<213>人工序列
<220>
<223>合成锌指蛋白zif268
<400>5
metgluargprotyralacysprovalglusercysaspargargphe
151015
serargseraspgluleuthrarghisileargilehisthrglygln
202530
lyspropheglncysargilecysmetargasnpheserargserasp
354045
hisleuthrthrhisileargthrhisthrglyglulyspropheala
505560
cysaspilecysglyarglysphealaargseraspgluarglysarg
65707580
histhrlysilehisleuargglnlysasp
8590
<210>6
<211>7
<212>prt
<213>人工序列
<220>
<223>合成的sv40病毒大t抗原的核定位信号序列(nls)
<400>6
prolyslyslysarglysval
15
<210>7
<211>16
<212>prt
<213>人工序列
<220>
<223>合成的来自核质蛋白的核定位信号序列(nls)
<400>7
lysargproalaalathrlyslysalaglyglnalalyslyslyslys
151015
<210>8
<211>9
<212>prt
<213>人工序列
<220>
<223>合成的c-myc核定位信号序列(nls)
<400>8
proalaalalysargvallysleuasp
15
<210>9
<211>11
<212>prt
<213>人工序列
<220>
<223>合成的c-myc核定位信号序列(nls)
<400>9
argglnargargasngluleulysargserpro
1510
<210>10
<211>38
<212>prt
<213>人工序列
<220>
<223>合成的hrnpaim9核定位信号序列(nls)
<400>10
asnglnserserasnpheglyprometlysglyglyasnpheglygly
151015
argserserglyprotyrglyglyglyglyglntyrphealalyspro
202530
argasnglnglyglytyr
35
<210>11
<211>42
<212>prt
<213>人工序列
<220>
<223>合成的来自输入蛋白-α的ibb结构域的核定位信号序列(nls)
<400>11
argmetargileglxphelysasnlysglylysaspthralagluleu
151015
argargargargvalgluvalservalgluleuarglysalalyslys
202530
aspgluglnileleulysargargasnval
3540
<210>12
<211>8
<212>prt
<213>人工序列
<220>
<223>合成的肌瘤t蛋白的的核定位信号序列(nls)
<400>12
valserarglysargproargpro
15
<210>13
<211>8
<212>prt
<213>人工序列
<220>
<223>合成的肌瘤t蛋白的的核定位信号序列(nls)
<400>13
proprolyslysalaarggluasp
15
<210>14
<211>8
<212>prt
<213>人工序列
<220>
<223>合成的人p53的核定位信号序列(nls)
<400>14
proglnprolyslyslysproleu
15
<210>15
<211>12
<212>prt
<213>人工序列
<220>
<223>合成的小鼠c-abliv的核定位信号序列(nls)
<400>15
seralaleuilelyslyslyslyslysmetalapro
1510
<210>16
<211>5
<212>prt
<213>人工序列
<220>
<223>合成的流感病毒ns1的核定位信号序列(nls)
<400>16
aspargleuargarg
15
<210>17
<211>7
<212>prt
<213>人工序列
<220>
<223>合成的流感病毒ns1的核定位信号序列(nls)
<400>17
prolysglnlyslysarglys
15
<210>18
<211>10
<212>prt
<213>人工序列
<220>
<223>合成的丁型肝炎病毒抗原的核定位信号序列(nls)
<400>18
arglysleulyslyslysilelyslysleu
1510
<210>19
<211>10
<212>prt
<213>人工序列
<220>
<223>合成的小鼠mx蛋白的核定位信号序列(nls)
<400>19
argglulyslyslyspheleulysargarg
1510
<210>20
<211>20
<212>prt
<213>人工序列
<220>
<223>合成的人多聚(adp-核糖)聚合酶的核定位信号序列(nls)
<400>20
lysarglysglyaspgluvalaspglyvalaspgluvalalalyslys
151015
lysserlyslys
20
<210>21
<211>17
<212>prt
<213>人工序列
<220>
<223>合成的甾族激素受体(人)糖皮质激素的核定位信号序列(nls)
<400>21
arglyscysleuglnalaglymetasnleuglualaarglysthrlys
151015
lys
- 还没有人留言评论。精彩留言会获得点赞!
- 阻断UBE2M介导Neddylation修饰在肾细胞癌治疗中的应用的制造方法与工艺
- 无载体共组装肿瘤靶向抗癌纳米药物及其制备方法与应用与流程
- 一种用于判断靶向单克隆抗体治疗肿瘤疗效的预测分子的制造方法与工艺
- 一种靶向CD47的免疫检查点抑制剂药物组合物及其制备方法与流程
- 具有淋巴靶向功能的生物自组装纳米晶注射剂及制备方法与流程
- 用于检测肝癌的基因标志物及其用途的制造方法与工艺
- 特异性shRNA筛选及其靶向Ang‑2基因抑制肺癌细胞的验证方法与流程
- 一种多功能自动化生物细胞提取分离控制系统及采用该系统实现的细胞提取分离方法与流程
- 一种融合蛋白及光敏剂复合物及其制备方法和应用与流程
- 具有可视化和显示光斑边界功能的光动力治疗系统的制造方法与工艺