猪HOXA1基因的两个CDS区及其检测方法与流程
本发明属于生物遗传学领域,涉及猪HOXA1基因的两个CDS区及其检测方法。
背景技术:
HOXA1基因是HOX同源异型盒基因家族中的一员。HOX基因高度保守,在染色体上成簇排列,是脊椎动物生长发育和细胞分化的主控基因。其中,HOXA1基因在基因簇中的位置靠近染色体的3’端,是最早表达的HOX基因,主要调控体轴近端的发育。近年来研究发现此基因与癌症的发生密切相关。
人和小鼠的HOXA1基因均具有一个可变CDS区,而猪仅有一个预测的可变CDS区。在小鼠的参考基因组(GRCm38.p4)上,该基因的CDS区和可变CDS区分别有1011 bp和417 bp碱基,分别编码336和138个氨基酸;在人的参考基因组(GRCh38.p5)上,该基因的CDS区和可变CDS区分别有1008 bp和414 bp碱基,分别编码335和137个氨基酸;而在猪的基因组(Sscrofa10.2)上,该基因的CDS区含有1011 bp碱基,编码336个氨基酸,预测的CDS区含有417 bp碱基,编码138个氨基酸。
利用小鼠在体外进行HOXA1可变CDS区的研究发现,可变CDS区编码的短蛋白可以通过影响正常HOXA1与pbx的结合能力,从而最终影响正常HOXA1的功能。同样,人的HOXA1可变CDS区编码的蛋白可导致人罹患BSA和ABDS综合征。BSAS综合征患者表现为眼睛水平凝视障碍和智力发育迟缓等,ABDS综合征的患者常出现肺换气不足,这一疾病若不经过医学干预,对人将是致命的。
技术实现要素:
本发明的目的在于为研究HOXA1基因提供新的可变CDS区,进而用于HOXA1基因对生长发育和癌症发生的机理研究。
为实现上述目的,本发明采用以下技术方案:
猪的两个CDS区,第一个CDS区的序列如SEQ ID NO:2所示,共372bp,与猪HOXA1基因的CDS区,如SEQ ID NO:1所示的序列相比:第一个CDS区5’端第213-238位碱基缺失,缺失了26个碱基;5’端第253-271位碱基缺失,缺失了19个碱基;5’端第358-560位碱基缺失,缺失了203个碱基,共缺失248个碱基;第二个CDS区的序列如SEQ ID NO:3所示,共183bp,与猪HOXA1基因的CDS区如SEQ ID NO:1所示的序列相比:第二个CDS区5’端的第124-560位碱基缺失,共缺失437个碱基。
所述第一个CDS区翻译的蛋白序列如SEQ ID NO:5所示,与猪HOXA1蛋白质序列如SEQ ID NO:4所示相比,SEQ ID NO:5编码122个氨基酸,且只有前71个氨基酸与SEQ ID NO:4相同,从第72位氨基酸开始发生改变,第72-122位氨基酸变为如SEQ ID NO:9所示蛋白序列;第二个CDS区翻译的蛋白序列如SEQ ID NO:6所示,编码60个氨基酸,与猪HOXA1蛋白质序列如SEQ ID NO:4所示的序列相比,SEQ ID NO:6编码60个氨基酸,且只有前41个氨基酸与SEQ ID NO:4相同,从第42位氨基酸开始发生改变,第42-60位氨基酸变为SEQ ID NO:10。
猪HOXA1基因的两个CDS区的检测方法,所述检测方法为PCR反应,反应体系:300 ng猪基因组cDNA,2×Taq PCR Mix 7.5μL,正向引物40 pmol,反向引物40 pmol,加水补足15μL;反应程序:94 ℃ 5 min;94 ℃ 30 s,68-54 ℃每个循环降1℃ 40s,72 ℃ 1 min,14个循环;94 ℃ 30 s,55 ℃ 40 s,72 ℃ 1 min,共25个循环;最后在72 ℃延伸8 min。
所述检测方法中采用的正向引物如SEQ ID NO:7所示、反向引物如SEQ ID NO:8所示。
本发明的有益效果在于:本发明对猪肾脏组织cDNA的扩增HOXA1基因全长转录本时发现,存在除已预测过的CDS区之外的两种新的CDS区域。这两个新的CDS区可通过降低已有HOXA1蛋白与其辅因子pbx1的结合能力,从而降低HOXA1的靶基因HOXB1的转录水平,最终影响有机体的体轴骨骼、神经和内脏器官原基的分化发育和癌细胞的增生。即,这两个新的CDS区可在分子和细胞水平上,用于挖掘影响生长发育及癌症发生的基因网络,探究该网络内基因间的互作。
附图说明
图1为肾脏组织RNA电泳图;其中1-6泳道分别表示6个个体,M代表DNA ladder。
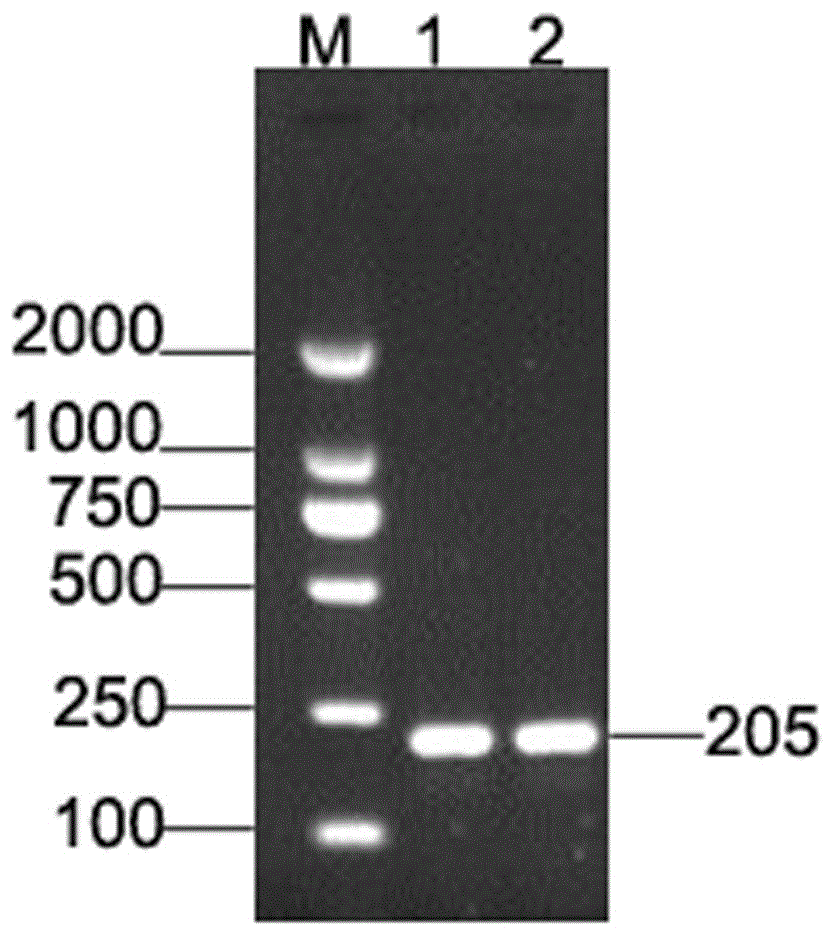
图2为肾脏cDNA的内参基因GAPDH的PCR扩增电泳图;其中PCR扩增的产物大小为205bp,1-2泳道分别表示2个个体,M表示DNA ladder。
图3为HOXA1基因的PCR凝胶电泳图。
图4为HOXA1基因第一种新的CDS区的测序图。
图5为HOXA1基因第二种新的CDS区的测序图。
具体实施方式
下面结合具体实施例,对本发明进行解释说明:
首先,利用Trizol提取猪肾脏组织的总RNA,提取结果如图1所示。取1ug RNA,利用Takara反转录试剂盒PrimeScript® 1st Strand cDNA Synthesis Kit,将RNA反转录为cDNA。利用内参基因GAPDH进行PCR扩增,检验cDNA的质量,扩增产物大小为205bp,扩增结果如图2所示。
其次,登陆网站Ensembl(http://asia.ensembl.org/index.html),下载猪HOXA1基因的全长转录本。利用软件Primer 3.0进行引物设计,正向引物如SEQ ID NO:7所示、反向引物如SEQ ID NO:8所示。15 μL的聚合酶链式反应(PCR)体系包括300 ng猪基因组cDNA,2×Taq PCR Mix 7.5μL,正反向引物各40 pmol,水5μL。引物的扩增条件为94 ℃ 5 min;94 ℃ 30 s,68 ℃(每个循环减1 ℃)40s,72 ℃ 1 min,14个循环;94 ℃ 30 s,55 ℃ 40 s,72 ℃ 1 min,共25个循环;最后在72 ℃延伸8 min。PCR产物见图3,与正常HOXA1相比,含有已预测的CDS区、两种新CDS区的cDNA,分别比含有全长CDS区的cDNA短293bp和248bp、437bp。猪的两个CDS区,第一个CDS区的序列如SEQ ID NO:2所示,共372bp,与猪HOXA1基因的CDS区,如SEQ ID NO:1所示的序列相比:第一个CDS区5’端第213-238位碱基缺失,缺失了26个碱基;5’端第253-271位碱基缺失,缺失了19个碱基;5’端第358-560位碱基缺失,缺失了203个碱基,共缺失248个碱基;第二个CDS区的序列如SEQ ID NO:3所示,共183bp,与猪HOXA1基因的CDS区如SEQ ID NO:1所示的序列相比:第二个CDS区5’端的第124-560位碱基缺失,共缺失437个碱基。
所述第一个CDS区翻译的蛋白序列如SEQ ID NO:5所示,与猪HOXA1蛋白质序列如SEQ ID NO:4所示相比,SEQ ID NO:5编码122个氨基酸,且只有前71个氨基酸与SEQ ID NO:4相同,从第72位氨基酸开始发生改变,第72-122位氨基酸变为如SEQ ID NO:9所示蛋白序列;第二个CDS区翻译的蛋白序列如SEQ ID NO:6所示,编码60个氨基酸,与猪HOXA1蛋白质序列如SEQ ID NO:4所示的序列相比,SEQ ID NO:6编码60个氨基酸,且只有前41个氨基酸与SEQ ID NO:4相同,从第42位氨基酸开始发生改变,第42-60位氨基酸变为SEQ ID NO:10。
将PCR扩增的每条产物带分别进行割胶回收,回收后,连接质粒,进行克隆测序,。
第一种新CDS区的测序结果见图3。图3(A)的测序结果显示,与家猪基因组(Sscrofa10.2)上HOXA1的CDS区(1011个bp,如SEQ ID NO:1所示)相比,在5’端第213-238位碱基缺失、第253-271位碱基缺失;图3(B)的测序结果显示第358-560位碱基缺失,共缺失248个碱基。
第二种新CDS区的测序结果见图4。测序结果显示,与家猪基因组(Sscrofa10.2)上HOXA1的CDS区(1011 bp,如SEQ ID NO:1所示)相比,在5’端第124-560位碱基缺失,共缺失437个碱基。
- 还没有人留言评论。精彩留言会获得点赞!