一种基于宿主‑肠道微生物互作基因的猪粪便污染特异性分子标记1‑38及其检测方法与流程
本发明涉及猪粪便污染物检测领域,尤其涉及一种基于宿主-肠道微生物互作基因的猪粪便污染特异性分子标记1-38及其检测方法。
背景技术:
猪肉制品作为深受国内消费者喜爱的肉制品之一,其庞大的需求量导致生猪产业迅猛发展。据农业部统计,我国生猪出栏量近年来一直占世界第一,占比维持在56%左右,并保持增长态势。由于缺乏对迅猛发展的集约化畜禽养殖业排放的有效管理,猪等畜禽粪便及其污水多以非点源的方式进入环境,引发污染并诱发健康威胁。猪粪便排泄物中不仅含有大量的病原微生物菌(戊型肝炎病毒、沙门氏菌(Salmonella)、李斯特菌(Listeria)、空肠弯曲菌(Campylobacter spp.),直接威胁到人类健康,而且含有氮,磷等物质,未经有效处理任意排放,会造成土壤,地下水,地表水及灌溉水的污染,并使水体富营养化,进而导致水产品蓄积大量的有害物质,影响水产品品质。因此,建立一种灵敏度高、特异性强并能高效指示粪便污染源的监测方法显得尤为迫切。
有研究从宿主粪便源菌群如厚壁菌门(Frimicutes)、双歧杆菌属(Bifidobacteria spp)、史氏产甲烷杆菌(Methanobrevibacter smithii)等的16S rRNA和毒力基因等筛选标签来判别污染来源。目前已鉴定的猪特异性分子标记大多基于猪肠道优势菌16S rRNA的保守区。也有从粪便中线粒体DNA、大肠埃希菌中筛选毒力基因[来判断猪粪便污染来源。然而,尽管上述特异性分子标记具有一定的特异性和灵敏性,但存在较大的局限性:1)宿主特异性不够高,2)不同宿主间存在交叉反应性,3)有地理地域差异性,4)判别动物来源的精确性不够高,因此会带来较高的错判率。
肠道微生物群落与其宿主是共同进化的,经宿主和肠道微生物之间强烈选择和协同进化可形成宿主-微生物相互作用的对宿主有益的功能保留区(互作靶点),这些互作靶点是形成肠道微生物多样性及特异性的重要因素。因此越来越多的研究从人、牛、鸡等的肠道微生物群落中筛选宿主-微生物互作基因作为宿主特异性分子标记,并证明此类分子标记具有较高的特异性和灵敏性。然而,目前尚未见以宿主-微生物互作基因作为特异性分子标记进行水体或食品中猪粪便非点源污染示踪的相关研究。
技术实现要素:
为了解决上述的技术问题,本发明的一个目的是提供一种基于宿主-肠道微生物互作基因的猪粪便污染特异性的分子标记1-38,本发明的另外一个目的是提供采用上述的分子标记1-38的检测方法,本发明针对猪特异性互作基因设计分子标记,建立了灵敏度高、特异性强并能高效指示粪便污染源的监测方法。
为了实现上述的第一个目的,本发明采用了以下的技术方案:
一种基于宿主-肠道微生物互作基因的猪粪便污染特异性分子标记1-38,其该分子标记由以下的引物和探针构成:
上游引物:GGAGGTGGTTAAGCCGATATGTT
下游引物:GCCCCTTTCTTGATACTTTGGA
探针:Fam-AAACTGATTGGAGAAGAATACAGGCG-Tam。
为了实现上述的第二个目的,本发明采用了以下的技术方案:
一种基于宿主-肠道微生物互作基因的猪粪便污染的检测方法,该方法采用所述的引物和探针与被检测样品混合,进行荧光PCR反应,在每个循环延伸结束后读数;样品无Ct值并且无扩增曲线,表示样品中无猪粪便污染;样品扩增Ct值≤35,且出现典型的扩增曲线,表示样品中存在猪粪便污染;样品Ct值>35的样本建议重做,重做结果无Ct值者为阴性,否则为阳性。
作为优选,所述的引物和探针浓度分别为0.25μM和0.3μM。
作为优选,所述的荧光PCR反应条件为:95℃预变性30s,40个循环的95℃变性5s和60℃退火延伸30s。
本发明采用竞争性杂交基因片段富集方法(GFE)富集猪特异性宏基因组,构建宏基因文库,对文库进行序列菌群及功能分类,从而靶向筛选了宿主(猪)-肠道微生物互作靶点有关特异性基因,并针对猪特异性互作基因设计分子标记,建立了灵敏度高、特异性强并能高效指示粪便污染源的监测方法。
附图说明
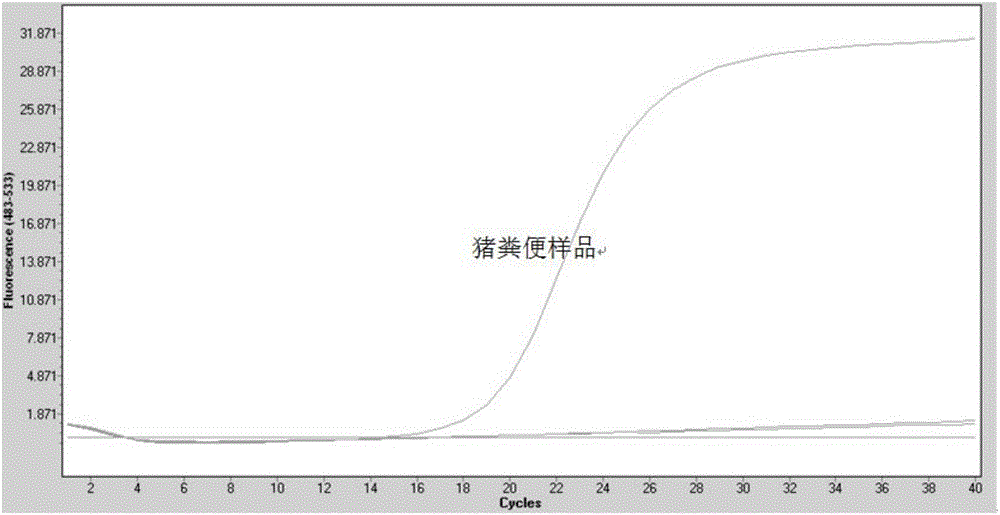
图1为分子标记1-38荧光PCR检测图谱。
图2为分子标记3-53荧光PCR检测图谱。
图3为分子标记1-38荧光PCR体系的标准曲线及扩增效率图。
图4为分子标记3-53荧光PCR体系的标准曲线及扩增效率图。
图5为分子标记1-38荧光PCR批间稳定性试验图。
图6为分子标记3-53荧光PCR批间稳定性试验图。
具体实施方式
下面结合附图对本发明的具体实施方式做一个详细的说明。
实施例1特异性猪-肠道微生物互作基因的富集筛选
1.1共采集6个物种145个粪便样品,其中包括猪粪便样品34个、奶牛牛粪便样品20份、山羊粪便样品12份、绵羊粪便样品8份,鸡粪便样品7份、鸭粪便样品20份、鹅粪便样品5份和人粪便样品13份。将样品置于无菌容器中,每200mg(湿重)样品中加入3mL GITC缓冲液(5M异硫氰酸胍、100Mm EDTA(pH 8.0)、十二烷基肌氨酸钠),低温保存。粪样DNA的提取按 Fast DNA Stool Kit(Qiagen,Valencia,CA)说明书进行。并在紫外分光光度计NanoDrop ND-2000UV上测定其浓度和纯度(NanoDrop Technologies,Thermofisher)。
1.2宿主(猪)特异性基因组富集(GFE)
将所有单个猪源样品基因组DNA混合形成基因组混合库以增加基因组的多样性(待富集组),以牛、羊、鸡、鸭、鹅等其它物种样品基因组混合库作为对照组。
1.2.1富集组DNA的准备
将待富集组又分为两个组(待富集组A和B)。
A组DNA的准备:将DNA声波震荡裂解后以Klenow I聚合酶标记上K9标签;被K9标签标记的DNA片段用 Multiwell PCR Purification Kit(Qiagen,Valencia,CA)纯化并扩增以获得足够量的带有K9随机引物的DNA片段。
B组DNA的准备(生物素标记DNA片段):将DNA经声波降解后标记生物素(PAB,Sigma-Aldrich,Atlanta,GA)。
1.2.2DNA预杂交及竞争性杂交
将待富集组B与对照组DNA分别变性后进行预杂交,随后将此预杂交混合液与A组猪源DNA竞争性杂交后,形成各种组合的DNA双链。经生物素-链亲和素(Streptavidin)酶联免疫吸附捕获试验,将标记有生物素的杂交双链DNA(即至少含一条待富集组单链的双链DNA)捕获并扩增富集。终将获得高丰度的猪源特异性粪样基因组DNA样品。
1.3构建宏基因组文库
将上述富集过程中每一轮的PCR产物经 Multiwel l PCR Purification Kit(Qiagen,Valencia,CA)进行纯化,按照pCR TOPO 4.0(Invitrogen)说明书将PCR纯化产物克隆至载体,并转入感受态大肠杆菌DH5α中。阳性克隆用含有有氨苄青霉素(50μg/mL)的LB固体培养基筛选获得,鉴定后随机挑选500个重组菌送上海生工股份有限公司进行测序。利用DNAstar对每轮所得序列进行拼接、筛选出382条猪源特异性非冗余序列,以构建宏基因组文库。
1.4DNA序列分析
在GeneBank数据库中利用BLASTX比对分析,根据相似序列的生物功能对每条非冗余序列进行蛋白质功能预测,其中E值≤10-3,识别率≥30%的序列被认为是相似蛋白序列。通过蛋白相邻类聚簇(COG)数据库将所得DNA序列进行功能基因分类。根据在GeneBank数据库BLASTX比对的结果(最小E值)对富集的非冗余序列进行菌群分类。最终筛选出60条与信息贮存与加工、细胞信息传导和代谢有关基因等可能的猪特异性互作靶点。
1.5猪-微生物特异性互作基因的鉴定
分别针对60条可能的特异性互作靶点设计引物进行鉴定,结果表明其中基因1-38和基因3-53具有猪源特异性,而不能扩增其它物种的粪便DNA。
基因1-38:
AAATACTAATGGATAAAATAGAAATAATCTCCGGCAAACTCGGAATTGAGCACCGGAAGGTAGCCAACACCGTAAAACTGCTTGAAGATGGCGCTACAGTGCCATTTATCTCGCGATACCGCAAAGAAGCAACCGGCTCACTCGATGAAGTTGCCATCATGAATATCAGCACGCTCTTGGGACAACTCGAAGAATTGGACAAGAGGCGTCGTTACATCCTCGAAAGCATTGAGGCAAGCGGTGCCTTGACTCCGGAATTGAATTCACGCATTATGGTGTGCGATGATGCAACGACACTCGAGGATATCTACCTCCCATTCAAGCCCAAACGCCGCACCAAGGCGGAAGTGGCACGAAACAACGGTCTCGAACCGTTGGCAAAAATAATTATGGCACAAAAATCGATCGATATTCATTCTCAAGCCGGTCGTTTTATTGGGGAAAATGTGCCTGACGAA。
基因3-53:
AAATACTAATGGATAAAATAGAAATAATCTCCGGCAAACTCGGAATTGAGCACCGGAAGGTAGCCAACACCGTAAAACTGCTTGAAGATGGCGCTACAGTGCCATTTATCTCGCGATACCGCAAAGAAGCAACCGGCTCACTCGATGAAGTTGCCATCATGAATATCAGCACGCTCTTGGGACAACTCGAAGAATTGGACAAGAGGCGTCGTTACATCCTCGAAAGCATTGAGGCAAGCGGTGCCTTGACTCCGGAATTGAATTCACGCATTATGGTGTGCGATGATGCAACGACACTCGAGGATATCTACCTCCCATTCAAGCCCAAACGCCGCACCAAGGCGGAAGTGGCACGAAACAACGGTCTCGAACCGTTGGCAAAAATAATTATGGCACAAAAATCGATCGATATTCATTCTCAAGCCGGTCGTTTTATTGGGGAAAATGTGCCTGACGAA。
实施例2分子标记的筛选和反应条件
采用竞争性杂交基因片段富集(GFE)方法建立猪特异性DNA基因文库。将文库克隆至pCR TOPO 4.0后,随机选取500个克隆进行测序分析,共获得382条非冗余猪源特异性序列。经BLAXTS比对分析发现其中60条非冗余序列与拟杆菌群(Bacteroidetes)、梭状杆菌群(Clostridials)等表面蛋白、膜分泌蛋白及碳水化合物代谢蛋白等宿主-微生物互作蛋白有关,可作为猪特异性分子标记筛选的靶点。针对这60条基因设计分子标记,经筛选后发现其中两套可作为猪源粪便污染失踪的分子标记(表1)。
表1分子标记序列
以阳性DNA为模板,多次重复性试验发现,分子标记1-38引物和探针浓度分别为0.2μM和0.3μM、3-53引物和探针浓度分别为0.25μM和0.3μM时,两套检测体系分别具有较高的扩增效率和敏感性。因此,20荧光PCR反应体系如下:
荧光PCR反应条件为:95℃预变性30s,40个循环的95℃变性5s和60℃退火延伸30s,在每个循环延伸结束后读数。图1为分子标记1-38荧光PCR检测图谱,图2为分子标记3-53荧光PCR检测图谱。样品无Ct值并且无扩增曲线,表示样品中无猪粪便污染;样品扩增Ct值≤35,且出现典型的扩增曲线,表示样品中存在猪粪便污染;样品Ct值>35的样本建议重做,重做结果无Ct值者为阴性,否则为阳性。
实施例3标准曲线和体系重复性
将1-38和分子标记3-53两种阳性DNA标准10倍倍比稀释至1.2×108至1.2×101拷贝/μl后分别进行荧光PCR反应,每个稀释度3个重复,绘制标准曲线计算扩增效率。结果表明两套分子标记均有较广的扩增线性范围和高扩增效率,分别为1.07和0.95(图3和图4)。同时,分子标记1-38和3-53的最低检测限分别为600拷贝/反应和60拷贝/反应,表明两套检测体系均具有良好的检测灵敏度。
对倍比稀释的两种阳性标准DNA按荧PCR分别进行10个重复检测,分子标记1-38和3-53反应体系的Ct值标准差分别在0.01-0.28和0.03-0.76之间,变异系数均低于2.17%(表2),表明两套基于特异性分子标记的荧光PCR均具有很高的重复性。
表2两种分子标记荧光PCR体系重复性试验
实施例4批间稳定性试验
为分析两套分子标记荧光PCR方法的稳定性,分别进行4次独立检测,每次试验时将阳性标准DNA倍比稀释至1.2×108至1.2×101拷贝/μl,每个稀释度设置4个重复,计算不同稀释度平均CT值及4次独立试验的标准差。结果表明分子标记1-38和3-53荧光PCR体系均具有良好的检测稳定性,其不同批次间检测Ct值标准差分别仅为0.32-1.56和0.07-0.51(图5和图6)。
实施例5实际样品检测的宿主特异性和敏感性
采集猪、奶牛、山羊、绵羊、鸡、鸭、鹅、狗和人粪便,提取DNA后对本发明中的猪特异性分子标记及其检测体系进行特异性(S)和敏感性验证(R)。荧光PCR反应体系和条件同上。并对检测结果按公式R=a/(a+b)和S=c/(c+d)计算,其中a和b分别代表分子标记针对特异性宿主的阳性样品数和假阴性样品数,c和d分别代表分子标记针对其它物种检测的阴性样品数和假阳性样品数。结果表明针对72份猪粪便样品或养殖场污水以及79份其它物种的粪便样品,分子标记1-38荧光PCR检测体系具有85%的宿主特异性和94%的宿主敏感性;3-53检测体系具有90%的宿主特异性和99%的宿主敏感性(表3),表明两套分子标记在实际应用中能高效特异的检测样品中是否含有猪粪便污染。
表3分子标记对实际样品检测的宿主特异性和敏感性
<110>浙江省检验检疫科学技术研究院;浙江工商大学
<120>一种基于宿主-肠道微生物互作基因的猪粪便污染特异性分子标记1-38及其检测方法
<160>8
<210>1
<211>458
<212>DNA
<213>猪-肠道微生物
<400>1
AAATACTAAT GGATAAAATA GAAATAATCT CCGGCAAACT CGGAATTGAG CACCGGAAGG 60
TAGCCAACAC CGTAAAACTG CTTGAAGATG GCGCTACAGT GCCATTTATC TCGCGATACC 120
GCAAAGAAGC AACCGGCTCA CTCGATGAAG TTGCCATCAT GAATATCAGC ACGCTCTTGG 180
GACAACTCGA AGAATTGGAC AAGAGGCGTC GTTACATCCT CGAAAGCATT GAGGCAAGCG 240
GTGCCTTGAC TCCGGAATTG AATTCACGCA TTATGGTGTG CGATGATGCA ACGACACTCG 300
AGGATATCTA CCTCCCATTC AAGCCCAAAC GCCGCACCAA GGCGGAAGTG GCACGAAACA 360
ACGGTCTCGA ACCGTTGGCA AAAATAATTA TGGCACAAAA ATCGATCGAT ATTCATTCTC 420
AAGCCGGTCG TTTTATTGGG GAAAATGTGC CTGACGAA 458
<210>2
<211>458
<212>DNA
<213>猪-肠道微生物
<400>2
AAATACTAAT GGATAAAATA GAAATAATCT CCGGCAAACT CGGAATTGAG CACCGGAAGG 60
TAGCCAACAC CGTAAAACTG CTTGAAGATG GCGCTACAGT GCCATTTATC TCGCGATACC 120
GCAAAGAAGC AACCGGCTCA CTCGATGAAG TTGCCATCAT GAATATCAGC ACGCTCTTGG 180
GACAACTCGA AGAATTGGAC AAGAGGCGTC GTTACATCCT CGAAAGCATT GAGGCAAGCG 240
GTGCCTTGAC TCCGGAATTG AATTCACGCA TTATGGTGTG CGATGATGCA ACGACACTCG 300
AGGATATCTA CCTCCCATTC AAGCCCAAAC GCCGCACCAA GGCGGAAGTG GCACGAAACA 360
ACGGTCTCGA ACCGTTGGCA AAAATAATTA TGGCACAAAA ATCGATCGAT ATTCATTCTC 420
AAGCCGGTCG TTTTATTGGG GAAAATGTGC CTGACGAA 458
<210>3
<211>23
<212>DNA
<213>人工序列
<400>3
GGAGGTGGTT AAGCCGATAT GTT 23
<210>4
<211>22
<212>DNA
<213>人工序列
<400>4
GCCCCTTTCT TGATACTTTG GA 22
<210>5
<211>26
<212>DNA
<213>人工序列
<400>5
AAACTGATTG GAGAAGAATA CAGGCG 26
<210>6
<211>21
<212>DNA
<213>人工序列
<400>6
GCGTCGTTAC ATCCTCGAAA G 21
<210>7
<211>18
<212>DNA
<213>人工序列
<400>7
GCGTTTGGGC TTGAATGG 18
<210>8
<211>29
<212>DNA
<213>人工序列
<400>8
TTCACGCATT ATGGTGTGCG ATGATGCAA 29
- 还没有人留言评论。精彩留言会获得点赞!