引物对及分子标记以及检测棉花植株对黄萎病菌抗性的方法和试剂盒与流程
本发明涉及农业生物技术领域,具体地,涉及一种检测棉花植株对黄萎病抗性的方法、一种引物对、一种检测棉花植株对黄萎病抗性的试剂盒和一种分子标记。
背景技术:
棉花黄萎病是世界性的重大植物病害,常年给棉花生产造成10-15%的产量损失,严重年份超过全国植棉面积的60%,给棉花生产造成沉重打击。其病原菌随种子、残枝败叶和土壤传播,菌核在土壤中可存活多年,化学药物、耕作栽培等手段对其无效,被植物病理界称为棉花的“癌症”,成为长期以来悬而未决的世界性难题。培育抗病品种被公认为是解决棉花黄萎病最经济有效的手段,究其一直未能成功的原因是棉花黄萎病的抗性受多个主效基因共同控制的多基因控制遗传模式,遗传机制较为复杂(祁伟彦,张永军,张天真,陈捷胤,戴小枫.基于人工病圃筛选和分子标记辅助的棉花抗黄萎病育种方法研究与应用.分子植物育种,2012,10(5):607-612;蒋锋,赵君,周雷,郭旺珍,张天真.陆地棉抗黄萎病基因的分子标记定位.中国科学,2009,39(9):849-861;葛海燕,汪业春,郭旺珍,张天真.陆地棉抗黄萎病性状的遗传及分子标记研究.棉花学报,2008,20(1):19-22;Jiang F,Zhao J,Zhou L,Guo WZ,Zhang TZ.Molecular mapping of Verticillium wilt resistance QTL on chromosomes D7and D9in upland cotton.Science China SerC-Life Science,2009,52(9):872-884;杨昶,郭旺珍,张天真.陆地棉抗黄萎病、纤维品质和产量等农艺性状的QTL定位.分子植物育种,2007,5(6):797-805),缺乏有效的可用于稳定筛选和准确跟踪其抗性基因的分子标记,鉴定十分困难,导致棉花抗黄萎病育种进展缓慢。
早在20世纪40年代我国就开始棉花育种,50年代后开展杂交育种,当时棉田病害轻,基本上根据产量表现选育品种。70年代后棉花枯萎病的发生逐渐加重,开始了抗枯萎病育种,但育种技术基本上采用自然感病试验田进行抗病性鉴定,由于病原菌不足,田间发病不均匀,年份间差异大,育种亲本和杂交后代材料鉴定结果不准确,材料当选或淘汰盲目性较大。90年代后黄萎病发生渐趋严重,迫切需要培育抗黄萎病品种,但抗病性鉴定与选育仍然采用与抗枯萎病类似的方法,育种效率低,后代材料选择不准确,育成品种抗病性差,且抗病品种产量较低。
棉花抗黄萎病分子标记辅助育种技术已经在纤维品种改良、不育系改良等得到应用,但是分子标记在不同的棉花品种之间的分布存在一定的不规律性,因此需要更多的分子标记来进行互相验证,以便从多个维度对棉花植株的抗黄萎病的能力进行考察,从而有利于提高棉花育种的效率。
技术实现要素:
本发明的目的是提供一种检测棉花植株对黄萎病菌抗性的方法,从而提高棉花育种的效率。
为了实现上述目的,一方面,本发明提供了一种检测棉花植株对黄萎病菌抗性的方法,该方法包括:(1)将待测棉花植株的种子用浸种液浸泡;所述浸种液是将死于黄萎病的棉花植株的组织粉碎,用水浸泡,过滤得到的菌悬液;(2)将浸泡后的种子种植于病圃中;所述病圃是将死于黄萎病的棉花植株的组织粉碎掺入土壤之后构建的;(3)选择能够在病圃中存活的棉花植株作为初筛棉花植株;(4)分别使用第一引物对和第二引物对初筛棉花植株的基因组DNA进行PCR扩增,得到第一扩增产物和第二扩增产物;所述第一引物对包括如SEQ ID NO:1所示的第一上游引物和如SEQ ID NO:2所示的第一下游引物,所述第二引物对包括如SEQ ID NO:3所示的第二上游引物和如SEQ ID NO:4所示的第二下游引物;如果所述第一扩增产物中具有SEQ ID NO:5所示的核酸且所述第二扩增产物中具有SEQ ID NO:6所示的核酸,则指示待测棉花植株为抗黄萎病植株。
再一方面,本发明提供了一种引物对,其中,该引物对包括第一引物对和第二引物对,所述第一引物对包括如SEQ ID NO:1所示的第一上游引物和如SEQ ID NO:2所示的第一下游引物,所述第二引物对包括如SEQ ID NO:3所示的第二上游引物和如SEQ ID NO:4所示的第二下游引物。
再一方面,本发明还提供了一种检测棉花植株对黄萎病菌抗性的试剂盒,其中,该试剂盒含有PCR试剂和如上所述的引物对。
再一方面,本发明还提供了一种分子标记,其中,该分子标记包括如SEQ ID NO:5所示的左侧抗病位点核酸和SEQ ID NO:6所示的右侧抗病位点核酸。
通过上述技术方案,本发明能够实现对棉花植株对黄萎病菌抗性进行方便、快速地检测,由此提高了棉花育种的效率。
本发明的其他特征和优点将在随后的具体实施方式部分予以详细说明。
附图说明
附图是用来提供对本发明的进一步理解,并且构成说明书的一部分,与下面的具体实施方式一起用于解释本发明,但并不构成对本发明的限制。在附图中:
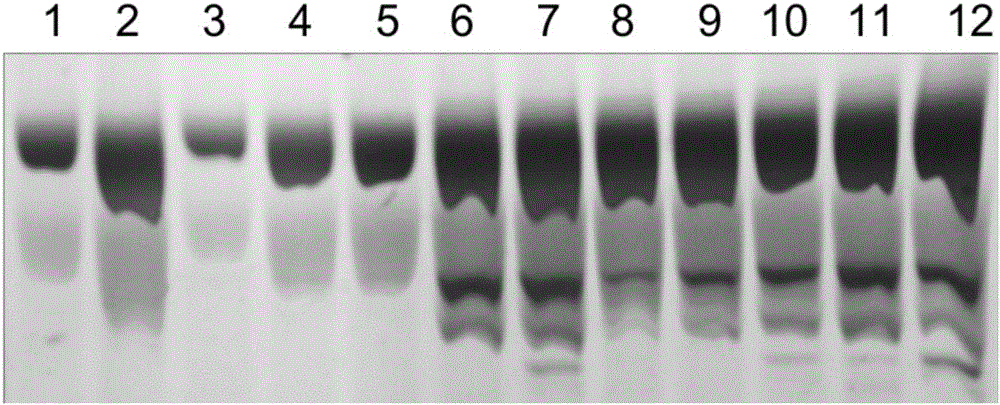
图1是12种棉种材料的第一扩增产物电泳图。
图2是12种棉种材料的第二扩增产物电泳图。
具体实施方式
以下对本发明的具体实施方式进行详细说明。应当理解的是,此处所描述的具体实施方式仅用于说明和解释本发明,并不用于限制本发明。
一方面,本发明提供了一种检测棉花植株对黄萎病菌抗性的方法,该方法包括:(1)将待测棉花植株的种子用浸种液浸泡;所述浸种液是将死于黄萎病的棉花植株的组织粉碎,用水浸泡,过滤得到的菌悬液;(2)将浸泡后的种子种植于病圃中;所述病圃是将死于黄萎病的棉花植株的组织粉碎掺入土壤之后构建的;(3)选择能够在病圃中存活的棉花植株作为初筛棉花植株;(4)分别使用第一引物对和第二引物对初筛棉花植株的基因组DNA进行PCR扩增,得到第一扩增产物和第二扩增产物;所述第一引物对包括如SEQ ID NO:1所示的第一上游引物和如SEQ ID NO:2所示的第一下游引物,所述第二引物对包括如SEQ ID NO:3所示的第二上游引物和如SEQ ID NO:4所示的第二下游引物;如果所述第一扩增产物中具有SEQ ID NO:5所示的核酸且所述第二扩增产物中具有SEQ ID NO:6所示的核酸,则指示待测棉花植株为抗黄萎病植株。
其中,进一步地,如果所述第一扩增产物中不具有SEQ ID NO:5所示的核酸且所述第二扩增产物中不具有SEQ ID NO:6所示的核酸,则指示待测棉花植株为黄萎病易感植株。
其中,所述PCR扩增的条件可以包括:变性温度为93.5-94.5℃;退火温度为54.5-55.5℃。
其中,所述PCR扩增的条件还可以包括:PCR循环数为28-32轮。
其中,制备浸种液时,相对于每重量份的死于黄萎病的棉花植株的组织,水的用量可以为4-6重量份。
其中,用浸种液浸泡种子时,相对于每重量份的种子,浸种液的用量可以为1-3重量份。
其中,用浸种液浸泡种子的时间可以为5-8小时。
其中,构建病圃时,相对于每平方米的土壤,死于黄萎病的棉花植株的组织的掺入量可以为1-5kg。
其中,待测棉花植株的基因组DNA可以通过常规的DNA提取方法从待测棉花植株中提取得到。
再一方面,本发明提供了一种引物对,其中,该引物对包括第一引物对和第二引物对,所述第一引物对包括如SEQ ID NO:1所示的第一上游引物和如SEQ ID NO:2所示的第一下游引物,所述第二引物对包括如SEQ ID NO:3所示的第二上游引物和如SEQ ID NO:4所示的第二下游引物。
其中,上述引物对中的各种引物可以通过常规的DNA引物合成方法合成得到,上述引物对中的各种引物可以分开独立存放。
再一方面,本发明还提供了一种检测棉花植株对黄萎病菌抗性的试剂盒,其中,该试剂盒含有PCR试剂和如上所述的引物对。
其中,所述PCR试剂可以包括耐高温DNA聚合酶、dNTP和PCR缓冲溶液。
再一方面,本发明还提供了一种分子标记,其中,该分子标记包括如SEQ ID NO:5所示的左侧抗病位点核酸和SEQ ID NO:6所示的右侧抗病位点核酸。
本发明可通过检测分子标记对棉花植株对黄萎病菌的抗性进行预测和筛选,淘汰病圃筛选过程中虽不发病但仍为感病的植株,减少人力物力的浪费,提高育种效率。
以下将通过实施例对本发明进行详细描述。
实施例1
收集12种棉种材料,按照文献(张兴华,棉花抗枯、黄萎病研究进展及其抗性鉴定方法,江西农业学报,2008,20(3):43-49)中的人工苗床病圃法,鉴定它们的抗病性,结果如表1中所列,其中,感病种质5份,在表1的感黄萎病材料一栏表示为“S”;抗病种质7份,在表1的抗黄萎病材料一栏表示为“R”。
按照《分子克隆实验指南》中的方法,分离每份种质的基因组DNA。以每份种质的基因组DNA为模板,采用正向引物如SEQ ID NO:1所示(5'-ATATAAACGATAACACAACGGTA-3')且反向引物如SEQ ID NO:2所示(5'-ATGGCACACAAAGTGACATGA-3')的第一引物对进行PCR扩增,变性温度为94℃;退火温度为55℃:PCR循环数为30轮;分别得到每份材料的第一扩增产物。
以每份种质的基因组DNA为模板,采用正向引物如SEQ ID NO:3所示(5'-GCTATTTACTGCTGTTTTACTGC-3')且反向引物如SEQ ID NO:4所示(5'-ACATTTTTTTCAACAAACTTGTG-3')的第二引物对进行PCR扩增,变性温度为94℃;退火温度为55℃:PCR循环数为30轮;分别得到每份材料的第二扩增产物。
按照《分子克隆实验指南》中的方法,将第一扩增产物和第二扩增产物在聚丙烯酰胺凝胶上进行电泳检测;图1是12种棉种材料的第一扩增产物电泳图;图2是12种棉种材料的第二扩增产物电泳图;将12种棉种材料的第一扩增产物和第二扩增产物进行测序,得到的测序结果如表1所示。
表1
表1的结果证明:如果第一扩增产物中具有SEQ ID NO:5所示的核酸且第二扩增产物中具有SEQ ID NO:6所示的核酸,则指示待测棉花植株为抗黄萎病植株;如果第一扩增产物中不具有SEQ ID NO:5所示的核酸或者第二扩增产物中不具有SEQ ID NO:6所示的核酸,则指示待测棉花植株为感黄萎病植株。
实施例2
本实施例中,待测的棉花植株为军棉1号棉花品种和军海1号棉花品种的F2代种子。军棉1号棉花品种和军海1号棉花品种的F2代种子对黄萎病的抗性是事先不知道的。
将死于黄萎病的棉花植株的组织粉碎,用等重量的水浸泡48小时,过滤得到含有黄萎病致病菌的浸种液。
军棉1号棉花品种和军海1号棉花品种的F2代种子浸泡于上述含有黄萎病致病菌的浸种液中24小时,得到浸泡后的种子,其中,相对于每重量份的种子,浸种液的用量为5重量份。将死于黄萎病的棉花植株的组织粉碎,掺入土壤中,相对于每平方米的土壤,死于黄萎病的棉花植株的组织的掺入量为3kg,构建病圃。将浸泡后的种子种植于病圃中,共长出236株棉苗,但仅有47株存活并最终收获种子,其余均死于黄萎病,得到47个抗黄萎病的棉花植株,分别记录为初筛棉花植株1-47。
提取47个初筛棉花植株的DNA,采用正向引物如SEQ ID NO:1所示(5'-ATATAAACGATAACACAACGGTA-3')且反向引物如SEQ ID NO:2所示(5'-ATGGCACACAAAGTGACATGA-3')的第一引物对进行PCR扩增,分别得到每份材料的第一扩增产物,并且采用正向引物如SEQ ID NO:3所示(5'-GCTATTTACTGCTGTTTTACTGC-3')且反向引物如SEQ ID NO:4所示(5'-ACATTTTTTTCAACAAACTTGTG-3')的第二引物对进行PCR扩增,分别得到每份材料的第二扩增产物,将第一扩增产物和第二扩增产物进行DNA测序。DNA测序结果显示,待测植株7、15、23、25、31、35-37和42的第一扩增产物中不具有SEQ ID NO:5所示的核酸且第二扩增产物中不具有SEQ ID NO:6所示的核酸,而待测植株1-6、8-14、16-22、24、26-30、32-34、38-41和43-46第一扩增产物中具有SEQ ID NO:5所示的核酸且第二扩增产物中具有SEQ ID NO:6所示的核酸。
根据上述结果,指示待测植株1-6、8-14、16-22、24、26-30、32-34、38-41和43-46为黄萎病抗病植株,而植株7、15、23、25、31、35-37和42为黄萎病感病植株。
对比例1
按照文献(张兴华,棉花抗枯、黄萎病研究进展及其抗性鉴定方法,江西农业学报,2008,20(3):43-49)中的人工苗床病圃法,对实施例2的初筛棉花植株1-47的种子种植后进行再次抗病性鉴定,植株1-6、8-14、16-22、24、26-30、32-34、38-41和43-46的黄萎病情指数均小于20,为抗黄萎病棉花植株,而植株7、15、23、25、31、35-37和42的黄萎病情指数均大于40,为黄萎病感病植株,与实施例2的指示结果相同。
通过实施例2和对比例1比较可见,本发明的方法能够实现对棉花植株对黄萎病抗性的准确的检测。
以上详细描述了本发明的优选实施方式,但是,本发明并不限于上述实施方式中的具体细节,在本发明的技术构思范围内,可以对本发明的技术方案进行多种简单变型,这些简单变型均属于本发明的保护范围。
另外需要说明的是,在上述具体实施方式中所描述的各个具体技术特征,在不矛盾的情况下,可以通过任何合适的方式进行组合,为了避免不必要的重复,本发明对各种可能的组合方式不再另行说明。
此外,本发明的各种不同的实施方式之间也可以进行任意组合,只要其不违背本发明的思想,其同样应当视为本发明所公开的内容。
SEQUENCE LISTING
<110> 中国农业科学院农产品加工研究所
<120> 引物对及分子标记以及检测棉花植株对黄萎病菌抗性的方法和试剂盒
<130> 1015CAAS
<160> 6
<170> PatentIn version 3.3
<210> 1
<211> 20
<212> DNA
<213> Artificial
<220>
<223> This sequence is synthesized in lab.
<400> 1
ggtcgaaaag catgaccagg 20
<210> 2
<211> 20
<212> DNA
<213> Artificial
<220>
<223> This sequence is synthesized in lab.
<400> 2
agtcgccctc ttttgggttt 20
<210> 3
<211> 21
<212> DNA
<213> Artificial
<220>
<223> This sequence is synthesized in lab.
<400> 3
actcgagaaa agcacggttt g 21
<210> 4
<211> 23
<212> DNA
<213> Artificial
<220>
<223> This sequence is synthesized in lab.
<400> 4
tcgattcaaa ttcacttccc act 23
<210> 5
<211> 225
<212> DNA
<213> Gossypium sp.
<400> 5
ggtcgaaaag catgaccagg acttgcctaa tctcatgagt ttagactaaa aaaaataaaa 60
aaatcaaaaa taaaaaaata aaaaaatcaa agtcaaaata gaaaaaaaga aaaagaaaaa 120
aaagagaaga aaaagaaaaa gaaaaaggag gggtcaaggc gaaaacccac aaagggcgct 180
ttgaccaaag gtggatctga gttgaaaacc caaaagaggg cgact 225
<210> 6
<211> 236
<212> DNA
<213> Gossypium sp.
<400> 6
actcgagaaa agcacggttt gtattactat aattcaagtt atttattatt aaatgttaaa 60
tttcaattta tgtgtttggt aaatgaaatg tgaggtaagt attattatta ttattattat 120
tattattatt attattatta ttattattat tatgagtgag aattaaattg aatagttgat 180
atgaattaat atttgaattg tttgttgatt gaaagtggga agtgaatttg aatcga 236
- 还没有人留言评论。精彩留言会获得点赞!