一种用于检测DNA错配修复系统的试剂盒及其用途的制作方法
本发明属于DNA错配修复系统领域,具体涉及一种用于检测DNA错配修复系统的试剂盒及其用途。
背景技术:
人类基因组含有短串联DNA重复序列,又称作微卫星序列(microsatellite),这些序列由于其重复性质,易发生复制错误,正常情况下,这些错误可以通过DNA的错配修复(MMR)系统来进行修复校正,DNA的MMR系统功能缺陷会引起微卫星不稳定性(microsatellite instability,MSI),MSI是林奇综合症(Lynch syndrome)的一个标志,在90%的林奇综合症中均有发生。
微卫星不稳定表现为同一微卫星位点在不同个体之间以及同一个体的正常组织与某些异常组织之间,微卫星位点的重复单位的数目不同。近年来的大量研究表明:MSI与肿瘤发生密切相关,如结直肠癌、胃癌、子宫内膜癌等,MSI是癌症发生的一个生物标记。美国国立癌症协会建议50岁以下的结直肠癌患者应该做MMR检测,在国外一些医学中心对所有的结直肠肿瘤组织进行MMR蛋白免疫组化检测或者MSI检测,以决定哪些患者需要进行Lynch综合征相关基因检测。
根据文献报道,大约15%的结直肠癌中存在MSI现象,与无MSI的结直肠癌相比,携带有MSI的结直肠癌其预后较好,已有大量证据表明,MMR蛋白表达缺失或者MSI-H是二期结直肠癌预后良好的一个标志物,而且存在MSI和无MSI的患者二者药物反应也不一样。此外,对于二期结直肠癌是否需要辅助化疗,临床上考量的一个重要信息就是微卫星不稳定性。
因此,MSI检测对结直肠癌患者的意义重大。在2011年,关于结直肠癌筛查的美国国家综合癌症网指南(NCCN Guidelines)中,MSI已成为首要检测项目,推荐了5个检测位点用于MSI稳定性的确认,两个及两个以上位点发生变化为MSI-H,一个位点发生变化为MSI-L,没有位点发生变化为MSI-S。
研究表明,二期/三期结肠癌患者发现MSI-L或者MSS通过辅助化疗确实会改善预后,但是MSI-H却不能从5-氟尿嘧啶(5-FU)辅助化疗中显著获益,5年生存率反而更低。结肠直肠癌是常见消化道恶性肿瘤,中国每年有13-16万人罹患结直肠癌,其中发病率占肿瘤发病率的第三位。具有MSI的散发性结直肠癌,具有不同的临床病理、分子生物学特征,表现为:发病年龄小;对某些化疗药物如5-FU、顺铂等有耐药性,MSI-H是二期结肠癌预后良好的一个标志物,也是患者不能从氟尿嘧啶单药辅助化疗获益的疗效预测指标。
此外,检测MSI还可以辅助PD-1抗体用药。2015年的ASCO中报道,结肠癌MMR基因表达情况可以作为PD-1抗体治疗的疗效预测指标。根据大会临床数据报告,MMR表达缺陷(dMMR)患者PD-1治疗的有效率达到62%,相关的实验结果在2015年底发表在全球最顶尖的医学杂志NEJM上,引起了医学界的轰动。MMR基因的情况对PD-1结直肠癌疗效的预测精准度远远高于了之前通用的疗效预测指标B7-H1(PD-L1)。
在美国和欧盟,NCCN已经将MSI检测纳入常规检测,但是在中国应用MSI检测的医院还非常少,用于结直肠癌筛查最常用的还是肠镜检查,粪便潜隐血检测,钡灌肠检测等。
目前国内外用于检测MSI的方法主要有:(1)免疫组化方法(IHC),主要检测MLH1,MSH2,MSH6,PMS2,这种方法不能检测MMR蛋白的生物学活性,所以有可能产生假阳性结果,而且IHC实验结果受抗体来源以及操作者的技能水平的影响,这会导致不同医院间结果的差异性,造成诊断不准确甚至是医疗纠纷;(2)直接测序法,对MMR基因各区域直接测序,这种方法耗时长,价格比较昂贵。
技术实现要素:
本发明的目的是提供一种用于检测DNA错配修复系统的试剂盒及其用途,该试剂盒的微卫星位点能够用于检测癌症DNA错配修复系统,其灵敏度比免疫组化方法(IHC)方法更高,本发明的试剂盒重复性更好,能够同时检测多个MSI位点,提高了检测效率。
为了达到上述目的,本发明提供了一种用于检测DNA错配修复系统的试剂盒,该试剂盒包含一扩增体系,该扩增体系包含PCR引物混合液和PCR反应液;所述的PCR引物混合液包含微卫星位点,该微卫星位点包含:NR-21、NR-24、BAT-25、BAT-26、MONO-27、BAT-48、BAT-49a、BAT-49b、BAT-50a、BAT-50b、BAT-51a、BAT-51b、BAT-51c、BAT-51d、BAT-51e、BAT-51f、BAT-52a、BAT-52b、BAT-53a、BAT-53b、BAT-53c、BAT-54、BAT-55、BAT-56a、BAT-56b、BAT-57、BAT-59、BAT-59b、BAT-60a、BAT-60b、BAT-60c、BAT-62、BAT-63a、BAT-63b、BAT-68a、BAT-68b、BAT-69、BAT-72、BAT-73、BAT-79、BAT-83、BAT-90、BGT-60、Penta C、Penta D、Penta E中的任意一种或两种以上串联重复位点单核苷酸A或者G大于40个重复。
所述的微卫星位点具体如下表所示:(Accession Number是Genebank的基因登录号)
;上表中核苷酸重复表示有多少个重复的核苷酸;微卫星位点为1-6个碱基的串联重复序列,这些位点为单核苷酸A或者G串联重复位点,或者五个核苷酸串联重复位点,(A)21中A代表腺嘌呤核糖核苷酸,数字21代表重复21个A。
所述的PCR引物混合液包含核苷酸序列SEQ ID NO:1-92中的任意一种或两种以上。
所述的核苷酸序列SEQ ID NO:1-92的5’末端用荧光标记物标记,该荧光标记物包含FL(Fluorescein)、JOE(6-carboxy-4′,5′-dichloro-2′,7′-dimethoxyfluorescein)、TMR(carboxy-tetramethylrhodamine)、ROX(Carboxyl-X-Rhodamine)、ET-CXR(Energy Transfer-carboxy-X-rhodamine)、ET-TMR(Energy Transfer-carboxy-tetramethylrhodamine)或FAM(Fluorescein amidite)中的任意一种或两种以上。
所述的扩增体系还包含DNA模板,该扩增体系的组分的体积比为PCR反应液:PCR引物混合液:DNA模板:水=1~2:1~2:1~4:2~7。
所述的PCR反应液包括:10-100mM Tris-HCl,10-250mM KCl,1-10mM MgCl2,0.1-10mM dNTP,0.1-10U/μL Taq DNA聚合酶。
该检测方法采用荧光标记毛细管电泳法确定微卫星位点突变的存在。
所述的突变包含:插入突变或缺失突变。
本发明还提供了一种根据所述的用于检测DNA错配修复系统的试剂盒的用途,所述的微卫星位点能够用于检测DNA错配修复系统,该DNA错配修复系统包括癌症组织中的DNA错配修复系统。
所述的癌症包括:结肠直肠癌,子宫内膜癌,胃癌,胰腺癌,肺癌,以及乳腺癌中的任意一种。
所述的微卫星位点用于检测癌症的微卫星的不稳定性,使该试剂盒能作为检测癌症DNA错配修复系统的试剂盒。
本发明提供的用于检测DNA错配修复系统的试剂盒及其用途,具有以下优点:
1、使用本发明的微卫星位点检测,其灵敏度比IHC方法更高。
2、本发明的长串联重复位点具有更高的灵敏度和准确度。
3、本发明的试剂盒采用荧光标记和毛细管电泳片段大小来区分不同的微卫星位点,实现了在一个PCR体系内同时检测多个MSI位点,提高了检测效率。
4、本发明的试剂盒检测能够从同一切片提取的DNA做多次重复实验,而传统的免疫组化同一切片无法做重复实验,比传统方法更具有可重复性,而且本发明的检测实验结果采用软件分析,减少了了人为主观判断和标准化。
5、美国国立癌症研究院提出的微卫星检测体系中并没有内对照,本发明使用Penta位点作为内对照,是本领域中是首次使用,其可以对整个检测体系进行内控制。
6、本发明的检测采用毛细管电泳检测方法,使结果更准确、直观。
7、本发明采用数据分析软件,使结果更好地被阐释。
8、本发明选择不同的微卫星位点可以适用于检测不同的癌症,包括结肠直肠癌,子宫内膜癌,胃癌,胰腺癌,肺癌以及乳腺癌。
附图说明
图1为本发明实验例2的短单核苷酸串联重复位点的DNA分型图谱。
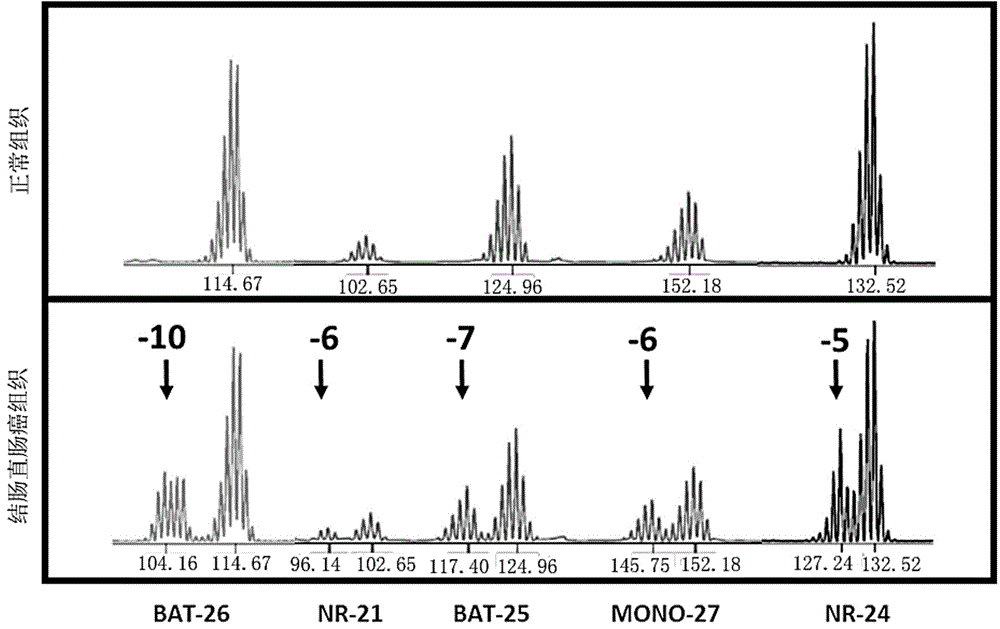
图2为本发明实验例2的正常组织和结肠直肠癌组织的短单核苷酸串联重复位点的对比的DNA分型图谱。
图3为本发明实验例2的长单核苷酸串联重复位点的DNA分型图谱。
具体实施方式
以下结合附图和实施例对本发明的技术方案做进一步的说明。
采用本发明的试剂盒对2例结肠直肠癌患者检测的具体实施情况具体如下:
实验例1PCR扩增反应
1、PCR反应体系
表1:反应具体组份
上述表1中的dNTP包括dATP、dGTP、dTTP和dCTP,dATP、dGTP、dTTP和dCTP的使用浓度为1mM。
使用的引物对的混合物指选用包含本发明的微卫星位点及其上游引物和下游引物的混合物,各引物对(上游引物和下游引物)的终浓度为0.1-1μM。如表2所示,为两个混合物的具体组份。
表2:两个混合液的具体组份
上述表2中,5’端荧光标记物JOE为6-carboxy-4′,5′-dichloro-2′,7′-dimethoxyfluorescein,FL为Fluorescein,ET-TMR为Energy Transfer-carboxy-tetramethylrhodamine,“/”表示不用荧光标记,5’端为羟基。其中,微卫星位点Penta C和Penta D作为内对照。
2、实验条件
PCR仪器选用ABI veriti PCR仪
表3:PCR反应的具体条件
将2μL PCR反应液和2μL PCR引物混合液混合,加入1-2μL DNA模板,并补加水4-6μL至PCR反应体系总体积为10μL。参照上述表3的具体反应条件进行PCR反应。
实验例2毛细管电泳实验
取扩增好的PCR产物,加入内标(Promega公司,分子内标ILS500)和甲酰胺,在95℃下干浴3min,冰水浴3min。ABI 3500Dx仪器上样,打开3500Dx数据收集软件,编辑上样板,导入片段分析程序,点击运行。具体步骤如下:
(1)准备上样样品:所得PCR产物1μL,加入甲酰胺10μL,DNA分子标准内标ILS500 1μL;
(2)毛细管电泳:按照ABI3500Dx仪器使用说明书进行毛细管电泳程序设置,具体如下:
A、准备仪器;B、预热加热炉至60℃;C、准备样本反应板并加载至仪器;D、检查仪器状态;E、创建或导入一个反应板;F、分配反应板内容;G、链接反应板;H、加载反应板以便运行,创建进样列表;I、开始运行。
片段分析运行完毕后,将原始数据,后缀名为.fsa的文件导入数据分析软件GeneMapper4.0,对荧光信号进行分析,导入Panel和Bin,点击Analyze,分析完毕得到毛细管电泳图谱及相关片段大小信息。
实验结果:
如图1所示,使用微卫星位点为:NR-21、NR-24、BAT-25、BAT-26、Mono-27、Penta C和Penta D的试剂盒对结肠直肠癌患者1的肿瘤组织及正常组织进行微卫星位点检测,凡是在癌症组织或者息肉组织中发现有一个位点的片段大小与正常组织不同,则说明该位点为微卫星不稳定,如图2所示,结肠直肠癌组织与正常组织5个微卫星位点相比,其核苷酸片段的大小有不同程度的减小,如BAT-26位点减小了10bp,NR-21位点减小了6bp,BAT-25位点减小了7bp,Mono-27位点减小了6bp,NR-24位点减小了5bp,表明结肠直肠癌组织在5个位点均出现MSI现象,说明该患者肿瘤组织为MSI-H。
如图3所示,使用微卫星位点为:BAT-52a、BAT-55、BAT-56a、BAT-57、BAT-59、Penta C和Penta D的试剂盒对结肠直肠癌患者2的肿瘤组织及正常组织进行微卫星位点检测,凡是在癌症组织或者息肉组织中发现有一个位点的片段大小与正常组织不同,则说明该位点为微卫星不稳定。结果显示癌症组织的微卫星位点的核苷酸片段的大小有不同程度的减小,如BAT-59位点减小了13bp,BAT-52位点减小了23bp,BAT-56位点减小了10bp,BAT-55位点减小了5bp,BAT-57位点减小了11bp,表明结肠直肠癌组织在上述位点均出现MSI现象,说明该患者肿瘤组织为MSI-H。
综上所述,本发明的用于检测DNA错配修复系统的试剂盒及其用途,该试剂盒包含微卫星位点,该微卫星位点能够用于检测癌症DNA错配修复系统,其灵敏度比免疫组化方法(IHC)方法更高,试剂盒重复性更好,能够同时检测多个MSI位点,提高了检测效率,而且还能判断肿瘤治疗预后以及指导5-氟尿嘧啶(5-FU)和程序性死亡受体-1(PD-1)抗体的用药。
尽管本发明的内容已经通过上述优选实施例作了详细介绍,但应当认识到上述的描述不应被认为是对本发明的限制。在本领域技术人员阅读了上述内容后,对于本发明的多种修改和替代都将是显而易见的。因此,本发明的保护范围应由所附的权利要求来限定。
<110> 上海普洛麦格生物产品有限公司
<120> 一种用于检测DNA错配修复系统的试剂盒及其用途
<130> 0
<160> 92
<170> PatentIn version 3.5
<210> 1
<211> 24
<212> DNA
<213> 人工合成
<400> 1
cggagtcgct ggcacagttc tatt 24
<210> 2
<211> 25
<212> DNA
<213> 人工合成
<400> 2
tcgcgtttac aaacaagaaa agtgt 25
<210> 3
<211> 20
<212> DNA
<213> 人工合成
<400> 3
ccattgctga attttacctc 20
<210> 4
<211> 20
<212> DNA
<213> 人工合成
<400> 4
attgtgccat tgcattccaa 20
<210> 5
<211> 20
<212> DNA
<213> 人工合成
<400> 5
tcgcctccaa gaatgtaagt 20
<210> 6
<211> 24
<212> DNA
<213> 人工合成
<400> 6
atttctgcat tttaactatg gctc 24
<210> 7
<211> 24
<212> DNA
<213> 人工合成
<400> 7
tgactacttt tgacttcagc cagt 24
<210> 8
<211> 24
<212> DNA
<213> 人工合成
<400> 8
aaccattcaa catttttaac cctt 24
<210> 9
<211> 24
<212> DNA
<213> 人工合成
<400> 9
tgtgaaccac ctatgaattg caga 24
<210> 10
<211> 26
<212> DNA
<213> 人工合成
<400> 10
attgcttgca gtgagcagag atcgtt 26
<210> 11
<211> 24
<212> DNA
<213> 人工合成
<400> 11
tataattagg tcccagatca ctta 24
<210> 12
<211> 24
<212> DNA
<213> 人工合成
<400> 12
ggcaatgttt aaagacatgg atac 24
<210> 13
<211> 24
<212> DNA
<213> 人工合成
<400> 13
aaacacagtg agactcccta tcta 24
<210> 14
<211> 24
<212> DNA
<213> 人工合成
<400> 14
acaggacaga gatggcacgg acag 24
<210> 15
<211> 24
<212> DNA
<213> 人工合成
<400> 15
ctgctgttgc atcgcggccc aatg 24
<210> 16
<211> 24
<212> DNA
<213> 人工合成
<400> 16
aagaagcccc tctcctccgg tctc 24
<210> 17
<211> 24
<212> DNA
<213> 人工合成
<400> 17
aggcatgggc aaggacttga tgtc 24
<210> 18
<211> 24
<212> DNA
<213> 人工合成
<400> 18
ctggatgtta gccgtttgtc agag 24
<210> 19
<211> 24
<212> DNA
<213> 人工合成
<400> 19
ggtttgcttg aggccagaac ttca 24
<210> 20
<211> 24
<212> DNA
<213> 人工合成
<400> 20
ctcatagcag ccttaaatta ctga 24
<210> 21
<211> 24
<212> DNA
<213> 人工合成
<400> 21
agcctgggcg acagagcaag actc 24
<210> 22
<211> 24
<212> DNA
<213> 人工合成
<400> 22
caagggcagc atcattatga caac 24
<210> 23
<211> 28
<212> DNA
<213> 人工合成
<400> 23
tgtgtgcaaa ttgtgaggga ggtaggta 28
<210> 24
<211> 24
<212> DNA
<213> 人工合成
<400> 24
agcggggtgc ggtggctcat atct 24
<210> 25
<211> 24
<212> DNA
<213> 人工合成
<400> 25
ctgaggcagg agaatggaga gtag 24
<210> 26
<211> 24
<212> DNA
<213> 人工合成
<400> 26
ctctgctacc cgggttcaaa cagt 24
<210> 27
<211> 28
<212> DNA
<213> 人工合成
<400> 27
gaggctgagg caggagaatg gcgtgaac 28
<210> 28
<211> 28
<212> DNA
<213> 人工合成
<400> 28
cgctgacgca gaacctgaaa ttgtgatt 28
<210> 29
<211> 24
<212> DNA
<213> 人工合成
<400> 29
aggttgcagt gagccaggat cata 24
<210> 30
<211> 24
<212> DNA
<213> 人工合成
<400> 30
atcacatcat ctgtcccacc taac 24
<210> 31
<211> 24
<212> DNA
<213> 人工合成
<400> 31
tgggcgacag agcgagactc cgtc 24
<210> 32
<211> 24
<212> DNA
<213> 人工合成
<400> 32
cagcggccca taaattctat gtta 24
<210> 33
<211> 28
<212> DNA
<213> 人工合成
<400> 33
ctaacttccc agcaacttcc tttacact 28
<210> 34
<211> 24
<212> DNA
<213> 人工合成
<400> 34
attgggcaga cactgaacta gctt 24
<210> 35
<211> 28
<212> DNA
<213> 人工合成
<400> 35
gggagaacct tgctgtcttt cagataat 28
<210> 36
<211> 24
<212> DNA
<213> 人工合成
<400> 36
agggctcctg gaatatggtt gtac 24
<210> 37
<211> 28
<212> DNA
<213> 人工合成
<400> 37
aacctccacc ttcccagctc aagtgaca 28
<210> 38
<211> 23
<212> DNA
<213> 人工合成
<400> 38
ggcgacagcg agactccgtc tca 23
<210> 39
<211> 24
<212> DNA
<213> 人工合成
<400> 39
ctgaggcagg agaatggcgt gaac 24
<210> 40
<211> 28
<212> DNA
<213> 人工合成
<400> 40
atgatgctgg cctcataaaa agagttag 28
<210> 41
<211> 26
<212> DNA
<213> 人工合成
<400> 41
tatcctagct tggcctgttt aagacc 26
<210> 42
<211> 22
<212> DNA
<213> 人工合成
<400> 42
tgaggcagga gaatggcgtg aa 22
<210> 43
<211> 26
<212> DNA
<213> 人工合成
<400> 43
tttaatatac ctgctgatca atgata 26
<210> 44
<211> 21
<212> DNA
<213> 人工合成
<400> 44
gacacatggg atcatagcaa a 21
<210> 45
<211> 24
<212> DNA
<213> 人工合成
<400> 45
ttgggcgaca gagcaagacg actc 24
<210> 46
<211> 26
<212> DNA
<213> 人工合成
<400> 46
atttggtcag tgggggctct gttaag 26
<210> 47
<211> 26
<212> DNA
<213> 人工合成
<400> 47
tcagcagctg aaagaaatct gagtac 26
<210> 48
<211> 22
<212> DNA
<213> 人工合成
<400> 48
gcgataccca aagtcaatag tc 22
<210> 49
<211> 24
<212> DNA
<213> 人工合成
<400> 49
gaagctgcag taagccgaga ttgt 24
<210> 50
<211> 24
<212> DNA
<213> 人工合成
<400> 50
gccctcttaa ctcccatgac attc 24
<210> 51
<211> 22
<212> DNA
<213> 人工合成
<400> 51
agcctgggcg acagagcgag tc 22
<210> 52
<211> 23
<212> DNA
<213> 人工合成
<400> 52
ctcggggctc gggagatgag tga 23
<210> 53
<211> 28
<212> DNA
<213> 人工合成
<400> 53
cagcctaggt aacagagcaa gacctttg 28
<210> 54
<211> 23
<212> DNA
<213> 人工合成
<400> 54
gtttgcgtga tttgcgtgga ctt 23
<210> 55
<211> 23
<212> DNA
<213> 人工合成
<400> 55
ctcctgcctc atcctcccga gta 23
<210> 56
<211> 24
<212> DNA
<213> 人工合成
<400> 56
ccgagatcac gccactgcac tcta 24
<210> 57
<211> 28
<212> DNA
<213> 人工合成
<400> 57
tctcatttga gtggtggaag tgactggt 28
<210> 58
<211> 23
<212> DNA
<213> 人工合成
<400> 58
tattctttcg ggatgtaatc tct 23
<210> 59
<211> 26
<212> DNA
<213> 人工合成
<400> 59
cccgtctcta ctaaaaatac taaaac 26
<210> 60
<211> 28
<212> DNA
<213> 人工合成
<400> 60
aaaccaacaa taaggcaacc tcttagtc 28
<210> 61
<211> 28
<212> DNA
<213> 人工合成
<400> 61
tgccagagta gggtggtcca tggtactt 28
<210> 62
<211> 25
<212> DNA
<213> 人工合成
<400> 62
gcccaaaatg tgtttagtta gcttc 25
<210> 63
<211> 28
<212> DNA
<213> 人工合成
<400> 63
aggctgaagc aggagaatca cttaaaac 28
<210> 64
<211> 25
<212> DNA
<213> 人工合成
<400> 64
gccaagtgtc gcttgtaatt ctatt 25
<210> 65
<211> 26
<212> DNA
<213> 人工合成
<400> 65
gaatcttgtt tcggcctttg acctta 26
<210> 66
<211> 25
<212> DNA
<213> 人工合成
<400> 66
cgagatcacg ccaccgcact ctagc 25
<210> 67
<211> 27
<212> DNA
<213> 人工合成
<400> 67
aaatctaccc agctctgtaa cgagaga 27
<210> 68
<211> 28
<212> DNA
<213> 人工合成
<400> 68
aagctctgtt tggcaagtgt taattgta 28
<210> 69
<211> 28
<212> DNA
<213> 人工合成
<400> 69
ttggaatgta ttctctgggt ttggcagt 28
<210> 70
<211> 27
<212> DNA
<213> 人工合成
<400> 70
ttcaggaggc tgaggtggga ggattgt 27
<210> 71
<211> 22
<212> DNA
<213> 人工合成
<400> 71
acctaggcaa taccatctaa ga 22
<210> 72
<211> 24
<212> DNA
<213> 人工合成
<400> 72
gttgcctgtt cactctgata gtct 24
<210> 73
<211> 23
<212> DNA
<213> 人工合成
<400> 73
agcctgggtg acagagcgag act 23
<210> 74
<211> 28
<212> DNA
<213> 人工合成
<400> 74
ttagagttat ttgttgggat gagaatct 28
<210> 75
<211> 22
<212> DNA
<213> 人工合成
<400> 75
ctgggcgaca gagcgagact cc 22
<210> 76
<211> 26
<212> DNA
<213> 人工合成
<400> 76
tctcctgcct tagcctcccg agtagc 26
<210> 77
<211> 27
<212> DNA
<213> 人工合成
<400> 77
tcctctccct aaaaagctcc ccctaag 27
<210> 78
<211> 28
<212> DNA
<213> 人工合成
<400> 78
aggtcaaggc tgcggtaagc tgtgatcg 28
<210> 79
<211> 28
<212> DNA
<213> 人工合成
<400> 79
tccccacttt gtcctgcaca ctcctacc 28
<210> 80
<211> 23
<212> DNA
<213> 人工合成
<400> 80
gggcgacaga gcgagactcc gtc 23
<210> 81
<211> 28
<212> DNA
<213> 人工合成
<400> 81
aagatttaat agacatgcgc agaacact 28
<210> 82
<211> 23
<212> DNA
<213> 人工合成
<400> 82
ccagcctggg caaaagagca agt 23
<210> 83
<211> 28
<212> DNA
<213> 人工合成
<400> 83
acaaacatga aaaggcaaat gatagaac 28
<210> 84
<211> 27
<212> DNA
<213> 人工合成
<400> 84
agaggttgca gtgagccaag attgtag 27
<210> 85
<211> 23
<212> DNA
<213> 人工合成
<400> 85
gagggatgaa gggggacaga tag 23
<210> 86
<211> 23
<212> DNA
<213> 人工合成
<400> 86
cattctcact ccacgccctc tat 23
<210> 87
<211> 24
<212> DNA
<213> 人工合成
<400> 87
catggcattg gggacatgaa caca 24
<210> 88
<211> 24
<212> DNA
<213> 人工合成
<400> 88
cactgagcgc ttctagggac ttct 24
<210> 89
<211> 24
<212> DNA
<213> 人工合成
<400> 89
cagcctaggt gacagagcaa gaca 24
<210> 90
<211> 24
<212> DNA
<213> 人工合成
<400> 90
atttgcctaa cctatggtca taac 24
<210> 91
<211> 33
<212> DNA
<213> 人工合成
<400> 91
tgggttatta attgagaaaa ctccttacaa ttt 33
<210> 92
<211> 26
<212> DNA
<213> 人工合成
<400> 92
attaccaaca tgaaagggta ccaata 26
- 还没有人留言评论。精彩留言会获得点赞!
- 一种新的多肽——人dna错配修复蛋白15和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复蛋白8.9和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复蛋白9.4和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复蛋白8和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复基因蛋白13和编码这种多肽的多核苷酸的制作方法
- 涉及miR-155对于错配修复和基因组稳定性的调节的材料和方法
- 一种新的多肽——人dna错配修复基因蛋白11和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复蛋白12.98和编码这种多肽的多核苷酸的制作方法
- 一种c端特异的人dna错配修复蛋白mlh1多肽及其抗体制备方法
- 作为抗癌化合物的蒽醌化合物的制作方法
- 自动缺陷修复系统的制作方法
- 一种新的多肽——人dna错配修复蛋白15和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复蛋白8.9和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复蛋白9.4和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复蛋白8和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复基因蛋白13和编码这种多肽的多核苷酸的制作方法
- 涉及miR-155对于错配修复和基因组稳定性的调节的材料和方法
- 一种新的多肽——人dna错配修复基因蛋白11和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复蛋白12.98和编码这种多肽的多核苷酸的制作方法
- 一种c端特异的人dna错配修复蛋白mlh1多肽及其抗体制备方法
- 一种c端特异的人dna错配修复蛋白mlh1多肽及其抗体制备方法
- 一种新的多肽——人碱基错配修复蛋白13.2和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽-dna错配修复蛋白11和编码这种多肽的多核苷酸的制作方法
- 人dna错配修复蛋白的制作方法
- 一种新的多肽——人dna错配修复基因蛋白9和编码这种多肽的多核苷酸的制作方法
- 与微rna-21、错配修复和结肠直肠癌相关的材料和方法
- 一种新的多肽——人dna错配修复基因蛋白10和编码这种多肽的多核苷酸的制作方法
- 一种多肽——碱基错配修正蛋白-11.22和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复蛋白12.76和编码这种多肽的多核苷酸的制作方法
- 测定dna错配修复功能的方法