提高同源重组效率的方法与流程
本发明涉及一种提高同源重组率的方法,特别涉及一种将foki-dcas9蛋白应用于基因编辑系统中以实现提高同源重组效率的方法。
背景技术:
随着生命科学研究进入基因组时代,越来越多物种的基因组完成测序,解读与改造基因组的功能就显得非常紧迫,近几年,生物学家们巧妙地利用蛋白质结构与功能领域的研究成果,将特异识别与结合dna的蛋白质结构与和核酸内切酶结构域融合,创造出能够按照人们意愿特异切割dna的序列特异核酸酶(sequence-specificnucleases,ssns),并藉此实现了对基因组特定位点的靶向修饰,及基因组编辑(genomeediting)。ssns主要包括3种类型:锌指核酸酶(zincfingernuclease,zfn)、类转录激活因子效应物核酸酶(transcriptionactivator-likeeffectornuclease,talen)和成簇的规律间隔的短回文重复序列及其相关系统(clusteredreg-ularlyinterspacedshortpalindromicrepeats/crispr-associated9,crispr/cas9system)。上述几类ssns的共同特征是能够作为核酸内切酶切割特定的dna序列,创造dna双链断裂(double-strandbreaks,dsbs)。
在真核生物中,dsbs的修复机制是高度保守的,主要包括两种途径:非同源末端链接(non-homologousendjoining,nhej)和同源重组(homologousrecombination,hr)。通过nhej方式,断裂的染色体会重新连接,但往往不是精确的,断裂位置会产生少量核苷酸的插入或删除,从而产生基因敲除突变体;通过hr方式,在引入同源序列的情况下,以同源序列为模板进行合成修复,从而产生精确的定点替换或者插入突变体。在这两种途径中,nhej方式占绝对主导,可以发生在几乎所有类型的细胞以及不同的细胞周期中(g1、s和g2期);然而,hr发生频率很低,主要发生在s和g2期。hr根据其发生方式不同可以分为两类:单链退火(single-strandannealing,ssa)和合成依赖式退火(synthesis-dependentstrandannealing,sdsa)。dsbs产生后,在这两种途径下dna断裂末端都会发生5’至3’方向的dna切除,形成3’单链末端。ssa途径类似nhej途径,dsbs两端各有一段同源序列,同源序列区域直接退火形成互补双链,再经过末端加工和连接修复dsbs,在基因组串联重复区域,ssa是主要的dsbs修复方式。sdsa途径是依赖dna合成的修复过程,基因组编辑过程中同源重组通常是指这种方式。dsb经过5’至3’方向的dna切除产生的一个3’单链末端入侵同源供体dna模板,形成d-loop环状结构,再利用同源供体dna的互补链作为模板进行dna合成修复,当延伸至可以与dsb另一个单链末端互补配对的位置时,脱离d-loop结构,两个单链dsb末端退火形成双链,完成修复过程。sdsa途径最终结果就是完成从同源dna至dsb遗传信息的转化过程。sdsa途径发生频率非常低,相同条件下只有ssa方式的10%-20%。由此可见,提高hr效率是基因组编辑研究最重要也是最迫切的任务之一。
在crispr/cas9基因编辑系统中,设计不同的sgrna以指导cas9内切酶完成对dna的定点切割,通过同源重组修复机制实现目标基因中不同类型的修饰,包括基因的删除、添加和替换等。因此明确dna修复机制特别是hr修复过程的研究将有助于人们采用适当方法提高基因组编辑中定点插入或替换的效率。
技术实现要素:
本发明的目的是提供一种提高同源重组效率的方法,首次提供了foki-dcas9融合蛋白可以提高同源重组效率。
为实现上述目的,本发明提供了一种提高同源重组效率的方法,包括向宿主细胞中引入foki-dcas9融合蛋白。
进一步地,所述foki-dcas9融合蛋白在宿主细胞中瞬时表达或稳定表达。
更进一步地,所述宿主细胞为植物细胞。
优选地,所述植物为玉米、水稻、大豆、拟南芥、棉花、油菜、高粱、小麦、大麦、粟、甘蔗或燕麦。
在上述技术方案的基础上,所述foki-dcas9融合蛋白的氨基酸序列具有seqidno:4和seqidno:5所示的氨基酸序列。
优选地,所述foki-dcas9融合蛋白的核苷酸序列具有seqidno:1第643-5523位所示的核苷酸序列。
为实现上述目的,本发明还提供了一种基因组编辑系统,包含foki-dcas9融合蛋白。
进一步地,所述foki-dcas9融合蛋白的氨基酸序列具有seqidno:4和seqidno:5所示的氨基酸序列。
更进一步地,所述foki-dcas9融合蛋白的核苷酸序列具有seqidno:1第643-5523位所示的核苷酸序列。
可选地,所述基因组编辑系统还包括编码序列操纵系统的多核苷酸序列。
优选地,所述序列操纵系统为crispr/cas系统。
为实现上述目的,本发明还提供了一种实现基因组编辑的方法,包括在生物体中表达所述基因组编辑系统。
为实现上述目的,本发明还提供了一种产生基因组编辑的植物的方法,包括向植物基因组中引入编码所述基因组编辑系统的核苷酸序列。
为实现上述目的,本发明还提供了一种产生基因组编辑植物种子的方法,包括将所述方法产生的基因组编辑的植物自交,从而获得具有基因组编辑植物种子。
为实现上述目的,本发明还提供了一种培育基因组编辑植物的方法,包括:
种植至少一粒所述方法产生的所述基因组编辑植物种子;
使所述种子长成植株。
为实现上述目的,本发明还提供了一种所述基因组编辑系统在提高同源重组效率和/或提高基因组编辑效率中的用途。
为实现上述目的,本发明还提供了一种foki-dcas9融合蛋白在提高同源重组效率中的用途。
进一步地,所述foki-dcas9融合蛋白的氨基酸序列具有seqidno:4和seqidno:5所示的氨基酸序列。
更进一步地,所述foki-dcas9融合蛋白的核苷酸序列具有seqidno:1第643-5523位所示的核苷酸序列。
本发明所述的foki是包括dna识别结构域和催化(内切核酸酶)结构域的ii型限制性内切核酸酶。本文所述的融合蛋白可包括所有foki或仅仅催化内切核酸酶结构域,例如,基因库登录号aaa24927.1的氨基酸388-583或408-583,例如,如li等,nucleicacidsres.39(1):359–372(2011);cathomen和joung,mol.ther.16:1200–1207(2008)中描述的,或如miller等natbiotechnol25:778–785(2007);szczepek等,natbiotechnol25:786–793(2007);或bitinaite等,proc.natl.acad.sci.usa.95:10570–10575(1998)中描述的foki的突变形式。
本发明中所述的cas9,ⅱ型crispr/cas系统中一种重要的蛋白质成分,可以从生物体如链球菌属物种(streptococcussp.),优选酿脓链球菌(streptococcuspyogenes.)中分离得到。当cas9与称为crisprrna(crrna)和反式激活crrna(tracrrna)的两个rna复合时,形成活性核酸内切酶,从而切断入侵噬菌体或质粒中的外源遗传元件,以保护宿主细胞。crrna从宿主基因组中的crispr元件转录,其中该crispr元件之前自外源入侵物捕获。研究表明通过融合crrna和tracrrna的必要部分产生的单链嵌合rna可以取代cas9/rna复合体中的两个rna以形成功能性核酸内切酶。cas9蛋白质的变体可以是cas9的突变体形式,其中催化性天冬氨酸残基改变为任何其它氨基酸。优选地,所述其它氨基酸可以是丙氨酸。
本发明中所述foki-dcas9融合蛋白,其中foki序列任选地通过间插接头,例如2-30个氨基酸,例如4-12氨基酸的接头,例如gly4ser与dcas9(优选地与dcas9的氨基末端,而且还任选地与羧基末端)融合。在一些实施方案中,所述融合蛋白包括dcas9与foki结构域之间的接头。可用于这些融合蛋白的接头(或串联结构中的融合蛋白之间)可包括不干扰融合蛋白的功能的任何序列。在优选实施方案中,所述接头是短的,例如2-20个氨基酸,并且通常是柔性的(即包含具有高度自由的氨基酸诸如甘氨酸、丙氨酸和丝氨酸)。在一些实施方案中,所述接头包含一个或多个由gggs或ggggs组成的单元,例如,2、3、4或更多个gggs或ggggs单元的重复,还可使用其它接头序列。
本发明所用2a肽(t2a)是一种可“自我剪切的短小肽链”,最初在手足口病毒(foot-and-mouthdiseasevirus,fmda)中发现,平均长度为18-22个氨基酸,2a肽可在蛋白翻译时通过核糖体跳跃从自身最后2个氨基酸c末端断裂(defelipeetal.,2003)。甘氨酸和脯氨酸之间的肽链结合群在2a为电视受损的,能引发核糖体跳跃而从第2个密码子开始翻译,从而使1个转录单元里2个蛋白独立表达。这中2a介导的剪切广泛存在与所有的真核动物细胞当中。利用2a较高的剪切效率及促使上下游基因平衡表达的能力,可以改进异源多聚蛋白(如细胞表面受体、细胞因子、免疫球蛋白等)的表达效率。
本发明中所述的向导rna或引导rna(guiderna,grna),也称为小向导rna(smallguiderna,sgrna),作用于动质体(kinetoplastid)体内一种称为rna编辑(rnaediting)的后转录修饰过程中,也是一种小型非编码rna。可与pre-mrna配对,并在其中插入一些尿嘧啶(u),产生具有作用的mrna。向导rna编辑的rna分子,长度大约是60-80个核苷酸,是由单独的基因转录的,在grna的5’末端有一段锚定区,以特殊的g-u配对方式与非编辑的pre-mrna序列互补,锚定序列促进grna与pre-mrna中的编辑区互补,特意结合;在grna分子的中间部位有一个编辑区负责在被编辑的pre-mrna分子中插入u的位置,其与被编辑mrna精确互补;在grna分子的3’末端,有一段转录后加入的由大约15个非编码的polyu序列,功能为把grna链接到pre-mrna的编辑区的5’上游富含嘌呤碱基的核苷酸序列上。在编辑时,形成一个编辑体(editosome),以grna内部的序列作为模板进行转录物的校正,同时产生编辑的mrna。
有三种类型crispr/cas系统,其中涉及cas9蛋白质和crrna、tracrrna的ⅱ型crispr/cas系统是代表性的。cas9蛋白质通过人工修饰的向导rna的导向作用可以打靶dna序列的5’-n20-ngg-3’(n代表任何脱氧核苷酸碱基),n20是与grna的5’序列相同的20个碱基,ngg是pam区(原型间隔子邻近基序,protospacer-adjacentmotif)。cas9剪切的位点就是pam附近的区域。相对于锌指和转录激活因子样效应物dna结合蛋白提供了优势-因为在核苷酸结合crispr-cas蛋白中的位点特异性由rna分子调控而不是dna结合蛋白调控。
本发明中所述重组,当用于例如细胞、核酸、蛋白质或载体时,表示该细胞、核酸、蛋白质或载体已通过引入异源核酸或蛋白质、或改变天然核酸或蛋白质而被修饰,或该细胞源自此修饰的细胞。
本发明中向导rna可以以编码该向导rna的rna或dna的形式转移到细胞或生物体中。向导rna可以是分离的rna、并入病毒载体的rna的形式、或者在载体中编码。优选地,载体可以是病毒载体、质粒载体、或农杆菌载体。
编码向导rna的dna可以是包含编码向导rna序列的载体。例如,可以通过用分离的向导rna或包含编码向导rna的序列和启动子的质粒dna转染细胞或生物体,将向导rna转染到细胞或生物体。
本发明中所述切割或剪切是指核苷酸分子共价骨架的断裂。向导rna可以制备为特异于任何待切割的靶,通过向导rna的靶特异性部分切割任何靶dna。
本发明中所述非同源末端连接(non-homologousendjoining,nhej)指完全不需要任何模版的帮助,修复蛋白可以直接将dna断裂的末端彼此拉近,再借由dna连接酶的帮助将断裂的末端重新接合的修复机制。
本发明中所述同源重组(homologousrecombination,hr)是指发生在非姐妹染色单体之间或同一染色体上含有同源序列的dna分子之间或分子之内的重新组合。同源重组需要一系列的蛋白质催化,如原核生物细胞内的reca、recbcd、recf、reco、recr等;以及真核生物细胞内的rad51、mre11-rad50等等。同源重组反应通常根据交叉分子或holliday结构的形成和拆分分为三个阶段,即前联会体阶段、联会体形成和holliday结构的拆分。同源重组反应依赖于dna分子之间的同源性,100%同源性的dna分子之间的重组常见于非姐妹染色体之间的同源重组,称为homologousrecombination,而小于100%同源性的dna分子之间或分子之内的重组,则被称为hemologusrecombination。后者可被负责碱基错配对的蛋白如原核细胞内的muts或真核生物细胞内的msh2-3等蛋白质“编辑”。同源重组可以双向交换dna分子,也可以单向转移dna分子,后者又被称为基因转换(geneconversion)。
本发明中,单链退火(ssa)模型是由lin等于1984年提出的。在ssa模型中,重组在dna双链断裂处开始,在单链特异性核酸外切酶的作用下,断裂点两侧逐渐形成dna单链区域,这一过程持续到两个断点处出现互补dna单链。互补dna单链退火,非互补末端被切除,单链缺口修复连接,完成dna重组。ssa模型没有其它模型所需的双链dna的识别配对过程,不形成holliday结构作为重组的中间过渡形式。因此,重组产生dna双链交换,并丢失非退火区域单链dna顺序。
本发明中,所述串联是指将二个或二个以上向导rna(sgrna)排成一串,每个sgrna的首端和前一个sgrna的尾端由csy4切割识别序列连接。
本发明中所述的植物、植物组织或植物细胞的基因组,是指植物、植物组织或植物细胞内的任何遗传物质,且包括细胞核和质体和线粒体基因组。
本发明中所述的多核苷酸和/或核苷酸形成完整“基因”,在所需宿主细胞中编码蛋白质或多肽。本领域技术人员很容易认识到,可以将本发明的多核苷酸和/或核苷酸置于目的宿主中的调控序列控制下。
如本申请,包括权利要求中使用的,除非上下文中清楚地另有说明,否则单数和单数形式的术语,例如“一”、“一个”和“该”,包括复数指代物。因此,例如“植物”、“该植物”或“一个植物”也指示多个植物。并且根据上下文,使用术语“植物”还可指示该植物在遗传上相似或相同的后代。类似地,术语“核酸”可以指核酸分子的许多拷贝。类似地,术语“探针”可以指代相同或相似的探针分子。
数字范围包括限定该范围的数字,并明确地包括所限定的范围内的每个整数和非整数分数。除非另外指出,否则本文所用的全部技术和科学术语具有与本领域普通技术人员普遍理解的相同的含义。
本发明中,术语“核酸”、“核苷酸”、“核苷酸序列”、“寡核苷酸”和“多核苷酸”可互换使用,它们是指具有任何长度的核苷酸的聚合形式,根据上下文含义,可指代dna或rna、或其类似物。其中dna包括但不限于cdna、基因组dna、合成dna(例如人工合成)和含核酸类似物的dna(或rna)。多核苷酸可具有任何三维结构,并且可以执行已知或未知的任何功能。核酸可为双链或单链(既有义链或反义单链)。多核苷酸的非限制性示例包括基因、基因片段、外显子、内含子、信使rna(mrna)、转移rna、核糖体rna、核酶、cdna、重组多核苷酸、分支多核苷酸、质粒、载体、任何序列的分离dna、任何序列的分离rna、核酸探针和引物、以及核酸类似物。
本发明中“野生型”表示生物、菌株、基因的典型形式或者当它在自然界存在时区别于突变体或变体形式的特征。
本发明中“突变体”或“变体”是指发生突变的个体,其具有与野生型不同的序列,可能会导致其中序列的至少部分功能已丢失的序列,例如,在启动子或增强子区域中序列的变化将至少部分地影响生物体中编码序列的表达。术语“突变”是指可由诸如缺失、添加、取代或重排引起的核酸序列中序列的任何变化。突变还可影响该序列参与的一个或多个步骤。例如,dna序列中的变化可导致有活性的、有部分活性的或无活性的改变的mrna和/或蛋白的合成。
本发明中“非天然存在的”表明人工的参与。当指核酸分子或多肽时,表示该核酸分子或多肽至少基本上从它们在自然界中或如发现于自然界中的与其结合的至少另一种组分游离出来。
本发明中“表达”指感兴趣的序列转录产生对应mrna并且该mrna翻译产生对应产物,即肽、多肽或蛋白。调节元件控制或调整感兴趣的序列表达,所述调节元件包括5’调节元件如启动子。
本发明中“多肽”、“肽”和“蛋白质”可互换地使用,是指具有任何长度的氨基酸聚合物。该聚合物可以是直链或支链的,它可以包含修饰的氨基酸,并且它可以被非氨基酸中断。这些术语还涵盖已经被修饰的氨基酸聚合物;这些修饰例如二硫键形成、糖基化、脂化、乙酰化、磷酸化、或任何其他操作,如与标记组分的结合。术语“氨基酸”包括天然的和/或非天然的或者合成的氨基酸,包括甘氨酸以及d和l旋光异构体、以及氨基酸类似物和肽模拟物。
本发明中术语“载体”是指能够在宿主细胞内复制的dna分子。质粒和粘粒是示例性载体。此外,术语“载体”和“媒介”可互换使用以指将dna片段从一种细胞转移至另一种细胞的核酸分子,因此细胞不必要属于相同的生物(例如将dna片段从农杆菌细胞转移至植物细胞)。
本发明中术语“表达载体”是指含有所需要的编码序列和在特定宿主生物中表达有效连接的编码序列所需要的适宜核酸序列的重组dna分子。
本发明中术语“重组表达载体”指来自任何来源、能够整合入基因组或自主复制的任何因子如质粒、粘粒、病毒、bac(细菌人工染色体)、自主复制型序列、噬菌体、或者线性或环状单链或双链dna或rna核苷酸序列,包括其中一种或多种dna序列使用众所周知的重组dna技术以功能性可操作方式连接的dna分子。
在本发明中,“定位结构域”可任选地添加为蛋白部分的部分,定位结构域可将蛋白部分或编程的蛋白部分或组装的复合物定位至活细胞中特定细胞或亚细胞定位。定位结构域可通过将蛋白部分的氨基酸序列融合到掺入包括以下结构域的氨基酸来构建:核定位信号(nls);线粒体前导序列(mls);叶绿体前导序列;和/或被设计以将蛋白运输或引导或定位至含有核酸的细胞器、细胞区室或细胞的任何细分部分的任何序列。在一些实施方案中,生物体是真核生物,且定位结构域包括允许蛋白进入细胞核和基因组dna内的核定位结构域(nls)。所述nls的序列可包括带正电荷序列的任何功能nls。在另一些实施方案中,定位结构域可包括使蛋白部分或编程的核蛋白进入细胞器的前导序列,使细胞器dna被修饰成为可能。
本发明中,真核生物有3类rna聚合酶,负责转录3类不同的启动子。由rna聚合酶i负责转录的rrna基因,启动子(i型)比较单一,由转录起始位点附近的两部分序列构成:第一部分是核心启动子(corepromoter),由-45—+20位核苷酸组成,单独存在时就足以起始转录;另一部分由-170—-107位序列组成,称为上游调控元件,能有效地增强转录效率。
由rna聚合酶ⅲ负责转录的是5srrna、trna和某些核内小分子rna(snrna),其启动子(ⅲ型)组成较复杂,又可被分为两个亚类:一类属于结构基因内启动子,一类属于结构基因外启动子。内启动子的有效启动依赖于基因内部包含的两个不连续的dna片段,这两个dna片段包括一些不同的连续dna序列a、b或c区,两个区之间被隔开。根据两个区的不同组合,可以分为i类和ii类两种:i类包括a和c区,目前发现其仅存在于5srrna的基因中;ii类包括a和b区,存在于trna的基因、7slrna的基因、腺病毒vai与vaii的rna中。a、b或a、c内部dna序列是转录因子tfⅲa和tfⅲc转录启动结合部位。在多数情况下,5’端还会有其它调节或关键元件,这些元件对rna的高效转录是必需的。这些序列的存在与否之间影响着转录效率。这些序列呈现复杂的多样性,不过大多数启动子5’端-30到-20处存在着tata盒样序列,与外启动子相似。外启动子缺乏相应的内部序列,仅在5’端有顺式作用元件,在基因的末端有一组由4个或更多胸腺嘧啶组成的终止信号,如脊椎动物u6核小rna和7skrna启动子,这些启动子都高度相似或完全相同,其位置和碱基序列高度保守,其结构与polii启动子也有一定的相似性。它们的5’端顺式作用元件包括几个控制元件,在其上游约-30处存在一个tata样序列,近-60处有一个snrnapse(snrna近似序列)和一个或多个被称为oct的修饰序列5’-atgcaaat-3’。tata样序列是poliii对snrna基因进行转录特异的。tata样元件和pse元件共同决定了转录起始位点的选择和转录效率。tata样元件和pse元件之间的距离决定了rna聚合酶转录的特异性,但似乎tata样元件更重要,因为在pse缺失的u6rna和7skrna基因转录中,只有转录效率的下降。而且,pse元件可能和b盒(boxb)相关,b盒在一定程度上可以替代pse元件。这些序列对下游基因的转录具有决定性的作用,而且它们与起始位点相距较远,往往超过150bp,而对poliii内启动子来说,一般在80bp以内;与poliii外启动子相比,polii启动子5’端各顺式作用元件的作用刚好相反,如果tata样元件缺失,pse则可以履行tata样元件功能,决定转录的起始部位。在pse上游,外启动子还有一个远端控制序列,其结构与polii增强子oct骨架相似,但较其复杂。而在-223处还有一个cacc序列与oct骨架相连。这些远端控制序列的存在可大大提高u6rna和7skrna的表达效率。
由rna聚合酶ii负责转录的ii型基因包括所有蛋白质编码基因和部分snrna基因,后者的启动子结构与iii型基因启动子中的第三亚类相似,编码蛋白质的ii型基因启动子在结构上有共同的保守序列。转录起始位点没有广泛的序列同源性,但第一个碱基为腺嘌呤,而两侧是嘧啶碱基。这个区域被称为起始子(initiator,inr),序列可表示为py2capy5。inr元件位于-3—+5。仅由inr元件组成的启动子是具有可被rna聚合酶ii识别的最简单启动子形式。多数ii型启动子有一个被称为tata盒的共有序列,通常处于-30区,相对于转录起始位点的位置比较固定。tata盒存在于所有真核生物中,tata盒是一个保守的七碱基对,也有一些ii型启动子不含有tata盒,这样的启动子称为无tata盒启动子。
本发明中所述“有效连接”或“可操作地连接”表示核酸序列的联结,所述联结使得一条序列可提供对相连序列来说需要的功能。在本发明中所述“有效连接”可以为将启动子与感兴趣的序列相连,使得该感兴趣的序列的转录受到该启动子控制和调控。当感兴趣的序列编码蛋白并且想要获得该蛋白的表达时“有效连接”表示:启动子与所述序列相连,相连的方式使得得到的转录物高效翻译。如果启动子与编码序列的连接是转录物融合并且想要实现编码的蛋白的表达时,制造这样的连接,使得得到的转录物中第一翻译起始密码子是编码序列的起始密码子。备选地,如果启动子与编码序列的连接是翻译融合并且想要实现编码的蛋白的表达时,制造这样的连接,使得5’非翻译序列中含有的第一翻译起始密码子与启动子相连结,并且连接方式使得得到的翻译产物与编码想要的蛋白的翻译开放读码框的关系是符合读码框的。可以“有效连接”的核酸序列包括但不限于:提供基因表达功能的序列(即基因表达元件,例如启动子、5’非翻译区域、内含子、蛋白编码区域、3’非翻译区域、聚腺苷化位点和/或转录终止子)、提供dna转移和/或整合功能的序列(即t-dna边界序列、位点特异性重组酶识别位点、整合酶识别位点)、提供选择性功能的序列(即抗生素抗性标记物、生物合成基因)、提供可计分标记物功能的序列、体外或体内协助序列操作的序列(即多接头序列、位点特异性重组序列)和提供复制功能的序列(即细菌的复制起点、自主复制序列、着丝粒序列)。
本发明中,调节元件可操作地连接至crispr系统的一个或多个元件,从而驱动该crispr系统的所述一个或多个元件的表达。一般而言,crispr(规律间隔成簇短回文重复),也称为spidr(spacer间隔开的同向重复),构成通常对于特定细菌物种而言特异性的dna基因座的家族。该crispr座位包含在大肠杆菌中被识别的间隔开的短序列重复(ssr)的一个不同类、以及相关基因。类似的间隔开的ssr已经鉴定于地中海富盐菌、化脓链球菌、鱼腥草属和结核分枝杆菌中。这些crispr座位典型地不同于其它ssr的重复结构,这些重复已被称为规律间隔的短重复(srsr)。一般而言,这些重复是以簇存在的短元件,其被具有基本上恒定长度的独特间插序列规律地间隔开。虽然重复序列在菌株之间是高度保守的,许多间隔开的重复和这些间隔区的序列一般在菌株与菌株之间不同,已经在40种以上的原核生物中鉴定出crispr座位。
本发明中,“靶序列”或“靶点序列”或“靶位点序列”或“靶多核苷酸”是待被作用的任何期望的预定的核酸序列,包括但不限于编码或非编码序列、基因、外显子或内含子、调节序列、基因间序列、合成序列和细胞内寄生物序列。在一些实施方案中,靶序列存在于靶细胞、组织、器官或生物体内。
术语“引物”是一段分离的核酸分子,其通过核酸杂交,退火结合到互补的目标dna链上,在引物和目标dna链之间形成杂合体,然后在聚合酶(例如dna聚合酶)的作用下,沿目标dna链延伸。本发明的引物对涉及其在目标核酸序列扩增中的应用,例如,通过聚合酶链式反应(pcr)或其他常规的核酸扩增方法。
引物的长度一般是11个多核苷酸或更多,优选的是18个多核苷酸或更多,更优选的是24个多核苷酸或更多,最优选的是30个多核苷酸或更多。这种引物在高度严格杂交条件下与目标序列特异性地杂交。尽管不同于目标dna序列且对目标dna序列保持杂交能力的引物是可以通过常规方法设计出来的,但是,优选的,本发明中的引物与目标序列的连续核酸具有完全的dna序列同一性。
本发明的引物在严格条件下与目标dna序列杂交。核酸分子或其片段在一定情况下能够与其他核酸分子进行特异性杂交。如本发明使用的,如果两个核酸分子能形成反平行的双链核酸结构,就可以说这两个核酸分子彼此间能够进行特异性杂交。如果两个核酸分子显示出完全的互补性,则称其中一个核酸分子是另一个核酸分子的“互补物”。如本发明使用的,当一个核酸分子的每一个核苷酸都与另一个核酸分子的对应核苷酸互补时,则称这两个核酸分子显示出“完全互补性”。如果两个核酸分子能够以足够的稳定性相互杂交从而使它们在至少常规的“低度严格”条件下退火且彼此结合,则称这两个核酸分子为“最低程度互补”。类似地,如果两个核酸分子能够以足够的稳定性相互杂交从而使它们在常规的“高度严格”条件下退火且彼此结合,则称这两个核酸分子具有“互补性”。从完全互补性中偏离是可以允许的,只要这种偏离不完全阻止两个分子形成双链结构。为了使一个核酸分子能够作为引物或探针,仅需保证其在序列上具有充分的互补性,以使得在所采用的特定溶剂和盐浓度下能形成稳定的双链结构。
术语“特异性结合(目标序列)”是指在严格杂交条件下引物仅与包含目标序列的样品中的目标序列发生杂交。
本发明中,“试剂盒”可包括本发明描述的基因组修饰系统与以下的任一种或全部:测定试剂、缓冲液、探针和/或引物、和无菌盐水或另一种药学上可接受的乳剂和悬液基底。此外,试剂盒可包括含有用于实践本发明描述的方法的用法说明(例如,操作方案)的说明性材料。
转化方案以及将核苷酸序列导入植物的方案依定向转化的植物或植物细胞类型而异,即单子叶植物或双子叶植物。将核苷酸序列导入植物细胞并随后插入植物基因组中的适合方法包括但不限于,农杆菌介导的转化、微量发射轰击、直接将dna摄入原生质体、电穿孔或晶须硅介导的dna导入。已经转化的细胞可按照常规的方式生长成植物。这些植物被培育,用相同的转化株或不同的转化株授粉,得到的杂交体表达所需的被鉴定的表现型特征。可培育二代或多代以保证稳定地保持和遗传所需表现型特征的表达,然后收获可保证得到所需表现型特征表达的种子。
本发明提供了一种提高同源重组效率的方法,具有以下优点:
1、本发明首次将foki-dcas9融合蛋白应用于提高同源重组效率,利用foki二聚体的内切酶活性,使得同源重组靶位点被切开的概率提高,以提高同源重组效率。
2、本发明为基因组编辑技术实现高效同源重组编辑提供了一种新的选择,提高同源重组效率的同时,也减少了转化受体的使用量。
下面通过附图和实施例,对本发明的技术方案做进一步的详细描述。
附图说明
图1为本发明提高同源重组效率方法中重组克隆剪刀载体dbn01-t构建流程图;
图2为本发明提高同源重组效率方法中重组表达载体dbn-get326构建流程图;
图3为本发明提高同源重组效率方法中重组表达载体dbn-get344载体结构示意图;
图4为本发明提高同源重组效率方法中重组表达载体dbn-get345载体结构示意图;
图5为本发明提高同源重组效率方法中水稻抗性愈伤gus染色标准图。
具体实施方式
下面通过具体实施例进一步说明本发明提高同源重组效率方法的技术方案。
第一实施例、剪刀载体构建
1.构建基础载体和重组克隆剪刀载体
将pcambia2300(cambia机构可以提供)载体进行改造,利用常规的酶切方法构建载体是本领域技术人员所熟知的,通过点突变去掉pcambia2300载体上的bsai位点,同时将卡那霉素表达盒去掉,得到pdbn骨架载体。向所述pdbn骨架载体引入pat表达盒,得到表达载体dbn-pat用于下述载体构建。
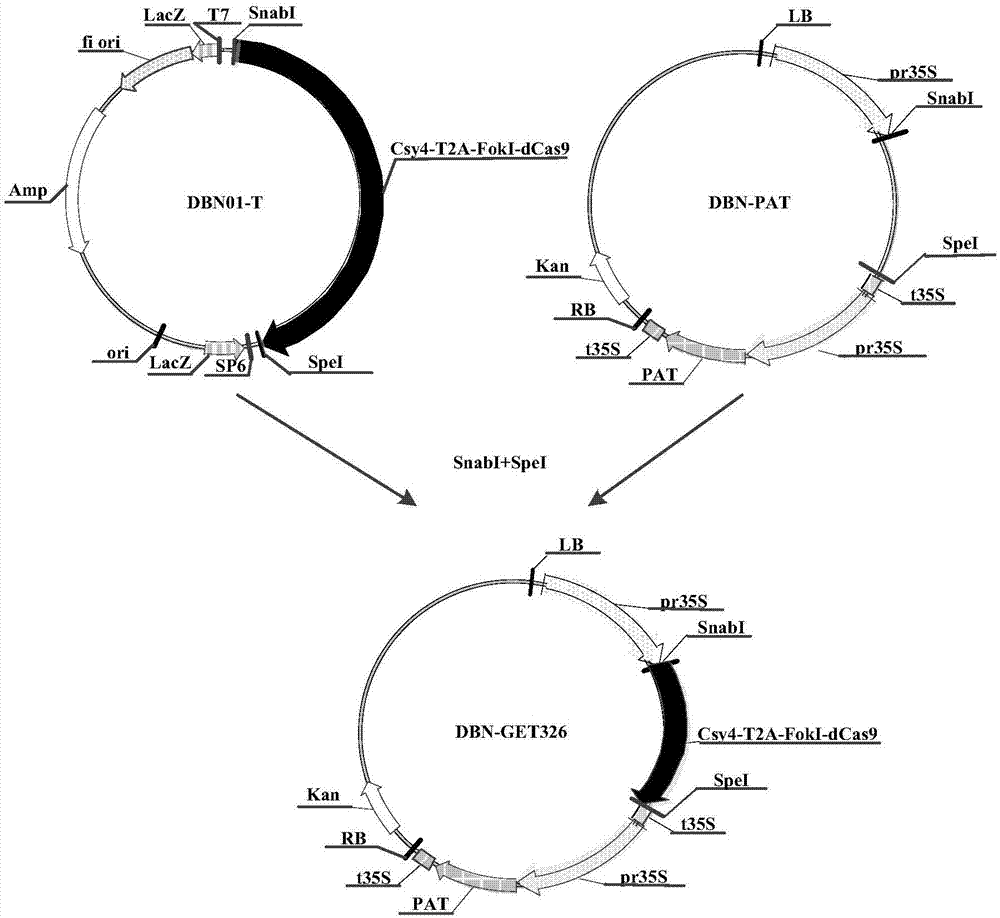
将合成的csy4-t2a-foki-dcas9核苷酸序列连入克隆载体pgem-t(promega,madison,usa,cat:a3600)上,操作步骤按promega公司产品pgem-t载体说明书进行,得到重组克隆剪刀载体dbn01-t,其构建流程如图1所示(其中,amp表示氨苄青霉素抗性基因;f1表示噬菌体f1的复制起点;lacz为lacz起始密码子;sp6为sp6rna聚合酶启动子;t7为t7rna聚合酶启动子;csy4-t2a-foki-dcas9为csy4-t2a-foki-dcas9核苷酸序列(csy4-t2a-foki-dcas9核苷酸序列如seqidno:1所示;csy4氨基酸序列如seqidno:2所示,t2a多肽序列如seqidno:3所示;foki的氨基酸序列如seqidno:4所示;dcas9氨基酸序列如seqidno:5所示);mcs为多克隆位点)。
然后将重组克隆剪刀载体dbn01-t用热激方法转化大肠杆菌t1感受态细胞(transgen,beijing,china,cat:cd501),其热激条件为:50μl大肠杆菌t1感受态细胞、10μl质粒dna(重组克隆剪刀载体dbn01-t),42℃水浴90秒;37℃振荡培养1小时(100rpm转速下摇床摇动),在表面涂有iptg(异丙基硫代-β-d-半乳糖苷)和x-gal(5-溴-4-氯-3-吲哚-β-d-半乳糖苷)的氨苄青霉素(100mg/l)的lb固体培养基(胰蛋白胨10g/l,酵母提取物5g/l,nacl10g/l,琼脂15g/l,用naoh调ph至7.5)上生长过夜。挑取白色菌落,在lb液体培养基(胰蛋白胨10g/l,酵母提取物5g/l,nacl10g/l,氨苄青霉素100mg/l,用naoh调ph至7.5)中于温度37℃条件下培养过夜。碱法提取其质粒:将菌液在12000rpm转速下离心1min,去上清液,沉淀菌体用100μl冰预冷的溶液i(25mmtris-hcl,10mmedta(乙二胺四乙酸),50mm葡萄糖,ph8.0)悬浮;加入200μl新配制的溶液ii(0.2mnaoh,1%sds(十二烷基硫酸钠)),将管子颠倒4次,混合,置冰上3-5min;加入150μl冰冷的溶液iii(3m醋酸钾,5m醋酸),立即充分混匀,冰上放置5-10min;于温度4℃、转速12000rpm条件下离心5min,在上清液中加入2倍体积无水乙醇,混匀后室温放置5min;于温度4℃、转速12000rpm条件下离心5min,弃上清液,沉淀用浓度(v/v)为70%的乙醇洗涤后晾干;加入30μl含rnase(20μg/ml)的te(10mmtris-hcl,1mmedta,ph8.0)溶解沉淀;于温度37℃下水浴30min,消化rna;于温度-20℃保存备用。
提取的质粒经snabi和spei酶切鉴定后,对阳性克隆进行测序验证,结果表明重组克隆剪刀载体dbn01-t中插入的所述foki-dcas9核苷酸序列为序列表中seqidno:1所示的核苷酸序列,即csy4-t2a-foki-dcas9核苷酸序列正确插入。
2.构建重组表达剪刀载体
用限制性内切酶snabi和spei分别酶切重组克隆剪刀载体dbn01-t和表达载体dbn-pat,将切下的csy4-t2a-foki-dcas9核苷酸序列插入表达载体dbn-pat,利用常规的酶切方法构建载体是本领域技术人员所熟知的,构建成重组表达载体dbn-get326,其构建流程如图2所示(rb:右边界;pr35s:花椰菜花叶病毒35s启动子(seqidno:6);csy4-t2a-foki-dcas9:csy4-t2a-foki-dcas9核苷酸序列(csy4-t2a-foki-dcas9核苷酸序列如seqidno:1所示;csy4氨基酸序列如seqidno:2所示;t2a多肽序列如seqidno:3所示;foki的氨基酸序列如seqidno:4所示;dcas9氨基酸序列如seqidno:5所示);t35s:花椰菜花叶病毒35s终止子(seqidno:7);pat:草丁膦乙酰转移酶基因(seqidno:8);lb:左边界)。
将重组表达载体dbn-get326用热激方法转化大肠杆菌t1感受态细胞,其热激条件为:50μl大肠杆菌t1感受态细胞、10μl质粒dna(重组表达载体dbn-get326),42℃水浴90秒;37℃振荡培养1小时(100rpm转速下摇床摇动);然后在含50mg/l卡那霉素(kanamycin)的lb固体培养基(胰蛋白胨10g/l,酵母提取物5g/l,nacl10g/l,琼脂15g/l,用naoh调ph至7.5)上于温度37℃条件下培养12小时,挑取白色菌落,在lb液体培养基(胰蛋白胨10g/l,酵母提取物5g/l,nacl10g/l,卡那霉素50mg/l,用naoh调ph至7.5)中于温度37℃条件下培养过夜。碱法提取其质粒。将提取的质粒用限制性内切酶snabi和spei酶切后鉴定,并将阳性克隆进行测序鉴定,结果表明重组表达载体dbn-get326在snabi和spei位点间的核苷酸序列为序列表中seqidno:1所示核苷酸序列,即csy4-t2a-foki-dcas9核苷酸序列。
第二实施例、水稻guus验证载体的构建
1、guus靶点的选择
将guus之间的靶序列信息输入到zifit网站
(http://zifit.partners.org/zifit/choicemenu.aspx)中,选择一对可用的靶点,即靶点1序列(如seqidno:9所示)和靶点2序列(如seqidno:10所示)。
2、水稻无靶点载体的构建
本实施例中,无靶点载体设计为prosu6+sgrna+t35s结构。向第一实施例中所述pdbn骨架载体引入pmi表达盒、guus表达盒,利用常规的酶切方法构建载体是本领域技术人员所熟知的,构建成水稻无靶点载体dbn-get344,其载体结构示意图如图3所示(lb:左边界;prosu6:水稻u6启动子(seqidno:14);csy4-r:csy4切割识别序列(如seqidno:11所示);sgrna:sgrna序列(如seqidno:15所示);t35s:花椰菜花叶病毒35s终止子(seqidno:7);pr35s:花椰菜花叶病毒35s启动子(seqidno:6);guus:含有靶点1序列和靶点2序列的gus基因(如seqidno:16所示);tnos:胭脂碱合成酶基因的终止子(seqidno:17);prubi:玉米泛素(ubiquitin)基因启动子(seqidno:18);pmi:磷酸甘露糖异构酶基因(seqidno:19);rb:右边界)。
按照第一实施例2中的方法,将无靶点载体dbn-get344用热激方法转化大肠杆菌;碱法提取的质粒经asci和avrii酶切鉴定后,对阳性克隆进行测序验证,结果表明无靶点载体dbn-get344构建正确。
3、水稻靶点载体的构建
本实施例中,靶点载体设计为prosu6+靶点+sgrna+t35s结构。两个靶点+sgrna之间连接有csy4切割识别序列。通过限制性内切酶bsai将各个片段无缝连接在一起。csy4切割识别序列如seqidno:11所示。本实施例中使用的2个靶点为:
靶点1序列,如seqidno:9所示;
靶点2序列,如seqidno:10所示。
引入靶点1和靶点2的引物如下:
正向引物:acatcaggtctccaaacggaggcattggtgcttcttggttttagagctagaaata,如seqidno:12所示;
反向引物:taggatggtctcgaaaacgtcgaggatgcctgggttgcctgcctatacggcagtgaacgcac,如seqidno:13所示;
其中,上述引物5’端的粗体小写字母为保护碱基,斜体小写字母为酶切位点bsai,带下划线的小写字母为酶切位点bsai的粘性末端。
以合成的sgrna+cys4识别序列为模板(扩增体系中<250ng),将靶点1序列和靶点2序列通过上述正向引物和反向引物带入模板,用pfu酶(neb)进行pcr扩增,pcr体系如下:
pcr反应条件为:98℃预变性30s,然后进入下列循环:98℃变性10s,56-60℃退火30s,72℃延伸30s/kb,共30-32个循环,最后72℃延伸5-10min;于4℃保存。
pcr扩增后得到含有靶位点序列+sgrna+csy4切割识别序列的产物,使用过柱纯化试剂盒(购自北京全式金生物技术有限公司)对上述pcr产物过柱纯化,具体方法参考其产品说明书;bsai酶切pcr产物和表达载体dbn-get344,切胶回收对应酶切产物后,将酶切后的表达载体dbn-get344产物和pcr产物按照1:10的比例用t4连接酶于温度16℃下连接30min,利用常规的酶切方法构建载体是本领域技术人员所熟知的,构建成水稻靶点载体dbn-get345,其载体结构示意图如图4所示(lb:左边界;prosu6:水稻u6启动子(seqidno:14);csy4-r:csy4切割识别序列(如seqidno:11所示);靶点1:靶1序列(seqidno:9);靶点2:靶点2序列(seqidno:10);sgrna:sgrna序列(如seqidno:15所示);t35s:花椰菜花叶病毒35s终止子(seqidno:7);pr35s:花椰菜花叶病毒35s启动子(seqidno:6);guus:含有靶点1序列和靶点2序列的gus基因(如seqidno:16所示);tnos:胭脂碱合成酶基因的终止子(seqidno:17);prubi:玉米泛素(ubiquitin)基因启动子(seqidno:18);pmi:磷酸甘露糖异构酶基因(seqidno:19);rb:右边界)。
按照第一实施例2中的方法,将靶点载体dbn-get345用热激方法转化大肠杆菌;碱法提取的质粒经kpni和asci酶切鉴定后,对阳性克隆进行测序验证,结果表明靶点载体dbn-get345中2个靶点(靶点1和靶点2)正确插入。
第三实施例、剪刀载体和guus验证载体转化农杆菌
将己经构建正确的重组表达载体dbn-get326、dbn-get344和dbn-get345用液氮法转化到农杆菌lba4404中,其转化条件为:100μl农杆菌lba4404、3μl质粒dna(重组表达载体);置于液氮中5分钟,37℃温水浴5分钟;将转化后的农杆菌lba4404接种于lb试管中于温度28℃、转速为200rpm条件下培养2小时,涂于含50mg/l的利福平(rifampicin)和50mg/l的卡那霉素的lb固体培养基上直至长出阳性单克隆,挑取单克隆培养并提取其质粒,用限制性内切酶进行酶切验证,结果表明重组表达载体dbn-get326、dbn-get344和dbn-get345结构完全正确。
按照以下组合将菌液等体积混合:dbn-get326和dbn-get345菌液(靶点处理)、dbn-get326和dbn-get344菌液(无靶点处理)、dbn-get344菌液(对照处理),室温静置3h,获得对应处理的农杆菌悬浮液。
第四实施例、获得稳定转化的水稻愈伤
对于农杆菌介导的水稻转化,简要地,把水稻种子(日本晴,中国农业大学提供)接种在诱导培养基(n6盐3.1g/l、n6维他命、干酪素300mg/l、麦芽糖30g/l、2,4-二氯苯氧乙酸(2,4-d)2mg/l、植物凝胶3g/l,ph5.8)上,从水稻成熟胚诱导出愈伤组织(步骤1:愈伤诱导步骤),之后,优选愈伤组织,用上述3种处理的农杆菌悬浮液接触愈伤组织,其中农杆菌能够将目的构建体传递至愈伤组织上的至少一个细胞(步骤2:侵染步骤)。在此步骤中,愈伤组织优选地浸入农杆菌悬浮液(od660=0.3,侵染培养基(n6盐3.1g/l、n6维他命、干酪素300mg/l、蔗糖30g/l、葡萄糖10g/l、乙酰丁香酮(as)40mg/l、2,4-二氯苯氧乙酸(2,4-d)2mg/l、ph5.4))中以启动接种。愈伤组织与农杆菌共培养一段时期(3天)(步骤3:共培养步骤)。优选地,愈伤组织在侵染步骤后在固体培养基(n6盐3.1g/l、n6维他命、干酪素300mg/l、蔗糖30g/l、葡萄糖10g/l、乙酰丁香酮(as)40mg/l、2,4-二氯苯氧乙酸(2,4-d)2mg/l、植物凝胶3g/l,ph5.8)上培养。在此共培养阶段后,有一个“恢复”步骤。在“恢复”步骤中,恢复培养基(n6盐3.1g/l、n6维他命、干酪素300mg/l、蔗糖30g/l、2,4-二氯苯氧乙酸(2,4-d)2mg/l、植物凝胶3g/l,ph5.8)中至少存在一种己知抑制农杆菌生长的抗生素(头孢霉素150-250mg/l),不添加植物转化体的选择剂(步骤4:恢复步骤)。优选地,愈伤组织在有抗生素但没有选择剂的固体培养基上培养,以消除农杆菌并为侵染细胞提供恢复期。接着,接种的愈伤组织在含选择剂(甘露糖和/或草铵膦)的培养基上培养并选择生长着的转化愈伤组织(步骤5:选择步骤)。优选地,所述靶点处理的愈伤组织和所述无靶点处理的愈伤组织在有甘露糖和草铵膦的筛选固体培养基(n6盐3.1g/l、n6维他命、干酪素300mg/l、蔗糖5g/l、甘露糖12.5g/l、草胺磷4mg/l、2,4-二氯苯氧乙酸(2,4-d)2mg/l、植物凝胶3g/l,ph5.8)上培养,所述对照处理的愈伤组织在有甘露糖的筛选固体培养基(n6盐3.1g/l、n6维他命、干酪素300mg/l、蔗糖5g/l、甘露糖12.5g/l、2,4-二氯苯氧乙酸(2,4-d)2mg/l、植物凝胶3g/l,ph5.8)上培养,导致转化的细胞选择性生长。将上述筛选得到的抗性愈伤组织进行gus染色分析。
第五实施例、水稻愈伤的gus染色测定
分别取稳定转化获得的靶点处理的抗性愈伤组织、无靶点处理的抗性愈伤组织和对照处理的抗性愈伤组织作为样品,参照jefferson等(jeffersonr.a.,burgesss.m.,hirshd.beta-glucuronidasefromescherichiacoliasagenefusionmarker.proc.natl.acad.sci.,1986,83:8447-8454)的方法,通过在gus染色液中37℃密封染色1-2天,从组织化学上检验gus的表达方式,即guus突变为gus酶后会将x-gluc原位分解产生蓝色沉淀,从而可以说明foki-dcas9有助于使guus恢复gus染色。每个处理设3次重复,每个重复做10个抗性愈伤组织,取平均值。具体方法如下:
步骤1、5-溴-4-氯-3-吲哚-β-d-葡糖苷酸(x-gluc)按照40mg/ml的浓度溶解到二甲基亚砜(dmso)中,用锡箔纸密封后置于-80℃冰箱中保存;
步骤2、配制gus染色液:100mmnah2po4、10mmna2edta、0.5mmk4[fe(cn)6]·3h2o、0.5mmk3[fe(cn)6]、体积比为1%的聚乙二醇辛基苯基醚(tritonx-100),用ph计调ph至7.0,加水定容至1l;
步骤3、在步骤2配制好的gus染色液中加入步骤1密封保存的x-gluc,使x-gluc终浓度为0.5mg/ml,用于gus染色;
步骤4、分别取靶点处理的抗性愈伤组织、无靶点处理的抗性愈伤组织和对照处理的抗性愈伤组织各30个,每3粒抗性愈伤组织放入1支2ml离心管中,加入步骤3获得的gus染色液并没过样品,于37℃恒温箱中放置24-48h后,目测染色情况。
gus染色结果如表1所示,在上述gus染色验证同源重组效率的实验中,将gus染色的程度分为四个等级,即+++、++、+、-,依次表示大部分细胞深蓝色、仅不到一半细胞蓝色、少数细胞蓝色、无蓝色),gus染色标准如图5所示,gus染色实验结果如表1所示,约有24%所述靶点处理的抗性愈伤组织发生了gus回复突变(染色程度为+的占14.00%),说明靶点与foki-dcas9融合蛋白共转化时可以促进同源重组的发生;仅有3.00%的所述对照处理的抗性愈伤组织的少数细胞被gus染成蓝色(染色程度为+);值得注意的是,在没有靶点存在的情况下,约有17.20%所述无靶点处理的抗性愈伤组织发生了gus回复突变(染色程度为+),比所述对照处理的抗性愈伤组织的同源重组效率提高4.7倍,说明当不存在切割(无靶点)时,foki-dcas9融合蛋白能单独促进同源重组的产生,并且通过过量表达能显著提高同源重组效率。
综上所述,本发明了首次提供了foki-dcas9融合蛋白能够促进同源重组的发生,并且显著提高细胞内同源重组效率,同时也大大的减少了对转化受体的需求,为高效同源重组编辑提供了一种新的选择。
最后所应说明的是,以上实施例仅用以说明本发明的技术方案而非限制,尽管参照较佳实施例对本发明进行了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的精神和范围。
sequencelisting
<110>北京大北农生物技术有限公司
<120>提高同源重组效率的方法
<130>dbnbc120
<160>19
<170>patentinversion3.3
<210>1
<211>5523
<212>dna
<213>artificialsequence
<220>
<223>csy4-t2a-foki-dcas9核苷酸序列
<400>1
atgggcgaccactacctggacatcaggctgaggccggacccggagttcccgccggcccag60
ctgatgagcgtgctgttcggcaagctgcaccaggcactggtggcccagggcggcgacagg120
atcggcgtgagcttcccggacctggacgagagcaggagcaggctgggcgagaggctgaga180
atccacgccagcgccgacgacctgagggcactgctggccaggccgtggctggagggcctg240
agggaccacctgcaattcggcgagccggccgtggtgccgcacccgaccccgtacaggcag300
gtgagcagggtgcaggccaagagcaacccggagaggctgaggaggaggctgatgaggagg360
cacgacctgagcgaggaggaggccaggaagagaatcccggacaccgtggcaagggccctg420
gacctgccgttcgtgaccctgaggagccagagcaccggccagcacttcaggctgttcatc480
aggcacggcccgctacaggtgaccgccgaggagggcggcttcacctgctacggcctgagc540
aagggcggcttcgtgccgtggttcgagggcaggggcagcctgctgacctgcggcgacgtg600
gaggagaacccgggcccgatgccgaagaagaagaggaaggtgtcctcccagctcgtgaag660
tccgagctcgaggagaagaagtccgagctccgccacaagctcaagtacgtgccgcacgag720
tacatcgagctcatcgagatcgcccgcaactccacccaggaccgcatcctcgagatgaag780
gtgatggagttcttcatgaaggtgtacggctaccgcggcaagcacctcggcggctcccgc840
aagccggacggcgccatctacaccgtgggctccccgatcgactacggcgtgatcgtggac900
accaaggcctactccggcggctacaacctcccgatcggccaggccgacgagatgcagcgc960
tacgtggaggagaaccagacccgcaacaagcacatcaacccgaacgagtggtggaaggtg1020
tacccgtcctccgtgaccgagttcaagttcctcttcgtgtccggccacttcaagggcaac1080
tacaaggcccagctcacccgcctcaaccacatcaccaactgcaacggcgccgtgctctcc1140
gtggaggagctcctcatcggcggcgagatgatcaaggccggcaccctcaccctcgaggag1200
gtgcgccgcaagttcaacaacggcgagatcaacttcggcggcggcggcagcatggactac1260
aaggaccacgacggggattacaaagaccacgacatagactacaaggatgacgatgacaaa1320
atggcaccgaagaaaaaaaggaaggtcggaatccatggcgttccagctgccgataagaaa1380
tattccatcggactcgccattggcacgaatagcgtcggatgggctgttattactgatgag1440
tacaaagttccgtctaagaagttcaaggtgctgggcaacacagaccgccacagcataaag1500
aaaaatctcatcggtgcactccttttcgatagtggggagactgcagaagcgacaagattg1560
aaaaggactgcgagaaggcgctatacacggcgtaagaatagaatctgctaccttcaggag1620
attttctctaacgaaatggctaaggtcgatgacagtttctttcatagacttgaggaatcg1680
ttcttggttgaggaggataagaaacatgagaggcacccgatatttggaaacatcgtggat1740
gaggtcgcatatcatgaaaagtaccccacaatctaccacctgagaaagaaactcgttgat1800
tccaccgacaaagcggatttgagactcatctacctcgctcttgcccatatgataaagttc1860
cgcggacactttctgatcgagggcgacctcaaccctgataatagcgacgtcgataagctc1920
ttcatccagttggttcaaacctacaatcagctctttgaggaaaacccaattaatgctagt1980
ggagtggatgcaaaagcgatactgtcggccagactctccaagagcagaaggttggagaac2040
ctgatcgctcaacttcctggagaaaagaaaaacggtctttttgggaatttgattgccttg2100
tctctgggcctcacaccaaacttcaagtcaaattttgacctcgctgaggatgccaaactt2160
cagttgtctaaggatacctatgatgacgatcttgacaatttgctggcacaaattggcgac2220
cagtacgcggatctgttcctcgcagcgaagaatctgagtgatgctattctcctttcggac2280
atactcagggttaacactgagatcacaaaagcacctttgagtgcgtcgatgattaagcgc2340
tatgatgaacatcaccaagacctcactttgctgaaggcccttgtgcggcagcaattgcca2400
gagaagtacaaagaaatcttctttgaccaatctaagaacggatacgctggctatattgat2460
ggaggagcttctcaggaggaattctataagtttatcaaacctatacttgagaagatggat2520
ggtacagaggaactccttgttaaattgaacagagaagatttgctgcgcaagcaacggacc2580
tttgacaacggatcaattccgcatcagatacacctcggcgagcttcatgccatccttcgc2640
cggcaggaagatttctacccctttttgaaggacaaccgcgagaagatagaaaaaatcctt2700
acgttccggattccttactatgtgggtccattggcaagggggaattcccgctttgcgtgg2760
atgactcggaaaagcgaggaaactatcacaccgtggaacttcgaggaagttgtggacaag2820
ggagcttctgcccaatcattcattgagaggatgactaacttcgataagaacctgccgaac2880
gagaaagttctccccaagcactccctcctttacgagtatttcaccgtgtataacgaactt2940
acgaaggttaaatacgtgactgagggtatgaggaagccagcattcttgagcggggaacaa3000
aagaaagcgattgttgatttgctgtttaaaactaatcgcaaggtgacagtcaagcagctc3060
aaagaggattatttcaagaaaattgaatgtttcgactctgtggagatatcaggagtcgaa3120
gataggtttaacgcttcccttggcacataccatgacctccttaagatcattaaggacaaa3180
gatttcctggataacgaggaaaatgaggacatcctcgaagatattgttcttaccttgacg3240
ctgtttgaggatcgcgaaatgatcgaggaacggcttaagacgtatgctcacttgttcgac3300
gataaggttatgaagcagctcaagcgtagaaggtacactggatggggccgtctgtctaga3360
aagctcatcaacggaatacgtgataaacaaagtggcaagacaattttggattttctgaag3420
tcggacggattcgccaacagaaattttatgcagctgattcatgacgatagtctcaccttc3480
aaagaggacatacagaaggctcaagtgagtggtcaaggggattcgctgcatgaacacatc3540
gcaaacctcgcgggttcaccggccataaagaaaggaatccttcaaactgttaaggtcgtt3600
gatgagttggttaaagtgatgggtaggcacaagcccgaaaacatagtgatcgagatggct3660
cgcgaaaatcagactacacaaaaagggcagaagaactctcgcgagcggatgaaaaggatt3720
gaggaaggaatcaaggaactgggctcacagattctcaaagagcatccagtcgaaaacaca3780
cagctgcaaaatgagaagctctatctttactatctccaaaatggccgggacatgtatgtt3840
gatcaggagcttgacatcaaccgtttgtccgactatgatgtggacgccattgtcccgcaa3900
tctttccttaaggacgattcaatcgataataaggtgttgacccggagcgataaaaaccgt3960
ggaaagtctgacaatgtcccttcagaggaagtggttaagaagatgaagaactactggaga4020
caattgctgaatgcaaaactgatcacacagagaaagttcgacaacctcaccaaagcagag4080
agaggtgggctcagtgaacttgataaagcgggcttcattaagcgtcagctcgttgagact4140
agacagatcacgaagcatgtcgcgcagattttggattcgcggatgaacacgaagtacgac4200
gagaatgataaactgatacgtgaagtcaaggttatcactcttaagtccaaattggtgagc4260
gatttcagaaaggacttccaattctataaggtcagggagatcaacaattatcatcacgct4320
cacgatgcctaccttaatgctgttgtggggaccgcccttattaagaaataccctaaattg4380
gagtctgaattcgtttacggggattataaggtctacgacgttaggaaaatgatagctaag4440
agtgagcaggagatcggtaaagcaactgcgaagtatttcttttactcgaacatcatgaat4500
ttctttaagaccgagataacgctggcaaatggcgaaattagaaagaggcctctcatagag4560
actaacggtgagacaggggaaatcgtctgggataagggtagggactttgcgacagtgcgc4620
aaggtcctctctatgccgcaagttaatattgtgaagaaaaccgaggtgcagacgggaggc4680
ttctccaaggaaagcatacttcccaaacggaactctgataagttgatcgctcgtaagaaa4740
gattgggaccctaagaaatatggtgggttcgattccccaactgttgcttacagcgtgctg4800
gtcgttgccaaggtcgagaagggtaaatccaagaaactcaaaagcgttaaggaactcctt4860
gggattactatcatggagagatcttcattcgaaaagaatcctatcgactttcttgaggcc4920
aaaggatataaggaagttaagaaagatctgataatcaaactcccaaagtactcattgttt4980
gagctggaaaacggcaggaagcgcatgcttgcttccgccggagagttgcagaaagggaac5040
gagttggctctgccttctaagtatgttaacttcctctatcttgcctctcattacgagaag5100
ctcaaaggctcaccagaggacaacgaacagaaacaactttttgtcgagcaacataagcac5160
tatttggatgagattatagaacagatcagtgaattctcgaaaagggttatccttgcagat5220
gcgaatcttgacaaggtgttgtctgcatacaacaaacatagagataagccgatcagggag5280
caagcggaaaatatcattcacctcttcactcttacaaacttgggtgctcccgctgccttc5340
aagtattttgataccacgattgaccggaaacgttacacctcaacgaaggaggtgctggat5400
gccaccctcatccaccaatctattaccggactctacgagactagaatcgatctctcacag5460
ctcggcggggataaaagaccagcagcgacgaaaaaggcaggacaggctaagaagaagaaa5520
tag5523
<210>2
<211>188
<212>prt
<213>pseudomonasaeruginosa
<400>2
metglyasphistyrleuaspileargleuargproaspprogluphe
151015
proproalaglnleumetservalleupheglylysleuhisglnala
202530
leuvalalaglnglyglyaspargileglyvalserpheproaspleu
354045
aspgluserargserargleuglygluargleuargilehisalaser
505560
alaaspaspleuargalaleuleualaargprotrpleugluglyleu
65707580
argasphisleuglnpheglygluproalavalvalprohisprothr
859095
protyrargglnvalserargvalglnalalysserasnprogluarg
100105110
leuargargargleumetargarghisaspleusergluglugluala
115120125
arglysargileproaspthrvalalaargalaleuaspleuprophe
130135140
valthrleuargserglnserthrglyglnhispheargleupheile
145150155160
arghisglyproleuglnvalthralaglugluglyglyphethrcys
165170175
tyrglyleuserlysglyglyphevalprotrpphe
180185
<210>3
<211>18
<212>prt
<213>foot-and-mouthdiseasevirus
<400>3
gluglyargglyserleuleuthrcysglyaspvalglugluasnpro
151015
glypro
<210>4
<211>198
<212>prt
<213>flavobacteriumokeanokoites
<400>4
serserglnleuvallyssergluleugluglulyslyssergluleu
151015
arghislysleulystyrvalprohisglutyrilegluleuileglu
202530
ilealaargasnserthrglnaspargileleuglumetlysvalmet
354045
gluphephemetlysvaltyrglytyrargglylyshisleuglygly
505560
serarglysproaspglyalailetyrthrvalglyserproileasp
65707580
tyrglyvalilevalaspthrlysalatyrserglyglytyrasnleu
859095
proileglyglnalaaspglumetglnargtyrvalglugluasngln
100105110
thrargasnlyshisileasnproasnglutrptrplysvaltyrpro
115120125
serservalthrgluphelyspheleuphevalserglyhisphelys
130135140
glyasntyrlysalaglnleuthrargleuasnhisilethrasncys
145150155160
asnglyalavalleuservalglugluleuleuileglyglyglumet
165170175
ilelysalaglythrleuthrleuglugluvalargarglyspheasn
180185190
asnglygluileasnphe
195
<210>5
<211>1423
<212>prt
<213>artificialsequence
<220>
<223>dcas9氨基酸序列
<400>5
metasptyrlysasphisaspglyasptyrlysasphisaspileasp
151015
tyrlysaspaspaspasplysmetalaprolyslyslysarglysval
202530
glyilehisglyvalproalaalaasplyslystyrserileglyleu
354045
alaileglythrasnservalglytrpalavalilethraspglutyr
505560
lysvalproserlyslysphelysvalleuglyasnthrasparghis
65707580
serilelyslysasnleuileglyalaleuleupheaspserglyglu
859095
thralaglualathrargleulysargthralaargargargtyrthr
100105110
argarglysasnargilecystyrleuglngluilepheserasnglu
115120125
metalalysvalaspaspserphephehisargleuglugluserphe
130135140
leuvalglugluasplyslyshisgluarghisproilepheglyasn
145150155160
ilevalaspgluvalalatyrhisglulystyrprothriletyrhis
165170175
leuarglyslysleuvalaspserthrasplysalaaspleuargleu
180185190
iletyrleualaleualahismetilelyspheargglyhispheleu
195200205
ilegluglyaspleuasnproaspasnseraspvalasplysleuphe
210215220
ileglnleuvalglnthrtyrasnglnleupheglugluasnproile
225230235240
asnalaserglyvalaspalalysalaileleuseralaargleuser
245250255
lysserargargleugluasnleuilealaglnleuproglyglulys
260265270
lysasnglyleupheglyasnleuilealaleuserleuglyleuthr
275280285
proasnphelysserasnpheaspleualagluaspalalysleugln
290295300
leuserlysaspthrtyraspaspaspleuaspasnleuleualagln
305310315320
ileglyaspglntyralaaspleupheleualaalalysasnleuser
325330335
aspalaileleuleuseraspileleuargvalasnthrgluilethr
340345350
lysalaproleuseralasermetilelysargtyraspgluhishis
355360365
glnaspleuthrleuleulysalaleuvalargglnglnleuproglu
370375380
lystyrlysgluilephepheaspglnserlysasnglytyralagly
385390395400
tyrileaspglyglyalaserglnglugluphetyrlyspheilelys
405410415
proileleuglulysmetaspglythrglugluleuleuvallysleu
420425430
asnarggluaspleuleuarglysglnargthrpheaspasnglyser
435440445
ileprohisglnilehisleuglygluleuhisalaileleuargarg
450455460
glngluaspphetyrpropheleulysaspasnargglulysileglu
465470475480
lysileleuthrpheargileprotyrtyrvalglyproleualaarg
485490495
glyasnserargphealatrpmetthrarglysserglugluthrile
500505510
thrprotrpasnpheglugluvalvalasplysglyalaseralagln
515520525
serpheilegluargmetthrasnpheasplysasnleuproasnglu
530535540
lysvalleuprolyshisserleuleutyrglutyrphethrvaltyr
545550555560
asngluleuthrlysvallystyrvalthrgluglymetarglyspro
565570575
alapheleuserglygluglnlyslysalailevalaspleuleuphe
580585590
lysthrasnarglysvalthrvallysglnleulysgluasptyrphe
595600605
lyslysileglucyspheaspservalgluileserglyvalgluasp
610615620
argpheasnalaserleuglythrtyrhisaspleuleulysileile
625630635640
lysasplysasppheleuaspasnglugluasngluaspileleuglu
645650655
aspilevalleuthrleuthrleuphegluaspargglumetileglu
660665670
gluargleulysthrtyralahisleupheaspasplysvalmetlys
675680685
glnleulysargargargtyrthrglytrpglyargleuserarglys
690695700
leuileasnglyileargasplysglnserglylysthrileleuasp
705710715720
pheleulysseraspglyphealaasnargasnphemetglnleuile
725730735
hisaspaspserleuthrphelysgluaspileglnlysalaglnval
740745750
serglyglnglyaspserleuhisgluhisilealaasnleualagly
755760765
serproalailelyslysglyileleuglnthrvallysvalvalasp
770775780
gluleuvallysvalmetglyarghislysprogluasnilevalile
785790795800
glumetalaarggluasnglnthrthrglnlysglyglnlysasnser
805810815
arggluargmetlysargileglugluglyilelysgluleuglyser
820825830
glnileleulysgluhisprovalgluasnthrglnleuglnasnglu
835840845
lysleutyrleutyrtyrleuglnasnglyargaspmettyrvalasp
850855860
glngluleuaspileasnargleuserasptyraspvalaspalaile
865870875880
valproglnserpheleulysaspaspserileaspasnlysvalleu
885890895
thrargserasplysasnargglylysseraspasnvalproserglu
900905910
gluvalvallyslysmetlysasntyrtrpargglnleuleuasnala
915920925
lysleuilethrglnarglyspheaspasnleuthrlysalagluarg
930935940
glyglyleusergluleuasplysalaglypheilelysargglnleu
945950955960
valgluthrargglnilethrlyshisvalalaglnileleuaspser
965970975
argmetasnthrlystyraspgluasnasplysleuilearggluval
980985990
lysvalilethrleulysserlysleuvalseraspphearglysasp
99510001005
pheglnphetyrlysvalarggluileasnasntyrhishisala
101010151020
hisaspalatyrleuasnalavalvalglythralaleuilelys
102510301035
lystyrprolysleuglusergluphevaltyrglyasptyrlys
104010451050
valtyraspvalarglysmetilealalyssergluglngluile
105510601065
glylysalathralalystyrphephetyrserasnilemetasn
107010751080
phephelysthrgluilethrleualaasnglygluilearglys
108510901095
argproleuilegluthrasnglygluthrglygluilevaltrp
110011051110
asplysglyargaspphealathrvalarglysvalleusermet
111511201125
proglnvalasnilevallyslysthrgluvalglnthrglygly
113011351140
pheserlysgluserileleuprolysargasnserasplysleu
114511501155
ilealaarglyslysasptrpaspprolyslystyrglyglyphe
116011651170
aspserprothrvalalatyrservalleuvalvalalalysval
117511801185
glulysglylysserlyslysleulysservallysgluleuleu
119011951200
glyilethrilemetgluargserserpheglulysasnproile
120512101215
asppheleuglualalysglytyrlysgluvallyslysaspleu
122012251230
ileilelysleuprolystyrserleuphegluleugluasngly
123512401245
arglysargmetleualaseralaglygluleuglnlysglyasn
125012551260
gluleualaleuproserlystyrvalasnpheleutyrleuala
126512701275
serhistyrglulysleulysglyserprogluaspasnglugln
128012851290
lysglnleuphevalgluglnhislyshistyrleuaspgluile
129513001305
ilegluglnileserglupheserlysargvalileleualaasp
131013151320
alaasnleuasplysvalleuseralatyrasnlyshisargasp
132513301335
lysproilearggluglnalagluasnileilehisleuphethr
134013451350
leuthrasnleuglyalaproalaalaphelystyrpheaspthr
135513601365
thrileasparglysargtyrthrserthrlysgluvalleuasp
137013751380
alathrleuilehisglnserilethrglyleutyrgluthrarg
138513901395
ileaspleuserglnleuglyglyasplysargproalaalathr
140014051410
lyslysalaglyglnalalyslyslyslys
14151420
<210>6
<211>328
<212>dna
<213>cauliflowermosaicvirus
<400>6
ccattgcccagctatctgtcactttattgtgaagatagtggaaaaggaaggtggctccta60
caaatgccatcattgcgataaaggaaaggccatcgttgaagatgcctctgccgacagtgg120
tcccaaagatggacccccacccacgaggagcatcgtggaaaaagaagacgttccaaccac180
gtcttcaaagcaagtggattgatgtgatatctccactgacgtaagggatgacgcacaatc240
ccactatccttcgcaagacccttcctctatataaggaagttcatttcatttggagaggac300
acgctgacaagctgactctagcagatct328
<210>7
<211>195
<212>dna
<213>cauliflowermosaicvirus
<400>7
ctgaaatcaccagtctctctctacaaatctatctctctctataataatgtgtgagtagtt60
cccagataagggaattagggttcttatagggtttcgctcatgtgttgagcatataagaaa120
cccttagtatgtatttgtatttgtaaaatacttctatcaataaaatttctaattcctaaa180
accaaaatccagtgg195
<210>8
<211>552
<212>dna
<213>streptomycesviridochromogenes
<400>8
atgagccctgaaagacggcctgtggagattagaccagcgacggcagcggacatggcggcg60
gtgtgcgacatcgtgaaccattacatcgaaacttcaacggtgaacttccgcacagagccc120
caaacaccacaggagtggatcgacgatctggagagacttcaagacagatacccgtggctt180
gttgcagaggtcgagggcgtggtcgcggggatcgcgtatgccggcccgtggaaggcgagg240
aacgcctacgattggacagtggaatccaccgtgtatgtcagccatcgccaccagaggctg300
ggcctcggcagcactctctacacccatctcctgaagagcatggaggcgcagggcttcaag360
tccgtggtcgcagtgattggcctgcctaacgatccatccgtgagactccatgaggccctc420
ggctacactgcgcgcggcactctgcgcgccgcgggctataagcacggcgggtggcatgac480
gtgggcttctggcagagagactttgaacttcccgctcccccaagacctgtcagacccgtt540
acgcagatctaa552
<210>9
<211>20
<212>dna
<213>oryzasativa
<400>9
ggaggcattggtgcttcttg20
<210>10
<211>20
<212>dna
<213>oryzasativa
<400>10
gcaacccaggcatcctcgac20
<210>11
<211>20
<212>dna
<213>pseudomonasaeruginosa
<400>11
gttcactgccgtataggcag20
<210>12
<211>55
<212>dna
<213>artificialsequence
<220>
<223>正向引物
<400>12
acatcaggtctccaaacggaggcattggtgcttcttggttttagagctagaaata55
<210>13
<211>62
<212>dna
<213>artificialsequence
<220>
<223>反向引物
<400>13
taggatggtctcgaaaacgtcgaggatgcctgggttgcctgcctatacggcagtgaacgc60
ac62
<210>14
<211>245
<212>dna
<213>oryzasativa
<400>14
ggatcatgaaccaacggcctggctgtatttggtggttgtgtagggagatggggagaagaa60
aagcccgattctcttcgctgtgatgggctggatgcatgcgggggagcgggaggcccaagt120
acgtgcacggtgagcggcccacagggcgagtgtgagcgcgagaggcgggaggaacagttt180
agtaccacattgcccagctaactcgaacgcgaccaacttataaacccgcgcgctgtcgct240
tgtgt245
<210>15
<211>76
<212>dna
<213>artificialsequence
<220>
<223>sgrna序列
<400>15
gttttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttgaaaaagt60
ggcaccgagtcggtgc76
<210>16
<211>3536
<212>dna
<213>artificialsequence
<220>
<223>含有靶点1序列和靶点2序列的gus基因
<400>16
atggtagatctgagggtaaatttctagtttttctccttcattttcttggttaggaccctt60
ttctctttttatttttttgagctttgatctttctttaaactgatctattttttaattgat120
tggttatggtgtaaatattacatagctttaactgataatctgattactttatttcgtgtg180
tctatgatgatgatgatagttacagaaccgacgaacttctctgtacccgatcaacaccga240
aacccgtggcgtcttcgacctcaatggcgtctggaacttcaagctggactacgggaaagg300
actggaagagaagtggtacgaaagcaagctgaccgacactattagtatggccgtcccaag360
cagttacaatgacattggcgtgaccaaggaaatccgcaaccatatcggatatgtctggta420
cgaacgtgagttcacggtgccggcctatctgaaggatcagcgtatcgtgctccgcttcgg480
ctctgcaactcacaaagcaattgtctatgtcaatggtgagctggtcgtggagcacaaggg540
cggattcctgccattcgaagcggaaatcaacaactcgctgcgtgatggcatgaatcgcgt600
caccgtcgccgtggacaacatcctcgacgatagcaccctcccggtggggctgtacagcga660
gcgccacgaagagggcctcggaaaagtcattcgtaacaagccgaacttcgacttcttcaa720
ctatgcaggcctgcaccgtccggtgaaaatctacacgaccccgtttacgtacgtcgagga780
catctcggttgtgaccgacttcaatggcccaaccgggactgtgacctatacggtggactt840
tcaaggcaaagccgaaaacctgaactgaactgaactgaaggttatgacattccaagcgga900
tggaagatcctgccggtgttagccgcggtgcatctggactcgtccctgtacgaggacccc960
cagcgcttcaatccctggagatggaaggtcagtcgcaataggattatcagtgtctcaagg1020
cgccattcagttccccgtgttccacaagaagcaccaatgcctccgcccatggtctgtccg1080
tgcaacccaggcatcctcgaccggagcatcaggagcaggaaaaggaggaggattgaacaa1140
tctacaggaagaggtctaaaaagctgcctgtgcggtggctggcttcctgcactgcatgca1200
ggtcgatctctgcgacgggcgacggcgcgcgtcgaggcgttggcggcatgcgcggtcatc1260
gctcacgcgtccgcggggatggtggcctgcggtgaccgcggagcttgtaaggataatgag1320
gtactggctggaaggcccaagagcgggcgaggtagaggtgttcgcgaacctgccgggctt1380
ccccgacaacgtgcgctccaacggcaggggccagttctgggtggcgatcgactgctgccg1440
gacgccggcgcaggaggtgttcgccaagaggccgtggctccggaccctatacttcaagtt1500
cccgctgtcgctcaaggtgctcacttggaaggccgccaggaggatgcacacggtgctcgc1560
gctcctcgacggcgaagggcgcgtcgtggaggtgctcgaggaccggggccacgaggtgat1620
gaagctggtgagtgaggtgcgggaggtgggcagcaagctgtggatcggaaccgtggcgca1680
caaccacatcgccaccatcccctaccctttagaggactaattttacccgtggcgtcttcg1740
acctcaatggcgtctggaacttcaagctggactacgggaaaggactggaagagaagtggt1800
acgaaagcaagctgaccgacactattagtatggccgtcccaagcagttacaatgacattg1860
gcgtgaccaaggaaatccgcaaccatatcggatatgtctggtacgaacgtgagttcacgg1920
tgccggcctatctgaaggatcagcgtatcgtgctccgcttcggctctgcaactcacaaag1980
caattgtctatgtcaatggtgagctggtcgtggagcacaagggcggattcctgccattcg2040
aagcggaaatcaacaactcgctgcgtgatggcatgaatcgcgtcaccgtcgccgtggaca2100
acatcctcgacgatagcaccctcccggtggggctgtacagcgagcgccacgaagagggcc2160
tcggaaaagtcattcgtaacaagccgaacttcgacttcttcaactatgcaggcctgcacc2220
gtccggtgaaaatctacacgaccccgtttacgtacgtcgaggacatctcggttgtgaccg2280
acttcaatggcccaaccgggactgtgacctatacggtggactttcaaggcaaagccgaaa2340
ccgtgaaagtgtcggtcgtggatgaggaaggcaaagtggtcgcaagcaccgagggcctga2400
gcggtaacgtggagattccgaatgtcatcctctgggaaccactgaacacgtatctctacc2460
agatcaaagtggaactggtgaacgacggactgaccatcgatgtctatgaagagccgttcg2520
gcgtgcggaccgtggaagtcaacgacggcaagttcctcatcaacaacaaaccgttctact2580
tcaagggctttggcaaacatgaggacactcctatcaacggccgtggctttaacgaagcga2640
gcaatgtgatggatttcaatatcctcaaatggatcggtgccaacagcttccggaccgcac2700
actatccgtactctgaagagttgatgcgtcttgcggatcgcgagggtctggtcgtgatcg2760
acgagactccggcagttggcgtgcacctcaacttcatggccaccacgggactcggcgaag2820
gcagcgagcgcgtcagtacctgggagaagattcggacgtttgagcaccatcaagacgttc2880
tccgtgaactggtgtctcgtgacaagaaccatccaagcgtcgtgatgtggagcatcgcca2940
acgaggcggcgactgaggaagagggcgcgtacgagtacttcaagccgttggtggagctga3000
ccaaggaactcgacccacagaagcgtccggtcacgatcgtgctgtttgtgatggctaccc3060
cggagacggacaaagtcgccgaactgattgacgtcatcgcgctcaatcgctataacggat3120
ggtacttcgatggcggtgatctcgaagcggccaaagtccatctccgccaggaatttcacg3180
cgtggaacaagcgttgcccaggaaagccgatcatgatcactgagtacggcgcagacaccg3240
ttgcgggctttcacgacattgatccagtgatgttcaccgaggaatatcaagtcgagtact3300
accaggcgaaccacgtcgtgttcgatgagtttgagaacttcgtgggtgagcaagcgtgga3360
acttcgcggacttcgcgacctctcagggcgtgatgcgcgtccaaggaaacaagaagggcg3420
tgttcactcgtgaccgcaagccgaagctcgccgcgcacgtctttcgcgagcgctggacca3480
acattccagatttcggctacaagaacgctagccatcaccatcaccatcacgtgtga3536
<210>17
<211>253
<212>dna
<213>agrobacteriumtumefaciens
<400>17
gatcgttcaaacatttggcaataaagtttcttaagattgaatcctgttgccggtcttgcg60
atgattatcatataatttctgttgaattacgttaagcatgtaataattaacatgtaatgc120
atgacgttatttatgagatgggtttttatgattagagtcccgcaattatacatttaatac180
gcgatagaaaacaaaatatagcgcgcaaactaggataaattatcgcgcgcggtgtcatct240
atgttactagatc253
<210>18
<211>1992
<212>dna
<213>zeamays
<400>18
ctgcagtgcagcgtgacccggtcgtgcccctctctagagataatgagcattgcatgtcta60
agttataaaaaattaccacatattttttttgtcacacttgtttgaagtgcagtttatcta120
tctttatacatatatttaaactttactctacgaataatataatctatagtactacaataa180
tatcagtgttttagagaatcatataaatgaacagttagacatggtctaaaggacaattga240
gtattttgacaacaggactctacagttttatctttttagtgtgcatgtgttctccttttt300
ttttgcaaatagcttcacctatataatacttcatccattttattagtacatccatttagg360
gtttagggttaatggtttttatagactaatttttttagtacatctattttattctatttt420
agcctctaaattaagaaaactaaaactctattttagtttttttatttaataatttagata480
taaaatagaataaaataaagtgactaaaaattaaacaaataccctttaagaaattaaaaa540
aactaaggaaacatttttcttgtttcgagtagataatgccagcctgttaaacgccgtcga600
cgagtctaacggacaccaaccagcgaaccagcagcgtcgcgtcgggccaagcgaagcaga660
cggcacggcatctctgtcgctgcctctggacccctctcgagagttccgctccaccgttgg720
acttgctccgctgtcggcatccagaaattgcgtggcggagcggcagacgtgagccggcac780
ggcaggcggcctcctcctcctctcacggcacggcagctacgggggattcctttcccaccg840
ctccttcgctttcccttcctcgcccgccgtaataaatagacaccccctccacaccctctt900
tccccaacctcgtgttgttcggagcgcacacacacacaaccagatctcccccaaatccac960
ccgtcggcacctccgcttcaaggtacgccgctcgtcctccccccccccccctctctacct1020
tctctagatcggcgttccggtccatggttagggcccggtagttctacttctgttcatgtt1080
tgtgttagatccgtgtttgtgttagatccgtgctgctagcgttcgtacacggatgcgacc1140
tgtacgtcagacacgttctgattgctaacttgccagtgtttctctttggggaatcctggg1200
atggctctagccgttccgcagacgggatcgatttcatgattttttttgtttcgttgcata1260
gggtttggtttgcccttttcctttatttcaatatatgccgtgcacttgtttgtcgggtca1320
tcttttcatgcttttttttgtcttggttgtgatgatgtggtctggttgggcggtcgttct1380
agatcggagtagaattctgtttcaaactacctggtggatttattaattttggatctgtat1440
gtgtgtgccatacatattcatagttacgaattgaagatgatggatggaaatatcgatcta1500
ggataggtatacatgttgatgcgggttttactgatgcatatacagagatgctttttgttc1560
gcttggttgtgatgatgtggtgtggttgggcggtcgttcattcgttctagatcggagtag1620
aatactgtttcaaactacctggtgtatttattaattttggaactgtatgtgtgtgtcata1680
catcttcatagttacgagtttaagatggatggaaatatcgatctaggataggtatacatg1740
ttgatgtgggttttactgatgcatatacatgatggcatatgcagcatctattcatatgct1800
ctaaccttgagtacctatctattataataaacaagtatgttttataattattttgatctt1860
gatatacttggatgatggcatatgcagcagctatatgtggatttttttagccctgccttc1920
atacgctatttatttgcttggtactgtttcttttgtcgatgctcaccctgttgtttggtg1980
ttacttctgcag1992
<210>19
<211>1176
<212>dna
<213>escherichiacoli
<400>19
atgcaaaaactcattaactcagtgcaaaactatgcctggggcagcaaaacggcgttgact60
gaactttatggtatggaaaatccgtccagccagccgatggccgagctgtggatgggcgca120
catccgaaaagcagttcacgagtgcagaatgccgccggagatatcgtttcactgcgtgat180
gtgattgagagtgataaatcgactctgctcggagaggccgttgccaaacgctttggcgaa240
ctgcctttcctgttcaaagtattatgcgcagcacagccactctccattcaggttcatcca300
aacaaacacaattctgaaatcggttttgccaaagaaaatgccgcaggtatcccgatggat360
gccgccgagcgtaactataaagatcctaaccacaagccggagctggtttttgcgctgacg420
cctttccttgcgatgaacgcgtttcgtgaattttccgagattgtctccctactccagccg480
gtcgcaggtgcacatccggcgattgctcactttttacaacagcctgatgccgaacgttta540
agcgaactgttcgccagcctgttgaatatgcagggtgaagaaaaatcccgcgcgctggcg600
attttaaaatcggccctcgatagccagcagggtgaaccgtggcaaacgattcgtttaatt660
tctgaattttacccggaagacagcggtctgttctccccgctattgctgaatgtggtgaaa720
ttgaaccctggcgaagcgatgttcctgttcgctgaaacaccgcacgcttacctgcaaggc780
gtggcgctggaagtgatggcaaactccgataacgtgctgcgtgcgggtctgacgcctaaa840
tacattgatattccggaactggttgccaatgtgaaattcgaagccaaaccggctaaccag900
ttgttgacccagccggtgaaacaaggtgcagaactggacttcccgattccagtggatgat960
tttgccttctcgctgcatgaccttagtgataaagaaaccaccattagccagcagagtgcc1020
gccattttgttctgcgtcgaaggcgatgcaacgttgtggaaaggttctcagcagttacag1080
cttaaaccgggtgaatcagcgtttattgccgccaacgaatcaccggtgactgtcaaaggc1140
cacggccgtttagcgcgtgtttacaacaagctgtaa1176
- 还没有人留言评论。精彩留言会获得点赞!