一套同时检测93种牛遗传缺陷基因和致死单倍型的引物组合及试剂盒的制作方法
本发明涉及畜牧业
技术领域:
,涉及一种用于确定牛是否携带93种牛遗传缺陷基因和致死单倍型的检测方法,具体而言,即利用dna提取、pcr多重扩增技术、单碱基延伸技术和飞行质谱技术,对93种遗传缺陷基因和致死单倍型同时进行基因分型,确定该牛是否携带隐性遗传缺陷基因和致死单倍型,涉及牛群隐性遗传缺陷基因和致死单倍型的剔除和个体标注、牛群的选种选配、β-酪蛋白特定型牛奶的奶牛等的筛选方法。
背景技术:
:迄今,牛已知的遗传缺陷有506种,属于孟德尔遗传的有233种,其中,已知因果突变的有133种(http://omia.angis.org.au/home/),大多数突变是有害的。一些导致遗传缺陷的单基因的(孟德尔的)突变已经被揭示。由于携带者很容易被鉴定并从育种群排除,显性突变很容易从群体中清除。而针对隐性突变的管理则更加困难,一小部分携带者必然会保存在群体中。根据表型选择(自然选择或人工选择)清除纯合子(缺陷表型)的情况下,携带者的频率将趋近于零,出现遗传缺陷的概率极低。当顶级公牛是遗传缺陷的携带者,缺陷突变就会在群体中保持更高的频率,相关的遗传缺陷就会成为一个严重的问题,对别是对专用的乳用牛品种群体就更加危险,因为他们有着特殊的群体结构,例如,高度近交、更少的奠基者、有效群体大小持续下降、人工授精的广泛使用,这些因素使得现代牛对遗传缺陷更加易感(pauschetal,2015)。对于隐性有害突变,通过鉴定携带者并从群体中剔除,或者应用有效的选种选配策略抵消有害突变的扩散,使其频率保持在较低的水平。遗传缺陷可严重影响牛的生产性能和经济价值,基因出现纯合时甚至导致早期死亡,引起重大的经济损失。有的遗传缺陷基因和致死单倍型的携带者就会影响牛生产性能,给养牛业造成重大的经济损失。例如,脊柱畸形综合征(hhc)影响荷斯坦奶牛的乳中的脂肪量,并趾(mulefoot)携带牛其体细胞评分更高,而且繁殖年限和基因组育种值更低。荷斯坦牛的单倍型0(hh0)携带者与脂肪和青母牛的受胎率有关,荷斯坦致死单倍型1(hh1)与产奶量、脂肪、蛋白、体细胞评分、单性状繁殖年限和繁殖力有关,荷斯坦牛致死单倍型2(hh2)与蛋白、繁殖年限、青年牛受胎率、母牛受胎率有关,荷斯坦牛致死单倍型4(hh4)与产奶、蛋白和繁殖力有关,荷斯坦单倍型5与蛋白量、女儿妊娠率有关。娟姗牛单倍型1(jh1)与表型效应无关,但脂肪、蛋白、繁殖年限、女儿妊娠率、成母牛受胎率和青年母牛受胎率的基因组育种值存在显著差异;娟姗牛单倍型2(jh2)仅与更低的成母牛受胎率有关,在美国奶牛群体中(约存栏900万头),每年因遗传缺陷导致的繁殖力下降和犊牛早期死亡产生的经济损失约为1100万美元,其中,每头牛导致的妊娠损失约为200美元,犊牛早起死亡的损失约为342美元(假设在第21天死亡),每延长1天,增加2美元的损失,另外,导致犊牛出生后或饲养早期死亡的遗传缺陷,其造成的经济损失尤其严重,因为其经济损失远远大于导致胚胎早期死亡的遗传缺陷。鉴定遗传缺陷基因的突变携带者,可在育种中制定专门的控制牛的近交和遗传缺陷的选配方案计划,指导选种和计划选配,以降低携带者在群体中的频率,避免携带者交配。我们还可以根据检测结果,筛选出不携带特定遗传缺陷基因的群体。对于像a2型β-酪蛋白基因(β-酪蛋白有a1,a2,a3,b,c,d,e,f,h1,h2,i,g等遗传变体型),还可以筛选出特定的突变体类型奶牛,组建单一基因或组合基因纯合的奶牛群体,进而培育新的品系、品种。而乳球蛋白aa型具有高的奶产量和乳清蛋白含量,而bb型具有高的酪蛋白和脂肪含量,其bb型更利于制备奶酪产品。除了育种之外,遗传缺陷的信息对于保种工作也有重要的价值,它可以避免携带者交配,特别是一些濒危品种,由于其近交的程度更大,出现遗传缺陷的风险也大(williamsetal,2016)。携带者特定突变或致死单倍型的鉴定传统是通过个体的实验室检测,例如,snp标记、微卫星标记、酪蛋白异构体、cvm的特异基因检测来完成,已知的遗传缺陷基因检测一般是针对特定的缺陷基因利用测序、pcr技术等方法来进行检测。这些方法一次一般只能检测单个的缺陷基因、检测的数量少、成本高,而且费事。而低密度的芯片可以一次性检测上百种的遗传缺陷基因型和单倍型型,一次检测的缺陷基因的种类多、成本更低,更加高效、方便和实用。中国授权发明专利,cn201010225605.2,应用as-pcr检测牛白细胞粘附缺陷的方法,该发明涉及应用as-pcr方法在体外检测牛白细胞粘附缺陷的方法,制备了一种针对牛粘附缺陷基因cd18的检测方法的特异性pcr引物用于在体外开展牛粘附缺陷的致病基因的筛选,该发明只能检测单一的遗传缺陷基因。中国发明申请专利cn201510569964.2,一种筛查荷斯坦奶牛白细胞粘附缺陷病携带者的分子检测方法,该发明提供了一种鉴定荷斯坦牛是否携带blad的竞争性等位基因特异性pcr成套引物,该发明利用kasp技术,通过设计一组针对blad突变位点的引物,进行反应,经过约1.5小时后通过配套的软件进行简单识别分析,即可准确获得所有待测样品的基因型,从而实现筛查blad携带者的目的,该发明只能检测单一的blad遗传缺陷基因。中国发明专利申请cn201610262293.x,一种用于检测奶牛四种遗传病的核酸质谱的检测产品及应用,该发明公开了一种可以同时检测奶牛是否具有脊椎畸形综合症(cvm)、白细胞粘附缺陷症(blad)、尿苷酸合酶缺乏症(dumps)、瓜氨酸血症(cn)四种遗传病的引物系统,基于该引物系统所制备的产品,只能实现同时对奶牛cvm、blad、dumps、cn4种遗传病基因相关的4处多态性位点进行检测。中国授权发明专利,cn200610150317.9,一种检测牛cvm有害基因的方法,该发明提供了一种检测牛脊椎畸形综合症(cvm)携带者的方法,该方法首先设计合成引物对牛的dna样品进行聚合酶链式反应(pcr),然后对pcr产物进行单链构象多态性(sscp)分析,根据聚丙烯酰胺凝胶电泳结果判断牛是否携带cvm有害基因,其不足之处是只能检测单一的cvm携带情况,且重复性较差。中国授权发明专利,cn201310617444.5,一种牛尿苷酸合酶缺乏症有害基因检测的引物组合物及其试剂盒和应用,该发明公开了一种牛尿苷酸合酶缺乏症有害基因检测的引物组合物及其试剂盒和应用,该发明的检测的方法为首先提取牛血液中的全组dna作为模板,进行巢式pcr(nestedpcr)扩增,所得pcr产物进行测序,根据测序结果可直接了解突变位点的碱基变化,其特点是,需要两次扩增、并对产物进行测序,易引入错误,且只能检测的单一缺陷基因。中国授权发明专利,cn200910272954.7,发明了一种应用as-pcr检测牛尿苷酸合酶缺乏症的方法,该发明属于动物疾病检测
技术领域:
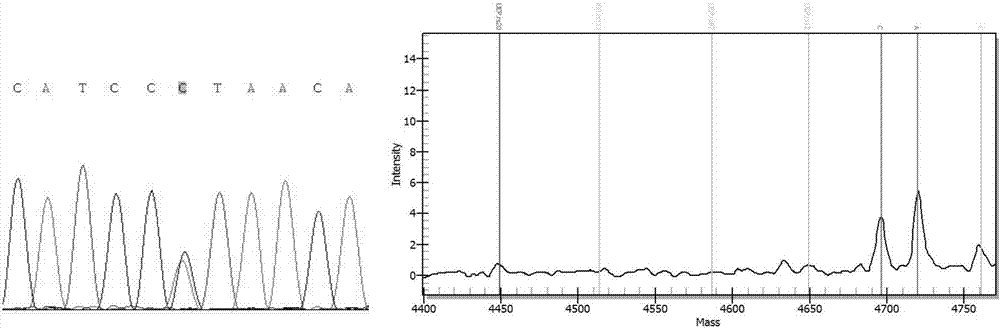
,具体涉及应用as-pcr检测牛尿苷酸合酶缺乏症的方法,制备了一种用于牛尿苷酸合酶缺乏症检测方法的特异性pcr引物,在扩增产物的中引入错配碱基,需要应用电泳进行基因分型,由于产物长度较短,易导致错判,且不适合高通量得、大量的牛群检测。中国授权发明专利cn201110440150.0,公开了一种筛查牛退化性轴突病携带者的方法及其试剂盒,该发明以待测牛的基因组dna为模板,设计上游引物和下游引物对其进行pcr扩增反应,然后回收pcr扩增片段,用fastdigest限制酶tspri进行酶切,在酶切反应体系酶切后进行凝胶检测,该筛查方法操作简单、快捷、费用低廉、但同样存在只能检测单一遗传缺陷基因的不足。目前国内外尚无有关检测其他遗传缺陷基因及致死单倍型方法研究,更没有能同时检测上百种已知牛遗传缺陷基因因果突变的高通量检测方法研究。技术实现要素:针对现有技术存在的问题,本发明人经过创造性的劳动整合了93种牛遗传缺陷病的因果突变位点(snp、短片段的插入或缺失)信息,得到了能够用于同时检测牛不同遗传缺陷病的引物组合及其试剂盒。所述引物组合及其试剂盒的特异性强、灵敏度高,能够快速、准确的检测93种牛遗传缺陷病,而且可以一次实验同时检测近百份奶牛样品,具有快速检测、通量高的特点。具体的,本发明涉及以下技术方案:首先,本发明公开了一套同时检测牛93种遗传缺陷101个相关因果突变位点的引物,所述引物分为四组,具体如表1-表4所示。本发明将检测93种遗传缺陷基因和致死单倍型相关101个因果突变位点所需的引物组合分成4个检测孔中,同时对不同遗传缺陷病进行检测。每个孔(1,2,3,4)所述引物的组合序列如下:1孔引物组合:用于同时检测牛的pirm综合征、球形红细胞症、β酪蛋白8、β酪蛋白4、ⅰ型白细胞粘附缺陷征、契-东综合征、隐性红毛、β酪蛋白9、双肌、β酪蛋白1、先天性鱼鳞病、致死单倍型(hh1)、致死单倍型(hh3)、致死单倍型(hh4)、软骨发育异常侏儒症、脊柱畸形综合征、软骨成长不全、瓜氨酸血症、安格斯牛侏儒症、肠病性肢端皮炎、流产slc37a2、生长激素缺乏型侏儒症、心肌病及绵状长毛病、伴有炎性病变合乎比例的侏儒症遗传缺陷基因的引物组合。其序列及相关信息如下表1所示:表12孔引物组合:用于同时检测牛的多种眼部缺陷、弯曲尾综合征、α型甘露糖苷贮积症、流产(jh1)、退行性轴突病、雄性不育、β乳球蛋白异常低表达、致死单倍型(hh5)、β酪蛋白11、无汗性外胚层发育不良、黄脂病、乳球蛋白、流产shbg、围产期弱犊牛综合征、先天性肌张力障碍i型、锌缺乏样综合征、并趾、蛋白多糖型侏儒症、致死性多器官发育不良、显性白与双侧耳聋、三甲基胺尿症、毛少色浅、蜘蛛腿综合征、先天性肌无力、隐性红毛373、β酪蛋白6遗传缺陷基因的引物组合。其序列及相关信息如下表2所示:表23孔引物组合:用于同时检测牛的大疱性表皮松解症、β型甘露糖苷贮积症、脊髓髓鞘形成障碍、血小板紊乱、粘多糖贮积症ⅲb型、无角/有角、隐性红毛ebr、多肢畸形、马凡综合征、隐性红毛358、原卟啉症、β酪蛋白12、甲状腺肿、α型甘露糖苷贮积症、槭糖尿病-h、蛋白多糖型侏儒症、先天性假性肌强直、二类黄嘌呤尿、显性红毛、前肢至腰部肌肉异常、营养不良型大疱性表皮松解症、五型肝醣储积症、脊髓性肌萎缩遗传缺陷基因的引物组合。其序列及相关信息如下表3所示:表34孔引物组合:用于同时检测牛的β酪蛋白3、隐性红毛、先天性假性肌强直、埃莱尔-当洛综合征、浅色少毛症、二型肝醣储积症(庞贝氏症)、a型血友病、β酪蛋白7、牙龈错构瘤型骨硬化病、kappa酪蛋白、扩张型心肌病、二型肝醣储积症(庞贝氏症)、先天性肌张力障碍ⅱ型、β酪蛋白2、少毛症、尿苷磷酸合酶缺乏症、f11因子缺乏、β酪蛋白5、皮肤脆裂症遗传缺陷基因引物组合。其序列及相关信息如下表4所示:表4本发明的设计思路为:在尽可能全面覆盖牛遗传缺陷基因和致死单倍型基因已知因果突变的前提下,设计一种高效、可应用且成本尽可能低的牛遗传缺陷基因因果突变的高通量检测方法。本发明基于牛不同遗传缺陷病对应的因果突变位点序列信息,经过独特设计和组合优化,获得pcr扩增反应引物和单碱基扩展引物,然后在384孔板上进行pcr反应扩增出待检样本突变位点的目的片段,再用虾碱性磷酸酶对pcr产物进行纯化反应,最后用单碱基扩展引物对pcr纯化的产物在384孔板上进行单碱基延伸反应并进行质谱检测,从而获得突变位点的基因型。本发明高通量检测引物和分组的设计主要包括三个方面的控制点。(1)针对牛遗传缺陷基因和致死单倍型的选择,发明人前期对已知因果突变的牛遗传缺陷基因和致死单倍型进行分析,发现针对骨硬化病、短脊椎综合症(hh0)、性腺发育不良、肾发育不良、流产、罗马诺拉肉牛白内障等牛的遗传缺陷病的因果突变位点信息,在评估设计引物设计过程中发现这几种牛遗传缺陷病的因果突变的插入或缺失,导致无法设计引物进行分型鉴定,预实验该6种遗传缺陷的检测引物设计与本发明93种牛遗传缺陷病引物设计整合性极差,经分析该6种遗传缺陷或为大片段的插入或缺失,或检测引物设计兼容性太差(正义链和反义链碱基设计无法满足要求),相较而言,肌肉肥大(双肌)、心肌病及绵状长毛病、无汗性外胚层发育不良、毛少色浅、蜘蛛腿综合征、先天性肌无力、隐性红毛373、二类黄嘌呤尿、二型肝醣储积症、庞贝氏症、f11因子缺乏、皮肤脆裂症等几种片段的插入或缺失遗传缺陷基因的引物设计可以有效的与本发明多snp位点多重引物设计整合和兼容。本发明实现了多插入或缺失突变检测结合多snp检测的引物设计。(2)对于本发明93种牛遗传缺陷病,总共涉及到101个因果突变位点,主要面临的是极限的整合难度。通常多重pcr结合多重单碱基扩增组合中位点数目越少其相互感染的可能性越低,本发明位点数量较多,因此从101个位点中筛选组合和优化,经初步筛选,每组约25重上下较优,进一步结合多重单碱基扩展引物的考察,初步确定了4孔检测引物的设计目标,并针对缺陷基因进行分组和试错分析;因整合难度大,发明人前期整理其相关位点信息后,发给北京某生物公司利用软件进行引物评估和设计,反馈分析发现,该生物公司能设计出检测87个突变位点的引物组合(另14个位点无法进行整合,包括并趾、三甲基胺尿症、隐性红毛373、粘多糖贮积症ⅲb型、先天性鱼鳞病、锌缺乏样综合征、营养不良型大疱性表皮松解症、牙龈错构瘤型骨硬化病、β酪蛋白3、β酪蛋白11、β酪蛋白12、β型甘露糖苷贮积症、无汗性外胚层发育不良、a型血友病),而且需要分成5个孔进行检测;在此基础上,发明人进行独特的策略进行的分组和整合(具体分组情况见表1-4),重新设计出本发明所述覆盖101个因果突变位点的4孔检测引物。(3)因为涵盖位点多、检测难度大,本发明着重对引物的特异性和检测准确性进行了考察,本发明对多重pcr引物采用assaydesigner3.1软件设计,通过对合成的引物使用质谱检测分子量以保证扩增准确且无杂质(由于位点太多,本发明对有杂质的引物对进行了剔除),本发明对软件设计的引物进行检测后,其中试错分析,大部分引物对复合理论分子量,12对存在杂质,2对分子量错误,发明人对其进行再设计和检测,获得多重pcr引物。在此基础上,发明人进一步对单碱基延伸引物进行设计和优化,并反馈调整多重pcr引物对,最终形成了本发明所述引物组(表1-表4)。其次,本发明公开了包含上述引物组的检测试剂盒。优选的实施方案中,所述试剂盒还包括pcr扩增反应试剂、碱性磷酸酶处理试剂、单碱基延伸反应。具体的,pcr扩增反应试剂包括dntp(25mmeach)、mgcl2(25mm)、hotstartaq(5u/μl)、pcrbμffer、ddh2o等;碱性磷酸酶处理试剂包括ddh2o、虾碱性磷酸酶buffer、虾碱性磷酸酶(1u/μl);单碱基延伸反应包括:iplexbμfferplus、iplexterminationmix、iplexenzyme、ddh2o。此外,本发明公开了一种同时检测93种牛遗传缺陷基因的方法,包括如下步骤:(1)提取牛dna;优选的实施方式中从牛血液、精液或毛囊中提取dna;(2)多重pcr反应:利用利用上述引物和步骤(1)所述dna进行pcr扩增反应;具体的,利用稀释好的引物和dna在384孔中进行pcr扩增反应,pcr扩增反应采用5.000μl体系,组成为:1μldna(20ng/μl)、1.000μl扩增引物混合物(500nmeach)、0.100μldntp(25mmeach)、0.325μlmgcl2(25mm)、0.100μlhotstartaq(5u/μl)、0.625μlpcrbμfferwith15mmmgcl2、1.850μlddh2o;优选的实施方案中,pcr扩增反应程序为:先94℃5min;接着94℃20sec,56℃30sec,72℃1min,此步45个循环;最后72℃3min;(3)产物碱性磷酸酶处理,去除剩余的dntps,具体的,上步中每个孔的5.000μlpcr产物各加2.00μl虾碱性磷酸酶处理,去除剩余的dntps,优选的,其中,2.00μl虾碱性磷酸酶处理反应液组成为:1.53μlddh2o、0.17μl虾碱性磷酸酶buffer、0.30μl虾碱性磷酸酶(1u/μl);其中,7μl产物碱性磷酸酶处理液反应程序为:37℃20min;85℃5min;4℃∞;(4)单碱基延伸反应,利用上述引物进行单碱基延伸反应,具体如下:虾碱性磷酸酶处理后的产物,进行单碱基延伸反应,优化的反应体系和程序,如下:其中,单碱基延伸反应采用9μl反应体系,组成为:7μl产物碱性磷酸酶处理产物、0.200μliplexbμfferplus、0.200μliplexterminationmix、0.940μl延伸引物混合物(7μm:14μm)、0.041μliplexenzyme、0.619μlddh2o;其中,单碱基延伸反应的程序为:先94℃30sec;接着94℃5sec,(52℃5sec,80℃5sec,5个循环),此步40个循环;72℃3min;(5)单碱基延伸反应后进行树脂纯化反应,具体如下:在384dimple板中均匀填充树脂并放置10min,在384样本板的每个孔中加16μl水,轻敲dimple使树脂落入样本板的每个孔中,然后将样本版室温旋转30min。(6)利用质谱检测及数据输出,具体操作如下:用点样仪将树脂纯化后的延伸产物移至384孔spectrochip芯片上,然后使用maldi-tof质谱仪对其分析,结果利用typer4.0软件进行数据处理,根据野生和突变位点对应的不同延伸产物分子量的大小,获得突变位点分型信息。本发明还公共开了一种牛遗传育种的方法,利用上述方法筛选去除遗传缺陷基因纯合和/携带者,配套选种选配技术,优化牛育种。本发明取得了以下有益效果:(1)本发明首次针对牛多种遗传缺陷基因及致死单倍型方法研究,首次提出了一种同时检测近百种已知牛遗传缺陷基因因果突变的高通量检测方法。(2)本发明的检测方法可同时检测93种牛遗传缺陷病101个因果突变位点,且4孔检测即可覆盖101个因果突变位点,本发明一次性检测样品通量高、费用低,而且开发出检测试剂盒,使操作更加简便、快捷。(3)本发明以经过直接测序鉴定的遗传缺陷病携带者的牛为检测对象,都能够准确地检出牛遗传缺陷病基因型(图1-图2),表明本发明的引物、反应体系、条件及检测步骤等准确、有效、重复性高、稳定性强,这一发明可以对牛遗传缺陷病进行早期(出生)检测,或者对双亲后代的遗传缺陷情况进行预测,消除有害基因型给养牛业带来的经济损失,能够产生显著的经济和社会效益。附图说明图1:脊柱畸形综合征(hhc)直接测序的遗传缺陷携带者的牛和本发明方法的鉴定结果,左图为测序结果,右图为本发明分析结果;图2:β酪蛋白a1/a2杂合型直接测序的遗传缺陷携带者的牛和本发明方法的鉴定结果,左图为测序结果,右图为本发明分析结果;图3:牛致死单倍型(hh1)携带者的检测结果图4:牛隐性红毛373携带者的检测结果图5:牛致死单倍型(hh5)携带者的检测结果图6:牛黄脂病携带者的检测结果图7:牛隐性红毛ebr携带者的检测结果图8:牛隐性红毛358携带者和纯合突变的检测结果,上图为携带者,下图为纯合突变图9:牛槭糖尿病纯合突变的检测结果图10:牛乳球蛋白不同型的检测结果,上图为aa型,中图为ab型,下图为bb型具体实施方式下面结合具体实施例对本发明作进一步的说明。实施例1:同时检测93种牛遗传缺陷基因的方法构建1.pcr扩增反应引物设计与条件优化整理snp序列信息成标准格式,进行93个因果突变位点(snp、短片段的插入或缺失)pcr扩增反应引物和单碱基延伸引物的设计,对影响检测反应体系条件和pcr程序条件进行优化,包括试剂浓度、温度、时间、反应循环数等,再进行384孔的pcr反应(93个因果突变位点分成4个pcr孔中同时检测)。pcr扩增反应具体如下:(1)采用assaydesigner3.1软件进行每个突变位点序列的引物设计,分析优化试验后的引物,分别用去离子水混合每个孔中检测突变位点所需的pcr引物,使得最终每个孔中的引物混合物浓度为0.5μm。(2)从牛血液、精液或毛囊中提取dna,用仪器测定其od值和1.25%琼脂糖凝胶电泳检测合格后,转移至96孔板,-20℃储存备用。(3)利用稀释好的引物和样品dna在384孔中进行pcr扩增反应,如下:pcr扩增反应采用5.000μl体系,组成为:1μldna(20ng/μl)、1.000μl扩增引物混合物(500nmeach)、0.100μldntp(25mmeach)、0.325μlmgcl2(25mm)、0.100μlhotstartaq(5u/μl)、0.625μlpcrbμfferwith15mmmgcl2、1.850μlddh2o。pcr扩增反应程序为:先94℃5min;接着94℃20sec,56℃30sec,72℃1min,此步45个循环;最后72℃3min。2.产物碱性磷酸酶处理,如下:上步中每个孔的5.000μlpcr产物各加2.00μl虾碱性磷酸酶处理,去除剩余的dntps,具体如下:其中,2.00μl虾碱性磷酸酶处理反应液组成为:1.53μlddh2o、0.17μl虾碱性磷酸酶buffer、0.30μl虾碱性磷酸酶(1u/μl)。其中,7μl产物碱性磷酸酶处理液反应程序为:37℃20min;85℃5min;4℃∞。3.单碱基延伸反应,如下:虾碱性磷酸酶处理后的产物,进行单碱基延伸反应,优化的反应体系和程序,如下:其中,单碱基延伸反应采用9μl反应体系,组成为:7μl产物碱性磷酸酶处理产物、0.200μliplexbμfferplus、0.200μliplexterminationmix、0.940μl延伸引物混合物(7μm:14μm)、0.041μliplexenzyme、0.619μlddh2o。其中,单碱基延伸反应的程序为:先94℃30sec;接着94℃5sec,(52℃5sec,80℃5sec,5个循环),此步40个循环;72℃3min。4.树脂纯化反应,如下:(1)在384dimple板中均匀填充树脂并放置10min。(2)在384样本板的每个孔中加16μl水。(3)轻敲dimple使树脂落入样本板的每个孔中,然后将样本版室温旋转30min。5.质谱检测及数据输出,如下:用点样仪将树脂纯化后的延伸产物移至384孔spectrochip芯片上,然后使用maldi-tof质谱仪对其分析,结果利用typer4.0软件进行数据处理,根据野生和突变位点对应的不同延伸产物分子量的大小,获得突变位点分型信息。实施例2:用于检测93种牛遗传缺陷基因的试剂盒试剂盒的组成包括:1-4孔扩增引物混合物(500nmeach)、1-4孔延伸引物混合物(7μm:14μm)、dntp(25mmeach)、mgcl2(25mm)、0.100μlhotstartaq(5u/μl)、pcrbμfferwith15mmmgcl2、虾碱性磷酸酶buffer、虾碱性磷酸酶(1u/μl)、iplexbμfferplus、iplexterminationmix、iplexenzyme、ddh2o、384dimple板、384样本板、树脂。其中,试剂盒所述的扩增引物混合物和延伸引物混合物,其序列如上述
发明内容部分所述。采用该试剂盒进行牛的组织样本的检测步骤为:(1)利用试剂盒中的试剂对质控合格的牛血液、精液或毛囊中的dna进行pcr扩增反应,每个样本的93种牛遗传缺陷因果突变位点分成4个孔,在384孔中进行检测,反应孔的条件具体如下:pcr扩增反应采用5.000μl体系,组成为:1μldna(20ng/μl)、1.000μl扩增引物混合物(500nmeach)、0.100μldntp(25mmeach)、0.325μlmgcl2(25mm)、0.100μlhotstartaq(5u/μl)、0.625μlpcrbμfferwith15mmmgcl2、1.850μlddh2o。pcr扩增反应程序为:先94℃5min;接着94℃20sec,56℃30sec,72℃1min,此步45个循环;最后72℃3min。(2)每个孔的5.000μlpcr产物各加2.00μl虾碱性磷酸酶反应液处理(组成为:1.53μlddh2o、0.17μl虾碱性磷酸酶buffer、0.30μl虾碱性磷酸酶(1u/μl)),去除剩余的dntps,7μl产物碱性磷酸酶处理液反应程序为:37℃20min;85℃5min;4℃∞。(3)虾碱性磷酸酶处理后,进行单碱基延伸反应,如下:单碱基延伸反应9μl反应体系,组成为:7μl产物碱性磷酸酶处理液、0.200μliplexbμfferplus、0.200μliplexterminationmix、0.940μl延伸引物混合物(7μm:14μm)、0.041μliplexenzyme、0.619μlddh2o。单碱基延伸反应的程序为:先94℃30sec;接着94℃5sec,(52℃5sec,80℃5sec,5个循环),此步40个循环;72℃3min。(4)在384dimple板中均匀填充树脂并放置10min;在384样本板的每个孔中加16μl水;轻敲dimple使树脂落入样本板的每个孔中,然后将样本版室温旋转30min。(5)用点样仪将树脂纯化后的延伸产物移至384孔spectrochip芯片上,然后使用maldi-tof质谱仪对其分析,结果利用typer4.0软件进行数据处理,根据野生和突变位点对应的不同延伸产物分子量的大小,获得位点分型信息。实施例3:牛不同遗传缺陷基因和特色奶基因的样本检测采集96头荷斯坦奶牛的血液样本,利用本发明实施例2的试剂盒中的试剂及其相关步骤,检测奶牛样本的遗传缺陷病的因果突变位点(snp、短片段的插入或缺失)。结果发现部分牛携带(杂合)遗传缺陷基因和致死单倍型(图3-图10),并经测序验证。具体样本的基因型检测结果如下表5所示:表5.牛遗传缺陷基因和致死单倍型(表型性状)检测结果遗传缺陷(表型性状)样本数(基因型)致死单倍型(hh1)3头携带致死单倍型(hh3)7头携带脊柱畸形综合征(hhc)4头携带隐性红毛3732头携带致死单倍型(hh5)4头携带黄脂病3头携带隐性红毛ebr4头携带隐性红毛35812头携带、1头纯合突变槭糖尿病1头纯合突变浅色少毛症8头携带、1头纯合突变乳球蛋白23头aa、25头bb、48头携带β酪蛋白35头a2、11头a1、49头携带sequencelisting<110>山东省农业科学院奶牛研究中心<120>一套同时检测93种牛遗传缺陷基因和致死单倍型的引物组合及试剂盒<130><160>303<170>patentinversion3.5<210>1<211>30<212>dna<213>人工序列<400>1acgttggatgactggaaatagaaaccccgc30<210>2<211>30<212>dna<213>人工序列<400>2acgttggatgacgtcacccacatcaccatc30<210>3<211>15<212>dna<213>人工序列<400>3cgacaagctcctcac15<210>4<211>30<212>dna<213>人工序列<400>4acgttggatgaggcctctctgtatctaacc30<210>5<211>30<212>dna<213>人工序列<400>5acgttggatggcaaacatcatccagatggg30<210>6<211>15<212>dna<213>人工序列<400>6tctaaccccacggag15<210>7<211>30<212>dna<213>人工序列<400>7acgttggatgaaaatctgcaccttcctctg30<210>8<211>30<212>dna<213>人工序列<400>8acgttggatggactgaggaggaaacatgac30<210>9<211>17<212>dna<213>人工序列<400>9cactcaccagcctcttc17<210>10<211>30<212>dna<213>人工序列<400>10acgttggatgtccctcctcttactcaaacc30<210>11<211>30<212>dna<213>人工序列<400>11acgttggatgccatagcctccttcactttg30<210>12<211>17<212>dna<213>人工序列<400>12ttgtggtgccgcctttc17<210>13<211>30<212>dna<213>人工序列<400>13acgttggatgttgacgttgacgaggtcatc30<210>14<211>30<212>dna<213>人工序列<400>14acgttggatgttcaatgtgaccttccggag30<210>15<211>17<212>dna<213>人工序列<400>15ccatcaggtagtacagg17<210>16<211>30<212>dna<213>人工序列<400>16acgttggatggctgctgtgtacagagtatg30<210>17<211>30<212>dna<213>人工序列<400>17acgttggatgctcaagggtaatctgcactg30<210>18<211>17<212>dna<213>人工序列<400>18gagtatgtgtgtgtggc17<210>19<211>30<212>dna<213>人工序列<400>19acgttggatgatgtcatcgacgtgctcatc30<210>20<211>30<212>dna<213>人工序列<400>20acgttggatgagaagatggagatgtagcgg30<210>21<211>18<212>dna<213>人工序列<400>21gcttcctgggtgccattg18<210>22<211>30<212>dna<213>人工序列<400>22acgttggatgagaggagggatgttttgtgg30<210>23<211>30<212>dna<213>人工序列<400>23acgttggatgtaaaatccacccctttgccc30<210>24<211>18<212>dna<213>人工序列<400>24ttggtgggaggctgttag18<210>25<211>30<212>dna<213>人工序列<400>25acgttggatgagttagagggtaacgacagc30<210>26<211>30<212>dna<213>人工序列<400>26acgttggatgagtcaaggtaacagacacac30<210>27<211>19<212>dna<213>人工序列<400>27cacgacagcatcgagattc19<210>28<211>30<212>dna<213>人工序列<400>28acgttggatgcaaattacctctgtttgctg30<210>29<211>30<212>dna<213>人工序列<400>29acgttggatgataaacagcatttctttatg30<210>30<211>20<212>dna<213>人工序列<400>30ttcggtttgctgctgttcct20<210>31<211>30<212>dna<213>人工序列<400>31acgttggatgacatgttaactcgcaggagg30<210>32<211>30<212>dna<213>人工序列<400>32acgttggatgacaggtatggtatgatccag30<210>33<211>20<212>dna<213>人工序列<400>33tgatgtaagctggaagggag20<210>34<211>30<212>dna<213>人工序列<400>34acgttggatgtgaaggtgtcctctatgctg30<210>35<211>30<212>dna<213>人工序列<400>35acgttggatgttactcacttggcactctgg30<210>36<211>21<212>dna<213>人工序列<400>36gacccaaagttctggaattta21<210>37<211>30<212>dna<213>人工序列<400>37acgttggatgatattgtgcaactgggcctc30<210>38<211>30<212>dna<213>人工序列<400>38acgttggatgattccgttatcgacctcctg30<210>39<211>21<212>dna<213>人工序列<400>39gaaacttcagaggtttatcgg21<210>40<211>30<212>dna<213>人工序列<400>40acgttggatgtctgaggctctttttggttc30<210>41<211>30<212>dna<213>人工序列<400>41acgttggatggccctggatctttctcatac30<210>42<211>21<212>dna<213>人工序列<400>42ggtcttacctgagaatgtgtg21<210>43<211>30<212>dna<213>人工序列<400>43acgttggatgtacagggtcacaaaggtgtc30<210>44<211>30<212>dna<213>人工序列<400>44acgttggatgaccctagtttgagagaggag30<210>45<211>22<212>dna<213>人工序列<400>45ttgactcttctagcacgacact22<210>46<211>30<212>dna<213>人工序列<400>46acgttggatgaagtaaaccccagcaaagcc30<210>47<211>30<212>dna<213>人工序列<400>47acgttggatgaaggaactttcagctggctc30<210>48<211>22<212>dna<213>人工序列<400>48gggtaaaaacatgctgtgagaa22<210>49<211>30<212>dna<213>人工序列<400>49acgttggatgcctttgtaccccttcctcag30<210>50<211>29<212>dna<213>人工序列<400>50acgttggatgtggaccccctggcgagaag29<210>51<211>23<212>dna<213>人工序列<400>51ctttacagaaggaccatcatctc23<210>52<211>30<212>dna<213>人工序列<400>52acgttggatgttgcttggtgcacaagatgg30<210>53<211>30<212>dna<213>人工序列<400>53acgttggatgtaaggccaattactgctctg30<210>54<211>23<212>dna<213>人工序列<400>54acttttgcaaaaatacaaattca23<210>55<211>30<212>dna<213>人工序列<400>55acgttggatgtgggcgatctccacctgctt30<210>56<211>30<212>dna<213>人工序列<400>56acgttggatgatccagtccagcgcactgta30<210>57<211>23<212>dna<213>人工序列<400>57gcgcgagaggtgcccaggaggta23<210>58<211>30<212>dna<213>人工序列<400>58acgttggatgacaatctgattctcaaaggc30<210>59<211>30<212>dna<213>人工序列<400>59acgttggatgattctcacctgcaaagcctc30<210>60<211>24<212>dna<213>人工序列<400>60tgcccagaaagataacaagacgac24<210>61<211>29<212>dna<213>人工序列<400>61acgttggatgagccacacccagaccccag29<210>62<211>29<212>dna<213>人工序列<400>62acgttggatggtgttctgccacgaggtgc29<210>63<211>24<212>dna<213>人工序列<400>63ggggatggaaccccgcaaggctca24<210>64<211>30<212>dna<213>人工序列<400>64acgttggatgactctctgctaacccagatg30<210>65<211>30<212>dna<213>人工序列<400>65acgttggatgacatgtgatagcaggtgtag30<210>66<211>25<212>dna<213>人工序列<400>66gatgaccaccttcccctgcaggtac25<210>67<211>30<212>dna<213>人工序列<400>67acgttggatgtccggaaggacctgcataag30<210>68<211>30<212>dna<213>人工序列<400>68acgttggatgagatggctggcaactagaag30<210>69<211>25<212>dna<213>人工序列<400>69aggacggacctgcataagacggaga25<210>70<211>30<212>dna<213>人工序列<400>70acgttggatgtaatgattcccgcctcacag30<210>71<211>30<212>dna<213>人工序列<400>71acgttggatgtgctctctctccagggtaac30<210>72<211>26<212>dna<213>人工序列<400>72ccgtatggatgtcttcccgcggcctg26<210>73<211>29<212>dna<213>人工序列<400>73acgttggatggaacaaaaacttaagcaag29<210>74<211>30<212>dna<213>人工序列<400>74acgttggatgagccccatgatgagtttgag30<210>75<211>26<212>dna<213>人工序列<400>75taacaaaaacttaagcaagatacatg26<210>76<211>30<212>dna<213>人工序列<400>76acgttggatgcgtcgatgacattgtccagc30<210>77<211>29<212>dna<213>人工序列<400>77acgttggatggtcagcaacgtgctggaga29<210>78<211>27<212>dna<213>人工序列<400>78acgtagatgacattgtccagctgctgc27<210>79<211>29<212>dna<213>人工序列<400>79acgttggatgaggtaggcggctcccggca29<210>80<211>29<212>dna<213>人工序列<400>80acgttggatgctggcagcgtgcagcctgg29<210>81<211>15<212>dna<213>人工序列<400>81cgctcgctgcccccc15<210>82<211>30<212>dna<213>人工序列<400>82acgttggatgaacagcaccagagagcagca30<210>83<211>30<212>dna<213>人工序列<400>83acgttggatgctactccctaccttgttcag30<210>84<211>15<212>dna<213>人工序列<400>84agacctgccgcccac15<210>85<211>30<212>dna<213>人工序列<400>85acgttggatgccaaacacactgtgatgacc30<210>86<211>30<212>dna<213>人工序列<400>86acgttggatgccaaacacactgtgatgacc30<210>87<211>15<212>dna<213>人工序列<400>87accatgggctcagac15<210>88<211>30<212>dna<213>人工序列<400>88acgttggatgagtgactttgccttgtcttg30<210>89<211>30<212>dna<213>人工序列<400>89acgttggatgcaactctctcctgaagtcac30<210>90<211>15<212>dna<213>人工序列<400>90gcccctgaagaggtt15<210>91<211>30<212>dna<213>人工序列<400>91acgttggatgtattttccctgctaggagtc30<210>92<211>30<212>dna<213>人工序列<400>92acgttggatgaggtgtgtttggatcccaag30<210>93<211>16<212>dna<213>人工序列<400>93gctaggagtcgctgtg16<210>94<211>30<212>dna<213>人工序列<400>94acgttggatgttcccctcatccgtaccttc30<210>95<211>30<212>dna<213>人工序列<400>95acgttggatgaagggtctctctactcactc30<210>96<211>17<212>dna<213>人工序列<400>96tccgtaccttctctgtt17<210>97<211>30<212>dna<213>人工序列<400>97acgttggatgccatctcctccaaaccatgt30<210>98<211>29<212>dna<213>人工序列<400>98acgttggatgtgaaccccatgtcaccctc29<210>99<211>17<212>dna<213>人工序列<400>99ccaaaccatgtgactgt17<210>100<211>17<212>dna<213>人工序列<400>100ccaaaccatgtgactgt17<210>101<211>30<212>dna<213>人工序列<400>101acgttggatgaagtaaaccccagcaaagcc30<210>102<211>17<212>dna<213>人工序列<400>102gtgtaggtctcatggca17<210>103<211>30<212>dna<213>人工序列<400>103acgttggatgtaaaatccacccctttgccc30<210>104<211>30<212>dna<213>人工序列<400>104acgttggatgagaggagggatgttttgtgg30<210>105<211>19<212>dna<213>人工序列<400>105cccgtccctggacccatcc19<210>106<211>29<212>dna<213>人工序列<400>106acgttggatgggagctgagtcccgcttca29<210>107<211>30<212>dna<213>人工序列<400>107acgttggatgtgatgggaccgttggggtcg30<210>108<211>19<212>dna<213>人工序列<400>108cttaaccctgagcagcccc19<210>109<211>30<212>dna<213>人工序列<400>109acgttggatgtggtgaagtaaagccattcc30<210>110<211>30<212>dna<213>人工序列<400>110acgttggatgtgaccccatgttttccttcc30<210>111<211>19<212>dna<213>人工序列<400>111gtaaagccattccatcaaa19<210>112<211>30<212>dna<213>人工序列<400>112acgttggatgatggagaacagtgctgagcc30<210>113<211>30<212>dna<213>人工序列<400>113acgttggatgaggaccacacagctggtctc30<210>114<211>19<212>dna<213>人工序列<400>114cagcccgagcaaagcctgg19<210>115<211>30<212>dna<213>人工序列<400>115acgttggatgcagacaactgaagactctcc30<210>116<211>30<212>dna<213>人工序列<400>116acgttggatggatcttggtgaggttaaagg30<210>117<211>20<212>dna<213>人工序列<400>117gacgcagcagtggtcctgga20<210>118<211>30<212>dna<213>人工序列<400>118acgttggatgacaatcccagccaaatcttc30<210>119<211>30<212>dna<213>人工序列<400>119acgttggatgggacatatacttgcagggac30<210>120<211>30<212>dna<213>人工序列<400>120acgttggatgggacatatacttgcagggac30<210>121<211>30<212>dna<213>人工序列<400>121acgttggatggtcggtgatcaaagagtggg30<210>122<211>30<212>dna<213>人工序列<400>122acgttggatggtcggtgatcaaagagtggg30<210>123<211>21<212>dna<213>人工序列<400>123cgtcctggccgggacaccctg21<210>124<211>30<212>dna<213>人工序列<400>124acgttggatggcgttttgcactccaagttc30<210>125<211>30<212>dna<213>人工序列<400>125acgttggatgtcggggactcacctgtgtca30<210>126<211>21<212>dna<213>人工序列<400>126ccgccgtgccaacatggactg21<210>127<211>30<212>dna<213>人工序列<400>127acgttggatgacacacaaagtccgaggttc30<210>128<211>30<212>dna<213>人工序列<400>128acgttggatgctttgctcatagggaccaac30<210>129<211>21<212>dna<213>人工序列<400>129gggccggtgagtgcatccgcc21<210>130<211>29<212>dna<213>人工序列<400>130acgttggatgtgacgatggggtagcggag29<210>131<211>29<212>dna<213>人工序列<400>131acgttggatgaccacaggccagctctatg29<210>132<211>22<212>dna<213>人工序列<400>132gggaggaggctgccgtcagcca22<210>133<211>30<212>dna<213>人工序列<400>133acgttggatgaagagaggttctgggacagg30<210>134<211>30<212>dna<213>人工序列<400>134acgttggatgcatgacagctaaaacctggc30<210>135<211>23<212>dna<213>人工序列<400>135tacctttttcctgaaaagcttat23<210>136<211>30<212>dna<213>人工序列<400>136acgttggatgcctagttccttaatgcggtc30<210>137<211>30<212>dna<213>人工序列<400>137acgttggatgggcaccattactaatagccc30<210>138<211>24<212>dna<213>人工序列<400>138atgcggtcatttatgttaaatctt24<210>139<211>30<212>dna<213>人工序列<400>139acgttggatgatgaaggctatccatgggac30<210>140<211>30<212>dna<213>人工序列<400>140acgttggatgccagttagaaatgactgtcg30<210>141<211>24<212>dna<213>人工序列<400>141ggagaggacatgctgtttatcact24<210>142<211>30<212>dna<213>人工序列<400>142acgttggatggagtctttggttgctggaag30<210>143<211>30<212>dna<213>人工序列<400>143acgttggatgcagaagaacacccatcagag30<210>144<211>24<212>dna<213>人工序列<400>144ggatatggtgctgagaaaatacct24<210>145<211>30<212>dna<213>人工序列<400>145acgttggatgggagaaactcactctgtgtc30<210>146<211>30<212>dna<213>人工序列<400>146acgttggatgtttgatgcgccaagattccc30<210>147<211>25<212>dna<213>人工序列<400>147gcacctcactctgtgtctcagaacc25<210>148<211>29<212>dna<213>人工序列<400>148acgttggatgccatctaccgcagtacctg29<210>149<211>30<212>dna<213>人工序列<400>149acgttggatgacaagtaagtcttcccaccg30<210>150<211>26<212>dna<213>人工序列<400>150ggcggggaggtcacctacttcccttt26<210>151<211>29<212>dna<213>人工序列<400>151acgttggatgtcagcaacgtgctggagac29<210>152<211>30<212>dna<213>人工序列<400>152acgttggatgacgtcgatgacattgtccag30<210>153<211>26<212>dna<213>人工序列<400>153aggccgtcatgcgctgctggaggccg26<210>154<211>30<212>dna<213>人工序列<400>154acgttggatgctctggctttcagtaaaggg30<210>155<211>30<212>dna<213>人工序列<400>155acgttggatgagtctccaaagtgaaggagg30<210>156<211>26<212>dna<213>人工序列<400>156gggtgtttagggaagggcatttcttt26<210>157<211>30<212>dna<213>人工序列<400>157acgttggatgtatcaaacgtgaggccactg30<210>158<211>30<212>dna<213>人工序列<400>158acgttggatgagatcgccacctacaggaag30<210>159<211>15<212>dna<213>人工序列<400>159cccacctgcactcct15<210>160<211>30<212>dna<213>人工序列<400>160acgttggatggctccaattcactcttgctg30<210>161<211>30<212>dna<213>人工序列<400>161acgttggatggggagattcagtgacaatgg30<210>162<211>15<212>dna<213>人工序列<400>162cttgctggtgggttt15<210>163<211>30<212>dna<213>人工序列<400>163acgttggatgtgtttcaagctctggaactc30<210>164<211>30<212>dna<213>人工序列<400>164acgttggatgatgaccaacggatactcagg30<210>165<211>16<212>dna<213>人工序列<400>165tctggaactcctacct16<210>166<211>30<212>dna<213>人工序列<400>166acgttggatgaagctgggccaaattctggg30<210>167<211>30<212>dna<213>人工序列<400>167acgttggatggcgtgttgaaattctgcagg30<210>168<211>16<212>dna<213>人工序列<400>168attgtcccccaggaac16<210>169<211>30<212>dna<213>人工序列<400>169acgttggatgaactccaccatggtaggcac30<210>170<211>30<212>dna<213>人工序列<400>170acgttggatgtccttctgccagcccagctc30<210>171<211>16<212>dna<213>人工序列<400>171gggcatcggccagaac16<210>172<211>30<212>dna<213>人工序列<400>172acgttggatgttgaccatcacacccatctg30<210>173<211>30<212>dna<213>人工序列<400>173acgttggatgctttgctctttgctaagcgg30<210>174<211>16<212>dna<213>人工序列<400>174gggcaggacttaggcg16<210>175<211>30<212>dna<213>人工序列<400>175acgttggatgttggcaatggcagccactac30<210>176<211>29<212>dna<213>人工序列<400>176acgttggatgagtgcctggaggtgtccat29<210>177<211>17<212>dna<213>人工序列<400>177gggctgagaaagagccc17<210>178<211>30<212>dna<213>人工序列<400>178acgttggatgtcatctcctttagctccacc30<210>179<211>30<212>dna<213>人工序列<400>179acgttggatgatagattgacctagaagctg30<210>180<211>18<212>dna<213>人工序列<400>180cctccagccacagtgctc18<210>181<211>18<212>dna<213>人工序列<400>181cctccagccacagtgctc18<210>182<211>30<212>dna<213>人工序列<400>182acgttggatgcaaacatgggccatcatcac30<210>183<211>18<212>dna<213>人工序列<400>183cgttcattatgctgcatt18<210>184<211>29<212>dna<213>人工序列<400>184acgttggatgtcagcaacgtgctggagac29<210>185<211>29<212>dna<213>人工序列<400>185acgttggatgtcagcaacgtgctggagac29<210>186<211>18<212>dna<213>人工序列<400>186gggagacggcagtcatgc18<210>187<211>30<212>dna<213>人工序列<400>187acgttggatgccaaatccttcttcaccagc30<210>188<211>29<212>dna<213>人工序列<400>188acgttggatgtcctcggaggtgtcgggag29<210>189<211>19<212>dna<213>人工序列<400>189atccccagccagcagctgt19<210>190<211>30<212>dna<213>人工序列<400>190acgttggatgggaaggtgcagattttcaac30<210>191<211>30<212>dna<213>人工序列<400>191acgttggatgcacaaagaaatgcccttccc30<210>192<211>19<212>dna<213>人工序列<400>192tagtgagagtcaggctctg19<210>193<211>30<212>dna<213>人工序列<400>193acgttggatgagcctgtttgtccctgcttg30<210>194<211>30<212>dna<213>人工序列<400>194acgttggatggtgtccacacagtagcactc30<210>195<211>20<212>dna<213>人工序列<400>195ccctttctccctgtccagtg20<210>196<211>30<212>dna<213>人工序列<400>196acgttggatgccatctgcagcgtctttttc30<210>197<211>30<212>dna<213>人工序列<400>197acgttggatgccatctccagatgggttttg30<210>198<211>20<212>dna<213>人工序列<400>198tcttgtcttgataatccagg20<210>199<211>30<212>dna<213>人工序列<400>199acgttggatgggagttcatagacaagctcg30<210>200<211>29<212>dna<213>人工序列<400>200acgttggatggttgatgatctggccctgc29<210>201<211>21<212>dna<213>人工序列<400>201cctcctccagcccaatgtcat21<210>202<211>30<212>dna<213>人工序列<400>202acgttggatgacggctgtcttctcaacatc30<210>203<211>30<212>dna<213>人工序列<400>203acgttggatgttcacacgttgccatatgtc30<210>204<211>21<212>dna<213>人工序列<400>204ttgcttctatctcaggttgat21<210>205<211>30<212>dna<213>人工序列<400>205acgttggatgtgcttccaagcccatgtcac30<210>206<211>30<212>dna<213>人工序列<400>206acgttggatgtttgaaattgggcctgatcc30<210>207<211>21<212>dna<213>人工序列<400>207aatggggaggacttctggata21<210>208<211>30<212>dna<213>人工序列<400>208acgttggatgtgcctcttggggtagttgag30<210>209<211>30<212>dna<213>人工序列<400>209acgttggatgacctatggtgaacactaccc30<210>210<211>22<212>dna<213>人工序列<400>210cgtcgttgaggcactaccccaa22<210>211<211>30<212>dna<213>人工序列<400>211acgttggatgtcccctgctacttcatcgac30<210>212<211>30<212>dna<213>人工序列<400>212acgttggatgaatgcggctccacttgattc30<210>213<211>23<212>dna<213>人工序列<400>213accgggaccaccgccccctccac23<210>214<211>30<212>dna<213>人工序列<400>214acgttggatggggtggtagacttgatggtg30<210>215<211>30<212>dna<213>人工序列<400>215acgttggatgttctctgctgacttcatgcc30<210>216<211>22<212>dna<213>人工序列<400>216aggcgacttgatggtgaggatg22<210>217<211>30<212>dna<213>人工序列<400>217acgttggatgactctctcgggacgtagaag30<210>218<211>30<212>dna<213>人工序列<400>218acgttggatgataaccgtttggctgctctg30<210>219<211>23<212>dna<213>人工序列<400>219cagaggccgtgcctcctccgaag23<210>220<211>30<212>dna<213>人工序列<400>220acgttggatgcagctcaccagaaatatccc30<210>221<211>30<212>dna<213>人工序列<400>221acgttggatgattacgtcatgtgcgctcag30<210>222<211>24<212>dna<213>人工序列<400>222tctaaccagaaatatcccaaacgc24<210>223<211>30<212>dna<213>人工序列<400>223acgttggatgaaagcagagaggaggcaatg30<210>224<211>30<212>dna<213>人工序列<400>224acgttggatgtctctctgcctttttcctcc30<210>225<211>24<212>dna<213>人工序列<400>225gagcggaggcaatgagcttacctt24<210>226<211>30<212>dna<213>人工序列<400>226acgttggatgtatgatgtcctgctggttcc30<210>227<211>30<212>dna<213>人工序列<400>227acgttggatgaaaaggcacccagtctgtag30<210>228<211>24<212>dna<213>人工序列<400>228ctggtatattagagagttagggtt24<210>229<211>30<212>dna<213>人工序列<400>229acgttggatgtcaaggacttctacgagctg30<210>230<211>30<212>dna<213>人工序列<400>230acgttggatgcagggttacacatcaccaac30<210>231<211>25<212>dna<213>人工序列<400>231caatcagaccaatgggatcacccct25<210>232<211>30<212>dna<213>人工序列<400>232acgttggatggatcgccaccatgaaggaac30<210>233<211>30<212>dna<213>人工序列<400>233acgttggatgatgctgtgtaaccaaacagg30<210>234<211>26<212>dna<213>人工序列<400>234gaagtgtcgtcttcgtgtcttcccag26<210>235<211>30<212>dna<213>人工序列<400>235acgttggatgtaaaatccacccctttgccc30<210>236<211>30<212>dna<213>人工序列<400>236acgttggatgagaggagggatgttttgtgg30<210>237<211>15<212>dna<213>人工序列<400>237tccctggacccatcc15<210>238<211>30<212>dna<213>人工序列<400>238acgttggatgaccgctacatctccatcttc30<210>239<211>30<212>dna<213>人工序列<400>239acgttggatgagatggccgcaatgatcctc30<210>240<211>15<212>dna<213>人工序列<400>240gccctgcggtaccac15<210>241<211>30<212>dna<213>人工序列<400>241acgttggatgccacggcgatcttaaagtag30<210>242<211>30<212>dna<213>人工序列<400>242acgttggatgtctggctcatcaacattggc30<210>243<211>15<212>dna<213>人工序列<400>243ccacggatccaggag15<210>244<211>30<212>dna<213>人工序列<400>244acgttggatggggaagaaccatcgatcagc30<210>245<211>30<212>dna<213>人工序列<400>245acgttggatgaaacagagagctcctcaccc30<210>246<211>15<212>dna<213>人工序列<400>246gcctgggggtagact15<210>247<211>15<212>dna<213>人工序列<400>247gcctgggggtagact15<210>248<211>30<212>dna<213>人工序列<400>248acgttggatgagacgtagccactgggttct30<210>249<211>16<212>dna<213>人工序列<400>249atgaagcaaggctcag16<210>250<211>30<212>dna<213>人工序列<400>250acgttggatgtggcttgggacagtgatgtg30<210>251<211>29<212>dna<213>人工序列<400>251acgttggatgcagggccatgtgctgcttg29<210>252<211>17<212>dna<213>人工序列<400>252tcctgccctcacgacca17<210>253<211>30<212>dna<213>人工序列<400>253acgttggatgtgggagaaggaagtacatgg30<210>254<211>30<212>dna<213>人工序列<400>254acgttggatgcaactcattacagcatccgc30<210>255<211>17<212>dna<213>人工序列<400>255ccatcaactccatgcga17<210>256<211>30<212>dna<213>人工序列<400>256acgttggatgagaggaaggtgcagattttc30<210>257<211>30<212>dna<213>人工序列<400>257acgttggatggcccttccctaaatatccag30<210>258<211>17<212>dna<213>人工序列<400>258gtgagagtcaggctctg17<210>259<211>29<212>dna<213>人工序列<400>259acgttggatgataccccccagcagtacct29<210>260<211>30<212>dna<213>人工序列<400>260acgttggatgtccacgtgtcacaggatgag30<210>261<211>18<212>dna<213>人工序列<400>261ccgtacctggggcaccgt18<210>262<211>30<212>dna<213>人工序列<400>262acgttggatgtgatcttctgctccaagtcg30<210>263<211>30<212>dna<213>人工序列<400>263acgttggatggagaaggtgaccatgcagaa30<210>264<211>18<212>dna<213>人工序列<400>264ggaggaggccaggcggtc18<210>265<211>30<212>dna<213>人工序列<400>265acgttggatgtccacgtgtcacaggatgag30<210>266<211>29<212>dna<213>人工序列<400>266acgttggatgataccccccagcagtacct29<210>267<211>19<212>dna<213>人工序列<400>267agtaatgaacccctcgccc19<210>268<211>30<212>dna<213>人工序列<400>268acgttggatgaatactgtgcctgccaagtc30<210>269<211>30<212>dna<213>人工序列<400>269acgttggatgtggtggaatggccataaatg30<210>270<211>19<212>dna<213>人工序列<400>270cagccaactaccatggcac19<210>271<211>29<212>dna<213>人工序列<400>271acgttggatggtgagcgtcagcaacgtgc29<210>272<211>30<212>dna<213>人工序列<400>272acgttggatgtgacattgtccagctgctgc30<210>273<211>19<212>dna<213>人工序列<400>273atccgcaacgtgctggaga19<210>274<211>30<212>dna<213>人工序列<400>274acgttggatgtcatcttctcacagtagccc30<210>275<211>30<212>dna<213>人工序列<400>275acgttggatggaaagctgtctagactctgg30<210>276<211>20<212>dna<213>人工序列<400>276ggggtgcccttcggatctca20<210>277<211>30<212>dna<213>人工序列<400>277acgttggatggatcttcccaatctcggtgc30<210>278<211>29<212>dna<213>人工序列<400>278acgttggatgcctgcctcttcttcccctt29<210>279<211>20<212>dna<213>人工序列<400>279cgcaggctgcgatgttggtg20<210>280<211>29<212>dna<213>人工序列<400>280acgttggatgcactcacggtcaccttggc29<210>281<211>30<212>dna<213>人工序列<400>281acgttggatgtgatggcagaggtggagtag30<210>282<211>21<212>dna<213>人工序列<400>282gctccccgttcatgccgccgt21<210>283<211>30<212>dna<213>人工序列<400>283acgttggatggtatatggctattaggacgg30<210>284<211>30<212>dna<213>人工序列<400>284acgttggatggtgtctgtgtggaaggccat30<210>285<211>21<212>dna<213>人工序列<400>285gctattaggacggagatgatc21<210>286<211>31<212>dna<213>人工序列<400>286acgttggatggaacaaattacctctgtttgc31<210>287<211>30<212>dna<213>人工序列<400>287acgttggatgataaacagcatttctttatg30<210>288<211>22<212>dna<213>人工序列<400>288actaactctgtttgctgctgtt22<210>289<211>30<212>dna<213>人工序列<400>289acgttggatgactcctggagtcaagtgaag30<210>290<211>30<212>dna<213>人工序列<400>290acgttggatgggtgcaaatggctgaagaac30<210>291<211>22<212>dna<213>人工序列<400>291aatgctggtttcatgcttactc22<210>292<211>30<212>dna<213>人工序列<400>292acgttggatgcaatatgtgcttctgctgtg30<210>293<211>30<212>dna<213>人工序列<400>293acgttggatgtgtccagttcttctcccaag30<210>294<211>22<212>dna<213>人工序列<400>294ggggattctgctgtgcagtgtt22<210>295<211>30<212>dna<213>人工序列<400>295acgttggatgtttgcttaggagccatagcc30<210>296<211>30<212>dna<213>人工序列<400>296acgttggatgttactcaaacccctgtggtg30<210>297<211>23<212>dna<213>人工序列<400>297gggcgtcactttggagactccca23<210>298<211>30<212>dna<213>人工序列<400>298acgttggatgtcgcatcagcattgagaacg30<210>299<211>30<212>dna<213>人工序列<400>299acgttggatgtctcagccgttgaaccaatc30<210>300<211>30<212>dna<213>人工序列<400>300acgttggatgtctcagccgttgaaccaatc30<210>301<211>30<212>dna<213>人工序列<400>301acgttggatgtctcagccgttgaaccaatc30<210>302<211>30<212>dna<213>人工序列<400>302acgttggatgtctcagccgttgaaccaatc30<210>303<211>24<212>dna<213>人工序列<400>303ggtgactctacgtcggagacgtcg24当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1