一种重组菌及其用途的制作方法
本发明涉及生物化工领域,尤其涉及一种重组菌及其用途,以及基于重组微生物法合成β-榄香烯。
背景技术:
β-榄香烯(beta-elemene)是一种具有郁金香味的挥发性倍半萜化合物,为国家一类肿瘤新药的原料药。目前主要从郁金和莪术等植物中分离提取得到,但这种方法有较多的缺点,包括含量低和差异大,产品纯化难,植物生长周期长,对生物资源尤其野生资源造成严重破坏等。
利用合成生物学的原理,设计和改造微生物菌株来生产天然产物已被国际认为是一种最有潜力的方法,如在大肠杆菌中生产紫杉醇的前体紫杉二烯已达到1000mg/l(parayilkumaranajikumaretal.,2010,science,330:70-74);银杏内酯类(ginkgolides)前体左旋海松二烯(levopimaradiene),在改造后的大肠杆菌工程菌中达到700mg/l的产量(effendileonardetal.,2010,pnas,107(31):13654–13659);在酵母工程菌中生产青蒿素(artemisinin)的前体青蒿酸(artemisinicacid)最高达到25g/l(paddoncjetal.,2013,nature,496(7446):528-531);目前国内在青蒿素,紫杉醇和丹参酮等药物分子的生物合成方面有相关研究。
自然界中,法尼基焦磷酸(fpp)能被吉玛烯a合成酶(gmas)催化合成吉玛烯a,吉玛烯a具有热不稳定性,易发生分子内热重排为β-榄香烯目前已有部分利用重组菌生产β榄香烯前体吉玛烯a的研究,但产量均较低,达不到工业应用的要求。例如:高允允等人通过在大肠杆菌中构建吉玛烯a的生物合成途径,得到的重组菌合成吉玛烯a的最高产量仅为6.32mg/l,距离工业化还有较大的差距[微生物生物合成β-榄香烯前体—吉马烯a研究,高允允,2012,杭州师范大学]。
技术实现要素:
本发明一个目的是提供一种重组菌。
本发明提供的重组菌,为体内含有或表达吉玛烯a合成酶或吉玛烯a合成酶融合蛋白的酵母菌;
所述吉玛烯a合成酶融合蛋白包括所述吉玛烯a合成酶和法尼基焦磷酸合酶。
上述重组菌中,根据融合蛋白基因宿主来源不同,分为1种或多种,所述融合蛋白的编码核酸包括所述吉玛烯a合成酶的编码核酸和所述法尼基焦磷酸合酶的编码核酸;
所述融合蛋白的编码核酸为1种或多种;
多种所述融合蛋白的编码核酸中,至少2种所述吉玛烯a合成酶编码核酸来源宿主不同,且至少2种所述法尼基焦磷酸合酶编码核酸来源宿主不同。
本发明中所述的基因来源宿主不同是指基因的原始来源宿主不同,本发明所述吉玛烯a合成酶(germacreneasynthase)基因可从已知含有吉玛烯a合成酶的植物或微生物中克隆得到,例如可选自向日葵(helianthusannuusl.)、小白菊(tanacetumparthenium)、莴苣(lactucasativalinn.)、青蒿(artemisiacarvifolia)、蓝细菌等,所述法尼基焦磷酸合酶(farnesyldiphosphatesynthase)基因可从已知含有吉玛烯a合成酶的植物或微生物中克隆得到,例如可选自丹参(salviamiltiorrhiza)、酵母菌(yeast)、刺五加(acanthopanaxsenticosus(rupr.maxim.)harms)、杜仲(eucommiaulmoidesoliv.)等。
所述吉玛烯a合成酶的编码核酸包括seqidno.3所示的核酸或seqidno.12第13-1686位所示的核酸;
所述法尼基焦磷酸合酶的编码核酸包括seqidno.2所示的核酸或seqidno.11第1-1056位所示的核酸。
上述重组菌中,所述融合蛋白还包括用于连接所述吉玛烯a合成酶和所述法尼基焦磷酸合酶的连接肽;
所述连接肽选自gggs、ygq(3a001)、pggh(4a001)、yrsqi(5a002)、vipfis(6a005)、flylkf(6b004)、wrfspklq(8a005)或hhvqesqcistv(12a003)。
上述重组菌中,所述体内含有或表达吉玛烯a合成酶或吉玛烯a合成酶融合蛋白为将所述吉玛烯a合成酶的编码核酸或所述融合蛋白的编码核酸导入所述酵母菌;
和/或,所述将吉玛烯a合成酶的编码核酸导入所述酵母菌为将含有所述吉玛烯a合成酶的编码核酸表达盒导入所述酵母菌;
所述将融合蛋白的编码核酸导入所述酵母菌为将含有所述融合蛋白的编码核酸表达盒导入所述酵母菌;
和/或,所述含有吉玛烯a合成酶的编码核酸表达盒包括启动子、所述吉玛烯a合成酶的编码核酸和终止子;
和/或,所述含有融合蛋白的编码核酸表达盒包括启动子、所述融合蛋白的编码核酸和终止子;
或所述启动子选自tef1或mf1或pgk1;所述终止子为cyc1或adh1;
或所述启动子为tef1,且终止子为cyc1;
或所述启动子为mf1,且终止子为cyc1;
或所述启动子为pgk1,且终止子为adh1。
上述中,启动子tef1包含seqidno.4所示的序列;所述启动子mf1包含seqidno.1所示的序列;所述终止子cyc1t包含seqidno.5所示的序列。
上述重组菌中,所述重组菌还表达1个或多个标记基因;和/或,所述标记基因选自his3或trp1。
上述重组菌中,所述含有吉玛烯a合成酶的编码核酸表达盒通过表达所述吉玛烯a合成酶的编码核酸表达盒的载体导入所述酵母菌;
所述含有融合蛋白的编码核酸表达盒通过表达所述含有融合蛋白的编码核酸表达盒的载体导入所述酵母菌。
上述重组菌中,所述吉玛烯a合成酶的编码核酸表达盒通过质粒形式导入所述酵母菌;
或,所述融合蛋白的编码核酸表达盒通过质粒形式和/或整合在染色体的形式导入所述酵母菌。
在本发明的实施例中,
融合蛋白选自如下中至少一种:synsmfps-gggs-stpgmas,synsmfps-ygq-stpgmas,synsmfps-pggh-stpgmas,synsmfps-yrsqi-stpgmas,synsmfps-vipfis-stpgmas,synsmfps-flylkf-stpgmas,synsmfps-wrfspklq-stpgmas,synsmfps-hhvqesqcistv-stpgmas,synsmfps-wrfspklq-stpgmas,erg20-gggs-lsltc2;
融合蛋白优选为synsmfps-8a005-stpgmas;
融合蛋白尤其优选为3种融合蛋白:synsmfps-wrfspklq(8a005)-stpgmas、erg20-gggs-lsltc2、synsmfps-gggs-stpgmas;
表达融合蛋白编码核酸的表达盒选自如下至少一种:
ptef1-synsmfps-gggs-stpgmas-tcyc1,
ptef1-synsmfps-ygq-stpgmas-tcyc1,
ptef1-synsmfps-pggh-stpgmas-tcyc1,
ptef1-synsmfps-yrsqi-stpgmas-tcyc1,
ptef1-synsmfps-vipfis-stpgmas-tcyc1,
ptef1-synsmfps-flylkf-stpgmas-tcyc1,
ptef1-synsmfps-wrfspklq(8a005)-stpgmas-tcyc1,
ptef1-synsmfps-hhvqesqcistv-stpgmas-tcyc1,
或pmf1-synsmfps-wrfspklq(8a005)-stpgmas-tcyc1;
表达融合蛋白编码核酸的表达盒优选为:pmf1-synsmfps-8a005-stpgmas-tcyc1;
表达融合蛋白编码核酸的表达盒尤其优选为3种:pmf1-synsmfps-8a005-stpgmas-tcyc1、ppgk1-erg20-gggs-lsltc2-tadh1和ptef1-synsmfps-gggs-stpgmas-tcyc1。
表达吉玛烯a合成酶编码核酸表达盒的载体选自如下:
prs313-leu2-ptef1-stpgmas-tcyc1,
prs425-leu2-ptef1-stpgmas-tcyc1,
表达吉玛烯a合成酶编码核酸表达盒的载体选自如下:
prs425-leu2-ptef1-synsmfps-gggs-stpgmas-tcyc1,
prs425-leu2-ptef1-synsmfps-ygq-stpgmas-tcyc1,
prs425-leu2-ptef1-synsmfps-pggh-stpgmas-tcyc1,
prs425-leu2-ptef1-synsmfps-yrsqi-stpgmas-tcyc1,
prs425-leu2-ptef1-synsmfps-vipfis-stpgmas-tcyc1,
prs425-leu2-ptef1-synsmfps-flylkf-stpgmas-tcyc1,
prs425-leu2-ptef1-synsmfps-wrfspklq-stpgmas-tcyc1,
prs425-leu2-ptef1-synsmfps-hhvqesqcistv-stpgmas-tcyc1,
或prs425-leu2-pmf1-synsmfps-wrfspklq-stpgmas-tcyc1;
上述整合在染色体上的融合蛋白基因表达盒选自ptef1-synsmfps-gggs-stpgmas-tcyc1和ppgk1-erg20-gggs-lsltc2-tadh1。
上述重组菌中,所述酵母菌为提高出发酵母菌中乙醇脱氢酶、乙醛脱氢酶和乙酰辅酶a合成酶的含量和/或活性得到的菌。
所述提高出发酵母菌中乙醇脱氢酶、乙醛脱氢酶和乙酰辅酶a合成酶的含量和/或活性得到的菌为增加所述出发酵母菌中乙醇脱氢酶的编码核酸、乙醛脱氢酶的编码核酸和乙酰辅酶a合成酶的编码核酸的拷贝数;
上述重组菌中,所述增加出发酵母菌中乙醇脱氢酶的编码核酸、乙醛脱氢酶的编码核酸和乙酰辅酶a合成酶的编码核酸的拷贝数为将乙醇脱氢酶的编码核酸表达盒、乙醛脱氢酶的编码核酸表达盒、乙酰辅酶a合成酶的编码核酸表达盒和另一个所述标记基因(his3)采用同源重组导入所述出发酵母菌。
上述重组菌中,所述出发酵母菌为酿酒酵母;和/或,所述酿酒酵母为nk2-sq。
1个所述标记基因为trp1;另一个所述标记基因为his3。
上述乙醇脱氢酶的基因adh2包含seqidno.6所示的序列,乙醛脱氢酶的基因ald6包含seqidno.7所示的序列,所述乙酰辅酶a合成酶的基因acs1包含seqidno.8所示的序列。
本发明的重组菌及各个所需载体和片段的构建见实施例。
在本发明的实施例中,重组菌具体如下:
重组菌ele-001,为将prs313-leu2-ptef1-stpgmas-tcyc1导入酵母fpp-001得到的菌;
重组菌ele-002,为将prs425-leu2-ptef1-stpgmas-tcyc1导入酵母fpp-001得到的菌;
重组菌ele-011,为将prs425-leu2-ptef1-synsmfps-gggs-stpgmas-tcyc1导入酵母fpp-001得到的菌;
重组菌ele-012,为将prs425-leu2-ptef1-synsmfps-3a001-stpgmas-tcyc1导入酵母fpp-001得到的菌;
重组菌ele-013,为将prs425-leu2-ptef1-synsmfps-4a001-stpgmas-tcyc1导入酵母fpp-001得到的菌;
重组菌ele-014,为将prs425-leu2-ptef1-synsmfps-5a002-stpgmas-tcyc1导入酵母fpp-001得到的菌;
重组菌ele-015,为将prs425-leu2-ptef1-synsmfps-6a005-stpgmas-tcyc1导入酵母fpp-001得到的菌;
重组菌ele-016,为将prs425-leu2-ptef1-synsmfps-6b004-stpgmas-tcyc1导入酵母fpp-001得到的菌;
重组菌ele-017,为将prs425-leu2-ptef1-synsmfps-8a005-stpgmas-tcyc1导入酵母fpp-001得到的菌;
重组菌ele-018,为将prs425-leu2-ptef1-synsmfps-12a003-stpgmas-tcyc1导入酵母fpp-001得到的菌;
重组菌ele-019,为将prs425-leu2-pmf1-synsmfps-8a005-stpgmas-tcyc1导入酵母fpp-001得到的菌;
重组菌ele-020,为将prs425-leu2-pmf1-synsmfps-8a005-stpgmas-tcyc1、导入酵母fpp-001后,再通过同源重组导入ppgk1-erg20-gggs-lsltc2-tadh1,ptef1-synsmfps-gggs-stpgmas-tcyc1,rdna-trp1-up和rdna-trp1-down得到的菌。
上述酵母fpp-001为将ndt80-his3-up、ppgk1-adh2-tadh1、ptdh3-acs1-ttpi1、ptef1-ald6-tcyc1和ndt80-his3-down导入酿酒酵母中得到的菌。
其中重组菌ele-020为酿酒酵母saccharomycescerevisiaecgmccno.14829也是本发明保护的范围。
该ele-020重组菌株于2017年10月20日保藏于中国微生物菌种保藏管理委员会普通微生物中心(cgmcc),保藏地址:北京市朝阳区北辰西路1号院3号,菌种名称:酿酒酵母,拉定名:saccharomycescerevisiae,保藏编号:cgmccno.14829。
上述重组菌在生产β-榄香烯和/或吉玛烯a中的应用也是本发明保护的范围。
本发明的第3个目的是提供一种生产吉玛烯a的方法。
本发明提供的方法,包括如下步骤:发酵上述的重组菌,得到吉玛烯a;
本发明的第4个目的是提供一种生产β-榄香烯的方法。
本发明提供的方法,包括如下步骤:
1)发酵上述的重组菌,得到发酵产物;
2)有机溶液萃取所述发酵产物,收集有机相;
3)将所述有机相加热,得到β-榄香烯。
上述方法中,所述发酵为先将所述重组菌在种子培养基中培养获得种子液;再将所述种子液接种到发酵培养基中发酵培养,将所述发酵培养所述产物记作发酵体系。
上述方法中,在所述发酵培养过程中,向所述发酵体系中添加补料培养基;优选地,待所述发酵体系中的溶氧值大于60%时,向所述发酵体系中加入补料培养基至所述发酵体系中葡萄糖浓度为5g/l。
上述方法中,每l体积所述种子培养基和所述发酵培养基配方包含:25g葡萄糖,15g硫酸铵,6.15g七水硫酸镁,0.72g七水硫酸锌,8g磷酸二氢钾,2ml氯化钙母液,10ml微量金属盐母液;12ml维他命母液,1g色氨酸,其余为水。
所述氯化钙母液为19.2g/l二水氯化钙水溶液;
每l体积所述微量金属盐母液的配方包含:19.1g乙二胺四乙酸二钠;10.2g七水硫酸锌;0.5g四水氯化锰;0.86g六水氯化钴;0.78g五水硫酸铜;0.56g二水钼酸钠;5.12g七水亚硫酸铁;其余为水。
每l体积维他命母液的配方包含:0.05g生物素;0.2g对氨基苯甲酸纳;1g烟酸;1g泛酸钙;1g盐酸吡哆醇;1g盐酸硫胺素;25g肌醇;其余为水。
每l体积所述补料培养基的配方含有800g葡萄糖,5.125g七水硫酸镁,3.5g硫酸钾,0.28g硫酸钠,9g磷酸二氢钾和1g色氨酸;其余为水。
在所述发酵前,还包括如下步骤:
a)在固体选择培养基中活化重组菌;
b)在液体选择培养基中震荡培养后,转入种子培养基培养,得到种子液;
其中,所述固体或液体选择培养基为sd-ura-his-leu培养基;
上述步骤b)中培养条件为30℃,250rpm;所述接种步骤为火焰环接种;
上述发酵方法中所述种子液的培养方法具体为重组菌活化后,挑取平板上的单克隆至装有sd-ura-his-leu培养基的试管,30℃,250rpm振荡培养过夜;吸取500μl菌液至装有50mlsd-ura-his-leu培养基的250ml三角瓶中,30℃,250rpm振荡培养24h;分别吸取2ml菌液至3个装有100ml种子培养基的1l三角瓶中,30℃,250rpm振荡培养48h。
上述生产β-榄香烯的方法中,所述有机溶剂为正十二烷;所述加热条件为:100-380℃加热1小时。
本发明的实验证明,本发明在宿主酵母中表达吉玛烯a合成酶基因或其融合蛋白基因,得到重组菌,可以大大提高吉玛烯a的产量,适用于工业化生产β-榄香烯和/或吉玛烯a,为抗癌原料β-榄香烯的生物合成方面提供有力菌株及研究基础。
附图说明
图1为吉玛烯a生物合成途径。
图2为gc-ms检测图。
具体实施方式
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
图1为吉玛烯a生物合成途径。
实施例1、目标基因和所用质粒制备
一、目标基因的制备
1、adh2、ald6、asc1、mf1、tef1和cyc1的获得
提取酵母nk2-sq(中国中药杂志,林庭庭,王冬,戴住波,张学礼,黄璐琦,2016,41(6):1008-1015)的基因组dna作为模板,用表1中基因扩增所需的引物进行扩增,得到符合预期大小的片段adh2、ald6、asc1基因、启动子mf1、tef1和终止子cyc1。
取pcr扩增试剂盒takara
表1为引物序列
2、来自丹参的法尼基焦磷酸合酶基因synsmfps和来自小白菊的吉玛烯a合成酶基因stpgma的获得
南京金斯瑞生物科技有限公司依照synsmfps(seqidno.2,来源于丹参)和stpgmas(seqidno.3,来源于小白菊)基因序列,设计全长引物,利用overlap方法形成模版dna,再利用pcr扩增的方法得到synsmfps(seqidno.2)和stpgmas(seqidno.3)的双链dna,然后将pcr产物转化克隆至克隆载体puc57(南京金斯瑞生物科技有限公司)中,分别构建了含synsmfps基因和stpgmas基因的克隆型质粒puc57-synsmfps和puc57-stpgmas。
3、来自酵母的法尼基焦磷酸合酶基因erg20-gggs和来自莴苣的吉玛烯a合成酶基因gggs-lsltc2的获得
提取莴苣叶片200mg用液氮研磨后ctab法(十六烷基三甲基溴化铵法)提取总rna:在1.5ml离心管中加入1ml2*ctab提取液(2%ctab,100mmtris-hclph8.0,20mmedta溶液(乙二胺四乙酸),1.4mnacl溶液),65℃预热之后,加入20μl2-mercaptoethanol(2-巯基乙醇);加入少量莴苣叶片粉末(约50mg),混合均匀,65℃保温10min,摇匀5次;4℃,12000rpm离心10min,移出上清,用等体积的氯仿/异戊醇抽提;4℃,12000rpm离心10min,移出上清,用等体积的氯仿/异戊醇抽提;4℃,12000rpm离心10min,移出上清,用1/6体积的氯仿/异戊醇抽提;4℃,15000rpm离心30min,移出上清,加入1/4体积的10mol/llicl,4℃放置过夜;4℃,15000rpm离心30min,弃去上清,用75%乙醇洗涤沉淀2次,无水乙醇洗涤沉淀1次,超净台放置15min(室温);用20μlmilliqdepc(溶剂为miliq纯水,溶质为diethylpyrocarbonate焦碳酸二乙酯;焦碳酸二乙酯:水的体积比为1:1000)处理水溶解,加入1/10体积的2mol/lnaac(ph4.0),加入2体积的无水乙醇,-20℃放置2h;4℃,12000rpm离心10min,弃上清,沉淀用75%乙醇洗涤两次,无水乙醇洗涤沉淀1次;超净台放置15min(室温),加15μlmilliqdepc处理水使沉淀充分溶解,-70℃保存。
第一链反转录-pcr:取无rna酶pcr管,按第一链反转录试剂盒(宝生物工程(大连)有限公司)配备体系:radom6mers2ul、dntp1ul、totalrna1ul(200ng)、h2o6ul、total10ul、瞬间离心,pcr65℃5min,冰上急冷;再加入以下体系中反应(第一链反转录试剂盒自带):5*primerbuffer4ul、rnasinhibiter0.5ul、r-transcription1ul、h2o4.5ul,瞬间离心,pcr仪进行反应:30℃10min、42℃60min、70℃15min、4℃保温。
分别以nk2-sq的基因组dna和莴苣cdna为模板,用表2中引物分别扩增得到约1068bperg20-gggs(seqidno.11中第13-1686为erg20)和1688bpgggs-lsltc2(seqidno.12中第1-1056为lsltc2)。
按pcr扩增试剂盒phusionhigh-fidelitypcrmastermixwithhfbuffer((购自neb(北京)有限公司)配置体系。扩增体系均为:5×phusionhfbuffer10μl、dntp(10mmeachdntp)1μl、dna模板20ng、引物(10μm)各1.5μl、phusionhigh-fidelitydnapolymerase(2.5u/μl)0.5μl、补加蒸馏水至总体积50μl。扩增条件为:98℃预变性3分钟(1个循环);98℃变性10秒、58℃退火10秒、72℃延伸1分钟(30个循环);72℃延伸10分钟(1个循环)。
表2引物序列
二、重组质粒的构建
1、pm2-adh2质粒
用sexa1(购自neb(北京)有限公司)和asc1(购自neb(北京)有限公司)分别对上述“一、目标基因的制备”中扩增得到的adh2以及质粒pm2-thmg1(记载在中国专利zl201310399947.x中)进行双酶切,得到1052bpadh2酶切产物和4738bp质粒pm2-thmg1酶切后骨架;再将adh2酶切产物与质粒pm2-thmg1酶切后骨架连接,得到重组质粒pm2-adh2。
2、pm4-acs1质粒
用sexa1和asc1分别对上述“一、目标基因的制备”中扩增得到的acs1以及质粒pm4-atcpr1(记载在中国专利zl201310399947.x中)进行双酶切;得到2201bpacs1酶切产物、和5061bp质粒pm4-atcpr1酶切后骨架;再将acs1酶切产物与质粒pm4-atcpr1酶切后骨架连接,得到重组质粒pm4-acs1。
3、pm3-ald6质粒
用sexa1和asc1分别上述“一、目标基因的制备”中扩增得到的ald6以及质粒pm3-erg9(记载在中国专利申请zl201310399947.x中)进行双酶切;得到1511bpald6酶切产物和4598bp质粒pm3-erg9酶切后骨架;再将ald6酶切产物与质粒pm3-erg9酶切后骨架连接,得到重组质粒pm3-ald6。
4、prs313-leu2-ptef1-stpgmas-tcyc1和prs425-leu2-ptef1-stpgmas-tcyc1质粒的构建
sexa1酶切上述“一、目标基因的制备”中扩增得到的tef1,得到440bptef1酶切产物;
asc1酶切上述“一、目标基因的制备”中扩增得到的cyc1,得到322bpcyc1酶切产物;
sexa1和asc1酶切puc57-stpgmas,回收1694bp的stpgmas;
将酶切产物tef1、cyc1和stpgmas各50ng加入连接体系:2μl10×t4dnaligasereactionbuffer(neb公司)、1μlt4dnaligase(neb公司,400,000cohesiveendunits/ml),补充蒸馏水至20μl,室温反应2小时得到连接产物;
取1μl连接产物加入pcr体系(phusionhigh-fidelitypcrmastermixwithhfbuffer试剂盒,neb公司):5×phusionhfbuffer10μl、dntp(10mmeachdntp)1μl、dna模板20ng、加入表3引物sac11-tef1和cyc1-sac11(10μm)各1.5μl、phusionhigh-fidelitydnapolymerase(2.5u/μl)0.5μl、补加蒸馏水至总体积50μl。扩增条件为:98℃预变性3分钟(1个循环);98℃变性10秒、58℃退火10秒、72℃延伸1.5分钟(30个循环);72℃延伸10分钟(1个循环)。得到2456bp的pcr扩增产物。
将扩增产物纯化,然后用sacii酶切,割胶回收目的片段sacii-tef1-stpgmas-cyc1-sacii,备用。
用sacii分别酶切质粒prs313(sikorski,r.s.andhieter,p.1989,genetics122(1):19-27)和prs425(sikorski,r.s.andhieter,p.1989,genetics122(1):19-27),得到4967bp的prs313载体片段和6849bp的prs425载体片段;再分别加入4μlnebbuffer、1μlcip去磷酸化酶(neb公司),补充蒸馏水至40μl,37℃处理1h,加入终浓度为10μmol的edta,65℃30min终止反应,割胶回收prs313-sacii载体片段和prs425-sacii载体片段。
将上述“一、目标基因的制备”步骤所得载体片段prs313-sacii、prs425-sacii分别和sacii-tef1-stpgmas-cyc1-sacii各50ng加入连接体系:2μl10×t4dnaligasereactionbuffer(neb公司)、1μlt4dnaligase(neb公司,400,000cohesiveendunits/ml),补充蒸馏水至20μl,室温反应2小时得到连接产物,转入trans10感受态细胞中和测序验证,得到prs313-his3-ptef1-stpgmas-tcyc1和prs425-leu2-ptef1-stpgmas-tcyc1质粒。
以prs313-his3-ptef1-stpgmas-tcyc1质粒为模板,用引物表3中引物,扩增6692bp质粒骨架prs313-tef1-stpgmas-cyc1;
用prs425为模板,用引物表3中引物,扩增leu2(1808bp)。
扩增体系均为:5×phusionhfbuffer10μl、dntp(10mmeachdntp)1μl、dna模板20ng、引物(10μm)各1.5μl、phusionhigh-fidelitydnapolymerase(2.5u/μl)0.5μl、补加蒸馏水至总体积50μl。扩增条件为:98℃预变性3分钟(1个循环);98℃变性10秒、58℃退火10秒、72℃延伸4分钟(30个循环);72℃延伸10分钟(1个循环)。
割胶纯化目的片段。在产物leu2片段中加入2μl10×t4dnaligasereactionbuffer(neb公司)、1μlt4polynucleotidekinase(neb公司),补充蒸馏水至20μl,37℃磷酸化1h,割胶回收后同prs313-ptef1-stpgmas-tcyc1用t4dna连接酶(neb公司)连接,转化,测序验证后得到质粒:prs313-leu2-ptef1-stpgmas-tcyc1。
表3引物序列
5、prs425-leu2-ptef1-synsmfps-gggs-stpgmas-tcyc1质粒的构建
以puc57-synsmfps和puc57-stpgmas为模板,用表4中引物分别扩增得到1080bpsynsmfps-gggs和1704bpgggs-stpgmas。
扩增体系均为:5×phusionhfbuffer10μl、dntp(10mmeachdntp)1μl、dna模板20ng、引物(10μm)各1.5μl、phusionhigh-fidelitydnapolymerase(2.5u/μl)0.5μl、补加蒸馏水至总体积50μl。扩增条件为:98℃预变性3分钟(1个循环);98℃变性10秒、58℃退火10秒、72℃延伸1分钟(30个循环);72℃延伸10分钟(1个循环)。
对synsmfps-gggs和gggs-stpgmas共同作为模板,用表4中引物(sexa1-synsmfps和stpgmas-asc1)扩增得到2767bpsynsmfps-gggs-stpgmas片段。
扩增体系均为:5×phusionhfbuffer10μl、dntp(10mmeachdntp)1μl、dna模板synsmfps-gggs和gggs-stpgmas各20ng、引物(10μm)各1.5μl、phusionhigh-fidelitydnapolymerase(2.5u/μl)0.5μl、补加蒸馏水至总体积50μl。扩增条件为:98℃预变性3分钟(1个循环);98℃变性10秒、58℃退火10秒、72℃延伸2分钟(30个循环);72℃延伸10分钟(1个循环)。
将扩增产物纯化,然后用sexa1和asc1酶切,割胶回收目的片段sexa1-synsmfps-gggs-stpgmas-asc1(2760bp),备用。
将前述第“4”项中所构建质粒prs425-leu2-ptef1-stpgmas-tcyc1用sexa1和asc1酶切,割胶回收7602bp大片段,得到载体prs425-leu2-ptef1-...-tcyc1;将载体prs425-leu2-ptef1-...-tcyc1与sexa1-synsmfps-gggs-stpgmas-asc1各50ng加入连接体系:2μl10×t4dnaligasereactionbuffer(neb公司)、1μlt4dnaligase(neb公司,400,000cohesiveendunits/ml),补充蒸馏水至20μl,室温反应2小时得到连接产物,转入trans10感受态细胞中,提取质粒测序验证,得到prs425-leu2-ptef1-synsmfps-gggs-stpgmas-tcyc1质粒。
表4引物序列
6、prs425-leu2-pmf1-synsmfps-gggs-stpgmas-tcyc1质粒的构建
用bamh1(购自takara公司)和sexa1分别对上述“一、目标基因的制备”得到的mf1和前述第“5”项所构建的质粒prs425-leu2-ptef1-synsmfps-gggs-stpgmas-tcyc1进行双酶切;割胶纯化814bp目标启动子基因mf1和9898bp载体片段prs425-leu2-...-synsmfps-gggs-stpgmas-tcyc1,并将二者加入连接体系(各50ng):2μl10×t4dnaligasereactionbuffer(neb公司)、1μlt4dnaligase(neb公司,400,000cohesiveendunits/ml),补充蒸馏水至20μl,室温反应2小时得到连接产物,转化trans10感受态细胞中,提取质粒测序验证,将得到的序列正确的质粒命名为prs425-leu2-pmf1-synsmfps-gggs-stpgmas-tcyc1。
7、.pm2-erg20-gggs-lsltc2质粒的构建
对erg20-gggs和gggs-lsltc2共同作为模板,用表5中引物(sexa1-erg20和lsltc2-asc1)扩增得到约2744bperg20-gggs-lsltc2片段。
扩增体系均为:5×phusionhfbuffer10μl、dntp(10mmeachdntp)1μl、dna模板erg20-gggs和gggs-lsltc2各20ng、引物(10μm)各1.5μl、phusionhigh-fidelitydnapolymerase(2.5u/μl)0.5μl、补加蒸馏水至总体积50μl。扩增条件为:98℃预变性3分钟(1个循环);98℃变性10秒、58℃退火10秒、72℃延伸2分钟(30个循环);72℃延伸10分钟(1个循环)。
将扩增产物纯化,然后用sexa1和asc1酶切,割胶回收目的片段sexa1-erg20-gggs-lsltc2-asc1(约2744bp),然后将其与载体与质粒pm2-thmg1酶切后骨架连接,得到重组质粒pm2-erg20-gggs-lsltc2。
表5引物序列
8、peasy-ndt80-his3质粒的构建
分别以nk2-sq的基因组dna和prs313模板,用引物表6中引物,扩增得到1252bpndt80(seqidno.13)和1168bphis3(seqidno.14)。
扩增体系为:5×phusionhfbuffer10μl、dntp(10mmeachdntp)1μl、dna模板20ng、引物(10μm)各1.5μl、phusionhigh-fidelitydnapolymerase(2.5u/μl)0.5μl、补加蒸馏水至总体积50μl。扩增条件为:98℃预变性3分钟(1个循环);98℃变性10秒、58℃退火10秒、72℃延伸1分钟(30个循环);72℃延伸10分钟(1个循环)。
将扩增产物ndt80克隆到peasy-bluntsimple克隆载体(peasy克隆载体,北京全式金生物技术(transgenbiotech)有限公司)转化trans10感受态细胞中,提取质粒测序验证,得到质粒peasy-ndt80。
表6引物
pmei(购自neb(北京)有限公司))酶切peasy-ndt80,割胶纯化5122bp目的片段(30ng),4μlnebbuffer(反应缓冲液,购自neb(北京)有限公司)、1μlcip去磷酸化酶(neb公司),补充蒸馏水至40μl,37℃处理1h,加入终浓度为10μmol的edta,65℃30min终止反应,割胶回收5122bp目的片段peasy-ndt80,备用。
割胶纯化his3(30ng),加入4μl10×t4dnaligasereactionbuffer(neb公司)、1μlt4polynucleotidekinase(neb公司),补充蒸馏水至40μl,37℃磷酸化1h,割胶回收后,与peasy-ndt80用t4dna连接酶连接(neb公司),转化trans10感受态细胞,测序验证,得到质粒peasy-ndt80-his3。
上述构建的各质粒信息如下表7所示:
表7质粒信息
9、peasy-rdna-trp1质粒的构建
分别以nk2-sq的基因组dna和prs314(sikorski,r.s.andhieter,p.1989,genetics122(1):19-27)模板,用引物表8中引物,扩增得到约rdna(seqidno.9)和trp1(seqidno.10)。
扩增体系为:5×phusionhfbuffer10μl、dntp(10mmeachdntp)1μl、dna模板20ng、引物(10μm)各1.5μl、phusionhigh-fidelitydnapolymerase(2.5u/μl)0.5μl、补加蒸馏水至总体积50μl。扩增条件为:98℃预变性3分钟(1个循环);98℃变性10秒、58℃退火10秒、72℃延伸1分钟(30个循环);72℃延伸10分钟(1个循环)。
将扩增产物rdna克隆到peasy-bluntsimple克隆载体转化trans10感受态细胞中,提取质粒测序验证,得到质粒peasy-rdna。
表8引物
pmei酶切peasy-rdna,割胶纯化5122bp目的片段(30ng),4μlnebbuffer、1μlcip去磷酸化酶(neb公司),补充蒸馏水至40μl,37℃处理1h,加入终浓度为10μmol的edta,65℃30min终止反应,割胶回收5122bp目的片段peasy-rdna,备用。
割胶纯化trp1(30ng),加入4μl10×t4dnaligasereactionbuffer(neb公司)、1μlt4polynucleotidekinase(neb公司),补充蒸馏水至40μl,37℃磷酸化1h,割胶回收后,与peasy-rdna用t4dna连接酶(neb公司)连接,转化trans10感受态细胞,测序验证,得到质粒peasy-rdna-trp1。
实施例2、重组菌的构建
一、酵母感受态的制备
将出发菌分别于相应培养基(表13)中30℃,250rpm过夜培养,取1ml(od约0.6-1.0)分装到1.5mlep管中,4℃、10000g离心1min,弃上清,沉淀用无菌水(4℃)洗涤,同样条件下离心,弃上清。菌体加入1ml处理液(10mmliac(醋酸锂);10mmdtt(二硫苏糖醇);0.6msorbitol(山梨糖醇);10mmtris-hcl(三羟甲基氨基甲烷盐酸缓冲溶液,ph7.5),处理液使用时才加dtt),25℃下放置20min。离心,弃上清,菌体中加入1ml1msorbitol(0.22μm水系膜过膜除菌)重悬,离心,弃上清(用1msorbitol重悬二次),到最终体积约为90μl。
二、fpp-001菌株的构建
1、ndt80-his3-up,ppgk1-adh2-tadh1,ptdh3-acs1-ttpi1,ptef1-ald6-tcyc1和ndt80-his3-down的制备
ppgk1-adh2-tadh1,ptdh3-acs1-ttpi1,ptef1-ald6-tcyc1分别为携带有乙醇脱氢酶2、乙酰辅酶a合成酶1和乙醛脱氢酶6的表达盒;ndt80-his3-up和ndt80-his3-down分别为his3的上游同源臂和下游同源臂;分别按照如下方法扩增片段:
分别用表9描述的pcr模板和引物进行pcr获得功能模块:698bpm1(ndt80-his3-up),2081bpm2(ppgk1-adh2-tadh1),3519bpm3(ptdh3-acs1-ttpi1),2376bpm4(ptef1-ald6-tcyc1),1835bpm5(ndt80-his3-down)。
扩增体系为:5×phusionhfbuffer10μl、dntp(10mmeachdntp)1μl、dna模板20ng、引物(10μm)各1.5μl、phusionhigh-fidelitydnapolymerase(2.5u/μl)0.5μl、补加蒸馏水至总体积50μl。扩增条件为:98℃预变性3分钟(1个循环);98℃变性10秒、58℃退火10秒、72℃延伸均用2分钟(30个循环);72℃延伸10分钟(1个循环),产物经割胶回收保存。
表9引物
2、fpp-001菌株的构建
出发菌酿酒酵母nk2-sq于sd-ura液体培养基(0.8%酵母选择培养基sd-ura-trp-his(北京泛基诺(功能基因组)科技有限公司),2%葡萄糖,0.005%his,0.01%trp)中过夜培养后制备感受态。然后,加入表9中转化用片段m1,m2,m3,m4和m5共5μg(摩尔比=1:1:1:1:1),混匀后转移至电转杯中,2.7kv电击5.7ms,加入1ml1msorbitol,30℃复苏1h,涂布于sd-ura-his培养基,30℃,培养36h以上。筛选培养基成分为:0.8%酵母选择培养基sd-ura-trp-his(北京泛基诺(功能基因组)科技有限公司),2%葡萄糖,0.01%trp。pcr鉴定出正确的阳性克隆,命名为菌株fpp-001。
三、ele-001和ele-002菌株的构建
出发菌酿酒酵母fpp-001于sd-ura-his液体培养基中过夜培养后制备感受态。然后,分别加入质粒prs313-leu2-ptef1-stpgmas-tcyc1和prs425-leu2-ptef1-stpgmas-tcyc1,混匀后转移至电转杯中,2.7kv电击5.7ms,加入1ml1msorbitol,30℃复苏1h,涂布于sd-ura-his-leu培养基,30℃,培养36h以上。筛选培养基成分为:0.8%酵母选择培养基sd-ura-trp-leu-his(北京泛基诺(功能基因组)科技有限公司),2%葡萄糖,0.01%trp。pcr鉴定出正确的阳性克隆,分别命名为菌株ele-001(转入质粒prs313-leu2-ptef1-stpgmas-tcyc1)和ele-002(转入质粒prs425-leu2-ptef1-stpgmas-tcyc1)。
四、ele-011菌株的构建
按照上述三中步骤制备fpp-001感受态。然后,加入质粒prs425-leu2-ptef1-synsmfps-gggs-stpgmas-tcyc1,混匀后转移至电转杯中,2.7kv电击5.7ms,加入1ml1msorbitol,30℃复苏1h,涂布于sd-ura-his-leu培养基30℃,培养36h以上。pcr鉴定出正确的阳性克隆,命名为菌株ele-011。
五、ele-012-ele-019菌株的构建
以prs425-leu2-ptef1-synsmfps-gggs-stpgmas-tcyc1质粒为模板,用表11的引物分別进行pcr扩增,得到不同引物对应的扩增产物;再分别将不同引物对应的扩增产物转入酵母fpp-001中进行自身同源重组,分别得到重组菌ele-012-ele-018,使该载体中的融合蛋白synsmfps-gggs-stpgmas之间的连接肽gggs分别替换成3a001、4a001、5a002、6a005、6b004、8a005、12a003(表10所示)。
以prs425-leu2-pmf1-synsmfps-gggs-stpgmas-tcyc1质粒为模板,用表10中连接肽为8a005的引物(表11)进行pcr扩增,得到不同引物对应的扩增产物;再分别将不同引物对应的扩增产物转入酵母fpp-001中进行自身同源重组,得到重组菌ele-019,使该载体中的融合蛋白synsmfps-gggs-stpgmas之间的连接肽gggs替换成8a005。
表10为连接肽核苷酸序列和氨基酸序列
具体反应条件如下:
上述扩增体系为:5×phusionhfbuffer10μl、dntp(10mmeachdntp)1μl、dna模板20ng、引物(表11所示)(10μm)各1.5μl、phusionhigh-fidelitydnapolymerase(2.5u/μl)0.5μl、补加蒸馏水至总体积50μl。扩增条件为:98℃预变性3分钟(1个循环);98℃变性10秒、58℃退火10秒、72℃延伸均用5.5分钟(30个循环);72℃延伸10分钟(1个循环)。
扩增产物经纯化后,利用fermentas公司的dpni酶进行消化处理,其体系为:5×fastdigestgreenbuffer4μl,纯化产物34μl,dpni2μl。酶切温度和反应时间分别为37℃和1h,最后割胶回收保存。
表11引物
按照上述三中步骤制备fpp-001感受态。然后,分别加入上步所得割胶回收产物,混匀后转移至电转杯中,2.7kv电击5.7ms,加入1ml1msorbitol,30℃复苏1h,分别涂布于sd-ura-his-leu培养基30℃,培养36h以上。pcr鉴定出正确的阳性克隆,分别命名为菌株ele-012-ele-019。
六、ele-020重组菌株的构建
1、ppgk1-erg20-gggs-lsltc2-tadh1,ptef1-synsmfps-gggs-stpgmas-tcyc1,rdna-trp1-up,和rdna-trp1-down的制备
ppgk1-erg20-gggs-lsltc2-tadh1和ptef1-synsmfps-gggs-stpgmas-tcyc1分别为携带有酵母法尼基焦磷酸合酶与莴苣来源吉玛烯a合酶的融合蛋白和密码子优化后的丹参来源的的法尼基焦磷酸合酶与密码子优化后的小白菊来源吉玛烯a合酶的融合蛋白表达盒;rdna-trp1-up和rdna-trp1-down分别为rdna的上游同源臂和下游同源臂;分别按照如下方法扩增片段:
分别用表12描述的pcr模板和引物进行pcr获得功能模块:
m1(rdna-trp1-up),
m2(ppgk1-erg20-gggs-lsltc2-tadh1),
m3(ptef1-synsmfps-gggs-stpgmas-tcyc1),
m4(rdna-trp1-down)。
扩增体系为:5×phusionhfbuffer10μl、dntp(10mmeachdntp)1μl、dna模板20ng、引物(10μm)各1.5μl、phusionhigh-fidelitydnapolymerase(2.5u/μl)0.5μl、补加蒸馏水至总体积50μl。扩增条件为:98℃预变性3分钟(1个循环);98℃变性10秒、58℃退火10秒、72℃延伸均用2分钟(30个循环);72℃延伸10分钟(1个循环),产物经割胶回收保存。
表12引物
出发菌酿酒酵母ele-019于sd-ura-his-leu液体培养基中过夜培养后制备感受态。然后,加入表12中转化用片段m1,m2,m3和m4共4μg(摩尔比=1:1:1:1),混匀后转移至电转杯中,2.7kv电击5.7ms,加入1ml1msorbitol,30℃复苏1h,涂布于sd-ura-his-leu-trp培养基,30℃,培养36h以上。筛选培养基成分为:0.8%酵母选择培养基sd-ura-his-leu-trp(北京泛基诺(功能基因组)科技有限公司),2%葡萄糖。pcr鉴定出正确的阳性克隆,命名为菌株ele-020。
该ele-020重组菌株于2017年10月20日保藏于中国微生物菌种保藏管理委员会普通微生物中心(cgmcc),保藏地址:北京市朝阳区北辰西路1号院3号,菌种名称:酿酒酵母,拉定名:saccharomycescerevisiae,保藏编号:cgmccno.14829。
上述所有的工程菌株信息如表13所示。
表13工程菌株信息
实施例3、重组菌在生产β-榄香烯中的应用
1、工程菌培养及产物提取
在相应固体选择培养基sd-ura-his-leu中活化实施例2制备的所有酵母工程菌株,于相应液体选择培养基sd-ura-his-leu中制备种子液(30℃,250rpm,16h),以1%的接种量接种于含15ml相应液体选择培养基的100ml三角瓶中,30℃,250rpm振荡培养1天,然后加入1.5ml正十二烷,继续震荡培养5天。最后,将三角瓶中液体转移至50ml离心管,5000rpm离心5min,收集有机相备用。
2、β-榄香烯转化及其定性定量分析
1)β-榄香烯转化
将上述有机相样品在通风厨内中经100-380℃(180℃)油浴加热1小时,得到转化后物质。
2)检测
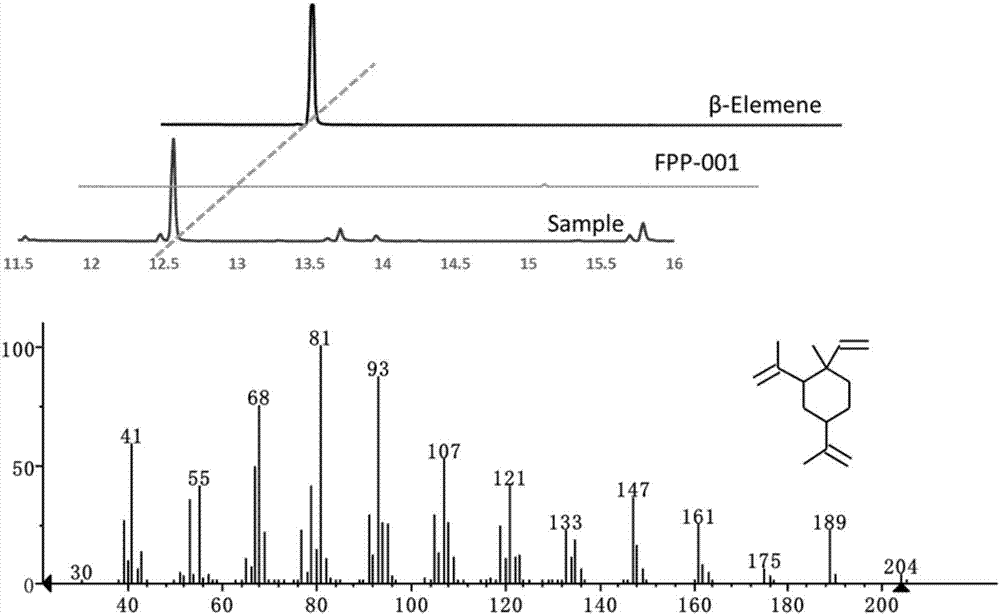
将转化后物质用正己烷稀释10倍,过有机尼龙膜(0.22μm),用gc-ms检测。检测仪器:安捷伦气质联用仪agilent7890a/5975c。gc-ms测定条件:进样口温度250℃,进样体积1μl,不分流,溶剂延时3min;色谱柱:hp-5ms(30m*0.25mm);色谱条件:45℃,1min,10℃/min到300℃保温5min;ms条件:fullscan:50-750amu。用β-榄香烯的标准品进行定性定量,β-榄香烯标准品在中国药品生物制品检定所购买(货号:100268)。图2为实施例2制备的所有酵母工程菌株生产β-榄香烯的gc-ms检测图。
结果各工程菌发酵6天时产量如下:
在fpp-001的基础上分别引入低、高拷贝数的stpgmas,得到工程菌株ele-001和ele-002。其中,ele-001的β-榄香烯产量达9.3mg/l,ele-002的β-榄香烯产量达22.1mg/l;
在fpp-001的基础上引入高拷贝数的融合蛋白基因synsmfps-gggs-stpgmas,得到工程菌株ele-011,其β-榄香烯的产量达101.1mg/l;
在fpp-001的基础上分别引入高拷贝数的融合蛋白基因synsmfps-linker-stpgmas,得到工程菌株ele-012-ele-019(启动子和linker分别为tef1和3a001、tef1和4a001、tef1和5a002、tef1和6a005、tef1和6b004、tef1和8a005、tef1和12a003、mf1和8a005)。
在ele-019的基础上重组导入融合蛋白基因ppgk1-erg20-gggs-lsltc2-tadh1和ptef1-synsmfps-gggs-stpgmas-tcyc1,得到工程菌株ele-020。
利用ele-012-ele-020菌株制备β-榄香烯的产量分别达2.2mg/l(相对于培养液)、35.5mg/l、110.4mg/l、108.6mg/l、73.6mg/l、109.7mg/l、48.3mg/l、158.1mg/l和469mg/l。
3、生物反应器发酵培养
1)培养基配置
氯化钙母液:19.2g/l二水氯化钙。
微量金属盐母液:19.1g/l乙二胺四乙酸二钠;10.2g/l七水硫酸锌;0.5g/l四水氯化锰;0.86g/l六水氯化钴;0.78g/l五水硫酸铜;0.56g/l二水钼酸钠;5.12g/l七水亚硫酸铁。
维他命母液:0.05g/l生物素;0.2g/l对氨基苯甲酸纳;1g/l烟酸;1g/l泛酸钙;1g/l盐酸吡哆醇;1g/l盐酸硫胺素;25g/l肌醇。
种子培养基和发酵培养基:25g/l葡萄糖,15g/l硫酸铵,6.15g/l七水硫酸镁,0.72g/l七水硫酸锌,8g/l磷酸二氢钾,2ml/l氯化钙母液,10ml/l微量金属盐母液;12ml/l维他命母液,1g/l色氨酸,其余为水。
补料培养基:800g/l葡萄糖,5.125g/l七水硫酸镁,3.5g/l硫酸钾,0.28g/l硫酸钠,9g/l磷酸二氢钾,1g/l色氨酸,其余为水。
2)工程菌ele-019发酵
按1方法活化工程菌ele-019。挑取平板上的单克隆至装有sd-ura-his-leu培养基的试管,30℃,250rpm振荡培养过夜;吸取500μl菌液至装有50mlsd-ura-his-leu培养基的250ml三角瓶中,30℃,250rpm振荡培养24h;
分别吸取2ml菌液至3个装有100ml种子培养基的1l三角瓶中,30℃,250rpm振荡培养48h;最后经火焰接种环,将种子液加入含3l发酵培养基的7l发酵罐(德国eppendorf公司,型号:
发酵过程中参数设定值分别为:温度30℃,ph5.0,溶氧30%,空气流量3-20l/min,搅拌转速300-1000rpm,溶氧与搅拌转速、通气级联。当溶氧值大于60%时,向发酵罐中加入补料培养基至发酵液中葡萄糖浓度为5g/l。
在发酵结束前3小时,添加10%(相对于培养液体积)的正十二烷,发酵结束后分离有机相。
按2中转化方法和检测方法处理,并进行定性定量分析,工程菌ele-019高密度发酵96小时后能得到2g/l(相对于培养液)β-榄香烯。
符合本发明目的的重组菌,包括但不限于表13中记载的具体实验例,均可按照“3”项下所述的发酵方法进行发酵培养,得到吉玛烯a。
序列表
<110>一种重组菌及其用途
<120>中国科学院天津工业生物技术研究所中国中医科学院中药研究所
<160>14
<170>patentinversion3.5
<210>1
<211>808
<212>dna
<213>人工序列
<220>
<223>
<400>1
gggaagacatgcttaacaagaagatataattatataatatatatattattaataataaca60
tccttactgcagtcctgttgtgggagaaaatggagagagactatgtttcgtatcaattcc120
taaaatcaaaaaaaaaaaaaaaaaaaagttaaacaagcactcgctgttcatttgttttac180
aagtattcatactctaataggtcattgagcttcttttcttgaggagagatccaatttgaa240
gtcggaataagatttgctttcattagcgtaggcaataattatgagataaatggtgcagca300
ctattaagtagtgtggatttcaataatttccgaattaggaataaatgcgctaaatagaca360
tcccgttctctttggtaatctgcataattctgatgcaatatccaacaactatttgtgcaa420
ttatttaacaaaatccaattaactttcctaattagtccttcaatagaacatctgtattcc480
ttttttttatgaacaccttcctaattaggccatcaacgacagtaaattttgccgaattta540
atagcttctactgaaaaacagtggaccatgtgaaaagatgcatctcatttatcaaacaca600
taatattcaagtgagccttacttcaattgtattgaagtgcaagaaaaccaaaaagcaaca660
acaggttttggataagtacatatataagagggccttttgttcccatcaaaaatgttactg720
ttcttacgattcatttacgattcaagaatagttcaaacaagaagattacaaactatcaat780
ttcatacacaatataaacgattaaaaga808
<210>2
<211>1071
<212>dna
<213>人工序列
<220>
<223>
<400>2
acctggtaaaacaatggctaatttgaatggtgaatctgctgatttgagagcaacattttt60
gggtgtttactctgttttgaagtcagaattgttgaatgatccagcatttgaatggacaga120
tggttcaagacaatgggttgaaagaatgttggattacaacgttccaggtggtaaattgaa180
cagaggtttgtctgttattgattcatacaaattgttgaagggtggtaaagatttgactga240
tgatgaagttttcttggcttctgcattaggttggtgtgttgaatggttacaagcatactt300
tttggttttggatgatatcatggataactcacatacaagaagaggtcaaccatgttggtt360
tagagttccaaaagttggtatgatcgcaattaatgatggtatcatcttgagaaatcatat420
tccaagaattttgaagaaacattttagaactaaaccatactacgttgatttgttggattt480
gtttaatgaagttgaattccaaacagcttctggtcaaatgatcgatttgatcactacaat540
cgaaggtgaaaaggatttgtctaagtactcattgccattgcatagaagaatcgttcaata600
caagactgcttattactcattttacttgccagttgcttgtgcattgttaatggcaggtga660
agatttggaaaaacatccaacagttaaggatgttttgattaatatgggtatctatttcca720
agttcaagatgattacttagattgttttggtgaaccagaaaagattggtaaaatcggtac780
tgatatcgaagatttcaagtgttcttggttggttgttaaagcattggaattgtgtaacga840
agaacaaaagaaaactttatttgaacattatggtaaagaagatccagctgatgttgcaaa900
gattaaagttttgtacaacgaaattaatttgcaaggtgttttcgcagaattcgaatctaa960
gtcatacgaaaaattgaattcttcaattgaagctcatccatctaagtcagttcaagcagt1020
tttgaaatcatttttgggtaaaatctataaaagacaaaaataaggcgcgcc1071
<210>3
<211>1701
<212>dna
<213>人工序列
<220>
<223>
<400>3
acctggtaaaacaatggcagcagtacaagcaaccacaggtattcaagcaaacacaaaaac60
ttccgcagaaccagtaagaccattagccaatttcccaccatccgtttggggtgacagatt120
tttatccttcagtttggacaagagtgaattcgaaagatacgctatcgcaatggaaaagcc180
aaaggaagatgttagaaagttaatcgttgattctactatggactcaaacgaaaaattggg240
tttgatctattccgttcatagagtcggtttgacatacatgttcttgcaagaaatagaatc300
ccaattggataaattgtttaatgaattcagtttgcaagattacgaagaagtagacttgta360
cactatctcaattaacttccaagttttcagacacttaggttacaaattgccttgtgatgt420
ttttaaaaagtttaaagacgctatatccggtacttttaaagaatccataaccagtgatgt480
tagaggcatgttgggtttgtacgaaagtgctcaattgagaattagaggtgaaaagatatt540
ggatgaagcatccgttttcattgagggtaaattgaagagtgttgtcaacacattggaggg600
taacttggcccaacaagtcaagcaatcattaagaagaccattccatcagggtatgcctat660
ggtagaagcaagattgtatttctctaactacgaagaagaatgctcttcacatgattcatt720
gtttaaattagcaaagttgcacttcaagtatttggaattgcaacaaaaggaagaattgag780
aatcgtcaccaagtggtacaaggatatgagattccaagaaactacaccatacatcagaga840
cagagttcctgaaatctacttatggattttgggtttgtacttcgaaccaagatactcttt900
ggctagaataatcgcaaccaagatcactttgttcttagtagttttggatgacacttatga960
tgcctacgctacaatcgaagaaatcagattgttgaccgatgctatgaataagtgggacat1020
ttctgcaatggaacaaatcccagaatacatcagacctttctacaaggttttgttggatga1080
atacgctgaaataggtaaaagaatggcaaaggaaggtagagccgatactgttatcgcctc1140
taaagaagcatttcaagacattgcaagaggttatttggaagaagccgaatggacaaactc1200
tggttatgttgcatcattcccagaatacatgaagaatggtttaatcacctcagcctataa1260
cgtcatttctaaatcagctttggtcggtatgggtgaaattgtatctgaagatgcattagc1320
ctggtacgaatcacacccaaagcctttgcaagcatctgaattgatcagtagattgcaaga1380
tgacgttatgacttaccaattcgaaagagaaagaggtcaatctgctaccggtgttgatgc1440
atacatcaagacttacggtgtctcagaaaagaaagcaatcgatgaattgaagatcatgat1500
cgaaaacgcctggaaggacattaacgaaggttgtttgaaaccaagacaagtttctatgga1560
tttgttagcccctatattgaatttggctagaatgatcgacgtcgtatatagatacgatga1620
cggttttacattcccaggttccacattgaaagaatacataaacttgttgttcgtagattc1680
cttgccagtctgaggcgcgcc1701
<210>4
<211>430
<212>dna
<213>人工序列
<220>
<223>
<400>4
agtgatcccccacacaccatagcttcaaaatgtttctactccttttttactcttccagat60
tttctcggactccgcgcatcgccgtaccacttcaaaacacccaagcacagcatactaaat120
ttcccctctttcttcctctagggtgtcgttaattacccgtactaaaggtttggaaaagaa180
aaaagagaccgcctcgtttctttttcttcgtcgaaaaaggcaataaaaatttttatcacg240
tttctttttcttgaaaatttttttttttgatttttttctctttcgatgacctcccattga300
tatttaagttaataaacggtcttcaatttctcaagtttcagtttcatttttcttgttcta360
ttacaactttttttacttcttgctcattagaaagaaagcatagcaatctaatctaagttt420
taattacaaa430
<210>5
<211>307
<212>dna
<213>人工序列
<220>
<223>
<400>5
ccgctgatcctagagggccgcatcatgtaattagttatgtcacgcttacattcacgccct60
ccccccacatccgctctaaccgaaaaggaaggagttagacaacctgaagtctaggtccct120
atttatttttttatagttatgttagtattaagaacgttatttatatttcaaatttttctt180
ttttttctgtacagacgcgtgtacgcatgtaacattatactgaaaaccttgcttgagaag240
gttttgggacgctcgaaggctttaatttgcaagctgcggccctgcattaatgaatcggcc300
aacgcgc307
<210>6
<211>1047
<212>dna
<213>人工序列
<220>
<223>
<400>6
atgtctattccagaaactcaaaaagccattatcttctacgaatccaacggcaagttggag60
cataaggatatcccagttccaaagccaaagcccaacgaattgttaatcaacgtcaagtac120
tctggtgtctgccacaccgatttgcacgcttggcatggtgactggccattgccaactaag180
ttaccattagttggtggtcacgaaggtgccggtgtcgttgtcggcatgggtgaaaacgtt240
aagggctggaagatcggtgactacgccggtatcaaatggttgaacggttcttgtatggcc300
tgtgaatactgtgaattgggtaacgaatccaactgtcctcacgctgacttgtctggttac360
acccacgacggttctttccaagaatacgctaccgctgacgctgttcaagccgctcacatt420
cctcaaggtactgacttggctgaagtcgcgccaatcttgtgtgctggtatcaccgtatac480
aaggctttgaagtctgccaacttgagagcaggccactgggcggccatttctggtgctgct540
ggtggtctaggttctttggctgttcaatatgctaaggcgatgggttacagagtcttaggt600
attgatggtggtccaggaaaggaagaattgtttacctcgctcggtggtgaagtattcatc660
gacttcaccaaagagaaggacattgttagcgcagtcgttaaggctaccaacggcggtgcc720
cacggtatcatcaatgtttccgtttccgaagccgctatcgaagcttctaccagatactgt780
agggcgaacggtactgttgtcttggttggtttgccagccggtgcaaagtgctcctctgat840
gtcttcaaccacgttgtcaagtctatctccattgtcggctcttacgtggggaacagagct900
gataccagagaagccttagatttctttgccagaggtctagtcaagtctccaataaaggta960
gttggcttatccagtttaccagaaatttacgaaaagatggagaagggccaaattgctggt1020
agatacgttgttgacacttctaaataa1047
<210>7
<211>1503
<212>dna
<213>人工序列
<220>
<223>
<400>7
atgactaagctacactttgacactgctgaaccagtcaagatcacacttccaaatggtttg60
acatacgagcaaccaaccggtctattcattaacaacaagtttatgaaagctcaagacggt120
aagacctatcccgtcgaagatccttccactgaaaacaccgtttgtgaggtctcttctgcc180
accactgaagatgttgaatatgctatcgaatgtgccgaccgtgctttccacgacactgaa240
tgggctacccaagacccaagagaaagaggccgtctactaagtaagttggctgacgaattg300
gaaagccaaattgacttggtttcttccattgaagctttggacaatggtaaaactttggcc360
ttagcccgtggggatgttaccattgcaatcaactgtctaagagatgctgctgcctatgcc420
gacaaagtcaacggtagaacaatcaacaccggtgacggctacatgaacttcaccacctta480
gagccaatcggtgtctgtggtcaaattattccatggaactttccaataatgatgttggct540
tggaagatcgccccagcattggccatgggtaacgtctgtatcttgaaacccgctgctgtc600
acacctttaaatgccctatactttgcttctttatgtaagaaggttggtattccagctggt660
gtcgtcaacatcgttccaggtcctggtagaactgttggtgctgctttgaccaacgaccca720
agaatcagaaagctggcttttaccggttctacagaagtcggtaagagtgttgctgtcgac780
tcttctgaatctaacttgaagaaaatcactttggaactaggtggtaagtccgcccatttg840
gtctttgacgatgctaacattaagaagactttaccaaatctagtaaacggtattttcaag900
aacgctggtcaaatttgttcctctggttctagaatttacgttcaagaaggtatttacgac960
gaactattggctgctttcaaggcttacttggaaaccgaaatcaaagttggtaatccattt1020
gacaaggctaacttccaaggtgctatcactaaccgtcaacaattcgacacaattatgaac1080
tacatcgatatcggtaagaaagaaggcgccaagatcttaactggtggcgaaaaagttggt1140
gacaagggttacttcatcagaccaaccgttttctacgatgttaatgaagacatgagaatt1200
gttaaggaagaaatttttggaccagttgtcactgtcgcaaagttcaagactttagaagaa1260
ggtgtcgaaatggctaacagctctgaattcggtctaggttctggtatcgaaacagaatct1320
ttgagcacaggtttgaaggtggccaagatgttgaaggccggtaccgtctggatcaacaca1380
tacaacgattttgactccagagttccattcggtggtgttaagcaatctggttacggtaga1440
gaaatgggtgaagaagtctaccatgcatacactgaagtaaaagctgtcagaattaagttg1500
taa1503
<210>8
<211>2142
<212>dna
<213>人工序列
<220>
<223>
<400>8
atgtcgccctctgccgtacaatcatcaaaactagaagaacagtcaagtgaaattgacaag60
ttgaaagcaaaaatgtcccagtctgccgccactgcgcagcagaagaaggaacatgagtat120
gaacatttgacttcggtcaagatcgtgccacaacggcccatctcagatagactgcagccc180
gcaattgctacccactattctccacacttggacgggttgcaggactatcagcgcttgcac240
aaggagtctattgaagaccctgctaagttcttcggttctaaagctacccaatttttaaac300
tggtctaagccattcgataaggtgttcatcccagaccctaaaacgggcaggccctccttc360
cagaacaatgcatggttcctcaacggccaattaaacgcctgttacaactgtgttgacaga420
catgccttgaagactcctaacaagaaagccattattttcgaaggtgacgagcctggccaa480
ggctattccattacctacaaggaactacttgaagaagtttgtcaagtggcacaagtgctg540
acttactctatgggcgttcgcaagggcgatactgttgccgtgtacatgcctatggtccca600
gaagcaatcataaccttgttggccatttcccgtatcggtgccattcactccgtagtcttt660
gccgggttttcttccaactccttgagagatcgtatcaacgatggggactctaaagttgtc720
atcactacagatgaatccaacagaggtggtaaagtcattgagactaaaagaattgttgat780
gacgcgctaagagagaccccaggcgtgagacacgtcttggtttatagaaagaccaacaat840
ccatctgttgctttccatgcccccagagatttggattgggcaacagaaaagaagaaatac900
aagacctactatccatgcacacccgttgattctgaggatccattattcttgttgtatacg960
tctggttctactggtgcccccaagggtgttcaacattctaccgcaggttacttgctggga1020
gctttgttgaccatgcgctacacttttgacactcaccaagaagacgttttcttcacagct1080
ggagacattggctggattacaggccacacttatgtggtttatggtcccttactatatggt1140
tgtgccactttggtctttgaagggactcctgcgtacccaaattactcccgttattgggat1200
attattgatgaacacaaagtcacccaattttatgttgcgccaactgctttgcgtttgttg1260
aaaagagctggtgattcctacatcgaaaatcattccttaaaatctttgcgttgcttgggt1320
tcggtcggtgagccaattgctgctgaagtttgggagtggtactctgaaaaaataggtaaa1380
aatgaaatccccattgtagacacctactggcaaacagaatctggttcgcatctggtcacc1440
ccgctggctggtggtgttacaccaatgaaaccgggttctgcctcattccccttcttcggt1500
attgatgcagttgttcttgaccctaacactggtgaagaacttaacaccagccacgcagag1560
ggtgtccttgccgtcaaagctgcatggccatcatttgcaagaactatttggaaaaatcat1620
gataggtatctagacacttatttgaacccttaccctggctactatttcactggtgatggt1680
gctgcaaaggataaggatggttatatctggattttgggtcgtgtagacgatgtggtgaac1740
gtctctggtcaccgtctgtctaccgctgaaattgaggctgctattatcgaagatccaatt1800
gtggccgagtgtgctgttgtcggattcaacgatgacttgactggtcaagcagttgctgca1860
tttgtggtgttgaaaaacaaatctagttggtccaccgcaacagatgatgaattacaagat1920
atcaagaagcatttggtctttactgttagaaaagacatcgggccatttgccgcaccaaaa1980
ttgatcattttagtggatgacttgcccaagacaagatccggcaaaattatgagacgtatt2040
ttaagaaaaatcctagcaggagaaagtgaccaactaggcgacgtttctacattgtcaaac2100
cctggcattgttagacatctaattgattcggtcaagttgtaa2142
<210>9
<211>1264
<212>dna
<213>人工序列
<220>
<223>
<400>9
atgagagtagcaaacgtaagtctaaaggttgttttatagtagttaggatgtagaaaatgt60
attccgataggccattttacatttggagggacggttgaaagtggacagaggaaaaggtgc120
ggaaatggctgattttgattgtttatgttttgtgtgatgattttacatttttgcatagta180
ttaggtagtcagatgaaagatgaatagacataggagtaagaaaacatagaatagttaccg240
ttattggtaggagtgtggtggggtggtatagtccgcattgggatgttactttcctgttat300
ggcatggatttccctttagggtctctgaagcgtatttccgtcaccgaaaaaggcagaaaa360
agggaaactgaagggaggatagtagtaaagtttgaatggtggtagtgtaatgtatgatat420
ccgttggttttggtttcggttgtgaaaagttttttggtatgatattttgcaagtagcata480
tatttcttgtgtgagaaaggtatattttgtatgttttgtatgttcccgcgcgtttccgta540
ttttccgcttccgcttccgcagtaaaaaatagtgaggaactgggttacccggggcacctg600
tcactttggaaaaaaaatatacgctaagatttttggagaatagcttaaattgaagttttt660
ctcggcgagaaatacgtagttaaggcagagcgacagagagggcaaaagaaaataaaagta720
agattttagtttgtaatgggagggggggtttagtcatggagtacaagtgtgaggaaaagt780
agttgggaggtacttcatgcgaaagcagttgaagacaagttcgaaaagagtttggaaacg840
aattcgagtaggcttgtcgttcgttatgtttttgtaaatggcctcgtcaaacggtggaga900
gagtcgctaggtgatcgtcagatctgcctagtctctatacagcgtgtttaattgacatgg960
gttgatgcgtattgagagatacaatttgggaagaaattcccagagtgtgtttcttttgcg1020
tttaacctgaacagtctcatcgtgggcatcttgcgattccattggtgagcagcgaaggat1080
ttggtggattactagctaatagcaatctatttcaaagaattcaaacttgggggaatgcct1140
tgttgaatagccggtcgcaagactgtgattcttcaagtgtaacctcctctcaaatcagcg1200
atatcaaacgtaccattccgtgaaacaccggggtatctgtttggtggaacctgattagag1260
gaaa1264
<210>10
<211>860
<212>dna
<213>人工序列
<220>
<223>
<400>10
tacaatcttgatccggagcttttctttttttgccgattaagaattaattcggtcgaaaaa60
agaaaaggagagggccaagagggagggcattggtgactattgagcacgtgagtatacgtg120
attaagcacacaaaggcagcttggagtatgtctgttattaatttcacaggtagttctggt180
ccattggtgaaagtttgcggcttgcagagcacagaggccgcagaatgtgctctagattcc240
gatgctgacttgctgggtattatatgtgtgcccaatagaaagagaacaattgacccggtt300
attgcaaggaaaatttcaagtcttgtaaaagcatataaaaatagttcaggcactccgaaa360
tacttggttggcgtgtttcgtaatcaacctaaggaggatgttttggctctggtcaatgat420
tacggcattgatatcgtccaactgcatggagatgagtcgtggcaagaataccaagagttc480
ctcggtttgccagttattaaaagactcgtatttccaaaagactgcaacatactactcagt540
gcagcttcacagaaacctcattcgtttattcccttgtttgattcagaagcaggtgggaca600
ggtgaacttttggattggaactcgatttctgactgggttggaaggcaagagagccccgaa660
agcttacattttatgttagctggtggactgacgccagaaaatgttggtgatgcgcttaga720
ttaaatggcgttattggtgttgatgtaagcggaggtgtggagacaaatggtgtaaaagac780
tctaacaaaatagcaaatttcgtcaaaaatgctaagaaataggttattactgagtagtat840
ttatttaagtattgtttgtg860
<210>11
<211>1068
<212>dna
<213>人工序列
<220>
<223>
<400>11
atggcttcagaaaaagaaattaggagagagagattcttgaacgttttccctaaattagta60
gaggaattgaacgcatcgcttttggcttacggtatgcctaaggaagcatgtgactggtat120
gcccactcattgaactacaacactccaggcggtaagctaaatagaggtttgtccgttgtg180
gacacgtatgctattctctccaacaagaccgttgaacaattggggcaagaagaatacgaa240
aaggttgccattctaggttggtgcattgagttgttgcaggcttacttcttggtcgccgat300
gatatgatggacaagtccattaccagaagaggccaaccatgttggtacaaggttcctgaa360
gttggggaaattgccatcaatgacgcattcatgttagaggctgctatctacaagcttttg420
aaatctcacttcagaaacgaaaaatactacatagatatcaccgaattgttccatgaggtc480
accttccaaaccgaattgggccaattgatggacttaatcactgcacctgaagacaaagtc540
gacttgagtaagttctccctaaagaagcactccttcatagttactttcaagactgcttac600
tattctttctacttgcctgtcgcattggccatgtacgttgccggtatcacggatgaaaag660
gatttgaaacaagccagagatgtcttgattccattgggtgaatacttccaaattcaagat720
gactacttagactgcttcggtaccccagaacagatcggtaagatcggtacagatatccaa780
gataacaaatgttcttgggtaatcaacaaggcattggaacttgcttccgcagaacaaaga840
aagactttagacgaaaattacggtaagaaggactcagtcgcagaagccaaatgcaaaaag900
attttcaatgacttgaaaattgaacagctataccacgaatatgaagagtctattgccaag960
gatttgaaggccaaaatttctcaggtcgatgagtctcgtggcttcaaagctgatgtctta1020
actgcgttcttgaacaaagtttacaagagaagcaaaggtggtggttct1068
<210>12
<211>1686
<212>dna
<213>人工序列
<220>
<223>
<400>12
ggtggtggttctatggcagcagttgacactaatgccaccatccaagaaaagaccaccgca60
gagccggtgcgtcctttagccaacttccctccttcggtatggggtgatcgcttcctatca120
ttcactcttgacaattcggaactcgaaggatatgcaaaagccatggaagccccaaaagaa180
gaattgagaagattgattgtagatcaaacaatggattcaaataagaaactaagtttgatt240
tattccgtccaccgtcttggtttgacatatctgttcttgcaagagattgaagcccagcta300
gacaaaattttcaaagagttcaacttgcaaaattatgatgaagttgatctttacacaact360
tctatcaactttcaagttttccgacaccttggttataaactaccttgtgatgtgtttaac420
aaattcaaagacaatacctccggcgctttcaaggaagatatttctacggatgtgaagggc480
atgctaggcttatacgaatcttcacaactaagaacaagaggagaatcgatactagatgag540
gcttcatcgttcactgaaactaaactcaagagtgtagtaaacaatcttgaaggaaatctt600
gcacaacaggtgttacaatcattgaggagaccatttcatcaagggatgccaatggtggag660
gcaaggctatatttctccaactatagtgaagagtgtgccacacatgagtgtttattaaag720
cttgcaaagctgcatttcagctatttggagctacagcaaaaggaagaacttcgcattgtc780
tcaaagtggtggaaagatatgagattccaggaaactacaccttatataagggatagagta840
ccagagatttacttatggattttgggattgtactttgagcctcgttactccttggcacga900
atcatcgccacaaaaattacattgtttcttgtggtgctagatgatacatatgacgcttac960
gctaccattgaagaaattcgacttttaactgatgccataaataggtgggacatgagtgct1020
atggagcaaattccggaatacattagaccattctacaaaattctcctagatgagtatgct1080
gagcttgagaaacaactagctatagaaggaagagcaaagagcgttattgcttcaaaagaa1140
gcgttccaagacattgctagaggctaccttgaagaagccgagtggacaaacagtggatat1200
gtggcatcatttcctgagtacatgaagaatgggttaatcacttcagcctacaatgttatt1260
tcgaaatctgctttagtgggtatgggcgacatagttagtgaaaatgcattggcatggtac1320
gaaagtcatccaaagactctacaagcttccgagttaatctcaagactccaagatgatgtc1380
atgacttaccagtttgagcgtgaaagaggacaatcagccactggagttgatgcgtatatc1440
aagacatacggcgtgtcagagaaggaagctattgatgagctaaagataatgattgaaaat1500
gcatggaaagatataaacgagggatgtctcaagccaagagaagtctcgatggatttgctc1560
gcgccaattcttaaccttgcaagaatgatagatgttgtgtatcgatatgatgatgggttc1620
acctttcctggaaaaaccatgaaagagtatattactcttttatttgttggttctgtatcc1680
atgtaa1686
<210>13
<211>1252
<212>dna
<213>人工序列
<220>
<223>
<400>13
gttcgaccatattgatgaagagtgggtaggttataaaagaaattattttaccttagtatc60
aacgtttgaaacggcaaattgtgatttggatacttttttaaagagcagttttgatcttct120
cgttgaagactcttcagtagaaagcagattaagagtgcaatatttcgctatcaaaataaa180
agctaagaatgacgacgacgacacggaaatcaatctcgtccagcatacagcgaaacgcga240
caaaggtcctcaattttgtccttcagtatgtccgttggtgccttcccctttgccaaaaca300
tcaaatcataagagaagcttcaaatgttcgaaatatcactaaaatgaaaaaatacgattc360
cactttttatttgcacagagaccacgttaattatgaagaatatggagtggactctttatt420
gttttcctatccagaagattctattcagaaagttgcccgttatgaaagagttcaatttgc480
ttcatcaattagcgtgaagaaaccatcccaacaaaataaacactttagcttgcatgtaat540
tttaggtgcagtggtagatccagatacctttcatggggagaatcccggaattccttatga600
tgaactggctttaaaaaatggatcaaaagggatgtttgtgtatttgcaagaaatgaaaac660
gcctcctcttattattagaggaagatcaccttctaactatgcgtcatctcagcgaataac720
tgtgagaacaccgtcgagtgtcaattcctcacaaaacagcacaaaaagaaaaatgccatc780
aatggcgcagccgttaaatgaaagttgcttaaatgcaagaccttcgaaaaggcgatccaa840
agtggcgctaggtgcaccgaactctggggcctccatctcgcctatcaaatctcgtcaatc900
cacaccaatggaagcttcgaaggaaaatgaggatccgttcttcaggccaaataaaagggt960
ggagactcttgaacatatccagaacaaactgggtgctttgaaaaatcaatgtccagattc1020
ctctctgaaatatccgagttcatcttcaagaggtatggaagggtgtttagaaaaggagga1080
tttagtttactcaagtagtttttctgttaatatgaagcaaatcgaactgaaaccggcacg1140
ctcttttgaacatgagaatattttcaaagtaggctcattagcattcaaaaaaatcaatga1200
attacctcatgaaaattatgatataacaatagagaagaaatcaatggaacag1252
<210>14
<211>1168
<212>dna
<213>人工序列
<220>
<223>
<400>14
tcgcgcgtttcggtgatgacggtgaaaacctctgacacatgcagctcccggagacggtca60
cagcttgtctgtaagcggatgccgggagcagacaagcccgtcagggcgcgtcagcgggtg120
ttggcgggtgtcggggctggcttaactatgcggcatcagagcagattgtactgagagtgc180
accataattccgttttaagagcttggtgagcgctaggagtcactgccaggtatcgtttga240
acacggcattagtcagggaagtcataacacagtcctttcccgcaattttctttttctatt300
actcttggcctcctctagtacactctatatttttttatgcctcggtaatgattttcattt360
ttttttttccacctagcggatgactctttttttttcttagcgattggcattatcacataa420
tgaattatacattatataaagtaatgtgatttcttcgaagaatatactaaaaaatgagca480
ggcaagataaacgaaggcaaagatgacagagcagaaagccctagtaaagcgtattacaaa540
tgaaaccaagattcagattgcgatctctttaaagggtggtcccctagcgatagagcactc600
gatcttcccagaaaaagaggcagaagcagtagcagaacaggccacacaatcgcaagtgat660
taacgtccacacaggtatagggtttctggaccatatgatacatgctctggccaagcattc720
cggctggtcgctaatcgttgagtgcattggtgacttacacatagacgaccatcacaccac780
tgaagactgcgggattgctctcggtcaagcttttaaagaggccctactggcgcgtggagt840
aaaaaggtttggatcaggatttgcgcctttggatgaggcactttccagagcggtggtaga900
tctttcgaacaggccgtacgcagttgtcgaacttggtttgcaaagggagaaagtaggaga960
tctctcttgcgagatgatcccgcattttcttgaaagctttgcagaggctagcagaattac1020
cctccacgttgattgtctgcgaggcaagaatgatcatcaccgtagtgagagtgcgttcaa1080
ggctcttgcggttgccataagagaagccacctcgcccaatggtaccaacgatgttccctc1140
caccaaaggtgttcttatgtagtgacac1168
- 还没有人留言评论。精彩留言会获得点赞!