一种调控水稻理想株型的基因OsVAS1的制作方法
本发明属于植物基因工程技术领域。具体涉及一种调控水稻理想株型的基因osvas1。本发明采用候选基因筛选的方法,克隆到同时参与水稻生长素和乙烯合成途径基因osvas1,发现用crispr技术定点突变osvas1基因,可以改良水稻株型并增加产量,并证实了该基因的功能及应用途径。
背景技术:
水稻理想株型是根据生产实际的需要在上世纪80年代由国际水稻所提出,相关领域成果就不断产出,受到广泛关注。比如osipa1,osnpt1等(jiaoetal.,2010;miuraetal.,2010;wangetal.,2017);理想株型主要包括茎的粗壮度,分蘖数,穗型等农艺性状。理想株型有助于水稻产量和水稻抗倒伏等性状的提升,对水稻粮食安全具有重要意义。
生长素是最早研究开发的激素之一,对植物的生长发育具有重要作用,近年来对生长素的研究越来越深入。生长素合成途径在拟南芥中研究较深,现已知生长素主要由色氨酸依赖途径中的ipa合成途径生成,即由taa酶催化色氨酸生成吲哚丙酮酸,再由yuc酶催化吲哚丙酮酸生成生长素(zhao,2001;chengetal.,2006;chengetal.,2007;mashiguchietal.,2011;wonetal.,2011;zhao,2012)。在2013年,zheng等在拟南芥中通过遗传筛选得到了一个能互补sav的突变体,命名为vas1,vas1同时参与生长素与乙烯的合称途径,直接证明了两种激素之间的紧密联系(zhengetal.,2013)。但是生长素研究在水稻中并不深入,尤其关于生长素与理想株型之间的关系的报道则更为少见。
本发明通过在水稻中分离筛选候选基因,分离并克隆了水稻中的osvas1基因,并利用crispr技术对该基因进行定点突变,后续再用超量表达等手段验证了该基因的功能,该基因突变之后,株型变化显著,产量提升明显,并且还未见报道,可以作为潜在的育种工具。
技术实现要素:
本发明的目的涉及一种调控水稻理想株型的基因osvas1。本发明的osvas1基因利用crispr技术定点突变以后能显著提高产量,减少分蘖数,茎杆强壮度等农艺性状。超量表达该基因则会显著增加籽粒大小,分蘖数增加,叶夹角增大等;其中,所述的含有osvas1基因的核苷酸序列如序列表seqidno:1所示,序列长度为5835bp,它对应的第一个转录本氨基酸序列如seqidno:2所示,其对应的氨基酸序列为394个。它的第二个转录本氨基酸序列如seqidno:3所示,其对应的氨基酸序列为208个。
携带有本发明osvas1基因的超表达载体可通过使用ti质粒使用农杆菌介导进行dna转化,也可以通过微注射、电穿孔等常规生物技术方法导入植物细胞。
可使用包括本发明的osvas1基因的表达载体转化宿主,其中宿主可以是包括水稻在内的多种植物,培育理想株型植物新材料。
下面结合附图和实施例对本发明做进一步的说明。
附图说明
序列表seqidno:1是本发明分离克隆的包含有osvas11基因开放阅读框(openreadingframe)的核苷酸序列,序列长度为5835bp。
序列表seqidno:2是osvas1基因的第一个转录本的核苷酸序列(长度为1185bp),也是该转录本的编码的氨基酸序列(1-1185bp),编码394个氨基酸。
序列表seqidno:3是osvas1基因的第一个转录本编码的蛋白质的序列,编码394个蛋白质。
序列表seqidno:4是osvas1基因的第二个转录本的核苷酸序列(长度为627bp),也是该转录本的编码的氨基酸序列(1-627bp),编码208个氨基酸。
序列表seqidno:5是osvas1基因的第二个转录本编码的蛋白质的序列,编码208个蛋白质。
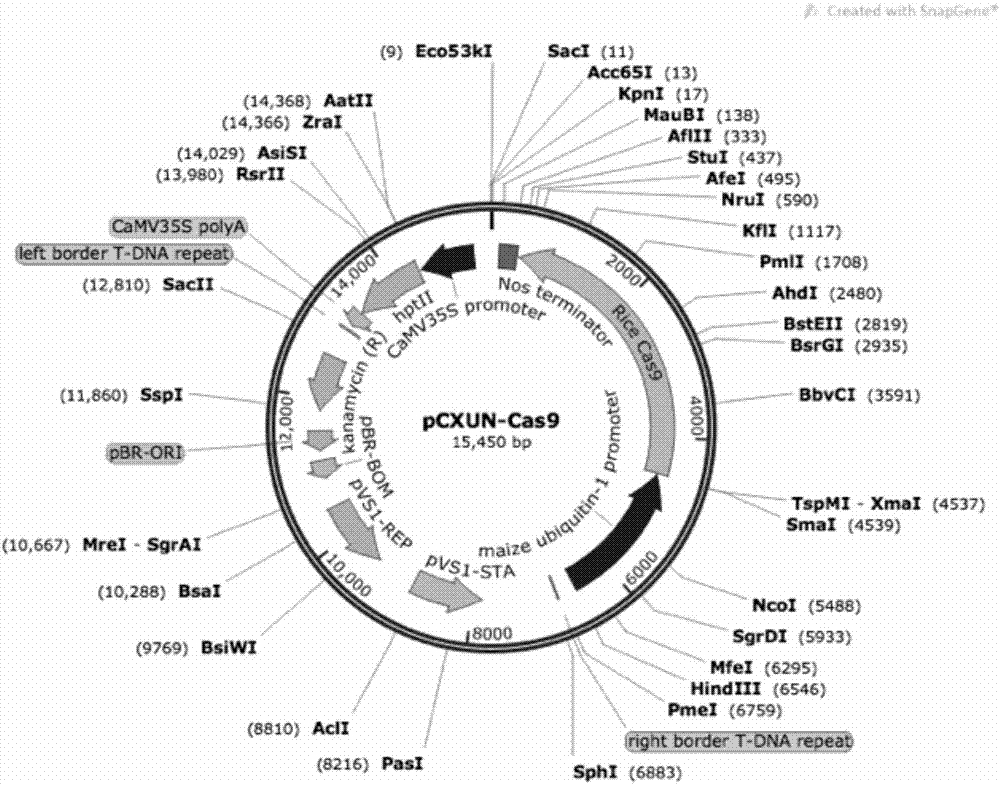
图1.crispr定点突变pcxun-cas9载体的图谱。附图标记说明:在ricecas9元件后的nosterminator之后的多克隆酶切位点引入sgrna可以达到基因编辑效应。本发明在kpni处引入。
图2.osvas1基因结构图以及定点突变靶位点位置。附图标记说明:图中上下两个图为该基因的两个不同的转录本。其中图2中的a图对应序列表序列表seqidno:2的转录本。图2中的b图对应序列表seqidno:3的转录本.红色箭头表示设计的crispr靶位点所在的位置。黑色实心部分为编码区外显子,白色空心部分为非编码区,实线部分为非编码内含子区。
图3.vcr-1和vcr-2突变体的植株表型。附图标记说明:图3中的a图表示vcr-1、vcr-2突变体与野生型zh11的整体植株表型对照;图3中的b图表示vcr突变体与野生型zh11的主茎的粗细对比;图3中的c图和d图分别表示株高和分蘖数的统计结果,n=15;e,f表示vcr突变体与野生型zh11的的一次枝梗数对比和统计结果,n=15。
图4.osvas1超量表达t0代的基因相对表达量。附图标记说明:纵坐标为相对倍数。
图5.osvas1超量表达阳性植株的表型。图5中的a有表示voe超表达家系与野生型zh11的整体植株表型对照;图5中的b图表示voe超表达与野生型水稻中花11(zh11)的主茎的粗细对比;图5中的c图和d图)分别表示株高和分蘖数的统计结果,n=15。
图6.用于osvas1基因超量表达的载体1301u的图谱。
具体实施方式
实施例1
1、osvas1候选基因的确定以及该基因定点突变载体的构建和转化
申请人为了研究水稻中生长素的合成机制,将拟南芥中相关同源基因atvas1的氨基酸序列在水稻数据库ricegenomeannotationproject中做了同源性比对分析,发现loc_os01g08270的氨基酸序列与其同源性最高,达到66.5%。确定该基因为水稻中的osvas1基因。
随后我们利用crispr-p网站(http://crispr.hzau.edu.cn/cgi-bin/crispr2/crispr)设计该基因crispr定点突变的靶位点,将该基因序列或者id输入网站提示的位置即可得到候选靶位点序列,最终我们选择靶位点序列如下:agatgcaggaattgcttcgaggg,其中下划线部分为pam(protospaceradjacentmotif)序列,该靶位点位于seqidno:1序列的615bp-636bp,可以同时对该基因的两个转录本进行定点突变,见图2。crispr载体的构建使用报道的pcxun-cas9载体(载体结构见图1),该载体已申请专利,申请号为2016106398543。该基因定点突变载体的构建以及转化水稻具体实施步骤如下:
1)pcxun-cas9载体(载体结构见图1)的构建
扩增水稻cas9基因,以克隆到ph-ubi-cas9-7的质粒(miaoetal.,2013)为模板,以cas9-f(5’-atggccccaaagaagaagcg-3’和cas9-r(5’-tcaatcgccgccgagttgtg-3’)为引物进行pcr扩增pcr扩增,获得了约4.1kb的cas9基因片段,将所获得的pcr产物克隆到pcxun载体上(chenetal.,2009),将阳性克隆对应样品测序(北京擎科生物科技有限公司测序),以验证目的pcr产物的正确性。
pcr过程如下:
cas9目的片段经凝胶电泳并挖胶回收(使用qiagen公司的回收柱)后用于载体pcxun-cas9的构建。
然后将质粒pcxun用xcmi酶酶切,形成t载体,反应体系如下:
37℃反应3个小时,然后挖胶、过柱、回收。
再将步骤1)中的cas9片段经t4连接酶16℃反应过夜连入xcmi酶酶切过的pcxun载体形成中间载体pcxun-cas9.
2)以携带u3启动子的质粒为模板,设计u3启动子前后接头引物(用kpni限制性内切酶消化载体,大写字母部分为载体末端序列,用于gibsonassembly一步法(gibsonetal.,2009)连接),引物序列如下:
osu3-f:cccctttcgccaggggtaccgtaattcatccaggtctccaag,
osu3-r:tacgaattcgagctcggtaccgctgtgccgtacgacggtacg;
3)将设计的特异性靶位点序列通过重叠pcr方法引入u3启动子中,据此设计的osvas1靶序列sgrna引物,序列如下:
u3osvas1cr-f:agatgcaggaattgcttcgagttttagagctagaaatagcaagtta
其中下划线部分为设计的osvas1的定点突变靶位点序列,斜体序列部分为靶位点互补配对序列。
重叠pcr程序如下:
pcr1反应,反应体系如下:
同时我们进行pcr2反应,如下:
4)以pcr1和pcr2产物为模板,进行pcr3反应,如下:
挖胶并利用qiagen试剂盒回收pcr3产物,并用gibsonassembly一步法(gibsonetal.,2009)将纯化过的pcr3产物和kpni切过的载体pcxun-cas9连接,50℃反应一小时,取1μl电转(1800v),并涂布在含卡那霉素(50μg/ml)抗性的lb培养基上。挑取单克隆菌落,抽提质粒并进行阳性检测,将质粒dna送测序公司测序核实。
将测序后证明序列正确的阳性质粒pcxun-cas9-sgrna(osvas1-cr)电转化到农杆菌(eha105)中,并侵染水稻愈伤组织。转化品种为水稻中花11(zh11,来自中国农业科学院作物科学研究所),具体转化步骤如下:
1)将水稻品种中花11的成熟胚去壳,用70%乙醇泡1min,0.15%升汞消毒20min,无菌水洗3~4次;将所得的外植体接种到诱导培养基上,于26℃暗培养诱导愈伤组织;
2)诱导培养35d后,取活力强、颗粒状的愈伤组织转入继代培养基进行继代培养;
3)取继代培养20d的愈伤组织的颗粒,接入预培养培养基上,于26℃下暗培养4d;
4)在预培养的第三天,用la(lb中加入1.5%琼脂)划线接种农杆菌菌株,28℃静止培养2d;之后,将农杆菌全部刮入悬浮培养基;于28℃,200r/m振荡培养0.5-1h;在分光光度计600nm波长光测定菌液浓度,调至od值=1.0;
5)将预培养后的愈伤组织接入100ml锥形瓶(大约到40ml处),加入调制好的农杆菌菌液,浸泡30min,期间摇动数次。配制悬浮培养基:(500μl乙酰丁香酮as(acetosringone)+5ml50%葡萄糖;
6)倒去菌液,将愈伤组织置于灭菌滤纸上吸干表面菌液(一定要吸干菌液,使愈伤组织的表面发白),但不能直接用电吹风吹干),接入共培养培养基(250μl乙酰丁香酮as+5ml50%葡萄糖),暗培养3d,再转入250ml共培养培养基上进行共培养;
7)将共培养后的愈伤组织用无菌水先快速摇动清洗两次;然后加入无菌水浸泡10min,使愈伤组织内部的菌体游离出来;倒去洗液,再加入含400mg/l的羧苄青霉素(cn)无菌水浸泡15min;倒干洗液,将愈伤组织置于灭菌滤纸上吸干,接入筛选培养基;26℃暗培养。每3周继代一次,共继代两次。每转化一个质粒,需灭菌单蒸水1~2瓶,其中第一次筛选时,在300ml筛选培养基中加入500ul羧苄青霉素(cn)和300ul潮霉素(hn);第二次继代筛选时,在筛选培养基中加入400ul羧苄青霉素(简称cn)和300ul潮霉素(简称hn)。
8)将筛选培养基培养的抗性愈伤组织接入预分化培养基,26℃暗培养一周。
9)将预分化培养一周的抗性愈伤组织转入分化培养基(50ml/瓶;改用三角瓶或平底试管做培养瓶);25℃,2000lux光照培养,通过再生获得转基因植株。
10)待小植株3~5cm;转入生根培养基上促进生根。
11)将根系健壮的植株移入盆钵,凉棚过渡3~5d;然后移到自然条件下生长,直至成熟。
以上各种培养基的具体配方及其配制方法见说明书的末尾的附录。
2、osvas1定点突变体的鉴定
具体步骤如下:
1)取成熟的水稻叶片,并经ctab法提取dna样品;
设计检测转基因植物的阳性引物的序列如下:
cas9check-f:gcatgaagaggatcgaggag,
cas9check-r:gatctcttgctcggacttgg;
用该引物进行pcr扩增,条带大小在750bp的即为阳性。随后挑取鉴定为阳性的植株进行测序,设计测序引物的序列如下所述:
osvas1cr-seqf:tgaccctaacatcgtatccaac,
osvas1cr-seqr:ttgacccaaacagtaaaggaac;
挑取测序为纯合的两个家系继续在田间种植,命名为vcr-1,vcr-2(见图2),其突变形式分别为:
参考序列:tgtgctcttttttcagatgcaggaattgcttcgagggaacaaggatgtga
野生型:ttttcagatgcaggaattgcttcgagggaacaaggat
vcr–1:ttttcagatgcaggaattgc--cgagggaacaaggat(2bp缺失)
野生型:cttttttcagatgcaggaattgcttcgagggaacaaggatgtgatgtcgct。
野生型:cttttttcagatgcaggaattgcttcgagggaacaaggatgtgatgtcgct
vcr–2:cttttttcagatgcaggaat------gtgatgtcgcttgcg(20bp缺失)
说明:上述引物序列的下划线部分为本发明设计的crispr靶位点序列。
2017年在湖北武汉的田间水稻自然生长发育整个阶段,申请人对突变体植株表型进行了考察,发现相比于野生型,突变体植株表现为理想株型所需要的农艺性状,具体表现为,分蘖数变少(见图3中的a图,d图);株高略有增加(见图3中a图,c图);主茎变粗(见图3中的b图);一次枝梗数增加(见图3中的e图,f图)。本发明的突变体具体表型图见图3所示。说明本发明克隆的基因功能缺失之后,符合理想株型的型态建成,有助于为后续育种改良提供有益的遗传资源。
3、osvas1基因的功能鉴定
基于该基因定点突变以后的表型,接下来通过构建超量表达载体来进一步研究该基因功能。具体实施步骤如下:
根据使用的超量表达载体1301u(载体图谱见图6),首先设计超表达载体osvas1的引物:
其中上述引物的下划线部分为kpni消化1301u产生的载体上的粘性末端重复序列,斜体部分为该基因转录区间上的序列。本发明取水稻品种中花11的水稻叶片立即存放于液氮中,按常规方法抽提水稻叶片的总rna,进行反转录。以反转录产物cdna为模板进行扩增,得到包含最长阅读框的osvas1全长序列。然后利用qiagen试剂盒纯化回收pcr产物和kpni消化酶切过的1301u载体,并用gibsonassembly一步法(gibsonetal.,2009)将这两者连接,电转然后挑单克隆,最后得到阳性质粒并转化水稻,后续的转化详细制备步骤参见本实施例1。
随后将得到的t0代植株,按常规方法提取t0代植株总dna并用gus引物进行阳性检测,gus引物序列如下:
gus-f:ccaggcagttttaacgatcagttcgc,
gus-r:gagtgaagatccctttcttgttacc;
抽提总rna并反转录来检测该基因表达量,设计rt引物如下:
vas1-rtf:gaattgctcatggaagcgatgtctcc
vas1-rtr:ctgtaccattccctcagtcacaagttctt
t0代植株相对表达量见图4所示;最终选取两个voe-2,voe-3,voe-4三个阳性家系进行后续的实验。通过对这两个家系在海南和武汉的连续表型考察,这两个家系植株相比于野生型主要表现为分蘖数增加(图5a,d),茎杆变细(图5b),植株变矮(图5a,c)。证实了该基因参与水稻各个时期的生长发育过程。
附录:本发明涉及的各种组培培养基配方
诱导培养基
继代培养基
预培养基
共培养培养基
悬浮培养基
筛选培养基
分化培养基
生根培养基
主要参考文献
cheng,y.,dai,x.,andzhao,y.(2006).auxinbiosynthesisbytheyuccaflavinmonooxygenasescontrolstheformationoffloralorgansandvasculartissuesinarabidopsis.genesdev.20:1790–1799.
cheng,y.,dai,x.,andzhao,y.(2007).auxinsynthesizedbytheyuccaflavinmonooxygenasesisessentialforembryogenesisandleafformationinarabidopsis.plantcell19:2430–2439.
gibson,d.g.,young,l.,chuang,r.-y.,venter,j.c.,hutchison,c.a.,andsmith,h.o.(2009).enzymaticassemblyofdnamoleculesuptoseveralhundredkilobases.nat.methods6:343–345.
jiao,y.,wang,y.,xue,d.,wang,j.,yan,m.,liu,g.,dong,g.,zeng,d.,lu,z.,zhu,x.,etal.(2010).regulationofosspl14byosmir156definesidealplantarchitectureinrice.nat.genet.42:541–544.
jinmiao,dongshuguo,jinzhezhang,qingpeihuang,genjiqin,xinzhang,jianminwan,hongyaguandli-jiaqu,targetedmutagenesisinriceusingcrispr-cassystem.cellresearch,2013,23:1233-1236
mashiguchi,k.,tanaka,k.,sakai,t.,sugawara,s.,kawaide,h.,natsume,m.,hanada,a.,yaeno,t.,shirasu,k.,yao,h.,etal.(2011).themainauxinbiosynthesispathwayinarabidopsis.proc.natl.acad.sci.u.s.a.108:18512–18517.
miura,k.,ikeda,m.,matsubara,a.,song,x.-j.,ito,m.,asano,k.,matsuoka,m.,kitano,h.,andashikari,m.(2010).osspl14promotespaniclebranchingandhighergrainproductivityinrice.nat.genet.42:545–549.
songbiaochen,pattaviphasongkumarn,jianliliu,andguo-liangwang,aversatilezerobackgroundt-vectorsystemforgenecloningandfunctionalgenomics.plantphysiology,2009,150:1111-1121
wang,s.,wu,k.,qian,q.,liu,q.,li,q.,pan,y.,ye,y.,liu,x.,wang,j.,zhang,j.,etal.(2017).non-canonicalregulationofspltranscriptionfactorsbyahumanotub1-likedeubiquitinasedefinesanewplanttypericeassociatedwithhighergrainyield.cellres.27:1142–1156.
won,c.,shen,x.,mashiguchi,k.,zheng,z.,dai,x.,cheng,y.,kasahara,h.,kamiya,y.,chory,j.,and
zhao,y.(2011).conversionoftryptophantoindole-3-aceticacidbytryptophanaminotransferasesofarabidopsisandyuccasinarabidopsis.proc.natl.acad.sci.u.s.a.108:18518–18523.
zhao,y.(2001).aroleforflavinmonooxygenase-likeenzymesinauxinbiosynthesis.science291:306–309.
zhao,y.(2012).auxinbiosynthesis:asimpletwo-steppathwayconvertstryptophantoindole-3-aceticacidinplants.mol.plant5:334–338.
zheng,z.,guo,y.,novák,o.,dai,x.,zhao,y.,ljung,k.,noel,j.p.,andchory,j.(2013).coordinationofauxinandethylenebiosynthesisbytheaminotransferasevas1.nat.chem.biol.9:244–246。
序列表
<110>华中农业大学
<120>一种调控水稻理想株型的基因osvas1
<141>2017-12-01
<160>5
<170>siposequencelisting1.0
<210>1
<211>5835
<212>dna
<213>水稻(oryzasativa)
<220>
<221>gene
<222>(1)..(5835)
<400>1
gtctcatgcttatcctccaacttcgcggcgcaccctccgcaaacgtaagccaaaaagatc60
aggaaaagagacagaaaaatcggaataaaagagtgatagatagatagatagatactccgt120
agatactagatcagcagcgatttttttttcagtttttgttttttcatttcttcctctgtc180
ttgtttagatgttggggttggtgagcccttggggggaatcagttggtgactgactcgaga240
agttcagaggttgttgtctccgaattctttaaccccaacctctcctcttctcttctcttc300
tctcctcttcttccttatactgcttcttgggctcgccgctgcgctgttctttggtcttct360
tggatctctgaccctaacatcgtatccaacttctccagttcttgttcttggaggggtgag420
gaagaggaggaggaggaggtgagatgggtagcttcgggaggctggcgaggagggccgtgg480
agacggaagcgccggtcatggtcaaggtgatggagaagaagctccctgcctcgtcgaaat540
cgcatctttcccttctgatttctttggtcttgattgatgagttctatctctcttgtgcct600
tgtgctcttttttcagatgcaggaattgcttcgagggaacaaggatgtgatgtcgcttgc660
gcaggtatcggatcgcaatttctttcggttatctgtccaatgattgtgtgtgtgtgtgtg720
tgttttcatgagagtaccaatgagtttgtgttgttgttggtacttggtcacaggcatttt780
gatgtttgcatttgattgggaagagtgttcgaaatggattagtgaaatgatcatttgtat840
tgttcctttactgtttgggtcaattgtctcagtaaactgttcacgttgtaaggtgaatca900
ggagagtttagtacatctgataacatattgtgctttgtataacacaactatttatcaagc960
atggaatttggaaactaaaatggttgtattcctgcttagttcctgaaaatggcgtttaac1020
tgcttatgctagccaaataagatatccaaatatatatattagtggcagtggtgtgtgtgc1080
tggcctcctattactgccaacagaacatgtgatatttactggaaattatttgctttcttt1140
taaactaatgtaaatcttggatgcagggagttgtttattggcagcctcctgaggcagcta1200
tgaataagattaaagaaattgtatgggaaccatcaatcagtaaatatggctctgatgatg1260
gtcttcctgaactccgagaagcacttctcgagaaggtaatttttcaattgggatttgtta1320
gataatggtttccatggcccatgttatcaagccttcccagttatcacaaagcaattacca1380
tgtgagacaaatgcacttggttgcctactgctaagcctttatcatgtgagacaaaggaac1440
ctgtgtgcctactactaagcctttgtcatgtgagaaattgattctggaattttttttacc1500
tcgatactgagagagtgagagtaaggccattcccaacccaaaacattagacatagtttcc1560
ataaactccacatcatcaagaaataagtactagacactactcttccaatgcaaacactac1620
tatttcatacttaaatttaatgctacttatctcacataatgtcttggatgttgtgtagaa1680
accatgtctcatgcaagacatagtttcctcctctttcctcatttattcacttgccacatc1740
agttttcatcctatgtgacagcttatttaatgctatggacactatcctagtcattgggtt1800
gggaatagcctaatatcaggaatcacatgctatccttacaaaattttatcaacgagaaag1860
tttcagaaagtatctctcagcttctgtgggggcttgggggggagggaagctaagggcccc1920
tttgaatcgtaggattgagaaaacatagtaatagaaaaaacgtaggattttgataggaat1980
gtaagtgtaaaatagaggattacaaaacgcaggaaaaacacaggaatgaccgtttgattg2040
aaccgcaggaaaaacgcaggatttggatgagagagatagactcaaagaaaagttactaag2100
aggttgaagctcttgcttaatttcctccaaaatttctataggattgttcattccatagga2160
attttaaaggattggataggattcaatcctttgattcaaaggacttcataggaaattttc2220
ctataggattgaaatcctccaaaattcctatgtttttcctccaaatcaaaggggccctaa2280
gctctgttgagagctataatagttcaatgctaccatgtaacagtgcagtagcacagttct2340
gagacattgcccttggtactaccgatgccccctcatgtagagatgggtgttatattcact2400
gatttctcttactgcgataatattttattgaaaaactaataatatgaatggtaattatta2460
taaacatgttcgttgccttccctctattttcttcctgaaatttttagcatacattttgca2520
gctgcgcagagagaataagctaaccaagtcgtcaattatggttacctctggtgcaaatca2580
ggtaatgcacatgctttccagtttccacctgatttattacagctttgtcatcttattccg2640
gttttcctctgtgtattttgccttgtttagctagactataggatgccaatttgtttttct2700
tcttcagttctatgagatgctttcttacttttctgtatgactgcttcacctaaaccttgt2760
ttaccaactaggttagccaaataaaagtcaaataaagtgatgtcttctatcaaattttgg2820
caggcctttgtaaatgtggtcctcaccctttgtgatgctggggatgcagttgtcatgttt2880
gcaccatattacttcaattcctacatgtcattccagatgacaggagttactgacatatta2940
gttggtgcaagcaatcctgagactcttcatcctgatgtcggtaagatagttcttcccttt3000
ttctttttgttggagaaattggttagatgtctattatcaacagtcaattataaactagtt3060
gccactttttttccgcttctgagatcttccactttgtaagttgataagttctatcacttg3120
atacattttctctcagttcaattgagagctatatggtttttgatcattcgatttggttca3180
cttaaggactgtaaaagaatagttcattctggtagtacaatttatatttgtttaataaaa3240
tgcagattggttggagaaggttctgcaagaaaacaaccctatccctaaacttgttagtgt3300
tgtaaatcctggaaacccctctggagctttcattccgaagccgatgctcgaagtaacctg3360
tctctctcattcacagttcacgcacacttgcattcgaggtgactttaattatttcttaat3420
attgcaccattgttccagagaatttcagaactgtgcagaaatgctggtgcatggcttgta3480
gttgacaatacctatgagtaagttagtactcaattactgattattgctcaactgcaacgt3540
cattttggtgcttcttttctttctgttgttgataaagctcctcagtcccttcagaaattg3600
atattacttaaagcaattgtaaagacgaggcactacgaaagaatgaactacacatggaca3660
tgatcaccttaagtagtagtttaaaaggacagttcaccataagcagttgagcaactgtgc3720
ttgacttcttgaagttgagtcctggatgacgccctctctgatagattaaataaataggac3780
ttcaagaatgtggtatctctttcaaaaagtatctgcaaaccacccgccagtaagggtttg3840
tcaaatgtagttatcttcacatgctcagcttattcaagtagggaaaatcttccatttctt3900
ctcaacactagaattggacacatggaacaactcgttttcttttgaaggtcccatttcagt3960
tcctcaatttcagtgatgcccaaaataaaccctccagtcacttacaggcagagtggaaga4020
ctcactgacaagagcctaaattgaatcacggattagactcttccctcaagaaaagtctta4080
ctttcagcagcctagcatccgctgtgcggcattgggaagaatatatctgttgttagttct4140
gatcaaatcagaaatatttgatctcttgctaatctacccttgctctatatttcagatcat4200
gtttaccttttcttgtctggctctgtagttgagtaatggaaattgatattagtttttgcc4260
atttccttaccctgatgttgatgttaaaaggatgctatgggtccttttcgaatataagct4320
tgatggacttcccaatgttaatacaccaccgaatttgacatgtgtaaatcttgtatatga4380
taaaagtataatttggaaactttcaattatcatcacattgatttttagataatctgtatt4440
caaaatgtttgcacgaatactgaaattcaagaaaacattttctcggaagaattcttttcc4500
ataaaaggtgactgaccttttactttctgtaaaataattctacatatggggctattttgg4560
attctgtataactttgatgttgaacttgcttgcaaaattggtgggtggtctccaggtact4620
ttatgtatgatggaatggagcactattgcttagagggtaatcacattgtcaacctcttct4680
cattctcgaaggcttacggaatgatgggctggcgtgtaggatacgtaagtttgcctctct4740
ctctctctctctctctctctctctctctctttctttcatcctctgcttaggcttgaaaat4800
gctgatgtaagagtcaatattgagttctttcagtttcatgaatttcccgcatccacttga4860
atcttttaccagggcattcccttaaattatcaccatagcaatgcagtataagtaacttag4920
tggctgcacaatctgtgttgatcagtgagtcttacaagaactgatgagctgactgctgca4980
gattgcccacccaaacgaagctgacggtcttcatgcacagctgctcaaagtgcaagataa5040
catacctatctgtgcttccatcatcgggcagcgcctggcgctctacgcattagaggctgg5100
tccagaatggatcagagaaagggtgagagatctagtgaaaaaccgtgaattgctcatgga5160
agcgatgtctccgcttggcaaggactctgtcaagggtggtgagggtgccatttacctctg5220
ggcaaaactacccgagaaatgctcagatgattttgaagttgtcagatggcttgcaaacaa5280
gcatggtgtggctgtgatccccgggagcgccagtggaggtcccggatatatccgggtttc5340
tttcggagggttgaaagaatcggatacaaggcttgctgctgagaggttaaggcgcggctt5400
gcaagaacttgtgactgagggaatggtacagtgacagcccaatggatgcaagaaatacat5460
gacatacaaatcacaaaagtgaaaattttcagttacataataattctgggattagttcaa5520
gagaagaaaagagccaaaatttcaagattacaacatggttgttagaaaatgctgaagctt5580
ttatttcagagaacttttgtcacattggatttcctagtggaaatttgatttggtgggttg5640
agtagagggaaaaaaaaagactcatccaccctgtggaataccattgtcattaaaggggta5700
ttcatgtttagtacttactgttgcttgataacctctttccctacaatcaggacagcagct5760
gtacacaagtaagatataataagatataatagcaacaataagtgttcatgatgtgctttt5820
atttcagctttttta5835
<210>2
<211>1185
<212>dna
<213>水稻(oryzasativa)
<220>
<221>gene
<222>(1)..(1185)
<220>
<221>cds
<222>(1)..(1185)
<400>2
atgggtagcttcgggaggctggcgaggagggccgtggagacggaagcg48
metglyserpheglyargleualaargargalavalgluthrgluala
151015
ccggtcatggtcaagatgcaggaattgcttcgagggaacaaggatgtg96
provalmetvallysmetglngluleuleuargglyasnlysaspval
202530
atgtcgcttgcgcagggagttgtttattggcagcctcctgaggcagct144
metserleualaglnglyvalvaltyrtrpglnproproglualaala
354045
atgaataagattaaagaaattgtatgggaaccatcaatcagtaaatat192
metasnlysilelysgluilevaltrpgluproserileserlystyr
505560
ggctctgatgatggtcttcctgaactccgagaagcacttctcgagaag240
glyseraspaspglyleuprogluleuargglualaleuleuglulys
65707580
ctgcgcagagagaataagctaaccaagtcgtcaattatggttacctct288
leuargarggluasnlysleuthrlysserserilemetvalthrser
859095
ggtgcaaatcaggcctttgtaaatgtggtcctcaccctttgtgatgct336
glyalaasnglnalaphevalasnvalvalleuthrleucysaspala
100105110
ggggatgcagttgtcatgtttgcaccatattacttcaattcctacatg384
glyaspalavalvalmetphealaprotyrtyrpheasnsertyrmet
115120125
tcattccagatgacaggagttactgacatattagttggtgcaagcaat432
serpheglnmetthrglyvalthraspileleuvalglyalaserasn
130135140
cctgagactcttcatcctgatgtcgattggttggagaaggttctgcaa480
progluthrleuhisproaspvalasptrpleuglulysvalleugln
145150155160
gaaaacaaccctatccctaaacttgttagtgttgtaaatcctggaaac528
gluasnasnproileprolysleuvalservalvalasnproglyasn
165170175
ccctctggagctttcattccgaagccgatgctcgaaagaatttcagaa576
proserglyalapheileprolysprometleugluargileserglu
180185190
ctgtgcagaaatgctggtgcatggcttgtagttgacaatacctatgag624
leucysargasnalaglyalatrpleuvalvalaspasnthrtyrglu
195200205
tactttatgtatgatggaatggagcactattgcttagagggtaatcac672
tyrphemettyraspglymetgluhistyrcysleugluglyasnhis
210215220
attgtcaacctcttctcattctcgaaggcttacggaatgatgggctgg720
ilevalasnleupheserpheserlysalatyrglymetmetglytrp
225230235240
cgtgtaggatacattgcccacccaaacgaagctgacggtcttcatgca768
argvalglytyrilealahisproasnglualaaspglyleuhisala
245250255
cagctgctcaaagtgcaagataacatacctatctgtgcttccatcatc816
glnleuleulysvalglnaspasnileproilecysalaserileile
260265270
gggcagcgcctggcgctctacgcattagaggctggtccagaatggatc864
glyglnargleualaleutyralaleuglualaglyproglutrpile
275280285
agagaaagggtgagagatctagtgaaaaaccgtgaattgctcatggaa912
arggluargvalargaspleuvallysasnarggluleuleumetglu
290295300
gcgatgtctccgcttggcaaggactctgtcaagggtggtgagggtgcc960
alametserproleuglylysaspservallysglyglygluglyala
305310315320
atttacctctgggcaaaactacccgagaaatgctcagatgattttgaa1008
iletyrleutrpalalysleuproglulyscysseraspasppheglu
325330335
gttgtcagatggcttgcaaacaagcatggtgtggctgtgatccccggg1056
valvalargtrpleualaasnlyshisglyvalalavalileprogly
340345350
agcgccagtggaggtcccggatatatccgggtttctttcggagggttg1104
seralaserglyglyproglytyrileargvalserpheglyglyleu
355360365
aaagaatcggatacaaggcttgctgctgagaggttaaggcgcggcttg1152
lysgluseraspthrargleualaalagluargleuargargglyleu
370375380
caagaacttgtgactgagggaatggtacagtga1185
glngluleuvalthrgluglymetvalgln
385390
<210>3
<211>394
<212>prt
<213>水稻(oryzasativa)
<400>3
metglyserpheglyargleualaargargalavalgluthrgluala
151015
provalmetvallysmetglngluleuleuargglyasnlysaspval
202530
metserleualaglnglyvalvaltyrtrpglnproproglualaala
354045
metasnlysilelysgluilevaltrpgluproserileserlystyr
505560
glyseraspaspglyleuprogluleuargglualaleuleuglulys
65707580
leuargarggluasnlysleuthrlysserserilemetvalthrser
859095
glyalaasnglnalaphevalasnvalvalleuthrleucysaspala
100105110
glyaspalavalvalmetphealaprotyrtyrpheasnsertyrmet
115120125
serpheglnmetthrglyvalthraspileleuvalglyalaserasn
130135140
progluthrleuhisproaspvalasptrpleuglulysvalleugln
145150155160
gluasnasnproileprolysleuvalservalvalasnproglyasn
165170175
proserglyalapheileprolysprometleugluargileserglu
180185190
leucysargasnalaglyalatrpleuvalvalaspasnthrtyrglu
195200205
tyrphemettyraspglymetgluhistyrcysleugluglyasnhis
210215220
ilevalasnleupheserpheserlysalatyrglymetmetglytrp
225230235240
argvalglytyrilealahisproasnglualaaspglyleuhisala
245250255
glnleuleulysvalglnaspasnileproilecysalaserileile
260265270
glyglnargleualaleutyralaleuglualaglyproglutrpile
275280285
arggluargvalargaspleuvallysasnarggluleuleumetglu
290295300
alametserproleuglylysaspservallysglyglygluglyala
305310315320
iletyrleutrpalalysleuproglulyscysseraspasppheglu
325330335
valvalargtrpleualaasnlyshisglyvalalavalileprogly
340345350
seralaserglyglyproglytyrileargvalserpheglyglyleu
355360365
lysgluseraspthrargleualaalagluargleuargargglyleu
370375380
glngluleuvalthrgluglymetvalgln
385390
<210>4
<211>627
<212>dna
<213>水稻(oryzasativa)
<220>
<221>gene
<222>(1)..(627)
<220>
<221>cds
<222>(1)..(627)
<400>4
atgggtagcttcgggaggctggcgaggagggccgtggagacggaagcg48
metglyserpheglyargleualaargargalavalgluthrgluala
151015
ccggtcatggtcaagatgcaggaattgcttcgagggaacaaggatgtg96
provalmetvallysmetglngluleuleuargglyasnlysaspval
202530
atgtcgcttgcgcagggagttgtttattggcagcctcctgaggcagct144
metserleualaglnglyvalvaltyrtrpglnproproglualaala
354045
atgaataagattaaagaaattgtatgggaaccatcaatcagtaaatat192
metasnlysilelysgluilevaltrpgluproserileserlystyr
505560
ggctctgatgatggtcttcctgaactccgagaagcacttctcgagaag240
glyseraspaspglyleuprogluleuargglualaleuleuglulys
65707580
ctgcgcagagagaataagctaaccaagtcgtcaattatggttacctct288
leuargarggluasnlysleuthrlysserserilemetvalthrser
859095
ggtgcaaatcaggcctttgtaaatgtggtcctcaccctttgtgatgct336
glyalaasnglnalaphevalasnvalvalleuthrleucysaspala
100105110
ggggatgcagttgtcatgtttgcaccatattacttcaattcctacatg384
glyaspalavalvalmetphealaprotyrtyrpheasnsertyrmet
115120125
tcattccagatgacaggagttactgacatattagttggtgcaagcaat432
serpheglnmetthrglyvalthraspileleuvalglyalaserasn
130135140
cctgagactcttcatcctgatgtcgattggttggagaaggttctgcaa480
progluthrleuhisproaspvalasptrpleuglulysvalleugln
145150155160
gaaaacaaccctatccctaaacttgttagtgttgtaaatcctggaaac528
gluasnasnproileprolysleuvalservalvalasnproglyasn
165170175
ccctctggagctttcattccgaagccgatgctcgaaagaatttcagaa576
proserglyalapheileprolysprometleugluargileserglu
180185190
ctgtgcagaaatgctggtgcatggcttgtagttgacaatacctatgag624
leucysargasnalaglyalatrpleuvalvalaspasnthrtyrglu
195200205
<210>5
<211>208
<212>prt
<213>水稻(oryzasativa)
<400>5
metglyserpheglyargleualaargargalavalgluthrgluala
151015
provalmetvallysmetglngluleuleuargglyasnlysaspval
202530
metserleualaglnglyvalvaltyrtrpglnproproglualaala
354045
metasnlysilelysgluilevaltrpgluproserileserlystyr
505560
glyseraspaspglyleuprogluleuargglualaleuleuglulys
65707580
leuargarggluasnlysleuthrlysserserilemetvalthrser
859095
glyalaasnglnalaphevalasnvalvalleuthrleucysaspala
100105110
glyaspalavalvalmetphealaprotyrtyrpheasnsertyrmet
115120125
serpheglnmetthrglyvalthraspileleuvalglyalaserasn
130135140
progluthrleuhisproaspvalasptrpleuglulysvalleugln
145150155160
gluasnasnproileprolysleuvalservalvalasnproglyasn
165170175
proserglyalapheileprolysprometleugluargileserglu
180185190
leucysargasnalaglyalatrpleuvalvalaspasnthrtyrglu
195200205
- 还没有人留言评论。精彩留言会获得点赞!