用于增溶目标蛋白质的组合物及其用途的制作方法
本发明涉及一种包含启动子及对伴娘蛋白和rna结合域的融合蛋白进行编码的基因的表达盒、包含所述表达盒的表达载体、含有所述表达盒或所述表达载体的用于增容目标蛋白质的组合物、转化体、试剂盒以及利用该试剂盒的目标蛋白质的生产方法。
背景技术:
在从异种细胞中生产重组蛋白的情况下存在重组蛋白形成不溶性凝聚体的问题,因此需要用于增加重组蛋白的可溶性的方法(currentopinioninbiotechnology,2001,12,202-207)。但是,尚未开发能够应用到多种重组蛋白的重组蛋白增溶方法。
在这种背景下,本发明人为了开发用于增加目标蛋白质的可溶性的方法而进行了不懈努力,其结果确认结合有伴娘蛋白的mrna(信使核糖核酸)支架(chaperone-recruitingmrnascaffold,cras)系统及伴娘蛋白-基质相同位置表达(chaperone-substrateco-localizedexpression,clex)系统增加各种目标蛋白质的可溶性,并且完成了本发明。
技术实现要素:
技术问题
本发明的目的在于提供一种用于增溶目标蛋白质的组合物,含有包含启动子及对伴娘蛋白和rna结合域的融合蛋白进行编码的基因的表达盒,或者含有包含所述表达盒的表达载体。
本发明的另一目的在于提供一种转化体,含有包含启动子及对伴娘蛋白和rna结合域的融合蛋白进行编码的基因的表达盒,或者含有包含所述表达盒的表达载体。
本发明的又一目的在于提供一种包含所述组合物或所述转化体的用于生产目标蛋白质的试剂盒。
本发明的又一目的在于提供一种包含启动子及对伴娘蛋白和rna结合域的融合蛋白进行编码的基因的表达盒,或者包含所述表达盒的表达载体。
本发明的又一目的在于提供一种包含启动子、对目标蛋白质进行编码的基因、以及在该基因的3'末端处具有发夹(haripin)结构的基因的表达盒,或者包含所述表达盒的表达载体。
本发明的又一目的在于提供一种目标蛋白质的生产方法,包括以下步骤:
(i)将(a)包含启动子及对伴娘蛋白和rna结合域的融合蛋白进行编码的基因的表达盒或者包含所述表达盒的表达载体、以及(b)包含启动子、对目标蛋白质进行编码的基因及在该基因的3'末端处具有发夹(haripin)结构的基因的表达盒或者包含所述表达盒的表达载体导入到微生物中;以及
(ii)培养所述微生物。
本发明的又一目的在于提供一种包含(a)对伴娘蛋白进行编码的顺反子及(b)对目标蛋白质进行编码的顺反子的表达盒,或者包含所述表达盒的表达载体。
本发明的又一目的在于提供一种用于增溶目标蛋白质的组合物,含有包含(a)对伴娘蛋白进行编码的顺反子及(b)对目标蛋白质进行编码的顺反子的表达盒,或者含有包含所述表达盒的表达载体。
本发明的又一目的在于提供一种转化体,含有包含(a)对伴娘蛋白进行编码的顺反子及(b)对目标蛋白质进行编码的顺反子的表达盒,或者含有包含所述表达盒的表达载体。
本发明的又一目的在于提供一种用于生产目标蛋白质的试剂盒,含有包含(a)对伴娘蛋白进行编码的顺反子及(b)对目标蛋白质进行编码的顺反子的表达盒、包含所述表达盒的表达载体、包含所述表达盒或所述表达载体的用于增溶目标蛋白质的组合物、或者包含所述表达盒或所述表达载体的转化体。
本发明的又一目的在于提供一种目标蛋白质的生产方法,包括以下步骤:
(i)准备转化体,所述转化体含有包含(a)对伴娘蛋白进行编码的顺反子及(b)对目标蛋白质进行编码的顺反子的表达盒,或者含有包含所述表达盒的表达载体;以及
(ii)培养所述转化体。
技术方案
为了实现上述目的,本发明的一方面提供一种用于增溶目标蛋白质的组合物,含有包含启动子及对伴娘蛋白和rna结合域的融合蛋白进行编码的基因的表达盒,或含有包含所述表达盒的表达载体。
由所述表达盒或所述表达载体表达的融合蛋白通过rna结合域部分与包含目标蛋白质的序列的mrna结合,其结果融合蛋白的伴娘蛋白部分位于经翻译的目标蛋白质附近,从而能够使目标蛋白质的折叠准确地进行。即,所述融合蛋白能够与目标蛋白质的种类无关地普遍增溶目标蛋白质。在该情况下,包含目标蛋白质的序列的mrna为了与融合蛋白结合而可在3'utr(3'非翻译区)上具有发夹结构(包含供融合蛋白的rna结合域结合的rna位点)。
在本发明中,术语“伴娘蛋白(chaperone)”是指参与其他蛋白质的结构性折叠(folding)和伸展(unfolding)或者组装(chaperone)和解体(disassembly)的蛋白质。具体而言,在本发明中伴娘蛋白发挥诱导或帮助目标蛋白质的折叠的功能,能够增加目标蛋白质的可溶性。在本发明中,伴娘蛋白以与rna结合域之间的融合蛋白形态结合到包含目标蛋白质的序列的mrna,其结果在经翻译的目标蛋白质附近帮助目标蛋白质的折叠。
在本发明中,只要伴娘蛋白参与目标蛋白质的折叠,则对其微生物来源及具体序列没有限制,具体而言伴娘蛋白可以是来源于原核生物(细菌等)、真核生物或古细菌(archaea)的蛋白质,可以在作为公知的数据库的ncbi(美国国家生物技术信息中心)的genbank(基因库)等中获取该蛋白质的序列。具体而言,所述伴娘蛋白可以是dnaj、dnak、groel、groes、htpg、clpa、clpx或clpp,更具体而言,可以是dnaj或dnak。所述dnaj或所述dnak可具有由序列号74或75的碱基序列编码的氨基酸序列,但并不限于此。此外,所述伴娘蛋白可以是hsp40、hsp60、hsp70、hsp90、hsp100或hsp104。
在本发明中,伴娘蛋白可以具有同种或异种的两个以上的伴娘蛋白单位结合而成的形态。具体而言,所述伴娘蛋白可以具有dnaj和dnak结合而成的形态(dnajk),但并不限于此。
在本发明中,术语“rna结合域(rnabindingdomain,rbd)”是指与rna结合的结构域。具体而言,在本发明中,rna结合域可被制作成通过与伴娘蛋白形成结合体或融合蛋白而使包含所述伴娘蛋白的结合体或融合蛋白与包含目标蛋白质的序列的mrna结合。即,所述rna结合域使融合蛋白的伴娘蛋白位于由核糖体翻译的目标蛋白质附近,从而通过位于附近的伴娘蛋白而使目标蛋白质的折叠被准确地进行。
在本发明中,rna结合域对能够结合到包含目标蛋白质的mrna的微生物来源及具体序列没有限制,具体而言,rna结合域可以是kh(khomology,k同源性)结构域、噬菌体ms2衣壳蛋白(coatprotein)、噬菌体pp7衣壳蛋白、不育α基序(sterilealphamotif,sam)或rrm(rnarecognitionmotif,rna识别基序)。所述rna结合域可从作为公知的数据库的ncbi的genbank中获取。此外,所述kh结构域可具有由序列号76的碱基序列编码的氨基酸序列,但并不限于此。
在本发明中,融合蛋白包含所述至少一种伴娘蛋白和所述rna结合域。此外,在所述融合蛋白中,伴娘蛋白可以直接或通过连接肽而连接到rna结合域的n-末端、c-末端或n-末端及c-末端。此外,所述融合蛋白的rna结合域可以直接或通过连接肽而连接到伴娘蛋白的n-末端、c-末端或n-末端及c-末端。
只要所述连接肽使融合蛋白的伴娘蛋白和rna结合域呈现活性,则不对此特别限制,具体而言,可使用甘氨酸、丙氨酸、亮氨酸、异亮氨酸、脯氨酸、丝氨酸、苏氨酸、天冬酰胺、天冬氨酸、半胱氨酸、谷氨酰胺、谷氨酸、赖氨酸、精氨酸等的氨基酸来连接融合蛋白的伴娘蛋白和rna结合域,更具体而言,可使用多个缬氨酸、亮氨酸、天冬氨酸、甘氨酸、丙氨酸、脯氨酸等来连接融合蛋白的伴娘蛋白和rna结合域,进一步更具体而言,可通过考虑基因操作的容易性而连接一个至十个甘氨酸、缬氨酸、亮氨酸、天冬氨酸等来使用。例如,在本发明中,通过g4s连接肽而连接伴娘蛋白的c-末端和rna结合域的n-末端而制备融合蛋白。
所述融合蛋白可包含具有一个以上的氨基酸残基与该融合蛋白中包含的各蛋白质或结构域的野生型氨基酸序列不同的序列的多肽。本领域中公知整体上不改变分子活性的、在蛋白质或多肽中的氨基酸交换。最普遍出现的交换为氨基酸残基ala/ser、val/ile、asp/glu、thr/ser、ala/gly、ala/thr、ser/asn、ala/val、ser/gly、thy/phe、ala/pro、lys/arg、asp/asn、leu/ile、leu/val、ala/glu、asp/gly之间的交换。此外,所述融合蛋白可包含通过氨基酸序列上的变异或修饰而具有增加的对热、ph等的蛋白质结构稳定性或增加的蛋白质活性的蛋白质。
可通过本领域公知的化学肽合成方法来制备所述融合蛋白或构成所述融合蛋白的多肽,或者通过利用pcr(polymerasechainreaction,聚合酶链式反应)扩增对所述融合蛋白进行编码的基因后或者利用公知方法合成对所述融合蛋白进行编码的基因后克隆到表达载体上并使其表达来制备所述融合蛋白或构成所述融合蛋白的多肽。
作为本发明的一实施方式,所述融合蛋白可以具有作为伴娘蛋白的dnaj及dnak(dnajk)和作为rna结合域的kh结构域结合而成的形态(dnajk-kh),但并不限于此。
本发明的所述蛋白质不仅可包含由以各序列号记载的碱基序列编码的氨基酸序列,而且可包含与所述序列相比具有80%以上,具体而言90%以上,更具体而言95%以上,进一步更具体而言97%以上的同源性的氨基酸序列。只要是表示呈现与所述各蛋白质实质上相同或相应的效能的蛋白质的氨基酸序列,则无限制地包含具有这种同源性的序列。此外,只要是具有这种同源性的氨基酸序列,则经部分序列的缺失、变形、置换或附加的氨基酸序列也包含在本发明的范围内,这是显而易见的。
此外,本发明的对所述蛋白质进行编码的基因不仅包含以各序列号记载的碱基序列,而且只要是对呈现与所述各蛋白质实质上相同或相应的效能的蛋白质进行编码的碱基序列,则无限制地包含与所述序列相比呈现80%以上,具体而言90%以上,更具体而言95%以上,进一步更具体而言98%以上,最具体而言99%以上的同源性的碱基序列。此外,只要是具有这种同源性的碱基序列,则经部分序列的缺失、变形、置换或附加的碱基序列也包含在本发明的范围内,这是显而易见的。
本发明中使用的术语“同源性”是指对蛋白质进行编码的基因的碱基序列或氨基酸序列的相似程度,在同源性充分高的情况下该基因的表达产物可具有相同或相似的活性。
在本发明中,术语“启动子”是指在合适的宿主细胞中对可操作地连接的其他核酸序列的表达进行调控的核酸序列,在本发明中,“可操作地连接(operablylinked)”是指以执行一般功能的方式功能性地连接(functionallinkage)核酸表达调控序列和对目标蛋白质或rna进行编码的核酸系列。例如,启动子与对蛋白质或rna进行编码的核酸序列被可操作地连接而有可能会影响编码序列的表达。作为使这种启动子与对蛋白质或rna进行编码的核酸序列可操作地连接的方法,可利用本技术领域熟知的基因重组技术,使用本技术领域普遍已知的酶等进行位点-特异性dna切断及连接。
在本发明中,术语“表达载体”是指以在合适的宿主内能够表达目标蛋白质的方式可操作地连接到合适的调控序列上的、含有所述表达盒的dna制备物。所述调控序列可包含能够开始转录的启动子、用于调控这种转录的任意操纵序列、对合适的mrna核糖体结合位点进行编码的序列及用于调控转录和翻译的终止的序列。载体在转化到合适的宿主细胞内之后能够与宿主基因组无关地被复制或发挥功能,并且能够整合到基因组自身中。
只要能够在宿主细胞内被复制,则对本发明中使用的表达载体并不特别限定,可利用本领域已知的任意载体。作为通常使用的载体的例,可列举具有天然状态或具有重组后的状态的质粒、粘粒、病毒及噬菌体。例如,作为噬菌体载体或粘粒载体可使用pwe15、m13、mbl3、mbl4、ixii、ashii、apii、t10、t11、charon4a及charon21a等,作为质粒载体可使用pbr类、puc类、pbluescriptii类、pgem类、ptz类、pcl类、pet类、pcp类等。具体而言,可使用pdz、pacyc177、pacyc184、pcl、peccg117、puc19、pbr322、pmw118、pcc1bac、pcp22、pamt7、pet16b载体等,但并不限于此。
能够在本申请中使用的载体并不特别限制,可使用公知的表达载体。此外,能够通过用于插入细胞内染色体中的载体来将对目标蛋白质进行编码的多核苷酸插入到染色体内。可通过本领域已知的任意方法,例如同源重组来将所述多核苷酸插入染色体内,但并不限定于此。本发明中使用的载体可进一步包括用于确认是否插入所述染色体内的筛选标记(selectionmarker)。筛选标记用于筛选利用载体转化的细胞,即用于确认是否插入目标核酸分子中,作为筛选标记可使用赋予抗药性、营养要求性、对细胞毒性药物的耐药性或如表面蛋白质的表达等可选择表型的标记。由于在经选择剂(selectiveagent)处理的环境下只有表达筛选标记的细胞能生存或呈现其他表型,因此能够筛选转化后的细胞。
本发明的组合物可进一步含有包含启动子、对目标蛋白质进行编码的基因及在该基因的3'末端处具有发夹(haripin)结构的基因的表达盒,或进一步含有包含所述表达盒的表达载体。所述启动子及所述表达载体如前所述。
由所述表达盒或所述表达载体转录的mrna具有目标蛋白质的rna且在该mrna的3'-utr上具有发夹(haripin)结构,能够使所述发夹和所述融合蛋白的rna结合域位点结合。通过所述结合而形成融合蛋白的伴娘蛋白部分结合到包含目标蛋白质的序列的mrna的3'-utr上而成的支架。在本发明中,将所述支架命名为结合有伴娘蛋白的mrna支架(chaperone-recruitingmrnascaffold,cras)。
在本发明中,所述“发夹(haripin)”是指通过单股dna或rna内部的碱基之间的氢结合而形成的、由双股部分(stem)和夹在该双股部分的环(loop)部分构成的结构。所述发夹与茎环(stem-loop)混用。作为所述发夹的序列可利用本领域已知的任意序列。
此外,所述发夹结构可具有供融合蛋白的rna结合域,即连接到伴娘蛋白的rna结合域结合的rna序列(位点)。因此,包含在所述表达盒或所述表达载体中的具有发夹结构的基因可包括对供所述结合域结合的rna序列进行转录的序列。
在本发明中,所述表达盒或所述表达载体在对目标蛋白质进行编码的基因与具有发夹结构的基因之间可进一步包含间隔(spacer)基因。所述间隔基因是指基因之间的非编码(non-coding)基因,可利用本领域已知的任意序列。
在本发明中,具有发夹结构的基因可具有一个以上的具有发夹结构的基因。在该情况下,在包含目标蛋白质序列的mrna中可存在一个以上的能够供本发明的融合蛋白结合的发夹,能够使更多的伴娘蛋白结合到所述mrna上。
所述一个以上的具有发夹结构的基因可以在发夹结构与发夹结构之间包含间隔基因,但并不限于此。
在本发明中,表达盒或表达载体可进一步包含核糖体结合位点。所述“核糖体结合位点(ribosomebindingsite,rbs)”是指当蛋白质生物合成开始时能够使从dna转录的mrna在宿主细胞内与核糖体结合的核糖体结合位点。所述核糖体结合位点可利用本领域已知的任意序列。
本发明的另一方面提供一种转化体,含有包含启动子及对伴娘蛋白和rna结合域的融合蛋白进行编码的基因的表达盒,或者含有包含所述表达盒的表达载体。具体而言,所述转化体可进一步含有包含启动子、对目标蛋白质进行编码的基因、以及在该基因的3'末端处具有发夹结构的基因的表达盒,或者进一步含有包含所述表达盒的表达载体。
所述启动子、伴娘蛋白、rna结合域、融合蛋白、具有发夹结构的基因、表达盒及表达载体与前述相同。
所述转化体可通过cras系统来生产可溶性增加的目标蛋白质。
在本发明中,术语“转化”是指通过使本发明的表达盒或包含所述表达盒的表达载体导入到宿主细胞内而在宿主细胞内能够表达由所述表达盒或所述表达载体的多核苷酸编码的蛋白质。只要能够在宿主细胞内表达,则可包括任何转化后的多核苷酸,而不管该多核苷酸被插入并设置于宿主细胞的染色体内还是设置于染色体外。此外,所述多核苷酸包含对标的蛋白质进行编码的dna和rna。只要所述多核苷酸能够被导入到所述宿主细胞内并表达,则可以任何形式被导入。例如,所述多核苷酸可以以作为包含自身表达所需要的所有要素的基因结构体的表达盒(expressioncassette)形态被导入到宿主细胞中。所述表达盒一般可包含可操作地连接到所述多核苷酸的启动子(promoter)、转录终止信号、核糖体结合位点及翻译终止信号。所述表达盒可以具有能够进行自我复制的表达载体形态。此外,所述多核苷酸也可以是通过以其自身形态被导入到宿主细胞中而在宿主细胞中与表达所需的序列可操作地连接的核酸,但并不限定于此。进行所述转化的方法也可以包含将核酸导入到细胞内的任何方法,可根据宿主细胞如本领域公知的那样选择合适的标准技术来执行进行所述转化的方法。例如,进行所述转化的方法具有电穿孔法(electroporation)、磷酸钾(capo4)沉淀法、氯化钾(cacl2)沉淀法、微细注入法(microinjection)、聚乙二醇(peg)法、deae-右旋糖苷法、阳离子脂质体法及醋酸锂-dmso法等,但并不限于此。
只要能够表达由本发明的表达盒或表达载体的多核苷酸编码的蛋白质,则所述转化体并不受限制,具体而言,所述转化体可以是植物、细菌、菌类、酵母或动物细胞,更具体而言,所述转化体可以是埃希氏菌(escherichia)属微生物。
本发明的另一方面提供一种用于生产目标蛋白质的试剂盒,包含用于增溶所述目标蛋白质的组合物或所述转化体。
本发明的另一方面提供一种包含启动子及对伴娘蛋白和rna结合域的融合蛋白进行编码的基因的表达盒,或者包含所述表达盒的表达载体。
所述启动子、伴娘蛋白、rna结合域、融合蛋白、表达盒及表达载体与前述相同。
本发明的另一方面提供一种包含启动子、对目标蛋白质进行编码的基因、以及在该基因的3'末端处具有发夹(haripin)结构的基因的表达盒,或者包含所述表达盒的表达载体。
所述启动子、具有发夹结构的基因、表达盒及表达载体与前述相同。本发明的另一方面提供一种包含所述组合物或所述转化体的用于生产目标蛋白质的试剂盒。
本发明的另一方面提供一种目标蛋白质的生产方法,包括以下步骤:
(i)准备转化体,所述转化体含有(a)包含启动子及对伴娘蛋白和rna结合域的融合蛋白进行编码的基因的表达盒或者包含所述表达盒的表达载体、以及(b)包含启动子、对目标蛋白质进行编码的基因及在该基因的3'末端处具有发夹(haripin)结构的基因的表达盒或包含所述表达盒的表达载体;以及
(ii)培养所述转化体。
所述启动子、伴娘蛋白、rna结合域、融合蛋白、具有发夹结构的基因、表达盒、表达载体及转化体与前述相同。
所述(i)准备转化体的步骤可以是在已具有(b)包含启动子、对目标蛋白质进行编码的基因及在该基因的3'末端处具有发夹结构的基因的表达盒或者包含所述表达盒的表达载体的宿主细胞中,进一步导入(a)包含启动子及对融合蛋白进行编码的基因的表达盒或包含所述表达盒的表达载体的步骤,但并不限于此。
此外,所述(i)准备转化体的步骤可以是在已具有(a)包含启动子及对融合蛋白进行编码的基因的表达盒或包含所述表达盒的表达载体的宿主细胞中,进一步导入(b)包含启动子、对目标蛋白质进行编码的基因及在该基因的3'末端处具有发夹结构的基因的表达盒或者包含所述表达盒的表达载体的步骤,但并不限于此。
此外,所述(i)准备转化体的步骤可以是将(a)包含启动子及对融合蛋白进行编码的基因的表达盒或者包含所述表达盒的表达载体和(b)包含启动子、对目标蛋白质进行编码的基因及在该基因的3'末端处具有发夹结构的基因的表达盒或者包含所述表达盒的表达载体顺序地、同时或逆序地导入到宿主细胞中的步骤,但并不限于此。
在所述生产方法中,所述培养转化体的步骤并不受特别限制,但可通过公知的分批式培养方法、连续式培养方法、分批补料式培养方法等来执行。此时,培养条件并不特别受限于此,可使用碱性化合物(例如,氢氧化钠、氢氧化钾或氨)或酸性化合物(例如,磷酸或硫酸)来调节至合适的ph(例如,ph5至9、具体而言ph6至8、最具体而言ph6.8),并且可通过将氧或含氧气体混合物导入到培养物中而维持好气性条件。培养温度可维持20至45℃,具体而言可维持25至40℃,并且可进行约10至160小时的培养,但并不限于此。通过所述培养生产出的目标蛋白质有可能会分泌到培养基中或者残留在细胞内。
此外,作为碳供应源,所使用的培养用培养基可以单独使用或混合使用糖及碳水化物(例如,葡萄糖、蔗糖、乳糖、果糖、麦芽糖、糖蜜、淀粉及纤维素)、油脂及脂肪(例如,大豆油、葵花油、花生油及椰子油)、脂肪酸(例如,软脂酸、硬脂酸及亚油酸)、醇类(例如,甘油及乙醇)以及有机酸(例如,乙酸)等,但并不限于此。作为氮供应源可单独使用或混合使用含氮有机化合物(例如,蛋白胨、酵母提取液、肉汁、麦芽提取液、玉米浆、大豆面粉及脲)或无机化合物(例如,硫酸铵、氯化铵、碳酸铵及硝酸铵)等,但并不限于此。作为磷供应源可单独使用或混合使用磷酸二氢钾、磷酸氢二钾、与此相应的含钠盐等,但并不限于此。此外,在培养基中可包含其他金属盐(例如,硫酸镁或硫酸铁)、氨基酸及如维生素等的必要促生长物质。
所述生产方法可进一步包括从转化体或该转化体的培养物中回收目标蛋白质的步骤。回收从所述培养步骤生产出的目标蛋白质的方法可根据培养方法利用本领域公知的适当方法来从转化体或该转化体的培养物中收集目标蛋白质。例如,该方法可使用细胞破碎、离心分离、过滤、阴离子交换色谱法、结晶化及hplc(高效液相色谱法)等,可利用本领域公知的适当方法来从转化体或该转化体的培养物中回收目标蛋白质。
本发明的另一方面提供一种包含(a)对伴娘蛋白进行编码的顺反子及(b)对目标蛋白质进行编码的顺反子的表达盒,或者包含所述表达盒的表达载体。
所述伴娘蛋白、表达盒及表达载体与前述相同。
伴娘蛋白和目标蛋白质通过所述表达盒或所述表达载体来一同被翻译,从而伴娘蛋白能够增加目标蛋白质的准确折叠。在本发明中,将所述系统命名为clex(chaperone-substrateco-localizedexpression)系统。
所述(a)及所述(b)可以在5'至3'方向上顺序连接或逆序连接,(a)或(b)的终止密码子可以与(b)或(a)的起始密码子重叠,但并不限于此。
本发明的另一方面提供一种用于增溶目标蛋白质的组合物,含有包含(a)对伴娘蛋白进行编码的顺反子及(b)对目标蛋白质进行编码的顺反子的表达盒,或者含有包含所述表达盒的表达载体。
所述伴娘蛋白、表达盒及表达载体与前述相同。
本发明的另一方面提供一种转化体,含有包含(a)对伴娘蛋白进行编码的顺反子及(b)对目标蛋白质进行编码的顺反子的表达盒,或者含有包含所述表达盒的表达载体。
所述伴娘蛋白、表达盒、表达载体及转化体与前述相同。
本发明的另一方面提供一种用于生产目标蛋白质的试剂盒,含有包含(a)对伴娘蛋白进行编码的顺反子及(b)对目标蛋白质进行编码的顺反子的表达盒、包含所述表达盒的表达载体、包含所述表达盒或所述表达载体的用于增溶目标蛋白质的组合物、或者包含所述表达盒或所述表达载体的转化体。
所述伴娘蛋白、表达盒、表达载体及转化体与前述相同。
本发明的另一方面提供一种目标蛋白质的生产方法,包括以下步骤:
(i)准备转化体,所述转化体含有包含(a)对伴娘蛋白进行编码的顺反子及(b)对目标蛋白质进行编码的顺反子的表达盒,或者含有包含所述表达盒的表达载体;以及
(ii)培养所述转化体。
所述伴娘蛋白、表达盒、表达载体、转化体、培养及生产方法与前述相同。
所述生产方法可进一步包括以下步骤:从转化体或该转化体的培养物中回收目标蛋白质。
发明效果
导入3'utr发夹序列的目标蛋白质的mrna及利用伴娘蛋白和rna结合域的融合蛋白的本发明的cras系统能提高多种目标蛋白质的可溶性。此外,具有两个顺反子的本发明的clex系统能提高多种目标蛋白质的可溶性。
附图说明
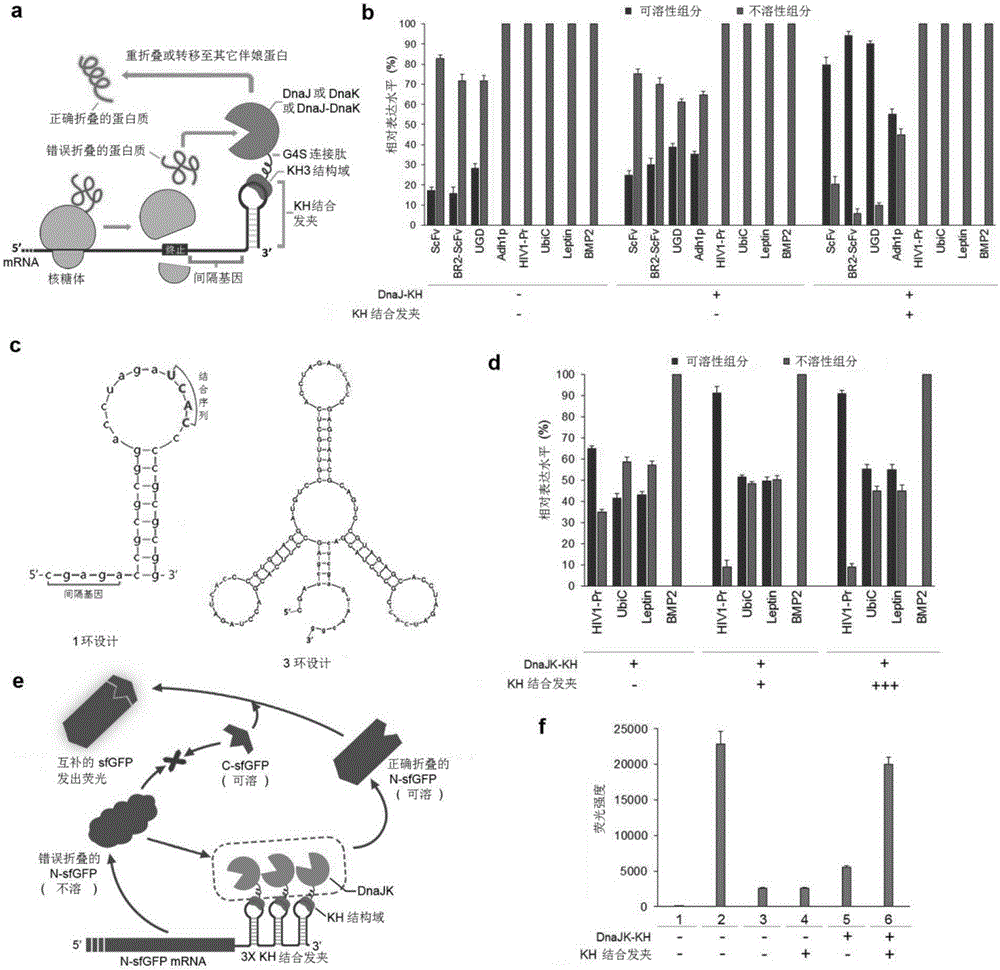
图1是表示结合有伴娘蛋白的mrna支架(chaperone-recruitingmrnascaffold,cras)系统下的重组蛋白的增溶的图:
(a)是呈现cras系统的图。伴娘蛋白分子(dnaj,dnak,或dnaj和dnak的结合体(dnaj-dnak))和nova-1的rna结合域kh的融合蛋白与插入有(c)中记载的kh同族(cognate)茎环序列的目标重组蛋白质一同被表达。通过在mrna的3'utr内导入kh-结合结构,伴娘蛋白结合体与早期蛋白结合而防止蛋白质的凝集,或者为了准确折叠而将蛋白质移向其他伴娘蛋白。
(b)是表示当八个已选择的容易凝集的重组蛋白(scfv、hiv-1protease、br2-scfv、ugd、adh1p、ubic、leptin、bmp2)与单个dnaj-kh或与dnaj-kh及3'utr发夹环一同被表达时的增溶结果的图。
(d)是表示在应用利用嵌合体dnaj-kh的cras系统的情况下,在利用dnaj-kh的cras系统中未增溶的四个重组蛋白根据一个(+)或三个(+++)3'utr序列被增溶的结果的图。
(e)是呈现为了观察cras系统的invivo(活体内)蛋白增溶活性而进行的分割gfp(绿色荧光蛋白)实验的图。
(f)是呈现利用cras系统的sfgfpn-末端的invivo增溶结果的图。(1)pet16b及pamt7(阴性对照组);(2)pet16b-sfgfp及pamt7-dnajk-kh(阳性对照组);(3)不存在kh结合发夹及dnaj-kh的条件下的sfgfpn-末端和c-末端的质粒;(4)存在kh结合发夹及不存在dnaj-kh的条件下的sfgfpn-末端和c-末端的质粒;(5)不存在kh结合发夹及存在dnaj-kh的条件下的sfgfpn-末端和c-末端的质粒;(6)测量包含有存在kh结合发夹及dnaj-kh的条件(cras系统)下的sfgfpn-末端和c-末端的质粒的大肠杆菌bl21(de3)细胞的荧光强度。在使用imagejv1.48software(nhi,usa)且通过库马西蓝色染色获得的sds-page图像中执行可溶性scfv的定量。(b)、(d)及(f)的误差柱表示从三次独立实验获得的±标准偏差。
图2是呈现在大肠杆菌bl21(de3)中应用具有一个环(a)或三个环(b)的cras系统时的scfv按不同时间的增溶的图。
泳道m,标记(elpis,daejeon,southkorea);泳道w,总体细胞溶解物;泳道s,可溶性组分;泳道i,不溶性组分。
图3是呈现随cras系统中的终止密码子与3'utr结合环之间的间隔长度而不同的增溶结果的图。在诱导表达四小时之后,与scfv单独表达的情况(a)相比,应用cras系统的情况(b)的scfv的可溶性增加。
泳道1,终止密码子与3'utr结合环之间不存在间隔时;泳道2,终止密码子与3'utr结合环之间存在5nt间隔时;泳道3,终止密码子与3'utr结合环之间存在30nt间隔时;泳道m,标记;泳道w,总体细胞溶解物;泳道s,可溶性组分;泳道i,不溶性组分。
图4是呈现在dnak敲除菌株中scfv的可溶性根据cras系统增加的图(泳道c-:具有pet16b及pamt7的大肠杆菌bl21(de3)的总体细胞溶解物(阴性对照组)、泳道1:不存在kh结合发夹及dnaj-kh的条件、泳道2:存在kh结合发夹及不存在dnaj-kh的条件、泳道3:不存在kh结合发夹及存在dnaj-kh的条件、泳道4:存在kh结合发夹及dnaj-kh的条件(rnascaffold)、泳道m:标记、泳道w:总体细胞溶解物、泳道s:可溶性组分、泳道i:不溶性组分)。
图5是表示clex系统下的重组蛋白的增溶的图。
(a)是呈现clex系统的图。为了从单个mrna转录物中进行临近dnaj和目标蛋白质的翻译而利用翻译上结合的两个顺反子表达系统。
(b)是呈现在第二个顺反子的起始密码子(atg)之前重叠第一个顺反子的终止密码子(taa)或者排成一列(tandem)或设计成间隔着nnt的距离的序列的图。第一个顺反子的3'序列内的12个核苷酸对第二个顺反子发挥核糖体结合位点(rbs)的作用。
(c)是在clex系统中伴娘蛋白dnaj用作第一个或第二个顺反子时,呈现容易凝集的五个重组蛋白的增溶的图。
(d)是为了确认准确折叠的瘦素的水平而测量从具有质粒的大肠杆菌bl21(de3)得到的样品od450的结果:pet16b(阴性对照组)、pet16b-leptin(廋素的过表达)及pet16b-dnaj/leptin(从clex系统中过表达dnaj及廋素、clex系统)。
(e)是在翻译上结合的两个顺反子表达结构中按dnaj-标的蛋白质的顺序呈现bmp2的增溶结果的图。
(f)是在翻译上结合的两个顺反子表达结构中按顺反子之间的距离呈现bmp2的增溶结果的图。
蛋白质之间的斜线(/)标志表示对mrna内的各蛋白质进行编码的基因的顺序。以sds-page结果为基础使用imagejv1.48software(nji,usa)来定量相对表达水平。(c)、(d)、(e)及(f)的误差柱是指从三次独立实验得到的±标准偏差。
图6是呈现在dnaj敲除菌株(a)或dnak敲除菌株(b)中bmp2的可溶性根据clex系统增加的图(泳道c-:具有pet16b及pamt7的大肠杆菌bl21(de3)的总体细胞溶解(阴性对照组)、泳道onlybmp2:不存在dnaj的条件下表达bmp2、泳道bmp2+dnaj:存在dnaj的条件下表达bmp2、泳道dnaj/bmp2:在clex系统中,在第一个顺反子上设置有dnaj,在第二顺反子上设置有bmp2的情况、泳道m:标记、泳道w:总体细胞溶解物、泳道s:可溶性组分、泳道i:不溶性组分)。
图7是表示在clex系统中随重组蛋白的大小而不同的增溶结果的图。
(a)是用红色长方形表示脂肪分解酶tlia内的dnaj/dnak结合序列的分布的图。使用limbo伴娘蛋白结合位点推测器来预测结合位点。
(b)是表示利用clex系统的tlia1片段的增溶的图。tlia1+dnaj表示tlia和dnaj的单纯同时表达,dnaj/tlia1表示当dnaj被用作第一个顺反子时的clex系统。以sds-page结果为基础使用imagejv1.48软件(nji,usa)来定量相对表达水平。(b)的误差柱是指从三次独立实验得到的±标准偏差。
具体实施方式
下面,通过实施例对本发明的结构及效果进行更详细说明。这些实施例只是用于举例说明本发明,本发明的范围并不受这些实施例的限制。
实验例1:微生物、酶及化学药品
使用大肠杆菌xl1-blue(stratagene,lajolla,ca,usa)及bl21(de3)(novagen,madison,wi,usa)菌株。将xl-1blue菌株用于所有克隆中,并且为了基因表达而使用bl21(de3)菌株。通过公知的p1转导(currentprotocolsinmolecularbiology,2007,chapter1,unit117)而制备没有dnaj和dnak基因的菌株。通过keiocollection获取具有单个敲除(δdnaj及δdnak)的bw25113菌株,并且作为用于bl21(de3)的供体菌株来使用。为了使用位于目标基因的周边的引物对来筛选缺失菌株而使用菌落pcr(colonypcr)(表1)。然后,通过来源于pcp22质粒的flp重组酶而从宿主基因组内的整合区域中去除卡那霉素抗性盒。将本发明中使用的寡核苷酸(genotech,大田,韩国)和基因(bioneer,大田,韩国)记载于表1中。从sigma-aldrich(st.louis,mo,usa)中购买化学药品。
[表1]
实验例2:制备用于cras系统的对伴娘蛋白和rna结合域的融合蛋白或目标重组蛋白进行编码的表达载体
为了结合有伴娘蛋白的mrna支架(chaperone-recruitingmrnascaffold,cras),制备对伴娘蛋白和rna结合域的融合蛋白或目标重组蛋白进行编码的表达载体。
首先,为了以具有两个强力的t7启动子的pacycduet-1载体(novagen,madison,wi,usa)为基础制备中间复制数载体(medium-copynumbervector),通过将作为低复制数的复制起点(originofreplication)的p15a替换为作为中间复制数的复制起点的pbr322,制备pamt7载体。
以大肠杆菌bl21(de3)的基因组dna为基础利用pcr来扩增伴娘蛋白基因dnaj(序列号74)及dnak(序列号75)。为了dnaj和dnaj-kh的高表达,利用核糖体结合位点(ribosomebindingsite,rbs)计算器来设计合成核糖体结合位点,并使其包含到dnajncof引物的5'末端。
为了制备伴娘蛋白和rna结合域的融合蛋白,合成具有适合大肠杆菌内的表达的序列的、nova-1kh3rna结合域(kh)的基因(序列号76),并利用recombinant(重组)pcr来使其结合到dnaj或dnajk的下游。
以各种原基为基础且利用pcr来扩增对目标重组蛋白进行编码的基因:以pbr2scfv为基础扩增scfv及br2-scfv(plosone,2013,8,e66084),以大肠杆菌bl21(de3)的基因组dna为基础扩增ugd及ubic,以s.cerevisae的基因组dna为基础扩增adh1p。利用recombinantpcr来制备hiv1-pr基因(proteinexpressionandpurification,2015,116,59-65)。以ptlia为基础扩增tlia(acscatal,2015,5,5016-5025)。为了绿色荧光蛋白质(gfp)互补分析法,以超折叠绿色荧光蛋白(sfgfp)(bioneer,大田,韩国)为模板扩增sfgfp的n-末端和c-末端。为了sfgfp的高表达,利用rbs计算器来设计合成rbs及sfgfp的n-末端,并且使其包含到sfgfpxbaf引物的5'末端。所使用的引物示于上述表1中。
以纯化为目的,在hiv1-pr的情况下将6×his-tag附加到正向引物的n-末端,在目标重组蛋白的情况下将6×his-tag附加到逆向引物的c-末端。为了hiv1-pr的高表达,利用rbs计算器来设计合成rba,并使其包含到hivprxbaf引物的5'-末端。使用表1所示的限制酶来切断pcr产物和与此相应的表达载体,然后在伴娘蛋白的情况下使其连接到pamt7,在重组蛋白的情况下使其连接到pet16b。
实验例3:利用cras系统制备rna支架(scaffold)
为了结合有伴娘蛋白的mrna支架(chaperone-recruitingmrnascaffold,cras)系统而制备rna支架。具体而言,使用定制rna(rnadesigner)及mfold来设计用于rna支架系统中使用的kh结构域的结合环的序列(journalofmolecularbiology,2004,336,607-624;science1989,244,48-52)。序列约束(constraint)如下所述:
5'-nnnnnnnnacctagatcacc(序列号77)nnnnnnnn-3',其中,n表示用于8bp茎结构的任意核苷酸,用下划线表示的序列表示包含用于khrna结合域的结合序列的茎结构。
在具有多个结合环的rna支架系统的情况下,利用用于将5nt间隔附加到各个系统环结构之间的类似临近(similarapproach)来设计茎环结构。通过利用具有3'utr内的茎环结构的序列的逆向引物的pcr,来制备关于目标蛋白质基因的茎环结构。
实验例4:制备clex系统
按dnaj配置于第一个或第二个顺反子上的两个不同的排列来制备dnaj伴娘蛋白和重组蛋白(scfv、bic、leptin、hiv1-pr、bmp2及tlia)的clex系统。对eeve的氨基酸序列(eeve,序列号78)进行编码,并且为了改善第二个顺反子内的基因的翻译开始,由包含核糖体结合位点((shine-dalgarno,sd)序列(下划线部分)的12nt构成的区域(5'-gaggaggtggaa-3',序列号79)被导入到第一个顺反子内基因的c-末端。此外,为了保障翻译偶联(translationalcoupling)并促进dnaj与重组蛋白之间的相互作用,通过1nt(5'-taatg-3')使第一个顺反子的终止密码子与第二个顺反子的起始密码子重叠。为了制备clex系统,利用重组pcr(recombinantpcr)来组装dnaj伴娘蛋白和重组蛋白。引物序列示于表2中。通过表2所记载的限制酶来切断pcr产物和pet16b后连接到表达载体。
[表2]
实验例5:蛋白质增溶(solubilization)测量方法
同时用伴娘蛋白的质粒和目标重组蛋白的质粒对大肠杆菌bl2(de3)进行转化。然后在补充有最终浓度分别为50μg/ml和25μg/ml的氨苄青霉素和氯霉素的、3ml的lb(lysogenicbroth,溶原性液)培养基(10g/l的非生物性浮游物、5g/l的酵母提取物及10g/l的氯化钠)中接种所述大肠杆菌,并在旋转振荡器(200rpm)中于37℃下培养一晚上。然后在补充有最终浓度分别为50μg/ml和25μg/ml的氨苄青霉素和氯霉素的、100ml的lb培养基中接种1ml的培养物之后,以200rpm于37℃下进行培养。当od600达到0.5~0.6时,通过添加0.5mm的异丙基-β-d-1-硫代吡喃半乳糖苷(isopropylβ-d-1-thiogalactopyranoside,iptg)而诱导伴娘蛋白和目标蛋白质的表达之后,进一步培养四小时(37℃、200rpm)。然后,当od600值为2时,对细胞培养液3ml(包含约1.6×109个细胞)进行离心分离之后,再悬浮于10mmtris-edta(te)缓冲液(ph7.6)500μl中,并通过超声波处理使其溶解。通过离心分离(21600×g、15分钟、4℃)而分离可溶性及不溶性组分。利用1%tritonx-100来清洗两次不溶性颗粒,并且使其再悬浮于10mm的te缓冲液(ph7.6)之后作为不溶性组分来使用。利用sds聚丙烯酰胺凝胶电泳法(sds-page)来测量目标重组蛋白质的可溶性。通过利用imagej软件来比较用考马斯蓝(coomassieblue)染色后的sds-page凝胶内的总体细胞溶解物及目标重组蛋白的可溶性组分的条带强度,从而计算可溶性目标重组蛋白的相对百分率。
实验例6:蛋白质纯化方法
在补充有最终浓度为25μg/ml的氯霉素的培养基中培养具有pamt7-dnaj-kh的大肠杆菌bl21(de3)。在旋转振荡器(200rpm)中于37℃下培养一晚上所述大肠杆菌。然后在补充有最终浓度为25μg/ml的氯霉素的100ml的lb培养基中接种培养物之后以200rpm于37℃下进行培养。当od600达到0.6时,进一步培养四小时(37℃、200rpm)。然后,通过离心分离获取20ml的细胞,并将该细胞再悬浮于1×nativeimac溶液缓冲液(biorad,hercules,ca,usa),经超声波处理使其溶解。对透明的细胞溶解物再次进行离心分离(21600×g、15分钟、4℃)。利用自动化profiniatm蛋白纯化系统(biorad,hercules,ca,usa)的nativeimac方法,在可溶性组分中对dnaj-kh进行纯化。然后利用含有ph7.4的磷酸缓冲盐溶液(pbs)和5mm的dtt、100mm的氯化钠(nacl)及10%的甘油的、25mm的tris-hcl缓冲液(ph8.8)来对经纯化的dnaj-kh进行透析。
实验例7:gfp互补分析法
在cras系统中对公知的gfp互补分析法(naturemethods,2012,9,671-675)进行局部变形而评价重组蛋白的准确折叠。在补充有最终浓度分别为50μg/ml和25μg/ml的氨苄青霉素和氯霉素的lb培养基中接种具有pamt7-dnajk-kh及pet16b-sfgfp/pet16b-csfgfp-nsfgfp/pet16b-csfgfp-nsfgfp3l的大肠杆菌bl21(de3),并在旋转振荡器(200rpm)中于37℃下培养一晚上。然后在补充有最终浓度分别为50μg/ml和25μg/ml的氨苄青霉素和氯霉素的、100ml的lb培养基中接种1ml的培养物之后,以200rpm于37℃下进行培养。当od600达到0.6时,通过添加0.5mm的iptg而诱导dnajk-kh和目标蛋白质的表达之后,进一步培养(37℃、200rpm)四小时。然后,通过离心分离获取1ml的细胞,并使其再悬浮于500μl的pbs(ph7.4)之后,直至od600达到1为止进行稀释。然后装载到96孔黑色板(spllifesciences,抱川,韩国)中。使用tecaninfinitef200pro仪器(tecangroupltd.,mannedorf,switzerland)按每个孔测量荧光强度(λexc=488nm/λem=530nm)。
实验例8:电泳变化分析法(electricmobilityshiftassay,emsa)
利用hiscribetmt7快速高效rna合成试剂盒(newenglandbiolab,beverly,ma)来制备没有环或具有一个及三个环的、antip21ras-scfvdml的mrna。混合含有0.1nm的mrna的pbs((ph7.4)和200μm的经纯化的dnaj-kh。然后在25℃下进行30分钟的混合物培养,并且利用含有2%的琼脂糖凝胶的tris-boricacid-edta缓冲液且通过电泳进行分析。在50v下执行20分钟的电泳,然后,使用凝胶层析系统(gel-docgeldocumentationsystem)(bio-rad,hercules,ca)对凝胶进行蚀刻化。
实验例9:瘦素活性测量方法
在补充有最终浓度为50μg/ml的氨苄青霉素的lb培养基中使用旋转振荡器(200rpm)于37℃下培养一晚上具有pet16b-dnajlep或pet16b-lep的大肠杆菌bl21(de3)。然后在补充有最终浓度为50μg/ml的氨苄青霉素的100ml的lb培养基中接种1ml的培养物之后以200rpm于37℃下进行培养。当od600达到0.6时,添加0.5mm的iptg之后进一步培养(37℃、200rpm)四小时,以诱导dnaj和瘦素的表达。然后,通过离心分离获取20ml的细胞,并使其再悬浮于1×pbs(ph7.4)之后经超声波处理使其溶解。对透明的细胞溶液再次进行离心分离(21600×g、15分钟、4℃)。使用elisa对纯化后的瘦素的活性进行分析。与碳酸盐-重碳酸盐缓冲液一同在37℃下将透明的细胞溶解物(8mg/ml)的40μg的可溶性组分装载到96孔板(thermofisherscientific,waltham,ma,usa)中一小时。然后利用1×pbs-t(含有0.5%的tween-20的1×pbs)清洗三次板之后,利用牛血清白蛋白(invitrogen,carlsbad,ca,usa)使各孔封闭一小时。然后将抗-瘦素抗体(abcam,cambridge,uk)添加到各孔中,并在常温下培养一小时。在清洗板之后与辣根过氧化物酶结合抗兔单克隆抗体(horseradishperoxide-conjugatedanti-rabbitmonoclonalantibody)(cellsignalingtechnology,beverly,ma,usa)一同在常温下培养一小时。然后利用1×pbs-t清洗四次板之后为了开始过氧化酶反应而添加100μl的tmb(3,3′,5,5′-tetramethylbenzidine,3,3',5,5'-四甲基联苯胺)过氧化酶基质(bd,franklinlakes,nj,usa)。为了停止反应,将50μl的2mh2so4(硫酸)添加到各孔中。利用酶联免疫检测仪(elisareader)(infinitem200pro;tecangroupltd.,mannedorf,switzerland)来测量各孔的450nm的吸光度。
实施例1:在多种不溶性蛋白质中应用cras系统的结果
作为细菌的分子伴娘蛋白系统的必要要素的dnaj与翻译或错误折叠的蛋白质相互作用,为了生产准确折叠的蛋白质而促进如dnak或groesl等的折叠酶(foldase)伴娘蛋白和蛋白质的相互作用。为了翻译后的快速折叠,通过将dnaj配置在目标蛋白质的翻译终止位点上,从而增加dnaj与目标蛋白质的空间接近性。为了固定(anchoring),使来源于人类nova-1蛋白质的序列特异性rna结合域(kh)和dnaj(图1a)结合。通过凝胶阻滞分析法(gelretardationassay)来确认dnaj-kh(dnaj与kh的结合体)和3'utr发夹环的相互作用。为了增加抗-rasscfv(singlechainvariablefragment,单链可变区片段)、scfv与抗菌肽br2的结合体(br2-scfv)、udp-6-葡萄糖脱氢酶(udp-6-glucosedehydrogenase,ugd)、ubic(pyruvatechorismatelyase丙酮酸分支酶)、bmp2(bonemorphogeneticprotein2,骨形态发生蛋白-2)、瘦素、adh1p(alcoholdehydrogenase1p,乙醇脱氢酶1p)及大肠杆菌的hiv-1蛋白质分解酶(hiv1-pr)等的不溶性重组蛋白的溶解度,将上述蛋白质应用到cras(chaperonerecruitingmrnascaffold,分子伴侣mrna支架)系统中。
其结果,与没有空间限制(没有结合环限制)地一同表达蛋白质和dnaj-kh或者单独表达蛋白质的情况相比,在应用cras系统的情况下,大幅增加scfv、br2-scfv及ugd的溶解度(图1b)。
实施例2:在cras系统中与3'utr发夹环的数量及终止密码子及第一个环之间的距离相关的增溶结果
但是,cras系统无法增加adh1p、hiv1-pr、ubic、leptin及bmp2的溶解度(图1b)。为了解决该问题,增加3'utr发夹环的数量。首先,为了将3'utr发夹环制作成所需的结构,通过计算机仿真进行设计及验证(图1c)。
但是,直至三个3'utr发夹环为止对蛋白质的溶解度增加没有显著影响。但是,在该情况下,在18小时的表达期间scfv的可溶性组分比率恒定,与此相反地,具有一个3'utr发夹环的蛋白质溶解度逐渐减少(图2)。这为了提高蛋白质的生产收率而非常重要。
此外,终止密码子与第一个环结构之间的30nt以下的空间对scfv的溶解度增加没有任何影响(图3)。该结果表示系统对终止密码子与dnaj之间的距离具有灵活性。
实施例3:确认在δdnaj菌株和δdnak菌株中应用cras系统的结果
dnak伴娘蛋白系统自然存在于大肠杆菌中,并且由于本发明的系统使dnak伴娘蛋白固定在翻译位点上,因此在大肠杆菌的基因组中使dnaj或dnak缺失之后,利用cras系统来测量scfv溶解度是否增加。只有以mrna为标的的dnaj能够与scfv有效地相互作用,并且由于在δdnak菌株中因折叠酶伴娘蛋白的缺乏而导致cras系统的功能减少,因此预测为在δdnaj菌株中溶解度增加效率相同。但是,虽然δdnaj菌株与预测一致,但δdnak菌株的scfv溶解度稍微增加(图4)。这给出了以下启示:为了能够使单个dnaj作为增加重组蛋白的溶解度的伴娘蛋白来发挥功能,cras系统增加dnaj的折叠酶活性。
实施例4:应用dnaj、dnak及kh结合体(dnaj-dnak-kh)的cras系统的结果
通过限制伴娘蛋白的空间范围的方法和增加各伴娘蛋白的转换数(turnovernumber)的方法,来增加cras系统的效率。为此,制备作为dnaj、dnak及kh结合体的嵌合伴娘蛋白dnaj-dnak-kh(dnajk-kh),并将其应用到cras系统中。
其结果,确认不溶性蛋白质的溶解度大幅增加而且甚至所表达的蛋白质的90%具有可溶性形态(图1d)。此外,确认dnajk-kh及kh发夹环的数量越多,则与dnaj-kh的情况相比目标蛋白质的溶解度在表达蛋白质之后增加得也越快(图1d)。这给出了以下启示:临近的伴娘蛋白的数量与翻译过程一同成为折叠反应的限制因素。
实施例5:cras系统中gfp互补分析结果
为了在invivo中观察目标蛋白质的功能性折叠,通过将超折叠绿色荧光蛋白(sfgfp)(bioneer,大田,韩国)分为n-末端(n-sfgfp)和c-末端(c-sfgfp)而设计gfp互补性分析法(图1e)。n-sfgfp的溶解度较低,被选定为证明cras系统功能的目标蛋白质。荧光水平是指增溶后的功能性n-sfgfp的水平。表达dnajk-kh及与此相互作用的n-sfgfp的同时表达c-sfgfp,则使同时表达两个片段的细胞荧光强度增加两倍,与此相反地,在与dnaj-kh相互作用的n-sfgfp的3'utr中导入三个环后,荧光强度增加约八倍。该数值为相当于由sfgfp的全长蛋白质放出的荧光强度的90%的数值。这表示cras系统不仅能增加溶解度,而且还能增加功能性活性蛋白质的表达(图1f)。
实施例6:clex系统应用结果
在不限制与翻译机制临近的伴娘蛋白的数量的情况下,为了证明关于伴娘蛋白和基质的空间制约的重要性,制备能够增溶不溶性非常强的蛋白质的、作为翻译上结合的两个顺反子表达系统(translationallycoupledtwo-cistronexpressionsystem)的clex(chaperone-substrateco-localizedexpression)系统(图5a)。通过第一个及第二个顺反子的终止密码子和起始密码子的重叠(5'-taatg-3'),核糖体复合体不会脱离转录物,并且能够对第二个顺反子进行翻译。此外,在clex系统中,dnaj被连接到第一个或第二个顺反子的对目标蛋白质进行编码的基因上,第一个顺反子为了第二个顺反子而被设计成具有包含核糖体结合位点(rbs)的5'-gaggaggtggaa-3'序列(序列号79)(下划线序列:rbs或sd序列(shine-dalgarnosequence))。关于两个顺反子之间的整合序列,设计重叠后的两个顺反子、串联形态的两个顺反子及具有nbp距离的两个顺反子(图5b)。
其结果,由于具有重叠后的终止-起始密码子的clex系统对不溶性非常强的包括未被cras系统增溶的bmp2在内的蛋白质的增溶非常有效。平均60%至90%以上的重组蛋白质可以以可溶性形态获得(图5c)。特别是,在瘦素的情况下,根据实验例9的方法利用结构特异性抗体来确认准确折叠后的状态(图5d)。
此外,确认到dnaj的作用根据具有表达水平及反应时间的差异的两个顺反子的顺序而不同(图5c)。当由第一个顺反子表达伴娘蛋白时确认到更多的可溶性组分。这是因为,在翻译第二个顺反子之前可以准备能够利用的伴娘蛋白。此外,在dnaj-bmp2排列的情况下呈现出比该排列的逆序更长的长期蛋白质表达(直至诱导后18小时为止)(图5e)。这给出了以下启示:伴娘蛋白与蛋白质之间的适时的相互作用比与伴娘蛋白反应相关联的结构成分量更重要。
此外,确认到能够通过使基因之间的序列增加至10nt而增加蛋白质的溶解度(图5f)。附加到两个基因之间的核苷酸不仅改变两个蛋白质之间的理论配比(stoichiometricratio),而且通过改变dnaj和目标重组蛋白的接近性而能够变换转录物的二次结构及用于开始第二个顺反子的翻译的亲和度。
实施例6:在δdnak菌株中应用clex系统的结果
在增溶clex系统的重组蛋白时,为了确认dnak的作用而利用δdnak菌株(图6)。
其结果,确认到在clex系统中dnaj能够充分增溶bmp2蛋白质而无需dnak。这给出了以下启示:dnaj对dnak具有非依赖性的折叠酶活性,或者在大肠杆菌内折叠蛋白质时参与其他伴娘蛋白系统。由此,可知利用单个伴娘蛋白dnaj的clex系统具有非常高的效率。
实施例7:在tlia1中应用clex系统的结果
虽然对多种重组蛋白成功进行了增溶,但在如tlia(52kda脂肪分解酶)的高分子蛋白质的溶解度增加中只能够获得微小的增加。被判断为这是因为高分子蛋白质有可能会具有容易错误折叠的多个结构域,并且本发明的系统在不可逆的错误折叠之前不足以促进伴娘蛋白与多个基质之间的相互作用。为了确认该判断,在将tlia基因切断成两片(tlia1、tlia2)之后应用到本发明的系统中。
其结果,tlia1具有很大的不溶性,与此相反地tlia2具有可溶性(图3a及图3b)。在第二个顺反子中表达tila1的clex系统的情况下增溶约60%的tlia1,与此相反地在单纯一同表达tlia1和dnaj的情况下增溶25%的tlia1(图3b)。该结果给出了以下启示:如蛋白质长度及结构域排列等的tlia的内在特性为不溶性的主要原因,能够通过使内部的容易错误折叠的区域充分暴露于伴娘蛋白的本发明的系统来增加高分子蛋白质的溶解度。
cras系统使伴娘蛋白位于mrna的3'utr上,与此相反地clex系统通过与目标蛋白质一同继续生产新的伴娘蛋白分子而促进它们的快速相互作用。当考虑增溶活性时,clex系统比cras系统更优异。但是,由于clex系统需要按各重组蛋白将dnaj克隆为两个顺反子系统,因此使多个蛋白质同时增溶是有限的。与此相反地,具有三个环及dnajk-kh的cras系统可通过在各mrna中简单导入3'utrkh结合发夹序列而提高细胞内表达的多个蛋白质的溶解度。因此,cras系统在增加参与代谢路径的功能酶的增溶组分时使用,clex系统更适合用于生成治疗用肽、酶或如肽等的一种重组蛋白。
本发明所属技术领域的技术人员应能够从上述说明中理解本发明在不变更其技术思想或必要特征的情况下可以以其他具体形态实施。与此相关联地,应理解为以上所述的实施例在所有方面是示例而不是限定性的。关于本发明的范围应解释为从所附的权利要求书的含义及范围以及其等同的概念导出的所有变更或变形后的形态包含在本发明的范围内。
<110>智能合成生物中心
<120>用于增溶目标蛋白质的组合物及其用途
<130>opa18006-cn
<160>79
<170>kopatentin3.0
<210>1
<211>54
<212>dna
<213>人工序列
<220>
<223>dnajncof
<400>1
tgataaccatggaagattctacggttaacacaatggctaagcaagattattacg54
<210>2
<211>30
<212>dna
<213>人工序列
<220>
<223>dnajxhor
<400>2
atttcactcgaggcgggtcaggtcgtcaaa30
<210>3
<211>94
<212>dna
<213>人工序列
<220>
<223>khncof1
<400>3
taatgaccatggaaaccgacggttctaaagacgttgttgaaatcgctgttccggaaaacc60
tggttggtgctatcctgggtaaaggtggtaaaac94
<210>4
<211>92
<212>dna
<213>人工序列
<220>
<223>khrecr1
<400>4
gcccggaacaaattcaccttttttagaaatctggatacgagcacctgtcagttcctggta60
ttcaaccagggttttaccacctttacccagga92
<210>5
<211>92
<212>dna
<213>人工序列
<220>
<223>khrecf2
<400>5
aaaaggtgaatttgttccgggcacccgtaaccgtaaagttaccatcacaggcaccccggc60
tgctacccaggctgctcagtacctgatcacac92
<210>6
<211>90
<212>dna
<213>人工序列
<220>
<223>khxhor2
<400>6
ttatcactcgagttaaccaactttctgcgggttagcagcacgaacaccctgttcgtaggt60
gatacgctgtgtgatcaggtactgagcagc90
<210>7
<211>48
<212>dna
<213>人工序列
<220>
<223>dnajlinkr
<400>7
gctgccgccaccaccgctaccgccaccgccgcgggtcaggtcgtcaaa48
<210>8
<211>42
<212>dna
<213>人工序列
<220>
<223>linkkhf
<400>8
ggtagcggtggtggcggcagcaccgacggttctaaagacgtt42
<210>9
<211>33
<212>dna
<213>人工序列
<220>
<223>khxhor
<400>9
atttcactcgagttaaccaactttctgcgggtt33
<210>10
<211>77
<212>dna
<213>人工序列
<220>
<223>kh-6xhis-ecor
<400>10
cgattaggatcctcatcattaatgatggtggtgatggtgagatccacgcggaaccagacc60
aactttctgcgggttag77
<210>11
<211>51
<212>dna
<213>人工序列
<220>
<223>dnajr
<400>11
agaacctccgccgccagaacccccgccaccgcgggtcaggtcgtcaaaaaa51
<210>12
<211>43
<212>dna
<213>人工序列
<220>
<223>dnakf
<400>12
tctggcggcggaggttctggtaaaataattggtatcgacctgg43
<210>13
<211>42
<212>dna
<213>人工序列
<220>
<223>dnakr
<400>13
gctaccgccaccgccttttttgtctttgacttcttcaaattc42
<210>14
<211>24
<212>dna
<213>人工序列
<220>
<223>khf
<400>14
gacaaaaaaggcggtggcggtagc24
<210>15
<211>28
<212>dna
<213>人工序列
<220>
<223>scfvndef
<400>15
tgataacatatgcaggtccaactgcagc28
<210>16
<211>59
<212>dna
<213>人工序列
<220>
<223>scfvxhor
<400>16
tcattactcgagtcatcattagtggtggtggtggtggtgtttgatctccagcttggtcc59
<210>17
<211>96
<212>dna
<213>人工序列
<220>
<223>scfv1lxhor
<400>17
ccgttactcgagccgcgcggggtgatctaggtccgcgcggtcgtcgtcgtcatcattagt60
ggtggtggtggtggtgtttgatctccagcttggtcc96
<210>18
<211>80
<212>dna
<213>人工序列
<220>
<223>3l-1r
<400>18
aggtgagcaacggacatccttcacgggtgatctaggtcgtgaaggctcgatcgtcatcat60
tagtggtggtggtggtggtg80
<210>19
<211>87
<212>dna
<213>人工序列
<220>
<223>3lxhor
<400>19
ccgttactcgagtcgtagagcggtgatctaggtgctctacggactgcgttgctcggtgat60
ctaggtgagcaacggacatccttcacg87
<210>20
<211>28
<212>dna
<213>人工序列
<220>
<223>br2scfvndef
<400>20
tgataacatatgcgtgctggtctgcagt28
<210>21
<211>28
<212>dna
<213>人工序列
<220>
<223>br2scfvndef
<400>21
tgataacatatgcgtgctggtctgcagt28
<210>22
<211>29
<212>dna
<213>人工序列
<220>
<223>ugdndef
<400>22
tgataacatatgaaaatcaccatttccgg29
<210>23
<211>55
<212>dna
<213>人工序列
<220>
<223>ugdxhor
<400>23
ccgttactcgagttattagtggtggtggtggtggtggtcgctgccaaagagatcg55
<210>24
<211>95
<212>dna
<213>人工序列
<220>
<223>ugd1lxhor
<400>24
ccgttactcgagccgcgcggggtgatctaggtccgcgcggtcgtcgtcgtcatcattagt60
ggtggtggtggtggtggtcgctgccaaagagatcg95
<210>25
<211>32
<212>dna
<213>人工序列
<220>
<223>adhndef
<400>25
tgataacatatgtctatcccagaaactcaaaa32
<210>26
<211>60
<212>dna
<213>人工序列
<220>
<223>adhxhor
<400>26
ccgttactcgagttattagtggtggtggtggtggtgtttagaagtgtcaacaacgtatct60
60
<210>27
<211>100
<212>dna
<213>人工序列
<220>
<223>adh1lxhor
<400>27
ccgttactcgagccgcgcggggtgatctaggtccgcgcggtcgtcgtcgtcatcattagt60
ggtggtggtggtggtgtttagaagtgtcaacaacgtatct100
<210>28
<211>80
<212>dna
<213>人工序列
<220>
<223>3l-1r
<400>28
aggtgagcaacggacatccttcacgggtgatctaggtcgtgaaggctcgatcgtcatcat60
tagtggtggtggtggtggtg80
<210>29
<211>87
<212>dna
<213>人工序列
<220>
<223>3lxhor
<400>29
ccgttactcgagtcgtagagcggtgatctaggtgctctacggactgcgttgctcggtgat60
ctaggtgagcaacggacatccttcacg87
<210>30
<211>33
<212>dna
<213>人工序列
<220>
<223>ubicndef
<400>30
tgataacatatgcgattgttgcgtttttgttgc33
<210>31
<211>56
<212>dna
<213>人工序列
<220>
<223>ubicxhor
<400>31
ccgttactcgagttattagtggtggtggtggtggtggtacaacggtgacgccggta56
<210>32
<211>96
<212>dna
<213>人工序列
<220>
<223>ubic1lxhor
<400>32
ccgttactcgagccgcgcggggtgatctaggtccgcgcggtcgtcgtcgtcatcattagt60
ggtggtggtggtggtggtacaacggtgacgccggta96
<210>33
<211>80
<212>dna
<213>人工序列
<220>
<223>3l-1r
<400>33
aggtgagcaacggacatccttcacgggtgatctaggtcgtgaaggctcgatcgtcatcat60
tagtggtggtggtggtggtg80
<210>34
<211>87
<212>dna
<213>人工序列
<220>
<223>3lxhor
<400>34
ccgttactcgagtcgtagagcggtgatctaggtgctctacggactgcgttgctcggtgat60
ctaggtgagcaacggacatccttcacg87
<210>35
<211>83
<212>dna
<213>人工序列
<220>
<223>hivprxbaf
<400>35
attctaaatctagattattcactacgcgttaaggaggtacgacatgcaccatcaccacca60
tcatcctcaaatcaccctgtggc83
<210>36
<211>85
<212>dna
<213>人工序列
<220>
<223>hivprxhor
<400>36
ccgaattactcgagtcatcattagaagttcagggtgcaaccgatctgggtcagcatgtta60
cgaccgatgatgttgatcggggtcg85
<210>37
<211>77
<212>dna
<213>人工序列
<220>
<223>hivpr1lxhor
<400>37
ccgttactcgagccgcgcggggtgatctaggtccgcgcggtcgtcgtcgtcatcattaga60
agttcagggtgcaaccg77
<210>38
<211>78
<212>dna
<213>人工序列
<220>
<223>hivprxho3lr
<400>38
ccgaattactcgagccgcgcggggtgatctaggtccgcgcggtcgtcgtcgtcatcatta60
gaagttcagggtgcaacc78
<210>39
<211>33
<212>dna
<213>人工序列
<220>
<223>lepndef
<400>39
tgataacatatgcgattgttgcgtttttgttgc33
<210>40
<211>56
<212>dna
<213>人工序列
<220>
<223>lepxhor
<400>40
ccgttactcgagttattagtggtggtggtggtggtggtacaacggtgacgccggta56
<210>41
<211>96
<212>dna
<213>人工序列
<220>
<223>lep1lxhor
<400>41
ccgttactcgagccgcgcggggtgatctaggtccgcgcggtcgtcgtcgtcatcattagt60
ggtggtggtggtggtggtacaacggtgacgccggta96
<210>42
<211>80
<212>dna
<213>人工序列
<220>
<223>3l-1r
<400>42
aggtgagcaacggacatccttcacgggtgatctaggtcgtgaaggctcgatcgtcatcat60
tagtggtggtggtggtggtg80
<210>43
<211>87
<212>dna
<213>人工序列
<220>
<223>3lxhor
<400>43
ccgttactcgagtcgtagagcggtgatctaggtgctctacggactgcgttgctcggtgat60
ctaggtgagcaacggacatccttcacg87
<210>44
<211>30
<212>dna
<213>人工序列
<220>
<223>bmp2ndef
<400>44
tgataacatatgcaagccaaacacaaacag30
<210>45
<211>54
<212>dna
<213>人工序列
<220>
<223>bmp2xhor
<400>45
ccgttactcgagttattagtggtggtggtggtggtggcgacacccacaaccctc54
<210>46
<211>93
<212>dna
<213>人工序列
<220>
<223>bmp21lxhor
<400>46
ccgttactcgagccgcgcggggtgatctaggtccgcgcggtcgtcgtcgtcatcattagt60
ggtggtggtggtggtgcgacacccacaaccctc93
<210>47
<211>80
<212>dna
<213>人工序列
<220>
<223>3l-1r
<400>47
aggtgagcaacggacatccttcacgggtgatctaggtcgtgaaggctcgatcgtcatcat60
tagtggtggtggtggtggtg80
<210>48
<211>30
<212>dna
<213>人工序列
<220>
<223>sfgfpndef
<400>48
tgataacatatgcaagccaaacacaaacag30
<210>49
<211>40
<212>dna
<213>人工序列
<220>
<223>sfgfpecor
<400>49
aggtcagaattctcatcattacgtaatacctgccgcattc40
<210>50
<211>68
<212>dna
<213>人工序列
<220>
<223>csfgfpxbaf
<400>50
ccatgatctagaaataattttgtttaactttaagaaggagatataccatggtgcccatcc60
aaaaagtc68
<210>51
<211>42
<212>dna
<213>人工序列
<220>
<223>csfgfpnder
<400>51
ccgttacatatgtcatcattatgtaatcccagcagcatttac42
<210>52
<211>30
<212>dna
<213>人工序列
<220>
<223>sfgfpndef
<400>52
tgataacatatgcaagccaaacacaaacag30
<210>53
<211>44
<212>dna
<213>人工序列
<220>
<223>sfngfpecor
<400>53
aggtcagaattctcatcattatttttcgttcggatctttagaca44
<210>54
<211>85
<212>dna
<213>人工序列
<220>
<223>sfngfp3l-1r
<400>54
aggtgagcaacggacatccttcacgggtgatctaggtcgtgaaggctcgatcgtcatcat60
tatttttcgttcggatctttagaca85
<210>55
<211>86
<212>dna
<213>人工序列
<220>
<223>sfngfp3lecor
<400>55
aggtcagaattctcgtagagcggtgatctaggtgctctacggactgcgttgctcggtgat60
ctaggtgagcaacggacatccttcac86
<210>56
<211>31
<212>dna
<213>人工序列
<220>
<223>dnajndef
<400>56
tgataacatatggctaagcaagattattacg31
<210>57
<211>55
<212>dna
<213>人工序列
<220>
<223>scfvfpol
<400>57
gttttttgacgacctgacccgcgaggaggtggaataatgcaggtccaactgcagc55
<210>58
<211>54
<212>dna
<213>人工序列
<220>
<223>dnajfpol
<400>58
caccaccaccaccaccacgaggaggtggaataatggctaagcaagattattacg54
<210>59
<211>60
<212>dna
<213>人工序列
<220>
<223>ubicfpol
<400>59
gttttttgacgacctgacccgcgaggaggtggaataatgcgattgttgcgtttttgttgc60
60
<210>60
<211>76
<212>dna
<213>人工序列
<220>
<223>hivprfpol
<400>60
gttttttgacgacctgacccgcgaggaggtggaataatgcaccatcaccaccatcatcct60
caaatcaccctgtggc76
<210>61
<211>55
<212>dna
<213>人工序列
<220>
<223>dnajfpolhivpr
<400>61
cggttgcaccctgaacttcgaggaggtggaataatggctaagcaagattattacg55
<210>62
<211>60
<212>dna
<213>人工序列
<220>
<223>lepfpol
<400>62
gttttttgacgacctgacccgcgaggaggtggaataatgcgattgttgcgtttttgttgc60
60
<210>63
<211>57
<212>dna
<213>人工序列
<220>
<223>bmp2fpol
<400>63
gttttttgacgacctgacccgcgaggaggtggaataatgcaagccaaacacaaacag57
<210>64
<211>59
<212>dna
<213>人工序列
<220>
<223>adhfpol
<400>64
gttttttgacgacctgacccgcgaggaggtggaataatgtctatcccagaaactcaaaa59
<210>65
<211>43
<212>dna
<213>人工序列
<220>
<223>tliandef
<400>65
tgataacatatgcatcatcatcatcatcatcatcatcacagca43
<210>66
<211>42
<212>dna
<213>人工序列
<220>
<223>tliaecor
<400>66
ctgagaattctcatcattaactgatcagcacaccctcgctcc42
<210>67
<211>70
<212>dna
<213>人工序列
<220>
<223>tliafpol
<400>67
gttttttgacgacctgacccgcgaggaggtggaataatgcatcatcatcatcatcatcat60
catcacagca70
<210>68
<211>59
<212>dna
<213>人工序列
<220>
<223>dnajtliafpol
<400>68
ggagcgagggtgtgctgatcagtgaggaggtggaataatggctaagcaagattattacg59
<210>69
<211>34
<212>dna
<213>人工序列
<220>
<223>dnajecor
<400>69
ctgagaattcttattagcgggtcaggtcgtcaaa34
<210>70
<211>39
<212>dna
<213>人工序列
<220>
<223>tlia1ecor
<400>70
ccgttagaattctcatcattagtcggtggtcgactcgtg39
<210>71
<211>71
<212>dna
<213>人工序列
<220>
<223>tlia1fpol
<400>71
agttttttgacgacctgacccgcaccggaggtacataatgcatcatcatcatcatcatca60
tcatcacagca71
<210>72
<211>39
<212>dna
<213>人工序列
<220>
<223>tlia1ecor
<400>72
ccgttagaattctcatcattagtcggtggtcgactcgtg39
<210>73
<211>31
<212>dna
<213>人工序列
<220>
<223>tlia2ndef
<400>73
tgataacatatgaacatcgtcagcttcaacg31
<210>74
<211>1131
<212>dna
<213>大肠杆菌k-12
<400>74
atggctaagcaagattattacgagattttaggcgtttccaaaacagcggaagagcgtgaa60
atcagaaaggcctacaaacgcctggccatgaaataccacccggaccgtaaccagggtgac120
aaagaggccgaggcgaaatttaaagagatcaaggaagcttatgaagttctgaccgactcg180
caaaaacgtgcggcatacgatcagtatggtcatgctgcgtttgagcaaggtggcatgggc240
ggcggcggttttggcggcggcgcagacttcagcgatatttttggtgacgttttcggcgat300
atttttggcggcggacgtggtcgtcaacgtgcggcgcgcggtgctgatttacgctataac360
atggagctcaccctcgaagaagctgtacgtggcgtgaccaaagagatccgcattccgact420
ctggaagagtgtgacgtttgccacggtagcggtgcaaaaccaggtacacagccgcagact480
tgtccgacctgtcatggttctggtcaggtgcagatgcgccagggattcttcgctgtacag540
cagacctgtccacactgtcagggccgcggtacgctgatcaaagatccgtgcaacaaatgt600
catggtcatggtcgtgttgagcgcagcaaaacgctgtccgttaaaatcccggcaggggtg660
gacactggagaccgcatccgtcttgcgggcgaaggtgaagcgggcgagcatggcgcaccg720
gcaggcgatctgtacgttcaggttcaggttaaacagcacccgattttcgagcgtgaaggc780
aacaacctgtattgcgaagtcccgatcaacttcgctatggcggcgctgggtggcgaaatc840
gaagtaccgacccttgatggtcgcgtcaaactgaaagtgcctggcgaaacccagaccggt900
aagctattccgtatgcgcggtaaaggcgtcaagtctgtccgcggtggcgcacagggtgat960
ttgctgtgccgcgttgtcgtcgaaacaccggtaggcctgaacgaaaggcagaaacagctg1020
ctgcaagagctgcaagaaagcttcggtggcccaaccggcgagcacaacagcccgcgctca1080
aagagcttctttgatggtgtgaagaagttttttgacgacctgacccgctaa1131
<210>75
<211>1917
<212>dna
<213>大肠杆菌k-12
<400>75
atgggtaaaataattggtatcgacctgggtactaccaactcttgtgtagcgattatggat60
ggcaccactcctcgcgtgctggagaacgccgaaggcgatcgcaccacgccttctatcatt120
gcctatacccaggatggtgaaactctagttggtcagccggctaaacgtcaggcagtgacg180
aacccgcaaaacactctgtttgcgattaaacgcctgattggtcgccgcttccaggacgaa240
gaagtacagcgtgatgtttccatcatgccgttcaaaattattgctgctgataacggcgac300
gcatgggtcgaagttaaaggccagaaaatggcaccgccgcagatttctgctgaagtgctg360
aaaaaaatgaagaaaaccgctgaagattacctgggtgaaccggtaactgaagctgttatc420
accgtaccggcatactttaacgatgctcagcgtcaggcaaccaaagacgcaggccgtatc480
gctggtctggaagtaaaacgtatcatcaacgaaccgaccgcagctgcgctggcttacggt540
ctggacaaaggcactggcaaccgtactatcgcggtttatgacctgggtggtggtactttc600
gatatttctattatcgaaatcgacgaagttgacggcgaaaaaaccttcgaagttctggca660
accaacggtgatacccacctggggggtgaagacttcgacagccgtctgatcaactatctg720
gttgaagaattcaagaaagatcagggcattgacctgcgcaacgatccgctggcaatgcag780
cgcctgaaagaagcggcagaaaaagcgaaaatcgaactgtcttccgctcagcagaccgac840
gttaacctgccatacatcactgcagacgcgaccggtccgaaacacatgaacatcaaagtg900
actcgtgcgaaactggaaagcctggttgaagatctggtaaaccgttccattgagccgctg960
aaagttgcactgcaggacgctggcctgtccgtatctgatatcgacgacgttatcctcgtt1020
ggtggtcagactcgtatgccaatggttcagaagaaagttgctgagttctttggtaaagag1080
ccgcgtaaagacgttaacccggacgaagctgtagcaatcggtgctgctgttcagggtggt1140
gttctgactggtgacgtaaaagacgtactgctgctggacgttaccccgctgtctctgggt1200
atcgaaacgatgggcggtgtgatgacgacgctgatcgcgaaaaacaccactatcccgacc1260
aagcacagccaggtgttctctaccgctgaagacaaccagtctgcggtaaccatccatgtg1320
ctgcagggtgaacgtaaacgtgcggctgataacaaatctctgggtcagttcaacctagat1380
ggtatcaacccggcaccgcgcggcatgccgcagatcgaagttaccttcgatatcgatgct1440
gacggtatcctgcacgtttccgcgaaagataaaaacagcggtaaagagcagaagatcacc1500
atcaaggcttcttctggtctgaacgaagatgaaatccagaaaatggtacgcgacgcagaa1560
gctaacgccgaagctgaccgtaagtttgaagagctggtacagactcgcaaccagggcgac1620
catctgctgcacagcacccgtaagcaggttgaagaagcaggcgacaaactgccggctgac1680
gacaaaactgctatcgagtctgcgctgactgcactggaaactgctctgaaaggtgaagac1740
aaagccgctatcgaagcgaaaatgcaggaactggcacaggtttcccagaaactgatggaa1800
atcgcccagcagcaacatgcccagcagcagactgccggtgctgatgcttctgcaaacaac1860
gcgaaagatgacgatgttgtcgacgctgaatttgaagaagtcaaagacaaaaaataa1917
<210>76
<211>276
<212>dna
<213>人工序列
<220>
<223>nova-1kh3rna结合域
<400>76
accgacggttctaaagacgttgttgaaatcgctgttccggaaaacctggttggtgctatc60
ctgggtaaaggtggtaaaaccctggttgaataccaggaactgacaggtgctcgtatccag120
atttctaaaaaaggtgaatttgttccgggcacccgtaaccgtaaagttaccatcacaggc180
accccggctgctacccaggctgctcagtacctgatcacacagcgtatcacctacgaacag240
ggtgttcgtgctgctaacccgcagaaagttggttaa276
<210>77
<211>12
<212>dna
<213>人工序列
<220>
<223>具有用于khrna结合域的结合位点的环
<400>77
acctagatcacc12
<210>78
<211>4
<212>prt
<213>人工序列
<220>
<223>导入第一个顺反子的c-末端处的区域
<400>78
glugluvalglu
1
<210>79
<211>12
<212>dna
<213>人工序列
<220>
<223>导入第一个顺反子的c-末端处的区域
<400>79
gaggaggtggaa12
- 还没有人留言评论。精彩留言会获得点赞!