circRNA_27455及其检测试剂与应用的制作方法
本发明涉及生物技术领域,具体涉及circrna_27455、含circrna_27455检测试剂及其在检测猪肌内脂肪中的应用。
背景技术:
circrna是一类由特殊选择性剪切产生的环形内源性分子,呈共价闭合的环形结构,没有5'和3'极性,不能被核糖核酸酶降解。在哺乳动物细胞中,circrna分布广泛,含量丰富,且具有稳定性,保守性和组织特异性。已经有研究发现circrna与多种疾病相关。但是,circrna是否参与脂肪沉积与代谢未见于报道。
随着分子生物学的发展,基因检测的技术在提高畜牧业育种、改善牲畜肉质等方面都显示出很大的潜力。目前研究已经发现部分和猪肉品质相关的基因,但是仍有大量的新基因有待发现,现有的基因远远无法满足实际生产应用的需求,故而,本申请结合最新的分子标志物环状rna,期望寻找一些新的和猪肉品质相关的基因。
肌内脂肪是指沉积在肌肉内的脂肪,它和肉质呈正相关。肌内脂肪之所以引起人们越来越大的兴趣,是因为它影响肉质的嫩度和多汁性,特别是肉的多汁性。当肌内脂肪含量低于2%时,肉的质地和口感都较差,因此,研究肌内脂肪的生成,对改善肉食香味、提高猪肉的食用价值以及生产有利于人体健康的肉产品具有重要的意义。
本申请选择了大白猪和莱芜猪,前者属于典型的瘦肉型猪种,肌内脂肪含量低,而后者肌内脂肪含量高;二者在脂肪沉积方面的显著差异为脂肪代谢的机制研究提供了良好模型。结合二代测序及生物信息学分析的方法,本申请寻找到部分调控猪脂肪沉积的circrnas,为选育具有不同肌肉品质的种猪提供理论基础和机理指导。
技术实现要素:
一种与猪肌内脂肪相关的环状rna,命名为circrna_27455,序列与seqidno.1具有95%以上序列同源性。
优选的,circrna_27455序列为seqidno.1。
一种检测肌内脂肪的试剂,检测circrna_27455的表达水平。
进一步,采用测序技术、核酸杂交技术或核酸扩增技术检测样品中circrna_27455的表达水平。
优选的,采用高通量测序技术、探针杂交技术、基因芯片技术或荧光定量pcr技术检测样品中circrna_27455的表达水平。
优选的,试剂包含检测circrna_27455的探针或用于荧光定量pcr的引物。
优选的,荧光定量pcr检测引物序列为seqidno.2和seqidno.3。
进一步,样品为组织,优选的,样品为肌内脂肪组织,更优选的,样品为猪肌内脂肪组织。
进一步,所述核酸扩增技术选自聚合酶链式反应(pcr)、逆转录聚合酶链式反应(rt-pcr)、转录介导的扩增(tma)、连接酶链式反应(lcr)、链置换扩增(sda)和基于核酸序列的扩增(nasba)。其中,pcr需要在扩增前将rna逆转录成dna(rt-pcr),tma和nasba直接扩增rna。
本发明中的核酸杂交技术包括但不限于原位杂交(ish)、微阵列和southern或northern印迹。
本发明的目的在于提供下述任意一项应用:
circrna_27455在预测或辅助预测猪肉品质中的应用;
circrna_27455在制备预测或辅助预测猪肉品质试剂中的应用;
circrna_27455在选育具有不同肌肉品质猪中的应用;
上述试剂在预测或辅助预测猪肉品质中的应用;
上述试剂在制备预测或辅助预测猪肉品质试剂中的应用;
上述试剂在选育具有不同肌肉品质猪中的应用。
一种检测猪肌内脂肪含量的方法,包括:(1)选取猪肌内脂肪组织;(2)检测样品中circrna_27455的表达量。
定义:
术语“同源”是主要是指序列上的同源,也就是用来说明两个或多个rna或dna序列具有相同的祖先。同源的序列一般有相似的功能。一般来说,当相似程度高于50%时,常推测检测序列和目标序列可能是同源序列;当相似性程度低于20%时,就难以确定其是否具有同源性。
环状rna(circularrna,circrna),亦称环形rna,是最近几年研究确认的一种新型的非编码rna(noncodingrna,ncrna)分子。根据rna构成的不同,环状rna可分为三类:外显子环状rna(exoncircularrna,ecircrna)、内含子环状rna(circularintronicrnas,cirnas)和外显子-内含子circrna(exon-introncircrna,eicirna)。
本发明中“探针”指能与另一分子的特定序列或亚序列或其它部分结合的分子。除非另有指出,术语“探针”通常指能通过互补碱基配对与另一多核苷酸结合的多核苷酸探针。根据杂交条件的严谨性,探针能和与该探针缺乏完全序列互补性的靶多核苷酸结合。探针可作直接或间接的标记,其范围包括引物。
所述探针具有与靶点基因的特定的碱基序列互补的碱基序列。这里,所谓“互补”,只要是杂交即可,可以不是完全互补。这些多核苷酸通常相对于该特定的碱基序列具有90%以上、更优选95%以上、特别优选100%的同源性。这些探针可以是dna,也可以是rna,另外,可以为在其一部分或全部中核苷酸通过pna(polyamidenucleicacid,肽核酸)、lna(注册商标,lockednucleicacid,bridgednucleicacid,交联化核酸)、ena(注册商标,2′-o,4′-c-ethylene-bridgednucleicacids)、gna(glycerolnucleicacid,甘油核酸)、tna(threosenucleicacid,苏糖核酸)等人工核酸置换得到的多核苷酸。
本发明中的术语“杂交”用于指代互补核酸的配对。杂交和杂交强度受诸如以下的因素影响:核酸之间的互补程度、所涉及的条件的严格性、形成的杂交体的tm和核酸内的g:c比率。
测序技术主要为高通量测序技术(high-throughputsequencing),又称下一代测序技术(nextgenerationsequencing),一次对几十万到几百万条dna分子进行序列测定,极大提高了测序效率。高通量测序平台的代表是罗氏公司(roche)的454测序仪(rochgsflxsequencer),illumina公司的solexa基因组分析仪(illuminagenomeanalyzer)和abi的solid测序仪(abisolidsequencer)。
附图说明
图1是肌内脂肪差异表达circrna分类图:a为上调的差异表达circrna分类图,b为下调的差异表达circrna分类图
图2是荧光定量验证候选基因差异表达图
具体实施方式
下面结合附图和实施例对本发明作进一步详细的说明。以下实施例仅用于说明本发明而不用于限制本发明的范围。
实施例1circrna测序及差异表达circrna挑选
1.1实验动物:
该试验以脂肪沉积存在明显差异的大白猪和莱芜猪为材料,饲养于莱芜市大千农牧有限公司,在相同饲养条件和环境下生长育肥,参照营养需要标准(nationalresearchcouncil,nrc,1998)饲喂日粮,选择180日龄、种内体重接近的大白猪(约100kg)和莱芜猪(约35kg)各3只,且健康、体质优良。
1.2样品采集:
屠宰实验猪后,迅速将肌内脂肪组织分离。为减少rna的降解,所有过程在冰上进行。之后用消毒剪刀将组织剪成小块,分别装入5ml冻存管,放入液氮冷冻,后转移至-80℃冰箱保存,用于提取总rna。实验设置分为2组,分别对大白猪肌内脂肪组织(d_jn)与莱芜猪肌内脂肪组织(l_jn)中的circrna进行鉴定及分析,每个样本设置3个生物学重复。
1.3样品总rna的提取及质控:
取出等量低温保存的脂肪组织,使用qiagen抽提试剂盒提取rna,提取的总rna保存于-80℃冰箱。用nanodrop2000紫外分光光度计测定rna样品的浓度和od260nm/od280nm比值,比值在1.9~2.1间,bioanalyzer2100检测总rna的质量,且rin≥7、28s/18s≥0.7,用rnase-freednaseⅰ除去基因组dna污染。
1.4circrna测序建库:
(1)ribo-zerokit去除rrna及rna的片段化;(2)核糖核酸酶r去除线性rna;(3)双链cdna的合成与纯化;(4)末端修复,加a碱基;(5)测序接头连接;(6)dna片段富集纯化;(7)文库的质检;(8)本试验共建立6个cdna文库(大白猪和莱芜猪肌内脂肪组织cdna文库),分别为d_jn_1、d_jn_2、d_jn_3、l_jn_1、l_jn_2、l_jn_3,文库质检合格后,运用illuminahiseqtm2500测序平台进行双端测序,对文库进行测序分析,下机得到的数据是原始测序数据。
1.5原始数据质控及过滤:
主要应用ngsqctoolkit进行质控并去除接头,后续的分析以cleanreads为基础。具体步骤如下:
(1)首先过滤低质量的reads,质量阈值设置为20,过滤长度阈值设置为70%。
(2)然后从5’端及3’端去除低质量碱基,质量阈值设置为20。
(3)最后切除reads中含n部分序列,长度阈值设置为35bp。
1.6circrna鉴定:
使用bwa软件与参考基因序列比对,生成sam文件,对文件中的cigar值进行分析,并从sam文件扫描pcc信号(pairedchiasticclippingsignals)。cigar值在junctionread的特征为xs/hym或者xmys/h,其中x,y代表碱基数目,m是mapping,s是softclipping,h是hardclipping。关于双端reads,ciri算法考虑一对reads,其中一条mapping到circrna上,另一条也需mapping到circrna的区间内。关于单外显子成环,或“长外显子1-短外显子-长外显子2”成环,cigar值应该是xs/hymzs/h以及(x+y)s/hzm或者xm(y+z)s/h,ciri软件能够将这两种情况分开。关于splicing信号(gt,ag)ciri会考虑其它弱splicing信息例如(at-ac)。算法:从gtf/gff文件中抽取外显子边界位置,用已知边界来过滤假阳性。
1.7circrna注释:
将circrna与基因元件进行比较,获取circrna在基因组上的位置信息。利用circrna位置信息与已知数据库中的蛋白编码基因注释信息,对circrna进行注释,获取序列信息,详细处理过程如下:
(1)利用intersectbed软件,根据环化位点是否落在转录本区域的数目及剪切位点之间的外显子数目,获取与circrna在基因组位置上有最大重叠区域的蛋白编码转录本;
(2)如果circrna的反向剪切位点落在或远离最大重叠转录本的外显子,则对相应的外显子进行相应的截断或延伸,作为circrna的边界外显子,同时取最大重叠转录本与circrna重叠区域的其他外显子,作为circrna的中间外显子;在该过程中需要保证circrna的反向剪切位点不变
(3)如果circrna与外显子没有重叠区域,则认为该circrna是一个单外显子circrna,取circrna的反向剪切位点之间的序列作为外显子序列。
1.8circrna分布统计:
统计各个组织中circrna的数目情况。统计预测到的所有circrna长度分布情况,在基因组染色体上的分布情况,circrna外显子数目情况。
1.9circrna表达水平定量:
利用rpm(splicedreadspermillonreads)对circrna进行定量,其中rpm计算公式如下:rpm=numberofcircularreads/numberoftotalreads(unitsinmillion)其中:numberofcircularreads表示比对到circrna的back-splicedjunctions区域的reads数,该数值来源于circrna预测软件中提供的支持circrna成环的reads数目;numberoftotalreads(unitsinmillion)表示每个样本测序数据中(clean_data)reads数目(单位为millon)。
1.10生物学重复样品间相关性分析:
样品间circrna表达水平相关性可以用来检验实验可靠性和样本选择合理性。相关系数越接近于1,说明样品间表达模式相似度则越高。当样本数目较多时(≥3),circrna的表达量情况,采用r语言中的cor函数及corrplot包,计算样本间的pearson相关系数,并绘制热图进行展示。
1.11样品组间差异circrna筛选:
由于实验有生物学重复,运用deseq包(huber&anders,2014),是基于readscount进行差异分析的r包,用负二项分布检验的方式对reads数进行差异的显著性检验,估算转录本表达量的方式采用basemean值来估算表达量,计算差异倍数。筛选差异的条件为p<0.05且|foldchange|>2。
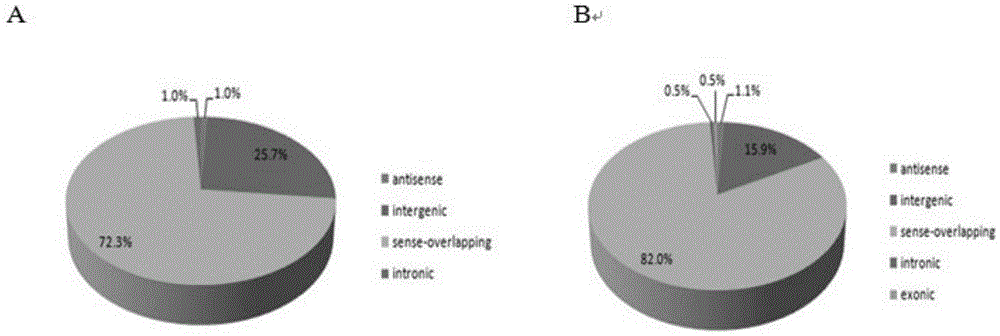
在莱芜猪和大白猪肌内脂肪组织(l_jnvsd_jn)中共鉴定得到283个差异表达circrnas(101个上调,182个下调),其中具有2倍以上差异的circrnas约占35.7%。在101个上调的circrna中,有26个intergeniccircrna,1个introniccircrna,73个sense-overlappingcircrna和1个antisensecircrna;在182个下调的circrna中,有1个exoniccircrna,29个intergeniccircrna,1个introniccircrna,149个sense-overlappingcircrna和2个antisensecircrna(图1)。其中,circrna_27455进入后续研究范围。
实施例2circrna深度分析
2.1差异表达circrna功能分析:
得到差异表达circrna之后,利用circrna来源基因的信息,对差异表达circrna进行go富集分析。统计每个go条目中所包括的差异circrna个数,并用超几何分布检验方法计算每个go条目中差异circrna富集的显著性。kegg是与pathway相关的重要公共数据库,利用kegg数据库对差异circrna来源基因进行pathway分析,可以找到富集差异circrna来源基因的pathway条目,寻找潜在的不同样品的差异circrna可能和哪些细胞通路的改变有关。大白猪和莱芜猪肌内脂肪沉积存在显著差异,通过go分析,差异表达circrna来源基因显著富集于脂质代谢与细胞分化的生物学过程,表明二者在肌内脂肪沉积、代谢的分子机制存在差异,受到不同的基因调控。go富集还显示circrna来源基因富集于与脂肪代谢相关的疾病过程,如胰岛素抵抗,氧化应激,流体剪切力,炎症反应等。
2.2circrna-mirna互作分析
选择在莱芜肌内脂肪组织上调的circrna_27455进行后续的分析,结合生物学信息学分析和本实验室前期做过的大白猪和莱芜猪肌内脂肪组织mirna的测序结果进行整合分析,发现与circrna_27455结合的2个mirna结合位点,分别为ssc-mir-140-3p和ssc-mir-671-5p。
实施例3实时荧光定量pcr验证
设置20个生物学重复(大白猪和莱芜猪各20例),以甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphatedehydrogenase,gapdh)基因为内参,qrt-pcr方法用于验证基因的表达水平。
3.1引物设计:
circrna_27455扩增:
正向引物:5’-ctcctccatcagttcttattagc-3’(seqidno.2)
反向引物:5’-taccataacttccatcctcaga-3’(seqidno.3)
3.2反转录体系:
将下列物质加入0.5ml的离心管中:rna5μl;oligo(dt)2μl;ddh2o(depc处理)4.5μl;
以上首先70℃孵育5min,然后迅速放在冰上。
5×buffer5μl;dntp(10mm)2μl;ribonucleaseinhibitor0.5μl;m-mlvrt1μl;
以上20μl体系42℃孵育60min,然后70℃10min。
3.3real-timercr反应
(1)将所得的cdna与下列物质配成20μl体系加入0.2ml的离心管中混匀。
real-timepcr体系:cdna2μl;引物1(10pmol/μl)0.5μl;引物2(10pmol/μl)0.5μl;
sybrmix10μl;ddh2o7μl。
(2)real-timepcr程序设置:94℃预变性10min;94℃15s,60℃60s,45个循环;72℃10min。
3.4结果统计分析:
为了验证测序分析结果,选取目标分子circrna_27455,进行验证。2-△△ct法分析每组样本间基因的相对表达情况,用t-检验统计分析相对表达量,数据结果展示为平均数±标准差(mean±sd),p<0.05为差异显著。
结果显示,circrna_27455在瘦肉型猪大白猪肌内脂肪组织中显著低表达,在脂肪含量较高的莱芜猪肌内脂肪组织中显著高表达,具体结果见图2。荧光定量pcr结果与测序结果一致。显示circrna_27455可以作为猪肌内脂肪含量检测的分子标志物,在预测或辅助预测猪肉品质、选育具有不同肌肉品质猪等领域具有很好的应用前景。例如,在选育瘦肉型猪的时候考虑circrna_27455表达量较低的个体。
序列表
<110>中国农业科学院北京畜牧兽医研究所
<120>circrna_27455及其检测试剂与应用
<160>3
<170>siposequencelisting1.0
<210>1
<211>1654
<212>dna
<213>人工序列(artificialsequence)
<400>1
accagaatccgtagaagctagccctgtggtagttgagaaatccaacagttatccccacca60
gttatataccagcagctcgcatcattcacacagttacattggtttgccttatgcggacca120
taattatggtgctcgtcctcctccaacacctccggcttcccctcctccatcagttcttat180
tagcaaaaatgaagtaggcatatttaccactcctaattttgatgaaacttccagtgcaac240
tacaatcagcacatctgaggatggaagttatggtactgatgtaaccaggtgcatatgtgg300
ttttacacatgatgatggatacatgatctgttgtgacaaatgcagtgtttggcaacatat360
cgactgcatggggattgacaggcagcatattcctgatacatatttatgtgaacgttgtca420
gcctaggagtttggataaagagagggcggtgctactccaacgccggaaaagggaaaatat480
gtcagatggtgataccagtgcaactgagagtggtgatgaggttcctgtggaattatatac540
tgcatttcagcataccccgacatcaattactttaactgcttcaagagtttccaaggttaa600
tgataaacgaaggaaaaaaagtggggagaaagaacaaaacatttccaaatgtaaaaaagc660
ctttcgtgaaggatctaggaaatcatcaagagttaagggctcagctccagagattgatcc720
ttcatctgatggttcaaattttggatgggagacaaaaatcaaagcatggatggatcgata780
tgaagaggcaaataataaccagtatagtgagggtgttcagagggaggcacaaagaatagc840
tctgagattaggcaatggaacggacaaaaaagagataaataaatcagatttgaataccaa900
caatttgctgttcaaacctcctgtagagagtcatatacaaaagaataagaaaattctgaa960
atctgcaaaagatttacctcccgatgcacttatcattgaatacagagggaagtttatgct1020
aagagaacagtttgaagcaaatggatatttctttaagagtagaccatacccttttgtttt1080
attctattctaaatttcatgggctggaaatgtgtgttgatgcaaggactttcggtaatga1140
agctcggttcatcaggcgttcttgtacacccaatgcagaggtgaggcatgaaattgaaga1200
tggaaccatacatctttacatttattctatacaaagtattccaaaaggaactgaaattac1260
tattgcctttgattttgactatgggaattgcaagtacaaggtggattgtgcatgcctcaa1320
agaaaatccagagtgccctgttctaaaacgtagttctgaatccatggaaaatatcaatag1380
cggttatgaaaccagaagaaaaaaaggaaaaaaagaaaaggatatttcaaaagaaaaaga1440
tacacaaaatcagaatattactttggattgtgaaggaacagtgaacaaaatgaagagccc1500
agaaactaaacagagaaagctttctccactgagactctcagtatcaaataatcaggaacc1560
agattttattgatgatatagaagaaaaaactcccattagcaatgaagtagaaatggagtc1620
agaagaacaaattgcagaaaggaaaaggaagatg1654
<210>2
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>2
ctcctccatcagttcttattagc23
<210>3
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>3
taccataacttccatcctcaga22
- 还没有人留言评论。精彩留言会获得点赞!