一种酶活提高的酮还原酶突变体及其应用的制作方法
本发明涉及一种酶活提高的酮还原酶突变体及其应用,属于生物
技术领域:
。
背景技术:
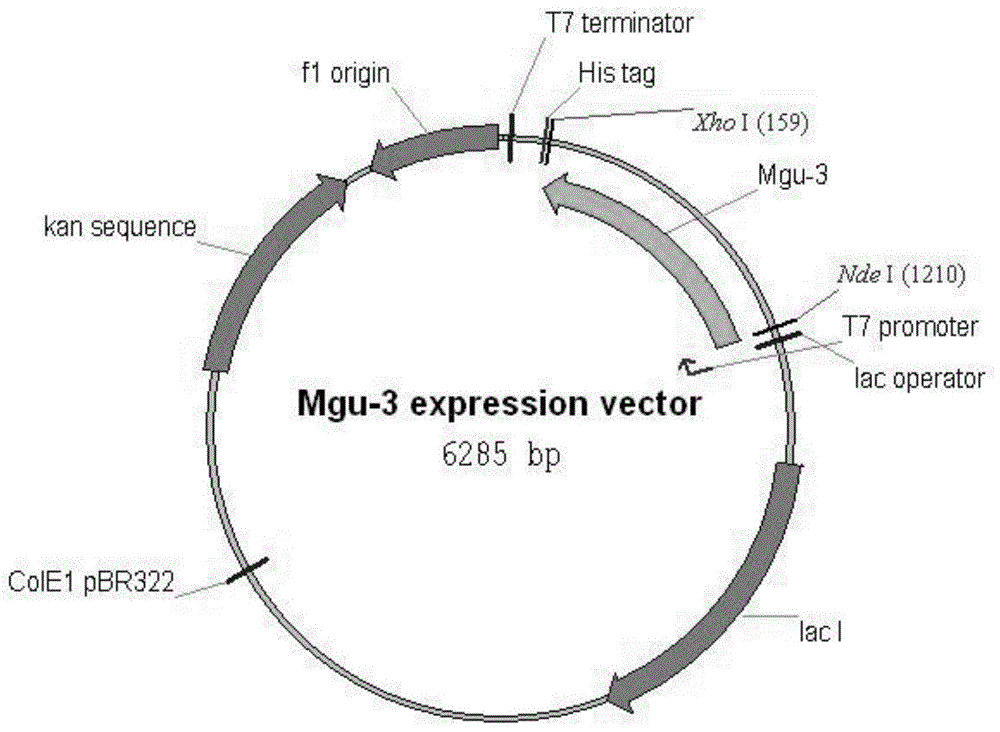
:酮还原酶是一种多用途催化剂,通过醛或酮的对映体选择性还原成对应醇。(r)特异性酮还原酶与(s)特异性酮还原酶具有不同特性,并且这些催化剂在光学活性醇的工业合成中被越来越频繁地用到。旋光性是许多医药和农药活性化合物选择性作用的必要条件,在一些情况下,一个对映体具有有益的药物活性,另一个对映体却具有基因毒性作用。因此,在药用和农药用活性化合物的合成中,需要使用具有所要求的立体特异性的催化剂合成光学活性醇。手性纯的(r)-4-氯-3-羟基丁酸乙酯是一种重要的手性醇。(r)-chbe(4-氯-3-羟基丁酸乙酯)已经被用作(r)-肉碱,r-4-氨基-3-羟基丁酸,(r)-4-羟基-2-吡咯烷酮和其他五烯化学品的主要前体。并且近年来有报道将(r)-4-氯-3-羟基丁酸乙酯用于超级他汀类药物的新合成路线,有望打破原合成工艺的专利垄断。一些属于sdrs的氧化还原酶已被筛选并应用于生物还原4-氯乙酰乙酸乙酯为chbe(4-氯-3-羟基丁酸乙酯)。大多数报道的微生物酶产生的都是(s)型chbe,并同时具有高对映选择性和高产率。但是,仅有极少有酶能产生(r)型chbe,并伴随立体选择性较低(如70%e.e.)或产率低等特性。许等人报道了一株产酮的芽孢杆菌还原酶,其中一个羰基还原酶能转化cobe为(r)-chbe(99.6%e.e.),收率为91.7%,但是需要用到水-甲苯两相系统,底物需要分三次加入反应体系,增加了生产成本并加重了生产负担;并且该催化体系活力偏低,需要依赖投入大量的酶或细胞才能使反应结束(0.5g干细胞催化2.158g底物),对于市场需用量大价格低廉的中间体chbe来说该工艺几乎不具有放大前景。日本人kataoka等使用sporobolomycessalmonicolor来源的酮还原酶被用来做(r)-4-氯-3-羟基丁酸乙酯,底物浓度高达300gl,但是只有92%的手性纯度,无法应用于对手性纯度高要求的药物合成。中国专利cn104988085a利用(r)4-氯-3-羟基丁酸乙酯及其衍生物的生物合成方法,以4-氯乙酰乙酸乙酯(cobe)及其衍生物为起始原料,利用重组e.colicczu-h15静息细胞为生物催化剂,以葡萄糖为辅底物,实现了(r)-chbe及其衍生物的合成。中国专利cn103160547中利用candidaalbicans来源的醇脱氢酶不对称还原4-氯乙酰乙酸乙酯制备(r)-4-氯-3羟基丁酸乙酯,以醇脱氢酶为催化剂,以4-氯乙酰乙酸乙酯为底物,以nadh为辅因子,不对称还原制备(r)-4-氯-3羟基丁酸乙酯。但是底物浓度仅为25g/l-50g/l,且投酶量过高(5g菌体/25ml体系),转化率低,不具备工业生产前景。技术实现要素:本发明的主要目的是为了提供一种酶活提高的酮还原酶突变体及其应用。本发明的目的可以通过采用如下技术方案达到:一种酶活提高的酮还原酶突变体,其源于meyerozymaguilliermondii的野生型酮还原酶,能将4-氯乙酰乙酸乙酯转化生成(r)-4-氯-3-羟基丁酸乙酯,酮还原酶突变体与野生型序列相比具有更高的醇脱氢酶活性,与seqidno.8具有90%以上相似性且具有以下特征中的一个或多个突变:r13k,g77d,n89g,t92a,l97n,f131y,t138p,l146i,g149n,l150v,m154v,v157a,l166y,v169i,h170k,l176v,a180s,n200a,y201f,c203a,c208v,c247v,g248s,l278f,s283l,r285l,g295w,w307l,t309s,q327e,k330e,r331k,该酮还原酶突变体的序列为seqidno.6。一种酶活提高的酮还原酶突变体,该酮还原酶突变体的酶活性比野生型酮还原酶的活性至少增强2-10倍。一种多核苷酸,该多核苷酸的编码由序列为seqidno.6的酮还原酶重组的多肽。一种多核苷酸,该多核苷酸的序列为seqidno.5。一种重组质粒,包括表达载体连接序列为seqidno.5的多核苷。一种宿主细胞,其包含所述的重组质粒。一种宿主细胞,该细胞是一种大肠杆菌。一种宿主细胞,所述重组质粒的密码子已经为在宿主细胞中表达而进行优化了的密码子。一种生产(r)-4-氯-3-羟基丁酸乙酯的方法,包括在与序列为seqidno.6的酮还原酶突变体的存在下,将4-氯乙酰乙酸乙酯转化生成(r)-4-氯-3-羟基丁酸乙酯。本发明的有益技术效果:本专利提供一种医药中间体(r)-4-氯-3-羟基丁酸乙酯的制备方法,整个体系使用单酶或双酶催化,使用葡萄糖或醇类用于辅酶循环,底物浓度高达120g/l-200g/l,手性纯度大于99%,底物用量/nad用量高达3017∶1,辅酶循环次数高,使得(r)-4-氯-3-羟基丁酸乙酯生产成本接近(s)-4-氯-3-羟基丁酸乙酯,有效的扩大了下游应用范围。本发明人发现,微生物meyerozymaguilliermondii中天然存在的野生型酮还原酶,具有将4-氯乙酰乙酸乙酯转化生成(r)-4-氯-3-羟基丁酸乙酯的潜力。本公开内容的发明人已发现包括在一定位置突变的酮还原酶与meyerozymaguilliermondii产生的野生型酮还原酶(seqidno:8)相比表现出增加的催化活性。″野生型酮还原酶″、″野生型kred酶″及″野生型kred酮还原酶″指由源自meyerozymaguilliermondii的具有seqidno:8氨基酸序列的酮还原酶。该酶可将4-氯乙酰乙酸乙酯转化生成(r)-4-氯-3-羟基丁酸乙酯。″野生型″指与自然中所发现的一样的材料或物质的形式。实施例如野生型的蛋白质或核酸序列是可以从自然界中的分离并且未经过人为修饰的,在生物体中存在的原始序列形式。″增加的催化活性″指在体外或体内试验中所测量的与野生型酮还原酶相比,表现出使底物(实施例如4-氯乙酰乙酸乙酯)向产物(实施例如(r)-4-氯-3-羟基丁酸乙酯)转化速率增加的酮还原酶。本发明提供一种酮还原酶突变体,其源于meyerozymaguilliermondii的野生型酮还原酶,表现出使底物(实施例如4-氯乙酰乙酸乙酯)向产物(实施例如(r)-4-氯-3-羟基丁酸乙酯)转化速率增加的酮还原酶。所述酮还原酶突变体,与seqidno.8的野生型酮还原酶相比表现出更强的催化活性。酮还原酶突变体和编码这种突变体的多核苷酸可以使用本领域技术人员通常使用的方法制备。突变体可以通过使编码该酶的体外重组、多核苷酸诱变、dna改组、易错pcr和定向进化方法等获得。上述酮还原酶突变体,具有选自以下特征中的一个或多个突变:r13k,g77d,n89g,t92a,l97n,f131y,t138p,l146i,g149n,l150v,m154v,v157a,l166y,v169i,h170k,l176v,a180s,n200a,y201f,c203a,c208v,c247v,g248s,l278f,s283l,r285l,g295w,w307l,t309s,q327e,k330e,r331k。上述酮还原酶突变体,优先自序列seqidno.4。全长突变的酮还原酶对于保持酶的催化活性并不是必需的。相应地,应考虑酮还原酶突变体的截短的类似物和有催化活性的片段。实施例如,在一些实施方案中,c端或n端的数个氨基酸可以被删去。任何特定的截短的类似物或片段可以利用相应的试验来评估催化活性。同样的,额外的氨基酸残基可以被添加到一个或两个末端而不影响催化活性。额外序列可以是功能性的或非功能性的。实施例如,额外氨基酸序列可以被用来帮助纯化,做为标记,或执行一些其它的功能。因此,本公开内容的酮还原酶突变体可以是融合蛋白的形式,其中酮还原酶突变体(或其片段)诸如通过助溶标签(如sumo蛋白)、纯化标签(如结合金属的his标签)和细菌定位信号(如分泌信号)的实施例而非限制的方式被融合到其它蛋白质。本发明提供一种酮还原酶突变体,其酮还原酶活性比野生型酮还原酶的活性至少增强2-10倍。上述酮还原酶突变体编码序列,其优选自seqidno.3,其已经过序列优化以适于在大肠杆菌中表达。在一些实施方案中,多核苷酸包括被优化用于在特定类型的宿主细胞中表达的密码子。用于各种不同类型的微生物的密码子的使用和偏好性是已知的,因为其是用于在这些微生物的表达特定的氨基酸的优化的密码子。本发明提供一种重组质粒,在一些实施方案中,控制序列包括启动子、前导序列、多腺苷酸化序列、前肽序列、信号肽序列和转录终止子等。对于细菌宿主细胞,指导编码序列的转录的适合的启动子包括但不限于从噬菌体t5、噬菌体t7、噬菌体lambda、大肠杆菌lacuv5操纵子、大肠杆菌trp操纵子、大肠杆菌tac操纵子等。附图说明图1为mgu-3的表达质粒图谱。图2为mgu-ck,mgu-1,mgu-2,mgu-3,生物转化反应21小时的tlc图谱,从左至右依次为mgu-ck,mgu-1,mgu-2,mgu-3,上方黄色条带为底物,下方白色条带为产物。图3为mgu-2在不同反应条件下生物转化20小时的tlc图谱,从左至右依次为实施例12,实施例13,实施例14,实施例15的反应结果。图4为mgu-3在200g/l底物浓度下生物转化5.5小时的tlc图谱,从左至右依次为实施例16,实施例17,实施例18的反应结果。图5为手性检测图谱,自上而下依次为s型标准品,r型标准品,对照反应实施例19产物,实施例10产物。具体实施方式为使本领域技术人员更加清楚和明确本发明的技术方案,下面结合实施例及附图对本发明作进一步详细的描述,但本发明的实施方式不限于此。如图1-图5所示,本实施例提供的酶活提高的酮还原酶突变体,通过以下是实施例实现。实施例1:通过全基因合成的方法,对基因的二级结构以及密码子偏好性进行调整,以实现在大肠杆菌中的高表达。利用primerpremier(http://primer3.ut.ee/)和optimizer(http://genomes.urv.es/optimizer/)进行设计,并保证tm差异控制在3℃以内,引物长度控制在60base以内,得到拼接引物。合成上述引物,并将获得的引物加双蒸水溶解后,加到如下的反应体系中,使得各引物的终浓度为30nm,首尾引物的终浓度为0.6μm。2mmdntpmix(2mmeachdntp)5μl10×pfubuffer5μlpfudnapolymerase(10u/μl)0.5μlddh2o使得反应体系总体积至50μl将配制好的pcr反应体系置于博日xpcycler基因扩增仪中,按下列程序进行扩增:98℃30s,55℃45s,72℃120s,35x。将pcr得到的dna片段进行切胶纯化,利用同源重组的方法克隆进pet30a的ndei/xhoi位点。挑取单克隆进行测序。测序成功的dna序列为seqidno.5,命名为mgu-1,其对应的氨基酸序列为seqidno.6。实施例2:通过全基因合成的方法,对基因的二级结构以及密码子偏好性进行调整,以实现在大肠杆菌中的高表达。利用primerpremier(http://primer3.ut.ee/)和optimizer(http://genomes.urv.es/optimizer/)进行设计,并保证tm差异控制在3℃以内,引物长度控制在60base以内,得到拼接引物。合成上述引物,并将获得的引物加双蒸水溶解后,加到如下的反应体系中,使得各引物的终浓度为30nm,首尾引物的终浓度为0.6μm。2mmdntpmix(2mmeachdntp)5μl10×pfubuffer5μlpfudnapolymerase(10u/μl)0.5μlddh2o使得反应体系总体积至50μl将配制好的pcr反应体系置于博日xpcycler基因扩增仪中,按下列程序进行扩增:98℃30s,55℃45s,72℃120s,35x。将pcr得到的dna片段进行切胶纯化,利用同源重组的方法克隆进pet30a的ndei/xhoi位点。挑取单克隆进行测序。测序成功的dna序列为seqidno.3,命名为mgu-2,其对应的氨基酸序列为seqidno.4。实施例3:通过全基因合成的方法,对基因的二级结构以及密码子偏好性进行调整,以实现在大肠杆菌中的高表达。利用primerpremier(http://primer3.ut.ee/)和optimizer(http://genomes.urv.es/optimizer/)进行设计,并保证tm差异控制在3℃以内,引物长度控制在60base以内,得到拼接引物。合成上述引物,并将获得的引物加双蒸水溶解后,加到如下的反应体系中,使得各引物的终浓度为30nm,首尾引物的终浓度为0.6μm。2mmdntpmix(2mmeachdntp)5μl10×pfubuffer5μlpfudnapolymerase(10u/μl)0.5μlddh2o使得反应体系总体积至50μl将配制好的pcr反应体系置于博日xpcycler基因扩增仪中,按下列程序进行扩增:98℃30s,55℃45s,72℃120s,35x。将pcr得到的dna片段进行切胶纯化,利用同源重组的方法克隆进pet30a的ndei/xhoi位点。挑取单克隆进行测序。测序成功的dna序列为seqidno.5,命名为mgu-3,其对应的氨基酸序列为seqidno.6。实施例4:参考蛋白ssa-ck基因序列的合成:根据u26463.1所示源自sporidiobolussalmonicolor的基因序列,委托上海捷瑞生物对该蛋白的编码序列进行全基因合成,并克隆至pet30a中,得到对照蛋白表达质粒ssa-ck,其对应的氨基酸序列为seqidno.7。实施例5:参考蛋白mgu-ck基因序列的合成:根据edk37381.2所示序列,委托上海捷瑞生物对该蛋白的编码序列进行全基因合成,并克隆至pet30a中,得到对照蛋白表达质粒mgu-ck,其对应的氨基酸序列为seqidno.8。实施例6:摇瓶表达测试:挑取含有表达载体的大肠杆菌单菌落接种于10ml高压灭菌后的培养基中:胰蛋白胨10g/l,酵母提取物5g/l,磷酸氢二钠3.55g/l,磷酸二氢钾3.4g/l,氯化铵2.68g/l,硫酸钠0.71g/l,七水硫酸镁0.493g/l,六水氯化铁0.027g/l,甘油5g/l,葡萄糖0.8g/l,添加卡那霉素至50mg/l。30℃,250rpm过夜培养。次日取1l三角瓶,按1∶100的接种比实施例接入到100ml高压灭菌后的培养基中:胰蛋白胨10g/l,酵母提取物5g/l,磷酸氢二钠3.55g/l,磷酸二氢钾3.4g/l,氯化铵2.68g/l,硫酸钠0.71g/l,七水硫酸镁0.493g/l,六水氯化铁0.027g/l,甘油5g/l,葡萄糖0.3g/l,添加卡那霉素至50mg/l。于30℃中培养至菌体od5-6,立刻将三角瓶置于25℃摇床中,250rpm培养1小时。加iptg至终浓度0.1mm,并于25℃,250rpm继续培养16小时。培养结束后,将培养液于4℃,12000g下离心20分钟收集湿菌体。然后将菌体沉淀用蒸馏水清洗两次,收集菌体,-70℃保存。同时取2克菌体加入6ml纯水超声破碎后进行sds-page检测,并将粗酶液-20℃保存。实施例7:分批补料发酵:分批补料发酵在计算机控制的生物反应器(上海国强)中进行,反应器容量为15l,工作体积为8l,所用到的培养基为酵母抽提物24g/l,蛋白胨12g/l,葡萄糖0.4%,2.31g/l磷酸二氢酶和12.54g/l磷酸氢二钾,ph7.0。初级接种菌种制备200ml培养物,od2.0时接入。在整个发酵过程中,温度保持在37℃,发酵过程中溶解氧浓度由搅拌速率(rpm)和通气供应级联控制在30%自动控制,而培养基的ph值由50%(v/v)正磷酸和30%(v/v)氨水维持在7.0。发酵过程中,当出现大幅的溶氧回升时,开始补料。补料溶液含有9%w/v蛋白胨、9%w/v酵母提取物、14%w/v甘油。当od600约为35.0(湿重约为60g/l)时,用0.2mmiptg诱导16小时。取2克菌体加入6ml纯水超声破碎后进行sds-page检测,并将粗酶液-20℃保存。实施例8:生物转化反应:在3l的三口烧瓶中依次加入680ml缓冲液(含0.1mpb7.0缓冲液,10%甘油,1mm氯化锌),120ml异丙醇,50mlmgu-ck粗酶液,终浓度1mm的nad,混匀2分钟,再加入取120g4-氯乙酰乙酸乙酯,混匀后调节水浴25℃反应。并在21小时取样保存。实施例9:生物转化反应:在3l的三口烧瓶中依次加入680ml缓冲液(含0.1mpb7.0缓冲液,10%甘油,1mm氯化锌),120ml异丙醇,50mlmgu-1酶,终浓度1mm的nad,混匀2分钟,再加入取120g4-氯乙酰乙酸乙酯,混匀后调节水浴25℃反应。并在21小时取样保存。实施例10:生物转化反应:在3l的三口烧瓶中依次加入680ml缓冲液(含0.1mpb7.0缓冲液,10%甘油,1mm氯化锌),120ml异丙醇,50mlmgu-2酶,终浓度1mm的nad,混匀2分钟,再加入取120g4-氯乙酰乙酸乙酯,混匀后调节水浴25℃反应。并在21小时取样保存。实施例11:生物转化反应:在3l的三口烧瓶中依次加入680ml缓冲液(含0.1mpb7.0缓冲液,10%甘油,1mm氯化锌),120ml异丙醇,50mlmgu-3酶,终浓度1mm的nad,混匀2分钟,再加入取120g4-氯乙酰乙酸乙酯,混匀后调节水浴25℃反应。并在21小时取样保存。将实施例8,实施例9,实施例10,实施例11样品进行薄层色谱分析,反应结果见图2。实施例12:生物转化反应:在3l的三口烧瓶中依次加入680ml缓冲液(含0.1mpb7.0缓冲液,10%甘油,1mm氯化锌),120ml异丙醇,100gmgu-2酶,终浓度0.3mm的nad,,混匀2分钟,再加入取160g4-氯乙酰乙酸乙酯,混匀后调节水浴25℃反应。并在20小时取样保存。实施例13:生物转化反应:在3l的三口烧瓶中依次加入680ml缓冲液(含0.1mpb7.0缓冲液,10%甘油,1mm氯化锌),90ml异丙醇,75gmgu-2酶,终浓度0.3mm的nad,,混匀2分钟,再加入取120g4-氯乙酰乙酸乙酯,混匀后调节水浴25℃反应。并在20小时取样保存。实施例14:生物转化反应:在3l的三口烧瓶中依次加入680ml缓冲液(含0.1mpb7.0缓冲液,10%甘油,1mm氯化锌),120ml异丙醇,100gmgu-2酶终浓度0.15mm的nad,,混匀2分钟,再加入取160g4-氯乙酰乙酸乙酯,混匀后调节水浴25℃反应。并在20小时取样保存。实施例15:生物转化反应:在3l的三口烧瓶中依次加入680ml缓冲液(含0.1mpb7.0缓冲液,10%甘油,1mm氯化锌),90ml异丙醇,75gmgu-2酶,终浓度0.15mm的nad,,混匀2分钟,再加入取120g4-氯乙酰乙酸乙酯,混匀后调节水浴25℃反应。并在20小时取样保存。将实施例12,实施例13,实施例14,实施例15样品进行薄层色谱分析,反应结果见图3。实施例16:生物转化反应:在3l的三口烧瓶中依次加入100ml1mpbph7.0缓冲液,300ml纯水,300g一水葡萄糖,200mgnad,1mm氯化锌,50mlmgu-3酶液,10ml枯草芽胞杆菌来源葡萄糖脱氢酶粗酶液,补水至总体系830ml,预混2分钟,加入200g4-氯乙酰乙酸乙酯,混匀后调节水浴25℃反应,并用5mnaoh控制ph约7.0。并在5.5小时取样保存。实施例17:生物转化反应:在3l的三口烧瓶中依次加入100ml1mpbph7.0缓冲液,300ml纯水,300g一水葡萄糖,66mgnad,1mm氯化锌,100mlmgu-3酶液,10ml枯草芽胞杆菌来源葡萄糖脱氢酶粗酶液,补水至总体系830ml,预混2分钟,加入200g4-氯乙酰乙酸乙酯,混匀后调节水浴25℃反应,并用5mnaoh控制ph约7.0。并在5.5小时取样保存。实施例18:生物转化反应:在3l的三口烧瓶中依次加入100ml1mpbph7.0缓冲液,300ml纯水,300g一水葡萄糖,100mgnad,1mm氯化锌,66mlmgu-3酶液,10ml枯草芽胞杆菌来源葡萄糖脱氢酶粗酶液,补水至总体系830ml,预混2分钟,加入200g4-氯乙酰乙酸乙酯,混匀后调节水浴25℃反应,并用5mnaoh控制ph约7.0。并在5.5小时取样保存。将实施例16,实施例17,实施例18的样品进行薄层色谱分析,反应结果见图4。实施例19:对照酶ssa-ck转化反应:由于ssa-ck在单一水相中催化4-氯乙酰乙酸乙酯的反应中酶活很低,无法有效催化150g/l或200g/l的底物浓度,故调整了反应体系使底物尽量反应完全。在3l的三口烧瓶中依次加入100ml1mpbph7.0缓冲液,300ml纯水,150g一水葡萄糖,终浓度1mm的nad,1mm氯化锌,100mlssa-ck粗酶液,10ml枯草芽胞杆菌来源葡萄糖脱氢酶粗酶液,补水至总体系830ml,预混2分钟,加入100g4-氯乙酰乙酸乙酯,混匀后调节水浴25℃反应,并用5mnaoh控制ph约7.0。手性纯度结果显示只有90.27%。实施例20:薄层色谱分析:取少量反应液用乙酸乙酯按照1∶3比实施例进行萃取,并取0.5ul在硅胶板上点样,展开剂为石油醚:乙酸乙酯按3∶1混匀,吹干后高锰酸钾显色。实施例21:酶活检测:取6个5ml离心管,分别标号1-6,分别加入3mm的nadh溶液0ul,40ul,80ul,100ul,120ul,160ul,然后每管补加0.1m,ph7.0的磷酸盐缓冲液补足3ml,混匀后在340nm下检测并记录吸光度的值;根据上述测得值,得到nadh的标准曲线y=k*x,其中y为吸光度的值,x为nadh的浓度(mm),曲线的r2>99.5%;用纯水按一定的倍数稀释酶液(参考稀释倍数:600-1000倍),稀释的倍数使每分钟吸光值变化0.02-0.04之间为合适;取5ml离心管,按以下比实施例取样加入离心管,迅速混合,立即倒入比色皿。检测试剂用量异丙醇500ul2%nad100ul100mmpbs(ph7.0)2.35ml稀释后的酶液50ul在340nm处检测吸光度的变化,每隔1min记录一个值,每分钟的变化率基本相同,其中0min的吸光度为s0,3min的吸光度为s3;酶活计算公式:酶活(u/ml)=[(s0-s3)*3ml*n]/[k×时间(t/min)×加酶量(ml)]其中,n为酶液的稀释倍数。检测结果如下:实施例22:手性纯度测定:以s型产物及r型产物的标准品做为参照,对上述反应产物进行提纯后检测手性纯度,测定条件如下:chiralpakad-3,4.6x100mm,3um;hold5%for0.5min;5-50%in2.5min;mecn;3ml/min;160bar;25℃;apci(+);0.05ulinjection;sim=167检测结果见下表,原始图谱见图5。反应手性纯度实施例1099.98%实施例19ssa-ck对照90.27%可见,本发明提供的酮还原酶突变体,其酮还原酶活性比野生型酮还原酶的活性至少增强2-10倍。与野生型酮还原酶相比,使底物4-氯乙酰乙酸乙酯向产物(r)-4-氯-3-羟基丁酸乙酯转化速率增加,且进一步的增加了单位体积的底物浓度,利于工业放大时提高生产效率。以上所述,仅为本发明进一步的实施例,但本发明的保护范围并不局限于此,任何熟悉本
技术领域:
的技术人员在本发明所公开的范围内,根据本发明的技术方案及其构思加以等同替换或改变,都属于本发明的保护范围。序列表<110>南京朗恩生物科技有限公司<120>一种酶活提高的酮还原酶突变体及其应用<130>2019<160>8<170>siposequencelisting1.0<210>1<211>1047<212>dna<213>人工序列()<400>1atgctgccgtctatccagaccgcttacaccttcaaacgtggttctcgtgaaatcgttcgt60cgtgacgactggccggttccgcacccggaagacaaccaggttctgctgaaagttgaagct120gttggtctgtgcatgtctgacctgcacatcctgatggctcaggaaacccacgttccggaa180accttcgttatgggtcacgaaatcgctggttctatcgctgctgttggtggtaaactggaa240tctgacccgcgttacaaagttggtggtcgtttcaccgtttgcatcggtctgacctgcggt300cgttgcgctaactgccgtaacggtcacgacaactgctgcaccggtaacggtaaattcccg360ggtgcttacggtctgaaccgtgacggtggtttccagcagtacctgctggttccggacctg420aacaccctgctgccgctgccggacggtctgtcttacgaaatggctgctgtttcttctgac480gctgttctgaccccgctgcacgctgttcacaaagttaaaccggacctggttccgaccgct540aaaatcctggttatgggtctgggtggtctgggttctaacgctgttcagatcatcaaaaac600tacggttgccacgttgttgctgttgacgttaaaccggaactggaagaattcgctcgtcag660tgcggtgctgacgaattctacaccgacatcaactcttctccgcacaaaccggaatctttc720gacgtttgcttcgacttctgcggtttccaggaaaccttcgacgtttgccagaaatacgct780cagtctggtggtaaaatcgttgttgttggtctgggtcgttctaaactgatgctgcgtaac840tacgactctgctctgcgttctctgcaggttatcttctctttctggggtaccgcttcttct900caggaacagtctctgcagtgggttaccaaaggtctggttaaaccgatggttaccaacggt960gacttcgctgaactgccgcagtacctgaaacgtctggctaaaggtgaagttaaaggtcgt1020gttgttttccgtccgtctaaactgtaa1047<210>2<211>348<212>prt<213>人工序列()<400>2metleuproserileglnthralatyrthrphelysargglyserarg151015gluilevalargargaspasptrpprovalprohisprogluaspasn202530glnvalleuleulysvalglualavalglyleucysmetseraspleu354045hisileleumetalaglngluthrhisvalprogluthrphevalmet505560glyhisgluilealaglyserilealaalavalglyglylysleuglu65707580seraspproargtyrlysvalglyglyargphethrvalcysilegly859095leuthrcysglyargcysalaasncysargasnglyhisaspasncys100105110cysthrglyasnglylyspheproglyalatyrglyleuasnargasp115120125glyglypheglnglntyrleuleuvalproaspleuasnthrleuleu130135140proleuproaspglyleusertyrglumetalaalavalserserasp145150155160alavalleuthrproleuhisalavalhislysvallysproaspleu165170175valprothralalysileleuvalmetglyleuglyglyleuglyser180185190asnalavalglnileilelysasntyrglycyshisvalvalalaval195200205aspvallysprogluleuglugluphealaargglncysglyalaasp210215220gluphetyrthraspileasnserserprohislysprogluserphe225230235240aspvalcyspheaspphecysglypheglngluthrpheaspvalcys245250255glnlystyralaglnserglyglylysilevalvalvalglyleugly260265270argserlysleumetleuargasntyraspseralaleuargserleu275280285glnvalilepheserphetrpglythralaserserglngluglnser290295300leuglntrpvalthrlysglyleuvallysprometvalthrasngly305310315320aspphealagluleuproglntyrleulysargleualalysglyglu325330335vallysglyargvalvalpheargproserlysleu340345<210>3<211>1047<212>dna<213>人工序列()<400>3atgctgccgtctatccagaccgcttacaccttcaaaaaaggttctcgtgaaatcgttcgt60cgtgacgactggccggttccgcacccggaagacaaccaggttctgctgaaagttgaagct120gttggtctgtgcatgtctgacctgcacatcctgatggctcaggaaacccacgttccggaa180accttcgttatgggtcacgaaatcgctggttctatcgctgctgttggtggtaaactggaa240tctgacccgcgttacaaagttggtaaccgtttcaccgtttgcatcggtctgacctgcggt300cgttgcgctaactgccgtaacggtcacgacaactgctgcaccggtaacggtaaattcccg360ggtgcttacggtctgaaccgtgacggtggtttccagcagtacctgctggttaccgacctg420aacaccctgctgccgatcccggacggtctgtcttacgaagttgctgctgtttcttctgac480gctgttctgaccccgtaccacgctgttcacaaagttaaaccggacctggttccgaccgct540aaaatcctggttatgggtctgggtggtctgggttctaacgctgttcagatcatcaaaaac600tacggttgccacgttgttgcttgcgacgttaaaccggaactggaagaattcgctcgtcag660tgcggtgctgacgaattctacaccgacatcaactcttctccgcacaaaccggaatctttc720gacgtttgcttcgacttcgtttctttccaggaaaccttcgacgtttgccagaaatacgct780cagtctggtggtaaaatcgttgttgttggtctgggtcgttctaaactgatgttccgtaac840tacgacctggctcgtcgttctctgcaggttatcttctctttcggtggtaccgcttcttct900caggaacagtctctgcagctggtttctaaaggtctggttaaaccgatggttaccaacggt960gacttcgctgaactgccgcagtacctgaaacgtctggctaaaggtgaagttaaaggtcgt1020gttgttttccgtccgtctaaactgtaa1047<210>4<211>348<212>prt<213>人工序列()<400>4metleuproserileglnthralatyrthrphelyslysglyserarg151015gluilevalargargaspasptrpprovalprohisprogluaspasn202530glnvalleuleulysvalglualavalglyleucysmetseraspleu354045hisileleumetalaglngluthrhisvalprogluthrphevalmet505560glyhisgluilealaglyserilealaalavalglyglylysleuglu65707580seraspproargtyrlysvalglyasnargphethrvalcysilegly859095leuthrcysglyargcysalaasncysargasnglyhisaspasncys100105110cysthrglyasnglylyspheproglyalatyrglyleuasnargasp115120125glyglypheglnglntyrleuleuvalthraspleuasnthrleuleu130135140proileproaspglyleusertyrgluvalalaalavalserserasp145150155160alavalleuthrprotyrhisalavalhislysvallysproaspleu165170175valprothralalysileleuvalmetglyleuglyglyleuglyser180185190asnalavalglnileilelysasntyrglycyshisvalvalalacys195200205aspvallysprogluleuglugluphealaargglncysglyalaasp210215220gluphetyrthraspileasnserserprohislysprogluserphe225230235240aspvalcyspheaspphevalserpheglngluthrpheaspvalcys245250255glnlystyralaglnserglyglylysilevalvalvalglyleugly260265270argserlysleumetpheargasntyraspleualaargargserleu275280285glnvalilepheserpheglyglythralaserserglngluglnser290295300leuglnleuvalserlysglyleuvallysprometvalthrasngly305310315320aspphealagluleuproglntyrleulysargleualalysglyglu325330335vallysglyargvalvalpheargproserlysleu340345<210>5<211>1047<212>dna<213>人工序列()<400>5atgctgccgtctatccagaccgcttacaccttcaaacgtggttctcgtgaaatcgttcgt60cgtgacgactggccggttccgcacccggaagacaaccaggttctgctgaaagttgaagct120gttggtctgtgcatgtctgacctgcacatcctgatggctcaggaaacccacgttccggaa180accttcgttatgggtcacgaaatcgctggttctatcgctgctgttggtgacaaactggaa240tctgacccgcgttacaaagttggtaaccgtttcgctgtttgcatcggtaacacctgcggt300cgttgcgctaactgccgtaacggtcacgacaactgctgcaccggtaacggtaaattcccg360ggtgcttacggtctgaaccgtgacggtggttaccagcagtacctgctggttaccgacctg420aacaccctgctgccgatcccggacaacgtttcttacgaagttgctgctgcttcttctgac480gctgttctgaccccgtaccacgctatcaaaaaagttaaaccggacgttgttccgacctct540aaaatcctggttatgggtctgggtggtctgggttctaacgctgttcagatcatcaaagct600ttcggtgctcacgttgttgcttgcgacgttaaaccggaactggaagaattcgctcgtcag660tgcggtgctgacgaattctacaccgacatcaactcttctccgcacaaaccggaatctttc720gacgtttgcttcgacttctgcggtttccaggaaaccttcgacgtttgccagaaatacgct780cagtctggtggtaaaatcgttgttgttggtctgggtcgttctaaactgatgctgcgtaac840tacgactctgctcgtcgttctctgcaggttatcttctctttctggggtaccgcttcttct900caggaacagtctctgcagtgggttaccaaaggtctggttaaaccgatggttaccaacggt960gacttcgctgaactgccggaatacctggaaaaactggctaaaggtgaagttaaaggtcgt1020gttgttttccgtccgtctaaactgtaa1047<210>6<211>348<212>prt<213>人工序列()<400>6metleuproserileglnthralatyrthrphelysargglyserarg151015gluilevalargargaspasptrpprovalprohisprogluaspasn202530glnvalleuleulysvalglualavalglyleucysmetseraspleu354045hisileleumetalaglngluthrhisvalprogluthrphevalmet505560glyhisgluilealaglyserilealaalavalglyasplysleuglu65707580seraspproargtyrlysvalglyasnargphealavalcysilegly859095asnthrcysglyargcysalaasncysargasnglyhisaspasncys100105110cysthrglyasnglylyspheproglyalatyrglyleuasnargasp115120125glyglytyrglnglntyrleuleuvalthraspleuasnthrleuleu130135140proileproaspasnvalsertyrgluvalalaalaalaserserasp145150155160alavalleuthrprotyrhisalailelyslysvallysproaspval165170175valprothrserlysileleuvalmetglyleuglyglyleuglyser180185190asnalavalglnileilelysalapheglyalahisvalvalalacys195200205aspvallysprogluleuglugluphealaargglncysglyalaasp210215220gluphetyrthraspileasnserserprohislysprogluserphe225230235240aspvalcyspheaspphecysglypheglngluthrpheaspvalcys245250255glnlystyralaglnserglyglylysilevalvalvalglyleugly260265270argserlysleumetleuargasntyraspseralaargargserleu275280285glnvalilepheserphetrpglythralaserserglngluglnser290295300leuglntrpvalthrlysglyleuvallysprometvalthrasngly305310315320aspphealagluleuproglutyrleuglulysleualalysglyglu325330335vallysglyargvalvalpheargproserlysleu340345<210>7<211>323<212>prt<213>sporidiobolussalmonicolor<400>7metvalglythrthrthrleuasnthrglyalaserleugluleuval151015glytyrglythrtrpglnalaalaproglygluvalglyglnglyval202530lysvalalailegluthrglytyrarghisleuaspleualalysval354045tyrserasnglnprogluvalglyalaalailelysglualaglyval505560lysarggluaspleupheilethrserlysleutrpasnasnserhis65707580argprogluglnvalgluproalaleuaspaspthrleulysgluleu859095glyleuglutyrleuaspleutyrleuilehistrpprovalalaphe100105110proprogluglyaspilethrglnasnleupheprolysalaasnasp115120125lysgluvallysleuaspleugluvalserleuvalaspthrtrplys130135140alametvallysleuleuaspthrglylysvallysalaileglyval145150155160serasnpheaspalalysmetvalaspalaileileglualathrgly165170175valthrproservalasnglnilegluarghisproleuleuleugln180185190progluleuilealahishislysalalysasnilehisilethrala195200205tyrserproleuglyasnasnthrvalglyalaproleuleuvalgln210215220hisprogluilelysargilealaglulysasnglycysthrproala225230235240glnvalleuilealatrpalailevalglyglyhisservalilepro245250255lysservalthrproserargileglygluasnphelysglnvalser260265270leuserglngluaspvalaspalavalserlysleuglygluglyser275280285glyargargargtyrasnileprocysthrtyrserprolystrpasp290295300ileasnvalpheglyglugluaspglulyssercyslysasnalaval305310315320lysilelys<210>8<211>348<212>prt<213>meyerozymaguilliermondii<400>8metleuproserileglnthralatyrthrphelysargglyserarg151015gluilevalargargaspasptrpprovalprohisprogluaspasn202530glnvalleuleulysvalglualavalglyleucysmetseraspval354045hisileleumetalaglngluthrhisvalprogluthrphevalmet505560glyhisgluilealaglyserilealaalavalglyglylysleuglu65707580seraspproargtyrlysvalglyasnargphethrvalcysilegly859095leuthrcysglyargcysalaasncysargasnglyhisaspasncys100105110cysthrglyasnglylyspheproglyalatyrglyleuasnargasp115120125glyglypheglnglntyrleuleuvalthraspleuasnthrleuleu130135140proleuproaspglyleusertyrglumetalaalavalserserasp145150155160servalleuthrproleuhisalavalhislysvallysproaspleu165170175valprothralalysileleuvalmetglyleuglyglyleuglyser180185190asnalavalglnileilelysasntyrglycyshisvalvalalacys195200205aspvallysprogluleuglugluphealaargglncysglyalaasp210215220gluphetyrthraspileasnserserprohislysprogluserphe225230235240aspvalcyspheaspphecysglypheglngluthrpheaspsercys245250255glnlystyralaglnserglyglylysilevalvalvalglyleugly260265270argserlysleumetleuargasntyraspseralaargargserleu275280285glnvalilepheserpheglyglythralaserserglngluglnser290295300leuglntrpvalthrlysglyleuvallysprometvalthrasngly305310315320aspphealagluleuproglntyrleulysargleualalysglyglu325330335vallysglyargvalvalpheargproserlysleu340345当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1