一种基于基因修饰得到人重组干扰素的融合基因、融合蛋白及其制备方法与流程
本发明属于生物制品技术领域,具体涉及一种基于基因修饰得到的人重组干扰素的融合基因、融合蛋白及其制备方法。
技术背景
干扰素是由诱导剂刺激网状内皮细胞、巨噬细胞、淋巴细胞等产生的一种具有多种功能的糖蛋白,具有属特异性。它们具有广谱的抗病毒活性及免疫调节功能。根据干扰素的基因类型、结构特点和功能作用以及在细胞表面的受体类型,干扰素目前分为三种类型。ⅰ型干扰素包括ifn-α,ifn-β,ifn-δ,ifn-ε,ifn-ω,ifn-κ,ifn-ζ和ifn-τ。其中,ifn-α又有20多种亚型;ifn-β只有一种亚型;ⅱ型干扰素是ifn-γ;ⅲ型干扰素为ifn-λ,又可分为ifn-λ1,ifn-λ2和ifn-λ3。干扰素可以结合特定的细胞表面受体并随后激活胞质信号转导通路,进而诱导一系列基因的表达,从而发挥其生物学作用。
天然ifn-β是含有166个氨基酸的糖基化产物,ifnβ-1b是天然ifn-β第十七位半胱氨酸突变为丝氨酸的变5异体,是大肠杆菌表达系统的非糖基化产物。作为首个fda批准的专门治疗复发缓解型多发性硬化症(rrms)的药物,干扰素β可以减缓rrms病人的病情恶化,改善病人的身体状况。大肠杆菌是目前应用最广泛、最成功的表达体系,常作为高效表达的首选体系。大肠杆菌作为外源基因表达的宿主,具有遗传背景清晰,技术操作容易,培养条件简单,大规模培养简单经济的优点。
虽然大肠杆菌系统可以用于目的蛋白的大量表达,但由于外源基因其中进行高效表达时,会形成由膜包裹的高密度、不溶性蛋白质颗粒(即包涵体)。包涵体的形成的原因复杂,主要与胞质内蛋白质生成速率有关,由于新生成的多肽浓度较高,没有充足的时间进行折叠,从而形成非结晶、无定形的蛋白质聚集体。目的蛋白表达为包涵体后通常不具有生物学活性,并且其复性与纯化均非常困难。目前通过大肠杆菌体系表达干扰素均为包涵体表达,这严重影响了其生产效率及生产成本。
技术实现要素:
基于此,本发明通过修饰基因序列引入带不同电荷的甘氨酸与赖氨酸,达到类似于两性离子聚合物的结构,以期实现ifn-β的可溶性表达。组氨酸标签是最为常用的亲和标签之一,通常由4-8个组氨酸构成,它的优点是表达方便且其较短的结构对蛋白质活性影响较低。组氨酸标签能够通过螯合作用特异性与表面固定的金属离子(ni2+,cu2+,co2+等)结合。因此,本发明在结构中增加了一个6聚的组氨酸标签以方便后期蛋白纯化。
本发明目的在于针对在原核表达系统中干扰素蛋白无法实现可溶性表达的问题,选取ifn-β型干扰素为例,通过在ifn-β序列中加入ek氨基酸重复单元使人ifn-β基因在大肠杆菌bl21(de3)中成功实现可溶性表达并通过镍柱亲和层析一步实现高纯度纯化。
本发明提供了一种基于基因修饰得到的人重组干扰素融合蛋白的制备方法,包括如下步骤:
(1)分别以干扰素基因和氨基酸序列为模板,分别进行pcr扩增,得到片段1和片段2;
(2)将片段1和片段2通过pcr进行基因融合,得到融合基因;
(3)分别酶切融合基因和表达质粒,再用dna连接酶连接;
(4)转化至大肠杆菌中扩增培养,iptg诱导表达后,得到粗融合蛋白;
(5)用镍柱亲和层析纯化步骤(4)所得粗融合蛋白,得到融合蛋白。
进一步地,上述技术方案中,步骤(1)所述干扰素基因包括ifn-α,ifn-β,ifn-δ,ifn-ε,ifn-ω,ifn-κ,ifn-ζ或ifn-τ。
进一步地,上述技术方案中,步骤(1)所述氨基酸序列由带有相反电荷的两种氨基酸的重复单元组成,所述重复单元包括赖氨酸(k)与谷氨酸(e)组成的重复单元。
进一步地,上述技术方案中,步骤(2)所述融合基因由干扰素基因与在干扰素基因c端或n端添加包含10到100个带有相反电荷的两种氨基酸的重复单元组成的氨基酸序列组成。
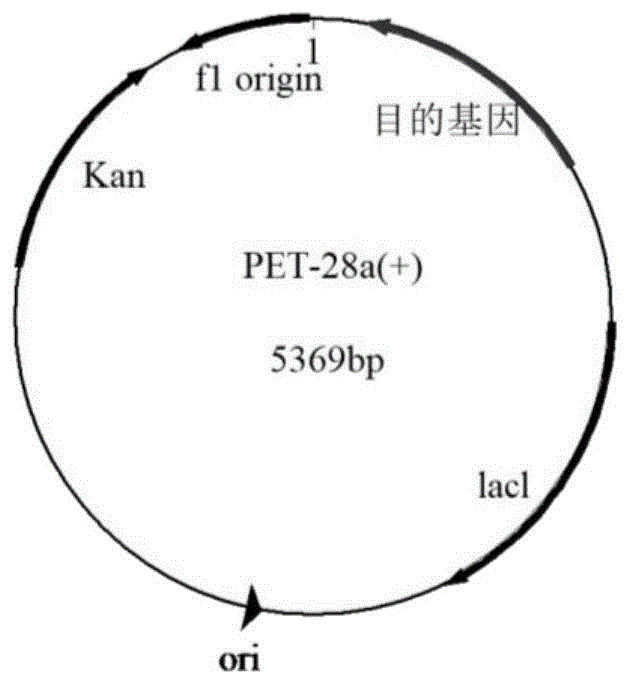
进一步地,上述技术方案中,步骤(3)所述表达质粒为pet系列质粒,所述pet系列质粒包括pet-28a(+)质粒。
进一步地,上述技术方案中,步骤(4)所述大肠杆菌包括bl21(de3)。
本发明还提供了一种基于基因修饰得到人重组干扰素ifn-β的融合蛋白,所述融合蛋白的氨基酸序列包括seqidno.2。
进一步地,氨基酸序列为seqidno.2的融合蛋白的制备方法,具体步骤为:
(1)分别以干扰素ifn-β基因序列和氨基酸序列为模板,分别进行pcr扩增,得到片段1和片段2;所述氨基酸序列由带有相反电荷的两种氨基酸的重复单元组成,所述重复单元为赖氨酸(k)与谷氨酸(e);
(2)将片段1和片段2通过pcr进行基因融合,得到融合基因;
(3)分别酶切融合基因和表达质粒,再用dna连接酶连接;
(4)转化至大肠杆菌中扩增培养,iptg诱导表达后,得到粗融合蛋白;
(5)用镍柱亲和层析纯化步骤(4)所得粗融合蛋白,得到融合蛋白。
进一步地,上述技术方案中,步骤(2)所述融合基因由干扰素基因与在干扰素基因c端或n端添加包含10到100个氨基酸基序列组成。
优选的,所述融合基因由干扰素基因与在干扰素基因c端或n端添加包含50个氨基酸基序列组成。
进一步地,上述技术方案中,步骤(3)所述表达质粒为pet系列质粒,所述pet系列质粒包括pet-28a(+)质粒。
本发明还提供了一种编码上述融合蛋白的人重组干扰素融合基因,所述融合基因的核苷酸序列如seqidno.1所示。
本发明还提供了一种含有所述融合蛋白的重组表达菌。
进一步地,上述技术方案中,所述重组表达菌为大肠杆菌,所述大肠杆菌包括bl21(de3)。
所述感受态细胞(competentcell)是用理化方法诱导细胞,使其处于最适摄取和容纳外来dna的生理状态的细胞。主要原理就是通过处理使细胞的通透性变大,直观的说,使得细胞膜表面出现一些孔洞,便于外源基因或载体进入感受态细胞。由于细胞膜的流动性,这种孔洞会被细胞自身所修复。
大肠杆菌对数生长期为od值在0.6-0.8之间,对数期的群体细胞具有生理特性比较一致、细胞各成分平衡增长和生长速率恒定等优点,是代谢、生理研究的良好材料,不仅是作为菌种的最佳材料,更会提高诱导表达效率。
有益效果:
本发明的有益效果在于:本发明解决了利用原核表达体系表达ifn-β蛋白时不能获得可溶性、具备天然活性的ifn-β蛋白的难题,通过设计ifn-β基因的序列成功在ifn-β蛋白c端或n端引入聚ek肽链,并成功得到可溶性的ifn-ke50融合蛋白,所得ifn-ke50融合蛋白纯度可达到94.8%,此类方法也可在其他类型干扰素在原核菌中可溶性表达制备方面的应用。
本发明对人重组干扰素ifn-β基因序列进行扩增,与质粒pet-28a(+)相连接得到融合基因,再将融合基因转染至大肠杆菌bl21(de3)中进行扩增和表达得到了可溶性的融合蛋白,解决了大肠杆菌中无法获得可溶性干扰素的问题。本发明还提供了从稳定细胞系获得高纯度的ifn-ke50蛋白的纯化方法,由于其具有组氨酸标签,用固定金属镍进行亲和层析可以对目标蛋白进行有效的纯化。简化了后续的纯化步骤,并可以获得更高的纯度。
附图说明
图1为本实验所用pet-28a(+)质粒图谱示意图。
图2为融合蛋白ifn-ke50在两种条件下诱导表达的电泳结果,其中1为marker;2为16℃ifn-ke50上清液;3为16℃ifn-ke50沉淀;4为37℃ifn-ke50上清液;5为37℃ifn-ke50沉淀;6为ifn-ke50诱导前菌液沉淀;7为ifn-ke50诱导前菌液上清液。
图3为融合蛋白ifn-ke50的纯化结果,其中1为marker;2为纯化的ifn-ke50;3为ifn-ke50原上清液。
具体实施方式
下述非限制性实施例可以使本领域的普通技术人员更全面地理解本发明,但不以任何方式限制本发明。
实施例1
通过pcr扩增的方式在原人重组干扰素ifn-β1b(genscriptbiotechcorporation)基因序列中引入组氨酸标签及谷氨酸赖氨酸重复单元,得到一种基于基因修饰得到人重组干扰素ifn-β的融合蛋白,所述融合蛋白的核苷酸序列如seqidno.1所示,氨基酸序列如seqidno.2所示。
pcr扩增反应体系如下:先由2.5μl50个重复ke氨基酸单元的基因序列seqidno.3作为模板dna,0.5μl基因序列seqidno.4:catatggaaaacctgtattt作为上游引物,0.5μl基因序列seqidno.5:ctttttcttaagctactactattt作为下游引物,蒸馏水补至50μl。反应过程如下:94℃预变性5min,94℃变性1min,5℃退火30s,72℃延伸40s,72℃延伸8min,结束后用1%琼脂糖凝胶回收,得到序列seqidno.6;同上,再以ifn-β1b基因序列(seqidno.7)为模板dna,05μl基因序列seqidno.8:gaattcgatgatgatgataaaggatcctataacctg作为上游引物,0.5μl基因序列seqidno.9:gacgcgttgattcgccggcg作为下游引物进行pcr扩增及回收,得到序列seqidno.10。最后混合序列seqidno.6、序列seqidno.10以及引物seqidno.11:catatggaaaacctgtattt和引物seqidno.12:gacgcgttgattcgccggcg,通过pcr进行基因融合,最终得到融合了ifnβ1b和ke50的序列并将其命名为ifn-ke50融合基因。所得ifn-ke50融合基因的核苷酸序列如seqidno.1所示,氨基酸序列如seqidno.2所示。
实施例2
通过用限制性内切酶ndeⅰ及notⅰ分别对实施例1所得ifn-ke50融合基因和2μgpet-28a(+)质粒(solarbio|货号:p3110)进行酶切。反应体系为:1.5μlndeⅰ酶,1.5μlnotⅰ酶,5μl10×buffer,2-5μgpet-28a(+)质粒或ifn-ke50融合基因,ddh2o补至50μl,37℃水浴30min。琼脂糖凝胶回收酶切后的ifn-ke50融合基因和pet-28a(+)质粒,利用t4连接酶16℃连接4hrs,得到ifn-ke50融合质粒室温放置2hrs,将ifn-ke50融合质粒转染至大肠杆菌感受态细胞中(bl21(de3)),涂布于含有50μg/ml卡那霉素的lb平板,37℃倒置培养过夜。在平板上挑取菌落于50ml含卡那霉素的lb液体培养基中,卡那霉素浓度为50μg/ml,于37℃温度下、培养12h扩培得到转染有ifn-ke50融合质粒的大肠杆菌菌液(重组表达菌)。
实施例3
按1比100的体积比取3ml转染有ifn-ke50融合基因的大肠杆菌菌液于300ml的2×yt培养基中,加入卡那霉素使其浓度为50μg/ml,于37℃温度下、培养2.5h后,加入iptg诱导剂使其在培养基中的浓度为0.1mm/l,于16℃温度下、培养表达12h后得到内含ifn-ke50融合蛋白的大肠杆菌菌液。
按上述步骤加入iptg诱导剂后于37℃温度下、培养表达12h,其余步骤不变,得到37℃下诱导得到的内含ifn-ke50融合蛋白的大肠杆菌菌液。
所述ifn-ke50融合蛋白的sds-page电泳如图1所示,考察了两个不同温度条件下的表达情况,其中1为marker;2为16℃ifn-ke50上清液;3为16℃ifn-ke50沉淀;4为37℃ifn-ke50上清液;5为37℃ifn-ke50沉淀;6为ifn-ke50诱导前菌液沉淀;7为ifn-ke50诱导前菌液上清液。ifn-ke50是在ifn-β1b基础上增加一条由50对赖氨酸/谷氨酸组成的多肽链得到的,其功能类似于两性离聚合物。通过和泳道6、7的原细菌表达蛋白条带对比,可以推测泳道2、4中位于分子量26.0kd和33.0kd之间的条带为目的蛋白的条带,分子量约为30kd左右。泳道3、5中未观察到目的蛋白包涵体形式的条带,可知ifn-ke50是以可溶性形式表达的。因此通过连接一个功能类似两性离子聚合物的聚ke肽链不但能增加蛋白分子量,降低表达速率,更可以有效的降低蛋白包涵体的表达含量,提高可溶性表达。
实施例4
利用ni柱纯化ifn-ke50融合蛋白,获得纯度较高的ifn-ke50融合蛋白。ni柱纯化的具体步骤为:先用bindingbuffer平衡ni柱10个柱体积,然后上样,再用bindingbuffer冲洗至紫外吸收线达到基线,换elutionbuffer洗脱,收集洗脱峰。其中,bindingbuffer:1l,含20mm的磷酸缓冲液、500mm的氯化钠和20mm的咪唑,ph为7.4;elutionbuffer:1l,含20mm的磷酸缓冲液、500mm的氯化钠和500mm的咪唑,ph为7.4。图2是ifn-ke50蛋白纯化结果图,其中1为marker;2为纯化的ifn-ke50;3为ifn-ke50原上清液。可以看出纯化效果较好,纯化后蛋白纯度可达到94.8%。其中所用仪器为aktaprimeplus。
将提纯后的蛋白溶液至于7kda分子量的透析袋中并置入盐浓度梯度递减的缓冲液中透析除盐,每12h换液,最后使得蛋白质处于水环境中,离心去除失活的蛋白沉淀,并将离心后蛋白溶液至于-20℃中冷冻,放入冻干机中冻干得到ifn-ke50融合蛋白粉末。其中盐浓度梯度为0.5m;0.4m;0.3m;0.2m;0.1m;去离子水。
对于任何熟悉本领域的技术人员而言,在不脱离本发明技术方案范围情况下,都可利用上述揭示的技术内容对本发明技术方案作出许多可能的变动和修饰,或修改为等同变化的等效实施例。因此,凡是未脱离本发明技术方案的内容,依据本发明的技术实质对以上实施例所做的任何简单修改、等同变化及修饰,均应仍属于本发明技术方案保护的范围内。
sequencelisting
<110>大连理工大学
<120>一种基于基因修饰得到人重组干扰素的融合基因、融合蛋白及其制备方法
<130>2020
<160>12
<170>patentinversion3.5
<210>1
<211>869
<212>dna
<213>人工序列(artificialsequence)
<400>1
catatggaaaacctgtattttcagggcgaaaaagaaaaagaaaaagaaaaagaaaaagaa60
aaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaa120
aaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaa180
aaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaa240
aaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaa300
aaagaaaaagaaaaagaaaaagaaaaagaattcgatgatgatgataaaggatcctataac360
ctgctgggctttctgcagcgcagcagcaactttcagagccagaaactgctgtggcagctg420
aacggccgcctggaatattgcctgaaagatcgcatgaactttgatattccggaagaaatt480
aaacagctgcagcagtttcagaaagaagatgcggcgctgaccatttatgaaatgctgcag540
aacatttttgcgatttttcgccaggatagcagcagcaccggctggaacgaaaccattgtg600
gaaaacctgctggcgaacgtgtatcatcagattaaccatctgaaaaccgtgctggaagaa660
aaactggaaaaagaagattttacccgcggcaaactgatgagcagcctgcatctgaaacgc720
tattatggccgcattctgcattatctgaaagcgaaagaatatagccattgcgcgtggacc780
attgtgcgcgtggaaattctgcgcaacttttattttattaaccgcctgaccggctatctg840
cgcaactaagcggccgcaagcttctcgag869
<210>2
<211>301
<212>prt
<213>人工序列(artificialsequence)
<400>2
metglyserserhishishishishishisserserglyleuvalpro
151015
argglyserhismetgluasnleutyrpheglnglyglulysglulys
202530
glulysglulysglulysglulysglulysglulysglulysglulys
354045
glulysglulysglulysglulysglulysglulysglulysglulys
505560
glulysglulysglulysglulysglulysglulysglulysglulys
65707580
glulysglulysglulysglulysglulysglulysglulysglulys
859095
glulysglulysglulysglulysglulysglulysglulysglulys
100105110
glulysglulysglulysglulysglulysglulysglulysglulys
115120125
glupheaspaspaspasplysglysertyrasnleuleuglypheleu
130135140
glnargserserasnpheglnserglnlysleuleutrpglnleuasn
145150155160
glyargleuglutyrcysleulysaspargmetasnpheaspilepro
165170175
glugluilelysglnleuglnglnpheglnlysgluaspalaalaleu
180185190
thriletyrglumetleuglnasnilephealailepheargglnasp
195200205
serserserthrglytrpasngluthrilevalgluasnleuleuala
210215220
asnvaltyrhisglnileasnhisleulysthrvalleugluglulys
225230235240
leuglulysgluaspphethrargglylysleumetserserleuhis
245250255
leulysargtyrtyrglyargileleuhistyrleulysalalysglu
260265270
tyrserhiscysalatrpthrilevalargvalgluileleuargasn
275280285
phetyrpheileasnargleuthrglytyrleuargasn
290295300
<210>3
<211>333
<212>dna
<213>人工序列(artificialsequence)
<400>3
catatggaaaacctgtattttcagggcgaaaaagaaaaagaaaaagaaaaagaaaaagaa60
aaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaa120
aaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaa180
aaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaa240
aaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaa300
aaagaaaaagaaaaagaaaaagaaaaagaattc333
<210>4
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>4
catatggaaaacctgtattt20
<210>5
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>5
ctttttcttaagctactactattt24
<210>6
<211>348
<212>dna
<213>人工序列(artificialsequence)
<400>6
catatggaaaacctgtattttcagggcgaaaaagaaaaagaaaaagaaaaagaaaaagaa60
aaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaa120
aaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaa180
aaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaa240
aaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaaaaagaa300
aaagaaaaagaaaaagaaaaagaaaaagaattcgatgatgatgataaa348
<210>7
<211>515
<212>dna
<213>人工序列(artificialsequence)
<400>7
tataacctgctgggctttctgcagcgcagcagcaactttcagagccagaaactgctgtgg60
cagctgaacggccgcctggaatattgcctgaaagatcgcatgaactttgatattccggaa120
gaaattaaacagctgcagcagtttcagaaagaagatgcggcgctgaccatttatgaaatg180
ctgcagaacatttttgcgatttttcgccaggatagcagcagcaccggctggaacgaaacc240
attgtggaaaacctgctggcgaacgtgtatcatcagattaaccatctgaaaaccgtgctg300
gaagaaaaactggaaaaagaagattttacccgcggcaaactgatgagcagcctgcatctg360
aaacgctattatggccgcattctgcattatctgaaagcgaaagaatatagccattgcgcg420
tggaccattgtgcgcgtggaaattctgcgcaacttttattttattaaccgcctgaccggc480
tatctgcgcaactaagcggccgcaagcttctcgag515
<210>8
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>8
gaattcgatgatgatgataaaggatcctataacctg36
<210>9
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>9
gacgcgttgattcgccggcg20
<210>10
<211>542
<212>dna
<213>人工序列(artificialsequence)
<400>10
gaattcgatgatgatgataaaggatcctataacctgctgggctttctgcagcgcagcagc60
aactttcagagccagaaactgctgtggcagctgaacggccgcctggaatattgcctgaaa120
gatcgcatgaactttgatattccggaagaaattaaacagctgcagcagtttcagaaagaa180
gatgcggcgctgaccatttatgaaatgctgcagaacatttttgcgatttttcgccaggat240
agcagcagcaccggctggaacgaaaccattgtggaaaacctgctggcgaacgtgtatcat300
cagattaaccatctgaaaaccgtgctggaagaaaaactggaaaaagaagattttacccgc360
ggcaaactgatgagcagcctgcatctgaaacgctattatggccgcattctgcattatctg420
aaagcgaaagaatatagccattgcgcgtggaccattgtgcgcgtggaaattctgcgcaac480
ttttattttattaaccgcctgaccggctatctgcgcaactaagcggccgcaagcttctcg540
ag542
<210>11
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>11
catatggaaaacctgtattt20
<210>12
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>12
gacgcgttgattcgccggcg20
- 还没有人留言评论。精彩留言会获得点赞!