梨转录因子PbHB.G7.2及其在促进果实成熟中的应用的制作方法
本发明属于植物基因工程领域,涉及梨转录因子pbhb.g7.2及其应用,具体涉及从‘翠冠梨’中分离、克隆得到一个与果实成熟相关的homeobox家族成员pbhb.g7.2基因及其应用。
背景技术:
果实成熟,是指果实完成生长发育之后,发生一系列生理生化变化的过程,其在分子水平上是相关基因的表达、转录及调控综合的结果。从生理角度来说,根据果实成熟过程中是否出现乙烯合成高峰以及呼吸速率显著上升的现象,可将其分为呼吸跃变型果实和非呼吸跃变型果实[1]。呼吸跃变型果实的成熟过程极大地依赖于乙烯的催化和诱导作用,而对于非呼吸跃变型果实的成熟,乙烯并不是必要的[2-3]。目前,乙烯在植物体内的合成路径已经十分清楚,s-蛋氨酸(s-adenosylmetionine)在1-氨基环丙烷-1-羧酸合成酶(1-aminocyclopropane-1-carboxylicacidsynthase,acs)的催化作用下生成1-氨基环丙烷-1-羧酸(1-aminocyclopropane-1-carboxylicacid,acc),而后acc被acc氧化酶(accoxidase,aco)氧化生成乙烯[4]。其中acs通常被认为是植物体合成的限速酶。
‘翠冠’梨(pyruspyrifoliacuiguan)属于呼吸跃变型果实,是一种重要的经济水果,商业价值极大,在我国南方地区广泛种植,具有果大皮薄、多汁味甜、肉质细嫩等优点[5]。因此研究‘翠冠’梨的成熟过程,克隆梨中与成熟有关的基因,对于延长梨果实的货架期,提高作物改良和育种的效率,指导农业生产具有非常重要的理论和实践意义。
同源异形盒蛋白(homeobox,hb)家族是植物细胞中的一类转录因子超家族,编码一个保守的dna结合蛋白域,称为同源域(homeodomain,hd)[6]。迄今为止,hb蛋白已经被报道参与多个植物生长发育过程。在拟南芥中,hb的亚家族wox中的成员被发现参与细胞器官的形成和维护,knox的成员则与分生组织活性、叶片发育和细胞分化有关[7-10]。athb-1对调节叶片发育至关重要,athb-2可调控植物的避荫反应[11-12]。此外,植物器官细胞外层和胚芽分生组织的形成、器官极性、顶端胚芽的形成和分生组织的功能也都受到hb蛋白的调控[13-14]。必须指出的是,尽管hb家族已经得到广泛研究,但是目前为止,对于调控梨果实成熟的hb基因尚未见报道。
技术实现要素:
本发明的目的在于提供一个能够促进梨果实乙烯的合成,加速成熟过程的转录因子pbhb.g7.2以及该基因的应用。
本发明的另一目的是提供一个受到转录因子pbhb.g7.2调控的基因pbacs1b,以及与转录因子pbhb.g7.2蛋白互作并同样能够调控pbacs1b基因的两个转录因子pbhb.g1和pbhb.g2.1。
本发明的目的可通过以下技术方案实现:
本发明提供了一种分离自‘翠冠梨’具有促进果实乙烯的合成,加速果实成熟过程功能的转录因子pbhb.g7.2基因,属于homeobox家族成员,其核苷酸序列如seqidno.1所示。包含912bp的开放阅读框,编码304个氨基酸,其编码的蛋白质的氨基酸序列如seqidno.2所示。
含有上述梨转录因子pbhb.g7.2的表达盒、重组载体、转基因细胞系或转基因重组菌。所述的重组载体为重组表达载体,优选的,含有所述pbhb.g7.2基因的重组表达载体以psak277为超表达载体,所述pbhb.g7.2基因在psak277载体上的插入位点为hindiii和xbai之间。
用于扩增上述梨转录因子pbhb.g7.2的引物对,它包括正向引物f1和反向引物r1;所述正向引物f1的序列如seqidno.5所示;所述反向引物r1的序列如seqidno.6所示。
本发明还提供了一个受到转录因子pbhb.g7.2调控的基因pbacs1b,属于acs家族成员,其核苷酸序列为seqidno.3所示,包含1488bp的开放阅读框,基因pbacs1b起始密码子上游2000bp启动子序列如seqidno.4所示。
本发明还提供了两个梨hb转录因子pbhb.g1和pbhb.g2.1,其所编码的蛋白能够与转录因子pbhb.g7.2编码的蛋白形成互作,且同样能够结合并激活pbacs1b基因的启动子,所述转录因子pbhb.g1的核苷酸序列如seqidno.15所示,所述转录因子pbhb.g2.1的核苷酸序列如seqidno.17所示。
上述的梨转录因子pbhb.g7.2、梨转录因子pbhb.g7.2所编码的蛋白质或上述的表达盒、重组载体、转基因细胞系或转基因重组菌在提高果实乙烯合成量,促进果实成熟中的应用。
上述的应用,构建瞬时过表达上述转录因子pbhb.g7.2的果实能够提高果实乙烯合成量,促进果实成熟。所述的果实以梨为例,构建瞬时过表达转录因子pbhb.g7.2的梨果实包括以下步骤:
1)以梨果肉cdna为模板,进行pcr扩增得到梨hb基因pbhb.g7.2;所述扩增用引物对包括正向引物f1和反向引物r1;所述正向引物f1的序列如seqidno.5所示;所述反向引物r1的序列如seqidno.6所示。
2)将所述梨hb基因pbhb.g7.2与载体连接获得重组载体;
3)将所述重组载体转入根癌农杆菌中获得重组根癌农杆菌;
4)将所述重组根癌农杆菌侵染梨果实,获得过表达pbhb.g7.2基因的梨果实。
本发明所述的梨hb基因pbhb.g7.2在促进梨果实成熟中的应用。
本发明所述的重组表达载体在促进梨果实成熟中的应用。
本发明还提供了所述的梨hb基因pbhb.g7.2在梨杂交育种中的应用。
本发明的有益效果体现在:
本发明提供的梨hb基因pbhb.g7.2,具有促进果实乙烯的合成,加速果实成熟过程的功能,能够应用于缩短果实成熟时间,所述梨hb基因pbhb.g7.2编码的蛋白能够结合并激活细胞中pbacs1b基因的启动子,促进pbacs1b基因的转录,从而促进了果实乙烯的合成,加速果实成熟过程。根据实施部分的记载,所述梨hb基因pbhb.g7.2转入到梨果实中获得的超表达梨果实,其乙烯释放量明显高于对照组,且成熟过程加快。在超表达梨果实中pbacs1b基因的表达水平与对照相比显著上升。这与超表达梨果实中pbhb.g7.2基因的表达趋势一致。此外,本发明还提供了两个梨hb转录因子pbhb.g1和pbhb.g2.1,pbhb.g1和pbhb.g2.1编码的蛋白能够与pbhb.g7.2编码的蛋白形成互作,且同样能够结合并激活pbacs1b基因的启动子。其中,本发明提供的梨hb基因pbhb.g7.2还可以作为分子标记应用于梨杂交育种,为促进梨果实成熟的分子育种提供新的基因资源,有利于增加农民收入降低农业成本。
附图说明
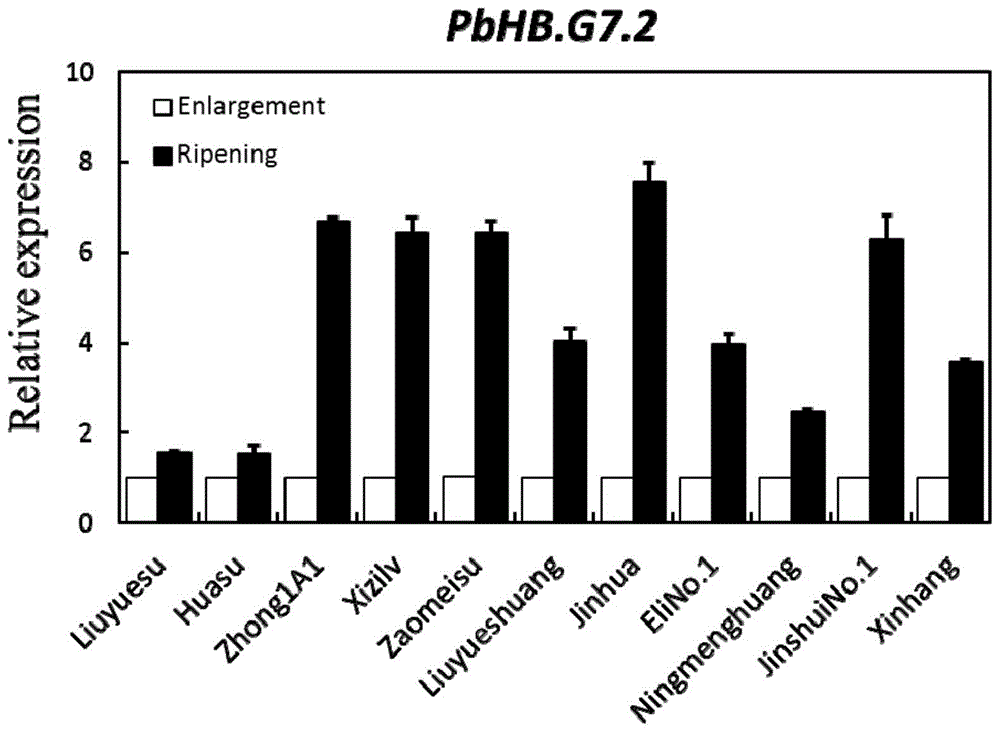
图1为本发明中转录因子pbhb.g7.2在11个梨品质的成熟与未成熟果实中基因表达模式图;
图2为超表达载体psak277-pbhb.g7.2的示意图;
图3为在梨果实中瞬时超表达转录因子pbhb.g7.2的果实表型图;
图4为在梨果实中瞬时超表达转录因子pbhb.g7.2的果实乙烯释放量示意图;
图5为在梨果实中瞬时超表达转录因子pbhb.g7.2,pbhb.g7.2和pbacs1b基因的表达量示意图;
图6为pbhb.g7.2与pbhb.g1蛋白互作的酵母双杂交实验结果展示图;
图7为pbhb.g7.2与pbhb.g2.1蛋白互作的酵母双杂交实验结果展示图;
图8为pbhb.g7.2与pbhb.g1、pbhb.g2.1蛋白互作的荧光互补实验结果展示图;
图9为转录因子pbhb.g7.2、pbhb.g1和pbhb.g2.1和pbacs1b基因启动子的双荧光素酶报告实验的载体构建示意图;
图10为转录因子pbhb.g7.2、pbhb.g1和pbhb.g2.1和pbacs1b基因启动子的双荧光素酶实验结果示意图;
图11为转录因子pbhb.g7.2、pbhb.g1和pbhb.g2.1和pbacs1b基因启动子的酵母单杂实验结果展示图;
图12为经生物素标记的pbacs1b基因启动子序列展示以及pbhb.g7.2蛋白和pbacs1b基因启动子的凝胶电泳迁移率实验结果展示图;
图13为经生物素标记的pbacs1b基因启动子序列展示以及pbhb.g1和pbhb.g2.1蛋白与pbacs1b基因启动子的凝胶电泳迁移率实验结果展示图;
具体实施方式
以下结合具体实施例对本发明做出详细的描述。根据以下的描述和这些实施例,本领域技术人员可以确定本发明的基本特征,并且在不偏离本发明精神和范围的情况下,可以对本发明做出各种改变和修改,以使其适用各种用途和条件。
实施例1pbhb.g7.2基因分离克隆及表达分析
本发明提供了一个梨hb基因pbhb.g7.2,所述pbhb.g7.2基因的序列如seqidno.1所示。
在本发明中所述梨hb基因pbhb.g7.2优选的来源于‘翠冠’梨(p.pyrifolianakaicv.‘cuiguan’)。
在本发明中所述梨hb基因pbhb.g7.2通过以下方法获得:从翠冠梨果肉中提取获得翠冠梨的rna;将所述rna反转录获得cdna;以所述cdna为模板,用正向引物f1和反向引物r1扩增获得所述pbhb.g7.2基因全长。
在本发明中所述翠冠梨的rna的提取采用本领域常规的植物组织rna提取方法即可,无其他特殊限定,在本发明具体实施过程中采用planttotalrnaisolationkitplus(购自福际生物技术有限公司,成都)试剂盒进行翠冠梨果肉的rna提取,按照试剂盒说明书操作。
本发明在获得‘翠冠’梨rna后,参照全式金公司transscriptone-stepgdnaremovalandcdnasynthesissupermix反转录试剂盒说明书进行反转录操作,反转录获得cdna。
本发明在获得所述cdna后,以获得的cdna为模板,用正向引物f1和反向引物r1扩增获得所述pbhb.g7.2基因全长。扩增基因引物对为:pbhb.g7.2-f1:5’-atgggttttgatcatgaagcttg-3’(seqidno.5);pbhb.g7.2-r1:5’-ctagcaagctgtagatggatgagtact-3’(seqidno.6)。在本发明具体实施过程中,优选的所述扩增体系为50μl体系,所述扩增体系包括100ng模板cdna,5×q5反应缓冲液(q5reactionbuffer),10mmdntps,1μlq5高保真聚合酶(q5high-fidelitydnapolymerase),1.0μm正向引物和1.0μm的反向引物。本发明中所述的5×q5缓冲液和q5高保真聚合酶优选的购自newenglandbiolabs公司。本发明中所述扩增反应的程序优选的为:98℃,30s;98℃变性10s、60℃退火30s、72℃延伸60s,35个循环;循环完成后,72℃延伸5min。本发明中所述扩增反应优选的在roche480(appliedbiosystem)扩增仪中完成。
本发明在扩增获得一条单一的pcr条带产物后,优选的进行1%的琼脂糖凝胶电泳检测,用axygen小量胶回收试剂盒(购自axygen公司,美国)回收dna片段,步骤参照使用说明。回收纯化的dna溶液回收纯化的dna溶液与peasy-bluntzero载体(购自trans公司)进行连接反应(参照使用说明操作),连接反应体系中pbhb.g7.2基因回收产物与peasy-bluntzero载体的摩尔比为4:1。连接反应总体积为5μl,包括4μl纯化的pcr回收产物和1μl的peasy-bluntzero载体。室温(20℃~37℃)反应5分钟,反应结束后,将连接产物置于冰上3分钟,然后将5μl连接产物,用热激法(参照《分子克隆实验手册》第三版,科学出版社,2002)转化大肠杆菌dh5α,在含有50mg/l卡那霉素的lb固体平板中筛选阳性克隆,挑取5个单克隆测序(由上海生工生物技术有限公司完成)。测序结果表明,pbhb.g7.2基因全长为912bp,其核苷酸序列为seqidno.1所示,blast的结果分析证明从梨中得到的基因为一个homeobox家族成员,申请人将这个基因命名为pbhb.g7.2。本发明克隆得到的基因是梨中homeobox基因。但是值得一提的是,还未见相关文献报道关于梨中homeobox基因的功能。
为分析pbhb.g7.2基因是否响应梨果实成熟过程,采用实时定量pcr分析该基因在不同梨品种的成熟和未成熟果实中表达量的变化。根据基因全长序列,设计1对该基因的荧光定量引物对,pbhb.g7.2-f2:5’-caagaaacctaaatctacgcccgc-3’(seqidno.7);pbhb.g7.2-r2:5’-cctgttgctgtttgtttcattgatttg-3’(seqidno.8)。实时荧光定量pcr在roche480iipcr仪上进行。20μl实时荧光定量pcr体系如下:syrb荧光染料10μl(购自roche公司)、300ngcdna1μl、上述上/下游引物各1μl和rnase-free水7μl;反应程序如下:95℃10min,95℃10s、65℃10s、72℃30s、45个循环,循环完成后,72℃10min。结果如图1所示,与未成熟果实相比,该基因在各个梨品种的成熟果中高度表达(图1)。
优选的,所述梨品种为‘六月酥’、‘华酥’、‘中1a1’、‘西子绿’、‘早美酥’、‘六月爽’、‘金华’、‘鄂梨1号’、‘柠檬黄’、‘金水1号’、‘新杭’。
实施例2:瞬时超表达pbhb.g7.2
在pbhb.g7.2基因扩增引物两端分别加上hindiii和xbai两个酶切位点,其扩增引物为(正向引物:pbhb.g7.2-f3:5’-tccaaagaattcaaaaagcttatgggttttgatcatgaagcttg-3’(seqidno.9);反向引物:pbhb.g7.2-r3:5’-tcattaaagcaggactctagactagcaagctgtagatggatgagtact-3’(seqidno.10))。50μl的扩增体系中包括100ngtemplatedna,5×q5反应缓冲液(q5reactionbuffer),10mmdntps,1uq5高保真聚合酶(q5high-fidelitydnapolymerase)(上述缓冲液和q5高保真聚合酶均购自newenglandbiolabs公司),1.0μm上述引物。pcr反应在roche480(appliedbiosystem)扩增仪上按以下程序完成:98℃,30s;98℃变性10s、60℃退火30s、72℃延伸60s,35个循环;循环完成后,72℃延伸5min,将产物回收。同时用hindiii和xbai双酶切psak277载体,回收产物并连接,从而得到psak277-pbhb.g7.2重组载体(如图2所示),随后转化大肠杆菌,取适量菌液涂布壮观霉素抗性的lb平板,平板置于37℃过夜培养,挑取单克隆培养10-12h,进行菌液pcr检测,提取阳性单克隆的质粒,送至北京擎科生物公司测序鉴定。质粒提取按照质粒提取试剂盒说明书(omega)进行。将上述提取的psak277-pbhb.g7.2质粒转化至农杆菌gv3101,每50μl感受态加入约1μg质粒,用手轻轻拨打管底混匀,依次于冰上静置5min、液氮5min、37℃水浴5min、冰浴5min,随后加入800μl无抗性lb液体培养基,于28℃振荡培养3小时。取100μl菌液涂布在壮观霉素和利福平抗性的lb固体平板上,平板倒置于28℃培养48h后挑取单克隆进行pcr扩增鉴定。
用农杆菌注射梨果实进行瞬时转化步骤如下:将上述含有psak277-pbhb.g7.2重组质粒农杆菌进行摇菌培养(含有壮观霉素50μg/ml,利福平25μg/mllb培养基),28℃振荡培养过夜,第二天4000rpm离心5min收集菌,随后用悬浮液(40mmmes,ph5.6,40mmmgcl2and300μmacetosyringone)悬浮至od600为0.8。将悬浮菌置于室温下3h后用于梨果实注射。用1ml注射器针头直接将菌液注射到成熟前10天的翠冠果实中。将含有不同载体的果实分别放置在不同的2l密封瓶中,每个包含至少4个果实。在黑暗的光线下放置24h后,置于正常光照条件下继续放置24h(如图3所示)。每两天测量一次乙烯的产量。乙烯的测定方法按照乙烯检测仪(购自普利通电子科技有限公司,深圳)说明书进行操作。注射部位于注射后8天取样,提取rna,将rna反转录成cdna,进行qrt-pcr分析,所用引物为pbhb.g7.2基因特异引物(pbhb.g7.2-f2:5’-caagaaacctaaatctacgcccgc-3’(seqidno.7);pbhb.g7.2-r2:5’-cctgttgctgtttgtttcattgatttg-3’(seqidno.8))和pbacs1b基因特异引物(pbacs1b-f1:5’-aaggctcgcaaagaggcacg-3’(seqidno.11);pbacs1b-r1:5’-aacaccttcctgctcaaaccttctg-3’(seqidno.12)),反应程序为94℃,3分钟;94℃变性30秒、65℃退火45秒、72℃延伸45秒,30个循环;72℃,延伸10分钟。用actin做内参基因,引物序列为:正向引物actin-f:5'-caatgtgcctgccatgtatg-3’(seqidno:13)';反向引物actin-r:5'-ccagcagcttccattccaat-3'(seqidno:14)。
结果表明,与对照组相比,过表达pbhb.g7.2基因的梨果实乙烯释放量显著增加(如图4所示),成熟加快,与乙烯合成相关的酶pbacs1b表达量也显著上升(如图5所示)。
实施例3:酵母双杂交实验
本发明提供两个与梨转录因子pbhb.g7.2蛋白互作的转录因子pbhb.g1和pbhb.g2.1,都属于homeobox家族成员。所述pbhb.g1基因的核苷酸序列如seqidno.15所示,包含1035bp的开放阅读框,编码345个氨基酸,其氨基酸序列如seqidno.16所示。所述pbhb.g2.1基因的核苷酸序列如seqidno.17所示,包含1107bp的开放阅读框,编码369个氨基酸,其氨基酸序列如seqidno.18所示。
在本发明中所述两个梨hb基因pbhb.g1和pbhb.g2.1优选的来源于‘翠冠’梨(p.pyrifolianakaicv.‘cuiguan’)。
在pbhb.g7.2基因扩增引物两端分别加上ecori和bamhi两个酶切位点,其扩增引物为(正向引物:pbhb.g7.2-f4:5’-atggccatggaggccgaattcgagatgggttttgatcatgaagcttg-3’(seqidno.19);反向引物:pbhb.g7.2-r4:5’-ccgctgcaggtcgacggatccgcagactagcaagctgtagatggatg-3’(seqidno.20));在pbhb.g1和pbhb.g2.1基因扩增引物两端分别加上ecori和bamhi两个酶切位点,pbhb.g1扩增引物为(正向引物:pbhb.g1-f1:5’-gccatggaggccagtgaattcatggagctagctctgagcttagg-3’(seqidno.21);反向引物:pbhb.g1-r1:5’-cagctcgagctcgatggatcctcatgcagcagcaccttgatgctg-3’(seqidno.22)),pbhb.g2.1基因扩增引物为(正向引物:pbhb.g2.1-f1:5’-gccatggaggccagtgaattcatggagctttgtttgagcttaggag-3’(seqidno.23);反向引物:pbhb.g2.1-r1:5’-cagctcgagctcgatggatcctcacgaagctgcggaagagggat-3’(seqidno.24))。
将pbhb.g7.2基因扩增产物连接至ecori和bamhi双酶切的pgbkt7载体上,将pbhb.g1和pbhb.g2.1基因扩增产物连接至ecori和bamhi双酶切的pgadt7载体上,都转化大肠杆菌并鉴定阳性结果后,分别提取质粒。扩增、连接和质粒提取方法与上述所示相同。酵母双杂实验方法和结果分析参照酵母单杂实验系统使用手册(美国clontech公司)。
结果显示,除阳性对照外,含有pbhb.g7.2和pbhb.g1与含有pbhb.g7.2和pbhb.g2.1转录因子组合的酵母菌落能够在缺乏腺嘌呤,组氨酸,亮氨酸和色氨酸的缺陷培养基(qod)上生长,而对照组不能,说明pbhb.g7.2蛋白与pbhb.g1和pbhb.g2.1蛋白互作(如图6~7所示)。
实施例4:烟草荧光素酶互补实验
在pbhb.g7.2基因扩增引物两端分别加上bamhi和sali两个酶切位点,其扩增引物为(正向引物:pbhb.g7.2-f5:5’-ctcggtacccggggatccgagatgggttttgatcatgaagcttg-3’(seqidno.25);反向引物:pbhb.g7.2-r5:5’-gtacgagatctggtcgacgcaagctgtagatggatg-3’(seqidno.26));在pbhb.g1和pbhb.g2.1基因扩增引物两端分别加上kpni和bamhi两个酶切位点,pbhb.g1扩增引物为(正向引物:pbhb.g1-f2:5’-gcgtcccggggcggtaccatggagctagctctgagcttagg-3’(seqidno.27);反向引物:pbhb.g1-r2:5’-agtccatttgttggatcctgcagcagcaccttgatgctg-3’(seqidno.28)),pbhb.g2.1基因扩增引物为(正向引物:pbhb.g2.1-f2:5’-gcgtcccggggcggtaccatggagctttgtttgagcttaggag-3’(seqidno.29);反向引物:pbhb.g2.1-r2:5’-agtccatttgttggatcccgaagctgcggaagagggat-3’(seqidno.30))。
将pbhb.g7.2基因扩增产物连接至bamhi和sali双酶切的pcambia1300-nluc载体上,将pbhb.g1和pbhb.g2.1基因扩增产物连接至kpni和bamhi双酶切的pcambia1300-cluc载体上,都转化大肠杆菌并鉴定阳性结果后,分别提取质粒。扩增、连接和质粒提取方法与上述所示相同。将重组质粒转化农杆菌gv3101,农杆菌的转化、培养和收集方法与上述相同,把悬浮液调整至od600=0.6,在28℃的黑暗中放置3-4h,按照不同的组合以1:1的体积比例混合,注射到野生烟叶片中。注射3天后,检测注射叶片中荧光素酶活性,检测方法依照用户使用说明书(美国promega公司)。
结果显示,注射了含有nluc-pbhb.g7.2和cluc-pbhb.g1的重组农杆菌与含有nluc-pbhb.g7.2和cluc-pbhb.g2.1的重组农杆菌叶片区域能够产生荧光,其他区域则不能。说明pbhb.g7.2蛋白与pbhb.g1和pbhb.g2.1蛋白互作(如图8所示)。
实施例5:烟草双荧光素酶实验
将pbacs1b基因起始密码子前2000bp启动子扩增引物两端分别加上kpni和hindiii两个酶切位点,其扩增引物为(pbacs1b-f2:5’-ctatagggcgaattgggtacctcaaacaaggaaattggtgtatg-3’(seqidno.31);pbacs1b-r2:5’-caggaattcgatatcaagctttttcttggttccaaagaacacac-3’(seqidno.32))。在pbhb.g1和pbhb.g2.1基因扩增引物两端分别加上hindiii和xbai两个酶切位点,pbhb.g1扩增引物为(正向引物:pbhb.g1-f3:5’-tgttccggattacgctgttaacatggagctagctctgagcttagg-3’(seqidno.33);反向引物:pbhb.g1-r3:5’-ctcattaaagcaggactctagatcatgcagcagcaccttgatgctg-3’(seqidno.34)),pbhb.g2.1扩增引物为(正向引物:pbhb.g2.1-f3:5’-tgttccggattacgctgttaacatggagctttgtttgagcttaggag-3’(seqidno.35);反向引物:pbhb.g2.1-r3:5’-ctcattaaagcaggactctagatcacgaagctgcggaagagggat-3’(seqidno.36)).
将pbacs1b基因启动子扩增产物连接至kpni和hindiii双酶切的pgreenii0800-luc载体上,将pbhb.g1和pbhb.g2.1基因扩增产物连接至hindiii和xbai双酶切的psak277载体上,都转化大肠杆菌并鉴定阳性结果后,分别提取质粒(如图9所示)。扩增、连接和质粒提取方法与上述所示相同。将重组质粒转化农杆菌gv3101,农杆菌的转化、培养和收集方法与上述相同,把悬浮液调整至od600=0.8,将悬浮菌置于室温黑暗处3h后用于烟草叶片注射。根据不同的组合,将含有转录因子重组质粒和启动子重组质粒的复悬液按9:1的比例混合,然后用1ml注射器注射到5-6周龄的野生烟草叶片中。将烟草在温室里培养3天后,按照双荧光素酶实验试剂盒使用说明书(美国promega公司)进行荧光酶的测定。通过实验组和对照组之间萤火虫荧光素酶(luc)和海肾荧光素酶(ren)的比值来判断pbhb.g7.2基因对pbacs1b启动子的作用。
结果显示,转录因子pbhb.g7.2、pbhb.g1和pbhb.g2.1与pbacs1b启动子的组合luc/ren比值显著高于对照组。而将pbhb.g7.2与pbhb.g1和pbhb.g2.1组合后再与pbacs1b启动子进行试验时,luc/ren比值与对照组相比没有显著差异。说明转录因子pbhb.g7.2、pbhb.g1和pbhb.g2.1对pbacs1b启动子都有激活作用,但是pbhb.g7.2与pbhb.g1和pbhb.g2.1互作后对pbacs1b启动子不再有激活作用(如图10所示)。
实施例6:酵母单杂实验
在pbacs1b基因起始密码子前2000bp启动子扩增引物两端分别加上kpni和xhoi两个酶切位点,其扩增引物为(pbacs1b-f3:5’-cttgaattcgagctcggtacctcaaacaaggaaattggtgtatg-3’(seqidno.37);pbacs1b-r3:5’-atacagagcacatgcctcgagtttcttggttccaaagaacacac-3’(seqidno.38))。在pbhb.g7.2基因扩增引物两端分别加上ecori和bamhi两个酶切位点,pbhb.g7.2基因扩增引物为(正向引物:pbhb.g7.2-f6:5’-gccatggaggccagtgaattcatgggttttgatcatgaagcttg-3’(seqidno.39);反向引物:pbhb.g7.2-r6:5’-cagctcgagctcgatggatccctagcaagctgtagatggatgagtact-3’(seqidno.40))。将pbacs1b启动子扩增产物连接至kpni和xhoi双酶切的pabai载体上,将pbhb.g7.2基因扩增产物连接至ecori和bamhi双酶切的pgadt7载体上,转化大肠杆菌并鉴定阳性结果后,分别提取质粒。扩增、连接和质粒提取方法如上述所示。用酵母单杂实验检测转录因子pbhb.g7.2、pbhb.g1和pbhb.g2.1与pbacs1b启动子之间的关系。酵母单杂实验操作方法和结果分析参照酵母单杂实验系统使用手册(日本takara公司)。
结果显示,转录因子pbhb.g7.2、pbhb.g1和pbhb.g2.1分别与pbacs1b启动子组合后,能够使重组酵母菌落在含有金担子素(aba)的亮氨酸缺陷培养基(-leu)上生长,而对照组不能,说明转录因子pbhb.g7.2、pbhb.g1和pbhb.g2.1都能够直接结合pbacs1b启动子(如图11)。
实施例7:凝胶电泳迁移率实验
在pbacs1b基因起始密码子前200-234bp、980-1020bp和1767-1800bp启动子序列在上海生工生物公司进行生物素标记,被标记的序列分别为(biotin-pbacs1b-1:5’-cttatgggtcggtttttctacatttgaattttga-3’(seqidno.41)、(biotin-pbacs1b-2:5’-cctcaaggaaatagtaataccctttagatttgaagtccta-3’(seqidno.42)和(biotin-pbacs1b-3:5’-ttatatgttgtatcttatttcatcgttgattac-3’(seqidno.43)。在pbhb.g7.2、pbhb.g1和pbhb.g2.1基因扩增引物两端分别加上xho1和xba1两个酶切位点,pbhb.g7.2扩增引物为(正向引物:pbhb.g7.2-f5:5’-gagctcggtacccggggatccatgagaaagaaactcagacttacaaaagaacaa-3’(seqidno.44);反向引物:pbhb.g7.2-r5:5’-cgcgtacgagatctggtcgacgcaagctgtagatggatgagtact-3’(seqidno.45))。pbhb.g1扩增引物为(正向引物:pbhb.g1-f4:5’-atggagctcggtaccctcgagatggagctagctctgagcttagg-3’(seqidno.46);反向引物:pbhb.g1-r4:5’-agcagagattacctatctagatgcagcagcaccttgatgctg-3’(seqidno.47)),pbhb.g2.1扩增引物为(正向引物:pbhb.g2.1-f4:5’-atggagctcggtaccctcgagatggagctttgtttgagcttaggag-3’(seqidno.48);反向引物:pbhb.g2.1-r4:5’-agcagagattacctatctagacgaagctgcggaagagggat-3’(seqidno.49))将pbhb.g7.2、pbhb.g1和pbhb.g2.1基因扩增产物连接至xho1和xba1双酶切的pcold载体上,转化大肠杆菌并鉴定阳性结果后,分别提取质粒。扩增、连接和质粒提取方法如上述所示。将pcold-pbhb.g7.2、pcold-pbhb.g1和pcold-pbhb.g2.1重组质粒按照使用手册转化至大肠杆菌bl21中(北京全式金公司),pcr鉴定阳性结果后,培养在37℃含有氨苄50μg/ml液体lb培养基中,培养至菌液od600为0.6时,加入iptg(isopropylb-d-1-thiogalactopyranoside;上海生工生物工程股份有限公司)至最终浓度为0.5mm并在15℃,150r.p.m环境下培养16-24h诱导蛋白表达。用离心机收集菌体,在1×pbs缓冲液中重悬(上海生工生物工程股份有限公司)。用超声波破碎后离心,取上清液用重力柱(美国clontech公司)纯化,纯化过程参照使用手册。
将纯化后得到的pbhb.g7.2、pbhb.g1和pbhb.g2.1蛋白分别与被生物素标记和未被生物素标记的pbacs1b-1、pbacs1b-3和pbacs1b-2基因启动子进行凝胶电泳迁移率实验,实验步骤和结果分析参照alightshiftchemiluminescentemsa试剂盒使用说明书(美国thermo公司)。
结果显示,pbhb.g7.2、pbhb.g1和pbhb.g2.1都蛋白能够特异结合pbacs1b基因启动子(如图12和13所示)。
综上结论可知,梨的pbhb.g7.2转录因子能够结合并激活乙烯合成通路中的pbacs1b基因启动子,促进pbacs1b基因的表达,合成更多的乙烯,从而加速梨果实成熟过程,而转录因子pbhb.g1和pbhb.g2.1能够与pbhb.g7.2发生蛋白互作,也能结合并激活pbacs1b基因启动子,与pbhb.g7.2共同调控pbacs1b基因。
由上述实施例可知,本发明提供的梨hb基因pbhb.g7.2,具有促进果实乙烯合成,加速果实成熟的功能。能够应用于缩短果实成熟时间。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
参考文献
[1]潘晓琪.slgr基因调控番茄果实成熟的机理研究[d].中国农业大学,2015.
[2]唐粉玲,林妃,李羽佳,李艳霞,许奕,黄东梅,李敬阳.果实成熟及品质调控遗传研究进展[j].分子植物育种,2018,16(04):1320-1326.
[3]李珊.番茄果实中重要转录因子mads-rin的功能分析[d].中国农业大学,2017.
[4]guo,h.w.,&ecker,j.r.(2003).plantresponsestoethylenegasaremediatedbyscf(ebf1/ebf2)-dependentproteolysisofein3transcriptionfactor.cell,115(6),667-677.
[5]阚超楠,高阳,陈明,陈楚英,万春鹏,刘善军,陈金印.不同采后处理对翠冠梨果实品质的影响[j].核农学报,2019,33(03):518-529.
[6]ariel,f.d.,p.a.manavella,c.a.dezar&r.l.chan(2007)thetruestoryofthehd-zipfamily.trendsinplantscience,12,419-426.
[7]vandergraaff,e.,t.laux&s.a.rensing(2009)thewushomeobox-containing(wox)proteinfamily.genomebiol,10,248.
[8]hake,s.,h.m.smith,h.holtan,e.magnani,g.mele&j.ramirez(2004)theroleofknoxgenesinplantdevelopment.annurevcelldevbiol,20,125-51.
[9]belles-boix,e.,o.hamant,s.m.witiak,h.morin,j.traas&v.pautot(2006)knat6:anarabidopsishomeoboxgeneinvolvedinmeristemactivityandorganseparation.plantcell,18,1900-7.
[10]du,j.,s.d.mansfield&a.t.groover(2009)thepopulushomeoboxgenearborknox2regulatescelldifferentiationduringsecondarygrowth.theplantjournal,60,1000-1014.
[11]aoyama,t.,c.h.dong,y.wu,m.carabelli,g.sessa,i.ruberti,g.morelli&n.h.chua(1995)ectopicexpressionofthearabidopsistranscriptionalactivatorathb-1altersleafcellfateintobacco.plantcell,7,1773-85.
[12]steindler,c.,a.matteucci,g.sessa,t.weimar,m.ohgishi,t.aoyama,g.morelli&i.ruberti(1999)shadeavoidanceresponsesaremediatedbytheathb-2hd-zipprotein,anegativeregulatorofgeneexpression.development,126,4235-45.
[13]prigge,m.j.,d.otsuga,j.m.alonso,j.r.ecker,g.n.drews&s.e.clark(2005)classiiihomeodomain-leucinezippergenefamilymembershaveoverlapping,antagonistic,anddistinctrolesinarabidopsisdevelopment.theplantcell,17,61-76.
[14]nakamura,m.,h.katsumata,m.abe,n.yabe,y.komeda,k.t.yamamoto&t.takahashi(2006)characterizationoftheclassivhomeodomain-leucinezippergenefamilyinarabidopsis.plantphysiology,141,1363-1375.
序列表
<110>南京农业大学
<120>梨转录因子pbhb.g7.2及其在促进果实成熟中的应用
<160>49
<170>siposequencelisting1.0
<210>1
<211>912
<212>dna
<213>翠冠梨(pyrusspp)
<400>1
atgggttttgatcatgaagcttgtaacactggccttggactcagcctaggttgtcaagct60
aatccagatcatcatacaaatttgcaggctttcgatctcgatcaccaaaagaagaagaaa120
atacagttgaaatacgatcacctattgccggctctaaccctaggtccatcatccggggat180
tcgaagattgaggccgccgaatcagatgatcatttgcaccaagtagtacatgtgcaagca240
aaagaacaggtctcttctccctgcagcggcgccgtttcgtcgttctccaactcatctagt300
ttcaagagggacagagatataggtgttgaagagatagagggagaagaagagagagtagtg360
aatatcactacttcatcaagagtaagcgatgaattatatgatcaagatcaagatcatgag420
ggcagtcccagaaagaaactcagacttacaaaagaacaatcttccactttagaagacaac480
ttcagagaacacactactctcaatccaaaacaaaagcaagaactagcaagaaagctaaat540
ctacgcccgcgacaagttgaagtctggttccaaaacaggagggccaggaccaagctgaag600
caaacagaagcagatttcgagttattgaagcaatgttgtgaaacattgaaagaagagaac660
agaaggctgcacaaggagctgcaagagctcaaatcaatgaaacaaacagcaacagcagca720
gcacccttttacatgcagtttccaactgccactctcaccatgtgtccctcctgtgagaaa780
atctgcaacggcggcgatcatcgtaatcataatcataacaacgggtcatcgacgagtcca840
ttcttaatcggatcgaataagactcacttgatcttcaacccctacagtactcatccatct900
acagcttgctag912
<210>2
<211>303
<212>prt
<213>翠冠梨(pyrusspp)
<400>2
metglypheasphisglualacysasnthrglyleuglyleuserleu
151015
glycysglnalaasnproasphishisthrasnleuglnalapheasp
202530
leuasphisglnlyslyslyslysileglnleulystyrasphisleu
354045
leuproalaleuthrleuglyproserserglyaspserlysileglu
505560
alaalagluseraspasphisleuhisglnvalvalhisvalglnala
65707580
lysgluglnvalserserprocysserglyalavalserserpheser
859095
asnserserserphelysargaspargaspileglyvalglugluile
100105110
gluglygluglugluargvalvalasnilethrthrserserargval
115120125
seraspgluleutyraspglnaspglnasphisgluglyserproarg
130135140
lyslysleuargleuthrlysgluglnserserthrleugluaspasn
145150155160
phearggluhisthrthrleuasnprolysglnlysglngluleuala
165170175
arglysleuasnleuargproargglnvalgluvaltrppheglnasn
180185190
argargalaargthrlysleulysglnthrglualaaspphegluleu
195200205
leulysglncyscysgluthrleulysglugluasnargargleuhis
210215220
lysgluleuglngluleulyssermetlysglnthralathralaala
225230235240
alaprophetyrmetglnpheprothralathrleuthrmetcyspro
245250255
sercysglulysilecysasnglyglyasphisargasnhisasnhis
260265270
asnasnglyserserthrserpropheleuileglyserasnlysthr
275280285
hisleuilepheasnprotyrserthrhisproserthralacys
290295300
<210>3
<211>1488
<212>dna
<213>人工序列(artificialsequence)
<400>3
atggcctcatctgcaagtgagaatcgcttactgctatccaagattgcaaccaatgaaaaa60
catggtgagaactcgccgtacttcgacggatggaataagtacaatcaaaacccatttcac120
ctaactgaaaatcctgaaggagttatccagatgggtctggcagaaaatcagctttccttc180
gacttggttgaagagtggattaggaaaaatcccaaagcctctatttgcactgctgaagga240
attgagaagttcagaaacgtggccaattttcaagactatcatggcttaccagagttcaga300
caggccattgctacgttcatgtcgaaagcaagaggtggtagagtcacatttgatcctcat360
cgcgtagttatgagtggaggagccaccggagcaaatgagctggtcatgttctgtttggcc420
gaccgcggcgatgctttccttatcccctcaccgtactatccagcattttaccgagacctc480
ggatggagaactggagtccaaattgtcccggtcgattgtgatagctccaacaatttcaaa540
ataaccaaggaagcactggaagcagcttatgaaaaagcccaaaacaacaacatcaatgtc600
aagggcttgatcataacaaacccatcaaatccattaggcacaaccctagacaaagacaca660
cttgaaagccttgtcacatttataaaccaaaagaacattcacttggtttgtgatgaaatc720
tatgcagccacagtcttcagctccccaaaattcacatgcatcaccgaggttatacaaaac780
atgaattgcaaccccaacctaatccacattgtctacagtttgtccaaggacatggggttc840
ccgggactgagagtcggcatcgtttactcctacaacgatgacgtggtgaacattggccga900
aaaatgtcaagttttgggctggtctcatcccaaactcagcacatgcttgcgtccatgctt960
tcggacgaagattttgtcgagaagttcctcaccacaagcgcaaaaaggctcgcaaagagg1020
cacggggtcttcactgatgggcttgaggaagtgggaatcaactgcttgaagagcaatgcc1080
ggccttttctgttggatggacttaaggaggctattgaaagatcaaacgtttgatggcgaa1140
atggtgttgtggcgtgtaatcgttaatgaagtgaagctcaacgtttctccgggctcttcg1200
tttaaatgcgtcgagcctggttggtttagggtttgcttcgccaacatggatgatgacaca1260
gtggaagttgcactcaaaaggattagggcatttgtagggcaaggaaagaaagctcaagaa1320
caagcacaagtgaaaagtcctaagaagcgttggcagagcaatctaaggctgagcttttca1380
tcatcatccagaaggtttgagcaggaaggtgttagtgttatgtcaccacgtatgatgtct1440
cctcattcaccaatgcctcactcacctctccttcgagctacgacttaa1488
<210>4
<211>2000
<212>dna
<213>人工序列(artificialsequence)
<400>4
tcaaacaaggaaattggtgtatgtcaatttataaattcataattttattcaaattctaag60
tttatttccctcatccaaacatagtgtaatggaattacaaaacggataaatggtcttatt120
aaagaggtgtgggtacagcaggtgtgtgattagattttataataaaaacttttgatgcaa180
tttgtagtaaaaccattcaattatatgttgtatcttatttcatcgttgattacataaact240
catatttgaatgtatacatttcgatagtctcagaaatataatgtaacataatgtttaaaa300
gtgtgatacggaaatgatttgagtgaatgattcgaaaggtagttaagtaacataaaaagt360
tgcaattatactattatacagcaaaccaatacataagtataacaatgaaaattagtaata420
tctcaagtgaaacaaatacatacagtcatgcaaaccaatacatacgtcagcaaatgtgaa480
agggatgcttcaaatcaatgactaatacaagtgagtgtatgcgctaaagtcaaaatacac540
aatcatcgcctttagttggtgctataccacttccggggcgtatgttttatcttttaaatc600
taaaatttgaaatttgtaccacccttatagagtaataagaagcaaccttctccaccctcc660
cacagtccacaaaccacctttttgatttgcttggcttttcggtactaccagagaccaccg720
cgtgaaatattacaacaaaaggttagggggccggaaagtacattttctgagatgccctca780
cttctcatcagctatttttggcaggatcatgatataagtcggttttccaactatttatga840
ggtatttaagtgcattcccatttgagcacacgagggatttaattagtaattttagaaaag900
atggcaaaataatgccatggagcacattaacgtggagaccaagttagtatctcacaaagc960
aaccgtggaggaaaaggaaacctcaaggaaatagtaataccctttagatttgaagtccta1020
ataatgtgtgagtcttgtgcatttactactcttggtgcacaagtccaaaaccgaccaaat1080
tccaaagttctaacttctaactatatgctttcagtcagcaccaaaatcttcattctttct1140
ttcatggtgcttcggggaagcttcattcctagaaacataacacgtccattgcagaaaaaa1200
ttaacacaaaagcaaatacctaactaatagaaaaatgaaaaaacaactttttatatacat1260
aaaaagaaccgtcaatgatgtgtctgtgtacatgttagtattttatttattagaaacaaa1320
tgaagttgagataagcccttatttgattaaatgatatattgttgtgtgacgatagagaaa1380
tgctatggagattcaatttgtaaataaaatttgcaaaccaaatgatgtatcactaatcgg1440
aaatgagtatgtttatcaacgattaagtaataatccactcattaactttcatgtcattta1500
gtttataaaattttgactataaatttactctccttaacattatttgttacaatattacca1560
tgtggcatgtcaattgagaaaaaattgatgagttataggaatgtttcatgaaaggtgatg1620
tatgaggaccaccaagaggtacgctgccggtgtacacgtggctgtgcaagtttttacatg1680
cttgatggttcggtatgatgtccaacacgcaagcaattacgattcattgaccaacactgg1740
cacaattaaattatgttatttcctttcttatgggtcggtttttctacatttgaattttga1800
agtggacttttcaatcaaattttgcttcctgtgcagcttgccatttaatcttctactgcc1860
tataaatctacaattgccttaccctttgcttcacacttcattgtttaaaaaacataagca1920
ctactgtcgttccagacttccagtagtataatcattccctccagctatatagcttgtgtg1980
tgttctttggaaccaagaaa2000
<210>5
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>5
atgggttttgatcatgaagcttg23
<210>6
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>6
ctagcaagctgtagatggatgagtact27
<210>7
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>7
caagaaacctaaatctacgcccgc24
<210>8
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>8
cctgttgctgtttgtttcattgatttg27
<210>9
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>9
tccaaagaattcaaaaagcttatgggttttgatcatgaagcttg44
<210>10
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>10
tcattaaagcaggactctagactagcaagctgtagatggatgagtact48
<210>11
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>11
aaggctcgcaaagaggcacg20
<210>12
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>12
aacaccttcctgctcaaaccttctg25
<210>13
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>13
caatgtgcctgccatgtatg20
<210>14
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>14
ccagcagcttccattccaat20
<210>15
<211>1035
<212>dna
<213>翠冠梨(pyrusspp)
<400>15
atggagctagctctgagcttaggggatgcttcaaagccgtttctctttctcgacaaagct60
ccaaagatgggcggcaacatggatatagggttttgcatgggcttggggactgctgctttc120
agtgctgcacgatcggatgagagtagcagaaagagcacctatgaagtttatgatcaggag180
gagagaagaagtaggatttcatcagatccacctcttcagcttaatctcttgccttctgct240
ccagttcctcgtagccacgtttcttctcagcttcgcttcccatggcttactgataatctg300
gctgtgtctgaaccggccggttcatcagatggaccgggaatagggttggatgtgaaccgg360
atgccaggagtgttggcctcgactgaggaggccgaagatgcggcggctcctctgtcatct420
ccgaacagcacggtttcatcgtttcagatggattttggaatgagaaatggaggaagatcg480
agcaagagagatttggatgttgacgccgacagagcgagtgatgacgaggagaacggatca540
actcggaagaaactcaggctctctaaagatcaatcggcttttcttgaagagagcttcaaa600
gagcacagcactctcaatcctaagcaaaaacttgctctggctaaacagttgaatcttcgt660
ccacggcaagtggaagtgtggtttcagaatcgaagagcaaggacaaagttgaagcagaca720
gaagtagattgtgagtacttaaagagatgctgtgaaacactgacagaagagaataggagg780
ttacagaaggaactgcaagacttgagagctttgaagacctctcagcctttctacatgcag840
ctgcctgccaccacactcaccatgtgcccctcatgtgaacgcgtggtcaccaccgcctca900
gccaacacctccaccaccaccaccaccaaccaccaccaccacataaagtctgctctgaac960
aacaacaagccaaggttaaatccatttactgctgctcaagtccacatacagcagcatcaa1020
ggtgctgctgcatga1035
<210>16
<211>344
<212>prt
<213>翠冠梨(pyrusspp)
<400>16
metgluleualaleuserleuglyaspalaserlyspropheleuphe
151015
leuasplysalaprolysmetglyglyasnmetaspileglyphecys
202530
metglyleuglythralaalapheseralaalaargseraspgluser
354045
serarglysserthrtyrgluvaltyraspglnglugluargargser
505560
argileserseraspproproleuglnleuasnleuleuproserala
65707580
provalproargserhisvalserserglnleuargpheprotrpleu
859095
thraspasnleualavalsergluproalaglyserseraspglypro
100105110
glyileglyleuaspvalasnargmetproglyvalleualaserthr
115120125
gluglualagluaspalaalaalaproleuserserproasnserthr
130135140
valserserpheglnmetasppheglymetargasnglyglyargser
145150155160
serlysargaspleuaspvalaspalaaspargalaseraspaspglu
165170175
gluasnglyserthrarglyslysleuargleuserlysaspglnser
180185190
alapheleuglugluserphelysgluhisserthrleuasnprolys
195200205
glnlysleualaleualalysglnleuasnleuargproargglnval
210215220
gluvaltrppheglnasnargargalaargthrlysleulysglnthr
225230235240
gluvalaspcysglutyrleulysargcyscysgluthrleuthrglu
245250255
gluasnargargleuglnlysgluleuglnaspleuargalaleulys
260265270
thrserglnprophetyrmetglnleuproalathrthrleuthrmet
275280285
cysprosercysgluargvalvalthrthralaseralaasnthrser
290295300
thrthrthrthrthrasnhishishishisilelysseralaleuasn
305310315320
asnasnlysproargleuasnprophethralaalaglnvalhisile
325330335
glnglnhisglnglyalaalaala
340
<210>17
<211>1107
<212>dna
<213>翠冠梨(pyrusspp)
<400>17
atggagctttgtttgagcttaggagatcatgaggctcttgcagagccatctgatcagacc60
accaccaccaaaacgacagggttttgcatgaacttatcaatcggtccaagctgtgcagct120
gctccttctcctcctcctcctcctagagatgaagaagcagataaaaaacaagatgatgat180
gatcatcatcatcatatccatgtagatgatcatacaccacttcaacttgatcttctccct240
tggcctacttcttcttgtaatcagattcaaaacgctggtgtgctgtccatgaacgtgatc300
aacaaagctcaggaagtggtggctgctgcgtcgtcatcgcatacaaacagtggttcgtcg360
tcgtttcatatcatggatatcggcttatgcagcaataataatcacagtaagggcagtgct420
ggtgatggtctgaataacaattaccacaacgtctacaactacaagaggagggctcactct480
gcagaaggacaaggcgatgatgagggagaaagggcatataattccagggtgggcagtgat540
gatgatgaggatgacatcatcaatggtaacgctaacacctgtagaaagaagctcaggctc600
tccaaggaacaatctgcttttcttgaagaaagcttcaaagaacaccatactctcaaccct660
aagcaaaagctagcactggcgaaacagcttaatcttcgtccaagacaagtagaagtttgg720
tttcagaatagaagagcaaggacaaagttgaagcaaacggaggtggactgtgagttcctg780
aagagatgttgtgagacactgacggaagagaacagaaggctaaacaaagagctacaggag840
ttgagggctttgaaggctactcatcactcaaacccgttctacatgcaatcaccagccacc900
accctcaccctgtgtccttcttgcgagcgcacagctaccacctctaacgccgccatcaac960
accaccaacaatacttataacaataataataataaagttatgtcaattagtactacaaaa1020
gttcaagcagctaagtttcctctcaacagacccagcttctatccgttttttcaacccaaa1080
tcccatccctcttccgcagcttcgtga1107
<210>18
<211>368
<212>prt
<213>翠冠梨(pyrusspp)
<400>18
metgluleucysleuserleuglyasphisglualaleualaglupro
151015
seraspglnthrthrthrthrlysthrthrglyphecysmetasnleu
202530
serileglyprosercysalaalaalaproserpropropropropro
354045
argaspgluglualaasplyslysglnaspaspaspasphishishis
505560
hisilehisvalaspasphisthrproleuglnleuaspleuleupro
65707580
trpprothrsersercysasnglnileglnasnalaglyvalleuser
859095
metasnvalileasnlysalaglngluvalvalalaalaalaserser
100105110
serhisthrasnserglyserserserphehisilemetaspilegly
115120125
leucysserasnasnasnhisserlysglyseralaglyaspglyleu
130135140
asnasnasntyrhisasnvaltyrasntyrlysargargalahisser
145150155160
alagluglyglnglyaspaspgluglygluargalatyrasnserarg
165170175
valglyseraspaspaspgluaspaspileileasnglyasnalaasn
180185190
thrcysarglyslysleuargleuserlysgluglnseralapheleu
195200205
glugluserphelysgluhishisthrleuasnprolysglnlysleu
210215220
alaleualalysglnleuasnleuargproargglnvalgluvaltrp
225230235240
pheglnasnargargalaargthrlysleulysglnthrgluvalasp
245250255
cysglupheleulysargcyscysgluthrleuthrglugluasnarg
260265270
argleuasnlysgluleuglngluleuargalaleulysalathrhis
275280285
hisserasnprophetyrmetglnserproalathrthrleuthrleu
290295300
cysprosercysgluargthralathrthrserasnalaalaileasn
305310315320
thrthrasnasnthrtyrasnasnasnasnasnlysvalmetserile
325330335
serthrthrlysvalglnalaalalyspheproleuasnargproser
340345350
phetyrprophepheglnprolysserhisproserseralaalaser
355360365
<210>19
<211>47
<212>dna
<213>人工序列(artificialsequence)
<400>19
atggccatggaggccgaattcgagatgggttttgatcatgaagcttg47
<210>20
<211>47
<212>dna
<213>人工序列(artificialsequence)
<400>20
ccgctgcaggtcgacggatccgcagactagcaagctgtagatggatg47
<210>21
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>21
gccatggaggccagtgaattcatggagctagctctgagcttagg44
<210>22
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>22
cagctcgagctcgatggatcctcatgcagcagcaccttgatgctg45
<210>23
<211>46
<212>dna
<213>人工序列(artificialsequence)
<400>23
gccatggaggccagtgaattcatggagctttgtttgagcttaggag46
<210>24
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>24
cagctcgagctcgatggatcctcacgaagctgcggaagagggat44
<210>25
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>25
ctcggtacccggggatccgagatgggttttgatcatgaagcttg44
<210>26
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>26
gtacgagatctggtcgacgcaagctgtagatggatg36
<210>27
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>27
gcgtcccggggcggtaccatggagctagctctgagcttagg41
<210>28
<211>39
<212>dna
<213>人工序列(artificialsequence)
<400>28
agtccatttgttggatcctgcagcagcaccttgatgctg39
<210>29
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>29
gcgtcccggggcggtaccatggagctttgtttgagcttaggag43
<210>30
<211>38
<212>dna
<213>人工序列(artificialsequence)
<400>30
agtccatttgttggatcccgaagctgcggaagagggat38
<210>31
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>31
ctatagggcgaattgggtacctcaaacaaggaaattggtgtatg44
<210>32
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>32
caggaattcgatatcaagctttttcttggttccaaagaacacac44
<210>33
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>33
tgttccggattacgctgttaacatggagctagctctgagcttagg45
<210>34
<211>46
<212>dna
<213>人工序列(artificialsequence)
<400>34
ctcattaaagcaggactctagatcatgcagcagcaccttgatgctg46
<210>35
<211>47
<212>dna
<213>人工序列(artificialsequence)
<400>35
tgttccggattacgctgttaacatggagctttgtttgagcttaggag47
<210>36
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>36
ctcattaaagcaggactctagatcacgaagctgcggaagagggat45
<210>37
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>37
cttgaattcgagctcggtacctcaaacaaggaaattggtgtatg44
<210>38
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>38
atacagagcacatgcctcgagtttcttggttccaaagaacacac44
<210>39
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>39
gccatggaggccagtgaattcatgggttttgatcatgaagcttg44
<210>40
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>40
cagctcgagctcgatggatccctagcaagctgtagatggatgagtact48
<210>41
<211>34
<212>dna
<213>人工序列(artificialsequence)
<400>41
cttatgggtcggtttttctacatttgaattttga34
<210>42
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>42
cctcaaggaaatagtaataccctttagatttgaagtccta40
<210>43
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>43
ttatatgttgtatcttatttcatcgttgattac33
<210>44
<211>54
<212>dna
<213>人工序列(artificialsequence)
<400>44
gagctcggtacccggggatccatgagaaagaaactcagacttacaaaagaacaa54
<210>45
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>45
cgcgtacgagatctggtcgacgcaagctgtagatggatgagtact45
<210>46
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>46
atggagctcggtaccctcgagatggagctagctctgagcttagg44
<210>47
<211>42
<212>dna
<213>人工序列(artificialsequence)
<400>47
agcagagattacctatctagatgcagcagcaccttgatgctg42
<210>48
<211>46
<212>dna
<213>人工序列(artificialsequence)
<400>48
atggagctcggtaccctcgagatggagctttgtttgagcttaggag46
<210>49
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>49
agcagagattacctatctagacgaagctgcggaagagggat41
- 还没有人留言评论。精彩留言会获得点赞!