鉴定改变iRhom多肽活性的化合物的方法及其用途与流程
本申请要求于2014年5月9日提交的美国临时专利申请No.61/991,423和于2014年5月12日提交的美国临时专利申请No.61/992,152的优先权,所述临时专利申请的全部内容以引用的方式纳入本文。
基金信息
本发明是在由国立卫生研究院所授予的基金No.CA034196、HLO77642和DK057199的政府支持下完成的。政府对本发明享有某些权利。
技术领域
本文总体上记载了用于鉴定改变iRhom多肽活性的化合物的方法及其用途。根据具体的方面,本文公开了用于鉴定调节iRhom多肽的蛋白水解活性(例如,增大、刺激、降低、抑制或消除iRhom多肽的蛋白水解活性)的化合物的方法,以及其用途。
背景技术:
菱形蛋白酶(rhomboid protease)是几乎存在于所有物种中的酶家族。菱形蛋白酶是膜内丝氨酸蛋白酶,菱形蛋白酶的蛋白水解裂解作用对细胞调控是重要的。膜内蛋白酶的活性位点埋在细胞膜的脂双层中,并且它们在其他跨膜蛋白的跨膜结构域内裂解这些跨膜蛋白。
无活性的菱形蛋白酶(iRhom)是高度保守的膜内蛋白。在本发明的发现之前,人们认为iRhom由于在推定的活性位点缺少丝氨酸残基而无蛋白水解活性。
技术实现要素:
一方面,本发明基于以下发现:iRhom是短寿命蛋白,但是显性突变增加它们的蛋白稳定性并刺激不依赖于金属蛋白酶活性的特定的EGF家族配体双调蛋白的分泌。另一方面,本发明基于以下发现:哺乳动物iRhom在调节EGFR信号传导事件中发挥作用,所述EGFR信号传导事件促进伤口加速愈合并引发肿瘤发生。另一方面,本发明至少部分基于以下发现:iRhom能够与金属蛋白酶并行调节EGFR信号传导,并且在受损的伤口愈合和癌症中可用作治疗靶标。
一方面,本发明提供了鉴定化合物的方法,所述化合物激活iRhom多肽对底物的蛋白水解活性,所述方法包括:a)使表达iRhom多肽的细胞与测试化合物接触;以及b)测定iRhom多肽稳定性相对于合适对照的增加,其中iRhom多肽稳定性的增加表明所述化合物激活了iRhom多肽对底物的蛋白水解活性。
另一方面,本发明提供了鉴定化合物的方法,所述化合物能够在受试者中加速伤口愈合或组织修复,所述方法包括:使表达iRhom多肽和EGFR配体的细胞与测试化合物接触;b)测定iRhom多肽稳定性相对于合适对照的增加;以及c)测定细胞分泌的EGFR配体相对于合适对照的增加,其中iRhom多肽稳定性的增加和细胞分泌的EGFR配体的增加表明所述化合物能够加速伤口愈合。
另一方面,本发明提供了鉴定化合物的方法,所述化合物抑制iRhom多肽的胞质结构域,所述方法包括:a)使表达iRhom多肽的细胞与测试化合物接触;b)测定iRhom多肽稳定性相对于合适对照的增加,其中iRhom多肽稳定性的增加表明所述化合物抑制iRhom多肽的胞质结构域。
在一个实施方案中,所述方法还提供了通过分析暴露于所述化合物后iRhom多肽的半衰期来测定iRhom多肽稳定性的增加。
在一些实施方案中,所述底物是EGFR配体或EGF样底物。
在另一个实施方案中,通过检测EGFR配体分泌的增加来测定iRhom多肽稳定性的增加。在另一个实施方案中,通过检测可溶性EGFR配体水平的增加来测定iRhom多肽稳定性的增加。在一些实施方案中,所述EGFR配体选自AREG、HB-EGF、TGFα和EPGN。
在另一个实施方案中,通过检测EGFR信号传导活性的增加来测定iRhom多肽稳定性的增加。
在另一个实施方案中,本发明的化合物抑制iRhom多肽和蛋白酶体之间的相互作用。
在另一个实施方案中,所述化合物使iRhom多肽的胞质结构域失活。在一些实施方案中,所述胞质结构域的失活是短暂的。
在一个实施方案中,本发明的化合物使iRhom多肽的胞质结构域裂解和/或缺失,以使所述多肽具有蛋白水解活性或改变的生物活性。在一些实施方案中,小鼠iRhom2在第1至268位氨基酸残基之间被裂解,或人iRhom2在第1至298位氨基酸残基之间被裂解。在其他实施方案中,小鼠iRhom1在第1至272位氨基酸残基之间被裂解,或人iRhom1在第1至316位氨基酸残基之间被裂解。在另一个实施方案中,所述化合物激活iRhom多肽的肽酶域。
在一个实施方案中,所述iRhom多肽是iRhom1或iRhom2。在另一个实施方案中,所述iRhom多肽是人或小鼠iRhom多肽。
在一个实施方案中,本发明的化合物选自小分子、肽或多肽诱饵(decoy)。在另一个实施方案中,所述化合物连接至细胞穿透肽。在另一个实施方案中,所述化合物是细胞可透过性的。在另一个实施方案中,所述化合物是泛素蛋白酶抑制剂。
在一个实施方案中,本发明的化合物加速角化细胞的迁移。在另一个实施方案中,所述化合物加速成纤维细胞的增殖。
一方面,本发明提供了鉴定化合物的方法,所述化合物抑制iRhom多肽对底物的蛋白水解活性,所述方法包括:a)使表达iRhom多肽的细胞与测试化合物接触;以及b)测定iRhom多肽的生理靶标的分泌相对于合适对照的减少,或EGFR活性相对于合适对照的降低,其中iRhom多肽的生理靶标分泌的减少或EGFR活性的降低表明所述化合物抑制iRhom多肽对底物的蛋白水解活性。
另一方面,本发明提供了鉴定化合物的方法,所述化合物能够在受试者中降低肿瘤生长和/或进展或治疗癌症,所述方法包括:a)使表达iRhom多肽的细胞与测试化合物接触;以及b)测定iRhom多肽的生理靶标的分泌相对于合适对照的减少,或EGFR活性相对于合适对照的降低,其中iRhom多肽的生理靶标分泌的减少或EGFR活性的降低表明所述化合物能够在受试者中降低肿瘤生长和/或进展或治疗癌症。
另一方面,本发明提供了鉴定化合物的方法,所述化合物能够在受试者中促进毛发生长,所述方法包括:a)使表达iRhom多肽的细胞与测试化合物接触;以及b)测定iRhom多肽的生理靶标的分泌相对于合适对照的减少,或EGFR活性相对于合适对照的降低,其中iRhom多肽的生理靶标分泌的减少或EGFR活性的降低表明所述化合物能够在受试者中促进毛发生长。
在一个实施方案中,所述肿瘤是实体瘤。在一些实施方案中,所述癌症是上皮癌。在其他实施方案中,所述癌症是食管癌、肺癌、脑癌、结肠癌、肾癌、前列腺癌、皮肤癌、肝癌、胰腺癌、胃癌、子宫癌、卵巢癌、乳腺癌、淋巴腺癌或膀胱癌。
在一个实施方案中,所述底物是EGFR配体或EGF样底物。
在另一个实施方案中,通过检测iRhom多肽的生理靶标分泌的减少来测定对iRhom多肽的蛋白水解活性的抑制。在一些实施方案中,所述iRhom多肽的生理靶标是EGFR配体。在一个实施方案中,通过检测可溶性EGFR配体水平的下降来测定对iRhom多肽的蛋白水解活性的抑制。在其他实施方案中,所述EGFR配体选自AREG、HB-EGF、TGFα和EPGN。
在一个实施方案中,通过检测EGFR活性的降低来测定对iRhom多肽的蛋白水解活性的抑制。
在另一个实施方案中,所述化合物抑制iRhom多肽的肽酶域。
在另一个实施方案中,所述化合物通过使iRhom多肽肽酶域的蛋白水解位点中的一个或多个氨基酸残基失活来影响iRhom多肽的活性。在一个实施方案中,所述iRhom多肽是小鼠iRhom2,并且所述化合物使小鼠iRhom2的蛋白水解位点中的一个或多个氨基酸残基失活,所述氨基酸残基选自TM螺旋2上的第635位组氨酸、TM螺旋4上的第695位谷氨酰胺、TM螺旋4上的第701位半胱氨酸和TM螺旋6上的第744位组氨酸。在其他实施方案中,所述iRhom多肽是人iRhom2,并且所述化合物使人iRhom2的蛋白水解位点中的一个或多个氨基酸残基失活,所述氨基酸残基选自TM螺旋2上的第664位组氨酸、TM螺旋4上的第724位谷氨酰胺、TM螺旋4上的第730位半胱氨酸和TM螺旋6上的第773位组氨酸。
在一个实施方案中,iRhom家族成员是iRhom1或iRhom2。在其他实施方案中,iRhom家族成员是人或小鼠iRhom家族成员。
在一个实施方案中,本发明的化合物选自小分子、多肽诱饵、miRNA分子、siRNA分子、shRNA分子、dsRNA分子、反义分子、特异性针对Rhbdf2的核酶;或编码miRNA、siRNA、shRNA、dsRNA的多聚核苷酸;或其各自的生物等价物。在其他实施方案中,所述化合物连接至细胞穿透肽。在一些实施方案中,所述化合物是细胞可透过性的。
一方面,本发明提供了分离的多肽,其包含变体iRhom多肽。
另一方面,本发明提供了分离的多肽,其包含缺失细胞溶质N-末端结构域的人iRhom2。
另一方面,本发明提供了分离的多肽,其包含缺失细胞溶质N-末端结构域的小鼠iRhom2。
另一方面,本发明提供了分离的小鼠iRhom2多肽,其在小鼠iRhom2的蛋白水解位点中的一个或多个氨基酸残基处包含突变,所述氨基酸残基选自TM螺旋2上的第635位组氨酸、TM螺旋4上的第695位谷氨酰胺、TM螺旋4上的第701位半胱氨酸和TM螺旋6上的第744位组氨酸。
另一方面,本发明提供了分离的人iRhom2多肽,其在人iRhom2的蛋白水解位点中的一个或多个氨基酸残基处包含突变,所述氨基酸残基选自TM螺旋2上的第664位组氨酸、TM螺旋4上的第724位谷氨酰胺、TM螺旋4上的第730位半胱氨酸和TM螺旋6上的第773位组氨酸。
在一个实施方案中,本发明提供了编码上述多肽中任一种的分离的核酸分子。在另一个实施方案中,本发明提供了包含上述核酸分子的载体。在另一个实施方案中,本发明提供了表达上述载体的宿主细胞。
附图说明
图1A是示出了使用本文所述的增殖测定对MEF进行定量的图;以X轴所示的数目接种细胞并孵育细胞24h,荧光程度与总细胞DNA的量成比例;数据以平均值±s.d.表示;
图1B是示出了使用本文所述的划痕(scratch-wound)试验对cub/cub mcub/mcub和+/+mcub/mcub MEF的迁移进行定量的图;在时间点0时测量划痕伤口的宽度(100%开口),以初始宽度的百分数计算在各时间点伤口闭合的增加;数据以平均值±s.d.表示;
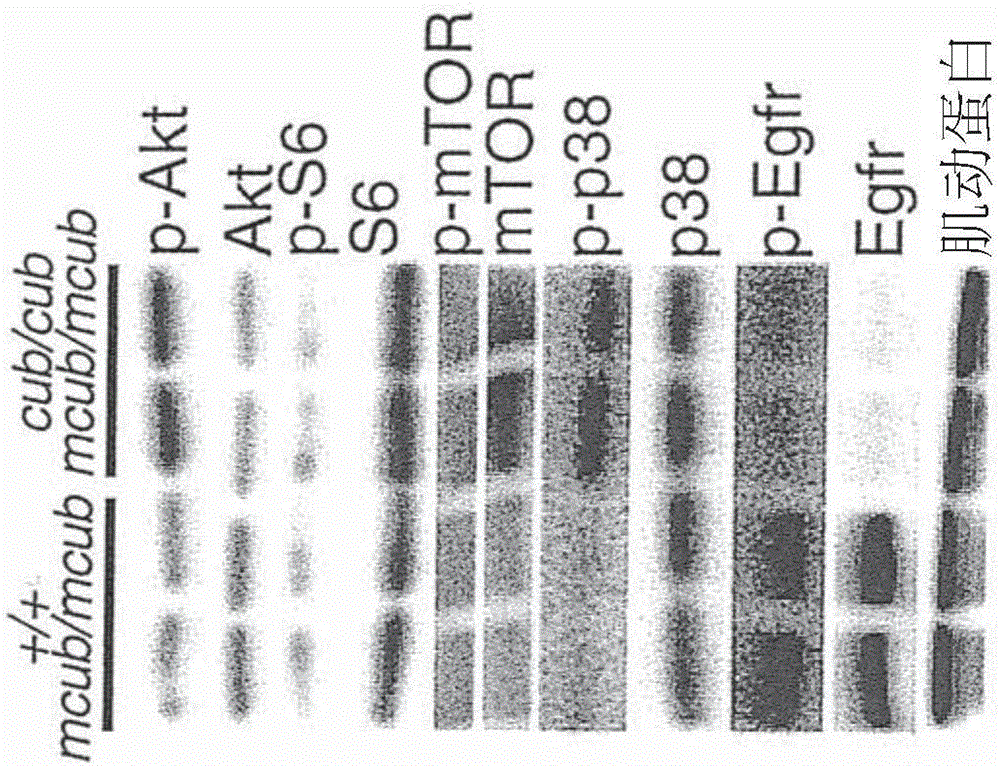
图1C是示出了对cub/cub mcub/mcub或+/+mcub/mcub MEF的EGFR信号传导的多个标记物的免疫印迹分析的图像,其中一式两份地测定(run)细胞裂解物,使用肌动蛋白充当内参照(loading control);
图1D示出了在受伤后第0、7、14、21和28天,6-40周龄雌性cub/cub mcub/mcub和+/+mcub/mcub小鼠(每组n=3)中再生耳组织的代表性图像。
图1E是示出了对图1D中所示的耳孔闭合的定量的图;数据以平均值±s.d.表示;
图1F示出了在受伤后第0、7和14天,cub/cub mcub/mcub和+/+mcub/mcub小鼠的耳横截面的图像,用苏木精/曙红进行染色(原始放大率,×10);注意cub/cub mcub/mcub小鼠的耳中未分化且增厚的表皮(E)(10-12个有核层)以及广泛程度的增殖(M);虚线表示切开的部位;
图2A是示出了cub突变为小鼠Rhbdf2基因中12681bp缺失的图,其中所述缺失开始于外显子1和2的中间并且包含外显子2-6,在外显子6之后随即结束;
图2B示出了使用外显子2、5、12和19的引物对+/+mcub/mcub、+/cub mcub/mcub和cub/cub mcub/mcub小鼠的MEF进行逆转录酶PCR的结果图;ntc代表无模板对照;
图2C示出了使用针对所示外显子边界的TaqMan基因表达测定,对从皮肤组织提取的cDNA进行qPCR的结果图,使用肌动蛋白充当内参照(endogenous control);将数据标准化为+/+mcub/mcub肌动蛋白水平,使用4种生物复制品,一式三份地测定样品;数据以平均值±s.d.表示;
图2D示出了使用常规TaqMan基因表达测定检测具有外显子1-外显子7边界的转录物的qPCR结果图;使用3种生物复制品,一式三份地测定样品(各柱代表单独的小鼠);数据以平均值±s.d.表示;
图2E是瞬时表达Flag标记的HuWt(人全长RHBDF2cDNA)和HuCub(人类版本的cub cDNA)的HEK293细胞的抗Flag免疫印迹的图像,使用肌动蛋白作为内参照;
图2F示出了使用Flag特异性抗体染色的表达Flag标记的HuWt和HuCub的B6原代MEF的代表性图像,DAPI用于对细胞核进行复染色;箭头代表全长蛋白和突变蛋白的细胞质表达;
图3A是示出了KOMP Rhbdf2敲除小鼠中野生型产物的PCR结果的图像,预期产物长度为2181bp;L代表从New England Biolabs获得的1kb DNA梯状标记物(ladder),ntc代表无模板对照;
图3B示出了对Rhbdf2表达的报告基因的分析的图像;全胚胎(whole-mount)X-gal染色的E18.5Rhbdf2-/-胚胎示出了表皮中β-gal的强表达(箭簇)(原始放大率,×2.5),X-gal染色的两周龄雌性Rhbdf2-/-皮肤示出了毛囊内鞘层和外鞘层中的β-gal阳性(箭头)(原始放大率,×5);毛干中未观察到染色;比例尺,1mm;
图3C是示出了具有Rhbdf2无效突变的小鼠缺少再生表型的图,如与Rhbdf2cub/cub小鼠相比,6至8周龄雌性Rhbdf2-/-小鼠的耳中显著延缓的伤口闭合所显示的;
图3D示出了证明了具有Rhbdf2无效突变的小鼠具有正常的皮肤和毛发形态的图像;在所示的创伤后时间点获取的来自Rhbdf2cub/cub和Rhbdf2-/-小鼠的成年皮肤的H&E染色切片,其中成年Rhbdf2cub/cub皮肤显示出薄的皮下脂肪层(F)、异常的毛囊(H)、厚的表皮(E)、增大的皮脂腺(SG)、致密的交织排列的胶原纤维束(C),以及毛囊和毛球的不完全分化(HF);成年Rhbdf2-/-皮肤显示出正常表皮和毛囊;比例尺,100μm;原始放大率,×10和×40;
图3E是示出了Rhbdf2-/-小鼠发育出正常的毛皮(箭簇),而复合突变的Rhbdf2-/cub小鼠表现出稀疏的毛皮(箭头)的图像;
图4A是示出了Mcub是T至G点突变的图,所述点突变破坏了Areg基因中的正常供体剪接位点(外显子1),导致使用可变的下游剪接位点;
图4B是示出了在28天中6-8周龄雌性Rhbdf2+/+Areg+/+、Rhbdf2cub/cub Areg+/+和Rhbdf2cub/cub AregMcub/Mcub小鼠中耳孔的愈合(每组n=3只小鼠)的图;数据以平均值±s.d.表示;
图4C示出了苏木精/曙红染色的切片图,其示出了Rhbdf2cub/cub AregMcub/Mcub小鼠耳孔切开后的愈合,其中虚线表示切开部位;原始放大率,×10;E代表表皮,M代表增殖;与图1F进行比较;
图4D是示出了同龄的Rhbdf2+/+Areg+/+、Rhbdf2cub/cub Areg+/+和Rhbdf2cub/cubAregMcub/Mcub雌性小鼠中血清AREG水平的图,其中在Rhbdf2cub/cub AregMcub/Mcub小鼠的血清中未检测到AREG(n.d.);数据以三次独立实验的平均值±s.d.表示;***p<0.001;
图 4E是对从Rhbdf2+/+Areg+/+、Rhbdf2cub/cub Areg+/+和Rhbdf2cub/cub AregMcub/Mcub小鼠中分离的培养的小鼠表皮角化细胞(MEK)的上清液中AREG水平的ELISA定量的图,其中在Rhbdf2cub/cub AregMcub/Mcub小鼠的MEK中未检测到AREG(n.d.);数据以三次独立实验的平均值±s.d.表示;***p<0.001;
图4F是示出了从EGFR配体的qPCR获得的结果:使用TaqMan基因表达测定,从Rhbdf2cub/cub Areg+/+和Rhbdf2+/+Areg+/+的皮肤组织提取cDNA,使用肌动蛋白充当内参照;将数据标准化为Rhbdf2+/+Areg+/+肌动蛋白水平,数据以三次独立实验的平均值±s.d.表示;*p<0.05;
图5A是示出了全长人RHBDF2(Hu Wt)基因和模拟小鼠Rhbdf2cub基因的人构建体(HuCub)的示意图;
图5B是示出了在293T细胞中共表达Hu Wt或HuCub与AREG基因后,对经裂解的AREG的ELISA定量的图;转染后24h,用DMSO或10μM马立马司他(MM)孵育细胞24h,并在条件培养基中分析AREG水平,重复进行转染并将条件培养基稀释5倍。
图5C是示出了对所示基因型的8-12周龄雌性小鼠进行LPS注射后3h,通过ELISA对血清TNFα水平进行定量的图,其中在没有进行LPS注射的Rhbdf2+/+小鼠的血清中未检测到TNFα(n.d.);*p<0.05;
图5D是示出了共表达人RHBDL2(HuRHBDL2)基因与AREG或HB-EGF或EGF基因后,对经裂解的AREG/HB-EGF/EGF的ELISA定量的图;转染后24h,用10μM马立马司他(MM)孵育细胞24h,并在条件培养基中分析AREG/HB-EGF/EGF水平,其中不稀释HB-EGF和EGF条件培养基,而将AREG培养基稀释5倍;数据以三次独立实验的平均值±s.d.表示;***p<0.001;
图5E是示出了在293T细胞中共表达Hu Wt或HuCub与HB-EGF基因后,对经裂解的HB-EGF的ELISA定量的图;转染后24h,用10μM马立马司他(MM)孵育细胞24h,并在条件培养基中分析HB-EGF水平;***p<0.001;
图5F是示出了在293T细胞中共表达Hu Wt或HuCub与EGF基因后,对经裂解的EGF的ELISA定量的图;转染后24h,用DMSO或10μM马立马司他(MM)孵育细胞24h,并在条件培养基中分析EGF水平;**p<0.01;
图5G是示出了在293T细胞中共表达HuCub或无肽酶域的HuCub(HuCubΔPD)与AREG基因后,对经裂解的AREG的ELISA定量的图;转染后24h,用10μM马立马司他(MM)孵育细胞24h,并在条件培养基中分析AREG水平;***p<0.001;
图5H是示出了在293T细胞中共表达HuCub或具有单个丙氨酸突变的HuCub与AREG基因后,对经裂解的AREG的ELISA定量的图;转染后24h,用10μM马立马司他(MM)孵育细胞24h,并在条件培养基中分析AREG水平;***p<0.001;
图5I是示出了在293T细胞中共表达HuCub或具有单个丙氨酸突变的HuCub与EGF基因后,对经裂解的EGF的ELISA定量的图;转染后24h,用10μM马立马司他(MM)孵育细胞24h,并在条件培养基中分析EGF水平;**p<0.001;
图5J是示出了Rhbdf2cub的膜拓扑结构的图,其中所示氨基酸对于Rhbdf2cub调控EGFR配体产生是关键的;
图6A是示出了在10μM马立马司他(MM)的存在下,通过ELISA对来自用AREG与人RHBDF2p.I186T突变体、HuWt或HuCub转染的HEK293细胞的条件培养基的经裂解的AREG进行定量的图;**p<0.01;
图6B是示出了HEK293细胞的蛋白质印迹的图像,所述HEK293细胞用AREG与HuCub、HuWt或p.I186T共转染,用DMSO或MM孵育,并且对HA-AREG进行免疫印迹,表明即使在10μM MM的存在下,与AREG/Hu Wt或单独的AREG的表达相比,AREG与HuCub或p.I186T的共表达显著降低了AREG前体的胞内水平,;
图6C是示出了表达各RHBDF2P.I186T点突变体并用AREG基因或空载体共转染的细胞中的AREG水平的图;各残基被突变为丙氨酸;用10μM MM孵育细胞24h并在条件培养基中测定AREG水平;
图6D是X-gal染色的2周龄雌性Rhbdf2-/-的肠的图像,其显示了报告基因β-gal在中部和上部绒毛区的表达;原始放大率,×2.5;
图6E是示出了ApcMin/+Rhbdf2+/+小鼠(n=28)和ApcMin/+Rhbdf2+/cub(n=23)小鼠的Kaplan-Meier存活曲线的图,其中ApcMin/+Rhbdf2+/+小鼠的中值存活期为172天,与之相比,ApcMin/+Rhbdf2+/cub小鼠的中值存活期为135天;
图6F示出了三月龄时所示基因型小鼠的肠组织的苏木精-曙红染色的切片的图像;原始放大率,×2.5;
图6G是示出了三月龄时所示基因型小鼠的息肉大小/每只小鼠的图;对于ApcMin/+Rhbdf2+/+小鼠和ApcMin/+Rhbdf2+/cub小鼠,平均肿瘤大小分别为1.8mm2和3.7mm2;
图6H是示出了三月龄时所示基因型小鼠的息肉数目/每只小鼠的图;对于ApcsMin/+Rhbdf2+/+小鼠和ApcMin/+Rhbdf2+/cub小鼠,平均息肉数目分别为12和21;
图7A是人iRhom2-AREG复合体的免疫共沉淀图,其中用Flag标记的HuWt或Flag标记的HuCub或Flag标记的p.I186T共转染来自COS7细胞的裂解物,对HA标记的AREG用抗Flag磁珠进行免疫沉淀并用抗Flag抗体和抗HA抗体进行探测;
图7B示出了用所示的Flag标记的基因转染并使用Flag特异性PE标记的抗体进行免疫标记的293T或COS7(图14)细胞的流式细胞术结果,其中在转染后48h对所述细胞进行免疫标记;
图7C是示出了对图7B中获得的数据的定量的图;
图7D示出了在所示时间用150μg/ml蛋白合成抑制剂环己酰胺对瞬时转染的COS7细胞进行追踪,并使用Flag特异性PE标记的抗体来评估蛋白表达的测试结果;数据代表总共三次实验中的一次;
图7E是示出了对图7D中获得的数据的定量的图;
图7F是示出了用10μM细胞可透过性的蛋白酶抑制剂MG-132预孵育瞬时转染的COS7细胞4h,然后在所示时间在MG-132存在下用150μg/ml环己酰胺追踪的测试结果的图;如图7D所述测定蛋白表达;***p<0.001;
图8示出了野生型iRhom2是短寿命蛋白,其N末端缺失或N-末端结构域中的突变(包括那些引起上皮癌的突变)增加了其蛋白稳定性,进而增加了选择性EGF家族配体(包括AREG)的分泌;抑制ADAM17对AREG分泌没有影响,而iRhom2的肽酶域中氨基酸H366、Q426、C432和H475的缺失终止了AREG分泌;相反,AREG分泌增加导致EGFR信号传导的过度激活,从而增加了细胞增殖和迁移;
图9A是示出了cub/cub mcub/mcub(箭簇)和cub/cub Mcub/mcub(箭头)小鼠的图像;
图9B是示出了+/cub mcub/mcub小鼠的伤口愈合似乎介于cub/cub mcub/mcub小鼠和+/+小鼠之间的图;在创伤(2mm通孔)后0、7、14和21天,在6至40周龄雌性+/+mcub/mcub、+/cub mcub/mcub和cub/cub mcub/mcub小鼠(每组n=3)中对耳孔闭合率进行定量;数据以平均值±s.d.表示;
图10A示出了B6原代MEF中Flag标记的全长(HuWt)和cub(HuCub)Rhbdf2克隆的体外表达,然后用Flag特异性抗体和细胞核的DAPI复染色进行蛋白表达的免疫化学评估,表明全长和突变蛋白都与ER标记物钙联蛋白共定位;
图10B示出了B6原代MEF中Flag标记的全长(HuWt)和cub(HuCub)Rhbdf2克隆的体外表达,然后用Flag特异性抗体和细胞核的DAPI复染色进行蛋白表达的免疫化学评估,表明全长和突变蛋白都不与高尔基体标记物巨蛋白(giantin)共定位;
图11是示出了T至G点突变破坏了原始剪接位点(外显子1)而导致可变剪接位点的图,所述可变剪接位点在Areg转录物上添加了22个额外的核苷酸并在蛋白质上添加了16个错误的氨基酸,引入了提前终止密码子;
图12A是对cub/cub Mcub/Mcub或cub/cub mcub/mcub MEF的所示标记物的免疫印迹分析图,其中一式两份地测定细胞裂解物,使用肌动蛋白充当内参照;
图12B是示出了对在scramble shRNA或小鼠Areg shRNA慢病毒感染的Rhbdf2+/+或Rhbdf2cub/cub MEF中Areg表达的定量RT-PCR测定的图;
图12C是示出了用scramble shRNA或小鼠Areg shRNA感染之后,使用本文所述的增殖测定对Rhbdf2+/+MEF进行定量的图,其中以X轴所示的数目接种细胞并孵育细胞24h,荧光程度与总细胞DNA的量成比例;
图12D是示出了用scramble shRNA或小鼠Areg shRNA感染之后,使用本文所述的增殖测定对Rhbdf2cub/cub MEF进行定量的图,其中以X轴所示的数目接种细胞并孵育细胞24h,荧光程度与总细胞DNA的量成比例;
图13A是示出了对来自在10μM马立马司他(MM)存在下用小鼠Rhbdf1或N-末端截短的小鼠Rhbdf1(ΔN-iRhom1)或ΔN-iRhom1突变体以及AREG基因转染的HEK 293T细胞的条件培养基的分泌的AREG进行ELISA定量的图;***p<0.001;
图13B是示出了对来自在10μM马立马司他(MM)存在下用小鼠Rhbdf1或N-末端截短的小鼠Rhbdf1(ΔN-iRhom1)以及EGF基因转染的HEK 293T细胞的条件培养基的经裂解的EGF进行ELISA定量的图;***p<0.001;
图13C是对人iRhom2和RHBDL2,以及人和小鼠iRhom的肽酶域序列的氨基酸进行比对的图像;
图13D是示出了在293T细胞中共表达HuCub或具有单个丙氨酸突变的HuCub与AREG基因后,对分泌的AREG进行ELISA定量的图;转染后24h时,用10μM马立马司他(MM)孵育细胞24h并在条件培养基中分析AREG水平;数据代表三次独立实验的平均值±s.d.;
图13E是示出了对iRHOM1和iRHOM2、活性菱形RHBDL2、秀丽线虫(C.elegans)无活性菱形ROM3和ROM4,以及果蝇属(Drosophila)iRhom的肽酶域的氨基酸的序列比对的图,其中星号代表对iRHOM2调控EGFR配体而言至关重要的保守氨基酸;以及
图14示出了用所示的Flag标记的基因转染并使用Flag特异性PE标记的抗体进行免疫标记的COS7细胞的流式细胞术结果的图像,其中在转染后48h对所述细胞进行免疫标记。
具体实施方式
本文使用的科技术语旨在具有本领域普通技术人员通常所理解的含义。发现这些术语在多篇标准参考文献的上下文中被定义和使用,所述参考文献示例性地包括J.Sambrook和D.W.Russell,Molecular Cloning:A Laboratory Manual,Cold Spring Harbor Laboratory Press;第3版,2001;F.M.Ausubel Ed.,Short Protocols in Molecular Biology,Current Protocols;第5版,2002;B.Alberts等人,Molecular Biology of the Cell,第4版,Garland,2002;D.L.Nelson and M.M.Cox,Lehninger Principles of Biochemistry,第4版,W.H.Freeman&Company,2004;Engelke,D.R.,RNA Interference(RNAi):Nuts and Bolts of RNAi Technology,DNA Press LLC,Eagleville,PA,2003;Herdewij n,P.(Ed.),Oligonucleotide Synthesis:Methods and Applications,Methods in Molecular Biology,Humana Press,2004;A.Nagy,M.Gertsenstein,K.Vintersten,R.Behringer,Manipulating the Mouse Embryo:A Laboratory Manual,第3版,Cold Spring Harbor Laboratory Press;2002年12月15日,ISBN-10:0879695919;Kursad Turksen(Ed.),Embryonic stem cells:methods and protocols in Methods Mol Biol.2002;185,Humana Press;Current Protocols in Stem Cell Biology,ISBN:9780470151808;Chu,E.和Devita,V.T.,Eds.,Physicians’Cancer Chemotherapy Drug Manual,Jones&Bartlett Publishers,2005;J.M.Kirkwood等人,Eds.,Current Cancer Therapeutics,第4版,Current Medicine Group,2001;Remington:The Science and Practice of Pharmacy,Lippincott Williams&Wilkins,第21版,2005;L.V.Allen,Jr.等人,Ansel’s Pharmaceutical Dosage Forms and Drug Delivery Systems,第8版,Philadelphia,PA:Lippincott,Williams&Wilkins,2004;以及L.Brunton等人,Goodman&Gilman’s The Pharmacological Basis of Therapeutics,McGraw-Hill Professional,第12版,2011。
除非另外明确说明或上下文中另外清楚说明,单数术语“一”、“一个”和“所述”不意为限制性的,且包括复数指代物。
无活性菱形蛋白酶(iRhom)是高度保守的膜内蛋白,其之前被认为不具有蛋白水解活性。iRhom的特征在于长的细胞溶质的N-末端结构域,保守的富含半胱氨酸的无活性菱形蛋白酶同源结构域(IRHD),在肽酶域内缺少活性位点丝氨酸残基的休眠的(dormant)蛋白水解位点。
在许多物种中,iRhom参与多种功能,包括在黑腹果蝇(Drosophila melanogaster)中调节表皮生长因子受体(EGFR)信号传导、人鳞状上皮癌细胞的存活、在哺乳动物细胞系中从内质网膜清除错误折叠的蛋白、诱导原代小鼠角化细胞的迁移、在小鼠中分泌可溶性TNFα、以及在小鼠胚胎成纤维细胞中调节受激的ADAM17介导的金属蛋白酶脱落(shedding)的底物选择性。EGF样配体可作为iRhom家族成员的底物发挥作用。
Rhbdf2基因编码无活性的菱形蛋白酶iRhom2——包含长细胞溶质的N-末端和之前功能未知的休眠的肽酶域的酶家族之一。本发明提供了iRhom2可通过EGFR信号传导的组成型激活在上皮再生和癌生长中发挥作用。
本发明的方面至少部分基于以下发现:Rhbdf2突变通过增加双调蛋白的分泌来提高iRhom2蛋白稳定性并驱使EGFR过度激活。
本发明的方面至少部分基于以下发现:iRhom2是短寿命蛋白,其稳定性可通过在N-末端结构域的选择性突变来提高。进而,这些稳定的变体增加EGF家族配体(包括双调蛋白)的分泌,此过程不依赖于金属蛋白酶ADAM17活性。在体内,N-末端iRhom2突变诱导伤口加速愈合以及肿瘤生成加速,但是不驱使自发性肿瘤发展。因此,本发明还至少部分基于iRhom2在调控创伤愈合和肿瘤生长中涉及的EGFR信号传导事件的生理作用,以及iRhom2关键结构域的功能。
表皮生长因子受体(EGFR)信号转导在哺乳动物细胞的生长、增殖和分化中发挥重要作用。典型的EGFR配体(包括EGF、AREG和HB-EGF)作为在细胞表面表达的前体蛋白而存在,为了结合EGFR,所述EGFR配体必须被排放(shed)至胞外区室。在不同的发育阶段,不同类别的蛋白酶使与膜相连的EGFR前配体裂解以调节多种生物活性。
一方面,本发明至少部分基于以下发现:小鼠中Rhbdf2基因中的自发性缺失是卷曲裸露(cub)突变的原因,在所述突变中iRhom2的细胞溶质的N-末端结构域的缺失导致随后对毛囊发育、创伤愈合和肿瘤发生的影响。此外,cub的修饰因子(Mcub)治愈或矫正了变秃和/或促进毛发生长(实施例1)。另一方面,本发明至少部分基于以下发现:iRhom2是短寿命蛋白,但是N-末端的功能获得性突变(胼胝症(tylosis))或N-末端缺失(cub突变)提高了突变蛋白稳定性,导致不依赖于金属蛋白酶的EGFR配体双调蛋白(AREG)的分泌。Cub表型的遗传修饰因子(Mcub)用于证明AREG是iRhom2的生理靶标。另一方面,本发明至少部分基于对AREG分泌所必需的iRhom2肽酶域中的关键氨基酸的鉴定,表明该假酶的肽酶域尽管在推定的活性位点缺少丝氨酸残基,仍可能是功能性的。本发明因此提供了iRhom关键结构域的功能并建立了框架,用于理解iRhom、EGFR信号传导、以及创伤愈合和肿瘤发生中涉及的生物过程之间的关系。
iRhom家族成员
iRhom1和iRhom2
菱形蛋白酶是存在于几乎所有物种中的酶家族。菱形蛋白酶是膜内丝氨酸蛋白酶,菱形蛋白酶的蛋白质水解裂解作用对细胞调控而言非常重要。膜内蛋白酶的活性位点埋在细胞膜的脂双层中,并且它们在其他跨膜蛋白的跨膜结构域内裂解这些蛋白。
iRhom或“无活性的菱形蛋白酶”是菱形蛋白酶的一个亚家族,在本发明之前,其被认为缺乏蛋白水解活性。iRhom是高度保守的膜内蛋白,其特征在于,长的细胞溶质的N-末端结构域,保守的富含半胱氨酸的无活性菱形蛋白酶同源结构域(IRHD),在肽酶域内缺少活性位点丝氨酸残基的休眠的蛋白水解位点。适用于本发明的iRhom家族成员包括iRhom1(RHBDF1)和iRhom2(RHBDF2)。用于本发明的iRhom1和/或iRhom2可以是真核的、原核的或哺乳动物的。此外,用于本发明的iRhom1和/或iRhom2可来自人、灵长类动物、啮齿类动物(例如,小鼠、大鼠、豚鼠)、牛、马、驴、兔、山羊、绵羊、狗、鸡、猪、果蝇、秀丽线虫或大肠杆菌(E.coli)。
在本文中举例说明的野生型iRhom为小鼠iRhom2(SEQ ID No:1)、小鼠iRhom1(SEQ ID No:3)、人iRhom2(SEQ ID No:5)和人iRhom1(SEQ ID No:7)。在本文中举例说明的分离的iRhom突变序列为SEQ ID NO:2、SEQ ID NO:4、SEQ ID NO:6、SEQ ID NO:8。
本领域技术人员将易于认识到编码本文所示的iRhom多肽的核酸序列为SEQ ID NO:1-8。应理解,由于遗传密码的简并性,多于一个的核酸序列编码具体的iRhom多肽或变体,并且所述核酸序列可被表达以产生所需的iRhom。
通常,iRhom1和iRhom2变体与本申请中所示的野生型iRhom1和iRhom2的序列的序列同一性为至少50%,例如至少60%、至少70%、至少80%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%,至少95%、至少96%、至少97%、至少98%、或至少99%。
一方面,与相应的野生型iRhom1或iRhom2相比,本发明的iRhom变体中改变(例如,置换、插入或缺失)的数目为1-20,例如1-10和1-5,例如1、2、3、4、5、6、7、8、9或10个改变。
作用机制
在许多物种中,iRhom参与多种功能,包括在黑腹果蝇中调节表皮生长因子受体(EGFR)信号传导、人鳞状上皮癌细胞的存活、在哺乳动物细胞系中从内质网膜清除错误折叠的蛋白、诱导原代小鼠角化细胞的迁移、在小鼠中分泌可溶性TNFα、以及在小鼠胚胎成纤维细胞中调节受激的ADAM17介导的金属蛋白酶脱落的底物选择性。
在一个实施方案中,本发明证明了小鼠中Rhbdf2基因内的自发性缺失是卷曲裸露(cub)突变的原因,在所述突变中iRhom2的细胞溶质的N-末端结构域的缺失导致随后对毛囊发育、创伤愈合和肿瘤发生的影响。在另一个实施方案中,本发明证明了iRhom2是短寿命蛋白,但是N-末端的功能获得性突变或N-末端缺失提高了突变蛋白稳定性,导致不依赖于金属蛋白酶的EGFR配体双调蛋白(AREG)的分泌。使用cub表型的遗传修饰因子(Mcub),本发明证明了AREG是iRhom2的生理靶标。在另一个实施方案中,鉴定了AREG分泌所必需的iRhom2的肽酶域中的关键氨基酸(H、C、Q、H),表明该假酶的肽酶域尽管在推定的活性位点中缺少丝氨酸残基,但仍可能是功能性的。
iRhom家族成员的突变形式
本发明还涉及iRhom家族成员的突变形式。在一个实施方案中,iRhom1或iRhom2的细胞溶质的N-末端被缺失。所述细胞溶质的N-末端的缺失激活了iRhom多肽的肽酶域中的休眠的蛋白水解位点。在一个实施方案中,iRhom1或iRhom2的细胞溶质末端被裂解,以使跨膜结构域具有蛋白水解活性。在一个实施方案中,小鼠iRhom1在第1至272位残基之间被裂解,和/或人iRhom1在第1至316位残基之间被裂解,以使跨膜结构域具有蛋白水解活性。在另一个实施方案中,小鼠iRhom2在第1至268位残基之间被裂解,和/或人iRhom2在第1至298位残基之间被裂解,以使跨膜结构域具有蛋白水解活性。
所述iRhom多肽也可在跨膜结构域的蛋白水解位点中包含突变。在一个实施方案中,小鼠iRhom2多肽在跨膜结构域的蛋白水解位点(肽酶域)中包含一个或多个关键氨基酸的突变,所述突变选自TM螺旋2上的第635位组氨酸、TM螺旋4上的第695位谷氨酰胺、跨膜螺旋4上的第701位半胱氨酸和第744位组氨酸,以使跨膜结构域具有蛋白水解活性。在另一个实施方案中,人iRhom2多肽在跨膜结构域的蛋白水解位点中包含一个或多个突变,所述突变选自TM螺旋2上的第664位组氨酸、TM螺旋4上的第724位谷氨酰胺、跨膜螺旋4上的第730位半胱氨酸和TM螺旋6上的第773位组氨酸,从而使跨膜结构域具有蛋白水解活性。
筛选测定
本发明提供了鉴定改变iRhom多肽(例如,iRhom1或iRhom2)活性的化合物的方法。所述iRhom多肽可以是突变的iRhom多肽。例如,所述iRhom多肽可在其N-末端胞质结构域包含缺失。所述iRhom多肽可在跨膜肽酶域中包含突变。此外,所述iRhom多肽可在其N-末端胞质结构域中包含缺失,以及在跨膜肽酶域中包含突变。
一方面,本发明的方法通常包括鉴定化合物,所述化合物激活iRhom多肽对底物的蛋白水解活性。在一个实施方案中,所述方法包括使表达iRhom多肽的细胞与测试化合物接触并测定iRhom多肽稳定性相对于合适对照的增加,其中iRhom多肽稳定性的增加表明所述化合物激活了iRhom多肽对底物的蛋白水解活性。在一个实施方案中,所述底物可以是EGFR配体或EGF样底物。
另一方面,本发明的方法通常包括鉴定化合物的方法,所述化合物抑制iRhom多肽对底物的蛋白水解活性。在一个实施方案中,所述方法包括使表达iRhom多肽的细胞与测试化合物接触并测定iRhom多肽的生理靶标的分泌相对于合适对照的减少或EGFR活性相对于合适对照的降低,其中iRhom多肽的生理靶标分泌的减少或EGFR活性的降低表明所述化合物抑制iRhom多肽对底物的蛋白水解活性。在一个实施方案中,所述底物可以是EGFR配体或EGF样底物。
人们认为,表皮生长因子受体(EGFR)信号转导在哺乳动物细胞的生长、增殖和分化中发挥重要作用。本发明提供了iRhom是短寿命蛋白,但是显性突变提高其蛋白稳定性并刺激不依赖于金属蛋白酶活性的特定的EGF家族配体双调蛋白的分泌。本发明证明了哺乳动物iRhom在调节促进创伤加速愈合并引发肿瘤形成的EGFR信号传导事件中的重要性。本发明还提供了iRhom可与金属蛋白酶并行调节EGFR信号传导。因此,本发明提供了使用iRhom作为受损的伤口愈合和癌症中的新型治疗靶标的新策略。
本发明的多方面涉及筛选和测定方法及手段,以及由此鉴定的物质,例如,用于激活或抑制iRhom多肽对底物的蛋白水解活性的化合物的测定。所述底物可以是EGFR配体或EGF样底物。
其他测定用于与iRhom多肽相互作用或结合并且调节(即提高、刺激、降低、抑制或消除)iRhom多肽的蛋白水解活性的化合物或物质。
对照是本领域熟知的,本领域技术人员易于识别出合适对照并且能够仅借助常规实验确定用于本发明方法的合适对照。
根据鉴定调节根据本发明方面的iRhom多肽活性的化合物的方法——其包括使表达iRhom多肽的细胞与测试化合物接触并测定所述测试化合物对iRhom多肽的活性的影响——的方面,当在相同或相似的条件下,表达iRhom多肽的细胞不与所述测试化合物接触时,用合适对照测定iRhom多肽的活性。
合适对照可以是变量的参照水平,例如iRhom多肽的活性、iRhom多肽的稳定性水平、iRhom多肽的生理靶标的分泌水平、相对于合适对照的EGFR活性水平和/或可溶性EGFR配体的水平,其被事先测定并存储在印刷品中或电子介质中,用于调用(recall)并与根据鉴定调节本发明的iRhom多肽活性的化合物的方法的方面而测定的测试化合物对iRhom多肽活性的影响进行比较。
鉴定iRhom多肽的调节剂的测定方法可包括使本文所述的iRhom多肽与测试化合物接触,确定所述测试化合物与iRhom多肽的结合,并在存在和不存在结合iRhom多肽的测试化合物的情况下测定iRhom多肽的蛋白水解活性。可通过下文所述的测定底物的裂解来测定蛋白水解活性。所述iRhom多肽可以是分离的或包含于脂质体或细胞中。
筛选和/或获得调节iRhom多肽活性的物质/化合物的方法可包括在合适的反应培养基中使一种或多种测试物质或化合物与iRhom多肽接触,测定经处理的多肽的活性,并将所述活性与在未用所述一种或多种测试物质处理的相当的反应培养基中所述多肽的活性进行比较。所述iRhom多肽可以以分离的形式存在于反应培养基中或可包含于脂质体或细胞中。
经处理和未经处理的iRhom多肽之间的活性差异指示相关的一种或多种测试物质的调节作用,例如,抑制或增强作用。
可通过测定经蛋白水解而裂解的底物的产生来测定iRhom多肽的活性。例如,所述iRhom多肽可作用于与膜结合的底物以产生被检测的可溶性产物。
根据本发明的另一方面,提供了鉴定和/或获得iRhom多肽的调节剂的测定方法,所述方法包括:(a)在存在多肽底物的情况下使iRhom多肽与测试化合物接触;和(b)测定所述多肽底物的蛋白水解裂解。
可在存在和不存在测试化合物的情况下测定对底物的裂解。存在测试化合物的情况相对于不存在测试化合物的情况下裂解的差异指示所述测试化合物是iRhom蛋白酶活性的调节剂。
任何被iRhom多肽经蛋白水解而裂解的多肽底物均可用于本文所述的测定方法。使用标准技术可容易地鉴定所述底物。所述多肽底物可以为EGFR配体,例如AREG或HB-EGF。
合适的底物可包括可检测的标记,例如绿色荧光蛋白(GFP)、荧光素酶或碱性磷酸酶。这允许便利地检测可溶性裂解产物,并且特别适用于自动化测定。
适用于本测定的EGFR配体在本领域被很好的表征,并且可具有包含一个或多个表皮生长因子(EGF)结构域和单一跨膜结构域的结构。
优选地,合适的EGFR配体与脊椎动物EGFR配体具有大于50%的同源性,大于60%的同源性,大于70%的同源性,大于80%的同源性,大于90%的同源性或大于95%的同源性。
在本文所述的方法中,嵌合配体可具有改进的特性,例如它可更有效地被iRhom多肽裂解,具有改进的分泌特性或更易于被检测。
本发明的另一方面提供了嵌合的EGFR配体,其包含来自两种或更多种EGFR配体的序列,例如嵌合配体可包含第一EGFR配体的跨膜结构域以及第二EGFR配体的胞内和胞外结构域。嵌合底物还可包含可检测的标签,例如荧光素酶、GFP或碱性磷酸酶。
本发明的获得或鉴定iRhom活性调节剂的测定方法或其他方法可以是基于细胞的体内测定或不基于细胞的体外测定。
可在巴马司他(Batimastat)存在下进行所述方法以抑制非菱形蛋白酶或非iRhom依赖的底物脱落,从而减少背景。
在体外测定中,所述iRhom多肽可以是分离的或包含于脂质体中。可使用本领域熟知的方法进行基于脂质体的试验。
用于体外测定的适合的细胞类型包括哺乳动物细胞,例如小鼠胚胎成纤维细胞、角化细胞、HEK293、CHO、HeLa和COS细胞。
对于本发明的体外或体内测定,不必使用完整的全长蛋白。可以本领域技术人员已知的任何合适的方法产生并使用保留全长蛋白活性的如本文所述的多肽片段。产生片段的合适的方法包括但不限于,来自编码DNA的片段的重组表达。可通过取编码DNA、鉴定待表达的部分任一侧的合适的限制性酶识别位点、以及从DNA切下所述部分来产生所述片段。然后可在标准的市售可得的表达系统中将所述部分与合适的启动子可操作地连接。另一种重组方法是使用合适的PCR引物来扩增DNA的相关部分。也可采用本领域熟知的肽合成法来产生小片段(例如,最多达约20或30个氨基酸)。
本发明的测定的准确形式可由本领域技术人员使用常规技术和知识而改变。例如,可通过用可检测的标记来标记一种多肽并使其与已固定在固体载体上的另一种多肽接触,在体外研究所述多肽之间的相互作用。合适的可检测的标记包括35S-甲硫氨酸,其可被掺入到通过重组产生的肽和多肽中。通过重组产生的肽和多肽也可被表达为融合蛋白,其包含可用抗体进行标记的表位。
可产生融合蛋白,其在所述重组蛋白的N-末端或C-末端纳入6个组氨酸残基。所述组氨酸标签可用于使用市售可得的包含金属离子(镍或钴)的柱来进行的蛋白纯化。这些标签也可用于检测蛋白,所述检测使用市售可得的针对所述6个组氨酸残基的单克隆抗体来进行。
本发明的测定可采用体外测定的形式。可在细胞系(例如酵母菌株、昆虫细胞系或哺乳动物细胞系)中进行体外测定,在所述细胞系中从被引入到细胞中的一个或多个载体表达相关的多肽或肽。
在所述实施方案的测定方法或其他方法中,在存在底物(例如EGFR配体)的情况下使iRhom多肽与测试化合物接触。在所述方法中,所述iRhom多肽和底物可存在于细胞中。例如,这可通过从已通过转化被引入到细胞中的一个或多个表达载体表达所述多肽而实现。
用于鉴定和/或获得iRhom蛋白水解活性的调节剂的测定方法因此可包括:(a)在存在EGFR配体多肽的情况下使iRhom多肽与测试化合物接触;和(b)测定EGFR配体的裂解。
可在存活和不存在测试化合物的情况下测定裂解。存在测试化合物的情况相对于不存在测试化合物的情况下裂解的差异指示所述化合物是iRhom活性的调节剂(即增强剂或抑制剂)。
可将编码上文所述的iRhom多肽和/或多肽底物的核酸提供为可复制载体(特别是可在合适条件下从其表达编码多肽的任何表达载体)和宿主细胞(其包含任何所述载体或核酸)的一部分。在本上下文中表达载体是包括编码目的多肽和合适调控序列的核酸的核酸分子,用于在体外表达系统(例如网织红细胞裂解物)或体内(例如,在真核细胞如HEK、COS或CHO细胞中或原核细胞如大肠杆菌中)表达所述多肽。这将在下文中进一步讨论。
组合文库技术提供了测试潜在的大量不同物质调节多肽活性的能力的有效方法。在活性调节的筛选之前或同时,可筛选测试物质与多肽相互作用的能力,例如在酵母双杂交系统(其需要多肽和测试物质都能够在酵母中由编码核酸表达)中进行筛选。这可用作测试物质调节多肽活性的真实能力之前的粗筛。
通常根据所使用的化合物的类型通过反复试验(trial and error)来确定可添加到本发明测定中的测试物质或化合物的量。通常,可使用约0.01至100nM浓度的推定抑制剂或激活剂化合物,例如0.1至10nM。当采用基于细胞的测定时,所述试验物质或化合物需要是膜可透过性的,以接近iRhom多肽。
测试化合物可以是用于药物筛选程序的天然或合成化合物。也可使用包含若干被表征或未被表征的组分的植物提取物。另一类推定的抑制剂或激活剂化合物可获得自iRhom多肽和/或结合其的配体。可测试5至40个氨基酸(例如6至10个氨基酸)的膜可透过性的的肽片段破坏所述相互作用或活性的能力。
在一个实施方案中,调节(例如提高或降低)iRhom多肽的蛋白水解活性的化合物可以为小分子化合物。在另一个实施方案中,所述化合物细胞可透过性的。例如,所述化合物可以为泛素蛋白酶抑制剂。在另一个实施方案中,所述化合物连接至细胞穿透肽。所述细胞穿透肽可包含富含赖氨酸或精氨酸的序列。例如,所述细胞穿透肽可以是tat。
其他测试化合物可基于多肽或肽片段的三维结构的建模并且使用合理的药物设计以提供具有特定分子形状、大小和电荷特征的潜在的抑制剂化合物。
在鉴定调节或影响多肽活性的物质之后,可进一步研究所述物质。此外,可制备所述物质和/或将其用于制备(即制备或配制)组合物(例如药剂、药物组合物或药物)。可将它们给予个体。
本发明的另一方面提供了本文所述的iRhom多肽在获得或鉴定iRhom蛋白水解活性的调节剂(例如抑制剂)的方法中的用途。也提供了iRhom多肽用于多肽底物的跨膜结构域的蛋白水解裂解的方法和用途。
iRhom活性调节剂(特别是抑制剂)可用于治疗癌症,例如食管癌、肺癌、脑癌、结肠癌、肾癌、前列腺癌、皮肤癌、肝癌、胰腺癌、胃癌、子宫癌、卵巢癌、淋巴腺癌或膀胱癌。
iRhom活性调节剂(特别是激活剂)可用于加速组织(例如,皮肤、肝脏、心脏、肌肉或肾)中受损的伤口愈合。
iRhom活性调节剂也可用于治疗炎症病症或疾病,例如类风湿性关节炎和自身免疫性疾病。
因此,本发明在多个方面不仅扩展至根据本文公开的内容被鉴定为iRhom活性调节剂的物质,还扩展至包含所述物质的药物组合物、药剂、药物或其他组合物;包括将所述组合物给予患者(例如用于治疗(其可包括预防性治疗)病原体感染或与异常ErbB或EGF受体活性相关的病症(例如癌症、冠状动脉粥样硬化、银屑病、伤口愈合、毛发生长)、治愈和/或矫正变秃)的方法;所述物质用于制备组合物的用途,所述组合物用于给予,例如用于治疗病原体感染或与异常ErbB或EGF受体活性相关的病症(例如癌症、冠状动脉粥样硬化、银屑病、伤口愈合、毛发生长)、治愈和/或矫正变秃;以及制备药物组合物的方法,包括将所述物质与可药用赋形剂、媒介物或载体以及任选的其他成分混合。
被使用本文所述的测定鉴定为多肽或启动子功能的调节剂的物质本质上可以为肽或非肽。对于许多体内药物用途而言,通常优选非肽“小分子”。因此,可针对药物用途设计所述物质(特别是如果是肽)的类似物或模拟物。
已知的药物活性化合物的类似物的设计是基于“先导(lead)”化合物开发药物的已知方法。当活性化合物难以合成或合成费用高时,或活性化合物不适合特定的给药方法时,可能需要设计其类似物。
类似物设计、合成和测试可用于避免针对靶标特性随机筛选大量分子。
iRhom家族多肽的必要催化残基对应于小鼠iRhom2多肽的TM螺旋2上的第635位组氨酸、TM螺旋4上的第695位谷氨酰胺、TM螺旋4上的第701位半胱氨酸和TM螺旋6上的第744位组氨酸。iRhom家族多肽的必要催化残基也对应于人iRhom2多肽的TM螺旋2上的第664位组氨酸、TM螺旋4上的第724位谷氨酰胺、TM螺旋4上的第730位半胱氨酸和TM螺旋6上的第773位组氨酸。
本发明的能够调节多肽活性的化合物、多肽、肽或物质可在试剂盒中提供,例如密封于适合的容器中,所述容器保护其内容物免受外部环境影响。这类试剂盒可包括使用说明书。
无论待给予个体的是本发明的多肽、抗体、肽、核酸分子、小分子或其他药学上有用的化合物,优选以“预防有效量”或“治疗有效量”(视情况而定,尽管预防可被认为是治疗)给予,这足以对个体显示出益处。实际给予的量、以及给予的速率和时间过程取决于被治疗的病症的性质和严重性。治疗处方(例如对剂量等的决定)在全科医师和其他医生的责任范围内。
根据待治疗的病症,可单独给予组合物或结合其他治疗同时或依次给予组合物。
本发明的化合物
改变iRhom多肽的蛋白水解活性的化合物
本发明的化合物或测试化合物可改变iRhom多肽的蛋白水解活性。在一个实施方案中,所述化合物包括iRhom多肽的蛋白水解活性的激活剂或抑制剂。
在一个实施方案中,所述化合物是结合iRhom多肽的小分子。在另一个实施方案中,所述化合物是抑制iRhom多肽活性的小分子。在另一个实施方案中,所述化合物是激活iRhom多肽活性的小分子。这些小分子可包括,例如,肽、类肽物(peptidomimetics)、有机化合物等。在一个实施方案中,所述小分子化合物能够转运通过质膜并与iRhom多肽相互作用。在一些实施方案中,所述小分子化合物与iRhom多肽的细胞质尾相互作用。在其他实施方案中,所述小分子化合物与iRhom多肽的蛋白水解结构域相互作用。
在一些实施方案中,所述化合物是泛素酶抑制剂。
在另一个实施方案中,改变iRhom多肽的蛋白水解活性的化合物可包括,例如,针对iRhom DNA或mRNA的miRNA、siRNA、shRNA、dsRNA或反义RNA,或编码所述miRNA、siRNA、shRNA、dsRNA或反义RNA的多聚核苷酸,包含所述多聚核苷酸的载体。在另一个实施方案中,改变iRhom多肽的蛋白水解活性的化合物包括,例如,抗体、抗体片段、肽或多肽诱饵。通过筛选可从组合化学抑制剂文库鉴定其他化合物,然后通过化学改变进一步优化这些化合物。
在另一个实施方案中,所述化合物还包括细胞穿透肽。一方面,所述细胞穿透肽包括HIV-TAT肽。
药物组合物
可使用药物组合物给予改变iRhom多肽的蛋白水解活性的本发明的化合物。合适的药物组合物包括本文所述化合物(或其可药用的盐或酯)的任一种,以及任选地包括可药用载体。在某些实施方案中,这些组合物任选地还包括一种或多种额外的治疗剂。
可接受的“药物载体”是本领域技术人员熟知的,并且可包括但不限于任一种标准药物载体,例如磷酸盐缓冲液、水和乳剂(例如油/水乳剂)和各种类型的湿润剂。
本文使用的术语“可药用的盐”是指在合理的医学判断的范围内,适用于与人和低等动物的组织接触而不导致明显的毒性、刺激性、过敏反应等,并且与合理的受益/风险比率相称的那些盐类。胺、羧酸和其他类型化合物的可药用的盐是本领域熟知的。例如,S.M.Berge等人在J.Pharmaceutical Sciences,66:1-19(1977)(以引用的方式纳入本文)中详细记载了可药用的盐。可在本发明化合物的最终分离和纯化过程中原位制备盐,或通过游离碱或游离酸官能团与合适试剂反应而单独地制备盐,如下文所概述的。
例如,游离碱官能团可与合适的酸反应。此外,当本发明的化合物携带酸性基团时,其适合的可药用的盐可包括金属盐,例如碱金属盐类,例如钠盐或钾盐;以及碱土金属盐,例如钙盐和镁盐。可药用的无毒的酸加成盐的实例为氨基与无机酸(例如盐酸、氢溴酸、磷酸、硫酸和高氯酸)或有机酸(例如乙酸、草酸、马来酸、酒石酸、柠檬酸、琥珀酸或丙二酸)形成的盐、或通过使用本领域中使用的其他方法如离子交换而形成的氨基盐。其他可药用的盐包括己二酸盐、海藻酸盐、抗坏血酸盐、天冬氨酸盐、苯磺酸盐、苯甲酸盐、硫酸氢盐、硼酸盐、丁酸盐、樟脑酸盐、樟脑磺酸盐、柠檬酸盐、环戊烷丙酸盐、二葡萄糖酸盐、十二烷基硫酸盐、乙磺酸盐、甲酸盐、延胡索酸盐、葡庚糖酸盐、甘油磷酸盐、葡糖酸盐、半硫酸盐、庚酸盐、己酸盐、氢碘化物、2-羟基-乙磺酸盐、乳糖酸盐、乳酸盐、月桂酸盐、月桂基硫酸盐、苹果酸盐、马来酸盐、丙二酸盐、甲磺酸盐、2-萘磺酸盐、烟酸盐、硝酸盐、油酸盐、草酸盐、棕榈酸盐、双羟萘酸盐(pamoate)、果胶酸盐(pectinate)、过硫酸盐、3-苯丙酸盐、磷酸盐、苦味酸盐、新戊酸盐、丙酸盐、硬脂酸盐、丁二酸盐、硫酸盐、酒石酸盐、硫氰酸盐、对甲苯磺酸盐、十一酸盐、戊酸盐等。代表性的碱金属或碱土金属盐包括钠盐、锂盐、钾盐、钙盐、镁盐等。其他可药用的盐包括,如果适合,使用抗衡离子(例如卤化物、氢氧化物、羧酸盐、硫酸盐、磷酸盐、硝酸盐、低烷基磺酸盐和芳基磺酸盐)形成的无毒的铵、季铵和胺阳离子的盐。
本文使用的术语“可药用的酯”是指在体内水解的酯且包括易于在人体中分解以离开母体化合物或其盐的那些酯。合适的酯基团包括,例如,来自可药用的脂肪族羧酸(特别是烷酸、烯酸、环烷酸和烷二酸)的那些,其中各烷基或烯基有利地具有不多于6个碳原子。具体的酯的实例包括甲酸酯、乙酸酯、丙酸酯、丁酸酯、丙烯酸酯和乙基丁二酸酯。
如上所述,所述药物组合物可额外地包括可药用的载体。所述载体包括任何或所有溶剂、稀释剂或其他液体载体、分散或悬浮助剂、表面活性剂、等渗剂、增稠剂或乳化剂、防腐剂、固体粘合剂、润滑剂等,其适合于所需的特定剂型。Remington’s Pharmaceutical Sciences,第16版,E.W.Martin(Mack Publishing Co.,Easton,Pa.,1980)公开了用于配制药物组合物的各种载体以及已知的制备它们的技术。除了与所述化合物不相容(例如通过产生任何不想要的生物效应或以有害方式与所述药物组合物的任何其他组分相互作用)的任何常规载体介质外,预期常规载体介质的使用落在本发明的范围内。可作为可药用载体的物质的一些实例包括但不限于糖,例如乳糖、葡萄糖和蔗糖;淀粉,例如玉米淀粉和马铃薯淀粉;纤维素及其衍生物,例如羧甲基纤维素钠、乙基纤维素和乙酸纤维素;黄芪胶粉;麦芽;明胶;滑石;赋形剂例如可可脂和栓剂蜡;油类,例如花生油、棉花籽油、红花油、芝麻油、橄榄油、玉米油和大豆油;二醇类,例如丙二醇;酯类,例如油酸乙酯和月桂酸乙酯;琼脂;缓冲剂,例如氢氧化镁和氢氧化铝;海藻酸;无热原的水;等渗盐水;林格氏溶液;乙醇和磷酸盐缓冲液,以及其他无毒的相容的润滑剂如十二烷基硫酸钠和硬脂酸镁,以及着色剂、释放剂、涂层剂、甜味剂、调味剂和芳香剂,防腐剂和抗氧化剂也可存在于所述组合物中,这依据调制人员的判断。
可将用于本发明的组合物配制成具有任意浓度的所需化合物。在一些实施方案中,配制所述组合物以使其包含至少治疗有效量的改变iRhom多肽的蛋白水解活性的化合物。治疗有效量是在给药条件下足以实现所需治疗效果的量,例如足以治疗癌症、加速伤口愈合或治疗炎性病症的量。在一些实施方案中,配制所述组合物以使其包含不会导致一种或多种不想要的副作用的量。在某些实施方案中,配制所述组合物以使所述化合物以约1mg/mL至约20mg/mL、约1mg/mL至约15mg/mL、约1mg/mL至约10mg/mL、约2mg/mL至约9mg/mL、约3mg/mL至约8mg/mL、约4mg/mL至约7mg/mL、约4mg/mL至约6mg/mL的浓度存在。在某些实施方案中,配制所述组合物以使所述化合物以约5mg/mL的浓度存在。
试剂盒
本发明还提供了组合物和试剂盒,用于预测改变iRhom多肽活性的化合物在受试者中加速伤口愈合、治疗癌症或治疗炎性疾病的能力,或用于确定受试者中的癌症是否对所述化合物的治疗敏感。这些试剂盒包括以下的一种或多种:获得和/或制备样品(例如,皮肤活检、肿瘤活检或血液样品)的试剂;确定样品是否表现出iRhom蛋白水解活性的试剂;确定样品是否表现出基因(例如,Rhbdf1或Rhbdf2基因)突变的探针和试剂;确定样品是否表现出野生型基因(例如,Rhbdf1或Rhbdf2基因)序列的探针和试剂;确定样品中iRhom多肽的半衰期的试剂;确定样品分泌EGFR配体的试剂;以及使用说明书。
本发明的试剂盒可任选地包括其他可用于实施本发明方法的组分。例如,所述试剂盒可包括适于使互补核酸退火或适于使抗体与其特异性结合的蛋白相结合的液体(例如,SSC缓冲液)、一个或多个样品室、描述本发明方法的实施的指导材料、正常细胞的样品、癌细胞的样品、创伤细胞的样品、炎症细胞的样品等。
适应症
伤口愈合
伤口愈合是一个过程,皮肤或另一器官组织在受伤后借助此过程自我修复。在一个实施方案中,本发明可用于鉴定在受试者中激活iRhom多肽的蛋白水解位点,从而加速伤口愈合的化合物。
本发明的方法可用于鉴定在受试者中加速伤口愈合的化合物。在一个实施方案中,所述化合物加速伤口愈合,以实现伤口闭合。在一个实施方案中,所述伤口可以是开放的皮肤伤口,例如烧伤、化学(例如碱)烧伤引起的伤口、物理创伤的伤口、神经性溃疡、压疮、静脉淤滞性溃疡和糖尿病性溃疡。
在另一个实施方案中,本发明提供了激活iRhom多肽的蛋白水解位点以在受试者中加速、促进或增强伤口愈合的化合物或药物组合物。
在一些实施方案中,本发明提供了上文所述或本文他处所述的化合物的任一种激活iRhom多肽的蛋白水解位点以在伤口周围或伤口附近加速伤口愈合或调节一种或多种细胞特性的用途。在一些实施方案中,本发明提供了上文所述的组合物在伤口周围或伤口附近增强伤口愈合或调节一种或多种细胞特性的用途。
癌症
本发明的化合物可用于在受试者中治疗癌症。本发明的化合物也可用于在受试者中降低肿瘤生长和/或进展。
在一个实施方案中,本发明可用于鉴定能够在受试者中降低肿瘤生长和/或进展或治疗癌症的化合物。在一个实施方案中,所述化合物抑制iRhom多肽的蛋白水解位点,从而减少iRhom多肽的生理靶标的分泌和/或降低EGFR活性,从而降低肿瘤生长和/或进展。在另一个实施方案中,所述化合物抑制iRhom多肽的蛋白水解位点,从而减少iRhom多肽的生理靶标的分泌和/或降低EGFR活性,从而可用于治疗癌症。
在一些实施方案中,编码iRhom2的基因RHBDF2的错义突变(p.I186T;p.P189L;p.D188N)可导致胼胝症及人食管癌,其特征在于掌跖和口的过度角化。
本发明的化合物也可用于治疗各种癌症。在一个实施方案中,所述癌症为上皮癌。在另一个实施方案中,所述癌症为食管癌、肺癌、脑癌、结肠癌、肾癌、前列腺癌、皮肤癌、肝癌、胰腺癌、胃癌、子宫癌、卵巢癌、乳腺癌、淋巴腺癌或膀胱癌。
本发明的化合物也可用于治疗各种肿瘤。在一个实施方案中,所述肿瘤是实体瘤。
炎症
本发明的化合物可用于在受试者中减轻炎症。本发明的化合物可用于治疗炎性疾病和/或病症。炎性疾病和/或病症包括,例如,系统性红斑狼疮(SLE)、狼疮肾炎、冷球蛋白血症、脉管炎、类风湿性关节炎、干燥综合征(Sjoegren’s syndrome)、葡萄膜炎、脊椎关节炎、流产、先兆子痫、寻常痤疮、哮喘、自身免疫性疾病、乳糜泻、慢性前列腺炎、肾小球性肾炎、超敏反应、炎性肠疾病(包括溃疡性结肠炎和节段性肠炎(Crohn’s disease))、盆腔炎性疾病、再灌注损伤、类风湿性关节炎、结节病、移植排斥、脉管炎、间质性膀胱炎、动脉粥样硬化、过敏反应、肌病、白细胞缺陷、银屑病、多发性硬化症和癌症。
在一个实施方案中,本发明可用于鉴定能够在受试者中减轻炎症或治疗炎性疾病的化合物。在一个实施方案中,所述化合物降低iRhom多肽的蛋白水解结构域的活性,以使iRhom多肽的生理靶标的分泌减少和/或TNFα分泌减少,从而使炎症减轻。在另一个实施方案中,所述化合物降低iRhom多肽的蛋白水解结构域的活性,以使iRhom多肽的生理靶标的分泌减少和/或TNFα分泌减少,所述化合物因此可用于于治疗炎性病症或疾病。在另一个实施方案中,本发明的化合物通过靶向细胞溶质结构域或跨膜肽酶域而减少TNFα分泌,从而可用于在受试者中减轻炎症。
可利用各种测试测定化合物治疗炎性病症的功效。
毛发生长
本发明的化合物可用于在受试者中治疗秃顶和/或促进毛发生长。毛发脱落或秃顶(例如,脱发症(alopecia))是毛发从头部或身体脱落。秃顶可以指一般的毛发脱落、男性模式的秃顶或女性毛发脱落。一些类型的秃顶可由斑秃(alopecia areata)——一种自身免疫性疾病——引起。斑秃的极端形式为全秃(alopecia totalis),其包括全部头发的脱落,以及普秃(alopecia universalis),其包括头部和身体的全部毛发脱落。
在一个实施方案中,本发明基于以下发现:cub突变导致变秃。在另一个实施方案中,本发明基于以下发现:cub(Mcub)的修饰因子促进毛发生长、治愈变秃、矫正变秃或减轻变秃(实施例1)。
在一个实施方案中,本发明可用于鉴定能够在受试者中促进毛发生长的化合物,其包括:使表达iRhom多肽的细胞与测试化合物接触;并测定iRhom多肽的生理靶标的分泌相对于合适对照的减少,或EGFR活性相对于合适对照的降低,其中iRhom多肽的生理靶标分泌的减少或EGFR活性的降低指示所述化合物能够在受试者中促进毛发生长。
在另一个实施方案中,本发明可用于鉴定能够在受试者中治愈和/或矫正秃顶的化合物,其包括:使表达iRhom多肽的细胞与测试化合物接触;并测定iRhom多肽的生理靶标的分泌相对于合适对照的减少,或EGFR活性相对于合适对照的降低,其中iRhom多肽的生理靶标分泌的减少或EGFR活性的降低指示所述化合物能够在受试者中治愈和/或矫正秃顶。
通过以下实施例进一步对本发明进行举例说明,所述实施例不应解释为限制性的。本申请通篇引用的所有文献、专利和公开的专利申请的全部内容由此以引用的方式纳入本文。
实施例
实施例1:cub突变导致EGFR信号传导通路的过度激活
被命名为卷曲-裸露(cub)的隐性小鼠突变的特征为无毛表型。显性Mcub等位基因的单拷贝与cub/cub基因型的组合产生丰满、卷曲的毛皮而非cub/cub mcub/mcub小鼠的无毛毛皮。我们研究了cub/cub mcub/mcub基因型是否可能与改变的EGFR信号传导方面(例如,细胞增殖和细胞迁移)有关。
我们对从cub/cub mcub/mcub小鼠和对照(+/+mcub/mcub)小鼠分离的小鼠胚胎成纤维细胞(MEF)进行增殖、细胞迁移(划痕-伤口愈合)和免疫印迹测定。图1A-1F示出了cub突变加速EGFR相关的细胞增殖和迁移以及皮肤愈合。
cub/cub mcub/mcub MEF相对于对照MEF具有显著更高的增殖率和迁移率(图1A和1B)。这些改变与EGFR通路的典型信号转导蛋白(包括Akt、S6、mTOR和p38)的磷酸化的显著增加有关(图1C)。此外,我们观察到cub/cub mcub/mcub MEF中细胞表面EGFR水平的显著下降(图1C),表明EGFR信号传导的内化和组成型激活。
为了测试EGFR信号传导的增加是否对应于上皮增殖的变化,我们进行了伤口-愈合测定,在测定中我们在6-40周龄cub小鼠的耳中穿出2mm通孔并在随后的多天监测闭合率(图1D-1E)。在14天中,与对照+/+mcub/mcub同窝幼仔相比,cub/cub mcub/mcub小鼠显示出加速闭合。相对于对照小鼠,+/cub mcub/mcub小鼠(其具有正常皮毛)还在受伤后14天显示出更快的愈合,参见图9A和9B,表明单一突变的cub等位基因能够引起EGFR信号传导的增加,尽管增加的水平不足以阻断毛囊诱导。这些数据表明,cub/cub mcub/mcub基因型产生了活性过高的EGFR表型。
实施例2:cub是编码iRhom2的Rhbdf2基因的突变
接下来,我们检验了作为cub突变的候选物的iRhom2基因Rhbdf2。cub/cub小鼠的DNA测序鉴定出Rhbdf2基因中12,681碱基对(bp)缺失,这导致外显子2-6的缺失(图2A)。为了测试所述缺失是否产生异常的Rhbdf2转录物,我们使用被设计以扩增外显子2、5、12和19的引物对来自野生型和cub/cub mcub/mcub MEF的RNA进行逆转录酶PCR(RT-PCR)。cub转录物含有外显子12和19,但是缺少外显子2和5(图2B)。另外,对来自cub/cub mcub/mcub和+/+mcub/mcub皮肤的RNA进行的定量RT-PCR(qPCR)扩增出cub转录物,其中外显子2-6被缺失但是其余外显子被表达,表明在cub小鼠中cub基因的其余部分被转录(图2C)。为了测试cub转录物是否导致外显子1至7的剪接,我们使用专门被设计以仅扩增包含剪接至外显子7的外显子1的转录物的探针进行qPCR。使用该探针,我们在cub/cub mcub/mcub小鼠中检测到PCR产物,但是未在+/+mcub/mcub小鼠中检测到(图2D)。这些发现确认了cub是Rhbdf2基因的突变,并将在下文中被称为Rhbdf2cub。
考虑到Rhbdf2cub转录物中外显子3中的正常翻译起始位点是缺失的,我们接下来测试这些突变转录物是否会产生突变的iRhom2蛋白。对Rhbdf2cub DNA的序列分析表明下一个框内翻译起始位点(ATG)在外显子8中,其将产生约63.5kDa的蛋白。由于我们缺少iRhom2的抗体,我们通过将全长野生型人RHBDF2cDNA(Hu Wt)以及模拟突变Rhbdf2cub转录物的版本(HuCub)同时克隆到C-末端Flag标记的表达载体中,测试了Rhbdf2cub转录物是否能够在体外产生蛋白产物。首先,使用免疫印迹法,我们确定Hu Wt克隆产生约100kDa的蛋白产物,而HuCub突变体构建体产生约67kDa的产物,这与预期的标记蛋白的分子量一致(图2E)。此外,Hu Wt克隆中没有任何缩短的蛋白产物的证据。第二,我们使用免疫染色法检验了用Hu Wt或HuCub克隆转染B6MEF之后两种蛋白形式的定位模式(图2F以及图10A和10B)。这两种形式都在内质网中表达,并且在高尔基体或细胞核中未观察到染色(图10A和10B),表明所述突变未导致蛋白定位改变。
实施例3:遗传非互补证实cub是Rhbdf2基因的突变等位基因,并且是功能获得性突变
为了测试Rhbdf2cub是否是功能获得性突变而非无效突变,我们使用来自敲除小鼠项目(KOMP)库的ES细胞产生了Rhbdf2敲除(Rhbdf2-/-)小鼠,其中lacZ表达是在内源Rhbdf2启动子的控制下(图3A)。主要在皮肤的表皮和毛囊的内鞘层和外鞘层中观察到Rhbdf2启动子驱动的lacZ表达(图3B)。但是,在Rhbdf2cub/cub小鼠中观察到的伤口愈合(图3C)和毛发脱落(图3D)表型未见于Rhbdf2-/-小鼠中,其在其他方面看起来是正常的。Rhbdf2cub/cub和Rhbdf2-/-小鼠的等位基因测试交配产生了具有稀疏毛皮的复合突变(Rhbdf2-/cub)小鼠,表明遗传非互补,并且证实cub是Rhbdf2基因的突变等位基因。另外,Rhbdf2-/cub复合突变小鼠的毛发生长水平介于Rhbdf2cub/cub小鼠和Rhbdf2+/+或Rhbdf2-/-小鼠之间,证实突变Rhbdf2cub蛋白产物的表达而非Rhbdf2缺乏导致Rhbdf2cub/cub表型。
实施例4:Rhbdf2cub表型的遗传修饰因子(Mcub)是Areg基因的功能丧失性突变
接下来我们试图定位引起Rhbdf2cub修饰基因(Mcub)的突变。我们检验了4个作为Mcub突变的候选基因座的编码EGFR配体的基因(Epng、Ereg、Areg和Btc)。通过对Mcub/Mcub Rhbdf2cub/cub小鼠中各基因的外显子和侧翼区进行测序(参见表II),我们发现Mcub是Areg(编码自分泌角化细胞生长因子双调蛋白(本文中称为AREG)的基因)中的功能丧失性突变(图4A)。
Mcub是T至G的点突变,所述突变破坏外显子1的典型供体剪接位点并导致排他使用可变的下游剪接位点,其向Areg转录物添加22个额外的核苷酸;这破坏了编码框并引入提前终止密码子(图11)。该突变将在下文中被称为AregMcub。值得注意的是,当存在单拷贝的显性AregMcub等位基因时,Rhbdf2cub/cub小鼠的活性过高的EGFR信号传导(图12A)和快速的伤口闭合的能力显著降低(图4B和4C),并阻止了毛发缺失表型的出现。显性AregMcub突变并不赋予Rhbdf2cub/cub小鼠正常毛皮,而是赋予卷曲毛发表型,表明EGFR途径中的其余异常情况。
我们接下来测量了Rhbdf2cub/cub Areg+/+、Rhbdf2cub/cub AregMcub/Mcub和Rhbdf2+/+Areg+/+小鼠中AREG的血清水平。我们发现Rhbdf2cub/cub AregMcub/Mcub小鼠的血清中无可检测的AREG,但是与Rhbdf2+/+Areg+/+小鼠相比在Rhbdf2cub/cub Areg+/+小鼠中观察到血清AREG水平的显著升高(图4D)。
对来自Rhbdf2cub/cub Areg+/+、Rhbdf2cub/cub AregMcub/Mcub和Rhbdf2+/+Areg+/+小鼠的经培养的小鼠表皮角化细胞(MEK)的上清液AREG水平进行的测量产生了相似的结果(图4E)。AREG在正常皮肤中大量表达;因此,我们接下来对来自Rhbdf2cub/cub Areg+/+和Rhbdf2+/+Areg+/+小鼠的皮肤样品进行qPCR以测量Areg以及6种其他已知编码EGFR配体的基因(Egf、Tgfα、Btc、Epgn、Ereg和Hbegf)的转录物水平。与对照相比,我们在Rhbdf2cub/cub Areg+/+小鼠中观察到Areg的4倍增加,以及Epgn和Hbegf mRNA的微小但统计学上显著的增加。此外,Btc和Egf转录物水平出现统计学上显著的降低(图4F)。
最后,为了检验AREG是否介导活性过高的EGFR表型,我们使用慢病毒shRNA使Rhbdf2+/+和Rhbdf2cub/cub MEF中的Areg表达沉默(图12B)并进行了增殖测定。Areg沉默对Rhbdf2+/+MEF的增殖具有微小的影响(图12C),而Rhbdf2cub/cub MEF的增殖率显著降低(图12D)。总之,这些数据表明提高的AREG水平通过增加EGFR信号传导来介导Rhbdf2cub/cub小鼠的突变表型。此外,我们的数据表明Rhbdf2cub表型由AregMcub表达修饰。
实施例5:N-末端截短的iRhom2(Rhbdf2cub)的肽酶域诱导不依赖于ADAM17的EGFR配体的底物特异性分泌
为了测试Rhbdf2cub是否能够——通过其短的N-末端结构域——诱导不依赖于金属蛋白酶活性的AREG分泌,我们在马立马司他(MM)——一种能够同时阻止ADAM17和ADAM10依赖性底物脱落的强效广谱金属蛋白酶抑制剂——存在或不存在的情况下进行了体外裂解测定。由于小鼠和人RHBDF2基因之间的显著同源性,我们单独使用人AREG或使用人AREG与人野生型(Hu Wt)或人Cub(HuCub)RHBDF2基因转染293T细胞(图5A),并在条件培养基中测量AREG水平。在不存在MM时,我们观察到表达AREG的细胞和共表达AREG/HuCub的细胞之间的AREG水平没有差异,而Hu Wt和AREG的共表达使AREG水平降低约60%(图5B)。在存在MM时,与用Hu Wt和AREG转染或单独用AREG转染的细胞相比,HuCub/AREG共转染的细胞中AREG水平约高出两倍(图5B)。这些数据表明,N-末端截短的iRhom2能够增强AREG分泌,该分泌仅在金属蛋白酶活性减小时变得明显。
可通过在用细菌内毒素脂多糖(LPS)刺激之后检查肿瘤坏死因子α(TNFα)分泌来测量小鼠中的ADAM17活性。我们使用该方法,通过将LPS注射到Rhbdf2cub/cub、Rhbdf2-/-和Rhbdf2+/+小鼠中并测量血清TNFα水平以测试Rhbdf2cub小鼠中ADAM17活性的变化。我们发现相对于注射LPS的Rhbdf2+/+小鼠,注射LPS的Rhbdf2cub/cub和Rhbdf2-/-小鼠均具有显著更低的TNFα诱导(图5C)。这些结果表明在Rhbdf2cub/cub和Rhbdf2-/-小鼠中ADAM17活性显著减弱,也显示了iRhom2的N-末端结构域在调节ADAM17依赖性TNFα释放中的作用。值得注意的是,LPS刺激的Rhbdf2cub/cub小鼠中血清TNFα水平并未完全消除,表明ADAM17活性减弱但并未消失。观察到的Rhbdf2cub/cub小鼠中的ADAM17活性减弱解释了与Adam17-/-小鼠相比或与过表达双调蛋白的转基因小鼠相比Rhbdf2-/-和Rhbdf2cub/cub小鼠中的非致死性或非炎性皮肤表型。
接下来,评估了活性菱形蛋白酶对其他EGF样底物的蛋白酶选择性,以及这些是否与HuCub有区别。我们发现,与空载体转染的对照相比,RHBDL2选择性地增加EGF的分泌但不增加AREG或HB-EGF的分泌(图5D)。相比之下,与空载体转染的细胞相比,HuCub选择性地增加AREG和HB-EGF的分泌但不增加EGF的分泌(图5E-5G)。这些结果表明,菱形蛋白酶肽酶域赋予底物选择性。N-末端截短的iRhom1能够诱导AREG分泌但不能诱导EGF分泌与该论断一致(图13A和13B)。
为了确定iRhom2肽酶域如何有助于调节EGF样底物的分泌,我们首先比对了人和小鼠iRhom的肽酶域的氨基酸序列。我们发现人iRhom2和RHBDL2的肽酶域之间的蛋白序列同源性具有显著差异(图13C)。相比之下,iRhom1和2的肽酶域显示出96%的同源性。这共同表明有活性和无活性的菱形蛋白酶之间形成活性位点或在底物识别中发挥作用的残基显著不同。此外,我们发现,与天然HuCub相比,HuCub肽酶域的缺失显著减少了AREG分泌(图5G),表明所述肽酶域对于EGF样底物的分泌是必需的。为了鉴定关键残基,我们进行了定点诱变,以使HuCub肽酶域中的关键残基突变为丙氨酸。我们发现第4跨膜结构域中的第426位谷氨酰胺和第432位半胱氨酸以及分别在第2和6跨膜结构域中的第336位和第475位组氨酸不仅对于介导AREG/HB-EGF的分泌增加是关键的,而且对于EGF的抑制也是关键的(图5H、I)。但是,丝氨酸突变S362A、402A和425A,谷氨酸突变E436A,以及谷氨酰胺突变Q439A不改变AREG分泌(图13D)。这些结果共同表明N-末端截短的iRhom的肽酶域中的关键残基(图5J和图13E)调节不依赖于金属蛋白酶活性的EGF样底物的分泌。
实施例6:N-末端截短的iRhom2(Rhbdf2cub)增强对上皮癌的易感性
RHBDF2的错义突变(p.I186T、p.P189L和p.D188N)可以是家族性胼胝症以及食管癌综合征(TOC)的起因。为了测试由于RHBDF2中的显性N-末端突变而增加的AREG分泌是否可能通过在293T细胞中共表达AREG与含有人错义突变(RHBDF2p.I186T)的HuWt克隆、HuCub或Hu Wt在体外驱动这些人类病理变化。与Hu Wt相比,RHBDF2p.I186T或HuCub的表达导致AREG在条件培养基中的更高水平和更低的细胞内水平(图6A和6B)。此外,RHBDF2p.I186T产生的AREG水平与HuCub所产生的那些相当,表明iRhom2N-末端的缺失或显性突变导致AREG分泌增加。另外,我们发现,RHBDF2p.I186T突变体的肽酶域中的4个关键残基(H、C、Q和H)中的至少一个的缺失导致AREG分泌显著减少(图6C)。
为了确定Rhbdf2cub等位基因是否增强肿瘤易感性,我们研究了在ApcMin/+小鼠(人家族性腺瘤性息肉病的小鼠模型)中其表达如何影响腺瘤形成。在ApcMin/+小鼠中,一个野生型Apc等位基因的自发缺失诱导肠内上皮腺瘤形成,以及在169天的中值年龄时的过早死亡。值得注意的是,在小肠中观察到Rhbdf2(图6D)和Areg表达,表明可能的功能性关系。我们产生并观察了ApcMin/+Rhbdf2+/cub和ApcMin/+Rhbdf2+/+小鼠,但是由于致死率增加我们无法产生足够的ApcMin/+Rhbdf2cub/cub小鼠用于有意义的比较。尽管如此,我们确实观察到ApcMin/+Rhbdf2+/+(172天)和ApcMin/+Rhbdf2+/cub(135天)小鼠的中值生存期的显著差异(图6E)。对3月龄的ApcMin/+Rhbdf2+/+和ApcMin/+Rhbdf2+/cub小鼠的尸检揭示ApcMin/+小鼠中单一Rhbdf2cub等位基因的存在显著增加了息肉的数目(图6F和6G)和腺瘤大小(图6F和6H),表明cub突变使上皮肿瘤生长增加。但是,在最长达2年龄的Rhbdf2cub/cub小鼠中没有自发的癌症发生,表明Rhbdf2cub突变产生了有利于肿瘤发展的环境,但是并不单独地驱使肿瘤发展。
实施例7:RHBDF2基因的细胞溶质N-末端的缺失或N-末端中的显性突变增加了其蛋白稳定性
iRhom2通过促进EGF样配体经蛋白酶体通路降解而负调控EGFR信号传导。我们确定,当缺少细胞溶质N-末端时,iRhom诱导AREG/HB-EGF的分泌(图5)。为了测试iRhom2的氨基端的功能获得性突变是否干扰蛋白酶体加工并由此增加其稳定性,我们检验了胼胝RHBDF2突变体p.I186T是否能够与AREG相互作用。我们发现,类似于Hu Wt和HuCub,p.I186T与AREG形成物理复合体(图7A)。我们之后通过免疫细胞化学和流式细胞术比较了293T和COS7细胞中Hu Wt、HuCub和p.I186T的蛋白表达水平。我们观察到,与HuCub和p.I186T相比,HuWt蛋白表达水平显著更低(图7B、C)。此外,当我们对COS7细胞进行所示时间的环己酰胺(一种蛋白合成抑制剂)追踪时,在1小时内我们观察到与HuCub或p.I186T相比Hu Wt的免疫反应性约下降50%(图7D、E)。
为了测试iRhom2是否是蛋白酶体降解的靶标,我们测定了在强效蛋白酶体抑制剂MG-132的存在下Hu Wt和HuCub的蛋白半衰期。意料之中的,Hu Wt的蛋白半衰期显著增大,但是HuCub的并没有(图7F),表明iRhom2的蛋白酶体降解可能影响其蛋白稳定性。我们得出结论,类似于Rhbdf2cub突变,iRhom2的氨基端的错义突变增加了其稳定性并有助于增加不依赖于金属蛋白酶活性的AREG的分泌。
实施例8
此处,我们报道了iRhom2(被熟知作为果蝇属中EGFR信号传导的调节子的菱形蛋白酶家族的成员)在皮肤愈合和肿瘤发展中能够调节EGFR信号传导。我们证明了iRhom2是短寿命蛋白,其稳定性可通过N-末端结构域的选择突变而增加。这些稳定的变体进而发挥增加不依赖于金属蛋白酶活性的AREG分泌的作用。我们鉴定了iRhom在EGFR依赖性细胞增殖和创伤愈合中的重要作用,并证明了在适当的情况下增加EGFR信号传导的iRhom2突变如何能够促进肿瘤发展。
N-末端结构域和肽酶域在调节EGFR信号传导中具有独立的功能
iRhom是复合多结构域酶,其包含长的细胞溶质N-末端、休眠的肽酶域和保守的iRhom同源结构域(IRHD);这些结构域的功能仍然是未知的。在正常情况下,iRhom通过促进EGF样底物的降解而负调控EGFR信号传导。但是,Rhbdf2cub突变不可能只是功能丧失性突变。Rhbdf2-/-小鼠无法重现(recapitulate)Rhbdf2cub表型。此外,我们证明,与Rhbdf2cub突变类似,iRhom2的N-末端的显性错义突变以由肽酶域的跨膜螺旋2、4和6中的关键氨基酸介导的方式诱导AREG和HB-EGF的分泌。因此,我们的结果表明,iRhom2的细胞溶质N-末端通过抑制肽酶域而负调控EGFR信号传导,从而负调控AREG/HB-EGF的分泌(图8)。我们的发现还揭示出iRhom2的更精细的调控功能,其在N-末端突变(例如Rhbdf2cub)中被揭示出。
Rhbdf2cub可被认为是功能获得性突变。该结论由若干证据支持。首先,iRhom2的负调控作用似乎是最小的,因为除非与Rhbdf2cub突变结合,Rhbdf2-/-小鼠并不表现出明显的“EGFR活性过高”的表型。第二,与Hu Wt和AREG的转染或单独的AREG转染相比,HuCub和AREG的共转染使AREG水平约高出2-3倍。第三,HuCub的表达诱导不依赖于金属蛋白酶活性的膜锚定的AREG和HB-EGF的分泌。第四,突变的iRhom2等位基因在不存在肽酶域的情况下无法诱导AREG/HB-EGF的分泌。与这些观察一致的是,N-末端截短的RHBDF1而非全长RHBDF1的转基因表达在果蝇属中诱导强EGFR信号传导相关的翅表型。另外,被截短的RHBDF1与HB-EGF的共表达在果蝇属中强化了改变的翅表型,表明被截短的iRhom1可能诱导HB-EGF的分泌,从而激活EGFR信号传导。这些结果验证了以下观点:细胞溶质N-末端有助于iRhom引起的EGF家族配体的泛素加工,而iRhom肽酶域在不被N-末端抑制时刺激EGFR信号传导。
我们发现在饱和浓度的马立马司他(一种强效、广谱的金属蛋白酶抑制剂)存在下,突变的iRhom2等位基因诱导AREG和HB-EGF的分泌。ADAM家族其他成员的成熟不受这两种iRhom的缺乏的影响。此外,我们证明了在Rhbdf2cub小鼠中ADAM17活性减弱。因此,我们得出结论,Rhbdf2cub和胼胝突变选择性地诱导不依赖于金属蛋白酶活性的AREG和HB-EGF分泌,并且跨膜肽酶域对该功能是必需的。
在Rhbdf2cub小鼠中活性过高的EGFR通路是皮肤加速愈合的原因
伤口愈合的生物学是复杂的。在人类中,成年人的伤口易受到非功能性纤维组织形成的影响,而胎儿期的伤口能实现完全再生,类似于脊椎动物(例如美西螈)和真涡虫中的无伤疤愈合。
组织再生和重塑领域仍处于初级阶段。在人角化细胞系中进行的体外伤口愈合测定表明,与未受伤的对照相比,受伤后RHBDL2的表达显著上调。
Rhbdf2cub小鼠能够快速使伤口愈合而不形成明显伤疤的原因可能是以下二者的结合:由于ADAM17活性减弱而使TNFα分泌减少,以及增加的AREG产生/EGFR过度激活所引起的快速上皮再生。我们提出,在Rhbdf2cub小鼠中尽管TNFα减少有助于使炎症程度更低,但AREG产生增加促进增殖加速和角化细胞迁移到伤口处。此外,iRhom2主要在皮肤中表达,使其成为受损的皮肤伤口愈合中潜在的新治疗靶标。
在一些上皮癌中iRhom2-AREG-EGFR通路具有组成型活性
在本研究中,我们发现N-末端截短的iRhom2促进不依赖于ADAM17活性的AREG产生的增加,从而诱导EGFR激活。特别地,ApcMin/+Rhbdf2+/cub小鼠的表型重现了在具有显性RHBDF2突变的患者中所见的对上皮癌的增强的易感性。但是,Rhbdf2cub/cub小鼠并未自发地发展肿瘤,表明Rhbdf2cub突变可能并不驱使癌症发展,而是可能通过产生有利环境而促进肿瘤生长和进展。
材料和方法
在Jackson实验室(Bar Harbor,ME)在改良的屏障(barrier)条件下获得、饲养并维持小鼠。将所有基因型(包括cub和Mcub)维持在C57BL/6J(B6)遗传背景上。为了产生Rhbdf2-/-小鼠,将从敲除小鼠项目(KOMP)库获得的ES细胞克隆(EPD0208_1_A09)注射到B6-Tyrc(B6白化病)胚泡中。使表现出>50%嵌合性的雄性与B6白化病雌性交配;通过PCR对黑色后代进行基因分型。使杂合体相互交配以产生纯合体,或使杂合体与Rhbdf2cub/cub小鼠交配以产生Rhbdf2-/cub小鼠。Jackson实验室的动物保护与使用委员会批准了全部实验操作。
细胞培养和试剂——如之前所述我们分离了MEF。根据制造商的说明书(CELLnTEC/Zenbio,Research Triangle Park,NC)分离小鼠表皮角化细胞(MEK)。使细胞生长在37℃、含有5%CO2的增湿室中。分别从Fisher Scientific(Boston,MA)和Zen-Bio(Research Triangle Park,NC)购买MEF试剂[(磷酸盐缓冲溶液(PBS)、HyClone Dulbeccos Modified Eagles培养基、高葡萄糖(DMEM)、青霉素-链霉素和HyClone胎牛血清)]和MEK试剂(祖细胞靶向培养基CnT-07、抗生素/抗真菌溶液ABM和蛋白酶Dispase)。从Life technologies(Chicago,IL)获得TrypLETM Select。使293T和COS7细胞分别生长在DMEM和RPMI培养基中。
组织学——如之前所述制备用于组织学检查的组织。
耳孔闭合:使用外科手术用的耳打孔装置Napox KN-292B使小鼠受伤以在左耳中央制造2mm通孔,并在28天期间分析闭合率。在所示的时间点,使小鼠安乐死并使用4×物镜(Olympus BX41显微镜)捕获耳孔及周围组织的图像。ImageJ软件分析受伤区域。在室温下将耳固定在10%中性缓冲福尔马林(NBF)中24h并进行石蜡包埋和切片处理。在第0天测量物理创伤面积并将在所有随后时间点的测量值计算为初始面积的百分数。
增殖和划痕测定:使用MEF进行这两组测定。对于增殖测定,在37℃使所示数目的细胞生长在胶原包被的96孔板中。孵育24h之后,移除生长培养基并在-80℃冷冻平板直至使用。使用细胞增殖试剂盒测定相对细胞数目(Life technologies)。使用Victor3多标记孔板读数仪(PerkinElmer,Boston,MA)测量荧光强度。对于划痕测定,使MEF在胶原包被的6孔板中生长至汇合,并通过将无菌p200吸管端拉过孔从汇合的单层除去一条细胞。使用Zeiss Observer相差显微镜上的5×物镜和Axio Vision 4.6.3软件在0、3、6、9和12h时获取划痕图像。使用GRID插件,我们在每个图像上覆盖了网格。所述网格大小使至少10条线等分划痕图像。汇集各划痕的宽度并计算平均值。用ImageJ软件分析来自0、3、6、9和12h时间序列的捕获图像。
人AREG、HB-EGF和EGF ELISA:在被接种于24孔板的293T细胞中进行瞬时转染。使用脂质体LTX以及Plus试剂,将16.5fmol HuWt或HuCub与25ng AREG或50ng HB-EGF或200ng EGF共转染。使每个孔中转染的DNA总量与pCMV6-AC-HA载体等量。24h之后,用溶于1ml新鲜培养基的DMSO或10μM MM替换旧培养基。24h之后,将100μl上清液不稀释(HB-EGF和EGF)或稀释5倍(AREG),并根据制造商的说明书进行ELISA(DY262、DY259和DY236)。
从OriGene(Rockville,MD)获得包含编码人AREG(RC203150)、人HB-EGF(SC108485)、人EGF(SC127840)、人RHBDF2(RC203923)、小鼠Rhbdf1(MC205414)和人RHBDL2(RC219882)的cDNA的表达质粒。根据制造商的说明书使用QuikChange II XL诱变试剂盒(Agilent Technologies,Santa Clara,CA)在全长RHBDF2或小鼠Rhbdf1中引入突变(缺失或插入),并通过DNA测序来验证。使用引物设计程序( Primer Design,Agilent Technologies)产生用于定向诱变的引物。
免疫化学:使铺在BD BioCoatTM胶原I 2-孔培养载片中的MEF生长至约60%汇合,并根据制造商的说明书用LipofectamineTM LTX(Life technologies)瞬时转染。48h之后,将MEF固定在丙酮中并进行免疫细胞化学。简言之,在-20℃将于1X冰冷的PBS中冲洗的细胞固定在丙酮中10min,然后在PBS中再冲洗3次。用3%H2O2阻断内源过氧化酶活性,然后在阻断缓冲液(PBST,10%山羊血清和1%BSA)中孵育1h,并在4℃下在DDK特异性抗体(Origene)中过夜孵育。第二天,将细胞在PBST中冲洗25min,并在室温下在HRP缀合的二级抗体中孵育30min。在PBST中冲洗三次之后,将细胞在DAPI溶液(IHC World,Woodstock,MD)中孵育10min,然后覆盖盖玻片并密封载玻片。从Abcam(Cambridge,MA)获得钙联蛋白和giatin抗体。从Life technologies获得二级抗体。
免疫沉淀:根据制造商的说明书(Life technologies)进行293T和COS7细胞转染。用400μl包含完全小蛋白酶抑制剂(Roche Applied Science,Indianapolis,IN)的细胞裂解缓冲液(Cell Signaling,Danvers,MA)处理铺在6孔板中的细胞,将细胞在冰上孵育10min并在冷室中以14,000g离心10min。在冷室中,在振荡平台上将细胞上清液和20μl与抗DDK抗体(Origene)缀合的磁珠孵育过夜。过夜孵育之后,将磁珠/浆体用TBS-T冲洗三次,使用2X SDS-PAGE上样缓冲液收集蛋白质。
SDS-PAGE和免疫印迹:将细胞用冰冷的PBS冲洗一次并用包含完全小蛋白酶抑制剂(Roche Applied Science,Indianapolis,IN)的RIPA缓冲液(Cell Signaling,Danvers,MA)在冰上孵育10min。在4℃将细胞裂解物以14,000g离心10min。收集上清液并贮存于-80℃直至进一步使用。使用Qubit荧光计(Life technologies)对蛋白总量进行定量,并将10μg蛋白上样到4-12%预制凝胶(Lonza,Allendale,NJ)上,在140V下运行直至低分子量Precision plus蛋白标准品(Bio-Rad)到达凝胶底部。使用iBLOT(Life technologies)将已分开的蛋白质通过电泳转移至PVDF膜上,然后在室温下在溶于TBST(20mM Tris、137mM氯化钠和0.1%Tween-20)的5%BSA(Cell Signaling Technology)中封闭1h。然后在4℃将膜在一级抗体(1:1000)中孵育过夜,然后在TBST中冲洗三次(每次15min),然后在室温下在二级抗体(1:2000)中孵育1h。在TBST中再冲洗90min之后,将膜暴露于SuperSignal West Femto Substrate(Fisher Scientific)5min以检测辣根过氧化物酶产生的信号。用肌动蛋白特异性抗体对膜进行预探测。所有的抗体购自Cell Signaling Technology(Danvers,MA)。DDK抗体购自Origene(Rockville,MD)。
原代MEF中的双调蛋白沉默:鼠Areg shRNA克隆TRCN0000089050(在慢病毒载体pLKO.1中)和pLKO.1中的scramble对照shRNA(质粒#1864)分别获得自Sigma Aldrich(St.Louis,MO)和Addgene(Cambridge,MA)。使用质粒pMDLg/pRRE、pRSV-rev和pMD2.g(Addgene)在293T/17细胞(ATCC)中产生慢病毒粒子。在48h和60h时收集慢病毒上清液,合并上清液并通过0.45μm PVDF过滤器过滤。在5μg/ml聚凝胺的存在下,连续两天用慢病毒上清液感染MEF两次,然后用嘌呤霉素(2μg/ml)选择72小时,之后用于下游实验。
息肉计数和肿瘤面积——对从12周龄的被安乐死的小鼠中摘除的肠道进行冲洗并将其固定于10%中性缓冲福尔马林中至少24h,进行息肉计数或组织学检查。使用数字扫描仪NanoZoomer 2.0HT(Hamamatsu,Japan)扫描H&E染色的载片。在解剖显微镜下对息肉直接计数或从扫描的数字图像进行计数。值得注意的是,所述两种方法之间息肉计数无显著差异。还使用NanoZoomer 2.0HT软件从扫描的数字图像测量肿瘤面积。
定量实时PCR(qPCR)——使用 Plus RNA纯化试剂盒(Life Technologies,Chicago,IL),根据制造商的说明书从皮肤分离纯化的RNA。用Agilent 2100生物分析仪测定纯化的RNA的质量。之前已公开了qPCR循环条件。我们从Life Technologies(Chicago,IL)获得以下预设计的TaqMan基因表达测定的列表,表I。
小鼠TNFα和AREG ELISA——对于小鼠TNFαELISA,对8-12周龄的所示基因型的雌性小鼠静脉注射70μg来自大肠杆菌O111:B4的脂多糖(Sigma-Aldrich,St.Louis,MO)。3h之后收集血清并冷冻于-80℃。对于AREG ELISA,将从所有基因型采集的血清立即使用或在-20℃贮存不超过24h。根据制造商的说明书,使用DuoSet ELISA Developmental试剂盒(R&D Systems,Minneapolis,MN)对样品进行测定(DY989和DY410)。
流式细胞术——使用LipofectamineTM LTX试剂,用600ng HuWt或HuCub或Hu Wt p.I186T cDNA瞬时转染铺于6孔板中的293T和COS7细胞。40h之后,在150μg/ml环己酰胺中孵育细胞0、0.5、1、2和4h。之后,在室温下将细胞固定于150μl新鲜制备的冰冷的4%PFA中40min。用2ml PBST冲洗细胞一次并在室温下在黑暗中将细胞重悬于150μl PBST中。再次冲洗后,将细胞重悬于在冰上的包含藻红蛋白标记的抗DDK一级抗体(1:50)的125μl PBST中30min。最后一次冲洗之后,将细胞重悬于FACS缓冲液并进行流式细胞术分析(BD FACSCalibur)。
逆转录酶PCR(RT-PCR)——根据制造商的方案使用
RNAqueousTM-4PCR(Ambion)从MEF中提取RNA。使用逆转录酶(Promega逆转录系统,Promega)从1μg RNA合成cDNA。使用特异性引物以及HotMaster TAQ(5-Prime Inc.)扩增cDNA。
统计学分析——使用GraphPad Prism v4(GraphPad Software,San Diego,CA)产生Kaplan-Meier存活曲线并计算蛋白质半衰期(单相指数衰减方程)。用单因素ANOVA和t-检验测定统计学上的差异。小于0.05P值的被认为是统计学上显著的。
项
1.一种鉴定化合物的方法,所述化合物激活iRhom多肽对底物的蛋白水解活性,所述方法包括:
a)使表达iRhom多肽的细胞与测试化合物接触;以及
b)测定iRhom多肽稳定性相对于合适对照的增加,其中iRhom多肽稳定性的增加表明所述化合物激活iRhom多肽对底物的蛋白水解活性。
2.第1项的方法,其中通过分析暴露于所述化合物之后iRhom多肽的半衰期来测定iRhom多肽稳定性的增加。
3.第1项的方法,其中所述底物是EGFR配体或EGF样底物。
4.第1项的方法,其中通过检测EGFR配体分泌的增加来测定所述iRhom多肽稳定性的增加。
5.第4项的方法,其中所述EGFR配体选自AREG、HB-EGF、TGFα和EPGN。
6.第1项的方法,其中通过检测可溶性EGFR配体水平的提高来测定iRhom多肽稳定性的增加。
7.第6项的方法,其中所述EGFR配体选自AREG、HB-EGF、TGFα和EPGN。
8.第1项的方法,其中通过检测EGFR信号传导活性来测定iRhom多肽稳定性的增加。
9.第1项的方法,其中所述化合物抑制iRhom多肽和蛋白酶体之间的相互作用。
10.第1项的方法,其中所述化合物使iRhom多肽的胞质结构域失活。
11.第10项的方法,其中所述胞质结构域的失活是短暂的。
12.第1项的方法,其中所述化合物使iRhom多肽的胞质结构域裂解和/或缺失,以使所述多肽具有蛋白水解活性或改变的生物活性。
13.第12项的方法,其中小鼠iRhom2在第1和268位氨基酸残基之间被裂解,或人iRhom2在第1和298位氨基酸残基之间被裂解。
14.第12项的方法,其中小鼠iRhom1在第1和272位氨基酸残基之间被裂解,或人iRhom1在第1和316位氨基酸残基之间被裂解。
15.第1项的方法,其中所述化合物激活iRhom多肽的肽酶域。
16.第1-15项中任一项的方法,其中所述iRhom多肽是iRhom1或iRhom2。
17.第15项的方法,其中所述iRhom多肽是人或小鼠iRhom多肽。
18.第1项的方法,其中所述化合物选自小分子、肽或多肽诱饵。
19.第18项的方法,其中所述化合物连接至细胞穿透肽。
20.第1项的方法,其中所述化合物加速角化细胞的迁移。
21.第1项的方法,其中所述化合物加速成纤维细胞的增殖。
22.第1项的方法,其中所述化合物是细胞可透过性的。
23.第1项的方法,其中所述化合物是泛素蛋白酶抑制剂。
24.一种鉴定化合物的方法,所述化合物抑制iRhom多肽对底物的蛋白水解活性,所述方法包括:
a)使表达iRhom多肽的细胞与测试化合物接触;和
b)测定iRhom多肽的生理靶标的分泌相对于合适对照的减少,或EGFR活性相对于合适对照的降低,其中iRhom多肽的生理靶标分泌的减少或EGFR活性的降低表明所述化合物抑制iRhom多肽对底物的蛋白水解活性。
25.第25项的方法,其中所述底物是EGFR配体或EGF样底物。
26.第24项的方法,其中通过检测iRhom多肽的生理靶标分泌的减少来测定对iRhom多肽蛋白水解活性的抑制。
27.第26项的方法,其中所述iRhom多肽的生理靶标是EGFR配体。
28.第27项的方法,其中所述EGFR配体选自AREG、HB-EGF、TGFα和EPGN。
29.第24项的方法,其中通过检测可溶性EGFR配体水平的降低来测定对iRhom多肽蛋白水解活性的抑制。
30.第29项的方法,其中所述EGFR配体选自AREG、HB-EGF、TGFα和EPGN。
31.第24项的方法,其中通过检测EGFR活性的降低来测定对iRhom多肽的蛋白水解活性的抑制。
32.第24项的方法,其中所述化合物抑制iRhom多肽的肽酶域。
33.第24项的方法,其中所述化合物通过使iRhom多肽肽酶域的蛋白水解位点中的一个或多个氨基酸残基失活来影响iRhom多肽的活性。
34.第33项的方法,其中所述iRhom多肽是小鼠iRhom2并且所述化合物使小鼠iRhom2的蛋白水解位点中的一个或多个氨基酸残基失活,所述氨基酸残基选自TM螺旋2上的第635位组氨酸、TM螺旋4上的第695位谷氨酰胺、跨膜螺旋4上的第701位半胱氨酸和TM螺旋6上的第744位组氨酸。
35.第33项的方法,其中所述iRhom多肽是人iRhom2并且所述化合物使人iRhom2的蛋白水解位点中的一个或多个氨基酸残基失活,所述氨基酸残基选自TM螺旋2上的第664位组氨酸、TM螺旋4上的第724位谷氨酰胺、跨膜螺旋4上的第730位半胱氨酸和TM螺旋6上的第773位组氨酸。
36.第24-33项中任一项的方法,其中所述iRhom家族成员为iRhom1或iRhom2。
37.第33项的方法,其中所述iRhom家族成员是人或小鼠iRhom家族成员。
38.第24项的方法,其中所述化合物选自小分子、肽或多肽诱饵。
39.第38项的方法,其中所述化合物连接至细胞穿透肽。
40.第24项的方法,其中所述化合物是细胞可透过性的。
41.一种鉴定化合物的方法,所述化合物能够在受试者中加速伤口愈合或组织修复,所述方法包括:a)使表达iRhom多肽和EGFR配体的细胞与测试化合物接触;b)测定iRhom多肽稳定性相对于合适对照的增加;和c)测定细胞分泌的EGFR配体相对于合适对照的增加,其中iRhom多肽稳定性的增加和细胞分泌的EGFR配体的增加表明所述化合物能够加速伤口愈合。
42.第41项的方法,其中所述iRhom家族成员是iRhom1或iRhom2。
43.第41项的方法,其中所述iRhom家族成员是人或小鼠iRhom家族成员。
44.一种鉴定化合物的方法,所述化合物能够在受试者中降低肿瘤生长和/或进展或治疗癌症,所述方法包括:a)使表达iRhom多肽的细胞与测试化合物接触;和b)测定iRhom多肽的生理靶标的分泌相对于合适对照的减少或EGFR活性相对于合适对照的降低,其中iRhom多肽的生理靶标分泌的减少或EGFR活性的降低表明所述化合物能够在受试者中降低肿瘤生长和/或进展或治疗癌症。
45.第44项的方法,其中所述肿瘤是实体瘤。
46.第44项的方法,其中所述癌症是上皮癌。
47.第44项的方法,其中所述癌症是食管癌、肺癌、脑癌、结肠癌、肾癌、前列腺癌、皮肤癌、肝癌、胰腺癌、胃癌、子宫癌、卵巢癌、乳腺癌、淋巴腺癌或膀胱癌。
48.一种鉴定化合物的方法,所述化合物抑制iRhom多肽的胞质结构域,所述方法包括:a)使表达iRhom多肽的细胞与测试化合物接触;b)测定iRhom多肽稳定性相对于合适对照的增加,其中iRhom多肽稳定性的增加表明所述化合物抑制iRhom多肽的胞质结构域。
49.第44-48项中任一项的方法,其中所述iRhom家族成员是iRhom1或iRhom2。
50.第44-48项中任一项的方法,其中所述iRhom家族成员是人或小鼠iRhom家族成员。
51.第41项的方法,其中所述化合物选自小分子、多肽诱饵、miRNA分子、siRNA分子、shRNA分子、dsRNA分子、反义分子、特异性针对Rhbdf2的核酶;或编码miRNA、siRNA、shRNA、dsRNA的多聚核苷酸;或其各自的生物等价物。
52.一种分离的多肽,其包含变体iRhom多肽。
53.一种分离的多肽,其包含细胞溶质N-末端结构域缺失的人iRhom2。
54.一种分离的多肽,其包含细胞溶质N-末端结构域缺失的小鼠iRhom2。
55.一种分离的小鼠iRhom2多肽,其在小鼠iRhom2的蛋白水解位点中的一个或多个氨基酸残基处包含突变,所述氨基酸残基选自TM螺旋2上的第635位组氨酸、TM螺旋4上的第695位谷氨酰胺、TM螺旋4上的第701位半胱氨酸和TM螺旋6上的第744位组氨酸。
56.一种分离的人iRhom2多肽,其在人iRhom2的蛋白水解位点中的一个或多个氨基酸残基处包含突变,所述氨基酸残基选自TM螺旋2上的第664位组氨酸、TM螺旋4上的第724位谷氨酰胺、TM螺旋4上的第730位半胱氨酸和TM螺旋6上的第773位组氨酸。
57.一种分离的核酸分子,其编码第52-56项中任一项的多肽。
58.一种载体,其包含第57项的核酸分子。
59.一种宿主细胞,其表达第58项的载体。
60.一种鉴定化合物的方法,所述化合物能够在受试者中促进毛发生长,所述方法包括:a)使表达iRhom多肽的细胞与测试化合物接触;和b)测定iRhom多肽的生理靶标的分泌相对于合适对照的减少或EGFR活性相对于合适对照的降低,其中iRhom多肽的生理靶标分泌的减少或EGFR活性的降低表明所述化合物能够在受试者中促进毛发生长。
序列
小鼠iRhom2蛋白序列全长SEQ ID NO:1
MASADKNGSNLPSVSGSRLQSRKPPNLSITIPPPESQAPGEQDSMLPERRKNPAYLKSVSLQEPRGRWQEGAEKRPGFRRQASLSQSIRKSTAQWFGVSGDWEGKRQNWHRRSLHHCSVHYGRLKASCQRELELPSQEVPSFQGTESPKPCKMPKIVDPLARGRAFRHPDEVDRPHAAHPPLTPGVLSLTSFTSVRSGYSHLPRRKRISVAHMSFQAAAALLKGRSVLDATGQRCRHVKRSFAYPSFLEEDAVDGADTFDSSFFSKEEMSSMPDDVFESPPLSASYFRGVPHSASPVSPDGVHIPLKEYSGGRALGPGTQRGKRIASKVKHFAFDRKKRHYGLGVVGNWLNRSYRRSISSTVQRQLESFDSHRPYFTYWLTFVHIIITLLVICTYGIAPVGFAQHVTTQLVLKNRGVYESVKYIQQENFWIGPSSIDLIHLGAKFSPCIRKDQQIEQLVRRERDIERTSGCCVQNDRSGCIQTLKKDCSETLATFVKWQNDTGPSDKSDLSQKQPSAVVCHQDPRTCEEPASSGAHIWPDDITKWPICTEQAQSNHTGLLHIDCKIKGRPCCIGTKGSCEITTREYCEFMHGYFHEDATLCSQVHCLDKVCGLLPFLNPEVPDQFYRIWLSLFLHAGIVHCLVSVVFQMTILRDLEKLAGWHRISIIFILSGITGNLASAIFLPYRAEVGPAGSQFGLLACLFVELFQSWQLLERPWKAFFNLSAIVLFLFICGLLPWIDNIAHIFGFLSGMLLAFAFLPYITFGTSDKYRKRALILVSLLVFAGLFASLVLWLYIYPINWPWIEYLTCFPFTSRFCEKYELDQVLH
Rhbdf2cub小鼠突变SEQ ID NO:2
MSSMPDDVFESPPLSASYFRGVPHSASPVSPDGVHIPLKEYSGGRALGPGTQRGKRIASKVKHFAFDRKKRHYGLGVVGNWLNRSYRRSISSTVQRQLESFDSHRPYFTYWLTFVHIIITLLVICTYGIAPVGFAQHVTTQLVLKNRGVYESVKYIQQENFWIGPSSIDLIHLGAKFSPCIRKDQQIEQLVRRERDIERTSGCCVQNDRSGCIQTLKKDCSETLATFVKWQNDTGPSDKSDLSQKQPSAVVCHQDPRTCEEPASSGAHIWPDDITKWPICTEQAQSNHTGLLHIDCKIKGRPCCIGTKGSCEITTREYCEFMHGYFHEDATLCSQVHCLDKVCGLLPFLNPEVPDQFYRIWLSLFLHAGIVHCLVSVVFQMTILRDLEKLAGWHRISIIFILSGITGNLASAIFLPYRAEVGPAGSQFGLLACLFVELFQSWQLLERPWKAFFNLSAIVLFLFICGLLPWIDNIAHIFGFLSGMLLAFAFLPYITFGTSDKYRKRALILVSLLVFAGLFASLVLWLYIYPINWPWIEYLTCFPFTSRFCEKYELDQVLH
小鼠iRhom1蛋白序列全长SEQ ID NO:3
MSEARRDSTSSLQRKKPPWLKLDIPAAVPPAAEEPSFLQPLRRQAFLRSVSMPAETARVPSPHHEPRRLVLQRQTSITQTIRRGTADWFGVSKDSDSTQKWQRKSIRHCSQRYGKLKPQVIRELDLPSQDNVSLTSTETPPPLYVGPCQLGMQKIIDPLARGRAFRMADDTADGLSAPHTPVTPGAASLCSFSSSRSGFNRLPRRRKRESVAKMSFRAAAALVKGRSIRDGTLRRGQRRSFTPASFLEEDMVDFPDELDTSFFAREGVLHEEMSTYPDEVFESPSEAALKDWEKAPDQADLTGGALDRSELERSHLMLPLERGWRKQKEGGPLAPQPKVRLRQEVVSAAGPRRGQRIAVPVRKLFAREKRPYGLGMVGRLTNRTYRKRIDSYVKRQIEDMDDHRPFFTYWLTFVHSLVTILAVCIYGIAPVGFSQHETVDSVLRKRGVYENVKYVQQENFWIGPSSEALIHLGAKFSPCMRQDPQVHSFILAAREREKHSACCVRNDRSGCVQTSKEECSSTLAVWVKWPVHPSAPDLAGNKRQFGSVCHQDPRVCDEPSSEDPHEWPEDITKWPICTKSSAGNHTNHPHMDCVITGRPCCIGTKGRCEITSREYCDFMRGYFHEEATLCSQVHCMDDVCGLLPFLNPEVPDQFYRLWLSLFLHAGILHCLVSVCFQMTVLRDLEKLAGWHRIAIIYLLSGITGNLASAIFLPYRAEVGPAGSQFGILACLFVELFQSWQILARPWRAFFKLLAVVLFLFAFGLLPWIDNFAHISGFVSGLFLSFAFLPYISFGKFDLYRKRCQIIIFQVVFLGLLAGLVVLFYFYPVRCEWCEFLTCIPFTDKFCEKYELDAQLH
小鼠iRhom1 N-末端截短的蛋白序列(等价于Rhbdf2cub突变——无细胞溶质结构域但保留跨膜肽酶域)SEQ ID NO:4
MSTYPDEVFESPSEAALKDWEKAPDQADLTGGALDRSELERSHLMLPLERGWRKQKEGGPLAPQPKVRLRQEVVSAAGPRRGQRIAVPVRKLFAREKRPYGLGMVGRLTNRTYRKRIDSYVKRQIEDMDDHRPFFTYWLTFVHSLVTILAVCIYGIAPVGFSQHETVDSVLRKRGVYENVKYVQQENFWIGPSSEALIHLGAKFSPCMRQDPQVHSFILAAREREKHSACCVRNDRSGCVQTSKEECSSTLAVWVKWPVHPSAPDLAGNKRQFGSVCHQDPRVCDEPSSEDPHEWPEDITKWPICTKSSAGNHTNHPHMDCVITGRPCCIGTKGRCEITSREYCDFMRGYFHEEATLCSQVHCMDDVCGLLPFLNPEVPDQFYRLWLSLFLHAGILHCLVSVCFQMTVLRDLEKLAGWHRIAIIYLLSGITGNLASAIFLPYRAEVGPAGSQFGILACLFVELFQSWQILARPWRAFFKLLAVVLFLFAFGLLPWIDNFAHISGFVSGLFLSFAFLPYISFGKFDLYRKRCQIIIFQVVFLGLLAGLVVLFYFYPVRCEWCEFLTCIPFTDKFCEKYELDAQLH
人iRhom2蛋白序列全长SEQ ID NO:5
MASADKNGGSVSSVSSSRLQSRKPPNLSITIPPPEKETQAPGEQDSMLPEGFQNRRLKKSQPRTWAAHTTACPPSFLPKRKNPAYLKSVSLQEPRSRWQESSEKRPGFRRQASLSQSIRKGAAQWFGVSGDWEGQRQQWQRRSLHHCSMRYGRLKASCQRDLELPSQEAPSFQGTESPKPCKMPKIVDPLARGRAFRHPEEMDRPHAPHPPLTPGVLSLTSFTSVRSGYSHLPRRKRMSVAHMSLQAAAALLKGRSVLDATGQRCRVVKRSFAFPSFLEEDVVDGADTFDSSFFSKEEMSSMPDDVFESPPLSASYFRGIPHSASPVSPDGVQIPLKEYGRAPVPGPRRGKRIASKVKHFAFDRKKRHYGLGVVGNWLNRSYRRSISSTVQRQLESFDSHRPYFTYWLTFVHVIITLLVICTYGIAPVGFAQHVTTQLVLRNKGVYESVKYIQQENFWVGPSSIDLIHLGAKFSPCIRKDGQIEQLVLRERDLERDSGCCVQNDHSGCIQTQRKDCSETLATFVKWQDDTGPPMDKSDLGQKRTSGAVCHQDPRTCEEPASSGAHIWPDDITKWPICTEQARSNHTGFLHMDCEIKGRPCCIGTKGSCEITTREYCEFMHGYFHEEATLCSQVHCLDKVCGLLPFLNPEVPDQFYRLWLSLFLHAGVVHCLVSVVFQMTILRDLEKLAGWHRIAIIFILSGITGNLASAIFLPYRAEVGPAGSQFGLLACLFVELFQSWPLLERPWKAFLNLSAIVLFLFICGLLPWIDNIAHIFGFLSGLLLAFAFLPYITFGTSDKYRKRALILVSLLAFAGLFAALVLWLYIYPINWPWIEHLTCFPFTSRFCEKYELDQVLH
人截短的iRhom2(等价于Rhbdf2cub突变——无细胞溶质结构域但保留跨膜肽酶域)SEQ ID NO:6
MSSMPDDVFESPPLSASYFRGIPHSASPVSPDGVQIPLKEYGRAPVPGPRRGKRIASKVKHFAFDRKKRHYGLGVVGNWLNRSYRRSISSTVQRQLESFDSHRPYFTYWLTFVHVIITLLVICTYGIAPVGFAQHVTTQLVLRNKGVYESVKYIQQENFWVGPSSIDLIHLGAKFSPCIRKDGQIEQLVLRERDLERDSGCCVQNDHSGCIQTQRKDCSETLATFVKWQDDTGPPMDKSDLGQKRTSGAVCHQDPRTCEEPASSGAHIWPDDITKWPICTEQARSNHTGFLHMDCEIKGRPCCIGTKGSCEITTREYCEFMHGYFHEEATLCSQVHCLDKVCGLLPFLNPEVPDQFYRLWLSLFLHAGVVHCLVSVVFQMTILRDLEKLAGWHRIAIIFILSGITGNLASAIFLPYRAEVGPAGSQFGLLACLFVELFQSWPLLERPWKAFLNLSAIVLFLFICGLLPWIDNIAHIFGFLSGLLLAFAFLPYITFGTSDKYRKRALILVSLLAFAGLFAALVLWLYIYPINWPWIEHLTCFPFTSRFCEKYELDQVLH
人iRhom1蛋白序列全长SEQ ID NO:7
MSEARRDSTSSLQRKKPPWLKLDIPSAVPLTAEEPSFLQPLRRQAFLRSVSMPAETAHISSPHHELRRPVLQRQTSITQTIRRGTADWFGVSKDSDSTQKWQRKSIRHCSQRYGKLKPQVLRELDLPSQDNVSLTSTETPPPLYVGPCQLGMQKIIDPLARGRAFRVADDTAEGLSAPHTPVTPGAASLCSFSSSRSGFHRLPRRRKRESVAKMSFRAAAALMKGRSVRDGTFRRAQRRSFTPASFLEEDTTDFPDELDTSFFAREGILHEELSTYPDEVFESPSEAALKDWEKAPEQAD
LTGGALDRSELERSHLMLPLERGWRKQKEGAAAPQPKVRLRQEVVSTAGPRRGQRIAVPVRKLFAREKRPYGLGMVGRLTNRTYRKRIDSFVKRQIEDMDDHRPFFTYWLTFVHSLVTILAVCIYGIAPVGFSQHETVDSVLRNRGVYENVKYVQQENFWIGPSSEALIHLGAKFSPCMRQDPQVHSFIRSAREREKHSACCVRNDRSGCVQTSEEECSSTLAVWVKWPIHPSAPELAGHKRQFGSVCHQDPRVCDEPSSEDPHEWPEDITKWPICTKNSAGNHTNHPHMDCVITGRPCCIGTKGRCEITSREYCDFMRGYFHEEATLCSQVHCMDDVCGLLPFLNPEVPDQFYRLWLSLFLHAGILHCLVSICFQMTVLRDLEKLAGWHRIAIIYLLSGVTGNLASAIFLPYRAEVGPAGSQFGILACLFVELFQSWQILARPWRAFFKLLAVVLFLFTFGLLPWIDNFAHISGFISGLFLSFAFLPYISFGKFDLYRKRCQIIIFQVVFLGLLAGLVVLFYVYPVRCEWCEFLTCIPFTDKFCEKYELDAQLH
人iRhom1 N-末端截短的蛋白序列(等价于Rhbdf2cub突变——无细胞溶质结构域但保留跨膜肽酶域)SEQ ID NO:8
MLPLERGWRKQKEGAAAPQPKVRLRQEVVSTAGPRRGQRIAVPVRKLFAREKRPYGLGMVGRLTNRTYRKRIDSFVKRQIEDMDDHRPFFTYWLTFVHSLVTILAVCIYGIAPVGFSQHETVDSVLRNRGVYENVKYVQQENFWIGPSSEALIHLGAKFSPCMRQDPQVHSFIRSAREREKHSACCVRNDRSGCVQTSEEECSSTLAVWVKWPIHPSAPELAGHKRQFGSVCHQDPRVCDEPSSEDPHEWPEDITKWPICTKNSAGNHTNHPHMDCVITGRPCCIGTKGRCEITSREYCDFMRGYFHEEATLCSQVHCMDDVCGLLPFLNPEVPDQFYRLWLSLFLHAGILHCLVSICFQMTVLRDLEKLAGWHRIAIIYLLSGVTGNLASAIFLPYRAEVGPAGSQFGILACLFVELFQSWQILARPWRAFFKLLAVVLFLFTFGLLPWIDNFAHISGFISGLFLSFAFLPYISFGKFDLYRKRCQIIIFQVVFLGLLAGLVVLFYVYPVRCEWCEFLTCIPFTDKFCEKYELDAQLH
本领域技术人员会理解或仅使用常规实验即能够确定本文所述的本发明的具体实施方案的许多等同方案。这样的等同方案意欲包含于以下权利要求中。
序列表
<110> The Jackson Laboratory
<120> 鉴定改变iRhom多肽活性的化合物的方法及其用途
<130> JLA-11352/47
<150> 61/991,423
<151> 2014-05-09
<150> 61/992,152
<151> 2014-05-12
<160> 90
<170> PatentIn版本3.5
<210> 1
<211> 827
<212> PRT
<213> 小家鼠(Mus musculus)
<400> 1
Met Ala Ser Ala Asp Lys Asn Gly Ser Asn Leu Pro Ser Val Ser Gly
1 5 10 15
Ser Arg Leu Gln Ser Arg Lys Pro Pro Asn Leu Ser Ile Thr Ile Pro
20 25 30
Pro Pro Glu Ser Gln Ala Pro Gly Glu Gln Asp Ser Met Leu Pro Glu
35 40 45
Arg Arg Lys Asn Pro Ala Tyr Leu Lys Ser Val Ser Leu Gln Glu Pro
50 55 60
Arg Gly Arg Trp Gln Glu Gly Ala Glu Lys Arg Pro Gly Phe Arg Arg
65 70 75 80
Gln Ala Ser Leu Ser Gln Ser Ile Arg Lys Ser Thr Ala Gln Trp Phe
85 90 95
Gly Val Ser Gly Asp Trp Glu Gly Lys Arg Gln Asn Trp His Arg Arg
100 105 110
Ser Leu His His Cys Ser Val His Tyr Gly Arg Leu Lys Ala Ser Cys
115 120 125
Gln Arg Glu Leu Glu Leu Pro Ser Gln Glu Val Pro Ser Phe Gln Gly
130 135 140
Thr Glu Ser Pro Lys Pro Cys Lys Met Pro Lys Ile Val Asp Pro Leu
145 150 155 160
Ala Arg Gly Arg Ala Phe Arg His Pro Asp Glu Val Asp Arg Pro His
165 170 175
Ala Ala His Pro Pro Leu Thr Pro Gly Val Leu Ser Leu Thr Ser Phe
180 185 190
Thr Ser Val Arg Ser Gly Tyr Ser His Leu Pro Arg Arg Lys Arg Ile
195 200 205
Ser Val Ala His Met Ser Phe Gln Ala Ala Ala Ala Leu Leu Lys Gly
210 215 220
Arg Ser Val Leu Asp Ala Thr Gly Gln Arg Cys Arg His Val Lys Arg
225 230 235 240
Ser Phe Ala Tyr Pro Ser Phe Leu Glu Glu Asp Ala Val Asp Gly Ala
245 250 255
Asp Thr Phe Asp Ser Ser Phe Phe Ser Lys Glu Glu Met Ser Ser Met
260 265 270
Pro Asp Asp Val Phe Glu Ser Pro Pro Leu Ser Ala Ser Tyr Phe Arg
275 280 285
Gly Val Pro His Ser Ala Ser Pro Val Ser Pro Asp Gly Val His Ile
290 295 300
Pro Leu Lys Glu Tyr Ser Gly Gly Arg Ala Leu Gly Pro Gly Thr Gln
305 310 315 320
Arg Gly Lys Arg Ile Ala Ser Lys Val Lys His Phe Ala Phe Asp Arg
325 330 335
Lys Lys Arg His Tyr Gly Leu Gly Val Val Gly Asn Trp Leu Asn Arg
340 345 350
Ser Tyr Arg Arg Ser Ile Ser Ser Thr Val Gln Arg Gln Leu Glu Ser
355 360 365
Phe Asp Ser His Arg Pro Tyr Phe Thr Tyr Trp Leu Thr Phe Val His
370 375 380
Ile Ile Ile Thr Leu Leu Val Ile Cys Thr Tyr Gly Ile Ala Pro Val
385 390 395 400
Gly Phe Ala Gln His Val Thr Thr Gln Leu Val Leu Lys Asn Arg Gly
405 410 415
Val Tyr Glu Ser Val Lys Tyr Ile Gln Gln Glu Asn Phe Trp Ile Gly
420 425 430
Pro Ser Ser Ile Asp Leu Ile His Leu Gly Ala Lys Phe Ser Pro Cys
435 440 445
Ile Arg Lys Asp Gln Gln Ile Glu Gln Leu Val Arg Arg Glu Arg Asp
450 455 460
Ile Glu Arg Thr Ser Gly Cys Cys Val Gln Asn Asp Arg Ser Gly Cys
465 470 475 480
Ile Gln Thr Leu Lys Lys Asp Cys Ser Glu Thr Leu Ala Thr Phe Val
485 490 495
Lys Trp Gln Asn Asp Thr Gly Pro Ser Asp Lys Ser Asp Leu Ser Gln
500 505 510
Lys Gln Pro Ser Ala Val Val Cys His Gln Asp Pro Arg Thr Cys Glu
515 520 525
Glu Pro Ala Ser Ser Gly Ala His Ile Trp Pro Asp Asp Ile Thr Lys
530 535 540
Trp Pro Ile Cys Thr Glu Gln Ala Gln Ser Asn His Thr Gly Leu Leu
545 550 555 560
His Ile Asp Cys Lys Ile Lys Gly Arg Pro Cys Cys Ile Gly Thr Lys
565 570 575
Gly Ser Cys Glu Ile Thr Thr Arg Glu Tyr Cys Glu Phe Met His Gly
580 585 590
Tyr Phe His Glu Asp Ala Thr Leu Cys Ser Gln Val His Cys Leu Asp
595 600 605
Lys Val Cys Gly Leu Leu Pro Phe Leu Asn Pro Glu Val Pro Asp Gln
610 615 620
Phe Tyr Arg Ile Trp Leu Ser Leu Phe Leu His Ala Gly Ile Val His
625 630 635 640
Cys Leu Val Ser Val Val Phe Gln Met Thr Ile Leu Arg Asp Leu Glu
645 650 655
Lys Leu Ala Gly Trp His Arg Ile Ser Ile Ile Phe Ile Leu Ser Gly
660 665 670
Ile Thr Gly Asn Leu Ala Ser Ala Ile Phe Leu Pro Tyr Arg Ala Glu
675 680 685
Val Gly Pro Ala Gly Ser Gln Phe Gly Leu Leu Ala Cys Leu Phe Val
690 695 700
Glu Leu Phe Gln Ser Trp Gln Leu Leu Glu Arg Pro Trp Lys Ala Phe
705 710 715 720
Phe Asn Leu Ser Ala Ile Val Leu Phe Leu Phe Ile Cys Gly Leu Leu
725 730 735
Pro Trp Ile Asp Asn Ile Ala His Ile Phe Gly Phe Leu Ser Gly Met
740 745 750
Leu Leu Ala Phe Ala Phe Leu Pro Tyr Ile Thr Phe Gly Thr Ser Asp
755 760 765
Lys Tyr Arg Lys Arg Ala Leu Ile Leu Val Ser Leu Leu Val Phe Ala
770 775 780
Gly Leu Phe Ala Ser Leu Val Leu Trp Leu Tyr Ile Tyr Pro Ile Asn
785 790 795 800
Trp Pro Trp Ile Glu Tyr Leu Thr Cys Phe Pro Phe Thr Ser Arg Phe
805 810 815
Cys Glu Lys Tyr Glu Leu Asp Gln Val Leu His
820 825
<210> 2
<211> 559
<212> PRT
<213> 小家鼠
<400> 2
Met Ser Ser Met Pro Asp Asp Val Phe Glu Ser Pro Pro Leu Ser Ala
1 5 10 15
Ser Tyr Phe Arg Gly Val Pro His Ser Ala Ser Pro Val Ser Pro Asp
20 25 30
Gly Val His Ile Pro Leu Lys Glu Tyr Ser Gly Gly Arg Ala Leu Gly
35 40 45
Pro Gly Thr Gln Arg Gly Lys Arg Ile Ala Ser Lys Val Lys His Phe
50 55 60
Ala Phe Asp Arg Lys Lys Arg His Tyr Gly Leu Gly Val Val Gly Asn
65 70 75 80
Trp Leu Asn Arg Ser Tyr Arg Arg Ser Ile Ser Ser Thr Val Gln Arg
85 90 95
Gln Leu Glu Ser Phe Asp Ser His Arg Pro Tyr Phe Thr Tyr Trp Leu
100 105 110
Thr Phe Val His Ile Ile Ile Thr Leu Leu Val Ile Cys Thr Tyr Gly
115 120 125
Ile Ala Pro Val Gly Phe Ala Gln His Val Thr Thr Gln Leu Val Leu
130 135 140
Lys Asn Arg Gly Val Tyr Glu Ser Val Lys Tyr Ile Gln Gln Glu Asn
145 150 155 160
Phe Trp Ile Gly Pro Ser Ser Ile Asp Leu Ile His Leu Gly Ala Lys
165 170 175
Phe Ser Pro Cys Ile Arg Lys Asp Gln Gln Ile Glu Gln Leu Val Arg
180 185 190
Arg Glu Arg Asp Ile Glu Arg Thr Ser Gly Cys Cys Val Gln Asn Asp
195 200 205
Arg Ser Gly Cys Ile Gln Thr Leu Lys Lys Asp Cys Ser Glu Thr Leu
210 215 220
Ala Thr Phe Val Lys Trp Gln Asn Asp Thr Gly Pro Ser Asp Lys Ser
225 230 235 240
Asp Leu Ser Gln Lys Gln Pro Ser Ala Val Val Cys His Gln Asp Pro
245 250 255
Arg Thr Cys Glu Glu Pro Ala Ser Ser Gly Ala His Ile Trp Pro Asp
260 265 270
Asp Ile Thr Lys Trp Pro Ile Cys Thr Glu Gln Ala Gln Ser Asn His
275 280 285
Thr Gly Leu Leu His Ile Asp Cys Lys Ile Lys Gly Arg Pro Cys Cys
290 295 300
Ile Gly Thr Lys Gly Ser Cys Glu Ile Thr Thr Arg Glu Tyr Cys Glu
305 310 315 320
Phe Met His Gly Tyr Phe His Glu Asp Ala Thr Leu Cys Ser Gln Val
325 330 335
His Cys Leu Asp Lys Val Cys Gly Leu Leu Pro Phe Leu Asn Pro Glu
340 345 350
Val Pro Asp Gln Phe Tyr Arg Ile Trp Leu Ser Leu Phe Leu His Ala
355 360 365
Gly Ile Val His Cys Leu Val Ser Val Val Phe Gln Met Thr Ile Leu
370 375 380
Arg Asp Leu Glu Lys Leu Ala Gly Trp His Arg Ile Ser Ile Ile Phe
385 390 395 400
Ile Leu Ser Gly Ile Thr Gly Asn Leu Ala Ser Ala Ile Phe Leu Pro
405 410 415
Tyr Arg Ala Glu Val Gly Pro Ala Gly Ser Gln Phe Gly Leu Leu Ala
420 425 430
Cys Leu Phe Val Glu Leu Phe Gln Ser Trp Gln Leu Leu Glu Arg Pro
435 440 445
Trp Lys Ala Phe Phe Asn Leu Ser Ala Ile Val Leu Phe Leu Phe Ile
450 455 460
Cys Gly Leu Leu Pro Trp Ile Asp Asn Ile Ala His Ile Phe Gly Phe
465 470 475 480
Leu Ser Gly Met Leu Leu Ala Phe Ala Phe Leu Pro Tyr Ile Thr Phe
485 490 495
Gly Thr Ser Asp Lys Tyr Arg Lys Arg Ala Leu Ile Leu Val Ser Leu
500 505 510
Leu Val Phe Ala Gly Leu Phe Ala Ser Leu Val Leu Trp Leu Tyr Ile
515 520 525
Tyr Pro Ile Asn Trp Pro Trp Ile Glu Tyr Leu Thr Cys Phe Pro Phe
530 535 540
Thr Ser Arg Phe Cys Glu Lys Tyr Glu Leu Asp Gln Val Leu His
545 550 555
<210> 3
<211> 856
<212> PRT
<213> 小家鼠
<400> 3
Met Ser Glu Ala Arg Arg Asp Ser Thr Ser Ser Leu Gln Arg Lys Lys
1 5 10 15
Pro Pro Trp Leu Lys Leu Asp Ile Pro Ala Ala Val Pro Pro Ala Ala
20 25 30
Glu Glu Pro Ser Phe Leu Gln Pro Leu Arg Arg Gln Ala Phe Leu Arg
35 40 45
Ser Val Ser Met Pro Ala Glu Thr Ala Arg Val Pro Ser Pro His His
50 55 60
Glu Pro Arg Arg Leu Val Leu Gln Arg Gln Thr Ser Ile Thr Gln Thr
65 70 75 80
Ile Arg Arg Gly Thr Ala Asp Trp Phe Gly Val Ser Lys Asp Ser Asp
85 90 95
Ser Thr Gln Lys Trp Gln Arg Lys Ser Ile Arg His Cys Ser Gln Arg
100 105 110
Tyr Gly Lys Leu Lys Pro Gln Val Ile Arg Glu Leu Asp Leu Pro Ser
115 120 125
Gln Asp Asn Val Ser Leu Thr Ser Thr Glu Thr Pro Pro Pro Leu Tyr
130 135 140
Val Gly Pro Cys Gln Leu Gly Met Gln Lys Ile Ile Asp Pro Leu Ala
145 150 155 160
Arg Gly Arg Ala Phe Arg Met Ala Asp Asp Thr Ala Asp Gly Leu Ser
165 170 175
Ala Pro His Thr Pro Val Thr Pro Gly Ala Ala Ser Leu Cys Ser Phe
180 185 190
Ser Ser Ser Arg Ser Gly Phe Asn Arg Leu Pro Arg Arg Arg Lys Arg
195 200 205
Glu Ser Val Ala Lys Met Ser Phe Arg Ala Ala Ala Ala Leu Val Lys
210 215 220
Gly Arg Ser Ile Arg Asp Gly Thr Leu Arg Arg Gly Gln Arg Arg Ser
225 230 235 240
Phe Thr Pro Ala Ser Phe Leu Glu Glu Asp Met Val Asp Phe Pro Asp
245 250 255
Glu Leu Asp Thr Ser Phe Phe Ala Arg Glu Gly Val Leu His Glu Glu
260 265 270
Met Ser Thr Tyr Pro Asp Glu Val Phe Glu Ser Pro Ser Glu Ala Ala
275 280 285
Leu Lys Asp Trp Glu Lys Ala Pro Asp Gln Ala Asp Leu Thr Gly Gly
290 295 300
Ala Leu Asp Arg Ser Glu Leu Glu Arg Ser His Leu Met Leu Pro Leu
305 310 315 320
Glu Arg Gly Trp Arg Lys Gln Lys Glu Gly Gly Pro Leu Ala Pro Gln
325 330 335
Pro Lys Val Arg Leu Arg Gln Glu Val Val Ser Ala Ala Gly Pro Arg
340 345 350
Arg Gly Gln Arg Ile Ala Val Pro Val Arg Lys Leu Phe Ala Arg Glu
355 360 365
Lys Arg Pro Tyr Gly Leu Gly Met Val Gly Arg Leu Thr Asn Arg Thr
370 375 380
Tyr Arg Lys Arg Ile Asp Ser Tyr Val Lys Arg Gln Ile Glu Asp Met
385 390 395 400
Asp Asp His Arg Pro Phe Phe Thr Tyr Trp Leu Thr Phe Val His Ser
405 410 415
Leu Val Thr Ile Leu Ala Val Cys Ile Tyr Gly Ile Ala Pro Val Gly
420 425 430
Phe Ser Gln His Glu Thr Val Asp Ser Val Leu Arg Lys Arg Gly Val
435 440 445
Tyr Glu Asn Val Lys Tyr Val Gln Gln Glu Asn Phe Trp Ile Gly Pro
450 455 460
Ser Ser Glu Ala Leu Ile His Leu Gly Ala Lys Phe Ser Pro Cys Met
465 470 475 480
Arg Gln Asp Pro Gln Val His Ser Phe Ile Leu Ala Ala Arg Glu Arg
485 490 495
Glu Lys His Ser Ala Cys Cys Val Arg Asn Asp Arg Ser Gly Cys Val
500 505 510
Gln Thr Ser Lys Glu Glu Cys Ser Ser Thr Leu Ala Val Trp Val Lys
515 520 525
Trp Pro Val His Pro Ser Ala Pro Asp Leu Ala Gly Asn Lys Arg Gln
530 535 540
Phe Gly Ser Val Cys His Gln Asp Pro Arg Val Cys Asp Glu Pro Ser
545 550 555 560
Ser Glu Asp Pro His Glu Trp Pro Glu Asp Ile Thr Lys Trp Pro Ile
565 570 575
Cys Thr Lys Ser Ser Ala Gly Asn His Thr Asn His Pro His Met Asp
580 585 590
Cys Val Ile Thr Gly Arg Pro Cys Cys Ile Gly Thr Lys Gly Arg Cys
595 600 605
Glu Ile Thr Ser Arg Glu Tyr Cys Asp Phe Met Arg Gly Tyr Phe His
610 615 620
Glu Glu Ala Thr Leu Cys Ser Gln Val His Cys Met Asp Asp Val Cys
625 630 635 640
Gly Leu Leu Pro Phe Leu Asn Pro Glu Val Pro Asp Gln Phe Tyr Arg
645 650 655
Leu Trp Leu Ser Leu Phe Leu His Ala Gly Ile Leu His Cys Leu Val
660 665 670
Ser Val Cys Phe Gln Met Thr Val Leu Arg Asp Leu Glu Lys Leu Ala
675 680 685
Gly Trp His Arg Ile Ala Ile Ile Tyr Leu Leu Ser Gly Ile Thr Gly
690 695 700
Asn Leu Ala Ser Ala Ile Phe Leu Pro Tyr Arg Ala Glu Val Gly Pro
705 710 715 720
Ala Gly Ser Gln Phe Gly Ile Leu Ala Cys Leu Phe Val Glu Leu Phe
725 730 735
Gln Ser Trp Gln Ile Leu Ala Arg Pro Trp Arg Ala Phe Phe Lys Leu
740 745 750
Leu Ala Val Val Leu Phe Leu Phe Ala Phe Gly Leu Leu Pro Trp Ile
755 760 765
Asp Asn Phe Ala His Ile Ser Gly Phe Val Ser Gly Leu Phe Leu Ser
770 775 780
Phe Ala Phe Leu Pro Tyr Ile Ser Phe Gly Lys Phe Asp Leu Tyr Arg
785 790 795 800
Lys Arg Cys Gln Ile Ile Ile Phe Gln Val Val Phe Leu Gly Leu Leu
805 810 815
Ala Gly Leu Val Val Leu Phe Tyr Phe Tyr Pro Val Arg Cys Glu Trp
820 825 830
Cys Glu Phe Leu Thr Cys Ile Pro Phe Thr Asp Lys Phe Cys Glu Lys
835 840 845
Tyr Glu Leu Asp Ala Gln Leu His
850 855
<210> 4
<211> 584
<212> PRT
<213> 小家鼠
<400> 4
Met Ser Thr Tyr Pro Asp Glu Val Phe Glu Ser Pro Ser Glu Ala Ala
1 5 10 15
Leu Lys Asp Trp Glu Lys Ala Pro Asp Gln Ala Asp Leu Thr Gly Gly
20 25 30
Ala Leu Asp Arg Ser Glu Leu Glu Arg Ser His Leu Met Leu Pro Leu
35 40 45
Glu Arg Gly Trp Arg Lys Gln Lys Glu Gly Gly Pro Leu Ala Pro Gln
50 55 60
Pro Lys Val Arg Leu Arg Gln Glu Val Val Ser Ala Ala Gly Pro Arg
65 70 75 80
Arg Gly Gln Arg Ile Ala Val Pro Val Arg Lys Leu Phe Ala Arg Glu
85 90 95
Lys Arg Pro Tyr Gly Leu Gly Met Val Gly Arg Leu Thr Asn Arg Thr
100 105 110
Tyr Arg Lys Arg Ile Asp Ser Tyr Val Lys Arg Gln Ile Glu Asp Met
115 120 125
Asp Asp His Arg Pro Phe Phe Thr Tyr Trp Leu Thr Phe Val His Ser
130 135 140
Leu Val Thr Ile Leu Ala Val Cys Ile Tyr Gly Ile Ala Pro Val Gly
145 150 155 160
Phe Ser Gln His Glu Thr Val Asp Ser Val Leu Arg Lys Arg Gly Val
165 170 175
Tyr Glu Asn Val Lys Tyr Val Gln Gln Glu Asn Phe Trp Ile Gly Pro
180 185 190
Ser Ser Glu Ala Leu Ile His Leu Gly Ala Lys Phe Ser Pro Cys Met
195 200 205
Arg Gln Asp Pro Gln Val His Ser Phe Ile Leu Ala Ala Arg Glu Arg
210 215 220
Glu Lys His Ser Ala Cys Cys Val Arg Asn Asp Arg Ser Gly Cys Val
225 230 235 240
Gln Thr Ser Lys Glu Glu Cys Ser Ser Thr Leu Ala Val Trp Val Lys
245 250 255
Trp Pro Val His Pro Ser Ala Pro Asp Leu Ala Gly Asn Lys Arg Gln
260 265 270
Phe Gly Ser Val Cys His Gln Asp Pro Arg Val Cys Asp Glu Pro Ser
275 280 285
Ser Glu Asp Pro His Glu Trp Pro Glu Asp Ile Thr Lys Trp Pro Ile
290 295 300
Cys Thr Lys Ser Ser Ala Gly Asn His Thr Asn His Pro His Met Asp
305 310 315 320
Cys Val Ile Thr Gly Arg Pro Cys Cys Ile Gly Thr Lys Gly Arg Cys
325 330 335
Glu Ile Thr Ser Arg Glu Tyr Cys Asp Phe Met Arg Gly Tyr Phe His
340 345 350
Glu Glu Ala Thr Leu Cys Ser Gln Val His Cys Met Asp Asp Val Cys
355 360 365
Gly Leu Leu Pro Phe Leu Asn Pro Glu Val Pro Asp Gln Phe Tyr Arg
370 375 380
Leu Trp Leu Ser Leu Phe Leu His Ala Gly Ile Leu His Cys Leu Val
385 390 395 400
Ser Val Cys Phe Gln Met Thr Val Leu Arg Asp Leu Glu Lys Leu Ala
405 410 415
Gly Trp His Arg Ile Ala Ile Ile Tyr Leu Leu Ser Gly Ile Thr Gly
420 425 430
Asn Leu Ala Ser Ala Ile Phe Leu Pro Tyr Arg Ala Glu Val Gly Pro
435 440 445
Ala Gly Ser Gln Phe Gly Ile Leu Ala Cys Leu Phe Val Glu Leu Phe
450 455 460
Gln Ser Trp Gln Ile Leu Ala Arg Pro Trp Arg Ala Phe Phe Lys Leu
465 470 475 480
Leu Ala Val Val Leu Phe Leu Phe Ala Phe Gly Leu Leu Pro Trp Ile
485 490 495
Asp Asn Phe Ala His Ile Ser Gly Phe Val Ser Gly Leu Phe Leu Ser
500 505 510
Phe Ala Phe Leu Pro Tyr Ile Ser Phe Gly Lys Phe Asp Leu Tyr Arg
515 520 525
Lys Arg Cys Gln Ile Ile Ile Phe Gln Val Val Phe Leu Gly Leu Leu
530 535 540
Ala Gly Leu Val Val Leu Phe Tyr Phe Tyr Pro Val Arg Cys Glu Trp
545 550 555 560
Cys Glu Phe Leu Thr Cys Ile Pro Phe Thr Asp Lys Phe Cys Glu Lys
565 570 575
Tyr Glu Leu Asp Ala Gln Leu His
580
<210> 5
<211> 856
<212> PRT
<213> 智人(Homo sapiens)
<400> 5
Met Ala Ser Ala Asp Lys Asn Gly Gly Ser Val Ser Ser Val Ser Ser
1 5 10 15
Ser Arg Leu Gln Ser Arg Lys Pro Pro Asn Leu Ser Ile Thr Ile Pro
20 25 30
Pro Pro Glu Lys Glu Thr Gln Ala Pro Gly Glu Gln Asp Ser Met Leu
35 40 45
Pro Glu Gly Phe Gln Asn Arg Arg Leu Lys Lys Ser Gln Pro Arg Thr
50 55 60
Trp Ala Ala His Thr Thr Ala Cys Pro Pro Ser Phe Leu Pro Lys Arg
65 70 75 80
Lys Asn Pro Ala Tyr Leu Lys Ser Val Ser Leu Gln Glu Pro Arg Ser
85 90 95
Arg Trp Gln Glu Ser Ser Glu Lys Arg Pro Gly Phe Arg Arg Gln Ala
100 105 110
Ser Leu Ser Gln Ser Ile Arg Lys Gly Ala Ala Gln Trp Phe Gly Val
115 120 125
Ser Gly Asp Trp Glu Gly Gln Arg Gln Gln Trp Gln Arg Arg Ser Leu
130 135 140
His His Cys Ser Met Arg Tyr Gly Arg Leu Lys Ala Ser Cys Gln Arg
145 150 155 160
Asp Leu Glu Leu Pro Ser Gln Glu Ala Pro Ser Phe Gln Gly Thr Glu
165 170 175
Ser Pro Lys Pro Cys Lys Met Pro Lys Ile Val Asp Pro Leu Ala Arg
180 185 190
Gly Arg Ala Phe Arg His Pro Glu Glu Met Asp Arg Pro His Ala Pro
195 200 205
His Pro Pro Leu Thr Pro Gly Val Leu Ser Leu Thr Ser Phe Thr Ser
210 215 220
Val Arg Ser Gly Tyr Ser His Leu Pro Arg Arg Lys Arg Met Ser Val
225 230 235 240
Ala His Met Ser Leu Gln Ala Ala Ala Ala Leu Leu Lys Gly Arg Ser
245 250 255
Val Leu Asp Ala Thr Gly Gln Arg Cys Arg Val Val Lys Arg Ser Phe
260 265 270
Ala Phe Pro Ser Phe Leu Glu Glu Asp Val Val Asp Gly Ala Asp Thr
275 280 285
Phe Asp Ser Ser Phe Phe Ser Lys Glu Glu Met Ser Ser Met Pro Asp
290 295 300
Asp Val Phe Glu Ser Pro Pro Leu Ser Ala Ser Tyr Phe Arg Gly Ile
305 310 315 320
Pro His Ser Ala Ser Pro Val Ser Pro Asp Gly Val Gln Ile Pro Leu
325 330 335
Lys Glu Tyr Gly Arg Ala Pro Val Pro Gly Pro Arg Arg Gly Lys Arg
340 345 350
Ile Ala Ser Lys Val Lys His Phe Ala Phe Asp Arg Lys Lys Arg His
355 360 365
Tyr Gly Leu Gly Val Val Gly Asn Trp Leu Asn Arg Ser Tyr Arg Arg
370 375 380
Ser Ile Ser Ser Thr Val Gln Arg Gln Leu Glu Ser Phe Asp Ser His
385 390 395 400
Arg Pro Tyr Phe Thr Tyr Trp Leu Thr Phe Val His Val Ile Ile Thr
405 410 415
Leu Leu Val Ile Cys Thr Tyr Gly Ile Ala Pro Val Gly Phe Ala Gln
420 425 430
His Val Thr Thr Gln Leu Val Leu Arg Asn Lys Gly Val Tyr Glu Ser
435 440 445
Val Lys Tyr Ile Gln Gln Glu Asn Phe Trp Val Gly Pro Ser Ser Ile
450 455 460
Asp Leu Ile His Leu Gly Ala Lys Phe Ser Pro Cys Ile Arg Lys Asp
465 470 475 480
Gly Gln Ile Glu Gln Leu Val Leu Arg Glu Arg Asp Leu Glu Arg Asp
485 490 495
Ser Gly Cys Cys Val Gln Asn Asp His Ser Gly Cys Ile Gln Thr Gln
500 505 510
Arg Lys Asp Cys Ser Glu Thr Leu Ala Thr Phe Val Lys Trp Gln Asp
515 520 525
Asp Thr Gly Pro Pro Met Asp Lys Ser Asp Leu Gly Gln Lys Arg Thr
530 535 540
Ser Gly Ala Val Cys His Gln Asp Pro Arg Thr Cys Glu Glu Pro Ala
545 550 555 560
Ser Ser Gly Ala His Ile Trp Pro Asp Asp Ile Thr Lys Trp Pro Ile
565 570 575
Cys Thr Glu Gln Ala Arg Ser Asn His Thr Gly Phe Leu His Met Asp
580 585 590
Cys Glu Ile Lys Gly Arg Pro Cys Cys Ile Gly Thr Lys Gly Ser Cys
595 600 605
Glu Ile Thr Thr Arg Glu Tyr Cys Glu Phe Met His Gly Tyr Phe His
610 615 620
Glu Glu Ala Thr Leu Cys Ser Gln Val His Cys Leu Asp Lys Val Cys
625 630 635 640
Gly Leu Leu Pro Phe Leu Asn Pro Glu Val Pro Asp Gln Phe Tyr Arg
645 650 655
Leu Trp Leu Ser Leu Phe Leu His Ala Gly Val Val His Cys Leu Val
660 665 670
Ser Val Val Phe Gln Met Thr Ile Leu Arg Asp Leu Glu Lys Leu Ala
675 680 685
Gly Trp His Arg Ile Ala Ile Ile Phe Ile Leu Ser Gly Ile Thr Gly
690 695 700
Asn Leu Ala Ser Ala Ile Phe Leu Pro Tyr Arg Ala Glu Val Gly Pro
705 710 715 720
Ala Gly Ser Gln Phe Gly Leu Leu Ala Cys Leu Phe Val Glu Leu Phe
725 730 735
Gln Ser Trp Pro Leu Leu Glu Arg Pro Trp Lys Ala Phe Leu Asn Leu
740 745 750
Ser Ala Ile Val Leu Phe Leu Phe Ile Cys Gly Leu Leu Pro Trp Ile
755 760 765
Asp Asn Ile Ala His Ile Phe Gly Phe Leu Ser Gly Leu Leu Leu Ala
770 775 780
Phe Ala Phe Leu Pro Tyr Ile Thr Phe Gly Thr Ser Asp Lys Tyr Arg
785 790 795 800
Lys Arg Ala Leu Ile Leu Val Ser Leu Leu Ala Phe Ala Gly Leu Phe
805 810 815
Ala Ala Leu Val Leu Trp Leu Tyr Ile Tyr Pro Ile Asn Trp Pro Trp
820 825 830
Ile Glu His Leu Thr Cys Phe Pro Phe Thr Ser Arg Phe Cys Glu Lys
835 840 845
Tyr Glu Leu Asp Gln Val Leu His
850 855
<210> 6
<211> 558
<212> PRT
<213> 智人
<400> 6
Met Ser Ser Met Pro Asp Asp Val Phe Glu Ser Pro Pro Leu Ser Ala
1 5 10 15
Ser Tyr Phe Arg Gly Ile Pro His Ser Ala Ser Pro Val Ser Pro Asp
20 25 30
Gly Val Gln Ile Pro Leu Lys Glu Tyr Gly Arg Ala Pro Val Pro Gly
35 40 45
Pro Arg Arg Gly Lys Arg Ile Ala Ser Lys Val Lys His Phe Ala Phe
50 55 60
Asp Arg Lys Lys Arg His Tyr Gly Leu Gly Val Val Gly Asn Trp Leu
65 70 75 80
Asn Arg Ser Tyr Arg Arg Ser Ile Ser Ser Thr Val Gln Arg Gln Leu
85 90 95
Glu Ser Phe Asp Ser His Arg Pro Tyr Phe Thr Tyr Trp Leu Thr Phe
100 105 110
Val His Val Ile Ile Thr Leu Leu Val Ile Cys Thr Tyr Gly Ile Ala
115 120 125
Pro Val Gly Phe Ala Gln His Val Thr Thr Gln Leu Val Leu Arg Asn
130 135 140
Lys Gly Val Tyr Glu Ser Val Lys Tyr Ile Gln Gln Glu Asn Phe Trp
145 150 155 160
Val Gly Pro Ser Ser Ile Asp Leu Ile His Leu Gly Ala Lys Phe Ser
165 170 175
Pro Cys Ile Arg Lys Asp Gly Gln Ile Glu Gln Leu Val Leu Arg Glu
180 185 190
Arg Asp Leu Glu Arg Asp Ser Gly Cys Cys Val Gln Asn Asp His Ser
195 200 205
Gly Cys Ile Gln Thr Gln Arg Lys Asp Cys Ser Glu Thr Leu Ala Thr
210 215 220
Phe Val Lys Trp Gln Asp Asp Thr Gly Pro Pro Met Asp Lys Ser Asp
225 230 235 240
Leu Gly Gln Lys Arg Thr Ser Gly Ala Val Cys His Gln Asp Pro Arg
245 250 255
Thr Cys Glu Glu Pro Ala Ser Ser Gly Ala His Ile Trp Pro Asp Asp
260 265 270
Ile Thr Lys Trp Pro Ile Cys Thr Glu Gln Ala Arg Ser Asn His Thr
275 280 285
Gly Phe Leu His Met Asp Cys Glu Ile Lys Gly Arg Pro Cys Cys Ile
290 295 300
Gly Thr Lys Gly Ser Cys Glu Ile Thr Thr Arg Glu Tyr Cys Glu Phe
305 310 315 320
Met His Gly Tyr Phe His Glu Glu Ala Thr Leu Cys Ser Gln Val His
325 330 335
Cys Leu Asp Lys Val Cys Gly Leu Leu Pro Phe Leu Asn Pro Glu Val
340 345 350
Pro Asp Gln Phe Tyr Arg Leu Trp Leu Ser Leu Phe Leu His Ala Gly
355 360 365
Val Val His Cys Leu Val Ser Val Val Phe Gln Met Thr Ile Leu Arg
370 375 380
Asp Leu Glu Lys Leu Ala Gly Trp His Arg Ile Ala Ile Ile Phe Ile
385 390 395 400
Leu Ser Gly Ile Thr Gly Asn Leu Ala Ser Ala Ile Phe Leu Pro Tyr
405 410 415
Arg Ala Glu Val Gly Pro Ala Gly Ser Gln Phe Gly Leu Leu Ala Cys
420 425 430
Leu Phe Val Glu Leu Phe Gln Ser Trp Pro Leu Leu Glu Arg Pro Trp
435 440 445
Lys Ala Phe Leu Asn Leu Ser Ala Ile Val Leu Phe Leu Phe Ile Cys
450 455 460
Gly Leu Leu Pro Trp Ile Asp Asn Ile Ala His Ile Phe Gly Phe Leu
465 470 475 480
Ser Gly Leu Leu Leu Ala Phe Ala Phe Leu Pro Tyr Ile Thr Phe Gly
485 490 495
Thr Ser Asp Lys Tyr Arg Lys Arg Ala Leu Ile Leu Val Ser Leu Leu
500 505 510
Ala Phe Ala Gly Leu Phe Ala Ala Leu Val Leu Trp Leu Tyr Ile Tyr
515 520 525
Pro Ile Asn Trp Pro Trp Ile Glu His Leu Thr Cys Phe Pro Phe Thr
530 535 540
Ser Arg Phe Cys Glu Lys Tyr Glu Leu Asp Gln Val Leu His
545 550 555
<210> 7
<211> 855
<212> PRT
<213> 智人
<400> 7
Met Ser Glu Ala Arg Arg Asp Ser Thr Ser Ser Leu Gln Arg Lys Lys
1 5 10 15
Pro Pro Trp Leu Lys Leu Asp Ile Pro Ser Ala Val Pro Leu Thr Ala
20 25 30
Glu Glu Pro Ser Phe Leu Gln Pro Leu Arg Arg Gln Ala Phe Leu Arg
35 40 45
Ser Val Ser Met Pro Ala Glu Thr Ala His Ile Ser Ser Pro His His
50 55 60
Glu Leu Arg Arg Pro Val Leu Gln Arg Gln Thr Ser Ile Thr Gln Thr
65 70 75 80
Ile Arg Arg Gly Thr Ala Asp Trp Phe Gly Val Ser Lys Asp Ser Asp
85 90 95
Ser Thr Gln Lys Trp Gln Arg Lys Ser Ile Arg His Cys Ser Gln Arg
100 105 110
Tyr Gly Lys Leu Lys Pro Gln Val Leu Arg Glu Leu Asp Leu Pro Ser
115 120 125
Gln Asp Asn Val Ser Leu Thr Ser Thr Glu Thr Pro Pro Pro Leu Tyr
130 135 140
Val Gly Pro Cys Gln Leu Gly Met Gln Lys Ile Ile Asp Pro Leu Ala
145 150 155 160
Arg Gly Arg Ala Phe Arg Val Ala Asp Asp Thr Ala Glu Gly Leu Ser
165 170 175
Ala Pro His Thr Pro Val Thr Pro Gly Ala Ala Ser Leu Cys Ser Phe
180 185 190
Ser Ser Ser Arg Ser Gly Phe His Arg Leu Pro Arg Arg Arg Lys Arg
195 200 205
Glu Ser Val Ala Lys Met Ser Phe Arg Ala Ala Ala Ala Leu Met Lys
210 215 220
Gly Arg Ser Val Arg Asp Gly Thr Phe Arg Arg Ala Gln Arg Arg Ser
225 230 235 240
Phe Thr Pro Ala Ser Phe Leu Glu Glu Asp Thr Thr Asp Phe Pro Asp
245 250 255
Glu Leu Asp Thr Ser Phe Phe Ala Arg Glu Gly Ile Leu His Glu Glu
260 265 270
Leu Ser Thr Tyr Pro Asp Glu Val Phe Glu Ser Pro Ser Glu Ala Ala
275 280 285
Leu Lys Asp Trp Glu Lys Ala Pro Glu Gln Ala Asp Leu Thr Gly Gly
290 295 300
Ala Leu Asp Arg Ser Glu Leu Glu Arg Ser His Leu Met Leu Pro Leu
305 310 315 320
Glu Arg Gly Trp Arg Lys Gln Lys Glu Gly Ala Ala Ala Pro Gln Pro
325 330 335
Lys Val Arg Leu Arg Gln Glu Val Val Ser Thr Ala Gly Pro Arg Arg
340 345 350
Gly Gln Arg Ile Ala Val Pro Val Arg Lys Leu Phe Ala Arg Glu Lys
355 360 365
Arg Pro Tyr Gly Leu Gly Met Val Gly Arg Leu Thr Asn Arg Thr Tyr
370 375 380
Arg Lys Arg Ile Asp Ser Phe Val Lys Arg Gln Ile Glu Asp Met Asp
385 390 395 400
Asp His Arg Pro Phe Phe Thr Tyr Trp Leu Thr Phe Val His Ser Leu
405 410 415
Val Thr Ile Leu Ala Val Cys Ile Tyr Gly Ile Ala Pro Val Gly Phe
420 425 430
Ser Gln His Glu Thr Val Asp Ser Val Leu Arg Asn Arg Gly Val Tyr
435 440 445
Glu Asn Val Lys Tyr Val Gln Gln Glu Asn Phe Trp Ile Gly Pro Ser
450 455 460
Ser Glu Ala Leu Ile His Leu Gly Ala Lys Phe Ser Pro Cys Met Arg
465 470 475 480
Gln Asp Pro Gln Val His Ser Phe Ile Arg Ser Ala Arg Glu Arg Glu
485 490 495
Lys His Ser Ala Cys Cys Val Arg Asn Asp Arg Ser Gly Cys Val Gln
500 505 510
Thr Ser Glu Glu Glu Cys Ser Ser Thr Leu Ala Val Trp Val Lys Trp
515 520 525
Pro Ile His Pro Ser Ala Pro Glu Leu Ala Gly His Lys Arg Gln Phe
530 535 540
Gly Ser Val Cys His Gln Asp Pro Arg Val Cys Asp Glu Pro Ser Ser
545 550 555 560
Glu Asp Pro His Glu Trp Pro Glu Asp Ile Thr Lys Trp Pro Ile Cys
565 570 575
Thr Lys Asn Ser Ala Gly Asn His Thr Asn His Pro His Met Asp Cys
580 585 590
Val Ile Thr Gly Arg Pro Cys Cys Ile Gly Thr Lys Gly Arg Cys Glu
595 600 605
Ile Thr Ser Arg Glu Tyr Cys Asp Phe Met Arg Gly Tyr Phe His Glu
610 615 620
Glu Ala Thr Leu Cys Ser Gln Val His Cys Met Asp Asp Val Cys Gly
625 630 635 640
Leu Leu Pro Phe Leu Asn Pro Glu Val Pro Asp Gln Phe Tyr Arg Leu
645 650 655
Trp Leu Ser Leu Phe Leu His Ala Gly Ile Leu His Cys Leu Val Ser
660 665 670
Ile Cys Phe Gln Met Thr Val Leu Arg Asp Leu Glu Lys Leu Ala Gly
675 680 685
Trp His Arg Ile Ala Ile Ile Tyr Leu Leu Ser Gly Val Thr Gly Asn
690 695 700
Leu Ala Ser Ala Ile Phe Leu Pro Tyr Arg Ala Glu Val Gly Pro Ala
705 710 715 720
Gly Ser Gln Phe Gly Ile Leu Ala Cys Leu Phe Val Glu Leu Phe Gln
725 730 735
Ser Trp Gln Ile Leu Ala Arg Pro Trp Arg Ala Phe Phe Lys Leu Leu
740 745 750
Ala Val Val Leu Phe Leu Phe Thr Phe Gly Leu Leu Pro Trp Ile Asp
755 760 765
Asn Phe Ala His Ile Ser Gly Phe Ile Ser Gly Leu Phe Leu Ser Phe
770 775 780
Ala Phe Leu Pro Tyr Ile Ser Phe Gly Lys Phe Asp Leu Tyr Arg Lys
785 790 795 800
Arg Cys Gln Ile Ile Ile Phe Gln Val Val Phe Leu Gly Leu Leu Ala
805 810 815
Gly Leu Val Val Leu Phe Tyr Val Tyr Pro Val Arg Cys Glu Trp Cys
820 825 830
Glu Phe Leu Thr Cys Ile Pro Phe Thr Asp Lys Phe Cys Glu Lys Tyr
835 840 845
Glu Leu Asp Ala Gln Leu His
850 855
<210> 8
<211> 539
<212> PRT
<213> 智人
<400> 8
Met Leu Pro Leu Glu Arg Gly Trp Arg Lys Gln Lys Glu Gly Ala Ala
1 5 10 15
Ala Pro Gln Pro Lys Val Arg Leu Arg Gln Glu Val Val Ser Thr Ala
20 25 30
Gly Pro Arg Arg Gly Gln Arg Ile Ala Val Pro Val Arg Lys Leu Phe
35 40 45
Ala Arg Glu Lys Arg Pro Tyr Gly Leu Gly Met Val Gly Arg Leu Thr
50 55 60
Asn Arg Thr Tyr Arg Lys Arg Ile Asp Ser Phe Val Lys Arg Gln Ile
65 70 75 80
Glu Asp Met Asp Asp His Arg Pro Phe Phe Thr Tyr Trp Leu Thr Phe
85 90 95
Val His Ser Leu Val Thr Ile Leu Ala Val Cys Ile Tyr Gly Ile Ala
100 105 110
Pro Val Gly Phe Ser Gln His Glu Thr Val Asp Ser Val Leu Arg Asn
115 120 125
Arg Gly Val Tyr Glu Asn Val Lys Tyr Val Gln Gln Glu Asn Phe Trp
130 135 140
Ile Gly Pro Ser Ser Glu Ala Leu Ile His Leu Gly Ala Lys Phe Ser
145 150 155 160
Pro Cys Met Arg Gln Asp Pro Gln Val His Ser Phe Ile Arg Ser Ala
165 170 175
Arg Glu Arg Glu Lys His Ser Ala Cys Cys Val Arg Asn Asp Arg Ser
180 185 190
Gly Cys Val Gln Thr Ser Glu Glu Glu Cys Ser Ser Thr Leu Ala Val
195 200 205
Trp Val Lys Trp Pro Ile His Pro Ser Ala Pro Glu Leu Ala Gly His
210 215 220
Lys Arg Gln Phe Gly Ser Val Cys His Gln Asp Pro Arg Val Cys Asp
225 230 235 240
Glu Pro Ser Ser Glu Asp Pro His Glu Trp Pro Glu Asp Ile Thr Lys
245 250 255
Trp Pro Ile Cys Thr Lys Asn Ser Ala Gly Asn His Thr Asn His Pro
260 265 270
His Met Asp Cys Val Ile Thr Gly Arg Pro Cys Cys Ile Gly Thr Lys
275 280 285
Gly Arg Cys Glu Ile Thr Ser Arg Glu Tyr Cys Asp Phe Met Arg Gly
290 295 300
Tyr Phe His Glu Glu Ala Thr Leu Cys Ser Gln Val His Cys Met Asp
305 310 315 320
Asp Val Cys Gly Leu Leu Pro Phe Leu Asn Pro Glu Val Pro Asp Gln
325 330 335
Phe Tyr Arg Leu Trp Leu Ser Leu Phe Leu His Ala Gly Ile Leu His
340 345 350
Cys Leu Val Ser Ile Cys Phe Gln Met Thr Val Leu Arg Asp Leu Glu
355 360 365
Lys Leu Ala Gly Trp His Arg Ile Ala Ile Ile Tyr Leu Leu Ser Gly
370 375 380
Val Thr Gly Asn Leu Ala Ser Ala Ile Phe Leu Pro Tyr Arg Ala Glu
385 390 395 400
Val Gly Pro Ala Gly Ser Gln Phe Gly Ile Leu Ala Cys Leu Phe Val
405 410 415
Glu Leu Phe Gln Ser Trp Gln Ile Leu Ala Arg Pro Trp Arg Ala Phe
420 425 430
Phe Lys Leu Leu Ala Val Val Leu Phe Leu Phe Thr Phe Gly Leu Leu
435 440 445
Pro Trp Ile Asp Asn Phe Ala His Ile Ser Gly Phe Ile Ser Gly Leu
450 455 460
Phe Leu Ser Phe Ala Phe Leu Pro Tyr Ile Ser Phe Gly Lys Phe Asp
465 470 475 480
Leu Tyr Arg Lys Arg Cys Gln Ile Ile Ile Phe Gln Val Val Phe Leu
485 490 495
Gly Leu Leu Ala Gly Leu Val Val Leu Phe Tyr Val Tyr Pro Val Arg
500 505 510
Cys Glu Trp Cys Glu Phe Leu Thr Cys Ile Pro Phe Thr Asp Lys Phe
515 520 525
Cys Glu Lys Tyr Glu Leu Asp Ala Gln Leu His
530 535
<210> 9
<211> 18
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 9
aaaaccctcc acccttcc 18
<210> 10
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 10
cggggaacaa ctctgaatcc 20
<210> 11
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 11
agagccactt tgggtctgtc 20
<210> 12
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 12
acgcaacgtt ctgtcaaaag 20
<210> 13
<211> 27
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 13
tgtattgaac atgtattatg aagttgg 27
<210> 14
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 14
caactgtgct gtgtgcatcc 20
<210> 15
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 15
ccttgtgcct tagcaaatgt 20
<210> 16
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 16
agacacacac atcaacaata ggg 23
<210> 17
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 17
tccatcacca tcaacacaca g 21
<210> 18
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 18
ccctgaaatt ccataaggac ag 22
<210> 19
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 19
tcaatcaagt ccaagtaatg cc 22
<210> 20
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 20
attccacatg ctgttgttcc 20
<210> 21
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 21
tcacacttgg tatccatcgc 20
<210> 22
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 22
aaaagaaata agcccatggt tc 22
<210> 23
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 23
ctagtaagtc ctcgcgtgcc 20
<210> 24
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 24
gtcctatgac aaatgcaccg 20
<210> 25
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 25
aacgaaagtt gctttcctgg 20
<210> 26
<211> 25
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 26
ttcattggca tgtacttata ttgac 25
<210> 27
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 27
gctatacagc cagagtccca g 21
<210> 28
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 28
cagaagaggc gaggaaagtg 20
<210> 29
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 29
ggtttcttat attgcagtgg gg 22
<210> 30
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 30
cgcagattcc actttgttcc 20
<210> 31
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 31
tgtctgggca ttttacataa gc 22
<210> 32
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 32
tgtgccaagc cataattcag 20
<210> 33
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 33
tgacattcct gcgagcttag 20
<210> 34
<211> 19
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 34
caagatctgg ttagcgccc 19
<210> 35
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 35
tctgaagaca aatttgaacc cc 22
<210> 36
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 36
ttccatgggt atggataatg aag 23
<210> 37
<211> 19
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 37
ggtgcccaga ttcaaaatg 19
<210> 38
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 38
tagtgcacca gaatgccttg 20
<210> 39
<211> 19
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 39
aagtgccaag ggaccaatc 19
<210> 40
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 40
taccccaagg ttcttgcatc 20
<210> 41
<211> 24
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 41
gaaaacaggc gttttagata ttgg 24
<210> 42
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 42
cacggagtgg atctactgga c 21
<210> 43
<211> 25
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 43
cactatactg agaaccaaag caacc 25
<210> 44
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 44
aagtgctgtt tatgctcccc 20
<210> 45
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 45
caaatttgtc acgtttggtt c 21
<210> 46
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 46
ccgacactac tttcaagccc 20
<210> 47
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 47
tccagcgggt ctataaaagc 20
<210> 48
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 48
gacgtttggg gaatgtcaag 20
<210> 49
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 49
tgtctacaca gcaatgctca c 21
<210> 50
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 50
ctttgcagag gtaaaacccg 20
<210> 51
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 51
aaaagagaac gtgggcagtc 20
<210> 52
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 52
ccttggttta cacactggaa aag 23
<210> 53
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 53
gtatgtttga gatgcagaaa acc 23
<210> 54
<211> 27
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 54
tgacagtaga gtctgatagg aatactg 27
<210> 55
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 55
gtcacaggag gaaagtcccc 20
<210> 56
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 56
cttcttttgc cttgagataa gtg 23
<210> 57
<211> 27
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 57
tggtggtttt ctataagaat atttagc 27
<210> 58
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 58
aagcccttgt aaatgattgt cc 22
<210> 59
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 59
aaggttcaca ggactccagc 20
<210> 60
<211> 18
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 60
aagtggtgtg cctctccg 18
<210> 61
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 61
aaaaggatta agcatttggg g 21
<210> 62
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 62
tgggtcatga cactaaaagg ac 22
<210> 63
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 63
tttccaatgc tgtcaaaccc 20
<210> 64
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 64
gaatcaatga ataaaacggg g 21
<210> 65
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 65
tgggaagagg agaaaagcaa g 21
<210> 66
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 66
gaaaacagtg ctaattactt ccaaag 26
<210> 67
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 67
attagacatg gtgaagcccc 20
<210> 68
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 68
tttgcatact gacaggaact cac 23
<210> 69
<211> 19
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 69
tgctgagcag acagtcacg 19
<210> 70
<211> 17
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 70
ttgggctcat gtgcgag 17
<210> 71
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 71
tgctttggtc agtcaaccag 20
<210> 72
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 72
ttttcccact cttgtggtcc 20
<210> 73
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 73
ttcccattgg ctgctgtaag 20
<210> 74
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 74
ccaactgaac cacactttgc 20
<210> 75
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 75
tccatgaagt caatcctgaa g 21
<210> 76
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 76
gaggcaggca aatctctgtg 20
<210> 77
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 77
tggttggttg gttggttagg 20
<210> 78
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 78
tgaagcacaa aagccaactc 20
<210> 79
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 79
cggtggaacc aatgagaact 20
<210> 80
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 80
ctcaccctcc tagcatgagc 20
<210> 81
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 81
cggtggaacc aatgagaact 20
<210> 82
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 82
gtcgtagtcc cctgtggaga 20
<210> 83
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 83
cggtggaacc aatgagaact 20
<210> 84
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 84
ttgcctccct ttttcttcct 20
<210> 85
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 85
cggtggaacc aatgagaact 20
<210> 86
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 86
ccacaccgtt caccaaagta 20
<210> 87
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 87
gttgctgcag agaccgagac 20
<210> 88
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 88
gtcgtagtcc cctgtggaga 20
<210> 89
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 89
ctggcagtga actctccaca 20
<210> 90
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> PCR引物
<400> 90
ccacaccgtt caccaaagta 20
- 还没有人留言评论。精彩留言会获得点赞!