修饰肽的展示的制作方法
本申请是申请号为2011800454742、申请日为2011年07月29日、名称为“修饰肽的展示”的发明专利申请的分案申请。
本发明涉及一种展示翻译后修饰肽的展示系统。
背景技术:
在许多情况下,人们利用大型化合物库来筛选和鉴定具有所需活性或特性的化合物,从而寻找新的化合物。组合肽库技术是发现和研制药物的宝贵资源。人们已经证明,在这种筛选中,采用在噬菌体或其它病毒颗粒上展示重组肽库尤其有用。为了探索人类许多疾病新的治疗方法,许多研究团体正在努力从肽库开发具有生物活性的肽。
噬菌体展示肽库技术已经在探索许多人类疾病新的治疗方法中得到了广泛的应用。在丝状噬菌体上展示的肽库已经作为一种筛选资源,用于鉴定与任何给定靶结合,从而显示有药理效用的肽。随后,可以采用传统合成化学方法大量合成出如此鉴定出来的肽。
噬菌体m13是丝状噬菌体科的一种带单链(ss)dna基因组的无包膜丝状大肠杆菌噬菌体。核衣壳由具有不同拷贝数的四种噬菌体蛋白组成:pviii的拷贝数是大约2700,而piii、pvi和pix的拷贝数是5。除t4、t7、fd和λ(lambda)等之外,m13噬菌体也已经成功用于噬菌体展示,在生物技术筛选方法中得到应用。在这种筛选方法中,将随机肽库在噬菌体m13的核衣壳表面上展示,以研究不同噬菌体与结合配体(蛋白质-蛋白质、蛋白质-dna等)的相互作用。通常,合成寡核苷酸被克隆到核衣壳蛋白的编码基因内,从而,将人们感兴趣的肽(或不同肽的库)在噬菌体m13核衣壳的表面上展示,用于后续的结合研究。
噬菌体展示方法通常涉及到将随机寡核苷酸插入到噬菌体基因组内,这样,它们可以引导细菌宿主表达与噬菌体外壳蛋白融合的肽库(如丝状噬菌体piii、pvi或pviii)。这种方法的优点是可以在较小的噬菌体中,处理包含高达1015个不同个体的库,以及展示肽与编码其的遗传信息的物理连锁也是这种技术的一个优点。
基础噬菌体展示技术已经扩展到包括从可复制基因包(如真核病毒、细菌及酵母细胞)展示而不是噬菌体展示的肽库。可复制基因包展示技术的原理和战略与噬菌体非常相似,也就是说,将核酸编码的展示肽插入到可复制基因包的基因组内,在待筛选肽和暴露在细胞或病毒表面的内源性蛋白之间产生融合蛋白。融合蛋白的表达及转运到细胞表面,使得肽在细胞或病毒表面得到展示。
某些团体试图通过肽的二级结构增加库的多样性,通过化学反应得到了构象限制的肽。
ep1187914b1公开了一种结构限制的肽库,包括利用半胱氨酸残基之间的二硫键稳定的多个环肽。
wo2009/098450a2公开了一种噬菌体颗粒,展示了经连接化合物连接的二硫化物稳定的双环肽。
us2009/0137424a1公开了展示多肽的噬菌体的翻译后修饰,所述多肽包含非天然氨基酸,为叠氮-炔烃[3+2]环加成反应和施陶丁格(staudinger)修饰提供靶。
ep2048155a1描述了缩酚酸肽天然化合物的生物合成。采用类atp-grasp酶对5-50个氨基酸之间的前体肽序列进行修饰。例如,在大肠杆菌中引入核酸分子,得到缩酚酸肽天然化合物,如microviridin。
oman等人(naturechemicalbiology6:9-18(2010))对几种核糖体肽的天然产品合成进行了综述。前体肽由前导肽进行翻译后加工得到的。这些前导肽帮助折叠前体肽,稳定前体肽防止其降解,并在宿主内部生物合成期间,保持前体在分泌和蛋白质水解的合适时间之前没有活性。
人们迫切需要利用肽库技术开发新的靶向肽。因此,本发明的目的是提供改进的筛选用结构限制的肽库。
技术实现要素:
通过提供具有权利要求所述特点的实施例,得到解决这种技术问题的方案。于是,本发明通过翻译后修饰(ptm)酶的酶促加工,很容易建立大型的多环肽库。
本发明提供了一种展示肽的可复制基因包,所述肽在氨基酸侧链的两个杂原子之间具有至少一个分子内环键,包括分子内异肽键、酯键或羊毛硫氨酸键。
本发明所述的可复制基因包优选选自由噬菌体颗粒、细菌、酵母、真菌、微生物孢子和核糖体组成的组。
特别地,所述可复制基因包选自由m13噬菌体、t4噬菌体、t7噬菌体、fd噬菌体和λ噬菌体组成的组。
根据本发明具体的一个方面,所述肽是包含前导肽和核心肽的前体肽。
所述前导肽优选包括天然核糖体肽的前导肽。
并且,所述核心肽优选源自天然核糖体肽。
特别地,所述环键连接两个选自由c、n、o和s的原子组成的组。
在一个优选的实施例中,所述环键连接两个选自由c、n、o和s的不同原子组成的组。
根据一个优选实施例,所述肽包括多环结构。
所述肽优选包括一随机序列。
根据本发明的另一个方面,提供了一种制备展示肽的可复制基因包的方法,所述肽在氨基酸侧链中具有至少一个分子内环键,如本发明所述可复制基因包,所述方法包括:
a)提供编码肽的核酸序列;
b)将所述核酸序列连接到可复制基因包的基因内;
c)在所述可复制基因包的表面上展示对应的初级肽序列,得到初级包;及
d)采用翻译后修饰(ptm)酶酶促加工所述初级包,得到展示所述肽环状结构的成熟包。
在一个具体的实施例中,所述方法在氨基酸侧链的两个杂原子之间提供一个分子内环键。
本发明优选的方法进一步包括构建一种肽来阻断肽的剪切。
优选的ptm酶选自由以下酶组成的组:羧酸酯-胺连接酶、环化酶、脱氢酶、脱羧酶、差向异构酶、羟化酶、肽酶、脱水酶、转移酶、酯酶、氧合酶和异构酶,尤其是羊毛硫氨酸键形成酶、溶细胞素形成酶、蓝细菌素(cyanobactin)形成酶、硫肽形成酶、芋螺肽形成酶、microviridin(microviridin)形成酶、环肽形成酶、细菌素形成酶和枯草菌素(subtilosin)形成酶。
根据一个具体的实施例,所述加工在前导肽,优选在前导肽的存在下进行。
根据本发明,进一步提供一种采用本发明所述方法可以获得的或获得的可复制基因包。
根据本发明的另一个方面,提供了一种建立展示肽的可复制基因包的文库的方法,所述肽在氨基酸侧链中具有至少一个分子内环键(如本发明的可复制基因包),所述方法包括:
a)提供编码肽变体的核酸序列集;
b)将所述核酸序列集连接到可复制基因包的基因内;
c)在所述可复制基因包表面上展示对应的初级肽序列,得到初级库;及
d)采用ptm酶对所述初级库进行酶促加工,得到展示所述肽变体环状结构的成熟库。
根据本发明的另一个方面,提供了一可复制基因包的文库,所述文库采用本发明所述方法建立,包括制备所述可复制基因包的方法和建立所述可复制基因包文库的方法。
根据本发明的另一个方面,提供了一可复制基因包的文库,如本发明所述的那些文库,其中所述包展示各种肽序列。
另外,根据本发明的另一个方面,提供了一种采用ptm酶翻译后修饰肽库而得到展示多环肽结构的成熟库的方法。
特别地,采用一酶,所述酶能够在选自c、n、o和s的两个相同或不同原子之间提供共价键。
因此,根据本发明,进一步提供了使用ptm酶加工肽库,以在肽变体中纳入多环结构的用途。
附图说明
图1:序列。与mvde(图1a,seqidno.2)融合的piii基因序列;翻译的氨基酸序列(图1b,seqidno.3);噬菌体蛋白piii,粗体字母指的是mvde前体肽,下划线字母指的是核心肽;
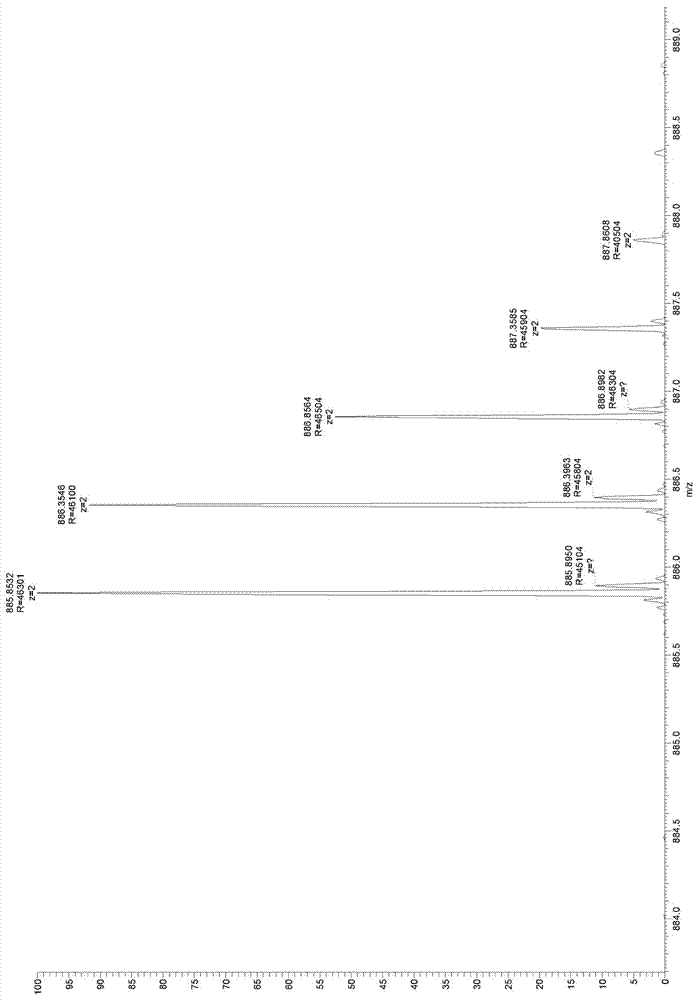
图2:糜蛋白酶处理的参考肽microviridink的高分辨率质谱图(ftms);图中所示质量885.8532da与microviridink的理论质量(885.85019da)相符。
图3:sds-page分离的糜蛋白酶酶切样品带的高分辨率质谱图(ftms);样品指的是ptm酶处理的m13kecmmvde噬菌体;检测得到的双电荷质量m/z885.8540da与肽microviridink的理论质量相符,以及与测得的参考肽microviridink的质量(图1)相符;质量准确度是5ppm。
具体实施方式
因此,本发明涉及一种基于可复制基因包的肽展示系统,所述可复制基因包展示具有特异性分子内环状结构的肽。肽的代谢工程优于化学交联,例如,构建二硫键,因为它利用杂原子作为杂环的桥墩(bridgepiers)。从而,通过代谢处理提供各种肽的初级结构和二级结构作为一个库,以用于专门特异性地选择携带所需结合基序(bindingmotifs)的合适的肽。
此处所使用的词语“可复制基因包”指的是原核或真核基因包,包括细胞、孢子、酵母、细菌、病毒、噬菌体、核糖体和多核糖体。优选的可复制基因包是噬菌体。肽在可复制基因包上展示,即它们与位于可复制基因包外表面上的基团或分子相连。可复制基因包是可筛选的单位,其包括与编码肽的核酸分子相连的待筛选肽。该核酸分子通常可在体内复制,如作为载体,或在体外复制,如通过pcr、转录和翻译进行复制。体内复制可以是自主进行的,如细胞,可以是在宿主因子的帮助下进行的,如病毒,或在宿主和辅助病毒的帮助下进行,如噬粒。在可复制基因包的基因组内引入待展示的编码异源肽的核酸分子,形成与通常表达在可复制基因包外表面的自体蛋白融合的融合蛋白,从而形成展示各种肽的可复制基因包。融合蛋白表达、运输到外表面及组装,从而在可复制基因包的外表面展示肽。根据本发明采用的展示系统通常指的是肽的集合,根据所需特点,如物理特点、化学特点或功能特点进行选择的肽的集合可以进入该展示系统,因此,很容易分离或回收编码所选肽的核酸。这种展示系统优选提供合适的生物系统中的肽的所有组成成分,有时称为生物展示系统,特别是可复制基因包。与体外展示系统相反的是,所述生物系统通常采用病毒或细胞表达系统,如在转化细胞、侵染细胞、转染细胞或转导细胞中表达核酸库,以及在可复制基因包的表面上展示编码肽。
本发明使用的词语“肽”指的是包含5个或更多个氨基酸,通常至少是10个,优选至少是20个,更优选至少是30个,更优选至少是40个,更优选至少是50个,更优选至少是60、70、80、90或100个氨基酸的肽或多肽。词语“肽”还可以指分子量更高的多肽,如蛋白质。
本发明使用的词语“分子内环键”指的是通过分子内异肽键构成的二级结构,所述异肽键利用肽序列中氨基酸侧链之间的共价键,如不包括分子外(外部)结构。该词语特别指的是那些通过翻译后酶加工得到的环和多环,所述酶加工优选不包括化学加工,如二硫键形成,如通过还原反应、环加成或施陶丁格(staudinger)反应。特别地,所述环是包括至少两个“杂原子”的杂环,杂原子是n、o或s等杂原子或在选自由c、n、o和s组成的组中的两个不同原子之间形成共价键的原子。特别是包括c-n、c-o、c-s、n-n、n-o、n-s、o-o、o-s和s-s键,在合适的化学意义上来说,包括双键。此类键合的任何桥墩被称为“杂原子”,并被视为“杂环”桥墩,特别是那些通过翻译后修饰或代谢处理得到的杂环。
此处使用的词语“多环”或“多环结构”指的是至少双环结构,优选是具有至少三个环键,更优选至少四个环键,甚至更优选至少五个环键的结构。根据本发明采用的肽的长度,可以实现更复杂的二级肽结构。
此处使用的词语“前体肽”指的是包括支持肽的翻译后酶加工(成熟)的元素的肽,如操作上,与核心肽连接的信号和/或前导序列。前体肽的羧基端通常编码酶修饰的序列,即“核心序列”。虽然前导物通常是在修饰后从成熟羧基端剪切掉得到较短的肽产物而得到的,但是,本发明展示的优选前体肽,即使在核心肽成熟后,仍然包括前体元素,如信号或前导序列。因此,构建优选的展示系统或构建体,以在成熟加工之前和/或之后阻断核心肽的剪切。通过突变而防止剪切或通过跨剪切点建立合适的桥,可影响剪切。
本发明使用的词语“前导”或“前导序列”指的是翻译后修饰(ptm)酶的识别基序。根据本发明一优选的方面,所述肽采用可复制基因包上整合的前导序列进行展示,例如前体肽的形式,或者前导可作为独立的实体提供,例如,根据本发明作为与可复制基因包无关的独立肽,如添加剂或通过辅助展示系统,所述辅助展示系统以展示前导序列或各种前导序列突变体的生物或体外展示系统为基础,充当ptm酶促作用的支持。
在本发明中,词语“天然核糖体肽”也称为“核糖体肽天然产物”,指的是结构多样、具有生物活性的核糖体合成肽,最常见的长度是大约100个氨基酸,所述肽由各种酶通过催化大量不同化学基序的结构进行翻译后修饰。这一类肽中,有许多带高变序列的(前体)肽。初级肽(primarypeptides)可作为加工途径的底物,因此,每个途径都有许多不同的成熟肽。这一类的成员在微生物学、环境、医学和科技方面具有可能比较重要的作用。通常,天然核糖体肽的前体肽包含相对保守的前导序列,所述序列通过修饰酶和/或输出机器(exportmachinery)至少部分负责识别。这些生物合成机制对于细菌核糖体肽天然产品来说几乎是普遍的,并且在其它生物,如古细菌、真菌、植物和动物的类似肽的生物合成中也常常发现。
根据本发明,优选采用的核糖体肽包括microviridins、乳链球菌素、硫肽(thiopeptides)、芋螺肽、小菌素、溶细胞素、羊毛硫抗生素、蓝细菌素(cyanobactins)、鹅膏毒素/毒伞七肽类(phallotoxins)、环肽和(环)细菌素,或它们功能上相当的变体。
词语“随机化”或“随机序列”指的是预定区域内特异性核苷酸或氨基酸序列修饰,例如,在代谢处理时,或位于改变杂环三维-结构的桥墩之间形成新的杂环桥墩。修饰通常造成氨基酸的随机插入、交换或删除。要取代或插入氨基酸,应采用本领域熟悉的方法及本专利申请中公开的方法,随机或半随机选择氨基酸或使用所有天然氨基酸或合成氨基酸。随机化将得到编码各种肽序列的核酸集。随机化优选采用天然氨基酸。在特定的实施例中,避免使用非天然氨基酸。
此处使用的词语“集”指的是以序列多样性表征的核酸或氨基酸序列集合。集中的各个成员具有共同的特征,如支架内具有共同的核心结构,和/或共同的功能,如特异性结合或生物活性。在集中,通常存在核酸或氨基酸序列的“变体”,如各种肽序列,它们是通过突变方法,如通过随机化技术由母序列衍生得到的。此处使用的词语“库”指的是异质的肽或核酸序列的混合物。所述库由多个成员组成,每个成员具有单个肽或核酸序列。在这方面,“库”是“集”的同义词。库成员之间的序列差异造成库具有多样性。
此处使用的母分子“功能相当的变体”或“功能活性变体”指的是通过插入、删除或替换序列中或序列任一个远端或两个远端中的一个或多个氨基酸或核苷酸,并对该序列进行不会影响(特别是损害)其活性的修饰而所得到的序列。对选定配体具有特异性的配体结合肽的情形,根据本发明所使用的功能活性肽变体将仍然具有预定的结合特异性,虽然这一点可能会改变,如改变对特定抗原表位的精细特异性、亲和性、亲合力、kon或koff率等。在优选的实施例中,功能活性变体a)是肽的生物活性片段,所述功能活性片段包括至少50%所述肽序列,优选至少70%,更优选至少80%,更优选至少90%,甚至更优选至少95%及最优选至少97%、98%或99%;b)是通过至少替换、添加和/或删除肽的一个氨基酸而衍生得到的,其中所述功能活性变体与所述肽的序列一致性是至少50%,优选至少60%,更优选至少70%,更优选至少80%,更优选至少90%,甚至更优选至少95%和最优选至少97%、98%或99%。所述功能活性变体可通过改变肽或核苷酸序列中的序列而得到,其中,当与本发明一起使用时,所述序列改变保留了未变动的肽或核苷酸序列的功能。此种序列改变包括但不限于,(保守)替换、添加、删除、突变和插入。
如果包括变体(但不是原序列)的多肽或核苷酸序列的肽制剂的活性是本发明使用的肽的生物活性的至少10%,优选至少25%,更优选至少50%,甚至更优选至少70%,更优选至少80%,尤其是至少90%,尤其至少95%,最优选至少99%,则称所述变体具有功能活性。其中,本发明使用的肽包括没有发生序列改变的肽或核苷酸序列(即原多肽或原核苷酸序列)。
保守替换是那些在其侧链和化学性质方面相关的氨基酸家族内发生的替换。这些氨基酸家族的实例有具有碱性侧链的氨基酸、具有酸性侧链的氨基酸、具有非极性脂肪族侧链的氨基酸、具有非极性芳香烃铡链的氨基酸、具有不带电极性侧链的氨基酸、具有小侧链的氨基酸及具有大侧链的氨基酸等。
在本发明的另一个实施例中,上文定义的肽或核苷酸序列可以采用各种化学方法进行修饰,得到具有与(上文定义的片段或变体)所述肽活性基本相同,及任选具有其它所需性质的衍生物。
此处使用的“同源物”或“功能同源物”指的是母核酸、肽、多肽或蛋白质的变体,在其初级、二级或三级结构中的对应位点具有相同的或保守的残基。该词语还可扩大到指编码同源肽的两个或多个核苷酸序列。特别是,同源化合物与其全长的天然序列或其任何片段的氨基酸序列的一致性通常是至少大约50%。同源化合物与天然化合物的氨基酸序列一致性,或是与全长化合物任何其它特别定义的片段的氨基酸序列一致性,优选是至少大约55%,更优选是至少大约60%,更优选是至少大约65%,更优选是至少大约70%,更优选是至少大约75%,更优选是至少大约80%,更优选是至少大约85%,更优选是至少大约90%,更优选是至少大约95%。当此类同源物作为生物活性肽的功能得到证明时,所述同源物被称为“功能同源物”。
本发明所述“同源核苷酸序列”指的是在核苷酸序列上与预期的核苷酸序列相关但并不相同,并且基本上执行相同功能的核苷酸序列。这也包括其核苷酸组成的变化,这些变化包括因遗传密码的简并性而出现的变化,从而所述核苷酸序列基本上执行相同的功能。
相对于鉴定的肽序列,此处的“氨基酸序列一致性的百分数(%)”定义为在比对序列和引入缺口后,如果必要的话,为了实现最大的序列一致性,而任何保守取代并不视为序列一致性的一部分时,候选序列中与特定多肽序列中氨基酸残基一致的百分数。本领域技术人员能够确定衡量比对的合适参数,包括待比较序列的全部长度实现最大对齐所需的任何算法。
此处使用的词语“翻译后修饰酶”或“ptm酶”指的是参与翻译肽的结构变化的酶,例如,特别是作为加工机器的一部分,在生物活性肽的生物合成中修饰天然核糖体肽。这类酶包括几种类型的酶,包括羧酸酯-胺连接酶、环化酶、脱氢酶、环化脱水酶、脱羧酶、差向异构酶、羟化酶、肽酶、脱水酶、转移酶、酯酶、氧合酶和异构酶,尤其是羊毛硫氨酸键形成酶、溶细胞素形成酶、蓝细菌素(cyanobactin)形成酶、硫肽形成酶、芋螺肽形成酶、microviridin形成酶、环肽形成酶、细菌素形成酶和枯草菌素(subtilosin)形成酶。
酶的进一步说明如下。
特别地,采用了羊毛硫氨酸键形成酶。willey和vanderdonkannu在rev.microbiol2007年的第61卷第477-501页中(willeyandvanderdonkannu.rev.microbiol.2007.61:477-501),将羊毛硫抗生素定义为含羊毛硫氨酸的抗生素。分子内键被称为羊毛硫氨酸或甲基羊毛硫氨酸键,它们均来自于不同氨基酸侧链的翻译后修饰。丝氨酸和苏氨酸的羟基脱水,分别得到2,3-双脱氢丙氨酸(dha)或(z)-2,3-双脱氢酪氨酸(dhb)。然后,半胱氨酸残基立体定向分子内加成到dha或dhb上,形成羊毛硫氨酸或甲基羊毛硫氨酸键。目前,已知的几种翻译后修饰酶及其基因如下:已经证明,lanb型脱水酶具有催化丝氨酸和苏氨酸脱水步骤的酶的c-端。lanc型环化酶催化半胱氨酸巯基加成。lanc是环化酶的组分,它是一种锌金属蛋白酶,它结合的金属被认为用于活化亲核加成反应的巯底物。lanm型融合脱水酶和环化酶:它的作用是在羊毛硫抗生素合成期间负责前体-肽的脱水和环化。land氧化脱羧酶类型的酶:这种酶从表皮素c-端内消旋-羊毛硫氨酸的半胱氨酸残基催化脱除两个还原当量(reducingequivalents),形成--c==c--双键。lanp类肽酶剪切掉羊毛硫抗生素上的前导肽。lant类肽酶与abc转运体融合;作为分泌过程的一部分,前体肽的剪切通过转运体调节。ltnm和ltnj类脱水酶和脱氢酶参与d-丙氨酸的形成。在肉桂霉素生物合成期间,cinx羟基化天门冬酰胺。
小菌素:(duquesne等人在natprodrep2007年8月第24(4)卷第708-734页(duquesneetal[natprodrep.2007aug;24(4):708-34]))大多数是通过肠细菌产生的,并且分为三类:i类、iia类和iib类。涉及的ptm酶及其基因是:mcbb类丝氨酸和半胱氨酸脱水酶(环化脱水酶)、mcbc类黄素依赖性脱氢酶(氧化还原酶)、参与抗菌素小菌素b17蛋白水解过程的tlde蛋白酶、pmba(tldd)小菌素加工肽酶2类、参与小菌素mccc7/c51生物合成的mccb类修饰酶、n-氨基丙醇基的mccd类转移酶(mccc7/c51)、参与小菌素j25加工和成熟的mcjb和mcjc、参与小菌素e492修饰的mcec类糖基转移酶、mced肠菌素酯酶、mcei乙酰转移酶。
优选采用其它溶细胞素形成酶。链球菌溶血素(mitchell等人在jbiolchem杂志2009年5月8日第284(19)卷第13004-13012页)(mitchelletal,jbiolchem.2009may8;284(19):13004-12)是由肉毒杆菌得到的翻译后修饰肽。具体的ptm酶有:脱氢酶sagb以及丝氨酸和半胱氨酸脱水酶(环化脱水酶)sagc。这两种酶均参与噻唑及(甲基)-恶唑的形成。
蓝细菌素(schmidt等人在美国procnatlacadsci2005年5月17日第102(20)卷第7315-7320页)(schmidtetalprocnatlacadsciusa.2005may17;102(20):7315-20)是从不同蓝藻属中分离得到的含杂环的环状肽。根据优选的实施例,可以采用蓝细菌素形成酶,如剪切前体肽的枯草菌素类丝氨酸蛋白酶肽酶pata、丝氨酸和半胱氨酸脱水酶(环化脱水酶)patd及脱氢酶patg(氧化还原酶)。
硫肽(morris等人在jamchemsoc杂志2009年4月29日第131(16)第5946-5955)(morrisetaljamchemsoc.2009apr29;131(16):5946-552009)是一类含杂环的翻译后修饰肽,在大环连接处具有特征的三-和四取代吡啶环。参与的ptm酶是参与吡啶环形成的tpdb脱水酶,参与吡啶环形成的tpdc脱水酶、参与噻唑啉形成的tpdg半胱氨酸脱水酶(环化脱水酶)、参与噻唑啉至形成噻唑的tpde脱氢酶(氧化还原酶)、tpdh肽酶、tpdi自由基sam蛋白、粪卟啉原iii氧化酶、tpdj1p450单加氧酶、tpdj2p450单加氧酶、参与碳甲基化的tpdl自由基sam蛋白、tpdm氧甲基转移酶、tpdn脱胺还原酶、tpdo环化脱水酶、tpdp脱水酶、tpdqp450单加氧酶、tpdt氮甲基转移酶及tpdu自由基sam蛋白。
优选还采用芋螺肽形成酶。芋螺肽(buczek等人在cell.mol.lifesci.2005年第62卷第3067-3079页)(buczeketalcell.mol.lifesci.62(2005)3067-3079)是一类由芋螺得到的翻译后修饰肽。据估计,此类芋螺肽构成了100,000种不同的肽。具体的ptm酶有:底物特异性内切蛋白酶tex31、特异性蛋白质二硫键异构酶mrpdi、维生素k-依赖性羧化酶(accno:af382823)。生物化学实验表明,下述酶反应参与了芋螺毒素的形成:脯氨酸、缬氨酸、赖氨酸羟基化,通过特异性单加氧酶进行的蛋白质酰胺化反应,通过特异性溴代过氧化物酶进行的色氨酸溴化形成6-溴色氨酸,通过差向异构酶进行的trp、leu、phe、val的差向异构化。此外,参与芋螺毒素生物合成的还有谷氨酰胺酰基环化酶、酪氨酰磺基转移酶及o-糖基转移酶。
鹅膏毒素/毒伞七肽类(walton等人在biopolymers2010年5月26日[epubaheadofprint];hallen等人在美国procnatlacadsci2007年10月27日第104(48)第19097-19101页)(waltonetalbiopolymers.2010may26.[epubaheadofprint],hallenetalprocnatlacadsciusa.2007nov27;104(48):19097-101)是从担子菌纲鹅膏属分离得到的翻译后修饰肽。特别有名的ptm酶是丝氨酸蛋白酶pop1。从生物化学实验可以推导出,环化酶、羟化酶及参与色氨酸-半胱氨酸锥虫胱甘肽(tryptathione)交联的酶参与了鹅膏毒素/毒伞七肽类的形成。
特别优选的是microviridin形成酶。microviridins(philmus等人2008年的文献,见上;ziemert等人在angewchemintedengl2008年第47(40)第7756-7759页)(philmusetal.,2008seeabove,ziemertetal.,angewchemintedengl.2008;47(40):7756-9):microviridins是一类从不同的蓝藻属中分离的三环肽。人们已经对参与microviridins成熟的ptm酶进行了生化特性表征。具体的ptm酶有乙酰转移酶mvdb、参与酰胺键形成且与rimkatp-结合蛋白类似的环化蛋白mvdc、atp抓握连接酶、参与二酯键形成且与rimkatp-结合蛋白和atp抓握连接酶类似的环化蛋白mvdd。
环肽(saska等人在jbiolchem杂志2007年10月5日第282(40)卷第29721-29728页)(saskaetaljbiolchem.2007oct5;282(40):29721-8)是从堇菜科、茜草科和葫芦科植物中分离的一类翻译后修饰肽。表征的ptm酶是参与环肽形成的两种肽酶:nbvpe1a和nbvpe1b。
圆形细菌素(maqueda等人在femsmicrobiolrev2008年1月第32(1)卷第2-22页)(maquedaetalfemsmicrobiolrev.2008jan;32(1):2-22)属于从革兰氏阳性细菌分离得到的一类翻译后修饰肽。虽然,对于负责圆形(圆)细菌素形成的酶进行编码的基因操纵子来说,人们已经对其进行了分离和说明,但是,利用候选酶催化的生化步骤进行鉴定的工作并没有完成。根据生物实验已经推导出,催化头-尾环化的肽酶参与了圆形细菌素的形成。l-丙氨酸向d-丙氨酸的差向异构化由特异性差向异构酶催化。优选的还有枯草菌素形成酶,如fe-s氧化还原酶alba、zn-依赖性肽酶albf和第二zn-依赖性肽酶albe。
优选的还有枯草菌素形成酶,如fe-s氧化还原酶albaap011541.1、zn-依赖性肽酶albfap011541.1和zn-依赖性肽酶albeap011541.1。
在优选的ptm酶中,还有从前导肽剪切肽产物的蛋白酶。通常,ptm酶修饰包含前导序列的前体肽,所述前导序列至少部分负责利用修饰酶和/或输出机器进行识别。然而,前导也可以作为独立的实体(如添加剂)提供,或采用含前导序列的共表达载体共表达。然后,肽和前导可以在同一重组宿主内共表达,并且翻译后修饰所述肽。或者,前导和酶可作为重组宿主细胞培养液的添加剂提供,然后,宿主能够表达成熟的肽。ptm酶通常修饰半胱氨酸、丝氨酸和苏氨酸残基,而且也修饰天门冬酰胺或谷氨酰胺的羧基,例如,形成杂环单元,如通过翻译后加工形成噻唑和恶唑单元。因此,ptm酶可作为单个蛋白质或单个-亚单位酶,以及包含至少两种、三种或四种不同酶的蛋白酶复合物,以支持杂环化。特别地,根据本发明,可以采用参与杂环核糖体肽生物合成的基因或基因簇,包括其功能活性变体或同源物。优选采用天然酶,例如,天然来源酶,如来源于生物体裂解物。特别地,优选采用来源于细菌的酶,如来源于细菌裂解物。或者,可以采用重组酶。根据另一个优选的实施例,采用底物特异性或酶活性被更改而构建的改善的酶或酶变体,优选包括天然来源酶的催化片段。
根据本发明,这是第一次可能提供由展示系统展示的肽的代谢加工,包括生物或体外展示,所述肽如天然核糖体肽或其功能活性变体或同源物。
结果证明,代谢的修饰加工、酶加工可以应用于展示肽的可复制基因包及应用于通常的肽库。这样会导致分子内交联,得到肽的(多)-环架构。线性肽的许多次级代谢产物是初始更大前体肽的衍生物。真核系统中的前体肽可以进一步细分功能上不同的段:在n-端,经加工形成成熟的次级代谢产物的核心肽,以及标明前体肽c-端的任选识别序列紧跟在信号肽和前导肽后面。所述不同的段的预期作用如下:信号肽引导转运至特定的亚细胞区室,信号肽是翻译后修饰(ptm)酶的识别基序。核心肽是经加工而成为成熟的次级代谢产物的段,而识别序列是核心肽段后在c-端的任何延伸。通常,这类次级代谢产物的产生涉及五个步骤。首先,采用核糖体和转运trna翻译前体肽的信使mrna编码。然后,由上述段组成的前体肽,通过翻译后修饰核心段的专门ptm酶识别,导致形成(多)-环架构的分子交联。蛋白酶剪切掉前导肽,从而将前导肽段与核心肽段分离。最后,转运蛋白将成熟的次级代谢产物移动到膜上。根据本发明,特定的突变可以阻断前导物的剪切,对于展示前体肽是有利的。
本发明尤其涉及可复制基因包,其在氨基酸侧链内,特别是在氨基酸侧链的两个杂原子之间含有至少一个分子内环键的肽的展示,以及制备所述基因包的方法,包括:
a)提供编码肽的核酸序列;
b)将所述核酸序列连接到可复制基因包的基因内;
c)在所述可复制基因包的表面上展示对应的初级肽序列,得到初级包;及
d)采用翻译后修饰(ptm)酶酶促加工所述初级包,得到展示所述肽环状结构的成熟包。
本发明的可复制基因包优选以库的成员提供,所述库的成员展示各种肽,亦称为肽库。因此,本发明还提供一种制备单独肽库的方法,所述肽库包含展示各种肽结构的可复制基因包集合。
制备这种库的优选方法包括:
a)提供编码肽变体的核酸序列集;
b)将所述核酸序列集连接到可复制基因包的基因内;
c)在所述可复制基因包表面上展示对应的初级肽序列,得到初级库;及
d)采用ptm酶对所述初级库进行酶促加工,得到展示所述肽变体环状结构的成熟库。
因此,编码各种肽的核酸序列集可以连接到可复制基因包的基因内。
为了将肽锚定到丝状噬菌体表面,采用将大多数基因与噬菌体外壳蛋白基因融合。优选与基因iii、基因viii、基因vi、基因vii和基因ix及它们的片段融合。此外,还在噬菌体λ上实现了噬菌体展示。此时,基因v蛋白、基因j蛋白和基因d蛋白均非常适合用于本发明的目的。除了采用基因融合外,外源肽或蛋白质,包括噬菌体上展示的标签及与待展示肽融合的标签结合配体已经通过相关区域与噬菌体表面连接,以实现非共价展示,还有包含用于共价展示的连接化合物的展示系统。
优选天然核糖体肽作为支架,用于制备在特异性位点具有不同修饰的变体集。母体结构,如核糖体肽支架的变体,优选分组形成肽库,其可以用于根据预定的功能选择库的成员。因此,支架序列优选是随机的,例如,通过突变方法。根据优选的战略,使肽序列中特异性位置发生突变,为杂环杂原子桥提供新的桥墩。或者,突变位置位于现有桥墩旁边,从而,在产生多样性的同时,保持(多)环肽结构。
根据优选的实施例,结合主体(agent)的环状区或末端区,如母体肽序列,优选发生突变或修饰而产生库,所述母体肽序列包括位于一个或多个环或端点内的可能贡献出配体结合点的位置。突变方法优选采用随机、半随机突变,或者尤其是定点随机突变,从而,得到随机序列,尤其是删除、交换或引入随机产生的插入。或者,优选采用组合方法。可以采用任何已知的突变方法,其中包括盒式诱变。在某些情况下,随机选择位置和氨基酸,例如,采用任何可能的氨基酸或选择优选的氨基酸,对序列进行随机化,或者采用简单的规则改变氨基酸。例如,优选对特定氨基酸(如丙氨酸)的所有残基进行突变,称为氨基酸或丙氨酸扫描。为了筛选更高水平的序列多样性,此类方法可与更精密的构建方法联合作为选择法来使用。
任何类型的肽库可采用ptm酶进行代谢加工和成熟化。从而,所述库通过肽的三维限制结构得到改善。这增加了高亲合性和/或高特异性结合剂的机会。
特别地,初级库可来源于作为支架的天然核糖体肽。然而,根据另一个优选的实施例,任何传统的肽库可以采用ptm酶加工。特别地,任何随机肽库的次级代谢产物可以采用体内或体外展示技术制备。
特别地,利用本发明所述ptm酶进行的肽库成熟可应用于许多展示系统,这些展示系统与核酸的编码功能以及肽的物理、化学和/或功能特点联系在一起,例如,生物展示系统,包括病毒,如噬菌体、腺病毒或杆状病毒展示、酵母展示、真菌展示、孢子展示、细菌展示或核糖体展示,而且还可应用于体外展示系统,包括固定肽展示、乳液区室化和展示、质粒展示、共价展示、固相展示、微矩阵展示等。
通常,本发明所述肽库包括至少106个库成员,更优选至少107个库成员,更优选至少108个库成员,更优选至少109个库成员,更优选至少1010个库成员,更优选至少1011个库成员,高达1012个库成员,甚至更高数量的库成员也是可行的。
包含8个单体单元(8-mer)随机肽序列的库的理论多样性是2.6×1010个成员。为了构建这种库,在基因多样性方面,需要1.1×1012个成员的转化子。为了避免构建全面肽库时的实验限制,可以采用限制的氨基酸集用于随机化,或在结构支架内采用随机化。环状结构通常在蛋白质-蛋白质或蛋白质-肽相互作用的分子识别中起着十分重要的作用,因此,根据本发明,优选为肽库制备候选支架,优选采用杂环的环状结构。除一个或多个酶环化预定的桥墩之外,为优选的库设计提供随机化。例如,在优选使用的microviridink内,随机化的优选位置是在一个或多个桥墩位置旁边,即t4、k6、s9、d10、e12和e13旁边。其它氨基酸可视为是必不可少的,因此,如果合适的话,可以选择不进行突变。
根据一个具体的实施例,利用化学加工而包括二级肽结构(如二硫桥)的限制性肽库可作为采用本发明所述ptm酶进行加工的一级肽库使用。因此,采用ptm酶进行肽库的酶促成熟可增加肽库的形态特点(topographiccharacteristics)和多样性。
在适合酶活性的条件下,肽库或肽集通常与一个或多个ptm酶结合。适合ptm酶活性的条件是本领域技术人员熟知的,或者是本领域技术人员很容易确定的。如果需要的话,例如,通过评估系列ph、酶浓度、温度条件下酶的活性,和/或改变允许库或集与酶反应的时间,可以确定或优化合适的条件。例如,在某些实施例中,如源自突变体阿氏浮丝藻(planktothrixagardhii)nivacya126/8(philmus等人在chembiochem(2008年)第3066-3073页)(philmusetal.chembiochem(2008)3066-3073)的裂解产物,酶与肽的比例(摩尔/摩尔比)至少是1:100,000,优选是至少1:10,000,更优选是至少1:1,000,或至少是1:100,或至少是1:10,或高达1:1(酶:肽,摩尔/摩尔)。优选的范围是大约0.01%到大约5%(酶和肽之比,w/w)。在一个实施例中,酶的浓度是至少0.01ng/ml,优选至少0.1ng/ml或至少0.5ng/ml。将混合物在适合酶活性的温度条件下培养,例如,在范围20℃到大约40℃条件下,或在室温,优选培养30分钟至24小时,特别优选培养1小时。
例如,可以使用单种酶,任何所需的不同酶的组合,或任何生物制剂、生物提取物,或具有酶活性的生物均匀混合物。并不一定需要采用已知的酶。可以单独使用或组合使用的酶的合适例子包括,羧酸酯-胺连接酶、环化酶、脱氢酶、环化脱水酶、羊毛硫氨酸键形成酶、脱羧酶、差向异构酶、羟化酶、肽酶、脱水酶、转移酶、酯酶、氧酶和异构酶。优选的ptm酶来源于生物或是纯化的酶制剂。在优选的实施例中,采用细胞裂解物或源自细胞裂解物的酶,如删除了mvde基因而无法得到microviridin的突变体阿氏浮丝藻cya126/8mvde菌株的裂解物(如2010年7月28日在dsmz保藏的菌株阿氏浮丝藻nivacya126/8,保藏号:dsm23872),或可以使用任何其它提供ptm酶的合适的菌株。
本发明优选采用的实例是mvdb(保藏号:acc54548.1)、mvdc(保藏号:acc54549.1)、mvdd(保藏号:acc54550.1)或它们的同源物,如mdnb(保藏号caq16122.1)和mdnc(保藏号caq16123.1)。
具有酶活性的合适的生物提取物、同源物及制剂包括含有机溶剂的含水提取物、裂解物等。
根据本发明具体的实例,克隆mvde基因,将蓝藻弹性蛋白酶抑制剂microviridin的前体肽编码到噬菌体m13genepiii内。然后,使这种突变噬菌体在其宿主大肠杆菌中增殖,对m13突变基因组进行测序,证明存在mvde与基因piii融合。随后,免疫化学分析表明,突变蛋白piii在聚丙烯酰胺凝胶电泳中的迁移速度与野生型piii不同,表明其与野生型piii相比存在物理扩展。这两个实验均证明,microviridin的前体肽mvde在噬菌体m13的表面上展示。microviridin是具有生物活性的次级代谢产物,是由不同的蓝细菌,如浮丝藻属(planktothrixspp.)产生的。它是一种n-端乙酰化的三环肽(14个单体单元),并具有氨基酸序列乙酸-ygntmkypsdweey(seqidno.1)(前体肽mvde保藏号eu438895.1)。
它是核糖体衍生生物合成的,包括采用ptm酶进行翻译后修饰。philmus等人(2008,参考上文)鉴定了负责microviridin生物合成的基因簇。philmus等人(参考上文)提供的突变阿氏浮丝藻cya126/8deltamvde菌株无法通过删除mvde基因得到microviridin。细胞裂解物是从microviridin缺乏菌株得到的,并与m13突变噬菌体颗粒培养。随后,对裂解物处理的突变噬菌体颗粒进行免疫化学分析,结果表明,裂解物处理的piii蛋白的迁移速度不同,表明展示的mvde前体肽存在截短。这一点与核糖体衍生的次级代谢产物的的成熟步骤一致,因为前导片段从核心肽被释放出来。因此,证明了在噬菌体核衣壳上展示的mvde前体肽成功成熟化了。采用质谱测定法,可以证明加工mvde前体肽得到的三环microviridin的乙酰化物理证据,所述mvde前体肽在噬菌体表面展示。采用聚丙烯酰胺凝胶电泳法分离和鉴定裂解物处理和截断的piii-mvde蛋白。将对应的带与凝胶分离之后,提取该蛋白,并用糜蛋白酶酶切,得到能够采用质谱法分析的肽片段。在对分离蛋白质进行质量分析后,检测质量,所得质量与乙酰化三环microviridin的质量相同。这些实验提供了具体的证据,证明了存在在噬菌体表面加工核糖体衍生的翻译后修饰次级代谢产物的可能性。
代谢加工后,可以分析具有合适肽结构的库成员。优选扩大或增加编码选定肽的核酸的拷贝数,以获得足量的核酸或肽,用于又一轮的选择或用于制备另一个库,如用于进一步酶加工,以进一步进行特定的随机化,如用于精化特异性或亲合力成熟的用途。例如,可以采用噬菌体扩增、细胞生长或pcr技术。在一个优选的实施例中,所述展示系统是噬菌体展示,而所述扩增是通过大肠杆菌表达。
采用可以将具有所需活性的肽与不具有所需活性的肽区分开来并进行选择的结合筛选方法或生物活性筛选方法,从本发明的肽库中选择配体结合肽。通常,要将感兴趣的可复制基因包从大量的非结合包中分离出来,需要进行一轮或多轮选择。可以采用合适的肽活性分析测定,选择用于进一步鉴定库成员。例如,可以采用合适的结合分析方法,如elisa、淘选法,评估配体结合的一般功能。例如,通过淘选可以选择和回收结合靶配体的肽。可以采用本领域熟悉的方法进行淘选。
优选地,采用被称为生物淘选的亲和性选择方法完成筛选噬菌体-展示肽库。生物淘选通常包括将靶与肽库一同培养、洗掉未结合的噬菌体、洗脱剩余的结合噬菌体以及扩增洗脱的噬菌体,用于随后的筛选回合。在经过几轮生物淘选后,可以富集靶-噬菌体结合体,并分离单个噬菌体,及进行测序,以显示任何富集的结合基序。
当采用噬菌体展示系统时,优选采用噬菌体elisa测试结合的效果。可以按照任何合适的程序进行噬菌体elisa测试。在一个实例中,为了鉴定展示配体结合肽的噬菌体,通过elisa与选择的配体结合,可以筛选每轮选择得到的噬菌体种群。根据常用的程序,可以测试可溶的肽与配体结合的情况,例如,采用试剂,如针对c-或n-端的标签,采用elisa进行测试。通过pcr产物的凝胶电泳法或通过载体dna的测序,还可以评估选定噬菌体的多样性。
根据靶应用,还可以基于催化或酶抑制活性选择特定的肽,采用酶活性分析方法可以测定其催化或酶抑制活性。根据要求的抗菌、抗真菌或生物活性,如抑制剂或辅因子活性,如采用合适的基于细胞分析方法的酶抑制剂或增强子活性,开展进一步的生物试验来筛选合适的肽。
合适的配体靶优选从微生物的(如细菌的、真菌的、寄生的或病毒的)结构或表位中选择,还可以从人类的或动物的细胞(包括蛋白质,特别是酶、酶的辅因子、受体、生长因子、dna结合蛋白质、核酸、脂类和糖类)的结构或表位中选择。
结合肽或编码肽的dna可以与可复制基因包分离并进行鉴定。然后,根据应用的形式,采用标准分子生物学技术合成或结合前导肽,构建编码肽融合的构建体。采用合适的方法制备肽或肽融合构建体,如采用重组表达技术,如通过重组细菌或酵母细胞表达。
本发明鉴定和提供的肽可作为前导物,用于开发治疗或诊断试剂,或可操作用于靶定独特的分子实体,用于特异性和差异性给药。尤其优选的应用是生物靶的模拟表位领域,如用作抑制剂,如抗菌、抗真菌、抗寄生虫、抗病毒、酶抑制剂和抗生素,或用于开发疫苗。还可以进一步应用于采用配体结合单元的工业、分析或环境应用。
包括本发明所述肽的药物组合物,通常进一步包括至少一种本领域技术人员熟悉的药学上可以接受的赋形剂。所述药物组合物可以进一步包括至少一种其它生物活性剂。合适的试剂是本领域技术人员熟知的。根据本发明,优选的肽组合物可包括稳定分子,如白蛋白或聚乙二醇,或盐。优选地,采用的是那些保持肽的所需生物活性,并且不会造成任何不希望存在的毒性影响的添加剂。
此处描述的实例是对本发明的说明,并不构成对本发明的限制。根据本发明,对本发明的各种实施例进行了描述。对此处描述和阐明的技术的许多改动和变化并不背离本发明的精神和范围。因此,应该理解的是,此处给出的实例仅仅是说明性的,并不会限制本发明的范围。
实例
实例1:microviridin的噬菌体展示
氯霉素乙酰转移酶(cat)基因克隆到噬菌体m13的基因组内
噬菌体m13的基因组dna购自newenglandbiolabs,gmbh。采用限制性内切酶hindiii(fermentasgmbh)与1μgdna在37℃培养1小时。按照生产商的建议,采用克列诺片段(fermentasgmbh)与酶切的dna在37℃培养20分钟。在培养之后,采用qiagengmbh的pcr纯化试剂盒提纯样品。采用nanodroptechnologies,inc的nd-1000分光光度计定量测定纯化的dna。
采用质粒dnapacyc184(newenglandbiolabsgmbh)作为扩增cat基因的模板dna,采用购自mwgeurofinsgmbh)的特异性寡核苷酸cat+和cat-和dna聚合酶(finnzymesoy,遵照生产商的建议,通过聚合酶链式反应(pcr)进行扩增。在pcr终止后,在采用溴化乙锭(sigma-aldrichgmbh)染色的琼脂糖凝胶(0.8%)上负载样品和分离样品。在终止后,通过紫外光使扩增带可视化,并采用凝胶提取试剂盒(qiagengmbh)从凝胶中分离和纯化扩增带。采用nanodroptechnologies,inc的nd-1000分光光度计定量测定纯化的dna。
采用t4dna连接酶(fermentasgmbh)连接等摩尔量的扩增的catdna和噬菌体m13dna。采用genepulser(bioradgmbh),使用预装在该装置上的大肠杆菌标准电击转化程序,对一份等分大肠杆菌k12菌株2738(newenglandbiolabs)样品进行电击转化。全部样品倒(plateout)在加入12.5μg/ml氯霉素(sigma-aldrichgmbh)的lb-琼脂平板上,并在37℃培养一个晚上。在培养后,单菌落接种到50ml液体lb培养液中,其中添加12.5μg/ml氯霉素,并在37℃剧烈摇动下培养一个晚上。在培养后,采用dupont,inc的sorvallrc5b离心分离机对悬浮液离心分离。采用genejet质粒小量提取试剂盒(plasmidminiprepkit)(fermentasgmbh)分离颗粒中的dna。采用特异性寡核苷酸m13cm+和mwgeurofinsgmbh应用的大荧光染料终止剂(bigdyeterminator)技术,通过dna测序证实了cat基因成功地融合到了噬菌体m13的基因组内。
随后,噬菌体被称为m13kecm。dna采用限制性核酸内切酶eco52i和acc65i(fermentasgmbh)进行双酶切。在终止后,采用pcr纯化试剂盒(qiagengmbh)纯化样品,并采用光谱法对dna进行定量。
寡核苷酸mvde+和mvde-用于扩增mvde基因,采用dna聚合酶(finnzymesoy)和来自蓝细菌阿氏浮丝藻(planktothrixagardhii)cya126/8(planktothrixagardhiinivacya126/8,dsmz保藏日期:28.07.2010,dsm23872)的dna模板,遵照生产商的建议利用pcr进行扩增。寡核苷酸mvde+在5`端带有6bp的延伸,产生一个acc65i的框内限制识别序列,而寡核苷酸mvde-在5`端带有6bp延伸,产生一个eco52i的框内限制识别序列。因此,采用寡核苷酸mvde+和mvde-进行pcr得到的扩增子携带了引入的eco52i和acc65i的限制性识别序列。在pcr终止后,在采用溴化乙锭染色的琼脂糖凝胶(3%)上负载样品和分离样品。在终止后,通过紫外光使扩增带可视化,并采用凝胶提取试剂盒(qiagengmbh)从凝胶中分离和纯化。纯化的dna采用nanodroptechnologies,inc的nd-1000分光光度计定量测定。1μgpcr产物用限制性核酸内切酶acc65i和eco52i(fermentasgmbh)进行双酶切。在培养之后,采用pcr纯化试剂盒(qiagengmbh)提纯样品。采用nd-1000分光光度计(nanodroptechnologies,inc)定量测定纯化的dna。
等摩尔量的双酶切的mvdedna和噬菌体m13kecmdna采用t4dna连接酶(fermentasgmbh)连接。采用genepulser(bioradgmbh),使用预装在该装置上的大肠杆菌标准电击转化程序,对一份等分大肠杆菌k12菌株2738(newenglandbiolabsgmbh)样品进行电击转化。全部样品倒在加入了12.5μg/ml氯霉素(sigma-aldrichgmbh)的lb-琼脂平板上,并在37℃培养一个晚上。在培养后,单菌落接种到50ml液体lb培养液中,其中添加12.5μg/ml氯霉素,并在37℃剧烈摇动下培养一个晚上。在培养后,在dupont,inc的sorvallrc5b离心分离机中,在4℃、10.000rpm条件下对悬浮液离心分离10分钟。采用fermentasgmbh的genejet质粒小量提取试剂盒分离颗粒dna。
采用特异性寡核苷酸m13ke+和mwgeurofinsgmbh应用的大荧光染料终止剂(bigdyeterminator)技术,通过dna测序证实了mvde基因成功地框内融合到了噬菌体m13kecm的piii基因内。
序列如图1所示:与mvde融合的piii基因序列,(图1a);噬菌体蛋白piii的翻译氨基酸序列(图1b):粗体字母指的是mvde前体肽,下划线字母指的是核心肽。
噬菌体m13kecmmvde混合(bulk)扩增
随后,噬菌体被称为m13kecmmvde。采用genepulser(bioradgmbh),使用预装在该装置上的大肠杆菌标准电击转化程序,对100ngdna进行大肠杆菌k12菌株2738(newenglandbiolabs)电击转化。全部样品倒到加入了12.5μg/ml氯霉素的lb-琼脂平板上,并在37℃培养一个晚上。在培养后,单菌落接种到3ml液体lb培养液中,其中添加了12.5μg/ml氯霉素,并在37℃剧烈摇动下培养一个晚上。在培养后,300μl悬浮液注种到300ml液体lb中,其中添加了12.5μg/ml氯霉素,剧烈摇动,在37℃培养4.5h。在培养后,悬浮液在dupont,inc的sorvallrc5b离心分离机中于4℃离心分离10分钟,然后,将上清液转移到新的桶(bucket)内,并在相同的条件下再进行离心分离。然后,将上清液(280ml)转移到新的桶内,加入45mlpeg-nacl缓冲液(20%(w/v)聚乙二醇-8000)、2.5mnacl(sigma-aldrichgmbh),剧烈混合,并在4℃培养一个晚上。样品在sorvallrc5b离心分离机(dupont,inc.)中在4℃、10.000rpm条件下离心分离15分钟。离心分离后,完全去掉上清液,将白色沉淀物在2mltbs缓冲液(50mmtris-hclph7.5、150mmnacl,sigma-aldrichgmbh)中重新悬浮,并在4℃贮存。
类似地,得到大量噬菌体m13kecm,并在4℃贮存。
噬菌体颗粒的聚丙烯酰胺凝胶电泳(page)
所有的page分析采用miniproteancell(bioradgmbh)和6%的凝胶强度开展。遵照生产商提供的指南,将总共10μl噬菌体样品m13kecm(阴性对照)和m13kecmmvde上样(load)到凝胶上并进行分离。结束后,遵照生产商建议的半干印迹程序,将凝胶印迹到pvdf膜(bioradgmbh)上。采用newenglandbiolabsgmbh提供的噬菌体m13的piii蛋白的一级抗体,对膜开展免疫化学分析。在膜显影后,可以清晰看到噬菌体m13kecm的piii带和噬菌体m13kecmmvde的piii带之间的大小差别,表明噬菌体m13kecmmvde中的piii出现了大小扩展。
噬菌体m13kecmmvde的裂解物处理
将150ml蓝细菌阿氏浮丝藻cya126/8delmvde(philmus等人在2008年的chembiochem第9(18)卷第3066-3073页)(philmusetal(2008)chembiochem.9(18):3066-73)的突变株离心分离,并用te缓冲液(10mmedta,ph8,1mmtris-hcl,ph8,sigma-aldrichgmbh)洗涤3次。在最后一次离心分离后,将细胞颗粒在1ml20mmtris-hclph8.3中重新悬浮,并新加入β-巯基乙醇,使其最终浓度达到10mm,两种试剂都是购自sigma-aldrichgmbh。接下来的所有步骤均在冰上进行。将悬浮液转移到细胞裂解容器(parrinstrumentgmbh)中,并施加110bar的压力,然后,使悬浮液通过阀门。进行三个这样的循环,完成后,将悬浮液在dupont,inc的sorvallrc5b离心分离机中于10.000rpm、4℃条件下离心分离10分钟。将上清液转移到新管中,并保持在冰上,以供下一步应用。
将100μl噬菌体m13kecmmvde悬浮液与500μl上述裂解物混合,并在22℃培养1小时。结束后,采用1/6体积的peg/nacl缓冲液处理悬浮液,并在4℃培养1个晚上。样品在dupont,inc的sorvallrc5b离心分离机中于10.000rpm、4℃条件下离心分离15分钟。离心分离后,将上清液全部去掉,并将绿色沉淀物在1mltbs缓冲溶液(50mmtris-hclph7.5,150mmnacl)中重新悬浮,并于4℃贮存。在上述步骤后,进行第二次洗涤步骤。最后,将颗粒在40μltbs中重新悬浮。遵照生产商提供的指南,将总共30μl未进行裂解物处理的噬菌体样品m13kecmmvde(阴性对照)和经裂解物处理的m13kecmmvde负载到凝胶上并进行分离。结束后,遵照生产商建议的半干印迹程序,将凝胶印迹到pvdf膜(bioradgmbh)上。采用newenglandbiolabsgmbh的噬菌体m13的piii蛋白一级抗体,对膜开展免疫化学分析。在膜显影后,可以清晰看到未采用裂解物处理的噬菌体m13kecmmvde的piii带和采用裂解物处理的噬菌体m13kecmmvde的piii带之间的大小差别,表明噬菌体m13kecmmvde的piii大小被截短。对这种大小差别的诠释是,在噬菌体衣壳上展示的前体肽成功进行翻译后修饰后,其差别应该是短了35个氨基酸。
按照上文所述,重复sdspage,而凝胶用于考马斯亮染色(bioradgmbh)。裂解物处理的噬菌体m13kecmmvde的piii蛋白带被分离并用于质谱分析。由proteomfactorygmbh进行质谱分析。加入100mmtris-hcl(sigmaaldrich)洗涤蛋白带20分钟,倒掉,然后加入100mmtris-hcl、10mmcacl2(sigmaaldrich)、80%乙腈(vwrinternational)(v/v),ph为8.0,洗涤蛋白带20分钟。重复洗涤步骤两次之后,去掉上清液,200ng糜蛋白酶加入到20μl100mmtris-hcl、10mmcacl2、5%acn(v/v),ph为8.0中,然后加到蛋白带中在25℃酶解一个晚上。采用1体积2%的甲酸酸化溶液,并培养30分钟。采用kosol等(2009年eur.j.phycol第44卷第49-62页)(2009eur.j.phycol.44:49-62)描述的梯度,通过高效液相色谱法仪(hewlett&packardinc.)将microviridink纯物质(philmus等2008年,参考上文)(philmusetal2008,ref.above)从菌株阿氏浮丝藻cya126/8中分离出来。
大约10ng纯肽与200ng糜蛋白酶在20μl100mmtris-hcl、10mmcacl2、5%乙腈(v/v),ph8.0的溶液中于25℃培养一个晚上。培养后,采用1体积2%的甲酸酸化溶液,并培养30分钟。对20μl的两种样品进行高分辨率nanolc-esi-ms(thermoft-icrms)分析,其中采用50分钟内的nanolc梯度仪(agilent1100nanolc系统),使用溶剂a(0.1%甲酸/5%乙腈/94.9%ddh2o)和溶剂b(甲酸/99.9%乙腈),程序从5%b开始梯度增加至40%b。ms数据的质量准确度是优于5ppm,msms数据的准确度优于0.5da。利用分析的蛋白质的氨基酸序列自定义数据库,通过mascotserver对ms数据进行分析。
理论质量计算:microviridink的单一同位素质量是1771.70038da(philmus等人,2008年)(philmusetal2008),结果双电荷质量m/z为885.85019da。
结果如图2(参照)和图3(m13kecmmvde)所示。
说明:
采用hplc分离乙酰化三环肽microviridink,并且糜蛋白酶处理大约10ng纯肽,然后采用ftms测定。检测到的质量是885.8532da,与microviridink的理论计算质量值885.85019da非常相符。因此,采用糜蛋白酶处理纯microviridin并不会剪切肽,这一点与microviridink的环状性质相符。
第二次ftms测定中所用的样品代表的是分离的噬菌体m13kecmmvde的piii蛋白。噬菌体m13kecmmvde展示n-端与mvde前体蛋白融合的piii蛋白,而所述噬菌体采用阿氏浮丝藻nivacya126/8delmvde的细胞裂解物处理。这样处理的噬菌体m13kecmmvde用于sdspage分离,并将代表修饰的piii-mvde蛋白的蛋白带从凝胶中分离和提取出来。通过裂解物中的ptm酶成功加工展示的piii-mvde之后,产生的三环microviridink预计与piii蛋白的n-端相连。microviridink的c-端氨基酸是酪氨酸。由于糜蛋白酶是一种蛋白水解酶,其优先剪切酰胺键,其中所述酰胺键的羧基端是酪氨酸,因此,它在microviridink后被准确地剪切,并将其从噬菌体蛋白piii中释放出来。事实上,图3所示质谱表明,在5ppm的质量准确度范围之内,质量与图2所示参考质量相同。这一点证明了采用包含在阿氏浮丝藻nivacya126/8delmvde裂解物中的ptm酶,展示的前体肽mvde被ptm酶完整地翻译后修饰,同时,连接和展示到噬菌体m13kecmmvde上。
此处的数据表明,可以产生翻译后修饰的肽(microviridin)成员,同时,该成员与病毒的核衣壳蛋白物理连接。
实例2:创建肽库
microviridink是带有氨基酸序列y1g2n3t4m5k6y7p8s9d10w11e12e13y14(参见seqidno.1)的三环肽,其中参与分子内交联的氨基酸残基是t4、k6、s9、d10、e12和e13。因此,位置1、2、3、5、7、8、11、14被认为是可以变化的,优选用于随机化,而不干扰桥的数量和桥的模式。位置1、2、3、5、7、8、11、14处的氨基酸残基的随机化是通过加工(elaboration)固相合成(mwgeurofinsgmbh)得到的寡核苷酸混合物完成的。
寡核苷酸序列如下:(nnk)n.act.nnk.aag.nnk.nnk.tcc.gat.nnk.gaa.gaa.(nnk)n(seqidno.4)。通常利用还原的遗传密码,通过随机化文库寡核苷酸dna,将多样性引入到噬菌体展示的肽库中。例如,在文库dna构建中,采用简并密码子nnk(其中n是腺嘌呤、胸腺嘧啶、鸟嘌呤和胞嘧啶核苷酸混合中的25%;k是胸腺嘧啶和鸟嘌呤核苷酸混合中的50%)。三联体,如编码t4可能是acc、acg、aca。k6以及在其它位置9、10、12和13可能采用同样的战略,k6可采用另外的三联体aaa。此外,通过加入(nnk)n三联体(n=gatc,k=gt,n是0-100之间的整数),加工5`端和3`端的不同三联体数量的延伸。遵照建立的操作规程(如sambrockandrussel2001,molecularcloning,3rdeditioncshlpress),将寡核苷酸融合到piii的基因编码内。噬菌体上展示的前体mvde肽的翻译后修饰还可以采用纯化的酶完成,如mvdb(保藏号:acc54548.1)、mvdc(保藏号:acc54549.1)、mvdd(保藏号:acc54550.1)或它们的同源物,如mdnb(保藏号caq16122.1)和mdnc(保藏号caq16123.1)。
oman和vanderdonk(2010年)(natchembiol.的2010年1月版第6(1)卷第9-18页)(natchembiol.2010jan;6(1):9-18)中描述的翻译后修饰肽主要可以细分为两类:一类是由microviridin、乳链球菌素、硫链丝菌肽、芋螺毒素等成员构成的,它们的共同特征是,前体肽的c-端氨基酸也是完全加工的核心肽的c-端氨基酸。第二类的成员,如海洋环肽(patellamides)、鹅膏菌素等,具有共同的特征,它们都是前体肽蛋白水解酶切得到的。对于提到的第一类成员,可以将其前体肽与病毒的核衣壳蛋白连接,并使用生产者生物裂解物或采用专用纯化ptm酶对其进行处理后,进行展示,并且使其与病毒连接,因为在核心肽与前体肽的c-端氨基酸分离时,它们的生物合成并不涉及这一步骤。因此,在其生物合成的所有步骤期间,对于所有一类ptm肽的代表来说,病毒表面上展示的遗传信息和结构之间的物理连锁均得到维持。
sequencelisting
<110>g·克里斯蒂安森
<120>修饰肽的展示
<130>ch001ep
<160>4
<170>patentinversion3.3
<210>1
<211>14
<212>prt
<213>浮游蓝丝藻属(planktothrix)
<400>1
tyrglyasnthrmetlystyrproserasptrpgluglutyr
1510
<210>2
<211>764
<212>dna
<213>人工序列
<220>
<223>修饰的噬菌体
<400>2
aaacgggttagatctcccctcgaaaagcaagctgataaaccgatacaattaaaggctcct60
tttggagccttttttttggagattttcaacgtgaaaaaattattattcgcaattccttta120
gtggaacctttctattctcactcgatgtccaaaaatgttaaagtctctgcgcccaaagca180
gtccccttctttgctcgtttcttagcagagcaagccgtggaagctaacaattctaattct240
gctccttacggtaatactatgaagtatccttccgattgggaagaatattcggccgaaact300
gttgaaagttgtttagcaaaatcccatacagaaaattcatttattaacgtctggaaagac360
gacaaaactttagatcgttacgctaactatgagggctgtctgtggaatgctacaggcgtt420
gtagtttgtactggtgacgcaactcagtgttacggtacatgggttcctattgggcttgct480
atccctgaaaatgagggtggtggctctgagggtggcggttctgagggtggaggttctgag540
ggtggcggtactaaacatcctgactacggtgatacacctatcccgggctatactttatca600
accctctcgacggcacgtatcctcctggtactgagcaaaacctcgctaatctaatccttc660
tcttgaggagtctcagcctcttaatactttcatgtttcagaataatatgttccacaaata720
ggcagggggcttaaactgtttatacggtatcctgtttactccaa764
<210>3
<211>244
<212>prt
<213>人工序列
<220>
<223>修饰的噬菌体
<400>3
lysargvalargserproleuglulysglnalaasplysproilegln
151015
leulysalapropheglyalaphepheleugluilepheasnvallys
202530
lysleuleuphealaileproleuvalgluprophetyrserhisser
354045
metserlysasnvallysvalseralaprolysalavalprophephe
505560
alaargpheleualagluglnalavalglualaasnasnserasnser
65707580
alaprotyrglyasnthrmetlystyrproserasptrpgluglutyr
859095
seralagluthrvalglusercysleualalysserhisthrgluasn
100105110
serpheileasnvaltrplysaspasplysthrleuaspargtyrala
115120125
asntyrgluglycysleutrpasnalathrglyvalvalvalcysthr
130135140
glyaspalathrglncystyrglythrtrpvalproileglyleuala
145150155160
ileprogluasngluglyglyglysergluglyglyglyserglugly
165170175
glyglysergluglyglyglythrlyshisproasptyrglyaspthr
180185190
proileproglytyrthrtyrileasnproleuaspglythrtyrpro
195200205
proglythrgluglnasnleualaasnleuileleuleuleuargser
210215220
leuserleuleuileleusercyspheargileilecysserthrasn
225230235240
argglnglyala
<210>4
<211>36
<212>dna
<213>人工序列
<220>
<223>随机化的肽
<220>
<221>misc_feature
<222>(1)...(2)
<223>n=a或c或g或t
<220>
<221>misc_feature
<222>(3)...(3)
<223>k=g或t
<220>
<221>misc_feature
<222>(7)...(8)
<223>n=a或c或g或t
<220>
<221>misc_feature
<222>(9)...(9)
<223>k=g或t
<220>
<221>misc_feature
<222>(13)...(14)
<223>n=a或c或g或t
<220>
<221>misc_feature
<222>(15)...(15)
<223>k=g或t
<220>
<221>misc_feature
<222>(16)...(17)
<223>n=a或c或g或t
<220>
<221>misc_feature
<222>(18)...(18)
<223>k=g或t
<220>
<221>misc_feature
<222>(25)...(26)
<223>n=a或c或g或t
<220>
<221>misc_feature
<222>(27)...(27)
<223>k=g或t
<220>
<221>misc_feature
<222>(34)...(35)
<223>n=a或c或g或t
<220>
<221>misc_feature
<222>(36)...(36)
<223>k=g或t
<400>4
nnkactnnkaagnnknnktccgatnnkgaagaannk36
- 还没有人留言评论。精彩留言会获得点赞!