异质双聚体型血管内皮生长因子及其应用的制作方法
本发明主张美国临时申请第62/262,630号(申请日为2015年12月3日)的优先权,其完整内文以引用方式纳入本说明书为参考文献。
背景技术:
血管生成是肿瘤发展过程中一个重要的限速过程(rate-limiting process),其通过将平衡倾向于促血管新生因子(proangiogenic factors),而驱动血管生长而被诱导发生。处在微环境中的癌细胞需倚赖血管生成来供给氧及营养物。因此,靶定血管生成途径的药剂已被研发作为具潜力的癌症药物。最初的努力主要集中于靶定内皮与肿瘤衍生的血管内皮生长因子A(VEGF-A)/VEGFR信号传导。已设计出数种不同的策略,用以抑制此种信号传导,作为单一疗法或辅助疗法,例如VEGFR信号传导的小分子抑制剂、VEGF-陷阱、抗-VEGFR抗体及抗-VEGF-A单株抗体贝伐单抗(Bevacizumab)(癌思停,Avastin),为第一种在2004年被美国FDA批准的VEGF-A标靶抗体。
但是,有许多患者对抗血管生成药物没有反应,或很快地对其产生抗性。肿瘤可通过适应性反应(adaptive response),例如增量调节替代的促血管新生信号传导、选择正常的肿瘤周围血管、通过招集免疫细胞与骨髓衍生的促血管新生细胞抑制免疫监控及活化侵袭性表现型,而对抗血管生成药物产生抗性。此等适应性反应是通过,因肿瘤血管修剪及广泛抑制血管生成所引起的肿瘤内缺氧而被诱导发生。
有效抑制VEGF-A/VEGFR信号传导,并同时减轻对抗血管生成治疗的抗性,仍然是一项主要的挑战。
技术实现要素:
于是,本发明的一方面关于一种融合蛋白,含有(i)第一血管内皮生长因子(VEGF)同功异形体,(ii)第二VEGF同功异形体,及(iii)介于该第一同功异形体与该第二同功异形体间的双聚化功能域。所述第一同功异形体及第二同功异形体选自VEGF121及VEGF165。VEGF121可具有与SEQ ID NO:2达至少80%(例如,至少99%、98%、97%、96%、95%、90%或85%)序列同一性的胺基酸序列。VEGF165可具有与SEQ ID NO:4达至少80%(例如,至少99%、98%、97%、96%、95%、90%或85%)序列同一性的胺基酸序列。
于一项具体实施状态中,所述的双聚化功能域含有两个Fc区域,及一介于该二Fc区域间的连结肽。所述的连结肽可为一由15至30个胺基酸组成的可挠性连结肽。例如,所述的连结肽可为(Gly-Gly-Gly-Gly-Ser)n,n为3、4、5或6。
于一项具体实施状态中,所述的融合蛋白包括(从N-端至C-端的方向)VEGF121、所述二Fc区域其中之一、所述的连结肽、所述二Fc区域其中另一个及VEGF165。
于一项具体实施状态中,所述的融合蛋白包括(从N-端至C-端的方向)VEGF165、所述二Fc区域其中之一、所述的连结肽、所述二Fc区域其中另一个及VEGF121。
所述的融合蛋白可具有与SEQ ID NO:11达至少80%序列同一性的胺基酸序列。
于另一方面,本发明关于一种核酸分子,包含一编码本发明所述融合蛋白的核酸序列。于一项具体实施状态,所述的核酸序列为编码一与SEQ ID NO:11达至少80%(例如,至少99%、98%、97%、96%、95%、90%或85%)序列同一性的胺基酸序列。
于又另一方面,本发明关于一种医药组成物,包含所述的融合蛋白及医药上可接受的载体。所述的医药组成物可用于治疗癌症,或抑制有需要个体中的血管生成。
于以下的实施方式及参照的附图,将详细描述本发明的一项或多项具体实施状态。该等具体实施状态的其它特色、标的及优点,将由下列叙述说明及附图,以及专利保护范围彰显出来。
附图说明
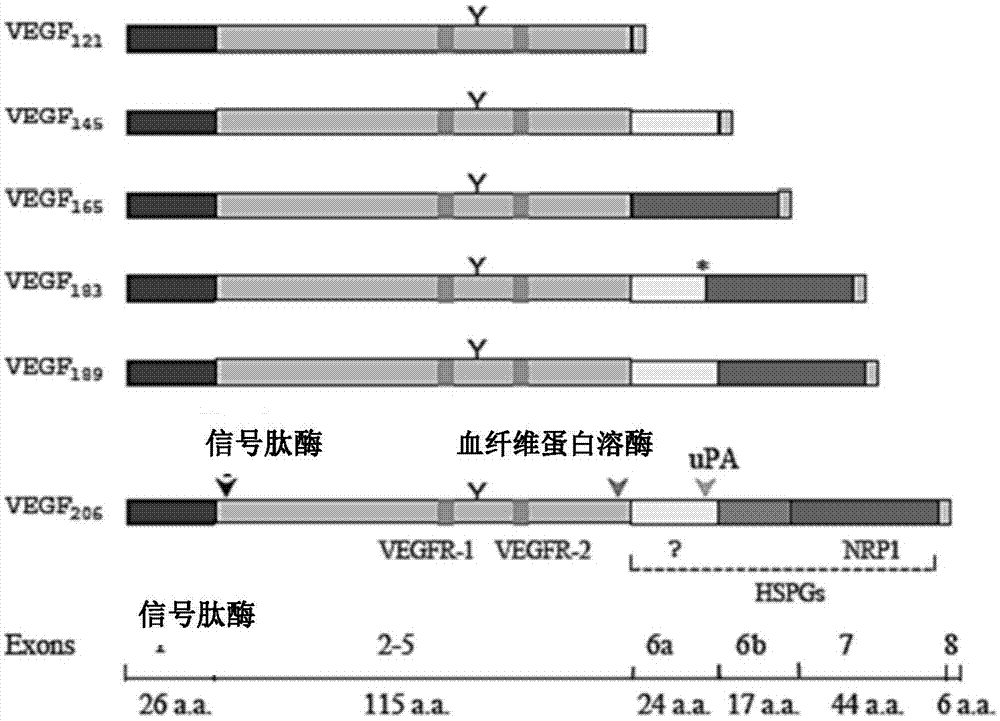
图1为所述血管内皮生长因子(VEGF)同功异形体的示意图。
图2为一种VEGF121-VEGF165融合蛋白的示意图。
图3为一组图显示所述VEGF121-VEGF165蛋白对于细胞增生的影响。总共1×104个3B-11细胞(A)及HCT-15细胞(B),于VEGF165(222pM)存在下以该融合蛋白(42、83、125pM)处理。细胞增生为以CCK-8套组进行测定(t-test,*P<0.05,**P<0.01,n=3)。1:对照组;2:VEGF165;3:VEGF165+VEGF121-VEGF165(42pM);4:VEGF165+VEGF121-VEGF165(83pM);5:VEGF165+VEGF121-VEGF165(125pM)。
图4为一组图显示所述VEGF121-VEGF165蛋白对于血管形成的影响。(A)3B-11(8×105)细胞接种于Matrigel,并于VEGF165(222pM)存在下以VEGF121-VEGF165(42、83、125pM)处理。管形成是经由在100×放大倍数下随机选择的视野中计数连接的细胞来定量。1:对照组;2:VEGF165;3:VEGF165+VEGF121-VEGF165(42pM);4:VEGF165+VEGF121-VEGF165(83pM);5:VEGF165+VEGF121-VEGF165(125pM)。(B)将VEGF121-VEGF165质体转感染至3B-11细胞中。使用3B-11细胞完成管形成分析。数据以平均值表示,基于三次独立实验。1:对照组;2:VEGF165(222pM);3:VEGF121-VEGF165质体。*P<0.05,**P<0.01,***p<0.001。
图5为显示VEGF121-VEGF165蛋白对于3B-11细胞移行的影响。将VEGF121-VEGF165质体转感染至3B-11细胞中。使用经转染的3B-11细胞测定细胞移行。3B-11细胞的细胞移行能力在VEGF165(222pM)存在时增强,但受到VEGF121-VEGF165质体的存在抑制。
图6为一组图显示所述VEGF121-VEGF165蛋白对于HCT-15细胞移行的影响。(A)HCT-15细胞接种于TranswellTM可通透嵌件中,并以VEGF121-VEGF165蛋白(42、83、125pM)或于VEGF165(222pM)存在下进行处理。于TranswellTM可通透嵌件中分析间隙之间的距离。以未经处理的HCT-15细胞为对照组。1:对照组;2:VEGF165;3:VEGF165+VEGF121-VEGF165(42pM);4:VEGF165+VEGF121-VEGF165(83pM);5:VEGF165+VEGF121-VEGF165(125pM)。(B)将VEGF121-VEGF165质体转感染至HCT-15细胞中。使用经转染的HCT-15细胞测定细胞移行。HCT-15细胞的细胞移行能力在VEGF165(222pM)存在时增强,但受到VEGF121-VEGF165质体的存在抑制。t-检定,*P<0.05,**P<0.01,***p<0.001,n=3。
图7显示VEGF121-VEGF165蛋白对于细胞侵袭性的影响。将HCT-15细胞于VEGF165(222pM)存在下以不同浓度的VEGF121-VEGF165(42、83、125pM)处理。通过小室分析(transwell chamber assay)测定细胞侵袭性。于进行细胞移行后48小时,经结晶紫染色后计数通过滤膜的细胞数目(原始放大倍率:400×)。1:对照组;2:VEGF165;3:VEGF165+VEGF121-VEGF165(42pM);4:VEGF165+VEGF121-VEGF165(83pM);5:VEGF165+VEGF121-VEGF165(125pM)。t-检定,*P<0.05,**P<0.01,***p<0.001。
图8为一组图显示以异种移植肿瘤发生分析VEGF121-VEGF165蛋白的功效。将HCT-15细胞经皮下注射入裸数的背侧(每只小鼠1部位)。受注射的小鼠每两天检查其肿瘤形成。将不同浓度的VEGF121-VEGF165蛋白(10、50或250ng/ml),或PBS对照组直接注射入小鼠体内的肿瘤中。(A)所侦测得的受注射小鼠的体重。(B)从肿瘤长度与宽度(以6-吋带表卡尺测量)估算的肿瘤体积,使用方程式:肿瘤体积=1x w2x 0.52。
具体实施方式
下列实施例将进一步说明本发明的其它特征和优点,但该等实施例仅为示例而用,并非对本发明的限制。
本发明意外发现,由两种不同的血管内皮生长因子(VEGF)同功异形体所组成的异质双聚体型VEGF,可通过以旁分泌及自体分泌的形式与VEGF165同质双聚体进行竞争,而减少细胞增生、移行、侵袭与管形成。
因此,本发明描述了一种融合型VEGF蛋白,含有通过一双聚化功能域连结的同功异形体VEGF121与同功异形体VEGF165。
VEGF-A基因包括八个外显子,可产生不同替换剪接的变异型,即VEGF121、VEGF145、VEGF165、VEGF183、VEGF189及VEGF206(参见图1)。
VEGF121为一种易溶且具弱酸性的多肽,缺乏肝素-结合功能域。于本文提供VEGF121的核酸序列(SEQ ID NO:1)及其所编码的胺基酸序列(SEQ ID NO:2)。SEQ ID NO:2的序列包括一N-端信号肽,此肽序列并不存在成熟型VEGF121中。
VEGF165含有碱性胺基酸及一与VEGF受体结合的缺乏肝素-结合功能域,可诱导信息传递及刺激内皮细胞增生。于本文提供VEGF165的核酸序列(SEQ ID NO:3)及其所编码的胺基酸序列(SEQ ID NO:4)。SEQ ID NO:4的序列包括一N-端信号肽,此肽序列并不存在成熟型VEGF165中。
本发明的融合型VEGF蛋白进一步包括一位于介于该二VEGF同功异形体之间,而使该融合型VEGF能形成一种异质双聚体的双聚化功能域。于一项具体实施状态,所述的双聚化功能域由两个经由一连结肽连接的Fc区域所组成。所述的Fc区域可为一人类IgG Fc区域。例如,所述的Fc区域可具有SEQ ID NO:6的胺基酸序列,其由SEQ ID NO:5的核酸序列所编码。
介于该二Fc区域间的连结肽可为任一该项技术已知的具挠性的连结肽。所述的连结肽可具有15至30个胺基酸。具挠性的连结肽可为富含Gly及Ser的连结肽。例如,所述的连结肽可为(Gly-Gly-Gly-Gly-Ser)n(SEQ ID NO:7),n为一整数(例如,1、2、3、4、5、6、7或8)。
本发明的融合蛋白可进一步包括一位于N-端的信号肽。所述信号肽可为对该等VEGF同功异形体系内源性的信号肽。例如,所述信号肽可具有SEQ ID NO:9的序列(其由SEQ ID NO:8的核酸序列所编码)。位于融合蛋白的C-端VEGF同功异形体可获可不包括一信号肽。
此外,本发明的融合蛋白可包括一有助于分离或鉴定该融合蛋白的C-端标记物。此类标记物可为一聚(His)标记、HA标记、Myc标记、V5或FLAG标记。
本发明的融合蛋白可含有(从N-端至C-端的方向)VEGF165、一Fc区域、一连结肽、一Fc区域及VEGF121。或者,本发明的融合蛋白可含有(从N-端至C-端的方向)VEGF121、一Fc区域、一连结肽、一Fc区域及VEGF165。各同功异形体可直接或经由一连结肽与一Fc区域连接,其中该连结肽可与介于该二Fc区域间的连结肽相同或不同。于一项具体实施状态中,所述融合蛋白可具有一与SEQ ID NO:11(其由SEQ ID NO:10的核酸序列所编码)达至少80%(例如,至少99%、98%、97%、96%、95%、90%或85%)序列同一性的胺基酸序列。
已知方法(例如重组技术)可使用于制造本发明的融合蛋白。例如,可制得一编码所述蛋白质的表现构体,并将其导入适合的宿主细胞(例如哺乳动物细胞)中。之后可将该于所述宿主细胞中表现的融合蛋白分离出。
本发明的融合蛋白可与一医药上可接受的载体混合,而形成一种医药组成物。所述组成物可投药予有其需要的个体用于治疗癌症或抑制血管生成。本发明的融合蛋白亦可用于与其它癌症治疗并用的组合疗法。
本发明的融合蛋白亦可与经设计用于将该融合蛋白靶定至肿瘤及/或其缔结血管的成分(例如,脂质、碳水化合物、聚合物或奈米粒子)结合或封装于其中。
所述的组成物可与一医药上可接受的载体,例如。磷酸盐缓冲食盐水、碳酸氢盐溶液及/或一佐剂调和。适宜的医药载体与稀释剂(以及其药用必需品)为该项技艺所已知。此类组成物可制备成一种可注射、液体溶液、乳剂或其它适合的配方。
佐剂的实例包括(但不限定于)明矾-沉淀物、弗氏完全佐剂、弗氏不完全佐剂、单磷酰脂质A/海藻糖双霉菌酸酯佐剂、含有短小棒杆菌和tRNA的油包水乳剂,及其它通过模拟进化上保守分子的特定集合来完成增加免疫反应任务的物质,包括微脂体、脂多醣(LPS)、抗原的分子笼、细菌细胞壁的组分及内吞的核酸,如双股RNA、单股DNA与未甲基化的含有CpG二核苷酸的DNA。其它实例包括霍乱毒素,大肠杆菌热-不稳定肠毒素,脂质体,免疫刺激性复合物(ISCOM),免疫刺激序列寡脱氧核苷酸及氢氧化铝。所述的组成物亦可包括一有助于活体内递送的聚合物。
可将有效量的前述组成物以非经肠道方式,例如皮下注射,静脉内注射或肌内注射进行投药。亦可使用其它投药途径。熟练的从业者将能够确定合适的剂量和投药途径。
可使用本发明的融合蛋进行治疗的癌症包括固体肿瘤,如胶质母细胞瘤、结肠直肠癌、肺癌、肾癌、肝癌,肾脏癌、神经内分泌肿瘤、乳腺癌、食道癌、胃肠道间质瘤、黑素瘤、卵巢癌、子宫颈癌、胰腺癌、前列腺癌,胃癌及头颈癌。
本发明以下所叙述的实施例仅作为辅助说明,并非用于以任何方式限制本发明所揭示的其它部分。无需进一步详细描述,相信习于本领域技术人员可基于本文的描述,最大程度地利用本发明所揭示的内容。所有于本文引用的公开文献皆以其完整内容通过引用方式并入本文。
实施例
本发明为制造一种与两个人类IgG1的Fc区域融合的新颖嵌合型VEGF121-VEGF165双聚体,作为有效的抗血管新生调制剂。经发现,本发明的嵌合型VEGF121-VEGF165双聚体可减低3B-11内皮细胞的管形成,并抑制HCT-15癌细胞的侵袭性。而且,本发明发现VEGF121-VEGF165蛋白可减弱癌细胞中通过PI3K-AKT-mTOR途径的VEGFR2-HIF-1α信号传递作用。数据证明,本发明的嵌合型VEGF121-VEGF165蛋白可拮抗血管生成及HIF-1α信号传递,显示该蛋白能对抗对于抗血管生成治疗产生的抗药性。
VEGF121-VEGF165融合蛋白的构筑及性质分析
经由将人类IgG1的Fc核酸序列分别与VEGF121序列的3’端及VEGF165序列的5’端融合,而产生一VEGF121-VEGF165融合体,其中该二Fc序列是通过一连结肽序列连接。将该VEGF121-VEGF165融合核酸序列选殖入pcDNA3.1载体,产生用以表现VEGF121-VEGF165的表现载体。使用DNA定序确认最终构筑体的完整性。所推导的蛋白质包括一段推定的26-aa信号肽(参见图2)。将含有VEGF121-VEGF165融合基因的质体转染至293T细胞株中。以西方转渍法确认VEGF121-VEGF165融合蛋白的表现及分泌。于本实例发现,在得自经转染的293T细胞的培养基及细胞溶解产物的样品中,存在对应于VEGF121-VEGF165融合蛋白的~120kDa的单一条带(数据未示)。重组蛋白于细胞系表现呈一种His-标记融合蛋白,并使用镍亲和性层析术进行纯化。通过西方转渍法测定所纯化得融合蛋白的纯度及分子量(数据未示)。此等结果表示,VEGF121-VEGF165经由片段与一多肽连结肽共价连接而形成双聚体。
VEGF121-VEGF165融合蛋白抑制由VEGF165所诱导的细胞增生
由于内皮细胞增生为早期血管生成反应所必需,本实例遂检测VEGF121-VEGF165蛋白是否会影响受VEGF165刺激的3B-11细胞增生,此为一种方便用于管形成分析的内皮细胞模式(参见Zhou等人,Methods 2008,44(2):190-195)。由图3A显示,VEGF165所诱导的3B-11细胞生长,以浓度-依赖方式受到VEGF121-VEGF165阻断。浓度222pM的VEGF165对于3B-11细胞增生的作用,容易地被浓度为42pM的重组VEGF121-VEGF165抑制,表示VEGF121-VEGF165蛋白能有效阻断VEGF165所诱导的细胞增生。而且,VEGF121-VEGF165蛋白对于VEGF165所诱导的HCT-15癌细胞生长,呈现相似的抑制功效(参见图3B)。吾等亦发现,当融合基因的质体转染至3B-11及HCT-15细胞时,VEGF121-VEGF165蛋白会以自分泌方式,对于此等细胞的细胞增生呈现相似的抑制活性,虽然该抑制作用在统计学上不具显著性(数据未示)。此等结果表示,VEGF121-VEGF165经由压制细胞增生(而非产生细胞毒性)来抑制细胞数量增加。再者,VEGF121-VEGF165可以自分泌方式抑制HCT-15的细胞转变(数据未示)。本发明的结果指出,VEGF121-VEGF165重组蛋白能以自分泌及旁分泌方式,阻断3B-11的细胞增生,及结肠癌细胞HCT-15的细胞增生与转变。
VEGF121-VEGF165融合蛋白抑制由VEGF165所诱导的管形成
血管生成后期阶段需要内皮细胞进行形态改变,而导致管腔形成。本实例检测在有VEGF121-VEGF165蛋白存在下的活体外管形成。使用被诱导侵入三维胶原凝胶,并在其中形成毛细管状管网络的3B-11内皮细胞,进行活体外管形成分析。参见Zhou等人,Methods2008,44(2):190-195。
结果显示,3B-11细胞能在正常条件下形成血管网络,而VEGF165会增加血管形成的数量(参见图4A)。然而,3B-11细胞的管状构造形成数,因添加VEGF121-VEGF165蛋白而以浓度-依赖方式受到抑制(参见图4A)。而且,VEGF121-VEGF165蛋白以旁分泌方式(图4A),及以自分泌方式(图4B),显著抑制VEGF165所诱导的管形成。此等结果证明,VEGF121-VEGF165嵌合型蛋白能于活体外抑制由VEGF165诱导的血管生成。
VEGF121-VEGF165融合蛋白抑制细胞移行
细胞移行为血管生成及肿瘤转移的一项关键过程。本实例研究细胞移行是否会受VEGF121-VEGF165嵌合型蛋白影响。使用间隙-闭合移行分析来检测细胞移行。结果与前述一致,VEGF121-VEGF165蛋白以自分泌方式,显著抑制3B-11细胞的移行作用(参见图5)。此外,结果亦显示,HCT-15细胞由VEGF165诱导的移行作用,因添加VEGF121-VEGF165蛋白而以浓度-依赖方式受到抑制(参见图6A)。VEGF121-VEGF165蛋白以旁泌方式(图6A),及以自分泌方式(图6B),显著抑制细胞移行。由此等结果表示,VEGF121-VEGF165嵌合型蛋白能于抑制内皮细胞及肿瘤细胞的移行作用。
VEGF121-VEGF165融合蛋白妨害肿瘤侵袭
为验证VEGF121-VEGF165嵌合型蛋白对于肿瘤细胞转移的效用,本实例遂使用Transwell分析来检测该蛋白对于细胞侵袭的影响。于无VEGF121-VEGF165存在下,VEGF165会诱导如密集穿透所呈现的侵袭能力(参见图7)。但是,由VEGF165诱导的细胞侵袭会因添加VEGF121-VEGF165蛋白,而以浓度-依赖方式受到抑制。以增加浓度的VEGF121-VEGF165蛋白处理时,穿透的细胞数目明显较单独以VEGF165处理时减少(参见图7)。此等结果表示,癌细胞的侵袭作用显著受到添加VEGF121-VEGF165蛋白的抑制,暗示该蛋白在压制癌细胞转移上扮演重要功能。
VEGF121-VEGF165嵌合型蛋白减弱通过PI3K-AKT-mTOR途径的VEGFR2-HIF-1α信号传递作用
为了验证VEGF121-VEGF165嵌合型蛋白是否会减弱HIF-1α信号传递作用,以减低对抗血管生成治疗的抗性,本实例首先检测VEGF121-VEGF165对于肿瘤细胞中VEGFR2-HIF-1α-VEGF165轴的影响。Lon受缺氧诱导因子-1α(HIF-1α)增量调节,且参与对于低氧可利用性的反应,使癌细胞能适应缺氧环境。本实例为检测该融合蛋白是否影响Lon蛋白酶的表现。使用西方转渍分析测定HIF-1α、VEGF165及Lon的表现。结果显示,HCT-15癌细胞中的VEGFR2-HIF-1α-VEGF165/Lon信号传递,受到VEGF165处理及氯化钴(CoCl2)刺激的缺氧而活化(数据未示)。然而,该信号传递的活化因添加VEGF121-VEGF165蛋白,而以浓度-依赖方式,在常氧及缺氧条件下受到抑制。重组VEGF121-VEGF165蛋白会减低由VEGF165及/或缺氧诱导的HIF-1α、VEGF165、Lon与二氧磷基-VEGFR2的表现量(数据未示)。就机制上来说,VEGF121-VEGF165蛋白通过阻制PI3K-AKT-mTOR途径,而抑制VEGFR2-HIF-1α-VEGF165/Lon信号传递的活化(数据未示),表示VEGF121-VEGF165蛋白会克服,在缺氧条件下由PI3K-AKT-mTOR触发至VEGFR2-HIF-1α-VEGF165/Lon轴的存活机制。此等数据暗示,VEGF121-VEGF165能抑制癌细胞中通过PI3K-AKT-mTOR途径的自分泌VEGFR2-HIF-1α-VEGF165/Lon信号传递作用。
VEGF121-VEGF165融合蛋白于活体内阻碍肿瘤生长
前述的数据证明,嵌合型VEGF121-VEGF165蛋白能于活体外抑制内皮细胞的管形成,及干扰肿瘤细胞生长、移行与侵袭。于本实例进行异种移植肿瘤发生分析,以证明VEGF121-VEGF165蛋白于活体内对肿瘤的抑制功效。于该项分析是使用BALB/c裸鼠(6-8周龄)。将1×106个HCT-15细胞悬浮于0.2ml的Matrigel中,以皮下注射入裸鼠的背侧(每只小鼠1个部位)。每两天检查小鼠体中的肿瘤形成。将不同浓度的VEGF121-VEGF165蛋白(10、50或250ng/ml),或PBS对照组溶液直接注射到小鼠体内的肿瘤中(参见图8B)。侦测小鼠体重(参见图8A)。然后将小鼠以CO2安乐死牺牲。
经VEGF121-VEGF165嵌合蛋白处理的小鼠体重并无明显变化,表示本发明蛋白质对该等动物不具毒性(参见图8A)。本发明的嵌合蛋白以剂量-依赖方式减低肿瘤生长(参见图8B)。因此,本实例的研究证明VEGF121-VEGF165嵌合蛋白能于活体内抑制肿瘤生长。
材料与方法
细胞株及细胞培养:3B-11细胞购自ATCC(#CRL-2160,马纳萨斯,VA,USA)。3B-11细胞维持于DMEM培养基中,HCT-15细胞维持于补充10%(v/v)热-不活化胎牛血清(FBS合格的;Invitrogen)、1%PSA(青霉素-链霉素-两性霉素B;Biological industries,NY,USA)的培养基,培养于具备5%CO2的37℃湿润化培养箱中。
VEGF121-VEGF165重组蛋白的纯化:使用Biomics转染试剂依pcDNA3.1载体无辅助剂(helper-free)系统(Biomics)的操作手册,将质体转染至293T细胞中。待培养36小时后,收集293T细胞培养物的上清液,并置于Ni-NTA树脂上,以250mmol/L咪唑溶析,依据操作手册进行纯化。将重组蛋白通过Microcon离心过滤单元(Millipore,Bedford,MA,USA)浓缩。最后,以10%SDS-PAGE与西方转渍法确认已纯化的蛋白。
细胞增殖分析:使用CCK-8染料减少分析(Enzo,USA)检测3B-11细胞及HCT-15细胞的增殖。将3B-11细胞或HCT-15细胞以不同浓度的VEGF121-VEGF165(42、83、125pM)进行前-处理30分钟,加入市售的VEGF165(222pM,Abcam,Cambridge,MA,USA),然后将细胞静置24小时。于处理结束,将10μl的CCK-8溶液加入平盘的每孔中,并将平盘至于培养箱中静置2~4小时。将平盘摇晃10秒后,经由使用微量平盘计读机测量于450nm下的吸光值分析细胞存活度。所有测量皆进行三次。使用T-测试比较各组的差异。数据以平均值±SD表示。
集落形成分析:集落生成分析是一项基于单细胞生长成为集落的能力的活体外转型分析。为检测此能力,将VEGF121-VEGF165的质体转染至HCT-15细胞(于10cm培养盘中处理过夜),并以VEGF165重组蛋白222pM处理做为阳性对照组。次日,将经处理的HCT-15细胞(每孔~1×103个)平铺于6-孔平盘中,并静置于37℃培养箱中。每48小时添加含有10%FBS的新鲜RPMI培养基。于第14天结束后,将细胞以冰冻PBS清洗两次,使用甲醇固定10分钟,的后以1%结晶紫甲醇溶液染色15分钟,随后以去离子水清洗。选出含有超过50个细胞的集落,并于显微镜下以200x倍率计量。
细胞移行分析:通过间隙-闭合移行分析,来测定细胞移行分析。将3B-11细胞或HCT-15细胞以不同浓度(42、83、125pM)的重组蛋白质处理16小时(37℃,5%CO2)。将此等细胞胰蛋白酶化,并悬浮于不含血清的DMEM或RPMI-1640培养基中。将总数8×105个悬浮于70μl不含血清的DMEM或RPMI-1640培养基中的细胞接种于各孔中(8x105个细胞/孔),并静置于37℃,5%CO2下培养。次日,使用灭菌的镊子,温和地将同上培养物-嵌件(Applied Biophysics,USA)取出,然后将每孔以2ml 0.1%FBS培养基填满。使用显微镜监测细胞移行达48小时。
血管形成分析:将Corning Matrix(BD Biosciences,San Jose,CA,USA)溶液置于冰上解冻过夜,取50μl等量覆盖于96-孔平盘并于37℃下静置1小时使其固化。将50μl含有约8x105 3B-11细胞的补充10%FBS的DMEM培养基接种于镀金属的Matrigel Matrix上,并静置于37℃下。将此等细胞以前述方法处理。该项分析进行三重复,并静置于37℃,5%CO2下。于2小时后,以结合计算机的显微镜(Olympus,东京,日本),于200x放大倍率下获取类毛细管构造的形成影像。经由手动计算于200x放大倍率下,随机选择视野中相连细胞的数目来定量管状构造。计数网络形成的总管数。
细胞侵袭分析:细胞侵袭为使用配备Matrigel-涂覆滤膜(孔径8μm)的小室(Corning Costar;Cambridge,MA,USA)进行评估。简言的,将滤膜预先涂覆以基底膜蛋白(Matrigel;BD Biosciences,San Jose,CA,USA),并静置于37℃,5%CO2下过夜令其干燥。将培养于不含FBS的培养基中的HCT-15细胞(8x105)接种于上层小室,并将下层小室充满10%FBS培养基。待于37℃下静置48小时后,将位于嵌件上面未移行的细胞以棉花棒去除。将已通过滤膜的细胞于甲醇中进行固定,并使用结晶紫染色。在显微镜下计数于随机选择视野中的细胞数量。
免疫转渍分析:将HCT-15细胞以10ml培养基含1.5×106个细胞的密度接种于10mm培养盘,并依如前述的方法于常氧及缺氧(CoCl2,氯化钴;150μM)条件下处理24小时。使用BCA蛋白分析法测定总体蛋白质浓度。将等量的总体蛋白质使用10%SDS-PAGE溶解,并经电转渍至聚偏二氟乙烯膜上。将膜以脱脂牛奶进行封阻,并于4℃下与第一级抗体进行探测反应过夜。然后将膜与适当的HRP-结合第二级抗体(GeneTex,新竹,台湾)进行反应,并使用增强化学发光方法(Bio-Rad,Hercules,CA,USA)将具有免疫反应的条带显色。用于本发明的抗体如说明书所述购得或制备得。对抗人类Lon的抗体制备方法如前所述。参见Wang等人,Cancer Sci 2010,101(12):2612-2620;及Cheng等人,Cell death&disease 2013,4,e681。Phospho-PI3K(Tyr458/Tyr199,#4228)、phospho-AKT(Ser473,#4060)及phoshpo-mTOR(Ser2448,#2971)抗体购自Cell Signaling Technology(Beverly,MA,USA);HIF-1α(#610958)抗体购自BD Biosciences(Franklin Lakes,NJ);phospho-VEGFR2(Tyr1054/Tyr1059,ab5473)、VEGF-165A(ab69479)抗体购自Abcam(Cambridge,MA,USA);beta-肌动蛋白抗体购自GeneTex(GTX109639,新竹,台湾)。
统计方法:于本发明使用参数Student氏t检定法判断所设定条件组之间的差异显著性。一般而言,P值<0.05认为在统计学上具有显著性(Student氏t检定,*p<0.05、**p<0.01及***p<0.001)。
其它具体实施状态
本说明书中所揭示的全部特征可以任何组合方式组合。于是,本说明书中所揭示的各别特征可由依相同、相等或类似目的的替代特征取代。因此,除非另行清楚地指示,所揭示的各特征仅为一系列同等物或类似特征的实例。
从前述的说明,习于本领域的普通技术人员可容易地确定本发明的基本特征,且在未偏离其范围的情况下,可进行本发明的各种改变与修饰,以使其适于各种不同用途与状况。因此,于本专利保护范围内亦包含其它具体状态。
SEQUENCE LISTING
<110> 财团法人国家卫生研究院 (National Health Research Institutes)
<120> 异质双聚体型血管内皮生长因子及其应用
<130> 218384-0006PCT
<150> US 62/262,630
<151> 2015-12-03
<160> 11
<170> PatentIn 3.5版
<210> 1
<211> 444
<212> DNA
<213> 人类
<220>
<221> CDS
<222> (1)..(444)
<223> VEGF121
<400> 1
atg aac ttt ctg ctg tct tgg gtg cat tgg agc ctt gcc ttg ctg ctc 48
Met Asn Phe Leu Leu Ser Trp Val His Trp Ser Leu Ala Leu Leu Leu
1 5 10 15
tac ctc cac cat gcc aag tgg tcc cag gct gca ccc atg gca gaa gga 96
Tyr Leu His His Ala Lys Trp Ser Gln Ala Ala Pro Met Ala Glu Gly
20 25 30
gga ggg cag aat cat cac gaa gtg gtg aag ttc atg gat gtc tat cag 144
Gly Gly Gln Asn His His Glu Val Val Lys Phe Met Asp Val Tyr Gln
35 40 45
cgc agc tac tgc cat cca atc gag acc ctg gtg gac atc ttc cag gag 192
Arg Ser Tyr Cys His Pro Ile Glu Thr Leu Val Asp Ile Phe Gln Glu
50 55 60
tac cct gat gag atc gag tac atc ttc aag cca tcc tgt gtg ccc ctg 240
Tyr Pro Asp Glu Ile Glu Tyr Ile Phe Lys Pro Ser Cys Val Pro Leu
65 70 75 80
atg cga tgc ggg ggc tgc tgc aat gac gag ggc ctg gag tgt gtg ccc 288
Met Arg Cys Gly Gly Cys Cys Asn Asp Glu Gly Leu Glu Cys Val Pro
85 90 95
act gag gag tcc aac atc acc atg cag att atg cgg atc aaa cct cac 336
Thr Glu Glu Ser Asn Ile Thr Met Gln Ile Met Arg Ile Lys Pro His
100 105 110
caa ggc cag cac ata gga gag atg agc ttc cta cag cac aac aaa tgt 384
Gln Gly Gln His Ile Gly Glu Met Ser Phe Leu Gln His Asn Lys Cys
115 120 125
gaa tgc aga cca aag aaa gat aga gca aga caa gaa aaa tgt gac aag 432
Glu Cys Arg Pro Lys Lys Asp Arg Ala Arg Gln Glu Lys Cys Asp Lys
130 135 140
ccg agg cgg tga 444
Pro Arg Arg
145
<210> 2
<211> 147
<212> PRT
<213> 人类
<400> 2
Met Asn Phe Leu Leu Ser Trp Val His Trp Ser Leu Ala Leu Leu Leu
1 5 10 15
Tyr Leu His His Ala Lys Trp Ser Gln Ala Ala Pro Met Ala Glu Gly
20 25 30
Gly Gly Gln Asn His His Glu Val Val Lys Phe Met Asp Val Tyr Gln
35 40 45
Arg Ser Tyr Cys His Pro Ile Glu Thr Leu Val Asp Ile Phe Gln Glu
50 55 60
Tyr Pro Asp Glu Ile Glu Tyr Ile Phe Lys Pro Ser Cys Val Pro Leu
65 70 75 80
Met Arg Cys Gly Gly Cys Cys Asn Asp Glu Gly Leu Glu Cys Val Pro
85 90 95
Thr Glu Glu Ser Asn Ile Thr Met Gln Ile Met Arg Ile Lys Pro His
100 105 110
Gln Gly Gln His Ile Gly Glu Met Ser Phe Leu Gln His Asn Lys Cys
115 120 125
Glu Cys Arg Pro Lys Lys Asp Arg Ala Arg Gln Glu Lys Cys Asp Lys
130 135 140
Pro Arg Arg
145
<210> 3
<211> 576
<212> DNA
<213> 人类
<220>
<221> CDS
<222> (1)..(576)
<223> VEGF165
<400> 3
atg aac ttt ctg ctg tct tgg gtg cat tgg agc ctt gcc ttg ctg ctc 48
Met Asn Phe Leu Leu Ser Trp Val His Trp Ser Leu Ala Leu Leu Leu
1 5 10 15
tac ctc cac cat gcc aag tgg tcc cag gct gca ccc atg gca gaa gga 96
Tyr Leu His His Ala Lys Trp Ser Gln Ala Ala Pro Met Ala Glu Gly
20 25 30
gga ggg cag aat cat cac gaa gtg gtg aag ttc atg gat gtc tat cag 144
Gly Gly Gln Asn His His Glu Val Val Lys Phe Met Asp Val Tyr Gln
35 40 45
cgc agc tac tgc cat cca atc gag acc ctg gtg gac atc ttc cag gag 192
Arg Ser Tyr Cys His Pro Ile Glu Thr Leu Val Asp Ile Phe Gln Glu
50 55 60
tac cct gat gag atc gag tac atc ttc aag cca tcc tgt gtg ccc ctg 240
Tyr Pro Asp Glu Ile Glu Tyr Ile Phe Lys Pro Ser Cys Val Pro Leu
65 70 75 80
atg cga tgc ggg ggc tgc tgc aat gac gag ggc ctg gag tgt gtg ccc 288
Met Arg Cys Gly Gly Cys Cys Asn Asp Glu Gly Leu Glu Cys Val Pro
85 90 95
act gag gag tcc aac atc acc atg cag att atg cgg atc aaa cct cac 336
Thr Glu Glu Ser Asn Ile Thr Met Gln Ile Met Arg Ile Lys Pro His
100 105 110
caa ggc cag cac ata gga gag atg agc ttc cta cag cac aac aaa tgt 384
Gln Gly Gln His Ile Gly Glu Met Ser Phe Leu Gln His Asn Lys Cys
115 120 125
gaa tgc aga cca aag aaa gat aga gca aga caa gaa aat ccc tgt ggg 432
Glu Cys Arg Pro Lys Lys Asp Arg Ala Arg Gln Glu Asn Pro Cys Gly
130 135 140
cct tgc tca gag cgg aga aag cat ttg ttt gta caa gat ccg cag acg 480
Pro Cys Ser Glu Arg Arg Lys His Leu Phe Val Gln Asp Pro Gln Thr
145 150 155 160
tgt aaa tgt tcc tgc aaa aac aca gac tcg cgt tgc aag gcg agg cag 528
Cys Lys Cys Ser Cys Lys Asn Thr Asp Ser Arg Cys Lys Ala Arg Gln
165 170 175
ctt gag tta aac gaa cgt act tgc aga tgt gac aag ccg agg cgg tga 576
Leu Glu Leu Asn Glu Arg Thr Cys Arg Cys Asp Lys Pro Arg Arg
180 185 190
<210> 4
<211> 191
<212> PRT
<213> 人类
<400> 4
Met Asn Phe Leu Leu Ser Trp Val His Trp Ser Leu Ala Leu Leu Leu
1 5 10 15
Tyr Leu His His Ala Lys Trp Ser Gln Ala Ala Pro Met Ala Glu Gly
20 25 30
Gly Gly Gln Asn His His Glu Val Val Lys Phe Met Asp Val Tyr Gln
35 40 45
Arg Ser Tyr Cys His Pro Ile Glu Thr Leu Val Asp Ile Phe Gln Glu
50 55 60
Tyr Pro Asp Glu Ile Glu Tyr Ile Phe Lys Pro Ser Cys Val Pro Leu
65 70 75 80
Met Arg Cys Gly Gly Cys Cys Asn Asp Glu Gly Leu Glu Cys Val Pro
85 90 95
Thr Glu Glu Ser Asn Ile Thr Met Gln Ile Met Arg Ile Lys Pro His
100 105 110
Gln Gly Gln His Ile Gly Glu Met Ser Phe Leu Gln His Asn Lys Cys
115 120 125
Glu Cys Arg Pro Lys Lys Asp Arg Ala Arg Gln Glu Asn Pro Cys Gly
130 135 140
Pro Cys Ser Glu Arg Arg Lys His Leu Phe Val Gln Asp Pro Gln Thr
145 150 155 160
Cys Lys Cys Ser Cys Lys Asn Thr Asp Ser Arg Cys Lys Ala Arg Gln
165 170 175
Leu Glu Leu Asn Glu Arg Thr Cys Arg Cys Asp Lys Pro Arg Arg
180 185 190
<210> 5
<211> 711
<212> DNA
<213> 人类
<220>
<221> CDS
<222> (1)..(711)
<223> Fc
<400> 5
gag ccc aaa tct tgt gac aaa act cac aca tgc cca ccg tgc cca gca 48
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
1 5 10 15
cct gaa ctc ctg ggg gga ccg tca gtc ttc ctc ttc ccc cca aaa ccc 96
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
20 25 30
aag gac acc ctc atg atc tcc cgg acc cct gag gtc aca tgc gtg gtg 144
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
35 40 45
gtg gac gtg agc cac gaa gac cct gag gtc aag ttc aac tgg tac gtg 192
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
50 55 60
gac ggc gtg gag gtg cat aat gcc aag aca aag ccg cgg gag gag cag 240
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
65 70 75 80
tac aac agc acg tac cgg gtg gtc agc gtc ctc acc gtc ctg cac cag 288
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
85 90 95
gac tgg ctg aat ggc aag gag tac aag tgc aag gtc tcc aac aaa gcc 336
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
100 105 110
ctc cca gcc ccc atc gag aaa acc atc tcc aaa gcc aaa ggg cag ccc 384
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
115 120 125
cga gaa cca cag gtg tac acc ctg ccc cca tcc cgg gat gag ctg acc 432
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr
130 135 140
aag aac cag gtc agc ctg acc tgc ctg gtc aaa ggc ttc tat ccc agc 480
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
145 150 155 160
gac atc gcc gtg gag tgg gag agc aat ggg cag ccg gag aac aac tac 528
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
165 170 175
aag acc acg cct ccc gtg ctg gac tcc gac ggc tcc ttc ttc ctc tac 576
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
180 185 190
agc aag ctc acc gtg gac aag agc agg tgg cag cag ggg aac gtc ttc 624
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
195 200 205
tca tgc tcc gtg atg cat gag gct ctg cac aac cac tac acg cag aag 672
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
210 215 220
agc ctc tcc ctg tct ccg ggt aaa gtc gac gag ggc ccg 711
Ser Leu Ser Leu Ser Pro Gly Lys Val Asp Glu Gly Pro
225 230 235
<210> 6
<211> 237
<212> PRT
<213> 人类
<400> 6
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
1 5 10 15
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
20 25 30
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
35 40 45
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
50 55 60
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
65 70 75 80
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
85 90 95
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
100 105 110
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
115 120 125
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr
130 135 140
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
145 150 155 160
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
165 170 175
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
180 185 190
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
195 200 205
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
210 215 220
Ser Leu Ser Leu Ser Pro Gly Lys Val Asp Glu Gly Pro
225 230 235
<210> 7
<211> 5
<212> PRT
<213> 人造序列
<220>
<223> 合成连结肽
<400> 7
Gly Gly Gly Gly Ser
1 5
<210> 8
<211> 78
<212> DNA
<213> 人造序列
<220>
<223> 合成聚核苷酸
<220>
<221> CDS
<222> (1)..(78)
<223> 信号肽
<400> 8
aac ttt ctg ctg tct tgg gtg cat tgg agc ctt gcc ttg ctg ctc tac 48
Asn Phe Leu Leu Ser Trp Val His Trp Ser Leu Ala Leu Leu Leu Tyr
1 5 10 15
ctc cac cat gcc aag tgg tcc cag gct gca 78
Leu His His Ala Lys Trp Ser Gln Ala Ala
20 25
<210> 9
<211> 26
<212> PRT
<213> 人造序列
<220>
<223> 合成构体
<400> 9
Asn Phe Leu Leu Ser Trp Val His Trp Ser Leu Ala Leu Leu Leu Tyr
1 5 10 15
Leu His His Ala Lys Trp Ser Gln Ala Ala
20 25
<210> 10
<211> 2388
<212> DNA
<213> 人造序列
<220>
<223> 合成聚核苷酸
<220>
<221> CDS
<222> (1)..(2388)
<223> VEGF121-Fc-连结肽-Fc-VEGF165-6xHis标记
<400> 10
atg aac ttt ctg ctg tct tgg gtg cat tgg agc ctt gcc ttg ctg ctc 48
Met Asn Phe Leu Leu Ser Trp Val His Trp Ser Leu Ala Leu Leu Leu
1 5 10 15
tac ctc cac cat gcc aag tgg tcc cag gct gca ccc atg gca gaa gga 96
Tyr Leu His His Ala Lys Trp Ser Gln Ala Ala Pro Met Ala Glu Gly
20 25 30
gga ggg cag aat cat cac gaa gtg gtg aag ttc atg gat gtc tat cag 144
Gly Gly Gln Asn His His Glu Val Val Lys Phe Met Asp Val Tyr Gln
35 40 45
cgc agc tac tgc cat cca atc gag acc ctg gtg gac atc ttc cag gag 192
Arg Ser Tyr Cys His Pro Ile Glu Thr Leu Val Asp Ile Phe Gln Glu
50 55 60
tac cct gat gag atc gag tac atc ttc aag cca tcc tgt gtg ccc ctg 240
Tyr Pro Asp Glu Ile Glu Tyr Ile Phe Lys Pro Ser Cys Val Pro Leu
65 70 75 80
atg cga tgc ggg ggc tgc tgc aat gac gag ggc ctg gag tgt gtg ccc 288
Met Arg Cys Gly Gly Cys Cys Asn Asp Glu Gly Leu Glu Cys Val Pro
85 90 95
act gag gag tcc aac atc acc atg cag att atg cgg atc aaa cct cac 336
Thr Glu Glu Ser Asn Ile Thr Met Gln Ile Met Arg Ile Lys Pro His
100 105 110
caa ggc cag cac ata gga gag atg agc ttc cta cag cac aac aaa tgt 384
Gln Gly Gln His Ile Gly Glu Met Ser Phe Leu Gln His Asn Lys Cys
115 120 125
gaa tgc aga cca aag aaa gat gag ccc aaa tct tgt gac aaa act cac 432
Glu Cys Arg Pro Lys Lys Asp Glu Pro Lys Ser Cys Asp Lys Thr His
130 135 140
aca tgc cca ccg tgc cca gca cct gaa ctc ctg ggg gga ccg tca gtc 480
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
145 150 155 160
ttc ctc ttc ccc cca aaa ccc aag gac acc ctc atg atc tcc cgg acc 528
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
165 170 175
cct gag gtc aca tgc gtg gtg gtg gac gtg agc cac gaa gac cct gag 576
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
180 185 190
gtc aag ttc aac tgg tac gtg gac ggc gtg gag gtg cat aat gcc aag 624
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
195 200 205
aca aag ccg cgg gag gag cag tac aac agc acg tac cgg gtg gtc agc 672
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
210 215 220
gtc ctc acc gtc ctg cac cag gac tgg ctg aat ggc aag gag tac aag 720
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
225 230 235 240
tgc aag gtc tcc aac aaa gcc ctc cca gcc ccc atc gag aaa acc atc 768
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
245 250 255
tcc aaa gcc aaa ggg cag ccc cga gaa cca cag gtg tac acc ctg ccc 816
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
260 265 270
cca tcc cgg gat gag ctg acc aag aac cag gtc agc ctg acc tgc ctg 864
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
275 280 285
gtc aaa ggc ttc tat ccc agc gac atc gcc gtg gag tgg gag agc aat 912
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
290 295 300
ggg cag ccg gag aac aac tac aag acc acg cct ccc gtg ctg gac tcc 960
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
305 310 315 320
gac ggc tcc ttc ttc ctc tac agc aag ctc acc gtg gac aag agc agg 1008
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
325 330 335
tgg cag cag ggg aac gtc ttc tca tgc tcc gtg atg cat gag gct ctg 1056
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
340 345 350
cac aac cac tac acg cag aag agc ctc tcc ctg tct ccg ggt aaa gtc 1104
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Val
355 360 365
gac gag ggc ccg ggt gga ggc ggg tcg ggc ggt ggg gga tct ggg ggc 1152
Asp Glu Gly Pro Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
370 375 380
ggt gga tcc gag ccc aaa tct tgt gac aaa act cac aca tgc cca ccg 1200
Gly Gly Ser Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
385 390 395 400
tgc cca gca cct gaa ctc ctg ggg gga ccg tca gtc ttc ctc ttc ccc 1248
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
405 410 415
cca aaa ccc aag gac acc ctc atg atc tcc cgg acc cct gag gtc aca 1296
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
420 425 430
tgc gtg gtg gtg gac gtg agc cac gaa gac cct gag gtc aag ttc aac 1344
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
435 440 445
tgg tac gtg gac ggc gtg gag gtg cat aat gcc aag aca aag ccg cgg 1392
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
450 455 460
gag gag cag tac aac agc acg tac cgg gtg gtc agc gtc ctc acc gtc 1440
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
465 470 475 480
ctg cac cag gac tgg ctg aat ggc aag gag tac aag tgc aag gtc tcc 1488
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
485 490 495
aac aaa gcc ctc cca gcc ccc atc gag aaa acc atc tcc aaa gcc aaa 1536
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
500 505 510
ggg cag ccc cga gaa cca cag gtg tac acc ctg ccc cca tcc cgg gat 1584
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp
515 520 525
gag ctg acc aag aac cag gtc agc ctg acc tgc ctg gtc aaa ggc ttc 1632
Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
530 535 540
tat ccc agc gac atc gcc gtg gag tgg gag agc aat ggg cag ccg gag 1680
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
545 550 555 560
aac aac tac aag acc acg cct ccc gtg ctg gac tcc gac ggc tcc ttc 1728
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
565 570 575
ttc ctc tac agc aag ctc acc gtg gac aag agc agg tgg cag cag ggg 1776
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
580 585 590
aac gtc ttc tca tgc tcc gtg atg cat gag gct ctg cac aac cac tac 1824
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
595 600 605
acg cag aag agc ctc tcc ctg tct ccg ggt aaa gtc gac gag ggc ccg 1872
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Val Asp Glu Gly Pro
610 615 620
gca ccc atg gca gaa gga gga ggg cag aat cat cac gaa gtg gtg aag 1920
Ala Pro Met Ala Glu Gly Gly Gly Gln Asn His His Glu Val Val Lys
625 630 635 640
ttc atg gat gtc tat cag cgc agc tac tgc cat cca atc gag acc ctg 1968
Phe Met Asp Val Tyr Gln Arg Ser Tyr Cys His Pro Ile Glu Thr Leu
645 650 655
gtg gac atc ttc cag gag tac cct gat gag atc gag tac atc ttc aag 2016
Val Asp Ile Phe Gln Glu Tyr Pro Asp Glu Ile Glu Tyr Ile Phe Lys
660 665 670
cca tcc tgt gtg ccc ctg atg cga tgc ggg ggc tgc tgc aat gac gag 2064
Pro Ser Cys Val Pro Leu Met Arg Cys Gly Gly Cys Cys Asn Asp Glu
675 680 685
ggc ctg gag tgt gtg ccc act gag gag tcc aac atc acc atg cag att 2112
Gly Leu Glu Cys Val Pro Thr Glu Glu Ser Asn Ile Thr Met Gln Ile
690 695 700
atg cgg atc aaa cct cac caa ggc cag cac ata gga gag atg agc ttc 2160
Met Arg Ile Lys Pro His Gln Gly Gln His Ile Gly Glu Met Ser Phe
705 710 715 720
cta cag cac aac aaa tgt gaa tgc aga cca aag aaa gat aga gca aga 2208
Leu Gln His Asn Lys Cys Glu Cys Arg Pro Lys Lys Asp Arg Ala Arg
725 730 735
caa gaa aat ccc tgt ggg cct tgc tca gag cgg aga aag cat ttg ttt 2256
Gln Glu Asn Pro Cys Gly Pro Cys Ser Glu Arg Arg Lys His Leu Phe
740 745 750
gta caa gat ccg cag acg tgt aaa tgt tcc tgc aaa aac aca gac tcg 2304
Val Gln Asp Pro Gln Thr Cys Lys Cys Ser Cys Lys Asn Thr Asp Ser
755 760 765
cgt tgc aag gcg agg cag ctt gag tta aac gaa cgt act tgc aga tgt 2352
Arg Cys Lys Ala Arg Gln Leu Glu Leu Asn Glu Arg Thr Cys Arg Cys
770 775 780
gac aag ccg agg cgg cac cac cac cac cac cac tga 2388
Asp Lys Pro Arg Arg His His His His His His
785 790 795
<210> 11
<211> 795
<212> PRT
<213> 人造序列
<220>
<223> 合成构体
<400> 11
Met Asn Phe Leu Leu Ser Trp Val His Trp Ser Leu Ala Leu Leu Leu
1 5 10 15
Tyr Leu His His Ala Lys Trp Ser Gln Ala Ala Pro Met Ala Glu Gly
20 25 30
Gly Gly Gln Asn His His Glu Val Val Lys Phe Met Asp Val Tyr Gln
35 40 45
Arg Ser Tyr Cys His Pro Ile Glu Thr Leu Val Asp Ile Phe Gln Glu
50 55 60
Tyr Pro Asp Glu Ile Glu Tyr Ile Phe Lys Pro Ser Cys Val Pro Leu
65 70 75 80
Met Arg Cys Gly Gly Cys Cys Asn Asp Glu Gly Leu Glu Cys Val Pro
85 90 95
Thr Glu Glu Ser Asn Ile Thr Met Gln Ile Met Arg Ile Lys Pro His
100 105 110
Gln Gly Gln His Ile Gly Glu Met Ser Phe Leu Gln His Asn Lys Cys
115 120 125
Glu Cys Arg Pro Lys Lys Asp Glu Pro Lys Ser Cys Asp Lys Thr His
130 135 140
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
145 150 155 160
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
165 170 175
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
180 185 190
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
195 200 205
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
210 215 220
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
225 230 235 240
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
245 250 255
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
260 265 270
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
275 280 285
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
290 295 300
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
305 310 315 320
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
325 330 335
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
340 345 350
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Val
355 360 365
Asp Glu Gly Pro Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
370 375 380
Gly Gly Ser Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
385 390 395 400
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
405 410 415
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
420 425 430
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
435 440 445
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
450 455 460
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
465 470 475 480
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
485 490 495
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
500 505 510
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp
515 520 525
Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
530 535 540
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
545 550 555 560
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
565 570 575
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
580 585 590
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
595 600 605
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Val Asp Glu Gly Pro
610 615 620
Ala Pro Met Ala Glu Gly Gly Gly Gln Asn His His Glu Val Val Lys
625 630 635 640
Phe Met Asp Val Tyr Gln Arg Ser Tyr Cys His Pro Ile Glu Thr Leu
645 650 655
Val Asp Ile Phe Gln Glu Tyr Pro Asp Glu Ile Glu Tyr Ile Phe Lys
660 665 670
Pro Ser Cys Val Pro Leu Met Arg Cys Gly Gly Cys Cys Asn Asp Glu
675 680 685
Gly Leu Glu Cys Val Pro Thr Glu Glu Ser Asn Ile Thr Met Gln Ile
690 695 700
Met Arg Ile Lys Pro His Gln Gly Gln His Ile Gly Glu Met Ser Phe
705 710 715 720
Leu Gln His Asn Lys Cys Glu Cys Arg Pro Lys Lys Asp Arg Ala Arg
725 730 735
Gln Glu Asn Pro Cys Gly Pro Cys Ser Glu Arg Arg Lys His Leu Phe
740 745 750
Val Gln Asp Pro Gln Thr Cys Lys Cys Ser Cys Lys Asn Thr Asp Ser
755 760 765
Arg Cys Lys Ala Arg Gln Leu Glu Leu Asn Glu Arg Thr Cys Arg Cys
770 775 780
Asp Lys Pro Arg Arg His His His His His His
785 790 795
- 还没有人留言评论。精彩留言会获得点赞!
- 血管内皮生长因子时间分辨荧光免疫分析方法及试剂盒的制作方法
- 基于模拟人血管内皮细胞生长因子vegf表位的疫苗及其制备方法
- 人血管内皮生长因子抗原表位及其表位疫苗的制作方法
- 重组人血管内皮生长因子165与免疫球蛋白Fc片段融合蛋白(VEGF165-Fc)的实验技术的制作方法
- 抗人促血液血管生长因子(hapo)的单克隆抗体、多克隆抗体及其应用的制作方法
- 针对血管内皮生长因子的氨基酸序列和包括其的多肽用于治疗特征在于过量和 /或病理 ...的制作方法
- 一种血管生长抑制因子Kringle 5融合和非融合基因的克隆、鉴定和表达方法
- 改造的血管内皮生长因子多肽片段及其应用的制作方法
- 一种抗血管内皮生长因子的单克隆抗体及应用的制作方法
- 人血管内皮生长因子165b与人血清白蛋白的融合蛋白及其制备的制作方法
- 用于血管内皮生长因子定量测定试剂盒及其发光底物溶液的制作方法
- 血管内皮生长因子b(vegf-b)缺陷动物的心脏异常及与这些心脏异常相关的方法
- 血管内皮生长因子时间分辨荧光免疫分析方法及试剂盒的制作方法
- 一种检测血清结缔组织生长因子用试剂盒的制作方法
- 人血管内皮生长因子抗原表位及其表位疫苗的制作方法
- 重组人血管内皮生长因子165与免疫球蛋白Fc片段融合蛋白(VEGF165-Fc)的实验技术的制作方法
- 抗人促血液血管生长因子(hapo)的单克隆抗体、多克隆抗体及其应用的制作方法
- 针对血管内皮生长因子的氨基酸序列和包括其的多肽用于治疗特征在于过量和 /或病理 ...的制作方法
- 改造的血管内皮生长因子多肽片段及其应用的制作方法
- 一种抗血管内皮生长因子的单克隆抗体及应用的制作方法