一种肾阳虚外感小鼠模型肺组织qPCR内参基因组合及其筛选方法与流程
本发明涉及分子生物学技术,特别涉及一种基于qpcr技术的内参基因组合及其筛选方法。
背景技术:
实时荧光定量pcr技术(quantitativereal-timepcr,qpcr)是一种实时监测pcr扩增产物并进行解析的定量方法,具有敏感性高、重复性好、检测速度快、自动化程度高等诸多优点,目前已被国内外学者广泛应用。该技术一般需要用表达稳定的内参基因对靶基因的表达情况进行校正和标准化。理想的内参基因应在各种试验条件下,各种类型的组织和细胞中均恒定表达,而且其表达量是近似的,无显著性差异。然而,不同类型的组织,不同的发育阶段和不同处理条件下内参基因转录水平均可能发生变化,因此,选择合适的内参基因以减少检测样本间的差异,决定了利用荧光定量pcr分析基因表达水平结果的可靠性。
以往的研究多以表达量众多的内参基因如核糖体rna18srrna或28srrna、3-磷酸甘油脱氢酶以及肌动蛋白等作为内参。然而,大量的研究表明,这些常用的内参基因在不同实验条件下的表达也有变化。不稳定内参基因的使用会导致qpcr的结果出现严重偏差。很多研究表明,一个广泛通用的内参基因是不存在的,研究者在每一个实验或物种中都需要寻找合适的稳定表达的基因作为内参。bestkeeper、genorm和normfinder等广泛用作内参基因筛选软件,可帮助研究人员在不同的研究中选择稳定的内参基因,但这些软件筛选到的单内参或双内参基因都不适合用于肾阳虚外感病证肺组织的内参基因。近年来,文献多见于玉兰[8]、泽泻[9]、川续断[10]等药用植物的rt-qpcr内参基因筛选的报道越来越多,但目前尚无关于肾阳虚外感小鼠模型肺组织rt-qpcr内参基因筛选的报道。
技术实现要素:
本发明的发明目的为引入了统计分析的组间t检验方法,能高效、准确地筛选到合适的双内参基因组合,可用于后续开展对肾阳虚外感病证肺组织目的基因表达的研究。
为实现上述发明目的,采用以下技术方案:
一种肾阳虚外感小鼠模型肺组织qpcr内参基因组合,其特征在于,所述内参基因组合为grcc10和ppia。
上述的肾阳虚外感小鼠模型肺组织qpcr内参基因组合的筛选方法,首先分别设计15个内参基因actb,β2m,gapdh,gusb,tuba,grcc10,eif4h,rnf187,nedd8,ywhea,18srrna,rpl13,ubc,rpl32,ppia的特异性引物,上下游引物序列依次如seqidno.3-32所示,然后以肾阳虚外感小鼠模型,cdna为模板,分别利用所述特异性引物对15个内参基因进行rt-qpcr分析,确立所述肾阳虚外感小鼠模型肺组织qpcr内参基因组合。
优选地,筛选方法包括以下步骤:
(1)复制肾阳虚模型;
(2)建立肾阳虚外感模型;
(3)rt-pcr检测肺组织中h1n1的mrna的表达;
(4)提取动物组织总rna;
(5)反转录合成cdna;
(6)设计扩增引物进行rt-pcr扩增;分别设计15个内参基因actb,β2m,gapdh,gusb,tuba,grcc10,eif4h,rnf187,nedd8,ywhea,18srrna,rpl13,ubc,rpl32,ppia,所述内参基因的特异性引物序列依次如seqidno.1-30所示;
(7)内参基因的rt-pcr分析;采用双基因两两组合的方法,并对两两组合的双基因在正常组和模型组之间的基因表达差异进行t检验,选择组合的p值大于0.05以上,又进行了这些组合的正常组与模型组差值的绝对值比较,筛选差值的绝对值最小的组合作为双参考基因的最优化组合,分析得grcc10+ppia组合基因为肾阳虚外感小鼠模型的稳定内参基因。
优选地,步骤(1)所述的复制肾阳虚模型的方法为以4ml/kg腹腔注射2mg/ml苯甲酸雌二醇稀释液,每日1次,连续7d,复制肾阳虚模型。
优选地,步骤(1)所述的建立肾阳虚外感模型的方法为给予麻醉后的肾阳虚小鼠滴鼻接种流感病毒h1n1鸡胚尿囊液20μl/只,正常组滴鼻等体积的生理盐水,接种病毒的小鼠隔离喂养,自由进食进水6d,解剖处死;其中鸡胚尿囊液的半数组织培养感染剂量为tcid502.34×108。
优选地,步骤(6)所述的rt-pcr扩增程序为:95℃15min下进行1个循环,95℃10s和60℃32s进行40个循环。
优选地,步骤(8)所述的内参基因的rt-pcr分析的具体步骤为:(1)15个内参基因引物pcr扩增;(2)15个内参基因的表达水平确定;(3)bestkeeper、genorm和normfinder软件对肾阳虚外感模型15个内参基因稳定性表达分析;(4)15个内参基因两两组合t检验;(5)内参基因稳定性验证;(6)得grcc10+ppia组合基因为肾阳虚外感小鼠模型的稳定内参基因。
优选地,步骤(5)所述的用于内参基因稳定性验证所选的目标基因为tlr3、tlr4、tle7、rig-1;目标基因的引物序列依次如seqidno.31-38所示。
本发明在软件计算得出的单参考基因和双参考基因组合在目的基因的表达水平上与我们所筛选出的双参考基因具有一定差异,甚至出现相反的结果,因此bestkeeper、genorm和normfinder等软件计算的最佳参考基因和双参考基因并不能作为最稳定的参考基因。本研究在双参考基因的筛选过程中,首先通过t检验筛选出p>0.05的双基因组合,在此基础上,进一步比较两两组合后正常组和模型组差值的绝对值,以差值最小的绝对值作为两两内参基因的最优组合。通过目标基因的验证表明内参基因选择的重要性。
本研究选择了15个常用的内参基因:基因丰富簇c10基因(generichcluster,c10gene,grcc10)、β-肌动蛋白(beta-actin,actb)、3-磷酸甘油脱氢酶(glyceraldehyde-3-phosphatedehydrogenase,gapdh)、真核翻译起始因子4h(eukaryotictranslationinitiationfactor4h,eif4h)、神经前体细胞表达发育下调基因8(neuralprecursorcellexpresseddevelopmentallydown-regulatedgene8,nedd8)、环指蛋白187(ringfingerprotein187,rnf187)、酪氨酸3-单加氧酶/色氨酸5-单加氧酶激活蛋白ε肽(tyrosine3-monooxygenase/tryptophan5-monooxygenaseactivationprotein,epsilon,ywhae)、β-葡萄糖醛酸(beta-glucuronidase,gusb)、α-微管蛋白(alpha-tubulin,tuba)、β-2微球蛋白(beta-2microglobulin,β2m)、18s核糖体rna(18sribosomalrna,18srrna)、核糖体蛋白l13(ribosomalproteinl-13,rpl13)、泛素c(ubiquitinc,ubc)、核糖体蛋白l32(ribosomalproteinl32,rpl32)、肽酰脯氨酰异构酶a(peptideacylpreservedammoniaacylisomerasea,ppia)。观察了这些基因在肾阳虚外感(流感病毒h1n1)过程中的表达变化,筛选适用于此阶段的稳定表达的内参基因。
有益效果
本发明首次发现了针对肾阳虚外感病证最合适的qpcr内参基因组合为grcc10和ppia。
本发明引入了统计分析的组间t检验方法,对15种候选的参考基因进行两两组合,并对组合后的双参考基因的表达水平进行组间差异性统计,能高效、准确地筛选到合适的双内参基因组合,最终得到grcc10+ppia组合基因在正常组和模型组之间差异最小,可作为肾阳虚外感小鼠模型的稳定内参基因,为后续在该病证结合模型上的开展系列目标基因表达研究奠定基础。
附图说明
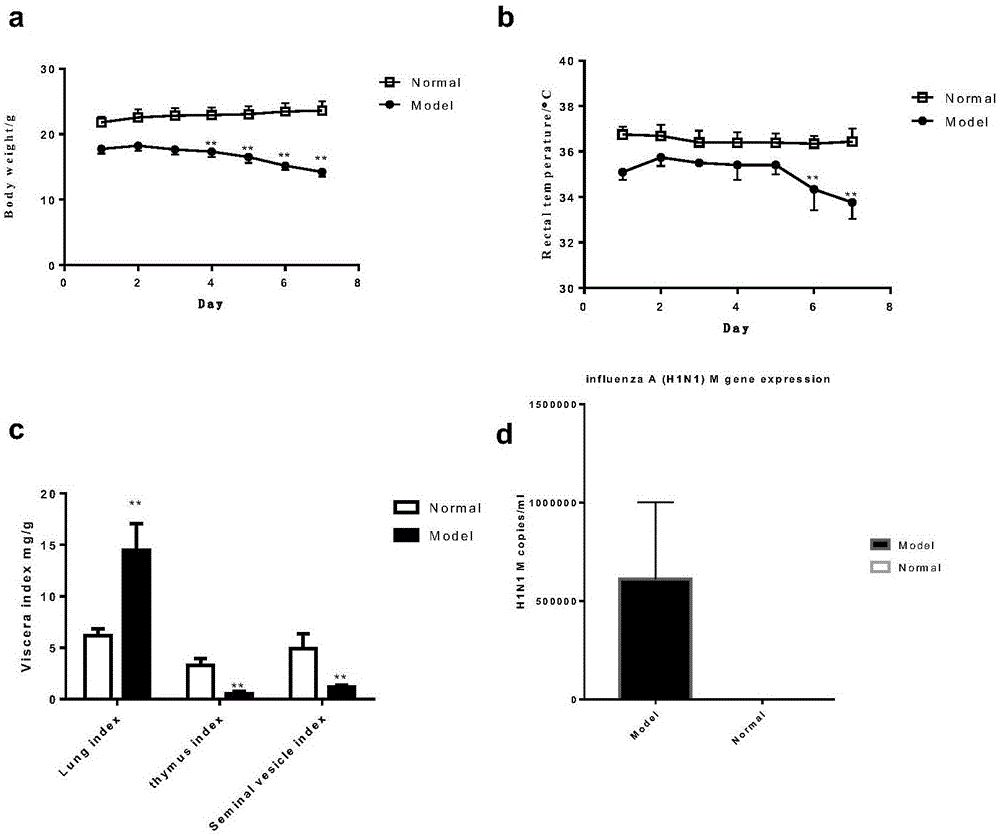
图1为正常组和模型组体重肛温、脏器指数及病毒载量;
图2为肾阳虚外感模型中15个参考基因的表达水平;
图3为bestkeeper、genorm和normfinder对肾阳虚外感模型中15个参考基因的稳定性表达分析;
图4为目标基因tlr3、tlr4、tle7、rig-1的相对表达量。
具体实施方式
下面结合附图对本发明原理和特征进行描述,所举实施例只用于解释本发明,并非限定本发明的范围。
实施例1
1材料与方法
1.1动物
spf级balb/c雄性小鼠,体重量18-20g,购买自济南朋悦实验动物繁育有限公司,许可证号:scxk(鲁)20140007。
1.2试药
苯甲酸雌二醇(批号:140627,购自宁波第二激素厂,2mg/ml)、h1n1流感病毒(北京地方株引自中国cdc病毒研究所),0.9%氯化钠注射液(辰欣药业股份有限公司,批号:1601042161),rnastore样品保存液-dp408、动物组织总rna提取试剂盒-dp431(rnapreppuretissuekit)、cdna反转录试剂盒-kr106、sybrgreen荧光定量pcr试剂盒-fp205(天根公司)。
1.3仪器
quawell超微量紫外分光光度计(usa),荧光定量pcr仪(cfxconnectreal-timesystem,伯乐),肛温测试器:bat-12微探头温度计(美国physitempinstruments,bat-12),小动物麻醉机(美国midmark公司,型号:vmr)。
1.4引物
本实验中选择了15个候选参考基因。候选基因是actb,β2m,gapdh,gusb,tuba,grcc10,eif4h,rnf187,nedd8,ywhea,18srrna,rpl13,ubc,rpl32,ppia。使用ncbiblast(http://blast.ncbi.nlm.nih.gov/blast.cgi)鉴定的核苷酸序列设计引物,上下游引物序列依次如seqidno.3-32所示;引物购自北京六合华大技术有限公司,见表1。
2实验方法
2.1动物分组
20只balb/c小鼠,根据体重、肛温随机分为2组,正常组、模型组。
2.2造模
2.2.1复制肾阳虚模型
参考文献相同性味不同归经中药对雌二醇致肾阳虚小鼠的影响中的方法以4ml/kg腹腔注射2mg/ml苯甲酸雌二醇稀释液,每日1次,连续7d,复制肾阳虚模型,测定自主活动、肛温、游泳时间,以此判定模型成功与否。
2.2.2建立肾阳虚外感模型
参照文献肾阳虚外感小鼠模型建立及麻黄细辛附子汤干预研究中的方法并改变病毒滴鼻剂量,给予麻醉后的肾阳虚小鼠滴鼻接种流感病毒h1n1鸡胚尿囊液(tcid502.34×108)20μl/只,正常组滴鼻等体积的生理盐水,接种病毒的小鼠隔离喂养,自由进食进水6d,解剖处死。
2.3指标测定
2.3.1各组小鼠体重、肛温检测每日测定小鼠的体重、肛温。
2.3.2脏器指数
解剖后取肺组织、心脏、肝脏,肾脏,用生理盐水冲洗后称重,用于计算脏器指数,称重后的组织样本以1:10比例浸入rnastore。
2.4realtimepcr检测肺组织中h1n1的mrna的表达
2.4.1总rna提取
称取10-20mg组织,按动物组织总rna提取试剂盒方法提取其总rna。通过quawell超微量紫外分光光度计测定rna的产率和纯度,将吸光度比od260/280在1.8-2.1之间的样品用于进一步分析。
2.4.2反转录合成cdna.
按照cdna反转录试剂盒方法,使用20μl逆转录系统42℃,3min去除基因组dna污染,将总rna与反转录酶fastquantrtenzyme,42℃,15min合成第一链cdna。cdna存储在-20℃备用。
2.4.3real-timepcr
对于每个反应,20μl混合物含有2μlcdna,正向和反向引物序列依次如seqidno.1-30所示,各6pmol、10μl2×superrealpremixplus(含sybrgreeni)。扩增程序如下:95℃15min下进行1个循环,95℃10s和60℃32s进行40个循环。扩增后,加热变性得到pcr产物的熔解曲线以验证扩增特异性。
使用标准曲线计算每个引物对的指数期qpcr效率(反转录的cdna进行4倍连续稀释),将每个连续稀释的平均阈值循环(ct)值对对数作图。根据方程式e=10(-1/斜率)×100计算cdna稀释因子,其中斜率是线性回归线的梯度。
2.4.4h1n1病毒定量
h1n1流感病毒m基因采用sybrgreen染料法的real-timepcr检测。h1n1流感病毒全长的m基因标准曲线采用基于引物(f:5’-ctgagaagcagatactgggc-3’,r:5’-ctgcattgtctccgaagaaat-3’)的real-timepcr扩增。pcr产物克隆到ptarget质粒,采用megashortscript试剂盒用于体外转录。h1n1流感病毒m基因的绝对拷贝数通过标准曲线计算。
2.5验证引物
基于在先前研究中作为内源对照基因的共同使用,选择了15个候选参考基因,候选基因分别是actb,β2m,gapdh,gusb,tuba,grcc10,eif4h,rnf187,nedd8,ywhea,18srrna,rpl13,ubc,rpl32和ppia。
用于验证所选参考基因的引物如下:tlr3-f:caggatacttctcgcctt和tlr3-r:tggccgctgagtttgttttc;tlr4-f:catggatcagaaactcagcaaagtc和tlr4-r:catgccatgccttgtcttca;tlr7-f:ctggagttcagcaaccatt和tlr7-r:gttatcaccgctccgctccatagaa;rig-1-f:gcagggggggggggttactgtggactttgtg和rig-1-r:tgccatctctcctttgtgtct。
2.7数据分析
这些数据通过三个常见的统计程序(即genorm、normfinder和bestkeeper)用来评估参考基因的稳定性。根据公式:2−δct(ct=对应的ct值-最小ct)将ct值转换为非标准化相对量。genorm和normfinder的计算基于这些转换的相对量;原始的ct值由bestkeeper软件直接分析。
2.8统计学分析
采用spss22.0软件对实验数据进行组间t检验,*p<0.05和**p<0.01认为具有统计学意义。
3结果
3.1体重、肛温、脏器指数及病毒载量
正常组小鼠皮毛干爽光滑,精神状态良好,活泼好动,体质量渐增。模型组小鼠感染h1n1病毒24h后,症状初期为不活泼,挠鼻子,精神萎靡,弓背萎缩等症状,体质量与正常组小鼠相比有显著性差异,且肛温明显低于正常组小鼠(图1a,b)。模型肺指数显著升高,精囊腺指数明显降低(图1c),同时模型小鼠的肺组织的流感病毒h1n1的m基因绝对拷贝数6.1×106/ml以上,表明肾阳虚外感模型构建成功(图1d)。
3.2参考基因引物pcr扩增效率及特异性
qrt-pcr反应后制作15个参考基因的标准曲线,结果显示,各相关系数r2均大于0.990,15对参看基因的扩增效率在91%-98.9%之间(表1)。上述结果表明,各参考基因引物特异性均符合qrt-pcr要求,可用于参考基因的稳定性评价。
表1qpcr引物及基因序列信息
3.3候选参考基因的表达水平
15个候选参考基因在肾阳虚外感模型小鼠中的ct值位于13.861-23.572之间,平均值为18.591。其中actb表达水平最高,ct值最小,为13.861。gusb的表达水平最低,ct值最大,为23.572(图2a)。15个候选基因的ct值经标准化处理后进行相对定量,分别用t检验统计学分析每个候选内参基因的在正常组和模型组之间的显著性差异。结果表明,这15个候选内参基因相对表达量在正常组和模型组之间都具有显著性差异(p<0.05,或p<0.01)(图2b)。
3.4bestkeeper、genorm和normfinder软件对肾阳虚外感模型参考基因的稳定性表达分析
由图可知,这三个常用的内参基因筛选软件所得出的不同参考基因的稳定性表达并不一致,经bestkeeper软件分析可发现,nedd8基因的稳定性最好,而β2m基因的稳定性最差;genorm软件分析发现,ywhea和tuba基因的稳定性最好,而β2m基因的稳定性最差;normfinder软件分析发现,18srrna基因的稳定性最好,而β2m基因的稳定性最差(图3)。另外,normfinder软件分析得到的最佳的双参考基因为eif4h+rpl32(表2)。
表2normfinder计算结果
3.5参考基因两两组合t检验
采用双基因两两组合的方法,并对两两组合的双基因在正常组和模型组之间的基因表达差异进行t检验,有15种组合的p值大于0.05以上,这些组间没有差异的两两基因组合又进行了各个组合的正常组与模型组差值的绝对值比较,我们认为以差值的绝对值最小的组合可作为双参考基因的最优化组合。实验结果显示,grcc10+ppia两参考基因组合的差值最小(0.009),故grcc10+ppia组合可作为肾阳虚外感病证肺组织标本内参的最优选择(表3、表4)。
表315种内参基因两两组合的t检验(p值)
表4筛选出的15种参考基因两两组合t检验p值及平均值的差值
3.6参考基因稳定性验证
为了验证双参考基因的稳定性和可靠性,我们选择了4个验证基因来分析靶基因的相对表达水平:toll样受体3(tlr3),toll样受体4(tlr4),toll样受体7(tlr7)和细胞溶质视黄酸诱导基因-1(rig-1)。结果发现,选用双参考基因grcc10+ppia与eif4h+rpl32及在不同软件中表现稳定的单参考基因(nedd8、ywhea、18srrna)在靶基因tlr3、tlr7、rig-1的表达水平上均出现了一定的差异,而在tlr4基因的表达上,eif4h+rpl32组合参考基因和18srrna在表达水平上出现相反的结果(见图4)。
在分子生物学研究中,基因表达分析是研究基因最常用的策略之一。qrt-pcr因其具有准确性而广泛用于相对基因的表达。在这一技术中,选择稳定表达的基因作为参考基因是至关重要的。细胞、组织或者其他应激处理后都能以恒定水平表达,并且具有与目标基因相当的表达水平。本研究发现,使用不同计算软件得出的最佳参考基因各不相同,且在肾阳虚外感小鼠模型中,使用一种参考基因无法准确判定靶向基因的表达水平,故对15种候选的参考基因进行两两组合,并对组合后的双参考基因的表达水平进行组间差异性统计,最终得到grcc10+ppia组合基因在正常组和模型组之间差异最小。通过验证目标基因tlr3、tlr4、tlr7和rig-1表明,软件计算得出的单参考基因和双参考基因组合在目的基因的表达水平上与我们所筛选出的双参考基因具有一定差异,甚至出现相反的结果,因此bestkeeper、genorm和normfinder等软件计算的最佳参考基因和双参考基因并不能作为最稳定的参考基因。本研究在双参考基因的筛选过程中,首先通过t检验筛选出p>0.05的双基因组合,在此基础上,进一步比较两两组合后正常组和模型组差值的绝对值,以差值最小的绝对值作为两两内参基因的最优组合。通过目标基因的验证表明内参基因选择的重要性。本研究发现的grcc10+ppia组合基因可作为肾阳虚外感小鼠模型的稳定内参基因,为后续在该病证结合模型上的开展系列目标基因表达研究奠定基础。
序列表
<110>山东中医药大学
<120>一种肾阳虚外感小鼠模型肺组织qpcr内参基因组合及其筛选方法
<160>38
<210>1
<211>23
<212>dna
<213>人工序列
ccttcttgggtatggaatcctgt23
<210>2
<211>26
<212>dna
<213>人工序列
cactgtgttggcatagaggtctttac26
<210>3
<211>18
<212>dna
<213>人工序列
catggctcgctcggtgac18
<210>4
<211>22
<212>dna
<213>人工序列
cagttcagtatgttcggcttcc22
<210>5
<211>19
<212>dna
<213>人工序列
tgcaccaccaactgcttag19
<210>6
<211>19
<212>dna
<213>人工序列
ggatgcagggatgatgttc19
<210>7
<211>19
<212>dna
<213>人工序列
ccgacctctcgaacaaccg19
<210>8
<211>21
<212>dna
<213>人工序列
gcttcccgttcataccacacc21
<210>9
<211>19
<212>dna
<213>人工序列
tgtcctggacaggattcgc19
<210>10
<211>19
<212>dna
<213>人工序列
ctccatcagcagggaggtg19
<210>11
<211>18
<212>dna
<213>人工序列
gcggaggtgattcaagcg18
<210>12
<211>18
<212>dna
<213>人工序列
tgaccaggcgggcaaact18
<210>13
<211>18
<212>dna
<213>人工序列
ccttggctcgggattgtc18
<210>14
<211>20
<212>dna
<213>人工序列
tccgcattggagatggatta20
<210>15
<211>18
<212>dna
<213>人工序列
ctggcaccacccttcatc18
<210>16
<211>18
<212>dna
<213>人工序列
acaagcccgagcacattc18
<210>17
<211>20
<212>dna
<213>人工序列
tgggaaggagattgagatag20
<210>18
<211>19
<212>dna
<213>人工序列
ttgcttgccactgtagatg19
<210>19
<211>18
<212>dna
<213>人工序列
cccattcgtttaggtctt18
<210>20
<211>18
<212>dna
<213>人工序列
tccacagcgtcaggttat18
<210>21
<211>21
<212>dna
<213>人工序列
ttgacggaagggcaccaccag21
<210>22
<211>21
<212>dna
<213>人工序列
gcaccaccacccacggaatcg21
<210>23
<211>20
<212>dna
<213>人工序列
gtacgctgtgaaggcatcaa20
<210>24
<211>20
<212>dna
<213>人工序列
atcccatccaacaccttgag20
<210>25
<211>20
<212>dna
<213>人工序列
gcccagtgttaccaccaaga20
<210>26
<211>20
<212>dna
<213>人工序列
cccatcacacccaagaaca19
<210>27
<211>17
<212>dna
<213>人工序列
gaactggcggaaaccca17
<210>28
<211>20
<212>dna
<213>人工序列
ggatctggcccttgaacctt20
<210>29
<211>20
<212>dna
<213>人工序列
cgcttgctgcagccatggtc20
<210>30
<211>20
<212>dna
<213>人工序列
cagctcgaaggagacgcggc20
<210>31
<211>18
<212>dna
<213>人工序列
caggatacttctcgcctt18
<210>32
<211>20
<212>dna
<213>人工序列
tggccgctgagtttgttttc20
<210>33
<211>25
<212>dna
<213>人工序列
catggatcagaaactcagcaaagtc25
<210>34
<211>20
<212>dna
<213>人工序列
catgccatgccttgtcttca20
<210>35
<211>19
<212>dna
<213>人工序列
ctggagttcagcaaccatt19
<210>36
<211>25
<212>dna
<213>人工序列
gttatcaccgctccgctccatagaa25
<210>37
<211>31
<212>dna
<213>人工序列
gcagggggggggggttactgtggactttgtg31
<210>38
<211>19
<212>dna
<213>人工序列
tgccatctctcctttgtgtct21
- 还没有人留言评论。精彩留言会获得点赞!