基于InDel标记的道地川穹鉴定用引物及鉴定方法与流程
本发明属于川穹鉴定方法
技术领域:
,具体涉及一种基于indel标记的道地川穹鉴定用引物及鉴定方法。
背景技术:
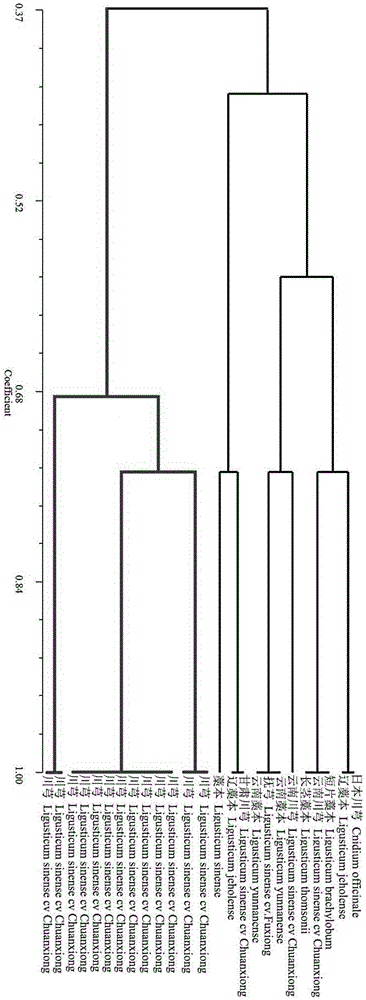
:川芎(ligusticumchuanxionghort)为伞形科藁本属植物,以干燥的根入药。其根茎含有藁本内酯、川芎嗪、阿魏酸、挥发油等多种有效药用成分,具有活血行气,祛风止痛之功效。主要用于治疗月经不调、经闭痛经、产后瘀滞腥痛、症瘕肿块、胸胁疼痛等。由于其疗效显著、用量极大,作为大宗药材,备受关注。四川、江西、云南、吉林、甘肃、贵州等省都有川芎分布。四川省川芎产量丰富,药效价值高,为川芎道地产区,其中都江堰、彭州、崇州市是主产区,产量占全国产量的90%以上。省农业厅调查统计2016年全省川芎种植面积达10万亩以上。随着大健康产业发展的深入和人们对中医药认识的不断提高,我国中药产业对中药材的需求不断增加,很多中药材的市场价格不断上升,当中药材生产不能完全满足市场需求的时候,部分易混品流入市场,充当正品中药材,严重影响其疗效。如川产川芎疗效好,市场价格稳定,随着川芎产品开发的深入,市场对川芎的需求越来越大,也出现了部分易混品在市场上充当川芎售卖,如抚芎、云芎、东川芎等。道地药材与非道地药材来源一致或十分相近,在形态、生物药性及化学成分等特征上也具有高度相似性,导致道地药材的鉴别不易。因此迫切地需要研究利用新的方法提高道地川芎的鉴别能力。dna条形码鉴定技术为中药材的物种鉴定研究提供了方便快捷的方法,dna条形码(dnabarcoding)是利用短的,标准的dna序列来鉴定物种的新技术。dna条形码技术可有效的将不同种、属之间的物种进行鉴别,且其操作的简便性和高效性推动物种鉴定与进化研究的发展,为解决中药行业对中药材基原物种鉴定的需求,有研究分析了中国川芎和日本川芎的its和matk序列,探讨中、日了川芎物种间鉴别,其结果显示中、日川芎关系,its测序难以区分中国川芎和日本川芎。更有试验通过its2研究了川芎与羽苞藁本等藁本属的遗传差异。但在对川芎进行its2测序时发现,川芎its2序列的杂合度高,存在插入缺失的现象,难以直接测序,上述标记均不满足dna条形码基本特征。目前研究中所采用的材料种类较少,并无市场中川芎常见的混伪品品种如抚芎、云南藁本等,并且少有结合道地川芎药材的研究报道。另外,在上述研究中所使用的多为常见通用条形码,缺乏开发新条形码的创新性研究,缺少关于市场上道地川芎及其混伪品问题的有效解决方法。普通用于鉴定植物的dna条形码,由于片段长度的限制,单一序列或多序列组合的条形码在物种鉴定特别是针对近缘物种之间的鉴定存在很大的局限性。叶绿体普遍存在于植物中,而相对于核基因组以及线粒体基因组,叶绿体基因组大多为母系遗传,进化速率较慢,序列较为保守。植物叶绿体基因组作为筛选dna条形码序列的研究热点,其本身也可以作为超级条形码用于系统进化、亲缘关系以及物种鉴定研究。根据叶绿体基因组中的插入缺失位点,设计一些扩增这些插入缺失位点的pcr引物,就是cpindel标记(叶绿体插入/缺失多态性chloroplastinsertion/delectionpolymorphismcpindel)。有学者通过在15个小麦(triticumaestivum)野生近缘属种基因组dna的扩增分析发现11对indel标记引物。也有研究发现在b73玉米(zeamays)全基因组序列中发现13个共显性indel标记,利用这些共显性indel标记可以进行快速准确的玉米杂交种纯度鉴定。通过比较2种粳稻(oryzasalivasubspkeng)的基因组序列,设计出634对indel候选标记,可以在涉及籼粳亚种的基因定位和分子育种中应用。但是对于川芎的indel标记尚未见相关研究。技术实现要素:针对现有技术中存在的上述问题,本发明提供一种基于indel标记的道地川穹鉴定用引物及鉴定方法,通过开发的4对特异性引物用以鉴定道地川芎及其常见混伪品,可高效、准确、可靠地得到鉴定结果。为实现上述目的,本发明解决其技术问题所采用的技术方案是:一种基于indel标记的道地川穹鉴定用引物,包括:cinp3:f:5’-tctgacactgcacaaacagga-3’;r:5’-catcttaggcggatagcggg-3’;cinp5:f:5’-ttcttgccagaggaatggtcc-3’;r:5’-ggttgaagatcacgaggcgt-3’;cinp19:f:5’-acgtagcagacatgcggatt-3’;r:5’-cgctgaagccgctattgga-3’;cinp22:f:5’-aaagcactcgcttttcgctg-3’;r:5’-acagccgttctaaacccgaa-3’。采用上述引物对道地川穹进行鉴定的方法,包括以下步骤:(1)提取待测样品的dna;(2)采用上述引物对提取的dna进行扩增;(3)对扩增产物进行凝胶电泳检测,若能同时扩增出4种引物对应的特异性条带,则为道地川穹。进一步地,从待测样品的新鲜叶片组织中提取dna。进一步地,扩增体系为20μl,包括:dna2μl、上下游引物各2μl、ddh2o6μl、2×taqpcrmastermix10μl;扩增程序:95℃预变性5min,95℃变性1min30s,58℃复性30s,72℃延伸30s,25个循环;最后72℃延伸10min。本发明提供的基于indel标记的道地川穹鉴定用引物及鉴定方法,具有以下有益效果:本发明利用12个伞形科植物的叶绿体全基因组序列和道地川穹基因组序列进行对比,获得插入/缺失差异片段所设计的4对特异dna引物,采用该引物来鉴定道地川芎,可根据引物扩增的特异性条带进行判断,即是否能够同时扩增出这4种引物对应的特异性条带,就能确定待测样品是否是道地川穹,该方法简便,样品用量少,准确性高,适合应用于大批量的品种鉴定,并在短时间内能精确鉴定道地川芎。附图说明图1为道地川芎及其混伪品种的聚类图。具体实施方式实施例11、实验材料本发明选取了藁本属的26个物种为材料,具体信息如下表:2、indel引物设计从genbank数据库中,下载12个伞形科植物的叶绿体全基因组序列,通过mafft在线比对软件进行比对,在细叶藁本的叶绿体全基因组序列上,找到插入(in)缺失(del)位点,在indel位点两侧设计引物,引物长度大于20bp,以25bp为最优选择,tm为50℃-60℃,以56℃为最优选择,gc含量为55%-60%之间,pcr产物大小在100-500bp之间。伞形科植物genbank登录号如下表:名称拉丁学名genbank登录号细叶藁本ligusticumtenuissimumnc_029394.1有喙欧芹anthriscuscerefoliumnc_015113.1阿尔泰柴胡bupleurumfalcatumnc_027834.1大齿山芹ostericumgrosseserratumnc_028618.1东当归angelicaacutilobanc_029391.1白芷angelicadahuricanc_029392.1朝鲜当归angelicagigasnc_029393.1野胡萝卜daucuscarotanc_008325.1茴香foeniculumvulgarenc_029469.1莳萝anethumgraveolensnc_029470.1芫荽coriandrumsativumnc_029850.1葛缕子carumcarvinc_029889.1所设计得到的24对特异dna引物情况如下所示:从24对indel引物中挑选出23对引物在26份藁本属材料中均能产生清晰条带的引物,通过对细叶藁本叶绿体基因注释及indel标记位置可知,在细叶藁本基因组上开发的引物标记分布广泛,覆盖了lsc区、ssc区和ir区。3、鉴定方法(1)采用ctab法提取实验材料新鲜叶片组织的总dna;(2)用上述24对引物对dna进行扩增;扩增体系为20μl,包括:dna2μl、上下游引物各2μl、ddh2o6μl、2×taqpcrmastermix10μl;扩增程序:95℃预变性5min,95℃变性1min30s,58℃复性30s,72℃延伸30s,25个循环;最后72℃延伸10min。(3)凝胶电泳检测,若能同时扩增出4种引物对应的特异性条带,则为道地川穹。通过对上述26个样本进行检测,只有道地川穹能同时扩增出4种引物对应的特异性条带,其余的样本均无法同时扩增出4种引物对应的特异性条带。此外,本发明还对扩增产物进行毛细管电泳检测,具体过程为:①取fa分离胶(fadsdnagel35-500bp)80ml加入5ul的插入染料(intercalatingdye),充分混匀,放入仪器试剂室gel1的锥形瓶中。确保液体管插入到瓶子的底部,勿吸入气泡,否则会引起压力性误差。gel1放好后,立即更新控制软件solutionlevels,输入正确的gel1体积。②准备毛细管清洗液(capillaryconditioningsolution)加入200ml的超纯水,放入仪器试剂室相应的锥形瓶中,其他过程同(1)。③取20ml的上样缓冲液(5x30dnainletbuffer)加入80ml超纯水,混匀并静止10min。将配好的液体加入96深孔板中,每孔1ml,切勿使孔内产生气泡。将96深孔板放入仪器的1号抽提中。④准备maker板,取96孔板一个,向其加入适量的maker液体,每孔24ul,并向其加入一滴石蜡油,适当离心,切勿使其中产生气泡。将配置好的maker板放入3号抽提中。⑤将pcr产物10ul,依次加入96孔反应板中,向其中加入14ul的1×te并混匀,最后一个孔加入24ul的300-1500bpdnaladder,3000r/min,离心1min。离心后静止片刻,切勿使孔内产生气泡。将样品板放入4号抽提中。⑥启动仪器,开始跑电泳。⑦根据毛细管电泳的结果,设置相应的参数,校对电泳峰图,并读取有效峰值。将有效峰值转换为谱带的0/1系统进行记录,有带计为1,无带计为0,数据缺失计为无数据。运用ntsys软件中qualitativedate计算材料间的遗传相似系数(sm系数),并采用upgma法进行聚类分析,绘制树状聚类图,结果见图1。由图1可知,道地川穹聚类为一个分支,可以准确地鉴定出道地川穹品种。本发明利用24对indel标记对26份道地川芎及其常见混伪品材料进行了分子标记的统计及聚类分析,结果显示,在4对indel引物(cinp3、cinp5、cinp19和cinp22)组合中,可以明显区分道地川穹及其混伪品为两个分支,26份道地川芎及其混伪品的遗传相似系数在0.37-1.0之间,表明品种间亲缘关系较为亲近。如图1所示,在相似系数为0.43,供试的26份藁本属材料划分为四大类。其中第一类可分为2个亚类,a类包括:日本川芎、辽藁本、长颈藁本和云南川芎2号。b类包括:云南川芎1号、藁本、短片藁本、抚芎和云南红杆藁本。第二类包括:甘肃川芎、辽藁本和云南绿杆藁本。第三类又分为两个亚类,a类包括:四川产川芎材料18、45号。b类包括:四川产川芎材料44、46、47、48、49、50、51、52、53。第四类包括:四川产川芎材料42、43、54号。从聚类结果可知:四川产川芎和其他藁本属材料可以明显看到分为两个分支。四川产川芎单独聚合为一类,遗传相似系数为0.68。其他藁本属材料均聚合为一类,遗传相似系数为0.43,其中日本川芎与辽藁本聚合为一个分支,短片藁本、长颈藁本和云南川芎2号聚合为一个分支,云南川芎1号、藁本、抚芎和云南红杆藁本聚合为一个分支,从形态特征上看,除了茎秆形状不同外,其叶型具有很相似的形态特征。甘肃川芎、辽藁本和云南绿杆藁本聚合为一个分支,亲缘关系较近。通过毛细管电泳检测、ntsys软件中qualitativedate计算材料间的遗传相似系数以及采用upgma法进行聚类分析,绘制树状聚类图,更进一步证明了本发明提供的4种引物可以准备地辨别出道地川穹和其他品种混伪品材料。序列表<110>四川省农业科学院经济作物育种栽培研究所四川农业大学<120>基于indel标记的道地川穹鉴定用引物及鉴定方法<160>48<170>siposequencelisting1.0<210>1<211>21<212>dna<213>人工序列(artificialsequence)<400>1acagaaagaagacccctttgc21<210>2<211>23<212>dna<213>人工序列(artificialsequence)<400>2acagccctttgtctttttgttgt23<210>3<211>25<212>dna<213>人工序列(artificialsequence)<400>3acaaacaaagtaaaaagtaacccgc25<210>4<211>21<212>dna<213>人工序列(artificialsequence)<400>4tggtgattccagagacgactt21<210>5<211>21<212>dna<213>人工序列(artificialsequence)<400>5tctgacactgcacaaacagga21<210>6<211>20<212>dna<213>人工序列(artificialsequence)<400>6catcttaggcggatagcggg20<210>7<211>22<212>dna<213>人工序列(artificialsequence)<400>7agtgtagtgatgctatccaccc22<210>8<211>20<212>dna<213>人工序列(artificialsequence)<400>8ttcaaatcccgtctccgcaa20<210>9<211>21<212>dna<213>人工序列(artificialsequence)<400>9ttcttgccagaggaatggtcc21<210>10<211>20<212>dna<213>人工序列(artificialsequence)<400>10ggttgaagatcacgaggcgt20<210>11<211>22<212>dna<213>人工序列(artificialsequence)<400>11ccaaaaagcccatttgactcct22<210>12<211>21<212>dna<213>人工序列(artificialsequence)<400>12caaatggggagtctttggtca21<210>13<211>20<212>dna<213>人工序列(artificialsequence)<400>13aatccgaactcaaacgcaga20<210>14<211>20<212>dna<213>人工序列(artificialsequence)<400>14ttccccggagaatgaactcc20<210>15<211>21<212>dna<213>人工序列(artificialsequence)<400>15ccacccttcctttcgtaggag21<210>16<211>20<212>dna<213>人工序列(artificialsequence)<400>16caccgggggcttagttgtat20<210>17<211>21<212>dna<213>人工序列(artificialsequence)<400>17cgtaaggtctgagacaaccca21<210>18<211>23<212>dna<213>人工序列(artificialsequence)<400>18acagagttgcaattatccagtgc23<210>19<211>20<212>dna<213>人工序列(artificialsequence)<400>19tgcgtgcgacacaatctact20<210>20<211>20<212>dna<213>人工序列(artificialsequence)<400>20ttgcccgggaattgagacag20<210>21<211>20<212>dna<213>人工序列(artificialsequence)<400>21acggagctggaactgctaac20<210>22<211>21<212>dna<213>人工序列(artificialsequence)<400>22tccctggagagatggttcact21<210>23<211>21<212>dna<213>人工序列(artificialsequence)<400>23tttcccattccttagccggaa21<210>24<211>21<212>dna<213>人工序列(artificialsequence)<400>24gcttgtactcttggagggtcc21<210>25<211>24<212>dna<213>人工序列(artificialsequence)<400>25acggtatttctgaacaagttcctg24<210>26<211>20<212>dna<213>人工序列(artificialsequence)<400>26gcagttccagctccgtatga20<210>27<211>21<212>dna<213>人工序列(artificialsequence)<400>27gctaggcaggaggatagcaag21<210>28<211>20<212>dna<213>人工序列(artificialsequence)<400>28cttatgtgtttccacgcccc20<210>29<211>20<212>dna<213>人工序列(artificialsequence)<400>29atgcttttcccggaggtctg20<210>30<211>20<212>dna<213>人工序列(artificialsequence)<400>30acacatcccattgcaccgat20<210>31<211>24<212>dna<213>人工序列(artificialsequence)<400>31agtcccaattttgcaaagatgaat24<210>32<211>24<212>dna<213>人工序列(artificialsequence)<400>32tggttaactccgtctataaacctt24<210>33<211>23<212>dna<213>人工序列(artificialsequence)<400>33aaagcacttcgataagaccccat23<210>34<211>20<212>dna<213>人工序列(artificialsequence)<400>34cggggctacttttgcccttg20<210>35<211>20<212>dna<213>人工序列(artificialsequence)<400>35tggagatgggggagtctttt20<210>36<211>20<212>dna<213>人工序列(artificialsequence)<400>36ggcttttgtcggttgggttg20<210>37<211>20<212>dna<213>人工序列(artificialsequence)<400>37acgtagcagacatgcggatt20<210>38<211>19<212>dna<213>人工序列(artificialsequence)<400>38cgctgaagccgctattgga19<210>39<211>20<212>dna<213>人工序列(artificialsequence)<400>39tcctttcggtttcacgcttt20<210>40<211>20<212>dna<213>人工序列(artificialsequence)<400>40tggagcacctaacaacgcat20<210>41<211>20<212>dna<213>人工序列(artificialsequence)<400>41ggataacggtaaccccacca20<210>42<211>20<212>dna<213>人工序列(artificialsequence)<400>42tggactctcatgtcgatccg20<210>43<211>20<212>dna<213>人工序列(artificialsequence)<400>43aaagcactcgcttttcgctg20<210>44<211>20<212>dna<213>人工序列(artificialsequence)<400>44acagccgttctaaacccgaa20<210>45<211>21<212>dna<213>人工序列(artificialsequence)<400>45gcttgtactcttggagggtcc21<210>46<211>20<212>dna<213>人工序列(artificialsequence)<400>46cgggtttaaccgctcgtttt20<210>47<211>20<212>dna<213>人工序列(artificialsequence)<400>47tgatacgggccttttccaca20<210>48<211>24<212>dna<213>人工序列(artificialsequence)<400>48acacggattcctatttctcaacga24当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1