小麦小穗数QTL连锁的SNP分子标记及其应用的制作方法
本发明涉及分子生物学及作物遗传育种领域,具体涉及小麦小穗数qtlqsns.sau-2d.1连锁的snp分子标记及其应用。
背景技术:
小麦是世界上最重要的粮食作物之一,同时也是我国重要的粮食作物,其常年种植的面积在2000万公顷以上,约占所有粮食作物面积的27%;总产量超过1亿吨,约占所有粮食作物产量的22%。小麦产量取决于粒重,每穗粒数和单位面积穗数。穗粒数是产量构成因素中最活跃的因素,可调节程度大,穗粒数的多少由小穗数、分化的小花数、小花结实率决定。小穗数是形成小花数的前提,增加的小穗数是提高穗粒数的基础。
小麦产量性状是复杂的数量性状,受多个数量性状基因位点(quantitativetraitlocus,qtl)控制,具有遗传力低、受环境影响大、选择难度高的特性,所以在选育过程中,传统的育种方法存在时间长、消费大、成本高、成果小的问题。分子标记辅助育种,不依赖于表型选择,即不受环境、基因互作、基因与环境互作等因素的影响,而是直接对基因型进行选择,因而能大大提高育种效率。
单核苷酸多态性(singlenucleotidepolymorphism,snp)指的是基因组内dna某一特定核苷酸位置上存在转换、颠换、插入、缺失等变化而引起的dna序列多态性。其技术是利用己知序列信息来比对寻找snp位点,再利用发掘的变异位点设计特异性的引物来对基因组dna或cdna进行pcr扩增,得到基于snp位点的特定的多态性产物,最后利用电泳技术分析产物的多态性。snp标记的优点是数量多,分布广泛;在单个基因和整个基因组中分布不均匀;snp等位基因频率容易估计。
kasp是由lgc公司(laboratoryofthegovernmentchemist)(http://www.lgcgenomics.com)研发的竞争性等位基因特异性pcr技术(kompetitiveallelespecificpcr,kasp)具有低成本、高通量特点的新型基因分型技术,通过引物末端碱基的特异匹配来对snp以及indel位点进行精准的双等位基因分型,在水稻、小麦、大豆等作物的分子标记辅助选择中得到广泛应用。
此前有学者对小穗数进行了qtl定位,发现与之相关的qtl在小麦中广泛存在,然而目前与小麦小穗数性状相关可用于实际分子育种的紧密连锁的分子标记却并不多。因此研究获得有关小穗数的qtl或基因,利用分子生物学技术,增加小穗数,进而增加穗粒数,最终达到选育增产小麦新品种的目的,在小麦育种工作中意义重大。
技术实现要素:
本发明的目的在于提供与小麦小穗数qtlqsns.sau-2d.1紧密连锁的分子标记。
本发明的另一目的在于提供所述分子标记的荧光定量pcr引物。
本发明的第三个目的在于提供上述小麦小穗数qtlqsns.sau-2d.1紧密连锁的分子标记的应用。
本发明的目的是通过以下技术方案实现的:
基于以上目的,申请人利用多小穗数小麦品系‘20828’为母本,以小麦品种‘cn16’为父本杂交,得到杂种f1,f1代单株自交获得f2,在f2使用单穗传法,一直到f8代,获得含有199个系的重组自交系,构成遗传作图群体。对重组自交系群体小穗数表型鉴定,提取亲本‘20828’、‘cn16’和重组自交系群体植株dna,本研究使用小麦55ksnp芯片来定位小麦小穗数qtl。小麦55ksnp芯片是在小麦660ksnp芯片的基础上开发的一款经济型中密度snp芯片。芯片包含55,000个左右的小麦snp标记,均匀分布在21条染色体上,每条染色体上平均有2600个标记,标记间的平均遗传距离约为0.1cm,平均物理距离小于300kb,适合于一般的种质资源多样性分析、遗传作图与新基因发掘、比较基因组分析、品种注册与鉴别(指纹分析)。
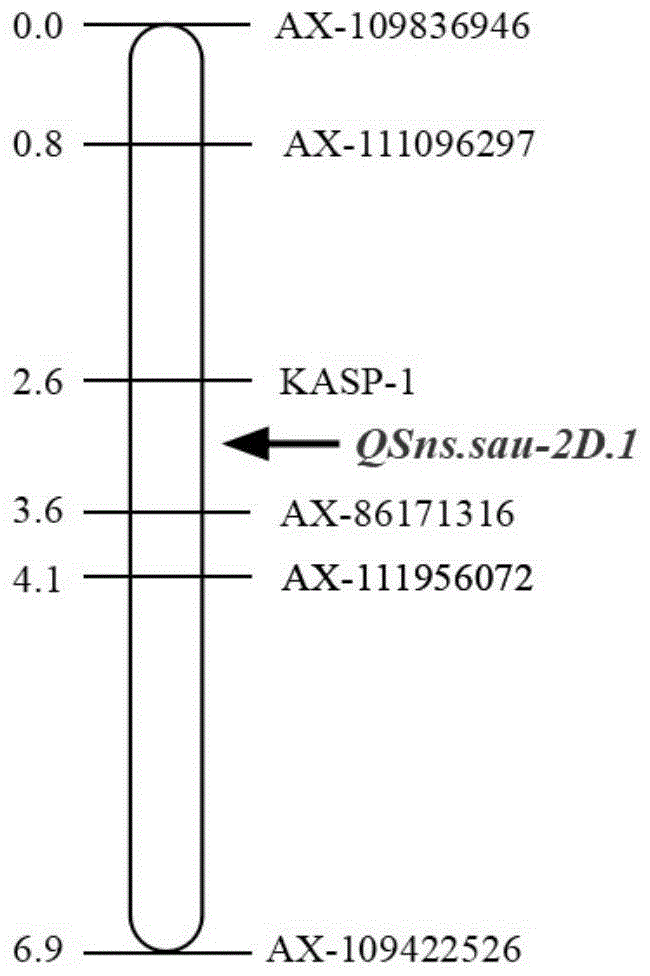
根据55ksnp芯片数据,利用joinmap4.0构建遗传图谱。结合群体的小穗数表型数据,用qtlicimapping4.0中的完备区间作图法(inclusivecompositeintervalmapping-add,icim-add),设置阀值lod≥2.5的条件下,用2016-2018三个年份共8个生态点及8个生态点小穗数的blup(最佳线性无偏预测,bestlinearunbiasedprediction)值来检测qtl,在2d染色体短臂上的1cm区间定位出稳定表达的小麦小穗数主效qtlqsns.sau-2d.1,对侧翼标记进行物理定位并每隔1mbp筛选位于区间内的基因,共筛选获得40个基因,并对这些基因在亲本‘20828’和‘cn16’进行克隆,对获得多态性位点并进行分子标记的开发,共设计了15对共45条kasp引物(表1),最终得到snp分子标记kasp-1与小穗数qtlqsns.sau-2d.1紧密连锁。
本发明所述的小麦小穗数qtlqsns.sau-2d.1,来自母本‘20828’,该qtl位于小麦染色体2d短臂上,在refseqv1.0基因组版本的物理位置为33735919。本发明提供了上述小麦小穗数qtlqsns.sau-2d.1在调控小麦小穗数性状中的应用,该小麦小穗数qtlqsns.sau-2d.1与分子标记kasp-1紧密连锁,可通过3条核苷酸序列分别如seqidno.1、2和3所示的引物扩增得到。具体地,该小麦小穗数qtlqsns.sau-2d.1能够提高小麦小穗数量。小麦小穗数qtlqsns.sau-2d.1显著增加小麦小穗数,平均lod值为19.86,解释约30.78%的表型变异。
进一步地,本发明提供了小麦小穗数qtlqsns.sau-2d.1的snp分子标记kasp-1,其与小麦小穗数qtlqsns.sau-2d.1紧密连锁,位于refseqv1.0基因组版本的2d染色体短臂上,与qsns.sau-2d.1之间的遗传距离小于1cm,其前后100bp序列如seqidno.46所示,所述snp分子标记位于seqidno.46所示序列的第101bp处,其多态性为c/t。
本发明的小麦小穗数qtlqsns.sau-2d.1的分子标记kasp-1由核苷酸序列如seqidno.1~3所示的3条引物对pcr扩增获得。所述3条引物包括2个上游引物和1条通用下游引物,所述2个上游引物5’端分别修饰不同的荧光基团;3条引物的核苷酸序列分别如seqidno.1、2和3所示。
本发明提供了上述分子标记kasp-1在作物分子辅助育种中的应用。
本发明提供了上述分子标记kasp-1在培育多小穗数性状的小麦中的应用。
本发明还提供了用于检测小麦小穗数qtlqsns.sau-2d.1的snp分子标记kasp-1的特异性引物对,其上、下游引物序列分别如seqidno.1~3所示。
本发明提供了上述特异性引物对在小麦种质资源改良中的应用。
本发明提供了上述特异性引物对在小麦多小穗数材料创制中的应用。
含有上述特异性引物对的试剂盒也属于本发明的保护范围。
本发明提供了一种鉴定小麦小穗数qtlqsns.sau-2d.1的分子标记方法,以待鉴定材料的dna作为模板,用荧光定量pcr引物进行荧光定量pcr扩增,利用扩增结果进行基因型分型,将能够读取到引物seqidno.2所修饰的荧光的植株鉴定为含有小麦小穗数qtlqsns.sau-2d.1的植株。
具体地,上述的应用,包括如下步骤:
1)提取待测植株的基因组dna;
2)以待测植株的基因组dna为模板,利用扩增分子标记kasp-1的引物,在仪器cfx96real-timesystem,进行pcr扩增反应并读取荧光值;
3)检测pcr扩增产物荧光,如果能够读取引物seqidno.2所修饰的荧光,则待测植株为具有多小穗数性状的小麦资源。
上述pcr扩增的扩增体系为:5μlmastermix、三条引物seqidno:1、2和3按照10ng/μl的浓度,分别加入120μl,120μl和300μl并添加ddh2o460μl进行混合后作为混合引物使用,混合引物1.4μl、5ng模板dna、双蒸水加至总量为10μl,同时需添加至少3个独立的以双蒸水代替dna模板的空白。
上述pcr扩增的程序为:94℃预变性15min;94℃变性20s、61℃复性/延伸60s,共10个循环;94℃变性20s、55℃复性/延伸60s,共26个循环;完成后进行荧光读值。
本发明公开了位于小麦2d染色体上的与小麦小穗数连锁的分子标记kasp-1,该分子标记是小麦2d染色体长臂上小穗数qtlqsns.sau-2d.1的侧翼标记,连锁度高。该标记可用来检测小麦2d染色体上的小穗数qtl,快速筛选具有该位点的植株,进而方便进行高产小麦的分子辅助育种。本发明提供的分子标记kasp-1与小麦2d上的小穗数qtlqsns.sau-2d.1紧密连锁,可用来对小麦小穗数这一性状进行定位,从而在育种过程中淘汰小穗数较少的植株,提高育种工作效率,并为小麦小穗数基因的研究提供基础。
本发明的有益效果在于:本发明首次公开了来自小麦‘20828’的小穗数qtlqsns.sau-2d.1,位于小麦2d染色体短臂上,显著增加小麦小穗数。该qtl在小麦产量(调控小穗数)育种中具有较高的利用价值。小麦小穗数qtlqsns.sau-2d.1显著增加小麦小穗数,平均lod值为19.86,解释约30.78%的表型变异。
本发明首次公开了基于荧光定量pcr平台精确检测小麦‘20828’的小穗数qtlqsns.sau-2d.1的分子标记kasp-1,且为共显性标记,检测准确高效、扩增方便稳定。本发明公开的分子标记kasp-1与小穗数qtlqsns.sau-2d.1极显著相关,呈现共分离标记特征,用于分子标记辅助选择的准确性高,提高适应不同环境的小麦多小穗数品种的选择鉴定效率,且成功率高。
附图说明
图1为本发明实施例1中小麦小穗数qtlqsns.sau-2d.1在2d染色体上的定位。
图2为本发明实施例1中‘20828’בcn16’的重组自交系株系植株分子标记kasp-1检测的荧光读值结果;其中,hex(方形,‘20828’)荧光为有较多小穗数的株系,fam(圆形,‘cn16’)荧光为较少小穗数株系;三角形荧光为杂合株系;菱形荧光为空白对照。
图3为本发明实施例2中‘20828’×普通小麦品系‘sy95-71’的重组自交系株系植株分子标记kasp-1检测的荧光读值结果;其中,hex(方形,‘20828’)荧光为有较多小穗数的株系,fam(圆形,‘sy95-71’)荧光为较少小穗数株系;三角形荧光为杂合株系;菱形荧光为空白对照。
具体实施方式
以下实施例进一步说明本发明的内容,但不应理解为对本发明的限制。在不背离本发明精神和实质的情况下,对本发明方法、步骤或条件所作的修改或替换,均属于本发明的范围。
本发明实施例中所用的小麦种质资源均来自四川农业大学小麦研究所兰秀锦研究员种植资源库。
若未特别指明,实施例中所用的技术手段为本领域技术人员所熟知的常规手段。
实施例1小麦小穗数qtlqsns.sau-2d.1及其分子标记kasp-1的获得
(1)利用小麦品系‘20828’为母本,以小麦品种‘cn16’为父本杂交,得到杂种f1,f1代单株自交获得f2,在f2使用单穗传法,一直到f8代,获得含有199个系的重组自交系,构成遗传作图群体。
(2)重组自交系群体小穗数表型鉴定:小麦成熟期对重组自交系小穗数进行分析鉴定,去除每行两端的单株,分别收取五个长势一致的单株,并选取主穗,计算小穗数,并得出平均值,代表该株系的小穗数。
(3)55ksnp芯片分析
a)dna提取:用ctab法提取亲本‘20828’、‘cn16’和重组自交系群体植株dna。
b)使用超微量分光光度计对提取的dna进行质量检测,合格后送样至公司进行基因型分析,在本研究中双亲和作图群体的基因型分析由北京博奥晶典生物技术有限公司(http://www.capitalbiotech.com)开发的55ksnp芯片完成,该芯片市售可得。
c)连锁图谱的构建:根据55ksnp芯片数据,利用joinmap4.0构建遗传图谱。结合群体的小穗数表型数据,用qtlicimapping4.0中的完备区间作图法(inclusivecompositeintervalmapping-add,icim-add),设置阀值lod≥2.5的条件下,用2016-2018三个年份共8个生态点及8个生态点小穗数的blup(最佳线性无偏预测,bestlinearunbiasedprediction)值来检测qtl,定位出小麦小穗数qtlqsns.sau-2d.1,并计算qsns.sau-2d.1的位置和分子标记之间的遗传距离。
d)遗传图谱的密化和紧密连锁分子标记的获得:为了密化遗传图谱并获得与小穗数qtlqsns.sau-2d.1紧密连锁的分子标记,利用55ksnp芯片数据定位结果对侧翼标记进行物理定位并筛选位于区间内的基因,对这些基因在亲本‘20828’和‘cn16’进行克隆,对获得多态性位点并进行分子标记的开发,利用dnaman设计kasp引物(共设计45条,15对kasp引物,引物序列见表2),最终得到snp标记kasp-1与小穗数qtlqsns.sau-2d.1紧密连锁。
表115对kasp引物序列
e)进行分析。设计的15对kasp引物中最终得到了2个分子标记,其中kasp-1与小穗数qtlqsns.sau-2d.1紧密连锁,。结果见图1,2。
实施例2分子标记kasp-1在选择控制小穗数qtlqsns.sau-2d.1上的应用
(1)利用小穗数多的普通小麦品系‘20828’为母本,小穗数少的普通小麦品系‘sy95-71’为父本构建重组自交系,在后代株系中随机选择80个株系。
(2)对所获得的80个株系进行kasp-1标记检测,具体方法为:提取80个株系的dna;将其作为模板,以分子标记kasp-1的特异性引物对为引物进行pcr扩增并进行荧光读值,所述引物为:
fam标签上引物:(下划线部分为fam标签序列)
5’-gaaggtgaccaagttcatgcttataaaccggtcgaactcgc-3’(seqidno.1)
hex标签上引物:(波浪线部分为hex标签序列)
5’-gaaggtcggagtcaacggatttataaaccggtcgaactcgt-3’(seqidno.2)
通用下游引物:
5’-tggtgcttctccttggcgag-3’(seqidno.3)
上述pcr扩增的扩增体系为:5μlmastermix、三条引物seqidno:1、2和3按照10ng/μl的浓度,分别加入120μl,120μl和300μl并添加ddh2o460μl进行混合后作为混合引物使用,混合引物1.4μl、5ng模板dna、双蒸水加至总量为10μl,同时需添加至少3个独立的以双蒸水代替dna模板的空白。
上述pcr扩增的程序为:94℃预变性15min;94℃变性20s、61℃复性/延伸60s,共10个循环;94℃变性20s、55℃复性/延伸60s,共26个循环;完成后进行荧光读值。
荧光读值结果(见图3),将检测到与‘20828’一致的hex(蓝色)荧光的植株基因型记为a,为多小穗数型株系,同‘sy95-71’一样表现为fam(橙色)荧光的植株基因型记为b,为少小穗数型株系。各个株系基因型与6个生态点小穗数的blup值如表2所示。与含有小穗数qtlqsns.sau-2d.1的‘20828’类型相同的植株平均小穗数为25.51,极显著高于与‘sy95-71’类型的植株小穗数(平均22.45)。实际结果与预期结果一致,说明本发明的小穗数qtlqsns.sau-2d.1确实有显著增高小穗数的作用;同时本发明的分子标记kasp-1可以用与跟踪鉴定小穗数qtlqsns.sau-2d.1。
表2‘20828’בsy95-71’重组自交系kasp-1基因型与表型对应结果
虽然,上文已经用一般性说明、具体实施方式及试验,对本发明作了详尽的描述,但在本发明基础上,可以对之做出一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
序列表
<110>四川农业大学
<120>小麦小穗数qtl连锁的snp分子标记及其应用
<130>khp191110805.6
<160>46
<170>siposequencelisting1.0
<210>1
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>1
gaaggtgaccaagttcatgcttataaaccggtcgaactcgc41
<210>2
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>2
gaaggtcggagtcaacggatttataaaccggtcgaactcgt41
<210>3
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>3
tggtgcttctccttggcgag20
<210>4
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>4
gaaggtgaccaagttcatgctggagggacttcttgatggac41
<210>5
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>5
gaaggtcggagtcaacggattggagggacttcttgatggat41
<210>6
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>6
gaagaggttggcagatgatt20
<210>7
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>7
gaaggtgaccaagttcatgctcaaacacatgacatggccg40
<210>8
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>8
gaaggtcggagtcaacggattcaaacacatgacatggcca40
<210>9
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>9
tccttattgctcttcttcttc21
<210>10
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>10
gaaggtgaccaagttcatgctggttaagtgcgtttatgaca41
<210>11
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>11
gaaggtcggagtcaacggattggttaagtgcgtttatgacc41
<210>12
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>12
atgccacatcagcgaaatagg21
<210>13
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>13
gaaggtgaccaagttcatgctcctgaaagtgtagggagcgt41
<210>14
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>14
gaaggtcggagtcaacggattcctgaaagtgtagggagcgc41
<210>15
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>15
ggttccaagattgatatgat20
<210>16
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>16
gaaggtgaccaagttcatgctacaggaggggcccaacccct41
<210>17
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>17
gaaggtcggagtcaacggattacaggaggggcccaacccca41
<210>18
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>18
attcctttttttaacgttc19
<210>19
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>19
gaaggtgaccaagttcatgctcacagccaaaaaagtccag40
<210>20
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>20
gaaggtcggagtcaacggattcacagccaaaaaagtccac40
<210>21
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>21
attattttttgcttttagggt21
<210>22
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>22
gaaggtgaccaagttcatgcttcccccgtaagacaagagct41
<210>23
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>23
gaaggtcggagtcaacggatttcccccgtaagacaagagca41
<210>24
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>24
acgcttagccagttggaagg20
<210>25
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>25
gaaggtgaccaagttcatgctaggactggcgttttaaccct41
<210>26
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>26
gaaggtcggagtcaacggattaggactggcgttttaacccg41
<210>27
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>27
caccagattccaaagtaac19
<210>28
<211>42
<212>dna
<213>人工序列(artificialsequence)
<400>28
gaaggtgaccaagttcatgctcatttctgtgcctacacgtac42
<210>29
<211>42
<212>dna
<213>人工序列(artificialsequence)
<400>29
gaaggtcggagtcaacggattcatttctgtgcctacacgtaa42
<210>30
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>30
cgagtgacacgtgaggcacgtg22
<210>31
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>31
gaaggtgaccaagttcatgctttcgccaaccacactcaaat41
<210>32
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>32
gaaggtcggagtcaacggattttcgccaaccacactcaaag41
<210>33
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>33
cagagtgtggctgtttgtatg21
<210>34
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>34
gaaggtgaccaagttcatgctgagagaaacgaggctatgct41
<210>35
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>35
gaaggtcggagtcaacggattgagagaaacgaggctatgcc41
<210>36
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>36
tggtccggcttcctgcttg19
<210>37
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>37
gaaggtgaccaagttcatgctgccagagcttaccggttgta41
<210>38
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>38
gaaggtcggagtcaacggattgccagagcttaccggttgtt41
<210>39
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>39
aagccggcagcattagagtc20
<210>40
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>40
gaaggtgaccaagttcatgctgctaattcgaattcgtctgt41
<210>41
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>41
gaaggtcggagtcaacggattgctaattcgaattcgtctgc41
<210>42
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>42
aataagaaagaattttacg19
<210>43
<211>42
<212>dna
<213>人工序列(artificialsequence)
<400>43
gaaggtgaccaagttcatgctccaatattattgtgggcaaac42
<210>44
<211>42
<212>dna
<213>人工序列(artificialsequence)
<400>44
gaaggtcggagtcaacggattccaatattattgtgggcaaag42
<210>45
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>45
gcccagctcgggaacgtaaac21
<210>46
<211>201
<212>dna
<213>人工序列(artificialsequence)
<400>46
gtatttttccttctcccgttgtaacgcacgggcatgtttgctagttatgtcataaaaagg60
agaaatagtaatttataaaccggtcgaactcgcgcagacgctctggtgcttctccttggc120
gagccaggcgtcttccatggcgtagtagttattcatcaattcggacagtccggccatggt180
tctcggccggtgatggatgag201
- 还没有人留言评论。精彩留言会获得点赞!