一种用于辅助诊断肝癌的试剂及其在制备试剂盒中的用途的制作方法
本发明属于分子生物学检测领域,具体地,属于肝癌检测领域,更具体地,涉及肝癌基因标志物的甲基化位点的检测。
背景技术:
肝癌是世界上最常见的和最致命的疾病之一,也是我国高发的、危害极大的恶性肿瘤。根据globocan数据统计,2012年全球新增癌症患者人数约为1400万人,其中肝癌患者排在常见癌种的第六位,全球约有78.2万人罹患肝癌,占所有癌症新增病人的5.6%。肝癌起病隐匿,早期无特异性症状,大多数肝癌患者在就诊时已是中晚期,手术治愈率低,生存期短。血清甲胎蛋白(alphafetalprotein,afp)、以及组织活检和超声检查是当前临床上诊断肝癌常用而又重要的方法,然而其灵敏度和特异度并不理想,并且活检还具有创伤性。
肿瘤标志物检测是近年发展起来的用于检测疾病的方法,而寻找准确、稳定、有效的肝癌分子标志物对肝癌进行早期诊断、早期治疗具有重要意义。而随着科学界对肿瘤的认识逐渐深入,越来越多的研究证实细胞表观遗传水平的改变是肿瘤发生与发展的关键事件。dna甲基化、组蛋白修饰及mirna表达异常均属于表观遗传学改变,而肿瘤发生的核心环节主要与dna异常甲基化相关。dna甲基化检测稳定性好,有组织特异性,易于检测且其异常程度常与癌症的进展相关。可以作为疾病的诊断、预后提供了新的潜在标记物。
专利cn106929567a公开了通过apc基因、cox2基因、rassf1a基因以及微小rna-203基因的甲基化位点的甲基化水平而预测肝癌,但其组合能获得的最高灵敏度和特异性为84.2%和83.0%。
因此,存在对特异性好、灵敏度高的涉及dna甲基化位点的新的肿瘤基因标志物,特别是肝癌基因标志物的需求。
技术实现要素:
本发明通过在tcga和geo数据库搜集和构建肝癌相关的甲基化大数据集,采用生物信息学方法筛选出有应用开发潜力的肝癌dna甲基化标记物,通过大量研究,申请人发现loc375196基因的甲基化位点的甲基化水平与肝癌有着密切的联系,而在此之前,本领域通常仅认为loc375196是一种具有氧化还原酶活性和锌离子结合功能的基因,尚无文献报导其与肝癌存在关联。有鉴于此,
第一方面,本发明提供了一种检测loc375196中甲基化位点的甲基化水平的试剂在制备辅助诊断肝癌的试剂盒中的用途,其中,所述甲基化位点为cg12206199。
第二方面,本发明提供了一种检测loc375196中甲基化位点的甲基化水平的试剂在制备辅助诊断肝癌的试剂盒中的用途,其中,所述甲基化位点为cg22158769。
进一步地,本发明提供了检测loc375196中甲基化位点的甲基化水平的试剂在制备辅助诊断肝癌的试剂盒中的用途,其中,所述甲基化位点为cg12206199和cg22158769。
在一些具体的实施方案中,所述试剂进一步包括用于检测选自cdkl2基因、efnb2基因以及sept9基因的一个或更多个甲基化位点的甲基化水平的试剂。
在一些具体的实施方案中,所述试剂进一步包括用于检测cdkl2基因甲基化位点cg02466113、cg03757145,efnb2基因甲基化位点cg1877135以及sept9基因甲基化位点cg17300544中的一个或多个的甲基化水平的试剂。
进一步地,所述甲基化水平是通过扩增-测序、芯片、pcr的方式测量。
所述扩增-测序测量甲基化,需要包括但不限于核酸引物、测序tag序列、亚硫酸氢盐等试剂。
所述芯片测量甲基化,需要包括但不限于例如安捷伦的humancpgislandmicroarrays和humandnamethylationmicroarrays、illumina的infiniumhumanmethylation27beadchip、infiniumhumanmethylation450beadchip和goldengatemethylationassay以及rochenimblegen的humandnamethylation2.1mdeluxepromoterarray、humandnamethylationarray等能够直接用于检测甲基化位点的甲基化水平的甲基化芯片。
所述pcr包括但不限于甲基化特异性pcr、实时甲基化特异性pcr、荧光定量pcr。
所述pcr测量甲基化,需要包括但不限于核酸引物以及核酸探针等试剂。
第三方面,本发明提供了一种用于辅助诊断肝癌的试剂,所述试剂包括用于检测loc375196的甲基化位点cg12206199和/或cg22158769的甲基化水平的试剂。
使用本发明的检测loc375196的甲基化位点的试剂,cg12206199能够以82.35%的灵敏度以及100%的特异性诊断肝癌,其曲线下面积(auc)为0.8720,cg22158769能够以85.29%的灵敏度以及100%的特异性诊断肝癌,其曲线下面积(auc)为0.9018,表明其具有良好的分类性能,能够诊断肝癌。
yezhou等(“clinicalsignificanceofaberrantcyclin-dependentkinase-like2methylationinhepatocellularcarcinoma”.gene,2019)已经报道,cdkl2甲基化是肝细胞癌的一个有价值的生物标志物;yutingkuang等(“guadecitabine(sgi-110)primingsensitizeshepatocellularcarcinomacellstooxaliplatin”.molecularoncology,2015)已经报道,efnb2在肝细胞癌的病人当中也发现其启动子区域的高甲基化以及中国专利cn201610621113公开了一种用于诊断肝癌的组合物,所述组合物包括用于检测sept9基因及其片段的至少一个区域内甲基化状态的核酸。因此,在一些具体的实施方案中,所述试剂进一步包括用于检测选自cdkl2基因、efnb2基因以及sept9基因的一个或更多个甲基化位点的甲基化水平的试剂。
在一些具体的实施方案中,所述试剂进一步包括用于检测cdkl2基因甲基化位点cg02466113、cg03757145,efnb2基因甲基化位点cg1877135以及sept9基因甲基化位点cg17300544中的一个或多个的甲基化水平的试剂。
使用上述的试剂,进一步提高了诊断肝癌的灵敏度为85.29%,特异性为100%,曲线下面积为0.9265,使得诊断肝癌的效果更佳。
在一些具体的实施方案中,所述核酸引物包括(1)-(4)中的一对或多对:
(1)cdkl2-f:5’-atgattggtttagggttaattat-3’;
cdkl2-r:5’-rtaaacatcacaatcrcctc-3’;
(2)efnb2-f:5’-gtgtyggttagtttagattgtgg-3’;
efnb2-r:5’-cccaaacrctaaaaactactatc-3’;
(3)loc375196-f:5’-tgtagtygttgtttaagagygt-3’;
loc375196-r:5’-aaacractcaacracttaacct-3’;
(4)sept9-f:5’-taatttttgtaggygtagag-3’;
sept9-r:5’-aatataaaaaccraaccataac-3’;
使用上述的引物,能够获得最佳的扩增效率,使得后续的检测过程更佳准确,更能够保证灵敏度和特异性。
第四方面,本发明提供了一种用于辅助诊断肝癌的试剂盒,所述试剂盒包括上述的试剂。
进一步地,所述试剂盒还包括提取核酸的试剂、dntp、mg2+、dna聚合酶、pcr缓冲液中的至少一种。
进一步地,各组分的终浓度的范围如下所示:mg2+2~6mm、dntp100~400mm、dna聚合酶0.01~30u、引物10~400nm。
附图说明
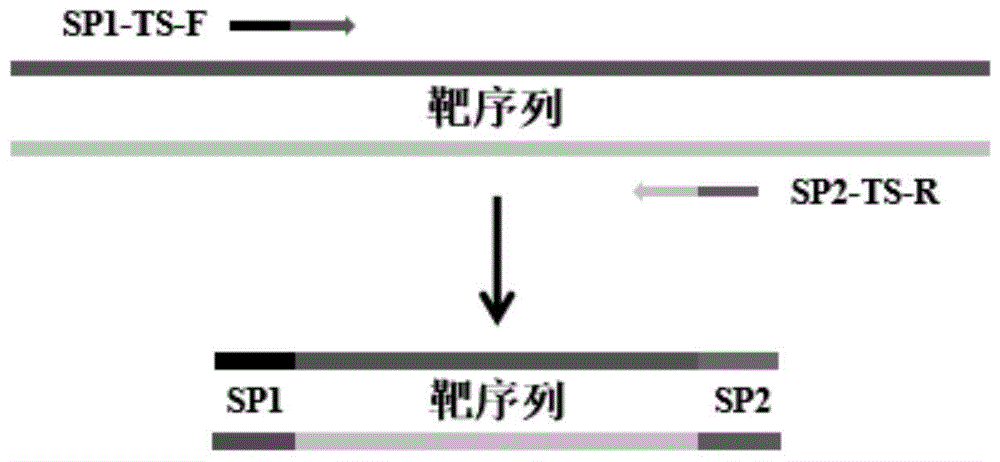
图1为靶向pcr扩增示意图;
图2为引物扩增凝胶电泳结果图;
图3为添加测序tag序列和带样本条形码的测序tag序列示意图;
图4为6个cpg位点甲基化水平鉴别癌组织和非癌组织的roc图。
具体实施方式
下文将结合具体实施方式和实施例,具体阐述本发明,本发明的优点和各种效果将由此更加清楚地呈现。本领域技术人员应理解,这些具体实施方式和实施例是用于说明本发明,而非限制本发明。
实施例1、基因甲基化位点的筛选
从ucscxena网站的tcga数据集(https://tcga.xenahubs.net)下载到379例癌组织和50例癌旁组织甲基化数据和临床信息。从从美国国立生物技术信息中心(ncbi)的geo数据库中下载到355例癌组织、66例癌旁组织、10例正常样本和9例肝硬化组织甲基化数据和临床信息。另外收集了原发性肝癌患者的34对肝癌组织及其癌旁正常组织的样本,用于检测分析dna甲基化标记物在样本中的甲基化水平。我们将该数据集标记为liverngs,见表1。
表1
对两个数据集gse以及lihc分别进行差异分析,挑选共有的差异甲基化位点,构建区分肝癌样本与非肝癌样本的分类器。以gse数据集作为训练集,采用logistics回归模型对训练集数据进行了分析,得到一个基于6个cpg位点的分类模型,分别为cdkl2(cg02466113、cg03757145)、efnb2(cg1877135)、loc375196(cg12206199、cg22158769)和sept9(cg17300544)。lihc数据为测试集,对模型进行性能测试。该6个cpg位点将它们筛选出来作为肝癌特异的dna甲基化标记物,其对应的基因是cdkl2、efnb2、loc375196和sept9。
实施例2、临床样本的分析方法
收集的原发性肝癌患者的34对肝癌组织及其癌旁正常组织的样本,用于检测分析dna甲基化标记物在样本中的甲基化水平。本发明中相关肝癌及癌旁正常组织均取自医院,通过了医学研究伦理委员会认可,所有受试者均签署了知情同意书。肝癌组织及癌旁正常组织的诊断均以最终的医院病理诊断为依据。样本存放于-80℃冰箱保存待用。实验过程如下所述:
1、样本制备
本发明的样本制备是通过tianampgenomicdnakit提取50mg的新鲜肝癌组织和新鲜的癌旁组织,100μl洗脱液洗脱。提取的基因组dna需满足以下三个质控条件:提取的核酸总量大于1μg;核酸的od260/280在1.8-2.0之间;核酸的od260/230大于2.0。
2、文库制备
本发明将200ng质控合格的基因组dna按照厂家操作规程采用ezdnamethylation-lightningtmkit(zymoresearch,irvine,ca,usa)进行重亚硫酸盐处理。随后,重亚硫酸盐处理后的样本dna采用双引物靶向pcr扩增,具体参见图1。
扩增程序为:95℃预变性10分钟;95℃变性15秒,58℃退火30秒,72℃延伸1分钟,扩增15个循环;最后72℃延伸4分钟。
其中,样本预混液包括1倍不含mgcl2的faststart高保真反应缓冲液、4.5mmmgcl2、200mmdntp、0.05u/μlfaststart高保真酶混合液(roche,indianapolis,indiana,usa);亚硫酸盐测序甲基化引物混合液包括0.01-0.4mm的前向靶向扩增引物和反向靶向扩增引物序列,其中,针对4个基因设计了多对引物(具体序列未示出),最终挑选如表2所示的引物,因为其扩增效率最佳,引物扩增胶图如图2所示,图2中泳道引物标记具体代表如下,c2:cdkl2,e2:efnb2,l6:loc375196,s9:sept9,下划线后字母代表该基因设计的不同引物对,箭头表示最终使用的引物,即表2中所列引物。针对每一个反应,加入10ng-50ng重亚硫酸盐处理后的样本dna。pcr反应完成后,采用agencourtampurexpsystem(beckmancoμlter,ca,usa)进行纯化处理以去除多余的引物和非特异性扩增产物。
表2
添加测序tag序列和带样本条形码的测序tag序列:pcr扩增完之后,得到每个样本的扩增产物(参见图3)。接下来通过普通的pcr扩增给每个样本的扩增产物添加上测序tag序列和带样本条形码的测序tag序列。添加样本条形码的pcr在25μl的反应体系内进行。25μl反应体系的组成包括:1倍pfx缓冲液、4mmmgcl2、200mmdntp、0.05u/μlplatinumpfxdna聚合酶(thermoscientific,de,usa)。扩增体系中包含0.4mm测序tag序列和0.4mm带样本条形码的测序tag序列,其中测序tag引物序列为:5'-aatgatacggcgaccaccgagatctacactgacgacatggttctaca-3,带样本条形码的测序tag引物序列为:5'-caagcagaagacggcatacgagat-[bc]-tacggtagcagagacttggtct-3',这里bc表示barcode,即样本条形码序列。
针对每一个反应,加入靶向扩增纯化后的全部样本。采用普通的pcr反应进行扩增,其热循环条件为:94℃预变性2分钟;94℃变性15秒、58℃退火30秒、72℃延伸60秒共进行15个循环的pcr扩增,最后在72℃下进行4分钟的延伸反应。pcr反应完成后,采用agencourtampurexpsystem(beckmancoμlter,ca,usa)进行纯化处理以去除多余的引物和非特异性扩增产物。同时,扩增产物的片段大小分布和引物二聚体比例采用labchipgx(perkinelmer,ca,usa)进行检测。纯化处理后的文库采用
3、测序
将文库总量、扩增产物的片段大小分布和引物二聚体比例质检均合格的待测文库,按照1:1的物质的量进行混合,使用
4、肝癌分类模型的建立和评价
对于测序得到的原始fastq数据,使用biqanalyzer3.0软件的默认分析参数,将测序reads比对到参考序列,并分析提取出6个cpg位点在各个样本中的甲基化水平,结果如表3所示。
表3
根据所选择用于检测的甲基化位点的甲基化程度计算出预测分数score,以及根据预测分数score评估检测样本患肝癌风险程度,
预测分数score计算公式,对于n个cpg位点构成的模型,其score计算公式为:常规的通用公式为:score=e(a+x1*cpg1+...+xi*cpgi+...+xn*cpgn),公式中各cpg位点,代表其相应的甲基化水平值(甲基化水平值介于0到1)。
肝癌风险判断阈值:若预测分数score>=0.5判断为肝癌阳性样本,预测分数score<0.5,判断为肝癌阴性样本
可以根据已收集的数据库来源和已检测的肝癌样本与对照样本的甲基化谱图来确定公式中每个cpg位点的系数,并可随着所检测的肝癌样本与对照样本的甲基化谱图的不断增加而更新模型中的系数。
例如,可使用下述公式来计算实施例3和4中的所使用到的cpg组合的预测分数:
cg22158769:score=e(20.99*cg22158769-2.91)
cg12206199:score=e(22.67*cg12206199-2.95)
cg17300544+cg12206199:score=e(10.73*cg17300544+20.05*cg12206199-3.63)
cg18771357+cg22158769:score=e(21.26*cg18771357+15.19*cg22158769-4.92)
cg03757145+cg22158769:score=e(28.97*cg03757145+12.57*cg22158769-5.21)
cg17300544+cg18771357+cg22158769:
score=e(7.88*cg17300544+19.70*cg18771357+13.14*cg22158769-5.25)
cg17300544+cg03757145+cg22158769:
score=e(9.65*cg17300544+24.14*cg03757145+13.14*cg22158769-5.51)
cg17300544+cg22158769+cg02466113:
score=e(10.71*cg17300544+15.54*cg22158769+7.99*cg02466113-4.57)
cg18771357+cg03757145+cg22158769:
score=e(17.47*cg18771357+15.99*cg03757145+13.14*cg22158769-5.71)
cg03757145+cg22158769+cg02466113:
score=e(52.62*cg03757145+17.51*cg22158769-15.68*cg02466113-5.70)
全部6个点的组合:
score=e(a+x1*cg02466113+x2*cg03757145+x3*cg1877135+x4*cg12206199+x5*cg22158769+x6*cg17300544),
上述公式中系数:a为-6.197599,x1为-49.597064,x2为69.590585,x3为29.773446,x4为40.284933,x5为-24.235368,x6为7.279583。
实施例3、单个甲基化位点用于预测肝癌
分别使用loc375196的甲基化位点cg22158769以及cg12206199位点而预测肝癌,基于这两个cpg位点logistics回归分析的分类模型,在临床肝癌组织及其癌旁正常组织样本liverngs测序数据为验证集中,显示该分类模型有较好的分类判别能力,其结果如表4所示,从表中可以看出,单个loc375196的甲基化位点也具有较好的分类性能。
表4
实施例4、甲基化位点的组合用于预测肝癌
使用loc375196基因的甲基化位点的组合,以及其与其余三个基因中的任意一个或两个的甲基化位点组合而预测肝癌,其结果如表5所示,loc375196与其余基因进行组合具有更好的分类性能。
表5
最终,使用所有6个cpg位点而预测肝癌,其构建的分类模型敏感性为88.24%,特异性为100%,曲线下面积为0.927,参见表6以及图4。上述结果进一步验证了所筛选出的6个高甲基化cpg位点构建的模型是很好的肝癌甲基化标记物,具备肝癌辅助诊断、甚至早期筛查的潜力。
表6
- 还没有人留言评论。精彩留言会获得点赞!