羰基还原酶突变体及在制备手性β’-羟基-β-氨基酸酯中的应用
本发明涉及酶工程和生物技术领域,具体涉及羰基还原酶突变体及其在制备手性β’-羟基-β-氨基酸酯及结构类似物中的应用。
背景技术:
碳青霉烯类抗生素是当前临床应用的抗生素中具有最广泛的抗菌谱和最高的抗菌活性的一类β-内酰胺类抗生素,且不存在与其他β-内酰胺类抗生素之间的交互耐药性,可以用来治疗对其他抗生素有耐药性的菌种。
碳青霉烯类抗生素结构通式
手性β’-羟基-β-氨基酸酯是合成碳青霉烯类抗生素的关键中间体。目前文献中报道较多的是利用金属配合物(dynamickineticresolutionofβ-keto-β-aminoestersusingru-dtbm-sunphoscatalyzedasymmetrichydrogenation,tetrahedron,2013,69,7152-7156;newchiraldiphosphineligandsdesignedtohaveanarrowdihedralangleinthebiarylbackbone,adv.synth.catal.2001,343,264-267)实现动态动力学拆分获得(2s,3r)-3-羟基-2-(保护的胺)甲基丁酸酯,但是金属配合物的催化往往需要比较苛刻的反应条件,金属的配体需要多步的合成步骤。
羰基还原酶可以实现羰基的立体选择性还原,在制备手性醇方面应用广泛,通过对羰基还原酶的改造可以实现提高酶活力和立体选择性,底物的氨基被苯甲酰基保护,野生型或改造后的羰基还原酶实现其还原(醛酮还原酶及其在合成(2s,3r)-2-苯甲酰氨甲基-3-羟基丁酸酯中的应用,cn201410706195.1;ketoreductasepolypeptidesfortheproductionofazetidinone,us20170002333),但是由于苯甲酰基脱保护时在水溶液中可能会被部分水解。使用邻苯二甲酰基保护氨基时可以使氨基在特定条件下水解脱除保护,但是使用生物法在转化β’-羰基-β-氨基酸酯时的效果较差,不仅底物浓度低,而且获得的还原产物是混合物(stereoselectiveprostereogenic3-oxoesterreductionmediatedbyanovelyeastalcoholdehydrogenasederivedfromkluyveromycesmarxianuscbs6556,adv.synth.catal.2007,349,1111–1118)。为了实现β’-羰基-β-(邻苯二甲酰亚胺)甲基酸酯的动态动力学的还原对碳青霉烯类抗生素的合成研究和工业应用具有重要意义。
技术实现要素:
为了解决上述问题,本发明提供了一种通过基因工程手段改造后的羰基还原酶突变体,具体地,改造后的羰基还原酶突变体合成手性(2s,3r)-3-羟基-2-(邻苯二甲酰亚胺)甲基丁酸酯的活力和立体选择性显著提高。
首先,本发明提供一种羰基还原酶突变体,所述的羰基还原酶突变蛋白与seqidno.:1所示的氨基酸序列具有至少90%同一性,且所述突变蛋白具有合成手性(2s,3r)-3-羟基-2-(邻苯二甲酰亚胺)甲基-丁酸甲酯的能力,并且催化活性和立体选择性显著提高。
优选地,本发明提供的羰基还原酶突变体是:在对应于seqidno.1的氨基酸序列的第1至342位中仅存在第207,224,226,242,245在内的一个或多个位点突变。
更具体地,本发明提供的羰基还原酶突变体是:在对应于seqidno.1的氨基酸序列的第1至342位中仅存在下述突变:s134i且t135i;t135r且s224a;y208i且s224a;m242a且q245s;m242v且q245l;m242f且q245t;m242v且q245s;p243f且q245g;s224a、m242v且q245s;m242v、q245s且w226i。更优选地,m242f且q245t;m242v且q245s;p243f且q245g;s224a、m242v且q245s;m242v、q245s且w226i,其氨基酸序列为seqidno.3-7之一所示。
本发明还提供上述突变体的编码基因。进一步提供含所述基因的表达载体、重组细胞。
本发明还提供本发明的羰基还原酶突变体或其编码基因在制备手性(2s,3r)-3-羟基-2-(邻苯二甲酰亚胺)甲基丁酸甲酯化合物中的应用。
在具体实施方式中,所述应用是以下式的β’-羰基-β-(保护)氨基酸酯化合物作为底物:
具体地,将本发明所述的羰基还原酶突变体与反应底物接触,进行催化反应,从而获得所述(2s,3r)-3-羟基-2-(邻苯二甲酰亚胺)甲基丁酸甲酯化合物;任选地,还包括分离并纯化所述(2s,3r)-3-羟基-2-(邻苯二甲酰亚胺)甲基丁酸甲酯化合物的步骤。
在一个具体实施方式中,所述催化反应以表达所述羰基还原酶突变体的编码基因的工程菌经发酵培养获得的湿菌体为催化剂,以β’-羰基-β-(邻苯二甲酰亚胺)甲基丁酸甲酯化合物为底物,以ph为6.0-11.0的缓冲液作为反应介质,在25℃-50℃条件下进行反应。
在更优选的实施方式中,所述反应中,反应体系中的催化底物浓度为50-250g/l,更佳地,底物浓度为100-150g/l;反应体系含有菌体量为10-150g/l,更佳地,30-100g/l;反应体系的ph为6.0-9.0,最佳为7.0;反应温度为25℃-35℃,最佳为30℃。
具体的反应体中加入nadp+,葡萄糖,葡萄糖脱氢酶,且在150rpm-250rpm的条件下反应,反应时间为5-25小时。更优选地,加入0.2g/lnadp+,2.5倍当量葡萄糖,2g/l葡萄糖脱氢酶冻干酶粉,于30℃,200rpm的摇床上反应,反应10h。
在另外的优选方式中,反应体系还添加有助溶剂,更具体地助溶剂为乙腈、丙酮、甲醇、乙醇、二甲基亚砜、n,n-二甲基甲酰胺、二氯甲烷、1,4-二氧六环,较佳地,无助溶剂、甲醇、乙醇、二甲基亚砜。
本发明的羰基还原酶突变体,催化获得的(2s,3r)-3-羟基-2-(邻苯二甲酰亚胺)甲基-丁酸甲酯的转化率≥70%,较佳地,≥90%,更佳地,≥97%;远高于野生型的羰基还原酶的转化率;催化获得的(2s,3r)-3-羟基-2-(邻苯二甲酰亚胺)甲基-丁酸甲酯ee值≥80%,较佳地,≥90%,更佳地,≥99%,de值≥80%,较佳地,≥90%,更佳地,≥98%;也远远优于野生型的羰基还原酶的转化率。
附图说明
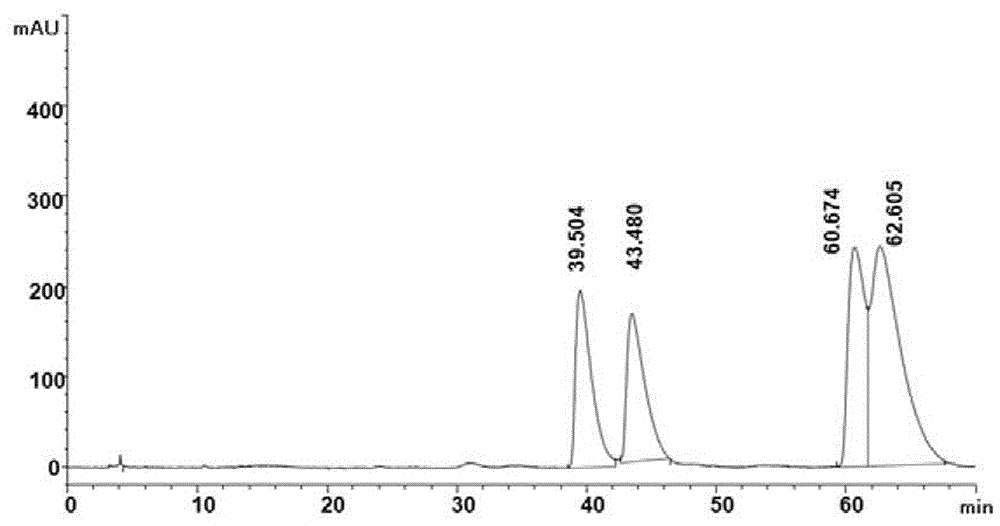
图1β’-羰基-β-(邻苯二甲酰亚胺)甲基丁酸甲酯的还原后消旋体混合物的液相谱图。其中,保留时间分别为39.5min,43.4min,60.6min,62.6min,对应的结构式如下:
图2突变体12转化粗品的液相谱图。
具体实施方式
下面通过具体实施方式对本发明作进一步的阐述,以期更好的理解本发明,但并不构成对本发明的限制。其中,如本文所用,术语“axxb”表示第xx位的氨基酸a变为氨基酸b,例如“q245h”表示第245位的缬氨酸q突变为丙氨酸h,以此类推。
在本发明的优选实施方式中,本发明的羰基还原酶突变体的制备方法如下:大肠杆菌为表达宿主。
具体地,该制备方法包括以下步骤:(1)羰基还原酶相应突变位点的基因构建到pet-21a表达载体上,获得带有目的酶基因的重组质粒。(2)将重组质粒转入宿主菌细胞(优选大肠杆菌bl21(de3)),获得相应的工程菌株。(3)将工程菌株接种至lb培养基中,37℃培养6小时,加入0.1mm的异丙基硫代半乳糖苷(iptg),25℃培养6-12小时。(4)离心收集菌体。
实施例1:羰基还原酶库的筛选
以β’-羰基-β-(邻苯二甲酰亚胺)甲基丁酸甲酯为筛选底物,对实验室现有156个的羰基还原酶进行筛选,筛选体系为1ml,羰基还原酶的浓度为2g/l,底物浓度为10g/l,葡萄糖20g/l,nad(p)+0.2mg/ml,葡萄糖脱氢酶(gdh)3u/l,反应缓冲液为100mmph7.5磷酸钾缓冲液,30℃反应15h后,乙酸乙酯萃取,萃取后风干,tlc初步检测产物生成情况,高效液相检测产物构型,检测方法:od-h正向柱,流动相95:5正己烷:异丙醇,流速1ml/min,检测波长230nm,经过液相检测,有产物生成的羰基还原酶有21个,其中活力最优的羰基还原酶是sscr,其核苷酸序列为seqidno.2,氨基酸序列为seqidno.1。
实施例2:羰基还原酶sscr突变位点的选择及突变体的构建
利用羰基还原酶sscr的晶体结构(pdbid:1y1p)与底物进行docking,使用的软件为discoverstudio4.1(accelrys,usa),经过分析,推测出与底物有相互作用的氨基酸为:134,135,207,208,224,226,242,243,245,278,280,将这些位点作为候选突变位点。
将seqidno.1的氨基酸序列对应的核苷酸序列seqidno.2全合成并克隆到pet-21a载体的限制性内切酶位点ndei和xhoi间,得到重组质粒pet-21a-sscr,进一步转化至表达宿主e.colibl21(de3),挑取阳性克隆,即获得重组表达转化体e.colibl21(de3)/pet-21a-sscr。
构建羰基还原酶单点突变体:以pet-21a-sscr为模板,选择通过软件推测与底物有相互作用的氨基酸134,135,207,208,224,226,242,243,245,278,280进行定点突变。设计引物(引物序列见表1),采用质粒滚换扩增的方法构建突变体,高保真聚合酶kod-plus进行pcr。pcr反应条件如下:总体积为50μl的pcr反应体系中,加入5μl10×kodbuffer,5μldntp(2mm),2μlmgso4(25mm),模板20~100ng,一对突变引物各1μl(10μm),1μlkod聚合酶,加灭菌蒸馏水至50μl。pcr反应程序:(1)94℃变性3min,(2)94℃变性30sec,(3)55℃退火30sec,(4)68℃延伸7min,步骤(2)~(4)共进行20~30个循环。4℃保存pcr产物。pcr产物经琼脂糖凝胶电泳分析验证后,加入限制性内切酶dpni在37℃消化2h。将消化产物转入e.colibl21(de3)感受态细胞并涂布于含有氨苄抗生素的平板中,置于37℃培养箱中静置培养约12h。挑取单克隆进行测序,测序正确即获得相应突变体。
组合突变体的构建方法同单点突变体。突变位点及突变后的氨基酸见表2。
表1突变位点的引物序列
实施例3:羰基还原酶突变体的诱导表达
将上述突变体的基因工程菌的单菌落分别接入4ml含有氨苄抗生素的lb液体培养基(蛋白胨10g/l,酵母粉5g/l,nacl10g/l)中,于37℃,200rpm摇床中培养过夜,即为种子液。将过夜培养的种子液以1%的接种量转接到50ml含有氨苄抗生素的lb培养基,37℃,200rpm培养至od600为0.6-1.0左右,加入0.5mm的iptg,并置于25℃,200rpm诱导8~12h。4℃,6000rpm条件下离心收集菌体。用磷酸钾缓冲液(100mm,ph7.5)重悬菌体,并用c超声或者高压匀浆机破碎,4℃,12000rpm条件下离心留取上清液,进行后续的sds-page及酶活检测。
实施例4:羰基还原酶酶活检测
通过检测340nm下nadph吸光值的变化来计算sscr突变体的催化活性,酶活检测底物为β’-羰基-β-(邻苯二甲酰亚胺)甲基丁酸甲酯,体系为底物0.875g/l,30%dmso助溶,0.5g/lnadph,缓冲液为100mmph7.5磷酸钾缓冲液。检测的比活力和催化得到的产物结果见表2。由表2中可知,单个位点的突变,比活力提高不明显,甚至与野生型的持平。但双位点或三位点的突变,比活力有明显的增加。但突变体12,13,14,15,16的比活力增加更为明显一些,其中,活力最高突变体是突变体16,粗酶液比活为54.02u/mg,其次是m242v/q245s的为粗酶液比活为42.89u/mg。1u表示每分钟内催化1微摩尔(μmol)底物转化为产物所需的酶量。
表2突变体活力及其催化结果
实施例5:羰基还原酶突变体催化合成(2s,3r)-3-羟基-2-(邻苯二甲酰亚胺)甲基丁酸甲酯
将野生型及比活力最高的突变体12,13,14,15,16,氨基酸序列为seqidno.3-7,分别按照实施例3培养诱导表达方法进行蛋白诱导表达后收集菌体,以菌体作为生物催化剂。
取20g/l菌体(野生型)重悬于磷酸钾缓冲液(ph7.5,100mm,100ml),称取底物20g/l,加入0.2g/lnadp+,2.5倍当量葡萄糖,2g/l葡萄糖脱氢酶冻干酶粉,于30℃,200rpm的摇床上反应,20h停止反应,反应结束后,tlc及hplc检测产物生成。底物转化率为12.3%,未进行后处理。
取20g/l菌体(突变体12)重悬于磷酸钾缓冲液(ph7.5,100mm,100ml),称取底物20g/l,加入0.2g/lnadp+,2.5倍当量葡萄糖,2g/l葡萄糖脱氢酶冻干酶粉,于30℃,200rpm的摇床上反应,4h停止反应,反应结束后,tlc及hplc检测产物生成。等体积乙酸乙酯萃取三次,无水硫酸钠干燥后,减压除去溶剂,获得产物粗品1.96g,收率为97.5%。粗品液相见附图2。
取20g/l菌体(突变体13)重悬于磷酸钾缓冲液(ph7.5,100mm,100ml),称取底物20g/l,加入0.2g/lnadp+,2.5倍当量葡萄糖,2g/l葡萄糖脱氢酶冻干酶粉,于30℃,200rpm的摇床上反应,4h停止反应,反应结束后,tlc及hplc检测产物生成。等体积乙酸乙酯萃取三次,无水硫酸钠干燥后,减压除去溶剂,获得产物粗品1.98g,收率为98.2%。
取20g/l菌体(突变体13)重悬于磷酸钾缓冲液(ph7.5,100mm,100ml),称取底物40g/l,加入0.2g/lnadp+,2.5倍当量葡萄糖,2g/l葡萄糖脱氢酶冻干酶粉,于30℃,200rpm的摇床上反应,8h停止反应,反应结束后,tlc及hplc检测产物生成。等体积乙酸乙酯萃取三次,无水硫酸钠干燥后,减压除去溶剂,获得产物粗品3.83g,收率为95.2%。
取20g/l菌体(突变体14)重悬于磷酸钾缓冲液(ph7.5,100mm,100ml),称取底物40g/l,加入0.2g/lnadp+,2.5倍当量葡萄糖,2g/l葡萄糖脱氢酶冻干酶粉,于30℃,200rpm的摇床上反应,12h停止反应,反应结束后,tlc及hplc检测产物生成。等体积乙酸乙酯萃取三次,无水硫酸钠干燥后,减压除去溶剂,获得产物粗品3.77g,收率为93.5%。
取20g/l菌体(突变体15)重悬于磷酸钾缓冲液(ph7.5,100mm,100ml),称取底物40g/l,加入0.3g/lnadp+,2.5倍当量葡萄糖,2g/l葡萄糖脱氢酶冻干酶粉,于30℃,200rpm的摇床上反应,8h停止反应,反应结束后,tlc及hplc检测产物生成。等体积乙酸乙酯萃取三次,无水硫酸钠干燥后,减压除去溶剂,获得产物粗品3.90g,收率为96.8%。
取40g/l菌体(突变体15)重悬于磷酸钾缓冲液(ph7.5,100mm,100ml),称取底物80g/l,加入0.3g/lnadp+,2.5倍当量葡萄糖,2g/l葡萄糖脱氢酶冻干酶粉,于30℃,200rpm的摇床上反应,12h停止反应,反应结束后,tlc及hplc检测产物生成。等体积乙酸乙酯萃取三次,无水硫酸钠干燥后,减压除去溶剂,获得产物粗品7.65g,收率为94.9%。
取40g/l菌体(突变体16)重悬于磷酸钾缓冲液(ph7.5,100mm,100ml),称取底物100g/l,加入0.2g/lnadp+,2.5倍当量葡萄糖,2g/l葡萄糖脱氢酶冻干酶粉,于30℃,200rpm的摇床上反应,12h停止反应,反应结束后,tlc及hplc检测产物生成。等体积乙酸乙酯萃取三次,无水硫酸钠干燥后,减压除去溶剂,获得产物粗品9.65g,收率为95.8%。
取80g/l菌体(突变体16)重悬于磷酸钾缓冲液(ph7.5,100mm,100ml),称取底物200g/l,加入0.2g/lnadp+,2.5倍当量葡萄糖,2g/l葡萄糖脱氢酶冻干酶粉,于30℃,200rpm的摇床上反应,20h停止反应,反应结束后,tlc及hplc检测产物生成。等体积乙酸乙酯萃取三次,无水硫酸钠干燥后,减压除去溶剂,获得产物粗品19.43g,收率为96.2%。
取100g/l菌体(突变体16)重悬于磷酸钾缓冲液(ph7.5,100mm,100ml),称取底物250g/l,加入0.2g/lnadp+,2.5倍当量葡萄糖,2g/l葡萄糖脱氢酶冻干酶粉,于30℃,200rpm的摇床上反应,28h停止反应,反应结束后,tlc及hplc检测产物生成。等体积乙酸乙酯萃取三次,无水硫酸钠干燥后,减压除去溶剂,获得产物粗品获得产物粗品24.27g,收率为96.7%。
取100g/l菌体(突变体12)重悬于磷酸钾缓冲液(ph7.5,100mm,100ml),称取底物250g/l,加入0.2g/lnadp+,2.5倍当量葡萄糖,4g/l葡萄糖脱氢酶冻干酶粉,于30℃,200rpm的摇床上反应,35h停止反应,反应结束后,tlc及hplc检测产物生成。等体积乙酸乙酯萃取三次,无水硫酸钠干燥后,减压除去溶剂,获得产物粗品23.92g,收率为95.3%。
取100g/l菌体(突变体13)重悬于磷酸钾缓冲液(ph7.5,100mm,100ml),称取底物250g/l,加入0.2g/lnadp+,2.5倍当量葡萄糖,4g/l葡萄糖脱氢酶冻干酶粉,于30℃,200rpm的摇床上反应,30h停止反应,反应结束后,tlc及hplc检测产物生成。等体积乙酸乙酯萃取三次,无水硫酸钠干燥后,减压除去溶剂,获得产物粗品24.12g,收率为95.8%。
序列表
<110>中国科学院天津工业生物技术研究所
<120>羰基还原酶突变体及在制备手性β’-羟基-β-氨基酸酯中的应用
<160>29
<170>siposequencelisting1.0
<210>1
<211>343
<212>prt
<213>ralstoniasp.
<400>1
metalalysileaspasnalavalleuprogluglyserleuvalleu
151015
valthrglyalaasnglyphevalglyserhisvalvalgluglnleu
202530
leugluhisglytyrlysvalargglythralaargseralaserlys
354045
leualaasnleuglnlysargtrpaspalalystyrproglyargphe
505560
gluthralavalvalgluaspmetleulysglnglyalatyraspglu
65707580
valilelysglyalaalaglyvalalahisilealaservalvalser
859095
pheserasnlystyraspgluvalvalthrproalaileglyglythr
100105110
leuasnalaleuargalaalaalaalathrproservallysargphe
115120125
valleuthrserserthrvalseralaleuileprolysproasnval
130135140
gluglyiletyrleuaspglulyssertrpasnleugluserileasp
145150155160
lysalalysthrleuprogluseraspproglnlysserleutrpval
165170175
tyralaalaserlysthrglualagluleualaalatrplysphemet
180185190
aspgluasnlysprohisphethrleuasnalavalleuproasntyr
195200205
thrileglythrilepheaspprogluthrglnserglyserthrser
210215220
glytrpmetmetserleupheasnglygluvalserproalaleuala
225230235240
leumetproproglntyrtyrvalseralavalaspileglyleuleu
245250255
hisleuglycysleuvalleuproglnilegluargargargvaltyr
260265270
glythralaglythrpheasptrpasnthrvalleualathrphearg
275280285
lysleutyrproserlysthrpheproalaasppheproaspglngly
290295300
glnaspleuserlyspheaspthralaproserleugluileleulys
305310315320
serleuglyargproglytrpargserileglugluserilelysasp
325330335
leuvalglysergluthrala
340
<210>2
<211>1029
<212>dna
<213>ralstoniasp.
<400>2
atggctaaaatcgataacgcagttctgccggaaggttccctggttctggttaccggtgct60
aacggtttcgttggttcccacgttgttgaacagctgctggaacacggttacaaagttcgt120
ggtaccgctcgttccgcttccaaactggctaacctgcagaaacgttgggacgctaaatac180
ccgggtcgtttcgaaaccgctgttgttgaagacatgctgaaacagggtgcttacgacgaa240
gttatcaaaggtgctgctggtgttgctcacatcgcttccgttgtttccttctccaacaaa300
tacgacgaagttgttaccccggctatcggtggtaccttgaacgctctgcgtgctgctgct360
gctaccccgtccgttaaacgtttcgttctgacctcctccaccgtttccgctctgattccg420
aaaccgaacgttgaaggtatctacctggacgaaaaatcctggaacctggaatccatcgac480
aaagctaaaaccctgccggaatccgacccgcagaaatccctgtgggtatacgctgcatcc540
aagaccgaagctgaactggctgcatggaaatttatggatgagaacaagccacacttcact600
ctgaacgctgtactgccaaactacactattggcactattttcgatccggaaactcagtcc660
ggttccacctccggttggatgatgtccctgtttaacggcgaggtttccccggctctggct720
ctgatgccaccgcagtactacgtttccgctgttgatattggcctgctgcacctgggttgc780
ctggttctgccacaaatcgaacgtcgtcgtgtttacggtacggctggtactttcgattgg840
aacaccgttctggctaccttccgtaaactgtacccgtccaaaaccttcccggctgacttc900
ccagatcaaggtcaggacctgtctaaattcgacaccgctccgtccctggaaattctgaaa960
tctctgggtcgcccaggttggcgttccatcgaagaatccatcaaagacctggttggttcc1020
gaaaccgct1029
<210>3
<211>343
<212>prt
<213>ralstoniasp.
<400>3
metalalysileaspasnalavalleuprogluglyserleuvalleu
151015
valthrglyalaasnglyphevalglyserhisvalvalgluglnleu
202530
leugluhisglytyrlysvalargglythralaargseralaserlys
354045
leualaasnleuglnlysargtrpaspalalystyrproglyargphe
505560
gluthralavalvalgluaspmetleulysglnglyalatyraspglu
65707580
valilelysglyalaalaglyvalalahisilealaservalvalser
859095
pheserasnlystyraspgluvalvalthrproalaileglyglythr
100105110
leuasnalaleuargalaalaalaalathrproservallysargphe
115120125
valleuthrserserthrvalseralaleuileprolysproasnval
130135140
gluglyiletyrleuaspglulyssertrpasnleugluserileasp
145150155160
lysalalysthrleuprogluseraspproglnlysserleutrpval
165170175
tyralaalaserlysthrglualagluleualaalatrplysphemet
180185190
aspgluasnlysprohisphethrleuasnalavalleuproasntyr
195200205
thrileglythrilepheaspprogluthrglnserglyserthrser
210215220
glytrpmetmetserleupheasnglygluvalserproalaleuala
225230235240
leupheproprothrtyrtyrvalseralavalaspileglyleuleu
245250255
hisleuglycysleuvalleuproglnilegluargargargvaltyr
260265270
glythralaglythrpheasptrpasnthrvalleualathrphearg
275280285
lysleutyrproserlysthrpheproalaasppheproaspglngly
290295300
glnaspleuserlyspheaspthralaproserleugluileleulys
305310315320
serleuglyargproglytrpargserileglugluserilelysasp
325330335
leuvalglysergluthrala
340
<210>4
<211>343
<212>prt
<213>ralstoniasp.
<400>4
metalalysileaspasnalavalleuprogluglyserleuvalleu
151015
valthrglyalaasnglyphevalglyserhisvalvalgluglnleu
202530
leugluhisglytyrlysvalargglythralaargseralaserlys
354045
leualaasnleuglnlysargtrpaspalalystyrproglyargphe
505560
gluthralavalvalgluaspmetleulysglnglyalatyraspglu
65707580
valilelysglyalaalaglyvalalahisilealaservalvalser
859095
pheserasnlystyraspgluvalvalthrproalaileglyglythr
100105110
leuasnalaleuargalaalaalaalathrproservallysargphe
115120125
valleuthrserserthrvalseralaleuileprolysproasnval
130135140
gluglyiletyrleuaspglulyssertrpasnleugluserileasp
145150155160
lysalalysthrleuprogluseraspproglnlysserleutrpval
165170175
tyralaalaserlysthrglualagluleualaalatrplysphemet
180185190
aspgluasnlysprohisphethrleuasnalavalleuproasntyr
195200205
thrileglythrilepheaspprogluthrglnserglyserthrser
210215220
glytrpmetmetserleupheasnglygluvalserproalaleuala
225230235240
leuvalproprosertyrtyrvalseralavalaspileglyleuleu
245250255
hisleuglycysleuvalleuproglnilegluargargargvaltyr
260265270
glythralaglythrpheasptrpasnthrvalleualathrphearg
275280285
lysleutyrproserlysthrpheproalaasppheproaspglngly
290295300
glnaspleuserlyspheaspthralaproserleugluileleulys
305310315320
serleuglyargproglytrpargserileglugluserilelysasp
325330335
leuvalglysergluthrala
340
<210>5
<211>343
<212>prt
<213>ralstoniasp.
<400>5
metalalysileaspasnalavalleuprogluglyserleuvalleu
151015
valthrglyalaasnglyphevalglyserhisvalvalgluglnleu
202530
leugluhisglytyrlysvalargglythralaargseralaserlys
354045
leualaasnleuglnlysargtrpaspalalystyrproglyargphe
505560
gluthralavalvalgluaspmetleulysglnglyalatyraspglu
65707580
valilelysglyalaalaglyvalalahisilealaservalvalser
859095
pheserasnlystyraspgluvalvalthrproalaileglyglythr
100105110
leuasnalaleuargalaalaalaalathrproservallysargphe
115120125
valleuthrserserthrvalseralaleuileprolysproasnval
130135140
gluglyiletyrleuaspglulyssertrpasnleugluserileasp
145150155160
lysalalysthrleuprogluseraspproglnlysserleutrpval
165170175
tyralaalaserlysthrglualagluleualaalatrplysphemet
180185190
aspgluasnlysprohisphethrleuasnalavalleuproasntyr
195200205
thrileglythrilepheaspprogluthrglnserglyserthrser
210215220
glytrpmetmetserleupheasnglygluvalserproalaleuala
225230235240
leumetpheproglytyrtyrvalseralavalaspileglyleuleu
245250255
hisleuglycysleuvalleuproglnilegluargargargvaltyr
260265270
glythralaglythrpheasptrpasnthrvalleualathrphearg
275280285
lysleutyrproserlysthrpheproalaasppheproaspglngly
290295300
glnaspleuserlyspheaspthralaproserleugluileleulys
305310315320
serleuglyargproglytrpargserileglugluserilelysasp
325330335
leuvalglysergluthrala
340
<210>6
<211>343
<212>prt
<213>ralstoniasp.
<400>6
metalalysileaspasnalavalleuprogluglyserleuvalleu
151015
valthrglyalaasnglyphevalglyserhisvalvalgluglnleu
202530
leugluhisglytyrlysvalargglythralaargseralaserlys
354045
leualaasnleuglnlysargtrpaspalalystyrproglyargphe
505560
gluthralavalvalgluaspmetleulysglnglyalatyraspglu
65707580
valilelysglyalaalaglyvalalahisilealaservalvalser
859095
pheserasnlystyraspgluvalvalthrproalaileglyglythr
100105110
leuasnalaleuargalaalaalaalathrproservallysargphe
115120125
valleuthrserserthrvalseralaleuileprolysproasnval
130135140
gluglyiletyrleuaspglulyssertrpasnleugluserileasp
145150155160
lysalalysthrleuprogluseraspproglnlysserleutrpval
165170175
tyralaalaserlysthrglualagluleualaalatrplysphemet
180185190
aspgluasnlysprohisphethrleuasnalavalleuproasntyr
195200205
thrileglythrilepheaspprogluthrglnserglyserthrala
210215220
glytrpmetmetserleupheasnglygluvalserproalaleuala
225230235240
leuvalproprosertyrtyrvalseralavalaspileglyleuleu
245250255
hisleuglycysleuvalleuproglnilegluargargargvaltyr
260265270
glythralaglythrpheasptrpasnthrvalleualathrphearg
275280285
lysleutyrproserlysthrpheproalaasppheproaspglngly
290295300
glnaspleuserlyspheaspthralaproserleugluileleulys
305310315320
serleuglyargproglytrpargserileglugluserilelysasp
325330335
leuvalglysergluthrala
340
<210>7
<211>343
<212>prt
<213>ralstoniasp.
<400>7
metalalysileaspasnalavalleuprogluglyserleuvalleu
151015
valthrglyalaasnglyphevalglyserhisvalvalgluglnleu
202530
leugluhisglytyrlysvalargglythralaargseralaserlys
354045
leualaasnleuglnlysargtrpaspalalystyrproglyargphe
505560
gluthralavalvalgluaspmetleulysglnglyalatyraspglu
65707580
valilelysglyalaalaglyvalalahisilealaservalvalser
859095
pheserasnlystyraspgluvalvalthrproalaileglyglythr
100105110
leuasnalaleuargalaalaalaalathrproservallysargphe
115120125
valleuthrserserthrvalseralaleuileprolysproasnval
130135140
gluglyiletyrleuaspglulyssertrpasnleugluserileasp
145150155160
lysalalysthrleuprogluseraspproglnlysserleutrpval
165170175
tyralaalaserlysthrglualagluleualaalatrplysphemet
180185190
aspgluasnlysprohisphethrleuasnalavalleuproasntyr
195200205
thrileglythrilepheaspprogluthrglnserglyserthrser
210215220
glyilemetmetserleupheasnglygluvalserproalaleuala
225230235240
leuvalproprosertyrtyrvalseralavalaspileglyleuleu
245250255
hisleuglycysleuvalleuproglnilegluargargargvaltyr
260265270
glythralaglythrpheasptrpasnthrvalleualathrphearg
275280285
lysleutyrproserlysthrpheproalaasppheproaspglngly
290295300
glnaspleuserlyspheaspthralaproserleugluileleulys
305310315320
serleuglyargproglytrpargserileglugluserilelysasp
325330335
leuvalglysergluthrala
340
<210>8
<211>37
<212>dna
<213>人工序列()
<400>8
ctgacctcctccnnkgtttccgctctgattccgaaac37
<210>9
<211>35
<212>dna
<213>人工序列()
<400>9
cagagcggaaacmnnggaggaggtcagaacgaaac35
<210>10
<211>37
<212>dna
<213>人工序列()
<400>10
ctgacctcctccaccnnktccgctctgattccgaaac37
<210>11
<211>35
<212>dna
<213>人工序列()
<400>11
cagagcggamnnggtggaggaggtcagaacgaaac35
<210>12
<211>37
<212>dna
<213>人工序列()
<400>12
cgctgtactgccannktacactattggcactattttc37
<210>13
<211>37
<212>dna
<213>人工序列()
<400>13
gtgccaatagtgtamnntggcagtacagcgttcagag37
<210>14
<211>37
<212>dna
<213>人工序列()
<400>14
cgctgtactgccaaacnnkactattggcactattttc37
<210>15
<211>37
<212>dna
<213>人工序列()
<400>15
gtgccaatagtmnngtttggcagtacagcgttcagag37
<210>16
<211>37
<212>dna
<213>人工序列()
<400>16
cggttccaccnnkggttggatgatgtccctgtttaac37
<210>17
<211>36
<212>dna
<213>人工序列()
<400>17
catcatccaaccmnnggtggaaccggactgagtttc36
<210>18
<211>37
<212>dna
<213>人工序列()
<400>18
cggttccacctccggtnnkatgatgtccctgtttaac37
<210>19
<211>34
<212>dna
<213>人工序列()
<400>19
gggacatcatmnnaccggaggtggaaccggactg34
<210>20
<211>37
<212>dna
<213>人工序列()
<400>20
ggctctggctctgnnkccaccgcagtactacgtttcc37
<210>21
<211>35
<212>dna
<213>人工序列()
<400>21
tactgcggtggmnncagagccagagccggggaaac35
<210>23
<211>37
<212>dna
<213>人工序列()
<400>23
ggctctggctctgatgnnkccgcagtactacgtttcc37
<210>24
<211>35
<212>dna
<213>人工序列()
<400>24
tactgcggmnncatcagagccagagccggggaaac35
<210>25
<211>36
<212>dna
<213>人工序列()
<400>25
ctctgatgccaccgnnktactacgtttccgctgttg36
<210>26
<211>35
<212>dna
<213>人工序列()
<400>26
ggaaacgtagtamnncggtggcatcagagccagag35
<210>27
<211>37
<212>dna
<213>人工序列()
<400>27
cggctggtactnnkgattggaacaccgttctggctac37
<210>28
<211>36
<212>dna
<213>人工序列()
<400>28
tgttccaatcmnnagtaccagccgtaccgtaaacac36
<210>29
<211>37
<212>dna
<213>人工序列()
<400>29
gtactttcgatnnkaacaccgttctggctaccttccg37
<210>30
<211>36
<212>dna
<213>人工序列()
<400>30
cagaacggtgttmnnatcgaaagtaccagccgtacc36
- 还没有人留言评论。精彩留言会获得点赞!