用于全面细胞裂解的组合裂解方案的制作方法
公开了用于裂解细胞以从细胞内部释放或提取基因组dna(gdna)的方法。所公开的方法将加热、洗涤剂和碱组合在单个管中,并且可以在几分钟内完成。该方法组合了通常不相容的洗涤剂和碱,有助于裂解后去除洗涤剂,而无需额外的步骤,并且该组合在无顺序处理的方案中产生了意想不到的协同作用,大大减少了步骤的数量和手动操作时间,同时提高了gdna的表现性(representation),例如,从微生物组样品中难以裂解的细菌中。
背景技术:
许多基于细胞和基于dna的分析方法需要从细胞内部释放dna以促进分析。打开细胞以释放dna被称为‘裂解’。例如,使用dna测序技术研究微生物组的方法首先需要裂解微生物,以便可以提取dna。大多数微生物组是细菌、古细菌和真菌的群落,它们对裂解技术的敏感性(susceptibility)差别很大。差异的易感性对研究人员来说是一个重大问题,研究人员希望确保最难裂解的细菌(通常是革兰氏阳性)和最容易裂解的细菌(通常是革兰氏阴性)与它们在原始样本中的数量成比例。遗憾的是,大多数微生物裂解方案对某些微生物很有效,但对其它微生物则很差。此外,用于回收质粒dna的快速且简单的碱裂解技术通常也会去除微生物的基因组dna,而基因组dna正是微生物组筛选的目标(alkalinelysisopenscellsbutremovesgdna-birnboim,h.c.anddoly,j,arapidalkalineextractionprocedureforscreeningrecombinantplasmiddna,nucleicacidsres.7(6),1979,1513-1524;kohlysisrecoversbacterialgenomicdna-raghunathan,arumughametal."genomicdnaamplificationfromasinglebacterium."appliedandenvironmentalmicrobiology71.6(2005):3342-3347.pmc.web.29sept.2016)。存在本领域已知的基于不同的生物化学方法攻击细胞完整性的多种裂解技术,包括溶菌酶(对肽聚糖细胞壁的酶促攻击)、强碱(化学攻击)、洗涤剂(溶解细胞膜)、珠磨(beadbeating)或摇动(机械破碎)和加热(comparisonoflysistechniquesformicrobiome-sanqingyuan,dorab.cohen,jacquesravel,zaidabdo,larryj.forney.evaluationofmethodsfortheextractionandpurificationofdnafromthehumanmicrobiome.plosone7(3):e33865.doi:10.1371/journal.pone.0033865;dnaextractionmethodsaffectmicrobiomeprofilingresults:wagnermackenzieb,waitedw,taylormw.evaluatingvariationinhumangutmicrobiotaprofilesduetodnaextractionmethodandinter-subjectdifferences.frontiersinmicrobiology.2015;6:130.doi:10.3389/fmicb.2015.00130)。大多数公开的或商业上可获得的dna制备方法使用这些方法中的一种或多种来裂解细胞,通常在顺序步骤中,这些方法可能花费大量时间的,特别是在一次处理多个样品时。虽然个别的裂解方法通常足以应用于不完全或部分裂解产生足够的dna,用于正在执行的方案,但它们通常不会与原始群落成比例地从微生物组样品中产生dna,并且可能不能完全裂解某些微生物。例如,基于洗涤剂的裂解可以破坏具有弱细胞壁和强细胞膜的细胞亚群,但不会打开具有强细胞壁的耐洗涤剂微生物,导致在所得dna制备中,耐洗涤剂细胞中dna的不充分表现或缺乏。在另一个示例中,微生物的珠磨足以裂解具有强细胞膜的细胞,其可以剪切或破坏在该过程早期从易于裂解的细胞释放的dna。另外,各种裂解方法往往彼此不相容,并且如果组合使用,则需要顺序进行。例如,溶菌酶在洗涤剂或强碱的存在下不起作用。某些洗涤剂在强碱的存在下会沉淀。珠磨难以与加热过程组合。虽然可以通过连续运行单独的裂解方案以克服个别的缺点,但这增加了所涉及的复杂性、时间和成本。重要的是,在裂解后必须去除洗涤剂如十二烷基硫酸钠(sds),因为sds会干扰下游的dna操作(manipulation)。另外,取决于方案顺序,某些微生物可能对顺序运行的裂解方案具有抗性。例如,具有坚韧的肽聚糖细胞壁的某些微生物可能具有脂质双层的外包膜,其保护免受强碱或溶菌酶的初始处理。只有多种方法的同时组合可能是有效的,或者是多个步骤的长顺序可能是有效的,以从样品中的所有微生物产生dna。
本文公开的方法通过将多种裂解方法组合成简单、快速的方案来简化用于需要或必须比例裂解的应用和技术(如微生物组的研究)的裂解,该方案在含有不同细胞成分(例如微生物组)的样品中产生更具表现性的dna谱。
技术实现要素:
公开了用于裂解细胞(例如存在于微生物组中的细菌)的方法,其将三个裂解步骤(1)加热、(2)洗涤剂和(3)碱组合成一步,并且可以在短时间内完成,例如几分钟。该方法组合了通常不相容的洗涤剂裂解和碱裂解,允许在裂解后简化洗涤剂的去除,并且重要的是,提高了从难以裂解的细菌中产生基因组dna(gdna)的量。
本文公开了用于裂解样品中细胞,以从细胞中释放dna的方法,包括:(a)将含有生物细胞的水溶液与(i)离子型洗涤剂和(ii)能够沉淀所述离子型洗涤剂的碱混合;(b)将该水溶液加热至至少50℃一段时间,以有效溶解离子型洗涤剂;(c)将该水溶液冷却至40℃或更低一段时间,以有效沉淀离子型洗涤剂;和(d)从水溶液中分离沉淀物,其中在分离沉淀物后,从生物细胞中释放的dna存在于水溶液中。
在一些实施方案中,离子型洗涤剂选自由以下项组成的组:十二烷基硫酸钠(sds)、n-月桂酰肌氨酸钠盐或脱氧胆酸钠。在一些实施方案中,离子型洗涤剂的浓度为约0.1%至约10%。在一些实施方案中,离子型洗涤剂为十二烷基硫酸钠(sds),浓度为约1%。
在一些实施方案中,碱选自由以下项组成的组:氢氧化钾(koh)、氢氧化锂(lioh)、氢氧化钠(naoh)、氢氧化铷(rboh)、氢氧化铯(csoh)、氢氧化钙(ca(oh)2)、氢氧化锶(sr(oh)2)、和氢氧化钡(ba(oh)2)。在一些实施方案中,碱的浓度为约0.05摩尔至约1摩尔。在一些实施方案中,碱为氢氧化钾(koh),浓度为约0.2mol。
在一些实施方案中,将洗涤剂与碱组合,该碱在低温下沉淀该洗涤剂,但允许在高温下溶解该洗涤剂。
在一些实施方案中,离子型洗涤剂为十二烷基硫酸钠(sds),浓度为1wt%,以及碱为含有氢氧化钾(koh)的水溶液,浓度为0.2mol。
在一些实施方案中,加热在约50℃至约100℃的温度范围内进行。在一些实施方案中,加热在约65℃的温度下进行。在一些实施方案中,加热在约95℃的温度下进行。在一些实施方案中,加热进行至少1分钟。
在一些实施方案中,冷却在约4℃至约40℃的温度范围内进行。在一些实施方案中,冷却在约20℃至约25℃的温度下进行。在一些实施方案中,冷却进行至少30秒。
在一些实施方案中,通过选自由以下项组成的组的方法进行分离:离心、过滤、重力沉降。
在一些实施方案中,生物细胞来源于选自由以下项组成的组的样本:粪便、细胞裂解物、组织、血液、肿瘤、舌头、牙齿、口腔拭子、痰、粘液、伤口拭子、皮肤拭子、阴道拭子、或最初从人体、动物、植物或环境样本(包括原始样本、复杂样本、混合物以及微生物组样本)中获得的任何其他生物材料或生物液体。
在一些实施方案中,生物细胞来源于选自由以下项组成的组的生物:多细胞生物、单细胞生物、原核生物、真核生物、微生物、细菌、古细菌、原生动物、藻类和真菌。
附图说明
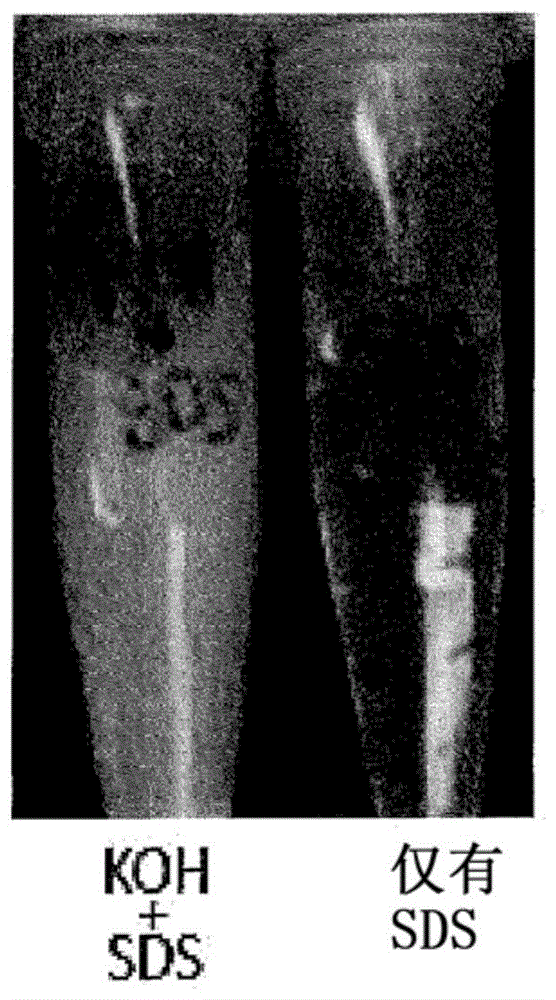
图1a示出了在室温下氢氧化钾中的白色sds沉淀物(koh+sds),管(左)。右边的管示出了koh不存在下的1%sds为澄清溶液。
图1b示出了在室温下氢氧化钾中的白色sds沉淀物(koh+sds),管(右)。左边的管示出了naoh存在下的1%sds为澄清溶液,表明naoh不会沉淀sds。
图2描绘了8个微生物组样品的16srrna基因的pcr条形码结果。8个泳道中每一个泳道的1500个碱基扩增子都标记有不同的dna条形码。
图3是示出了使用洗涤剂和珠磨的顺序裂解步骤或本文所述的组合裂解方法所裂解的多个样品在门水平(phylumlevel)上的平均微生物丰度的比较图。使用海岸线生物群方法(shorelinebiomemethod),更难以裂解的厚壁菌门存在更高的丰度。
图4是示出了使用洗涤剂和珠磨的顺序裂解步骤或本文所述的组合裂解方法所裂解的多个样品在属水平(genuslevel)上的平均微生物丰度的比较图。这表明图3中的门水平丰度对应于属水平的厚壁菌门增加的数量和多样性。
具体实施方式
在说明书结论中,权利要求特别指出并清楚地要求保护被视为本发明的主题。通过以下结合附图的详细描述,本发明的前述和其他目的、特征和优点将变得显而易见。
位于细胞内(例如微生物组中的细菌和古细菌)的dna,可以通过裂解细胞来释放。为了研究微生物组,将目标微生物组中的细胞裂解后,本说明书中所得的dna可以直接测序(‘鸟枪’(shotgun)测序,未示出),或者用作靶向遗传区域(例如16srrna基因,其存在于所有细菌和古细菌中)的pcr扩增的模板。16s基因在本文用作为示例,因为它可以用作微生物的‘指纹’识别方法,需要的测序比鸟枪法少约~1000x。微生物可以使用其16srrna基因序列进行识别,16srrna基因序列在大多数(如果不是全部的话)细菌和古细菌中略有不同。16s基因序列的变异意味着细菌和古细菌的各种物种在16srrna基因中具有特征性dna变异(‘指纹’),其作为那些物种或菌株的标识符。试剂盒、方案和软件能够对样品中的微生物进行全面的指纹分析,并允许使用全长的16srrna基因,以高分辨率一次对多个样品同时进行16srrna指纹分析(参见,例如,markdriscoll和thomasjarvie的标题为“用于多元高通量靶标筛选的dna制备的方法(methodsfordnapreparationformultiplexhighthroughputtargetedscreening)”的美国临时专利申请号62/266,072,其通过引用整体并入本文。)通过将16s基因的dna序列映射到已知读数的数据库,可以在测序后识别已知的微生物。未知的微生物将含有不同于数据库中的任何微生物的16sdna序列,但可以使用其独特的16s序列进行跟踪。此外,样品中每种微生物获得的读数数量可以揭示样品中每种微生物的相对丰度。特定微生物的相对丰度可以作为每个微生物组状态的重要指标。改变微生物的相对丰度,或完全遗漏某些微生物的dna的裂解技术,可能导致测序结果错误地表征所研究的微生物组的状态。本文描述的方法可以用于实现从样品中获得微生物的正确相对丰度。
裂解过程也可用于‘鸟枪’微生物组测序,其中dna在裂解后进行测序,而无需16srrna基因扩增。当研究人员想要读取样品中所有的dna序列,而不仅仅是来自细菌和古细菌的16s基因时,使用鸟枪法。例如,高深度的鸟枪微生物组dna测序可以揭示来自未知细菌/古细菌以及真菌、或多细胞真核生物、病毒dna或任何其他含有dna的生物的完整dna基因组序列。由于完整的细菌基因组可以长达数百万个碱基(比16s基因大数千倍),真菌基因组可以超过一亿个碱基,真核基因组可以长达数十亿个碱基,鸟枪微生物组图谱可能比16srrna基因微生物组谱图需要数千次更多的测序,相应地更多的时间和成本。尽管在该示例中仅讨论了16s图谱方法,但本文描述的裂解方案为鸟枪和16srrna微生物组测序方法提供了相同的益处。
以下是所公开方法的示例,用于16srrna基因微生物组测序方法:
步骤1.将微生物组样本分散到含有2wt%十二烷基硫酸钠(sds)的水溶液中。
步骤2.加入0.4mkoh,sds洗涤剂沉淀为白色絮状物。在该示例中,洗涤剂(1%sds)通过碱(0.2mkoh)沉淀。
步骤3.将管加盖并加热(温度可以在约50℃至约100℃的范围内)。sds在高于50℃的温度下溶解。加热和koh攻击肽聚糖细胞壁,以及sds溶解保护微生物免受koh和加热的破坏性影响的膜。这种步骤的组合是协同的,因为顺序暴露于koh、sds和加热(这与本文描述的组合暴露相反)可能因为微生物细胞壁和膜的构成方式而不会产生相同的结果。加热实际上允许sds在强碱的存在下起作用,从而实现三种不同的裂解技术的独特的同时组合。
步骤4.加热后,将样品恢复到室温(例如,低于40℃)以沉淀sds洗涤剂。
步骤5.将样品短暂离心以将sds洗涤剂颗粒化(pelletize)(无添加必要,快速去除洗涤剂)。
步骤6.将上清液移至含有500mmtris缓冲液或等效物的管中,ph8.5。释放的dna现在可以通过16srrnapcr(如下文所述)进行分析,或者可以进一步储存或纯化用于其他用途。
对每个dna样品进行pcr扩增,该方法为每个样品分配独特的dna条形码。8种不同微生物组的示例性pcr反应在图2中示出,其中根据上述步骤1-6中描述的方案,或者通过具有顺序洗涤剂/珠磨步骤的标准方案裂解人粪便样品1-8。使用1500bp16srrna基因的引物对每个样品进行pcr扩增,每个样品具有不同的dna条形码。合并样品用于pcr后的dna测序。由于每个样品的读数含有独特的识别dna条形码,因此可以通过在样品测序后对其进行分类。通过使用条形码按样品对测序仪的读数输出进行分类,并将其映射到数据库以识别已知的微生物、未知的微生物以及它们在每个样品中的相对比例。
通过条形码分类到来源样品中、通过属的识别、以及通过读数的软件分析定量每个属的读数数量后,对每个微生物组的读数进行比较。根据实验设计,有许多输出的方式可以比较。在图3中,微生物组中每种微生物的数量包括在两个样品的100%堆积条形图中。该方法允许简单、直接地比较微生物组。其他有用的比较包括门水平差异、物种或菌株水平差异,或其他分类水平。
对于多个样品,将使用洗涤剂和珠磨的顺序裂解步骤的标准方法与本文所述的组合裂解方法进行比较。如图3所示,革兰氏阳性厚壁菌门的丰度从微生物组的-30%增加到超过60%。厚壁菌门是具有牢固细胞壁的革兰氏阳性细菌,往往难以裂解。这表明本文所述的裂解方法在裂解具有牢固细胞壁的微生物方面更好。容易裂解的拟杆菌门和疣微菌门按比例地减少,正如在使用100%堆积条形图时所预期的那样。
图4描绘了图3中所示的相同样品在属水平的平均丰度。图4是使用利用洗涤剂和珠磨的顺序裂解步骤的标准方法而未充分表现的厚壁菌门的更高分辨率视图。在丰度上的属水平差异的条形图显示,使用标准顺序裂解法的五种厚壁菌门属(李斯特菌属(listeria)、布劳特氏菌属(blautia)、毛螺菌属(lachnospiraceaincertaesedis)、丁酸球菌属(butyrococcus)、瘤胃球菌属(ruminococcus))未充分表现,并且使用标准方法,拟杆菌属(bacteroides)和阿克曼氏菌属(akkermansia)的相对表现虚高。这与图3中的门水平差异相似,同时显示了使用标准方法可以显著未充分表现个别厚壁菌门属水平。
已经描述了本发明的一个或多个实施方案。然而,应该理解,在不脱离本发明的精神和范围的情况下,可以进行各种修改。因此,其他的实施方案在以下权利要求的范围内。
- 还没有人留言评论。精彩留言会获得点赞!