预测氨基酸变异对蛋白质结构稳定性影响的系统及其方法与流程
本发明属于生物医学数据分析技术领域,具体涉及一种预测氨基酸变异对蛋白质结构稳定性影响的系统及其方法。
背景技术:
预测氨基酸变异对蛋白质稳定性影响的重要指标是野生型蛋白质和变异后蛋白质的自由能变化值ddg。目前已有的预测方法分为两种:一种是直接基于能量用物理公式计算,但由于蛋白质物理结构的不明确,这样计算结果并不准确,且泛化性弱;
另一种是基于已有实验数据,运用机器学习的方法来预测,但这种方法会存在以下这些问题:
(1)准确性差,目前通用的实验数据库protherm中的数据错、漏较多,导致训练数据集质量差,严重影响预测结果的准确性;
(2)泛化性差,该方法使用了大量蛋白质结构相关的输入属性,但对于蛋白质结构未知的情况,则无法预测。
(3)实用性差,该方法缺乏一个支持单个和批量输入,并能将预测结果分为三类(变异导致蛋白质稳定性升高、降低、不变)的系统。
技术实现要素:
为了解决上述问题,本发明旨在提供一种预测氨基酸变异对蛋白质结构稳定性影响的系统及其方法,该系统及其方法可以依据用户提供的氨基酸变异和对应的蛋白质序列,准确预测该氨基酸变异会导致所在蛋白质的结构稳定性升高、降低或不变,以及相应概率,并将结果存储并发送用户保存。
为实现上述技术目的,达到上述技术效果,本发明通过以下技术方案实现:
一种预测氨基酸变异对蛋白质结构稳定性影响的系统,由氨基酸变异信息输入模块、氨基酸变异位点属性计算模块、蛋白质序列属性计算模块、预测稳定性变化模块、预测结果输出模块组成,其中,所述氨基酸变异信息输入模块分别与所述氨基酸变异位点属性计算模块和所述蛋白质序列属性计算模块连接,所述氨基酸变异位点属性计算模块和所述蛋白质序列属性计算模块同时与所述预测稳定性变化模块连接,所述预测稳定性变化模块与所述预测结果输出模块连接;
所述氨基酸变异信息输入模块的功能为获取用户提交的单个或成组的氨基酸变异及其蛋白质序列,并进行用户信息及数据的存储;
所述氨基酸变异位点属性计算模块的功能为根据野生型和变异型位点上的氨基酸情况,提取相对应的aaindex属性特征值,并根据氨基酸变异数据,计算变异后的氨基酸位点物理化学属性特征;
所述蛋白质序列属性计算模块的功能为根据氨基酸变异数据计算相关蛋白质的保守性以及蛋白质属性特征;
所述预测稳定性变化模块的功能为通过基于随机森林的两层三分类算法将氨基酸变异对蛋白质稳定性的影响进行计算和分类,并给出相应概率,作为预测结果;
所述预测结果输出模块的功能为将预测结果生成excel和pdf文件形式,存储并自动邮件发送用户,同时支持用户查询统计。
一种预测氨基酸变异对蛋白质结构稳定性影响的方法,包括以下步骤:
步骤1)所述氨基酸变异信息输入模块首先根据用户输入的氨基酸变异信息,获取其中的氨基酸变异及其蛋白质序列,然后所述氨基酸变异信息输入模块将获取到的氨基酸变异数据以及与氨基酸变异对应的蛋白质序列数据分别传输至所述氨基酸变异位点属性计算模块和所述蛋白质序列属性计算模块,同时,所有输入数据以及提交数据的用户信息将被系统存储;
步骤2)在收到所述氨基酸变异数据后,所述氨基酸变异位点属性计算模块一方面从aaindex数据库中,根据野生型和变异型位点上的氨基酸情况,提取相对应的aaindex属性特征值,另一方面以该氨基酸变异位点为中心,计算相邻位点中各类氨基酸的分布情况,并换算成相应的氨基酸位点物理化学属性特征;然后,所述氨基酸变异位点属性计算模块将提取到的所述aaindex属性特征值和计算出的所述氨基酸位点物理化学属性特征同时传输至所述预测稳定性变化模块;
步骤3)在收到所述与氨基酸变异对应的蛋白质序列数据后,所述蛋白质序列属性计算模块一方面调用blast方法寻找该蛋白质序列的同源序列,然后构造pssm矩阵,计算该蛋白质序列的保守性,作为预测的输入属性特征;另一方面调用protdcal算法,计算该蛋白质序列的蛋白质属性特征;然后,所述蛋白质序列属性计算模块将计算出的该蛋白质序列的保守性和蛋白质属性同时传输至所述预测稳定性变化模块;
步骤4)在收到所述aaindex属性特征值、所述氨基酸位点物理化学属性特征、所述蛋白质序列的保守性以及所述蛋白质属性特征后,所述预测稳定性变化模块采用基于随机森林的两层三分类模型预测方法,将氨基酸变异对蛋白质结构稳定性的影响归为影响蛋白质稳定性降低、升高和不变三类中的一类,并计算出相应的概率,作为预测结果;然后,所述预测稳定性变化模块将计算出的所述预测结果传输至所述预测结果输出模块;
步骤5)在收到所述据测结果后,所述预测结果输出模块首先将所述预测结果进行存储,然后将所述预测结果生成excel和pdf文件形式,并按照任务发送邮件给对应的提交数据的用户;对于注册系统的用户,可输入任务名称以查看对应的预测结果,亦或输入某一个特定蛋白质,统计其上面所有变异对稳定性产生的影响。
进一步的,步骤1)中,输入氨基酸变异信息的方法具体包括以下三种方式:
1)输入单个变异及变异所在的蛋白质序列、实验温度和ph值;
2)一次批量输入多个氨基酸变异及每个变异对应的蛋白质序列、实验温度和ph值;
3)输入指定的蛋白质序列、实验温度和ph值(目的是预测该蛋白质上所有可能的氨基酸变异对蛋白质稳定性的影响)。
进一步的,步骤4)中,所述的基于随机森林的两层三分类模型预测方法的具体步骤如下:
1)按照所述氨基酸变异位点属性计算模块和所述蛋白质序列属性计算模块中的计算方法以及特征提取算法,分别构造两个基于随机森林的分类预测器,第一个分类预测器基于部分重要输入属性,将氨基酸变异分为导致蛋白质稳定性降低和不降低两类;第二个分类预测器基于另一部分输入属性,将变异分为导致蛋白质稳定性升高和不变两类;
2)对所有待预测的氨基酸变异数据分别抽取对应的输入属性、运用所述第一个分类预测器,将氨基酸变异分类成导致蛋白质稳定性降低和不降低两类;
3)对上一步中被预测为导致蛋白质稳定性不降低的变异数据,再次抽取相应的输入属性,运用所述第二个分类预测器,将这部分氨基酸变异分类为导致蛋白质稳定性升高和不变两类;
由此,待预测的氨基酸变异就被分为了影响蛋白质稳定性降低、升高和不变三类。
与现有技术相比,本发明的有益效果是:
本发明的系统及其方法可以依据用户提供的氨基酸变异和对应的蛋白质序列,准确预测该氨基酸变异会导致所在蛋白质的结构稳定性升高、降低或不变,以及相应概率,并将结果存储并发送用户保存。这种三类预测实用性强,且具有较高的预测准确率,特别是在蛋白质结构未知的情况下也能预测变异的影响,泛化性强。对于蛋白质功能分析、辅助蛋白质工程和设计、药物设计等方面具有重大的意义。
上述说明仅是本发明技术方案的概述,为了能够更清楚了解本发明的技术手段,并可依照说明书的内容予以实施,下面以本发明的较佳实施例,并结合附图进行详细说明。本发明的具体实施方式由以下实施例及其附图详细给出。
附图说明
此处所说明的附图用来提供对本发明的进一步理解,构成本申请的一部分,本发明的示意性实施例及其说明用于解释本发明,并不构成对本发明的不当限定。在附图中:
图1为本发明的预测氨基酸变异对蛋白质结构稳定性影响的系统的结构示意图;
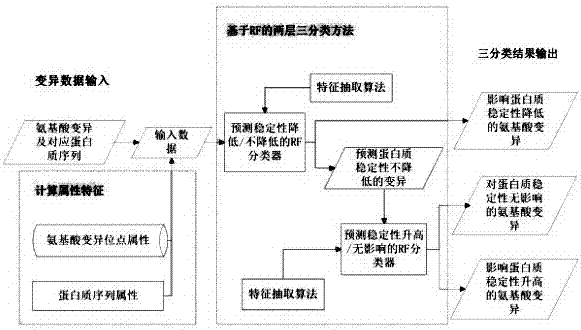
图2为本发明的预测氨基酸变异对蛋白质结构稳定性影响方法中两层三分类算法的示意图;
图3为本发明中预测模型属性特征抽取算法的流程图。
具体实施方式
下面将参考附图并结合实施例,来详细说明本发明。
参见图1所示,一种预测氨基酸变异对蛋白质结构稳定性影响的系统,由氨基酸变异信息输入模块1、氨基酸变异位点属性计算模块2、蛋白质序列属性计算模块3、预测稳定性变化模块4、预测结果输出模块5组成,其中,所述氨基酸变异信息输入模块1分别与所述氨基酸变异位点属性计算模块2和所述蛋白质序列属性计算模块3连接,所述氨基酸变异位点属性计算模块2和所述蛋白质序列属性计算模块3同时与所述预测稳定性变化模块4连接,所述预测稳定性变化模块4与所述预测结果输出模块5连接;
所述氨基酸变异信息输入模块1的功能为获取用户提交的单个或成组的氨基酸变异及其蛋白质序列,并进行用户信息及数据的存储;
所述氨基酸变异位点属性计算模块2的功能为根据野生型和变异型位点上的氨基酸情况,提取相对应的aaindex属性特征值,并根据氨基酸变异数据,计算变异后的氨基酸位点物理化学属性特征;
所述蛋白质序列属性计算模块3的功能为根据氨基酸变异数据计算相关蛋白质的保守性以及蛋白质属性特征;
所述预测稳定性变化模块4的功能为通过基于随机森林的两层三分类算法将氨基酸变异对蛋白质稳定性的影响进行计算和分类,并给出相应概率,作为预测结果;
所述预测结果输出模块5的功能为将预测结果生成excel和pdf文件形式,存储并自动邮件发送用户,同时支持用户查询统计。
参见图1和图2所示,一种预测氨基酸变异对蛋白质结构稳定性影响的方法,包括以下步骤:
步骤1.1)用户通过所述氨基酸变异信息输入模块1输入氨基酸变异信息,具体包括以下三种方式:
(1)输入单个变异及变异所在的蛋白质序列、实验温度和ph值;
(2)一次批量输入多个氨基酸变异及每个变异对应的蛋白质序列、实验温度和ph值;
(3)输入指定的蛋白质序列、实验温度和ph值(目的是预测该蛋白质上所有可能的氨基酸变异对蛋白质稳定性的影响);
步骤1.2)所述氨基酸变异信息输入模块1获取用户提交的变异信息并校验:如果用户提交的是单个变异和对应的蛋白质名称、序列、实验温度、ph值,检查变异位点与序列是否一致,检查温度取值范围为-20至100之间(缺省值为25),ph范围为0至14之间(缺省值为7),否则报错并要求用户重新提交;如果用户提交的是批量氨基酸变异及对应蛋白质名称、序列等信息的文件,检查文件格式与标准格式是否一致,如一致,则逐一检查变异位点与序列是否一致,检查温度取值范围为-20至100之间(缺省值为25),ph范围为0至14之间(缺省值为7),否则报错并要求用户重新提交;
步骤1.3)所述氨基酸变异信息输入模块1获取用户提交的个人信息并校验,构造任务存储:对用户提交的个人信息,主要是邮件地址进行校验,不符合格式规范的报错,以用户邮件地址+编号构造任务名称,与用户提交的变异信息建立链接,一起存储到数据库中;
步骤1.4)所述氨基酸变异信息输入模块1将获取到的氨基酸变异数据以及与氨基酸变异对应的蛋白质序列数据分别传输至所述氨基酸变异位点属性计算模块2和所述蛋白质序列属性计算模块3;
步骤2.1)在收到所述氨基酸变异数据后,所述氨基酸变异位点属性计算模块2从aaindex数据库中,读取变异位点的野生型和变异型氨基酸,然后根据野生型和变异型位点上的氨基酸情况,从aaindex数据矩阵中提取相对应的aaindex属性特征值;
步骤2.2)所述氨基酸变异位点属性计算模块2读取变异位点的残基位置,在氨基酸序列中以该位置为中心,定义一个长度为21的窗口(变异位点前后各取10位),计算窗口中相邻位点中各类氨基酸(按其物理化学属性分类)的分布情况,并换算成相应的氨基酸位点物理化学属性特征;
20种氨基酸按物理化学属性可具体分为6组,包括:
疏水性(hydrophobic):v、i、l、f、m、w、y、c;
带负电荷(negativelycharged):d、e;
带正电荷(positivelycharged):r、k、h;
构象特殊(conformational):g、p;
极性(polar):n、q、s;
其它:a、t;
步骤2.3)所述氨基酸变异位点属性计算模块2将提取到的所述aaindex属性特征值和计算出的所述氨基酸位点物理化学属性特征同时传输至所述预测稳定性变化模块4;
步骤3.1)在收到所述与氨基酸变异对应的蛋白质序列数据后,所述蛋白质序列属性计算模块3通过调用blast方法寻找该蛋白质序列的同源序列,然后构造pssm矩阵,计算该蛋白质序列的保守性,构造3个输入属性特征;
步骤3.2)所述蛋白质序列属性计算模块3通过调用protdcal算法,计算该蛋白质序列的其他19个能量、结构相关的蛋白质属性特征;
步骤3.3)所述蛋白质序列属性计算模块3将计算出的该蛋白质序列的保守性和蛋白质属性同时传输至所述预测稳定性变化模块4;
步骤4.1)在收到所述aaindex属性特征值、所述氨基酸位点物理化学属性特征、所述蛋白质序列的保守性以及所述蛋白质属性特征之前,所述预测稳定性变化模块4首先运用机器学习和人工阅读结合的方法,获得训练数据集并反复校验,保证数据准确性;
步骤4.2)接着所述预测稳定性变化模块4计算预测所需的输入属性,即按所述氨基酸变异位点属性计算模块2和所述蛋白质序列属性计算模块3中的计算方法,计算出训练数据集中氨基酸变异的位点属性和蛋白质序列属性;
步骤4.3)然后所述预测稳定性变化模块4运用特征提取算法,将上一步计算出的属性特征反复迭代,分别获得图2中两个分类预测器所需的输入属性集合;
参见图3所示,所述的特征提取算法的详细描述如下:
阶段一
4.3.1)开始;
4.3.2)对于5重交叉验证中的5个训练数据集,每次留下1个作为测试集,另外4个作为训练集;基于所有输入属性分别在5个训练集上建立基于随机森林(rf)的分类预测器;
4.3.3)计算5重交叉验证的预测准确率,并按照rf中的重要性将输入属性降序排列;
4.3.4)去掉排列在最末的输入属性;
4.3.5)基于剩余输入属性重新训练分类预测器;
4.3.6)判断是否仅有8个输入属性剩余,若是,则进行下一步,若否,则返回至步骤4.1.3);
4.3.7)在5个训练集上分别计算预测准确率,并将对应的分类预测器上抽取出来的输入属性保留并储存;
阶段二
4.3.8)建立一个输入属性集合,包含阶段一种在5个训练集上抽取出的所有输入属性;
4.3.9)基于输入属性集合中所有属性训练一个分类预测器,并将这些属性按照rf中的重要性排序,选取重要性最高的属性进入最终的属性集;
4.3.10)基于最终属性集中的所有属性训练一个分类预测器;
4.3.11)将输入属性集里面剩下的属性里排序最高的一个加入最终属性集;
4.3.12)重新训练分类预测器,如果能够增加预测准确率,则留下最新加入的特征属性,否则去掉;
4.3.13)判断属性集合中所有属性是否都已遍历过,若是,则进行下一步,若否,则返回至步骤4.3.10);
4.3.14)得到最终抽取到的属性集合;
4.3.15)结束;
步骤4.4)紧接着,在输入属性集合中提取出两部分重要输入属性,分别构造两个基于随机森林的分类预测器;第一个分类预测器将氨基酸变异分为导致蛋白质稳定性降低和不降低两类;第二个分类预测器将变异分为导致蛋白质稳定性升高和不变两类;
步骤4.5)参见图2所示,在收到所述aaindex属性特征值、所述氨基酸位点物理化学属性特征、所述蛋白质序列的保守性以及所述蛋白质属性特征之后,所述预测稳定性变化模块4首先对所有待预测的氨基酸变异数据分别抽取对应的输入属性,并且运用所述第一个分类预测器,将氨基酸变异分类成导致蛋白质稳定性降低和不降低两类;
步骤4.6)然后所述预测稳定性变化模块4对上一步中被预测为导致蛋白质稳定性不降低的变异数据,再次抽取相应的输入属性,并且运用所述第二个分类预测器,将这部分氨基酸变异分类为导致蛋白质稳定性升高和不变两类;
由此,待预测的氨基酸变异就被分为了影响蛋白质稳定性降低、升高和不变三类;
步骤4.7)所述预测稳定性变化模块4将计算出的所述预测结果传输至所述预测结果输出模块5;
步骤5.1)在收到所述据测结果后,所述预测结果输出模块5将所述预测结果进行存储;
步骤5.2)根据任务,所述预测结果输出模块5将所述预测结果生成excel和pdf文件形式,并按照任务发送邮件给对应的提交数据的用户;
步骤5.3)对于注册系统的用户,可输入任务名称以查看对应的预测结果,亦或输入某一个特定蛋白质,统计其上面所有变异对稳定性产生的影响。
以上所述仅为本发明的优选实施例而已,并不用于限制本发明,对于本领域的技术人员来说,本发明可以有各种更改和变化。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!