基于计算云平台的微生物多样性交互分析系统及其方法与流程
本发明涉及生物信息分析技术领域,尤其涉及一种基于计算云平台的微生物多样性交互分析系统及其方法。
背景技术:
微生物作为生物类群的重要组成部分,具有巨大的种群数量,广泛的分布范围,极强的适应与繁殖能力。一些非培养的研究微生物多样性的方法,如dna指纹图谱技术、微阵列基因芯片等,克服了传统培养技术的一些缺点,获得了一部分不能分离培养的土壤微生物信息。高通量测序方法实现了大规模微生物基因直接测序,极大的提高了测序通量,在微生物物种和结构多样性研究中可获得更多的信息。
生物信息学数据分析是高通量测序应用于微生物多样性研究的最关键步骤。illuminahiseq运行一次产生的数据量高达1000g,个人计算机和工作站显然无法完成这些数据的处理工作。
高通量数据处理时要对数据进行调整、筛选、比对,需要生物信息学工作者掌握脚本程序编写能力。现有微生物多样性生物信息分析主要由三部分组成。标准生物信息学分析是整个多样性分析的基础,其结果呈现包括有效测序序列结果统计、优质序列统计、各样本序列数目统计、otu(操作分类单元)生成、稀释曲线分析、多样性指数分析、样品otu分布及分类学信息。高级生物信息学分析呈现方式包括:聚类分析、多样品群落结构分析以及组间显著性差异分析等。个性化生物信息学分析包括:pca分析、rda/cca分析等。
现有技术的业务流程采用手动的方式,工作效率较低,因此无法满足市场的需求。
技术实现要素:
针对上述技术中存在的不足之处,本发明提供一种基于计算云平台的微生物多样性交互分析系统及其方法,以解决个人计算机和工作站无法完成数据处理和现有手动业务流程效率低的问题。
为实现上述目的,本发明提供一种基于计算云平台的微生物多样性交互分析系统,包括
项目管理模块,用于对项目信息进行查看和管理,通过项目、任务、应用和文件对所有状态的分析项目进行综合管理;
基础分析任务提交模块,用于设置基础参数运行任务,并将结果以及原始数据按照预设格式整合封装成相应的项目文件;
交互式结果分析模块,用于根据用户请求生成分析结果,并将结果可视化展示;
所述项目管理模块通过基础分析任务提交模块与交互式结果分析模块相连接;
首先,向本地集群服务器上传测序数据,在项目管理模块中建立项目,同时在本地集群服务器上传客户的数据库或者选择使用线上的数据库,并且在该项目管理模块中可以进行项目锁定或者共享给他人操作;
然后,在基础分析任务提交模块中,用户可以通过可视化界面对测序数据进行参数分析,分析之后产生项目文件;分析之前,先进行判定数据质控是否合格,若合格则进行参数分析;若不合格则直接返回报错;
最后,产生的项目文件传送至交互式结果分析模块中进行交互式分析,根据用户需求对项目文件进行二次分析和统计,得到直观呈现的报告。
为实现上述目的,本发明还提供一种基于计算云平台的微生物多样性交互分析方法,包括以下步骤:
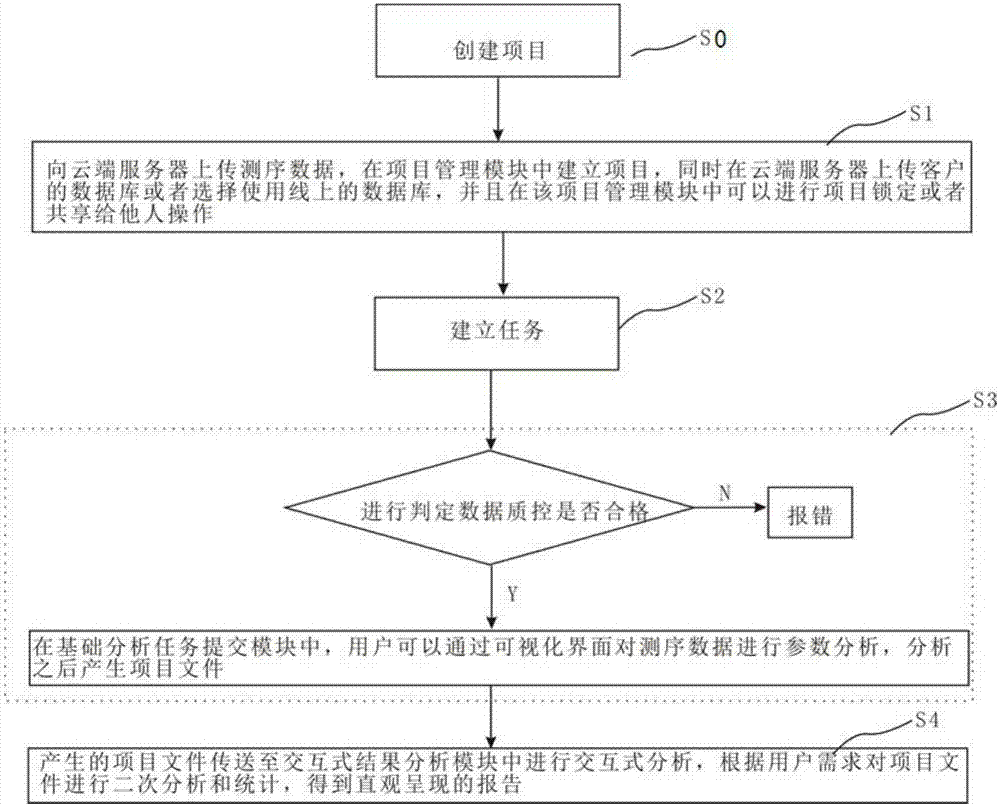
步骤0,创建项目;
步骤1,向本地集群服务器上传测序数据,在项目管理模块中建立项目,同时在本地集群服务器上传客户的数据库或者选择使用线上的数据库,并且在该项目管理模块中可以进行项目锁定或者共享给他人操作;
步骤2,建立任务;
步骤3,在基础分析任务提交模块中,用户可以通过可视化界面对测序数据进行参数分析,分析之后产生项目文件;分析之前,先进行判定数据质控是否合格,若合格则进行参数分析;若不合格则直接返回报错;
步骤4,产生的项目文件传送至交互式结果分析模块中进行交互式分析,根据用户需求对项目文件进行二次分析和统计,得到直观呈现的报告。
其中,所述步骤3中在进行测序数据参数分析时,用户可以自行选择分组方案,并且配置环境因子,选择需要的数据库,选择完成之后再进行分析。
其中,所述项目管理模块,还可用于查看和管理上传的相关文件或由分析产生的相关文件,可对相关文件进行上传、查找、复制、移动、删除和下载操作;项目管理模块还用于标记项目状态进度,项目状态进度可为未开始、进行中、已完成、已终止和问题;项目管理模块还用于查看任务运行的状态与日志信息;且项目管理模块,还用于共享项目,并管理成员权限。
其中,物种分类数据库存储在本地集群服务器内,使用线上的数据库包括16s、18s、its和fungi,也可上传自定义数据库。
其中,所述基础分析任务提交模块可用于数据质控统计、otu聚类、物种分类、alpha多样性和beta多样性研究。
其中,所述交互式结果分析模块还用于高级生物信息学分析和个性化生物信息学分析,包括群落组成分析、组间显著性差异分析、样本分组分析、lefse多级物种差异判别分析和进化分析;交互式结果分析模块还用于更改分组方案和挑选分析样品。
其中,所述交互式结果分析模块包括图表工具,可实现更改颜色方案、形状方案、柱形方向;可显示图例、点名称、合并或排序功能;交互式结果分析模块分析结果可存入报告,在报告中展示;交互式结果分析模块分析结果图可以png、jpeg、pdf、svg格式下载;交互式结果分析模块的报告格式可为html和pdf。
其中,所述项目管理模块、基础分析任务提交模块和交互式结果分析模块的操作均基于html+css+jquery的前端页面和php+alpha服务器后台,交互分析模块接收到任务执行命令后调动perl、c、python、r计算机语言的服务器端脚本对测序数据进行基本分析。
其中,所述基础分析任务提交模块在对所述测序数据进行分析的不同阶段,从其存储的分析软件中选取相应的分析软件对所述测序数据进行分析。
本发明的有益效果是:与现有技术相比,本发明提供的基于计算云平台的微生物多样性交互分析系统及其方法,该发明主要包括项目管理模块、基础分析任务提交模块和交互式结果分析模块三大模块,该系统及方法借助云计算技术,通过网络方便的获取大量测序数据分析所需要的基础计算资源,满足了大数据背景下研究人员对于基础计算资源的巨大需求。同时,基于计算云平台的微生物多样性交互分析方法提供了高集成化的数据分析平台,无需用户自己整合各类分析软件,搭建分析流程,真正意义上实现了一键化生物信息分析。此外,基于计算云平台的微生物多样性分析可灵活设置分组,支持多种算法,呈现可视化的结果和交互式的报告。最后,基于计算云平台的微生物多样性分析内容全面,不仅涵盖微生物多样性分析的基础分析和高级分析,还包括部分个性化分析,更好满足用户需求。
附图说明
图1为本发明的基于计算云平台的微生物多样性交互分析系统方框图;
图2为本发明的基于计算云平台的微生物多样性交互分析方法流程图;
图3为本发明中基础分析任务提交模块的微生物多样性分析的示意图;
图4为本发明中微生物多样性分析新建项目示意图;
图5为本发明中微生物多样性基础任务参数提交示意图。
图6为本发明中微生物多样性交互分析示意图;
图7为本发明中微生物多样性pca图表工具示意图;
图8为本发明中微生物多样性分析报告示意图。
主要元件符号说明如下:
10、项目管理模块11、基础分析任务提交模块
12、交互式结果分析模块。
具体实施方式
为了更清楚地表述本发明,下面结合附图对本发明作进一步地描述。
请参阅图1,本发明的基于计算云平台的微生物多样性交互分析系统,包括
项目管理模块10,用于对项目信息进行查看和管理,通过项目、任务、应用和文件对所有状态的分析项目进行综合管理;
基础分析任务提交模块11,用于设置基础参数运行任务,并将结果以及原始数据按照预设格式整合封装成相应的项目文件;
交互式结果分析模块12,用于根据用户请求生成分析结果,并将结果可视化展示;
项目管理模块通过基础分析任务提交模块与交互式结果分析模块相连接;
首先,向本地集群服务器上传测序数据,在项目管理模块中建立项目,同时在本地集群服务器上传客户的数据库或者选择使用线上的数据库,并且在该项目管理模块中可以进行项目锁定或者共享给他人操作;测序数据可以是fastq格式的数据;
然后,在基础分析任务提交模块中,用户可以通过可视化界面对测序数据进行参数分析,分析之后产生项目文件;分析之前,先进行判定数据质控是否合格,若合格则进行参数分析;若不合格则直接返回报错;
最后,产生的项目文件传送至交互式结果分析模块中进行交互式分析,根据用户需求对项目文件进行二次分析和统计,得到直观呈现的报告。
请进一步参阅图2,本发明还提供一种基于计算云平台的微生物多样性交互分析方法,包括以下步骤:
步骤s0,创建项目;
步骤s1,向本地集群服务器上传测序数据,在项目管理模块中建立项目,同时在本地集群服务器上传客户的数据库或者选择使用线上的数据库,并且在该项目管理模块中可以进行项目锁定或者共享给他人操作;
步骤s2,建立任务;
步骤s3,在基础分析任务提交模块中,用户可以通过可视化界面对测序数据进行参数分析,分析之后产生项目文件;分析之前,先进行判定数据质控是否合格,若合格则进行参数分析;若不合格则直接返回报错;
步骤s4,产生的项目文件传送至交互式结果分析模块中进行交互式分析,根据用户需求对项目文件进行二次分析和统计,得到直观呈现的报告。
相较于现有技术的情况,本发明提供的基于计算云平台的微生物多样性交互分析系统及其方法,该发明主要包括项目管理模块10、基础分析任务提交模块11和交互式结果分析模块12三大模块,该系统及方法借助云计算技术,通过网络方便的获取大量测序数据分析所需要的基础计算资源,满足了大数据背景下研究人员对于基础计算资源的巨大需求。同时,基于计算云平台的微生物多样性交互分析方法提供了高集成化的数据分析平台,无需用户自己整合各类分析软件,搭建分析流程,真正意义上实现了一键化生物信息分析。此外,基于计算云平台的微生物多样性分析可灵活设置分组,支持多种算法,呈现可视化的结果和交互式的报告。最后,基于计算云平台的微生物多样性分析内容全面,不仅涵盖微生物多样性分析的基础分析和高级分析,还包括部分个性化分析,更好满足用户需求。
在本实施例中,步骤s2中在进行测序数据参数分析时,用户可以自行选择分组方案,并且配置环境因子,选择需要的数据库,选择完成之后再进行分析。
在本实施例中,项目管理模块,还可用于查看和管理上传的相关文件或由分析产生的相关文件,可对相关文件进行上传、查找、复制、移动、删除和下载操作;项目管理模块还用于标记项目状态进度,项目状态进度可为未开始、进行中、已完成、已终止和问题;项目管理模块还用于查看任务运行的状态与日志信息;且项目管理模块,还用于共享项目,并管理成员权限。
在本实施例中,物种分类数据库存储在本地集群服务器内,使用线上的数据库包括16s、18s、its和fungi,也可上传自定义数据库。
请进一步参阅图3,基础分析任务提交模块可用于数据质控统计、otu聚类、物种分类、alpha多样性和beta多样性研究。数据质控统计根据所选fastq文件判定文件中包含的样本数目,有效序列数目,有效碱基数目,序列平均长度,序列长度分布等信息。otu聚类根据参数otu序列相似度对序列进行归类操作,而物种分类则通过选择数据库,通过rdpclassifier贝叶斯算法对otu代表序列进行分类学比对,得到每个otu对应的物种分类信息。alpha多样性反映微生物群落的丰度和多样性,包括一系列统计学分析指数估计环境群落的物种丰度和多样性,可选择多个指数进行分析。beta多样性表示的是微生物群落构成的比较,可选择某种分析方法进行运算,在交互分析页面可进一步设置样本分组和设置不同的距离算法进行分析。在此生成的结果可在交互式分析模块中可视化查看,也可在项目文件中查看对应结果文件。
在本实施例中,交互式结果分析模块还用于高级生物信息学分析和个性化生物信息学分析,包括群落组成分析、组间显著性差异分析、样本分组分析、lefse多级物种差异判别分析和进化分析;交互式结果分析模块还用于更改分组方案和挑选分析样品。交互式结果分析模块包括图表工具,可实现更改颜色方案、形状方案、柱形方向;可显示图例、点名称、合并或排序功能;交互式结果分析模块分析结果可存入报告,在报告中展示;交互式结果分析模块分析结果图可以png、jpeg、pdf、svg格式下载;交互式结果分析模块的报告格式可为html和pdf。项目管理模块、基础分析任务提交模块和交互式结果分析模块的操作均基于均基于html+css+jquery的前端页面和php+alpha服务器后台,交互分析模块接收到任务执行命令后调动perl、c、python、r计算机语言的服务器端脚本对测序数据进行基本分析。基础分析任务提交模块在对测序数据进行分析的不同阶段,从其存储的分析软件中选取相应的分析软件对测序数据进行分析。
请进一步参阅图4,为基于本发明创建项目和任务步骤,点击分析平台进入我的项目,点击新建项目,填写项目名称,项目描述,选择领域标签,物种标签。点击已建立的项目名称,新建任务。请参阅图5,参数的设置主要包括数据质控统计、otu聚类、物种分类、alpha多样性和beta多样性参数设置。在此可选择输入fastq格式序列文件也可选择包含每个样本序列的fastq序列文件夹。在此可对fastq文件中的样本信息进行检测,可编辑样本名称,并选择分析使用的样本,若有多行样本的分析名称相同则分析中合并为同一样本。与此同时,可选择样本分组文件与环境因子表,otu序列相似度,一般默认为0.97,物种分类置信度,一般默认为0.7,采用rdpclassifier贝叶斯算法对97%相似水平的otu代表序列进行分类学比对,系统已有数据库包括16s,18s,its,fungi等,也可上传自定义数据库,满足格式要求即可。alpha多样性可选择多样性估计指数和稀释曲线多样性指数进行分析,beta多样性可选择多元分析方法和距离算法。
交互式结果分析模块的分析参阅图6,主要包括数据质控,物种注释与评估,alpha多样性研究,beta多样性研究,物种差异分析。其中数据质控包括样本信息统计和样本的序列长度分布两个模块。样本信息统计表统计了每个样本的序列数、碱基数、平均长度、最短序列长度和最长序列长度等信息。样本的序列长度分布通过统计和绘制柱形图查看样本序列在各个长度区间范围中的序列数分布情况。在柱形图的图表工具中可选择样本、分组方案、颜色以及步长。在序列长度分布图上可以选中实现图形的部分区域的放大,也可使用图形右下角的放大工具对整个图形进行放大。点击存入报告按钮,可将该图片存到报告相应位置。
物种注释与评估包括otu分析、venn图分析、群落组成分析和样本与物种关系图四个模块。其中otu分析分为otu统计和pan/coreotu。其中otu统计用于显示otu统计的相关信息,可对otu表进行抽平和物种筛选等操作,其中抽平可按照最小样本序列数也可自定义样本序列数进行,物种筛选可按照不同分类水平的物种(otu)进行,也可根据物种(otu)在样本中的分布进行,也可根据物种(otu)的序列数总和进行。将参数设置完成后,点击运行可生成新的otu表格,otu分析的结果显示分为otu物种分类统计和等级丰度曲线(rank-abundance)。otu物种分类统计包括信息统计和otu表,信息统计可统计各个分类水平的物种(otu)数目。otu表可显示物种(otu)名称和数目,并可根据物种(otu)名称进行查找。rank-abundance曲线和otu分类统计都可选择上述运行生成的结果表和选择当前显示的最低级别的分类水平。pan/coreotu的参数设置与otu统计基本一致,选择otu表、分类水平、分组方案后点击运行进行pan/coreotu的分析。分析结果分为pan/core曲线图和pan/core曲线表,运行结束后在pan/core结果选择运行后的panotu或coreotu结果,计算类别中选择显示panotu还是coreotu。venn图分析可点击otu表的下拉框选择要分析的otu表,分类水平的下拉框选择进行分析的分类水平,venn分析还需要分组信息,点击分组方案下拉框可选择分组或创建分组,点击运行进行分析。venn结果包括venn统计表和venn图以及venn物种分布饼图。点击保存可将图片保存至本地,点击存入报告则将图片存入静态报告中。群落组成分析,同上选择otu表、分类水平、分组方案后即可运行,产生的结果可绘制柱图(多样本bar图)、热图(heatmap图)、pie图和多级物种饼图。样本与物种分布关系以样本与物种共现性弦图展示,只需要选择otu表即可生成对应的样本与物种共现性弦图。
alpha多样性研究包括多样性指数和稀释性曲线两个模块,其中多样性指数分析包括多样性指数和组间t检验,多样性指数选择相应的otu表、分类水平、分组方案和指数类型即可运行。可供选择的指数有:ace、bergerparker、boneh、bootstrap、bstick、chao、coverage、efron、geometric、goodscoverage、heip、invsimpson、jack、logseries、npshannon、nseqs、qstat、shannon、shannoneven、shen、simpsoneven、smithwilson、sobs、solow。结果为多样性指数柱状图和多样性指数表,多样性指数柱状图点击切换指数可切换显示某一指数的柱状图。针对alpha多样性指数表,用组间t检验来比较两组样本中各指数的差异是否显著,计算的方法可为student’sttest,welch’sttest和wilcoxonrank-sumtest。若选择的分组超过两个,则将所有分组类别进行两两组合,分别分析每个两两分组的多样性指数差异是否显著,可点击切换组别的下拉按钮进行切换显示某个两两分组比较的结果表,点击指数类型旁边的图表即可显示该指数类型的柱状图。稀释性曲线分析包括稀释性曲线表和稀释性曲线图,选择otu表,分类学水平,分组方案和指数类型即可运行产生结果。指数类型包括:ace、bootstrap、chao、coverage、heip、invsimpson、jack、npshannon、shannon、shannoneven、simpson、simpsoneven、smithwilson、sobs。针对稀释性曲线表,可选择切换指数按钮选择显示相应的稀释性曲线表。
beta多样性研究包括beta多样性分析、环境因子分析和样本分组分析三个模块。其中,beta多样性分析包括hcluster层级聚类、pca主成分分析、pcoa主坐标分析和nmds非度量多维分析。hcluster层级聚类选择otu表、分类水平、分组方案、距离算法和物种层级聚类方式等参数后即可运行。结果展示为hcluster聚类图和样本距离热图。pca主成分分析,选择otu表、分类水平和分组方案后即可运行,也可选择环境因子数据进行分析,结果分为pca图和pca相关数据表。在pca图,可利用图表工具设置x轴、y轴,改写x轴、y轴和图主标题名称,颜色方案,形状方案,显示环境因子和点名称。pca相关数据表包括物种/环境因子相关数据和pca图坐标表,其中物种/环境因子相关数据又包括物种主成分贡献度表和数量型环境因子表,pca图坐标表又包括样本坐标表和数量型环境因子坐标表。pcoa主坐标分析需设置的参数有otu表、分类水平、分组方案和距离算法,结果展示为pcoa图和pcoa相关数据表。pcoa图表工具可设置x轴、y轴,颜色方案、形状方案和显示点名称,pcoa相关数据包括样本坐标表和矩阵特征值表。nmds非度量多维分析输入的参数同pcoa一样,得到的结果分为nmds图和nmds相关数据表。环境因子分析包括rda/cca、db-rda、manteltest和pearsoncorrelation分析4个部分。rda/cca分析参数设置包括otu表、分类水平、分组方案、环境因子数据,结果也包括rda/cca图和rda/cca相关数据表包括样本坐标表、物种坐标表、dca分析结果表和数量型环境因子坐标表。db-rda分析参数设置包括otu表、分类水平、分组方案、环境因子数据和距离算法,结果也分为db-rda图和db-rda相关数据表。manteltest分析输入的参数有otu表、分类水平、分组方案、群落距离矩阵算法、环境因子表、环境因子矩阵算法和控制单位,结果展示为manteltest判断结果和结果矩阵表,结果矩阵表又包括样本群落距离矩阵、样本环境因子距离矩阵和样本控制矩阵。pearsoncorrelation分析输入的参数有otu表、分类水平、分组方案和环境因子表,展示结果为相关性热图和相关性矩阵。样本分组分析模块包括anosim/adonis分析和pls-da分析。anosim相似性分析输入的参数有otu表、分类水平、分组方案、距离算法和置换次数,结果为组间距离盒状图和anosim/adonis分析结果表。pls-da分析只需输入otu表、分类水平和分组方案即可,结果为pls-da图和pls-da相关数据表。
物种差异分析模块包括组间显著性差异检验和lefse多级物种差异判别分析。其中组间显著性差异检验包括多组比较、两组比较和两样本比较。多组比较需要输入的参数有otu表、分类水平、分组方案、检验方法、多重检验校正和ci计算方法,结果展示为多物种差异检验柱形图和差异检验统计表,点击差异检验统计表中某一物种的柱形图、箱线图或post-hoc图图标即可查看相应的单物种比较图。两组比较输入的参数为otu表、分类水平、分组方案、检验方法、单双尾检验、显著性水平、多重检验校正和ci计算方法。结果展示为两组比较多物种差异检验柱形图和差异性检验统计表,点击差异性检验统计表中某一物种(otu)的柱形图或箱线图图表即可查看相应的单物种比较图。两样本比较参数设置包括otu表、分类水平、样本1、样本2、检验方法、单双尾检验、显著性水平、多重检验校正和ci计算方法。结果展示为多物种差异检验柱形图和差异检验统计表。lefse多级物种差异判别分析参数设置包括otu表、分组方案、二级分组、lda阈值和多组比较策略,生成的结果展示为lefse多级物种层级树图和lda判别柱形图和lda判别结果表。
进化分析模块主要是系统发生进化树,参数设置包括otu表、分类水平、颜色设置和样本分组,结果展示为进化树图和环形进化树图。
本发明的分析报告参阅图7-8,在交互分析页面点击存入报告按钮,即可将分析所得结果存入报告相应位置。在报告中可查看使用软件及方法,以及生物学意义上的解释。
本发明所述的一种基于计算云平台的微生物多样性交互分析方法交互分析所产生的结果文件按照预设格式整合封装成相应的项目文件。产生的结果文件可下载,供进一步的分析。
本发明所述的一种基于计算云平台的微生物多样性交互分析方法,用户可以自由选择测序数据,自定义所需参数,利用所述配置文件对所述测序数据进行基本分析,并以图表和报告的形式呈现,因而相较于采用手动方式进行分析的现有技术,本发明采用自动的方式进行分析,能够提高微生物多样性分析的效率。
本发明实施例中,还包括高级生信分析和个性化生信分析,高级生信分析和个性化生信分析是在基本分析的基础上做进一步分析,提高基本分析所得数据的利用效率,有针对的、更深层的挖掘数据信息,使得微生物多样性分析方式不再局限于传统业务线流程的单一性,提高了微生物多样性分析的效率和数据利用率,一个基本数据可做无限次高级分析和个性化分析,节省了时间和实验成本。
本实施例中,基于计算云平台的微生物多样性交互分析方法集成业内普遍认可的qiime、mothur对测序数据进行分析,生成的图表符合专业期刊的要求。在工作流程界面,按照步骤一到步骤六的顺序,选择感兴趣的拼接后的fastq格式的文件,上传分组信息表以及环境因子表,选择比对数据库,设定分析参数,点击保存并运行,即可实现微生物多样性项目的交互分析。在交互分析页面中,用户可以自由执行挑选样本、更改分组、改变配色等操作,不必和分析员反复沟通等待,大大缩短了项目周期。
以上公开的仅为本发明的几个具体实施例,但是本发明并非局限于此,任何本领域的技术人员能思之的变化都应落入本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!