氨基酸序列特征提取方法、装置、存储介质及电子设备与流程
本公开涉及生物信息领域,具体地,涉及一种氨基酸序列特征提取方法、装置、存储介质及电子设备。
背景技术:
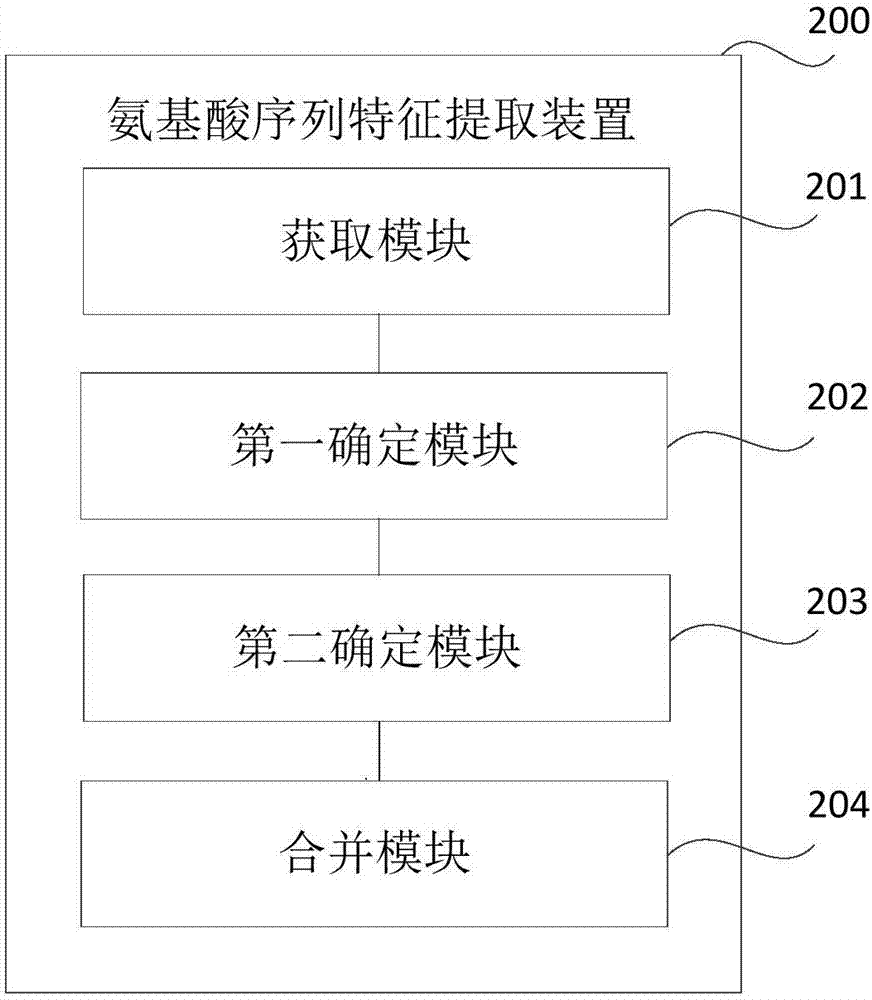
:在生物信息学中,蛋白质氨基酸序列的信息起到了关键作用,并且,蛋白质由20种天然氨基酸经过线性组合而成,这些线性序列中包含着大量生命体特征信息,通常被认为能够解释和表达所有机体内的生命活动。因此,氨基酸序列通常作为信息源与机器学习技术结合应用于预测蛋白质功能。具体来说,首先收集由已知具有某类功能和未具有某类功能的氨基酸序列组成的数据集;然后从未具有某类功能的氨基酸序列中进行特征提取,并表示成特征向量;最后根据该特征向量,利用机器学习算法对蛋白质功能进行预测。由于,机器学习预测中,需要输入固定维度的特征向量,因此目前的氨基酸序列特征提取方法,仅仅能根据计算出的氨基酸单个出现频率或者利用氨基酸理化性质参数来对氨基酸序列进行特征提取,能够提取的特征非常有限,并不能充分表达该氨基酸序列中承载的大量信息。技术实现要素:为了解决相关技术中存在的问题,本公开提供一种氨基酸序列特征提取方法、装置、存储介质及电子设备。为了实现上述目的,本公开提供一种氨基酸序列特征提取方法,所述方法包括:获取待进行特征提取的氨基酸序列;确定所述氨基酸序列的位置特异性得分矩阵PSSM,其中,所述位置特异性得分矩阵PSSM的大小为N×M,N表示所述氨基酸序列的序列长度,M表示预设氨基酸的总数,所述位置特异性得分矩阵PSSM中的每个分数PSSM(n,m)表示在第n行的氨基酸的位置下,第m列的预设氨基酸出现的倾向程度,且-S≤PSSM(n,m)≤S,其中,N、M和S均为正整数,且1≤n≤N,1≤m≤M;确定每种预设氨基酸在[-S,S]分数区间内的得分分布;合并M种预设氨基酸的所述得分分布,以获得所述氨基酸序列的特征向量。可选地,所述确定每种预设氨基酸在[-S,S]分数区间内的得分分布,包括:针对每种预设氨基酸,根据该预设氨基酸的全部分数,统计在[-S,S]分数区间内的每个整数分数的出现频率,并按照所述整数分数由小及大的顺序,将所述整数分数的出现频率组合成1×(2S+1)的特征向量,所得特征向量作为该预设氨基酸在[-S,S]分数区间内的得分分布。可选地,所述确定每种预设氨基酸在[-S,S]分数区间内的得分分布,包括:设每种预设氨基酸在[-S,S]分数区间内的得分分布为f(m),其中,f(m)为1×(2S+1)的特征向量,通过以下公式计算f(m)中的每个元素值fj(m),其中,j=0,…,2S:可选地,所述M种预设氨基酸为20种天然氨基酸。本公开还提供一种氨基酸序列特征提取装置,所述装置包括:获取模块,用于获取待进行特征提取的氨基酸序列;第一确定模块,用于确定所述获取模块获取到的所述氨基酸序列的位置特异性得分矩阵PSSM,其中,所述位置特异性得分矩阵PSSM的大小为N×M,N表示所述氨基酸序列的序列长度,M表示预设氨基酸的总数,所述位置特异性得分矩阵PSSM中的每个分数PSSM(n,m)表示在第n行的氨基酸的位置下,第m列的预设氨基酸出现的倾向程度,且-S≤PSSM(n,m)≤S,其中,N、M和S均为正整数,且1≤n≤N,1≤m≤M;第二确定模块,用于确定每种预设氨基酸在[-S,S]分数区间内的得分分布;合并模块,用于合并M种预设氨基酸的所述得分分布,以获得所述氨基酸序列的特征向量。可选地,所述第二确定模块用于:针对每种预设氨基酸,根据该预设氨基酸的全部分数,统计在[-S,S]分数区间内的每个整数分数的出现频率,并按照所述整数分数由小及大的顺序,将所述整数分数的出现频率组合成1×(2S+1)的特征向量,所得特征向量作为该预设氨基酸在[-S,S]分数区间内的得分分布。可选地,所述第二确定模块用于:设每种预设氨基酸在[-S,S]分数区间内的得分分布为f(m),其中,f(m)为1×(2S+1)的特征向量,通过以下公式计算f(m)中的每个元素值fj(m),其中,j=0,…,2S:可选地,所述M种预设氨基酸为20种天然氨基酸。本公开还提供一种计算机可读存储介质,其上存储有计算机程序,该程序被处理器执行时实现本公开提供的所述氨基酸序列特征提取方法的步骤。本公开还提供一种电子设备,包括:本公开提供的所述计算机可读存储介质;以及一个或者多个处理器,用于执行所述计算机可读存储介质中的程序。在上述技术方案中,由于确定出的氨基酸序列的位置特异性得分矩阵PSSM中蕴含有同源蛋白质信息,因此该蕴含有同源蛋白质信息的位置特异性得分矩阵PSSM表示成的特征向量中能够融入更多的序列保守信息,更有利于氨基酸序列与其他生物高分子物质之间(例如,氨基酸序列之间、氨基酸序列与DAN/RNA之间、氨基酸序列与糖类及脂质类之间)的相互作用的预测,且预测效率高。并且,能够将位置特异性得分矩阵PSSM转换成固定长度的特征向量,为氨基酸序列特征提供了一种多维空间表达方式。本公开的其他特征和优点将在随后的具体实施方式部分予以详细说明。附图说明附图是用来提供对本公开的进一步理解,并且构成说明书的一部分,与下面的具体实施方式一起用于解释本公开,但并不构成对本公开的限制。在附图中:图1是根据一示例性实施例示出的一种氨基酸序列特征提取方法的流程图。图2是根据一示例性实施例示出的一种氨基酸序列特征提取装置的框图。图3是根据一示例性实施例示出的一种电子设备的框图。图4是根据一示例性实施例示出的一种电子设备的框图。具体实施方式以下结合附图对本公开的具体实施方式进行详细说明。应当理解的是,此处所描述的具体实施方式仅用于说明和解释本公开,并不用于限制本公开。图1是根据一示例性实施例示出的一种氨基酸序列特征提取方法的流程图。如图1所示,该方法可以包括以下步骤。在步骤101中,获取待进行特征提取的氨基酸序列。在本公开中,该方法可以用于氨基酸序列特征提取系统。当用户需要提取氨基酸序列特征时,可将待进行特征提取的氨基酸序列输入到氨基酸序列特征提取系统,该氨基酸序列特征提取系统接收该待进行特征提取的氨基酸序列。在步骤102中,确定氨基酸序列的位置特异性得分矩阵PSSM。在本公开中,可以采用位置特异迭代搜索工具(Position-SpecificIterativeBlastLocalAlignmentSearchTool,PSI-BLAST)进行多序列比对来建立上述待进行特征提取氨基酸序列的位置特异性得分矩阵(Position-SpecificScoringMatrix,PSSM)。并且,上述位置特异性得分矩阵PSSM的大小为N×M,其中,N表示上述氨基酸序列的序列长度,M表示预设氨基酸的总数;并且,该位置特异性得分矩阵PSSM中的每个分数PSSM(n,m)表示在第n行的氨基酸的位置下,第m列的预设氨基酸出现的倾向程度,且-S≤PSSM(n,m)≤S,其中,N、M和S均为正整数,且1≤n≤N,1≤m≤M。示例地,如表1所示,该位置特异性得分矩阵PSSM为:表1如表1中所示,纵列{A,R,N,D,C,Q,E,…,V}为M种预设氨基酸,横行M,K,I,S,F,H…为待进行特征提取的氨基酸序列,每个分数PSSM(n,m)表示在第n行的氨基酸的位置下,第m列的预设氨基酸出现的倾向程度。上述位置特异性得分矩阵PSSM的矩阵形式为:此外,上述采用PSI-BLAST算法来建立氨基酸序列的位置特异性得分矩阵PSSM时所采用的氨基酸数据库可以例如是NCBI(NationalCenterforBiotechnologyInformation)提供的非冗余氨基酸序列数据库,其中,该数据库包含95,563,598条完全解析的氨基酸序列信息。并且,PSI-BLAST算法的迭代次数可以例如是2次,默认域值E-value可以例如是0.05,上述M种预设氨基酸可以为20种天然氨基酸,即,甘氨酸、丙氨酸、缬氨酸、亮氨酸、异亮氨酸、苯丙氨酸、脯氨酸、色氨酸、丝氨酸、酪氨酸、半胱氨酸、蛋氨酸、天冬酰胺、谷氨酰胺、苏氨酸、天冬氨酸、谷氨酸、赖氨酸、精氨酸和组氨酸。另外,需要说明的是,上述采用PSI-BLAST算法来建立氨基酸序列的位置特异性得分矩阵PSSM的方式,是本领域技术人员公知的,在本公开中不再赘述。在步骤103中,确定每种预设氨基酸在[-S,S]分数区间内的得分分布。在本公开中,可以通过多种方式来确定每种预设氨基酸在[-S,S]分数区间内的得分分布。在一种实施方式中,可以针对每种预设氨基酸,根据该预设氨基酸的全部分数,统计在[-S,S]分数区间内的每个整数分数的出现频率,并按照各整数分数由小及大的顺序,将各整数分数的出现频率组合成1×(2S+1)的特征向量,所得特征向量作为该预设氨基酸在[-S,S]分数区间内的得分分布。下面以S=13、第m列预设氨基酸为例来具体说明如何确定每种预设氨基酸在[-S,S]分数区间内的得分分布:针对第m列预设氨基酸,根据上述步骤102中确定出的位置特异性得分矩阵PSSM中第m列中的各分数,统计在[-13,13]分数区间内的每个整数分数的出现频率,其中,每个分数的出现频率等于该分数的出现次数除以表示所述氨基酸序列的序列长度N,示例地,统计结果如表2中所示(其中,score表示每个分数值,count表示每个分数的出现次数,freq表示每个分数的出现频率):表2score-13-12-11-10-9…910111213count31582…51302freq0.030.010.050.080.02…0.050.010.0300.02如表2中所示,上述步骤102中确定出的位置特异性得分矩阵PSSM中第m列的各分数中,整数分数-13的出现频率为0.03,整数分数-12的出现频率为0.01,整数分数-11的出现频率为0.05,整数分数-10的出现频率为0.08,整数分数-9的出现频率为0.02,…,整数分数9的出现频率为0.05,整数10的出现频率为0.01,整数11的出现频率为0.03,整数分数12的出现频率为0,整数分数13的出现频率为0.02。在统计出在[-13,13]分数区间内的每个整数分数的出现频率后,可以按照各整数分数由小及大的顺序,将各整数分数的出现频率组合成1×27的特征向量,即,[0.030.010.050.080.02…0.050.010.0300.02],并将该特征向量[0.030.010.050.080.02…0.050.010.0300.02]作为该第m列的预设氨基酸在[-13,13]分数区间内的得分分布。在另一种实施方式中,可以通过以下等式(1)来计算每种预设氨基酸在[-S,S]分数区间内的得分分布:其中,f(m)为所述每种预设氨基酸在[-S,S]分数区间内的得分分布,且f(m)为1×(2S+1)的特征向量;fj(m)为f(m)中的每个元素值,且j=0,…,2。在步骤104中,合并M种预设氨基酸的得分分布,以获得待进行特征提取的氨基酸序列的特征向量。在确定出M种预设氨基酸在[-S,S]分数区间内的得分分布后,可以将它们合并,即获得了上述待进行特征提取的氨基酸序列的特征向量。示例地,预设氨基酸的总数M=20,分数区间为[-13,13],在上述步骤103中确定出20种预设氨基酸在[-13,13]分数区间内的得分分布(1×27的特征向量)后,可以将该20种预设氨基酸在[-13,13]分数区间内的得分分布(1×27的特征向量)合并,即将上述步骤103中确定出的20个1×27的特征向量合并,从而得到20×27的固定维度的特征向量,即获得了上述待进行特征提取的氨基酸序列的特征向量。在上述技术方案中,由于确定出的氨基酸序列的位置特异性得分矩阵PSSM中蕴含有同源蛋白质信息,因此该蕴含有同源蛋白质信息的位置特异性得分矩阵PSSM表示成的特征向量中能够融入更多的序列保守信息,更有利于氨基酸序列与其他生物高分子物质之间(例如,氨基酸序列之间、氨基酸序列与DAN/RNA之间、氨基酸序列与糖类及脂质类之间)的相互作用的预测,且预测效率高。并且,能够将位置特异性得分矩阵PSSM转换成固定长度的特征向量,为氨基酸序列特征提供了一种多维空间表达方式。图2是根据一示例性实施例示出的一种氨基酸序列特征提取装置的框图。参照图2,该装置200可以包括:获取模块201,用于获取待进行特征提取的氨基酸序列;第一确定模块202,用于确定所述获取模块201获取到的所述氨基酸序列的位置特异性得分矩阵PSSM,其中,所述位置特异性得分矩阵PSSM的大小为N×M,N表示所述氨基酸序列的序列长度,M表示预设氨基酸的总数,所述位置特异性得分矩阵PSSM中的每个分数PSSM(n,m)表示在第n行的氨基酸的位置下,第m列的预设氨基酸出现的倾向程度,且-S≤PSSM(n,m)≤S,其中,N、M和S均为正整数,且1≤n≤N,1≤m≤M;第二确定模块203,用于确定每种预设氨基酸在[-S,S]分数区间内的得分分布;合并模块204,用于合并M种预设氨基酸的所述得分分布,以获得所述氨基酸序列的特征向量。可选地,所述第二确定模块203可以用于:针对每种预设氨基酸,根据该预设氨基酸的全部分数,统计在[-S,S]分数区间内的每个整数分数的出现频率,并按照所述整数分数由小及大的顺序,将所述整数分数的出现频率组合成1×(2S+1)的特征向量,所得特征向量作为该预设氨基酸在[-S,S]分数区间内的得分分布。可选地,所述第二确定模块203可以用于:通过以上等式(1)来确定每种预设氨基酸在[-S,S]分数区间内的得分分布为f(m)中的每个元素值fj(m)。可选地,所述M种预设氨基酸为20种天然氨基酸。关于上述实施例中的装置,其中各个模块执行操作的具体方式已经在有关该方法的实施例中进行了详细描述,此处将不做详细阐述说明。图3是根据一示例性实施例示出的一种电子设备300的框图。如图3所示,该电子设备300可以包括:处理器301,存储器302,多媒体组件303,输入/输出(I/O)接口304,以及通信组件305。其中,处理器301用于控制该电子设备300的整体操作,以完成上述的氨基酸序列特征提取方法中的全部或部分步骤。存储器302用于存储各种类型的数据以支持在该电子设备300的操作,这些数据例如可以包括用于在该电子设备300上操作的任何应用程序或方法的指令,以及应用程序相关的数据,例如联系人数据、收发的消息、图片、音频、视频等等。该存储器302可以由任何类型的易失性或非易失性存储设备或者它们的组合实现,例如静态随机存取存储器(StaticRandomAccessMemory,简称SRAM),电可擦除可编程只读存储器(ElectricallyErasableProgrammableRead-OnlyMemory,简称EEPROM),可擦除可编程只读存储器(ErasableProgrammableRead-OnlyMemory,简称EPROM),可编程只读存储器(ProgrammableRead-OnlyMemory,简称PROM),只读存储器(Read-OnlyMemory,简称ROM),磁存储器,快闪存储器,磁盘或光盘。多媒体组件303可以包括屏幕和音频组件。其中屏幕例如可以是触摸屏,音频组件用于输出和/或输入音频信号。例如,音频组件可以包括一个麦克风,麦克风用于接收外部音频信号。所接收的音频信号可以被进一步存储在存储器302或通过通信组件305发送。音频组件还包括至少一个扬声器,用于输出音频信号。I/O接口304为处理器301和其他接口模块之间提供接口,上述其他接口模块可以是键盘,鼠标,按钮等。这些按钮可以是虚拟按钮或者实体按钮。通信组件305用于该电子设备300与其他设备之间进行有线或无线通信。无线通信,例如Wi-Fi,蓝牙,近场通信(NearFieldCommunication,简称NFC),2G、3G或4G,或它们中的一种或几种的组合,因此相应的该通信组件305可以包括:Wi-Fi模块,蓝牙模块,NFC模块。在一示例性实施例中,电子设备300可以被一个或多个应用专用集成电路(ApplicationSpecificIntegratedCircuit,简称ASIC)、数字信号处理器(DigitalSignalProcessor,简称DSP)、数字信号处理设备(DigitalSignalProcessingDevice,简称DSPD)、可编程逻辑器件(ProgrammableLogicDevice,简称PLD)、现场可编程门阵列(FieldProgrammableGateArray,简称FPGA)、控制器、微控制器、微处理器或其他电子元件实现,用于执行上述的氨基酸序列特征提取方法。在另一示例性实施例中,还提供了一种包括程序指令的计算机可读存储介质,例如包括程序指令的存储器302,上述程序指令可由电子设备300的处理器301执行以完成上述的氨基酸序列特征提取方法。图4是根据一示例性实施例示出的一种电子设备400的框图。例如,电子设备400可以被提供为一服务器。参照图4,电子设备400包括处理器422,其数量可以为一个或多个,以及存储器432,用于存储可由处理器422执行的计算机程序。存储器432中存储的计算机程序可以包括一个或一个以上的每一个对应于一组指令的模块。此外,处理器422可以被配置为执行该计算机程序,以执行上述的氨基酸序列特征提取方法。另外,电子设备400还可以包括电源组件426和通信组件450,该电源组件426可以被配置为执行电子设备400的电源管理,该通信组件450可以被配置为实现电子设备400的通信,例如,有线或无线通信。此外,该电子设备400还可以包括输入/输出(I/O)接口458。电子设备400可以操作基于存储在存储器432的操作系统,例如WindowsServerTM,MacOSXTM,UnixTM,LinuxTM等等。在另一示例性实施例中,还提供了一种包括程序指令的计算机可读存储介质,例如包括程序指令的存储器432,上述程序指令可由电子设备400的处理器422执行以完成上述的氨基酸序列特征提取方法。以上结合附图详细描述了本公开的优选实施方式,但是,本公开并不限于上述实施方式中的具体细节,在本公开的技术构思范围内,可以对本公开的技术方案进行多种简单变型,这些简单变型均属于本公开的保护范围。另外需要说明的是,在上述具体实施方式中所描述的各个具体技术特征,在不矛盾的情况下,可以通过任何合适的方式进行组合。为了避免不必要的重复,本公开对各种可能的组合方式不再另行说明。此外,本公开的各种不同的实施方式之间也可以进行任意组合,只要其不违背本公开的思想,其同样应当视为本公开所公开的内容。当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1