一种利用水稻基因组学技术快速精准选育三系水稻恢复系的方法与流程
本发明涉及的是水稻育种技术领域,特别是涉及一种利用水稻基因组学技术快速精准选育三系水稻恢复系的方法。
背景技术:
随着人们生活水平的不断提高,产量高、外观和适口性好的优质大米的需求量越来越大。杂交水稻的优质化尤为重要,而选育高配合力优质恢复系,是提高杂交水稻米质的有效途径。为了提高水稻恢复系的整体水平,实现所配组合在优质、高产、稳产、抗倒、抗病、高配合力、广适性等方面的重大突破,在恢复系选育上,需进一步提高恢复力及其选配组合的优势。目前杂交水稻育种实践证明快速、精准选育高配合力、米质优、多抗、适用于机械化生产需要的恢复系和不育系,是育成强优组合的关键。然而多年来,中国三系水稻恢复系育种主要采用常规技术方法进行,虽然取得重大进展,但仍存在的主要问题有以下几点:1、恢复系育种年限较长;2、三系恢复系之间的遗传距离不大,与不育系之间的差异也比较小,这同时也成为选育超级杂交水稻的一个障碍;3、采用恢复系材料和不具有恢复基因的优良常规水稻材料杂交,选育新的优良恢复系时,因技术方法较为传统,新的优良恢复株系的选育时间长且测交群体大,难以在早代确定新的优良恢复系单系,造成已应用于生产的恢复系的综合品质难以利用目前优良的育种新材料,导致具有综合优异性状的恢复系缺乏,制约了三系杂交水稻的发展突破。
三系水稻恢复系要克服上述问题实现优质多抗发展目标,必须进行常规育种技术与现代高新技术相结合。现代生物技术的迅速发展特别是水稻全基因组测序的完成,使水稻恢复系育种能从基因水平上进行操作;为快速、精准选育聚合优异多抗性状的三系水稻恢复系奠定了坚实的基础。
技术实现要素:
本发明的目的在于克服现有技术的不足,提供了一种利用水稻基因组学技术快速精准选育三系水稻恢复系的方法,旨在解决现有选育三系恢复系的年限较长、综合优良性状聚合不够等问题。
本发明是通过以下技术方案实现的:
本发明提供了一种利用水稻基因组学技术快速精准选育三系水稻恢复系的方法,包括如下步骤:
(1)以待改良的水稻品种为母本,以含有目标性状的常规水稻品种为父本,进行常规有性杂交后,与父本回交一次,获得的BC1F1代再自交一次,获得BC1F2分离群体,其中,所述母本和/或父本为含有水稻育性恢复基因的恢复系;
(2)对BC1F2代分离群体进行田间单株选择,择优选择含有父母本双亲优良性状的单株进行混收,并种植BC1F2代混选株系群体种子,得到BC1F3群体;
(4)同时,利用二代全基因组测序技术对父本、母本及步骤(3)的自交单株进行全基因组测序和基因型分析,筛选含有父母本双亲优良基因且遗传背景更接近父本的单株收种;
(5)重复步骤(4),再自交2代以上,即育成稳定的恢复系。
进一步地,所述步骤(2)还包括对BC1F3群体进行田间单株选择,择优选择含有双亲优良性状的单株进入下一步分子标记筛选步骤。
进一步地,利用全基因组测序筛选后,还包括配合力筛选步骤,即对经全基因组测序筛选后的单株进行配合力测定,选择配合力好的单株。
进一步地,所述目标性状包括株叶形态好、早熟、优质、高产、抗病、抗倒伏及配合力高等一种或多种优良性状,但不限于此。
本发明相比现有技术具有以下优点:
1)有效选择育性恢复基因:在选育过程中,采用分子标记辅助选择,将含有和不含育性恢复基因的植株有效的区分开,而常规的方法是很难实现的。
2)有效扩大三系恢复系的遗传背景,利于创制新型三系恢复系新资源。
3)快速精准:利用基于水稻全基因组测序的基因分型技术进行遗传背景选择,从而缩短了恢复系的改良世代,仅3-4年就能育成稳定的恢复系,大大加快了育种速度,减少育种的盲目性。
4)聚合优良性状:田间农艺性状观察结合全基因组测序,筛选聚合双亲优良性状的单株。
附图说明
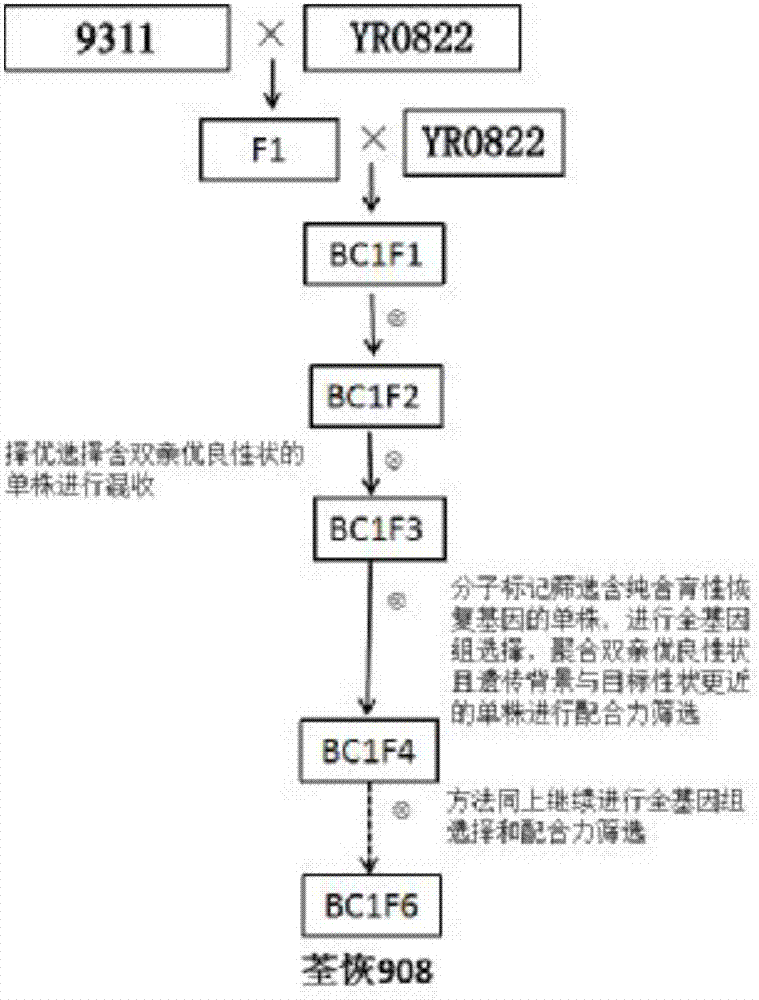
图1:实施例1的技术路线图;
图2:实施例2的技术路线图。
具体实施方式
下面通过两组具体实施例对本发明的技术方案进行详细说明,其中涉及的方法如无特别说明均为本领域的技术人员所知晓的常规方法,所用的试剂等材料,如无特别说明,均为市售购买产品,所用的种质资源,均为市售常规品种,可直接从安徽荃银高科种业股份有限公司购买或从国家种质库直接获取。
实施例1
本实施例提供了一种利用水稻全基因组学技术,以株型较好、耐肥、抗倒、结实率高的“黄华占”为待改良亲本,进行快速精准选育具有更多优良性状的水稻恢复系新品种的方法,其技术路线图如图1所示,包括以下步骤:
(1)杂交
以“黄华占”为母本,以农艺性状优良、早熟、含抗稻瘟病Pi2基因、品质优良的恢复系“华占”为父本,进行常规有性杂交后,与父本回交一次,与父本回交一次,获得的BC1F1代再自交一次,获得1200株BC1F2分离群体。
(2)田间农艺性状筛选
对步骤(1)的BC1F2代分离群体进行田间农艺性状筛选,选择株叶形态较好的优良单株126个,在这126个单株中继续筛选,择优选择含有父母本双亲优良性状的单株107个,对这107个单株的种子进行混收,并种植BC1F2代混选株系群体,得到BC1F3群体。
对BC1F3群体继续进行田间农艺性状筛选,择优选择含有父母本双亲优良性状的单株176个。
(3)分子标记筛选和全基因组测序分析
以育性恢复基因RF3基因为分子标记基因,以RF3基因标记筛选引物RM10338为特异性扩增引物,对上述步骤(2)筛选获得的176个单株进行分子标记筛选,步骤包括:
1)DNA制备:对上述176个F3单株进行植株编号,利用CTAB法提取基因组DNA;
2)PCR反应:
PCR反应体系为20μl,含:2mM Mg2+的10×Buffer 2.0ul,2.5mM dNTPs 2.0ul,1U/μl Taq酶0.5ul,2μM正反引物各0.5ul,100ng左右基因组DNA 2ul,ddH2O 12.5ul;
RM10338引物序列为:
正向:GTGAAGTTTCCCTCGGAATCACG
反向:AGCTAGGGAGAAGAAGCGGAAGC
反应条件为94℃预变性3min后,按94℃变性30s,56℃退火30s,72℃延伸1min循环35次,72℃延伸5min;
扩增后的PCR产物利用8%的非变性聚丙烯酰胺凝胶电泳检测60min(250V),电泳结果在胶片观测灯上观察、拍照。
3)结果统计:记录PCR产物条带,剔除含纯合及杂合育性恢复基因的单株,筛选得到不含恢复基因的单株。结果:有45份含有纯合育性恢复基因,88份含杂合育性恢复基因及43份不含恢复基因的单株,筛选得到45份单株。
(4)全基因组测序
利用二代全基因组测序技术,对双亲“黄华占”、“华占”和上述筛选获得的45个单株的全基因组进行测序,步骤包括:
i)对“黄华占”、“华占”和上述筛选获得的45个单株进行编号,在苗期取对应的植株叶片,利用Qiaqen公司的N96新型植物基因组DNA提取试剂盒提取植株叶片基因组DNA;
ii)将基因组DNA打碎,用DNA聚合酶修复后连上接头,多个样品混合进行电泳,并回收300-400bp的片段,建立PE400测序文库;
iii)利用Illumina Hiseq X10测序平台,用2×150bp双末端测序法进行全基因组重测序,亲本测序深度约50×、后代单株测序深度约0.1×,约在一周左右时间内便可完成测序工作。
通过将子代单株和双亲进行全基因组序列比对和遗传变异分析鉴定,获得子代单株的精细基因型图谱,通过子代单株的精细基因型图谱,分析优异基因位点的基因型,选择优异基因杂合程度低、含双亲优异基因(包括抗稻瘟病Pi2基因)且遗传背景更接近“华占”的单株6个,将这6个单株进行套袋自交,获得BC1F4。
(5)连续自交
重复步骤(4),获得BC1F5,利用步骤(4)的全基因组测序方法筛选优异基因杂合程度低、含双亲优异基因(包括抗稻瘟病Pi2基因)且遗传背景更接近“华占”的单株,获得的单株同时与II-32A、荃9311A、荃香9A和中9A不育系进行配合力测定,筛选配合力高的单株继续套袋自交并收种,即得到稳定的优良恢复系,命名为荃丰占。
实施例2
本实施例提供了一种利用水稻全基因组学技术,以株型较好、耐肥、抗倒、结实率高的“9311”为待改良亲本,进行快速精准选育具有更多优良性状的水稻恢复系新品种的方法,其技术路线图如图1所示,包括以下步骤:
(1)杂交
以“9311”为母本,以艺性状优良、早熟、含抗稻瘟病Pi2基因、品质优良的恢复系“YR0822”为父本,进行常规有性杂交后,与父本回交一次,与父本回交一次,获得的BC1F1代再自交一次,获得1100株BC1F2分离群体。
(2)田间农艺性状筛选
对步骤(1)的BC1F2分离群体进行田间农艺性状筛选,选择株叶形态较好的优良单株119个,在这119个单株中继续筛选,择优含有父母本双亲优良性状的单株99个,对这99个单株的种子进行混收,并种植BC1F2代混选株系群体,得到BC1F3群体。
对BC1F3群体继续进行田间农艺性状筛选,择优选择株叶形态符合父本性状的单株185个。
(3)分子标记筛选和全基因组测序分析
通过国家水稻数据中心查询恢复系“YR0822”的育性恢复基因为RF3,以RF3基因为分子标记基因,以RF3基因标记筛选引物RM10338为特异性扩增引物,对上述步骤(2)筛选获得的185个单株进行分子标记筛选,步骤包括:
1)DNA制备:对上述185个F3单株进行植株编号,利用CTAB法提取基因组DNA;
2)PCR反应:
PCR反应体系为20μl,含:2mM Mg2+的10×Buffer 2.0ul,2.5mM dNTPs 2.0ul,1U/μl Taq酶0.5ul,2μM正反引物各0.5ul,100ng左右基因组DNA 2ul,ddH2O 12.5ul;
RM10338引物序列为:
正向:GTGAAGTTTCCCTCGGAATCACG
反向:AGCTAGGGAGAAGAAGCGGAAGC
反应条件为94℃预变性3min后,按94℃变性30s,56℃退火30s,72℃延伸1min循环35次,72℃延伸5min;
扩增后的PCR产物利用8%的非变性聚丙烯酰胺凝胶电泳检测60min(250V),电泳结果在胶片观测灯上观察、拍照。
3)结果统计:记录PCR产物条带,剔除含纯合及杂合育性恢复基因的单株,筛选得到不含恢复基因的单株。结果:有44份含有纯合育性恢复基因,93份含杂合育性恢复基因及48份不含恢复基因的单株,筛选得到44份单株。
(4)全基因组测序
利用二代全基因组测序技术,对双亲“9311”、“YR0822”和上述筛选获得的44个单株的全基因组进行测序,步骤同实施例1的步骤(3),在此不做重复描述。通过将子代单株和双亲进行全基因组序列比对和遗传变异分析鉴定,获得子代单株的精细基因型图谱,分析子代单株的精细基因型图谱中优良基因位点的基因型,选择含双亲优异基因(包括抗稻瘟病Pi2基因)、杂合程度低且遗传背景更接近“YR0822”的单株5个。
将筛选获得的5个单株同时与II-32A、荃9311A、荃香9A和中9A不育系进行配合力测定,筛选配合力高的单株继续套袋自交并收种。
(5)连续自交
重复步骤(4),再自交2次,即得到稳定的优良恢复系,命名为荃恢908。
以上为本发明一种详细的实施方式和具体的操作过程,是以本发明技术方案为前提下进行实施,但本发明的保护范围不限于上述的实施例。
- 还没有人留言评论。精彩留言会获得点赞!