与前列腺癌有关的基因组重排和使用所述基因组重排的方法与流程
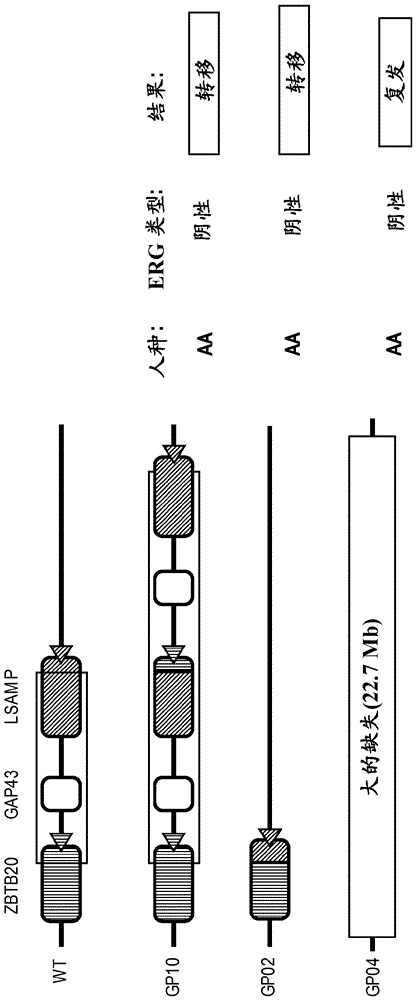
本申请要求2013年12月30日提交的美国临时专利申请号61/921,780的权益,并依赖于其提交日期,其整个内容通过引用结合到本申请中。政府利益本发明部分在政府支持下完成。政府在本发明中享有一定权益。序列表本申请含有电子提交的ASCII格式的序列表,并通过引用以其整体结合到本文中。2014年12月30日创建的所述ASCII副本被命名为HMJ-149-PCT_SL.txt,且大小为205,868字节。发明背景到2013年,估计238,590名男性将被诊断患有前列腺癌(CaP),并且估计29,720名男性将列于该疾病[1]。这种恶性肿瘤是美国男性癌症相关死亡的第二主要原因。另外,与其它人种相比,非裔美国(AA)男性具有因CaP所致的最高发病率和死亡率[1]。从表现和诊断至治疗、生存和生命质量来看都存在人种差异[2]。研究人员提出社会经济地位(SES)对包括CaP特异性死亡率在内的这些差异产生显著影响[3]。同样,有证据显示,获得护理的机会减少与CaP结局差有关,所述结局差在AA男性中比在高加索美国(CA)男性中更普遍[4]。然而,存在其中AA男性具有与CA男性类似的结局的群体。Sridhar和同事[5]发表了元分析,其中他们得出结论,当说明SES时,在AA和CA男性之间在总体生存和CaP特异性生存方面没有差异。类似地,军人和退伍军人群体(同样的访问和筛选系统)在种族间在生存方面未观察到差异[6],且到2000年代早期在退伍军人组群中在诊断时病理期的差异缩小[7]。值得注意的是,这两项研究表明,与CA男性相比,AA男性更可能具有较高的Gleason评分和PSA水平[6,7]。虽然社会经济因素可能对CaP结局产生影响,但是它们似乎无法说明与诊断和疾病风险有关的所有变量。若干研究支持与CA男性相比AA男性具有较高的CaP发病率[1,8,9]。研究还显示AA男性具有诊断时显著较高的PSA、活组织检查中较高等级的疾病、各阶段较大的肿瘤体积和根治性前列腺切除术前较短的PSA倍增时间[10-12]。在有关应激和炎性反应的肿瘤微环境中注意到CA和AA男性的前列腺癌之间的生物学差异[13]。虽然在生物学差异的作用方面仍有争议,但是所表现的发病率和疾病侵袭性中观察到的差异表明AA和CA男性之间前列腺癌病变的不同途径的潜在作用。在过去十年内,大量研究集中在癌基因的改变及其在CaP中的作用[14-16]。在CaP中过量表达并且是CaP中最普遍已知的癌基因的TMPRSS2/ERG基因融合物中注意到种族性和人种间流行率的变化[17,18]。累计数据表明在种族性间ERG致癌变化有差异[17,19-21]。在描述ERG过量表达和ERG剪接变体的最初的论文中注意到与AA男性相比CA男性中显著较大的ERG表达[17,21]。该差异在有高Gleason等级(8-10)肿瘤的患者中在CA和AA之间甚至更明显(50%与16%)。[Ferrell等,手稿]。因此,ERG是这些人种群(ethnicgroup)间的主要体细胞基因改变。但除TMPRSS2/ERG以外,对有关AA和CA男性间的CaP差异的遗传基础知之甚少,甚至未知[24]。因此,需要对不同种族人群有特异性并提供更准确的诊断和/或预后潜力的新的生物标志物和治疗标志物。发明概述本公开内容提供发生在染色体3q13区域并包括ZBTB20基因和LSAMP基因的基因组排列和根据在包含前列腺细胞的生物样品中ZBTB20/LSAMP基因组排列的检测诊断和预测前列腺癌的方法。ZBTB20/LSAMP基因组排列可以是ZBTB20基因和LSAMP基因间的基因融合、基因倒位、基因缺失或基因重复。在来自受试者的前列腺细胞中检出ZBTB20/LSAMP基因组重排表明受试者患有前列腺癌或发生前列腺癌的可能性加大或将受试者的前列腺癌表征为侵袭形式的前列腺癌或发展成侵袭形式的前列腺癌的风险增加。ZBTB20/LSAMP基因重排可在核酸或蛋白质水平上测量。在一个实施方案中,受试者的前列腺癌不表达ERG和雄激素调节基因(例如TMPRSS2)间的基因融合物。在另一个实施方案中,受试者具有非洲血统。这样,ZBTB20/LSAMP基因组重排可用于预测特定人种群内前列腺癌的严重程度,因为实例显示在前列腺细胞中具有ZBTB20/LSAMP基因组重排,但无TMPRSS2/ERG融合物的非洲血统受试者,始终发生侵袭形式的前列腺癌。鉴于ZBTB20/LSAMP基因组重排的预测价值,所述方法可进一步包括根据ZBTB20/LSAMP基因组重排的检出选择用于受试者的治疗方案或如果在获自受试者的生物样品中检出基因组重排则对受试者进行治疗的步骤。或者,所述方法可进一步包括增加针对发展前列腺癌或更具侵袭形式的前列腺癌而监测受试者的频率的步骤。另一方面涉及用于诊断或预测前列腺癌的组合物。在一个实施方案中,组合物包含多核苷酸探针,其中多核苷酸探针在高严格性条件下与嵌合核酸的连接处杂交,其中嵌合核酸包含来自ZBTB20基因的第一部分和来自LSAMP基因的第二部分(“ZBTB20/LSAMP多核苷酸探针”)。在一个实施方案中,第一部分包含ZBTB20基因的外显子1和第二部分包含LSAMP的外显子3*或外显子4。在其它实施方案中,多核苷酸探针在高严格性条件下与LSAMP基因的外显子3*或嵌合核酸的连接处杂交,其中嵌合核酸包含来自LSAMP基因的外显子3*的第一部分和来自LSAMP基因的外显子4的第二部分(“外显子3*/外显子4多核苷酸探针”)。在其它实施方案中,多核苷酸探针在高严格性条件下与LSAMP基因的外显子0*或嵌合核酸的连接处杂交,其中嵌合核酸包含来自LSAMP基因的外显子0*的第一部分和来自LSAMP基因的外显子1的第二部分(“外显子0*/外显子1多核苷酸探针”)。多核苷酸探针被任选标记。另一方面涉及包含具有ZBTB20/LSAMP多核苷酸探针、外显子3*/外显子4多核苷酸探针或外显子0*/外显子1多核苷酸探针的组合物和包含在高严格性条件下与选自COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1的基因杂交的多核苷酸探针的第二组合物的试剂盒。多核苷酸探针被任选标记。或者,试剂盒包含具有ZBTB20/LSAMP多核苷酸探针、外显子3*/外显子4多核苷酸探针或外显子0*/外显子1多核苷酸探针的组合物和包含在高严格性条件下与选自ERG、AMACR、PCA3和PSA的基因杂交的多核苷酸探针的第二组合物。多核苷酸探针被任选标记。又一方面涉及包含双链寡核苷酸双链体的组合物,其中寡核苷酸双链体包含与第二核酸杂交的第一核酸,其中第一核酸包含与来自LSAMP基因的第二部分融合的来自ZBTB20基因的第一部分,且其中第二核酸是与来自ZBTB20基因的第一部分和来自LSAMP基因的第二部分之间的连接处杂交的多核苷酸探针。在一个实施方案中,第一部分包含ZBTB20基因的外显子1,第二部分包含LSAMP的外显子3*或外显子4。在另一个实施方案中,第一核酸包含与来自LSAMP基因的外显子4的第二部分融合的来自LSAMP基因的外显子3*的第一部分,且其中第二核酸是与来自LSAMP基因的外显子3*的第一部分和来自LSAMP基因的外显子4的第二部分之间的连接处杂交的多核苷酸探针。在另一个实施方案中,第一核酸包含与来自LSAMP基因的外显子1的第二部分融合的来自LSAMP基因的外显子0*的第一部分,且其中第二核酸是与来自LSAMP基因的外显子0*的第一部分和来自LSAMP基因的外显子4的第二部分之间的连接处杂交的多核苷酸探针。多核苷酸探针被任选标记。另一方面涉及与由基因融合物编码的多肽结合的分离的抗体,其中基因融合物具有来自ZBTB20基因的第一部分和来自LSAMP基因的第二部分。在一个实施方案中,多肽是截短的LSAMP多肽。在另一个实施方案中,抗体与不存在于野生型ZBTB20蛋白或野生型LSAMP蛋白中的基因融合物编码的多肽中存在的表位结合。抗体任选被标记。另一方面涉及用于扩增基因融合物的组合物,其中基因融合物具有来自ZBTB20基因的第一部分和来自LSAMP基因的第二部分。在一个实施方案中,第一部分包含ZBTB20基因的外显子1,第二部分包含LSAMP的外显子3*或外显子4。在一个实施方案中,组合物包含第一和第二引物,其中第一和第二引物能够扩增来自跨越来自ZBTB20基因的基因融合物的第一部分和来自LSAMP基因的基因融合物的第二部分之间的连接处的基因融合物的核苷酸序列。在一个实施方案中,第一引物与来自ZBTB20基因的基因融合物的第一部分杂交,第二引物与来自LSAMP基因的基因融合物的第二部分杂交。LSAMP基因座的外显子3*是因ZBTB20和LSAMP基因的基因组重排而产生的最近鉴定的LSAMP外显子。因此,另一方面涉及用于扩增LSAMP基因或基因融合物的外显子3*的组合物,其中基因融合物具有来自LSAMP基因的外显子3*的第一部分和来自LSAMP基因的外显子4的第二部分。在一个实施方案中,组合物包含第一和第二引物,其中第一和第二引物能够扩增是外显子3*特有的且不存在于其它基因序列中的LSAMP的外显子3*内的核苷酸序列,或来自跨越来自LSAMP基因的外显子3*的第一部分和来自LSAMP基因的外显子4的第二部分之间的连接处的基因融合物的核苷酸序列。LSAMP基因座的外显子0*是因ZBTB20和LSAMP基因的基因组重排而产生的最近鉴定的LSAMP外显子。因此,另一方面涉及用于扩增LSAMP基因或基因融合物的外显子0*的组合物,其中基因融合物具有来自LSAMP基因的外显子0*的第一部分和来自LSAMP基因的外显子1的第二部分。在一个实施方案中,组合物包含第一和第二引物,其中第一和第二引物能够扩增是0*外显子特有的且不存在于其它基因序列中的LSAMP的外显子0*内的核苷酸序列,或来自跨越来自LSAMP基因的外显子0*的第一部分和来自LSAMP基因的外显子1的第二部分之间的连接处的基因融合物的核苷酸序列。与前列腺癌有关的另一个目标基因组重排是PTEN缺失。虽然PTEN基因是常见的肿瘤抑制物,且已知其缺失与癌症有关,但是出人意料地发现,PTEN缺失以显著不同的频率出现在不同的人种群中,并且在非洲血统的受试者中明显不存在。了解不同患者群间的癌症相关基因组重排(例如PTEN缺失)的分层提供指导前列腺癌患者的治疗选择的重要信息。因此,一方面涉及为非洲血统患者选择定向前列腺癌治疗的方法,其中所述方法包括(a)排除靶向PI3K/PTEN/Akt/mTOR途径的前列腺癌疗法作为治疗选择;并选择合适的前列腺癌治疗。在一个实施方案中,该方法进一步包括测试患者的生物样品的步骤,其中生物样品包含前列腺细胞以证实前列腺细胞不含PTEN基因缺失。附图简述并入且构成本说明书的一部分的附图阐明某些实施方案,并且与书面描述一起,用来解释本文公开的抗体和方法的某些原理。图1是野生型染色体3q13区域的图谱,显示ZBTB20、GAP43和LSAMP基因,以及在3名不同AA患者中鉴定的染色体3q13区域的基因组重排,全部患者均发生侵袭形式的前列腺癌。图2显示患者GP10中染色体3q13区域的基因组重排,并提供产生于基因组重排的ZBTB20的外显子1和LSAMP的外显子4之间的连接处的cDNA序列。图2按出现顺序分别公开了SEQIDNOS49-50。图3显示本发明一些实施方案的系统的示意图。具体地说,该图说明按照所公开的系统和方法可用于计算机系统106执行的各种硬件、软件和其它资源。在所示实施方案中,计算机系统106可包括与在操作系统控制下或与操作系统联合操作的随机存储器连接的一个或多个处理器110。实施方案中的处理器110可包括在一个或多个服务器、群集器或其它计算机或硬件资源中,或可采用基于云的资源执行。操作系统可为例如LinuxTM操作系统、UnixTM操作系统或其它开放源码或专有操作系统或平台的分配。处理器110可与数据存储112(例如保存在硬盘驱动器或驱动器阵列上的数据库)通信以访问或保存程序指令或其它数据。处理器110可进一步通过网络接口108通信,网络接口108进而可通过一个或多个网络104(例如互连网或其它公共或专用网络)通信,使得可从客户机102或其它装置或服务程序中接受查询或其它要求。另外,处理器110可利用网络接口108通过一个或多个网络104向用户发送信息、指令、工作流查询、部分工作流或其它数据。网络接口104可包括一个或多个服务器或与一个或多个服务器通信连接。客户机102可为例如与互连网连接的个人计算机。一般而言,可对处理器110进行编程或配制以执行实施本文公开的方法的控制逻辑和控制操作。处理器110可与协处理器114进一步通信连接(即通过通信信道连接)。协处理器114可为经配制以实施本文公开的方法的专用硬件和/或固件部件。因此,可通过处理器110和/或协处理器114实施本文公开的方法。计算机系统106的其它配置、相关网络连接和其它硬件、软件和服务程序资源是可能的。图4A显示2个PTENFISH探针和2个染色体10着丝粒探针的杂交,表明ADCaP染色体10上存在2个(二倍体)野生型PTEN等位基因。通过丢失CDCaP核中的1拷贝PTEN(缺少1个PTEN特异性FISH信号)表明杂合PTEN缺失。缺失可发生在母本或父本染色体的任一个或两个上。图4B显示通过丢失ADCaP核内的1拷贝信号表明ZBTB20-LSAMP区域之间的杂合缺失。着丝粒探针检出2拷贝的染色体3。图5显示10种不同的ZBTB20-LSAMP融合转录物的外显子(E)结构(以5’-3’方向)和可变剪接的LSAMP(LPCS1)转录物的外显子结构。E1、E1A、E1B和E1C表示ZBTB20的外显子1的4种变体。E0、E1、E2、E3、E3*、E4、E5、E6和E7表示LSAMP的外显子。括号中的数字表示各个外显子的核苷酸数目。星号表示之前未被诠释的外显子变体。发明详述下面将详细参考不同的示例性实施方案,在附图中说明了其实例。要了解下面的详细描述为了使读者更全面地理解本发明方面的某些实施方案、特征和详细资料而提供,不应解释为限制本发明的范围。定义为了更容易地理解本发明,首先定义了某些术语。在整个详细描述中给出了其它定义。术语“非洲血统的”是指自认为具有非洲血统的个人,包括自认为是非洲裔美国人的个人和已确定具有与非洲祖先相关的遗传标记(亦称祖先信息标记(AIM))的个人,所述祖先信息标记例如JudithKidd等,Analysesofasetof128ancestryinformativesingle-nucleotidepolymorphismsinaglobalsetof119populationsamples(整套119群样品中一套128种提供祖先信息的单核苷酸多态性的分析),InvestigativeGenetics,(2):1,2011(所述参考资料通过引用以其整体予以结合)中鉴定的AIM。术语“高加索血统的”是指自认为具有高加索血统的个人,包括自认为是高加索美国人的个人和已确定具有与高加索(例如欧洲、北非或亚洲(西亚、中亚或南亚)祖先相关的遗传标记(亦称祖先信息标记(AIM))的个人,祖先信息标记例如JudithKidd等,Analysesofasetof128ancestryinformativesingle-nucleotidepolymorphismsinaglobalsetof119populationsamples(整套119群样品中一套128种提供祖先信息的单核苷酸多态性的分析),InvestigativeGenetics,(2):1,2011(所述参考资料通过引用以其整体予以结合)中鉴定的AIM。术语“抗体”是指免疫球蛋白或其抗原结合片段,并包括包含抗原结合片段或抗原结合结构域的任何多肽。该术语包括但不限于多克隆、单克隆、单特异性、多特异性、人源化、人、单链、嵌合、合成、重组、杂合、突变、移植和体外产生的抗体。除非之前有措词“完整”,否则术语“抗体”包括抗体片段例如Fab、F(ab')2、Fv、scFv、Fd、dAb和保留抗原结合功能的其它抗体片段。除非另有说明,否则抗体不必来自任何特殊来源,也不通过任何特殊方法产生。术语“抗原结合结构域”和“抗原结合片段”是指包含负责抗体和抗原之间特异性结合的氨基酸的抗体分子的一部分。对于某些抗原,抗原结合结构域或抗原结合片段可只与抗原的一部分结合。被抗体特异性识别和结合的抗原的一部分称为“表位”或“抗原决定簇”。抗原结合结构域和抗原结合片段包括Fab(Fragmentantigen-binding);F(ab')2片段,一种具有被铰链区处的二硫桥连接的2个Fab片段的二价片段;Fv片段;单链Fv片段(scFv),参见例如Bird等(1988)Science242:423-426和Huston等(1988)Proc.Natl.Acad.Sci.USA85:5879-5883);具有2个VH和CH1结构域的Fd片段;dAb(Ward等(1989)Nature341:544-546)和保留抗原结合功能的其它抗体片段。Fab片段具有通过恒定区间的二硫键共价连接的VH-CH1和VL-CL结构域。Fv片段较小,并具有非共价连接的VH和VL结构域。为了克服非共价连接的结构域解离的趋势,可构建scFv。scFv含有将(1)VH的C端与VL的N端,或(2)VL的C端与VH的N端连接的柔性多肽。15聚体(Gly4Ser)3肽(SEQIDNO:48)可用作接头,但其它接头是本领域已知的。采用本领域技术人员已知常规技术,获得这些抗体片段,并以与完整抗体相同的方式评价片段的功能。术语“检测”意指本领域已知的用于测定核酸或蛋白质的存在或量的各种方法的任一种。如本说明书中所用,术语“检测”包括定性或定量检测。术语“治疗有效量”是指足以治疗指定疾病或病况的剂量或量。术语“基因表达谱”是指样品中多个基因的表达水平。如本领域所理解的一样,可通过测量核酸(例如基因组DNA或mRNA)或由核酸编码的多肽的表达,分析基因的表达水平。术语“分离的”当在多肽或核酸的情况下使用时是指其天然环境基本上没有,因此可与可能天然存在的多肽或核酸区分开来的多肽或核酸。例如,分离的多肽或核酸基本上不含细胞物质或其来源于其中的细胞或组织源的其它多肽或核酸。术语“多肽”、“肽”和“蛋白质”在本文可互换使用,是指氨基酸的聚合物。术语“多肽探针”本文所用是指可用于蛋白质检测测定法(例如质谱法)以定量测定生物样品中的目标多肽的标记的(例如同位素标记的)多肽。术语“引物”意指能够与靶核酸的区域或其互补序列结合,并促进靶核酸的核酸扩增的多核苷酸。一般来讲,引物可具有可通过核酸聚合酶延伸的游离3’端。引物一般还包括能够通过直接与靶核酸的至少1条链或与该靶序列互补的链进行互补碱基相互使用而杂交的碱基序列。引物可包含靶标特异性序列和任选不与靶序列互补的其它序列。这些非互补序列可包含例如启动子序列或限制性内切核酸酶识别位点。“变异”或“变体”是指在小至单一碱基或较长间隔上不同于参比的等位基因序列。术语“ZBTB20和LSAMP基因的基因组重排”等是指与前列腺癌有关的ZBTB20和LSAMP基因的任何重排,可包括ZBTB20基因和LSAMP基因间的基因融合、包括ZBTB20基因和LSAMP基因的基因倒位、包括ZBTB20和LSAMP基因(或一个或两个基因的一部分)的基因缺失或包括ZBTB20和LSAMP基因的基因重复。术语“ERG”或“ERG基因”是指Ets相关基因(ERG),其被指定唯一的HugoGeneNomenclatureCommittee(HGNC)标识码:HGNC:3446,并包括在前列腺癌中是普遍的ERG基因融合产物,包括TMPRSS2-ERG融合产物。分析ERG或ERG基因的表达包括分析与前列腺癌有关的ERG基因融合产物(例如TMPRSS2-ERG)的表达。本文所用术语“侵袭形式的前列腺癌”是指主要Gleason评分为4或5的前列腺癌(亦称“分化差的”前列腺癌或已转移或在前列腺切除术后复发的前列腺癌)。本文所用术语“Gleason6-7”是指Gleason3+3和3+4级。在本领域中它还指主要模式3(primarypattern3)或主要Gleason模式3。ZBTB20/LSAMP基因组重排采用下一代测序技术以鉴定CaP的新的生物标志物和治疗靶标。对7名高加索血统患者和7名非洲血统患者(包括各患者的匹配对照的共28个样品)的组群间的获自组织学上确定的和精确解剖的原发CaP样本(80-95%肿瘤,主要Gleason模式3)的高质量基因组序列数据和覆盖度进行了比较,以评价所观察到的2个人种群之间CaP发病率和死亡率的差异。这些数据和分析提供来自针对临床病理特征匹配的CaP非洲血统患者(“AD”)和高加索血统(“CD”)的前列腺癌基因组的首次评价。这些前列腺癌样品中的全基因组序列分析鉴定出ZBTB20(含有锌指和BTB的20)和LSAMP(边缘系统相关性膜蛋白)基因之间的新的基因组重排。来自非洲血统受试者的7个样品的4个对TMPRSS2/ERG融合(至今在前列腺癌中鉴定的最普遍的基因融合)为阴性。在来自非洲血统受试者的4个TMPRSS2/ERG阴性样品的3个中,ZBTB20-LSAMP区域重排或缺失。ZBTB20/LSAMP阳性、TMPRSS2/ERG阴性受试者的所有3个发生侵袭形式的前列腺癌,其中2个经历转移,1个发生复发,表明ZBTB20/LSAMP基因组重排是更多攻击性前列腺癌表型、特别是在不表达TMPRSS2/ERG融合的患者中的指示物。由于与CD的患者相比,AD的患者以较低频率表达TMPRSS2/ERG融合,因此ZBTB20和LSAMP基因的基因组重排可表示用于检测AD患者中更多攻击性前列腺癌表型的特别有用的生物标志物。由HGNC指定的LSAMP基因的唯一标识码是HGNC:6705。LSAMP的EntrezGene代码是4045。LSAMP的核苷酸和氨基酸序列是已知的,并由NCBI参考序列NM_002338.3,GI:257467557(SEQIDNO:9和SEQIDNO:10)表示,所述序列通过引用以其整体予以结合。LSAMP基因的染色体位置是3q13.2-q21。LSAMP基因编码存在于边缘系统的皮质区和皮质下区的神经元表面糖蛋白。LSAMP被报道为肿瘤抑制物基因(Baroy等,2014,MolCancer28;13:93)。例如,Kuhn等报告了在核心结合因子急性髓系白血病的亚组中含有LSAMP基因的染色体3q13.31区域的常发生的缺失[29]。在骨肉瘤中,染色体3q13.31区域被鉴定为最多变化的基因组区,其最多的变化呈缺失的形式,在某些情况下,包括含有LSAMP基因的区域的缺失[30]。在肾透明细胞癌中鉴定出具有包括染色体区1q32.1的NORE1基因和染色体3q13.3区域的LSAMP基因的断裂点的染色体易位(t1;3)[31]。还鉴定出上皮卵巢癌中的染色体易位[32]。虽然已表明LSAMP的单一核苷酸变异是前列腺癌特异性死亡率的一个重要预测因子[33],但是从未报告过前列腺癌中LSAMP的基因组重排,并且从未在任何癌症类型中描述为与ZBTB20的融合。由HGNC指定的ZBTB20基因的唯一标识码是HGNC:13503。ZBTB20的EntrezGene代码为26137。ZBTB20是DNA结合蛋白,且被认为是转录因子。有至少7种备选转录物变体。有可在ZBTB20基因座内的至少4个不同位点启动转录的至少4个不同的启动子,产生ZBTB20的外显子1的4种变体:E1、E1A、E1B和E1C。ZBTB20变体1的代表性核苷酸和氨基酸序列是已知的,并由NCBI参考序列NM_001164342.1,GI:257900532(SEQIDNO:11和SEQIDNO:12)表示,所述序列通过引用以其整体予以结合。与变体1相比,变体2在5’非翻译区中不同于变体1,缺乏5’编码区的一部分,并在下游起始密码子处启动翻译。与同种型1相比,所编码的同种型(2)具有较短的N端。变体2-7编码相同的同种型(2)。ZBTB20变体2的代表性核苷酸和氨基酸序列是已知的,并由NCBI参考序列NM_015642.4,GI:257900536(SEQIDNO:13和SEQIDNO:14)表示,所述序列通过引用以其整体予以结合。ZBTB20基因的染色体位置为3q13.2。某些实施方案涉及收集数据用于诊断或预测CaP的方法,所述方法包括检测包含前列腺细胞的生物样品(或从前列腺细胞分离的核酸或多肽)中ZBTB20和LSAMP基因的基因组重排。所述方法可任选包括利用所收集的基因表达数据诊断或预测CaP的额外步骤。在一个实施方案中,检出ZBTB20和LSAMP基因的基因组重排表明生物样品中存在CaP或发生CaP的可能性增加。在另一个实施方案中,检出ZBTB20和LSAMP基因的基因组重排表明生物样品中攻击形式的CaP的存在或发生攻击形式的CaP的可能性加大。在一个实施方案中,基因组重排包含ZBTB20基因和LSAMP基因间的基因融合,例如ZBTB20基因的外显子1(例如E1、E1A、E1B或E1C)和LSAMP基因的外显子4之间的融合。在另一个实施方案中,基因组重排包含包括ZBTB20基因和LSAMP基因的基因倒位。在另一个实施方案中,基因组重排包含染色体3q13区域的缺失,其中缺失跨越两个ZBTB20和LSAMP基因(或一个或两个基因的一部分)。在另外又一个实施方案中,基因组重排包含包括ZBTB20和LSAMP基因的基因重复。收集数据或诊断和/或预测CaP的方法可进一步包括检测与前列腺癌有关的其它基因的表达,包括但不限于COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1。表1提供在非洲血统患者中较频繁过量表达的这些基因的由HGNC和EntrezGene指定的唯一标识码和代表性序列的登记号,所述序列通过引用以其整体结合到本文中。表1在另一个实施方案中,收集数据或诊断和/或预测CaP的方法可进一步包括检测与前列腺癌有关的其它基因的表达,包括但不限于ERG、PSA和PCA3。PTEN缺失PTEN(磷酸酶和张力蛋白同源物)是一种在多种癌症中以高频率突变的肿瘤抑制物基因。由该基因编码的蛋白质是一种磷脂酰肌醇-3,4,5-三磷酸3-磷酸酶。它含有张力蛋白样结构域以及类似于二重特异性蛋白质酪氨酸磷酸酶的催化结构域。与大部分蛋白质酪氨酸磷酸酶不一样,PTEN优先使磷酸肌醇基底物脱去磷酸。它负调节细胞中的磷脂酰肌醇-3,4,5-三磷酸的胞内水平,并通过负调节AKT/PKB信号转导途径起肿瘤抑制物的作用。通过生长因子与其受体的结合所致生长因子受体激活或通过生长因子受体的突变导致PI3K/PTEN/Akt/mTOR级联的激活,这特别导致某些转录因子的激活[28],所述参考文献通过引用以其整体结合到本文中。PTEN通常起下调该途径的作用。因此,在含有PTEN基因缺失的癌症中,Akt基因的表达和mTOR的激活常常增加。PTEN基因的由HGNC和EntrezGene指定的唯一标识码分别为HGNC:9588和EntrezGene:5728。代表性PTEN核酸和多肽序列的登记号为NM_000314.4,GI:257467557(SEQIDNO:15和SEQIDNO:16),所述序列通过引用以其整体予以结合。PTEN基因的染色体位置为10q23。CD和AD前列腺癌样品的全基因组序列分析公开了不同人种群中PTEN基因座的基因组重排之间的显著差异。更准确地说,只在高加索血统患者中检出PTEN缺失。组织微阵列的其它FISH分析证实与CD男性相比,PTEN缺失是AD男性前列腺癌的发生中的稀有事件。因此,一方面涉及利用有关人种群间PTEN缺失差异的发现,以做出有关患有前列腺癌的受试者可得到的治疗选择的知情决定。具体地说,鉴于所公开的来自高加索美国和非洲血统患者前列腺癌中PTEN缺失的差异,对于非洲血统患者一般来说不应选择靶向PI3K/PTEN/Akt/mTOR途径的前列腺癌疗法[28]。或至少对于非洲血统患者,不应考虑靶向PI3K/PTEN/Akt/mTOR途径的前列腺癌疗法[28],除非首先通过基因测试证实患者的前列腺细胞含有PTEN缺失。因此,一个实施方案涉及为非洲血统患者选择定向前列腺癌治疗的方法,其中所述方法包括排除靶向PI3K/PTEN/Akt/mTOR途径的前列腺癌疗法[28]作为治疗选择;并选择合适的前列腺癌治疗。在一个实施方案中,该方法进一步包括测试患者的生物样品的步骤,其中生物样品包含前列腺细胞以证实前列腺细胞不含PTEN基因缺失。有多种靶向PI3K/PTEN/Akt/mTOR途径的抑制剂,包括PI3K抑制剂、Akt抑制剂、mTOR抑制剂和双重PI3K/mTOR抑制剂。PI3K抑制剂包括但不限于LY-294002、渥曼青霉素、PX-866、GDC-0941、CAL-10、XL-147、XL-756、IC87114、NVP-BKM120和NVP-BYL719。Akt抑制剂包括但不限于A-443654、GSK690693、VQD-002(a.k.a.API-2、曲西立滨)、KP372-1、KRX-0401(哌立福辛)、MK-2206、GSK2141795、LY317615(恩扎啕林)、瓢儿菜基磷酸胆碱(ErPC)、瓢儿菜基磷酸高胆碱(erucylphosphohomocholine,ErPC3)、PBI-05204、RX-0201和XL-418。mTOR抑制剂包括但不限于雷帕霉素、改性雷帕霉素类(rapalogs,例如CCI-779、afinitor、torisel、坦罗莫司)、AP-23573(ridaforolimus)和RAD001(afinitor、依维莫司)、二甲双胍、OSI-027、PP-242、AZD8055、AZD2014、palomid529、WAY600、WYE353、WYE687、WYE132、Ku0063794和OXA-01。双重PI3K/mTOR抑制剂包括但不限于PI-103、NVP-BEZ235、PKI-587、PKI-402、PF-04691502、XL765、GNE-477、GSK2126458和WJD008。检测ZBTB20/LSAMP或PTEN缺失在本文所述方法中测量或检测ZBTB20和LSAMP基因的基因组重排的表达包括测量或检测其任何核酸转录物(例如mRNA、cDNA或基因组DNA)或由所述核酸转录物编码的任何蛋白质。因此,在一个实施方案中,检测生物样品中ZBTB20/LSAMP基因组重排的存在包括检测具有来自ZBTB20基因的第一部分和来自LSAMP基因的第二部分的基因组DNA的染色体重排。在一个实施方案中,染色体重排产生ZBTB20基因的外显子1(例如E1、E1A、E1B或E1C)和LSAMP基因的外显子3*或外显子4之间的融合。在一个实施方案中,染色体重排包含SEQIDNO:1的核苷酸序列。在另一个实施方案中,染色体重排导致ZBTB20和LSAMP基因的缺失。因此,在一个实施方案中,检测生物样品中ZBTB20/LSAMP基因组重排的存在包括检测染色体3q13区域的缺失,其中缺失跨越ZBTB20和LSAMP基因。在另一个实施方案中,检测生物样品中ZBTB20/LSAMP基因组重排的存在包括检测具有来自ZBTB20基因的第一核酸部分和来自LSAMP基因的第二核酸部分的嵌合mRNA或cDNA转录物。在一个实施方案中,嵌合mRNA或cDNA转录物包含ZBTB20基因的外显子1(例如E1、E1A、E1B或E1C)和LSAMP基因的外显子4之间的融合。例如,嵌合mRNA或cDNA转录物可包含SEQIDNO:4、SEQIDNO:32、SEQIDNO:34、SEQIDNO:35、SEQIDNO:36、SEQIDNO:37、SEQIDNO:38或SEQIDNO:40的核苷酸序列。在另一个实施方案中,嵌合mRNA或cDNA转录物包含ZBTB20基因的外显子1(例如E1、E1A、E1B或E1C)和LSAMP基因的外显子3*之间的融合。例如,嵌合mRNA或cDNA转录物可包含SEQIDNO:33、SEQIDNO:39或SEQIDNO:41的核苷酸序列。外显子3*(SEQIDNO:45)表示来自之前未被诠释并且现已表明在ZBTB20和LSAMP基因的基因组重排后产生的LSAMP基因座的一个新的外显子序列。因此,检测生物样品中ZBTB20/LSAMP基因组重排的存在可包括检测对应于其它基因中不存在的外显子3*的区域的mRNA或cDNA转录物,并因此可用于明确鉴定出LSAMP基因的外显子3*。或者,检测生物样品中ZBTB20/LSAMP基因组重排的存在可包括检测包含跨越LSAMP的外显子3*和外显子4之间的连接处且不存在于其它基因中的LSAMP基因的外显子3*和外显子4或其部分的mRNA或cDNA转录物,并因此可用于将转录物明确鉴定为来自LSAMP的外显子3*和外显子4的融合。外显子0*(SEQIDNO:47)表示来自之前未被诠释并且在具有ZBTB20和LSAMP基因的基因组重排的患者的可变剪接的转录物中鉴定出的LSAMP基因座的一个新的外显子序列。因此,检测生物样品中ZBTB20/LSAMP基因组重排的存在可包括检测对应于其它基因中不存在的外显子0*的区域的mRNA或cDNA转录物,并因此可用于明确鉴定出LSAMP基因的外显子0*。或者,检测生物样品中ZBTB20/LSAMP基因组重排的存在可包括检测包含跨越LSAMP基因的外显子0*和外显子1之间的连接处且不存在于其它基因中的LSAMP基因的外显子0*和外显子1或其部分的mRNA或cDNA转录物,并因此可用于将转录物明确鉴定为来自LSAMP的外显子0*和外显子1的融合。可通过测量或检测对应于基因的基因组重排的基因组序列或mRNA/cDNA转录物或对应于与ZBTB20和LSAMP基因的基因组重排相关的所有基因组序列或mRNA/cDNA转录物的一个或多个,来测量或检测ZBTB20/LSAMP基因组重排的表达。检测PTEN基因中的缺失包括检测染色体10q23区域的缺失,其中缺失跨越PTEN基因或其部分。可通过测量或检测对应于PTEN缺失或对应于与PTEN基因有关的所有基因组序列或mRNA/cDNA转录物的基因组序列或mRNA/cDNA转录物的一个或多个,来测量或检测PTEN缺失。染色体重排可采用已知技术检测。例如,荧光原位杂交(FISH)分析可用于检测染色体重排。在这些实施方案中,使在高严格性条件下与染色体重排(例如ZBTB20/LSAMP染色体重排或PTEN缺失)杂交的核酸探针与包含前列腺细胞的生物样品(或获自其中的核酸)一起温育。其它已知的原位杂交技术可用于检测染色体重排,例如ZBTB20/LSAMP或PTEN缺失。核酸探针(DNA或RNA)可与DNA或mRNA杂交,并可被设计成检测ZBTB20和LSAMP基因或PTEN基因中的基因组重排,例如基因融合事件、扩增、缺失或突变。通常,可根据mRNA或cDNA水平检测或测量基因表达,尽管适当时也可使用蛋白质水平。可采用用于测量mRNA水平、cDNA或蛋白质水平的任何定量或定性方法。检测或测量mRNA或cDNA水平的合适方法包括例如RNA印迹法、RNA酶保护测定法、微阵列分析或核酸扩增程序,例如反转录PCR(RT-PCR)或实时RT-PCR,亦称定量RT-PCR(qRT-PCR)。所述方法是本领域众所周知的。参见例如Sambrook等,MolecularCloning:ALaboratoryManual,第4版,ColdSpringHarborPress,ColdSpringHarbor,N.Y.,2012。其它技术包括基因表达的数字式多路复用分析,例如nCounter®(NanoStringTechnologies,Seattle,WA)基因表达测定法,其进一步描述于[22]、[23]、US20100112710和US20100047924,以上所有通过引用以其整体结合到本文中。检测目标核酸通常包括靶标(例如mRNA、cDNA或基因组DNA)和探针之间的杂交。本领域技术人员可容易地设计杂交探针用于检测ZBTB20和LSAMP基因的基因组重排或PTEN基因的缺失。参见例如Sambrook等,MolecularCloning:ALaboratoryManual,第4版,ColdSpringHarborPress,ColdSpringHarbor,N.Y.,2012。各个探针基本上对其靶标应有特异性,以避免任何交叉杂交和假阳性。在从转录物衍生物质时,使用特异性探针的一个替代是使用特异性试剂(例如在cDNA产生期间,或在扩增期间使用靶标特异性引物)。在两种情况下,可通过与在所分析的基因组内基本上是唯一的靶标的部分杂交,实现特异性,例如与聚A尾杂交不会提供特异性。如果靶标具有多个剪接变体,则有可能设计识别对各变体是共同的区域的杂交试剂和/或使用一种以上试剂,所述试剂的每一种可识别一个或多个变体。杂交反应的严格性可容易地通过本领域普通技术人员确定,一般根据探针长度、洗涤温度和盐浓度任经验计算。一般而言,对于适当退火,较长的探针需要较高的温度,则较短的探针需要较低的温度。当互补链存在于低于其解链温度的环境下时,杂交一般取决于变性的核酸序列再退火的能力。探针和可杂交序列之间所需同源性的程度越高,可使用的相对温度越高。因此,因此得出结论,较高的相对温度往往使反应条件较严格,而较低的相对温度则较不严格。有关杂交反应严格性的其它详情和解释参见Ausubel等,CurrentProtocolsinMolecularBiology,WileyIntersciencePublishers,(1995)。本文所用“严格条件”或“高严格性条件”根据以下识别,但不限于此:(1)对于洗涤使用低的离子强度和高的温度,例如0.015M氯化钠/0.0015M柠檬酸钠/0.1%十二烷基硫酸钠,在50℃下;(2)在杂交期间使用变性剂,例如甲酰胺,例如50%(v/v)甲酰胺与0.1%牛血清白蛋白/0.1%聚蔗糖(Ficoll)/0.1%聚乙烯吡咯烷酮/50mM磷酸钠缓冲液(pH6.5)与750mM氯化钠、75mM柠檬酸钠,在42℃下;或(3)使用50%甲酰胺、5XSSC(0.75MNaCl、0.075M柠檬酸钠)、50mM磷酸钠(pH6.8)、0.1%焦磷酸钠、5XDenhardt溶液、声波处理的鲑精DNA(50μg/ml)、0.1%SDS和10%硫酸葡聚糖,在42℃下,其中在42℃下在0.2XSSC(氯化钠/柠檬酸钠)中并在50%甲酰胺中在55℃下洗涤,接着包括含有0.1XSSC的EDTA、在55℃下的高严格性洗涤。Sambrook等,MolecularCloning:ALaboratoryManual,NewYork:ColdSpringHarborPress,1989描述了“中等严格条件”,但不限于此,包括使用不比上述那些严格的洗涤溶液和杂交条件(例如温度、离子强度和%SDS)。中等严格条件的实例是在包含以下的溶液中在37℃下温育过夜:20%甲酰胺、5XSSC(150mMNaCl、15mM柠檬酸三钠)、50mM磷酸钠(pH7.6)、5XDenhardt溶液、10%硫酸葡聚糖和20mg/mL变性已剪切鲑精DNA,接着在1XSSC中在约37-50℃下洗涤过滤器。技术人员应了解需要时如何调节温度、离子强度等以适应例如探针长度等因素。在某些实施方案中,采用微阵列分析或基于PCR的方法。在这方面,测量前列腺癌细胞中ZBTB20和LSAMP基因的基因组重排或PTEN缺失的表达可包括例如使含有或疑似含有前列腺癌细胞的样品与对ZBTB20/LSAMP基因组重排或PTEN缺失有特异性的多核苷酸探针或与经设计以扩增ZBTB20/LSAMP的一部分或PTEN基因组重排的引物接触,并分别检测探针与核酸靶标的结合或核酸的扩增。设计PCR引物的详细方案是本领域已知的。参见例如Sambrook等,MolecularCloning:ALaboratoryManual,第4版,ColdSpringHarborPress,ColdSpringHarbor,N.Y.,2012。类似地,制备和使用微阵列以分析的详细方案是本领域已知和本文描述的。因此,一方面涉及测定生物样品是否包含含有基因融合的核酸的方法,其中基因融合具有来自ZBTB20基因的第一部分和来自LSAMP基因的第二部分,所述方法包括:将含至少第一和第二多核苷酸引物的生物样品在杂交条件下混合,其中第一多核苷酸引物包含与来自ZBTB20基因的基因融合的第一部分(例如外显子1)杂交的序列,第二多核苷酸引物包含与来自LSAMP基因的基因融合的第二部分(例如外显子3*或外显子4)杂交的序列,其中第一和第二多核苷酸引物能够扩增来自跨越来自ZBTB20基因的基因融合的第一部分和来自LSAMP基因的基因融合的第二部分之间的连接处的基因融合的靶序列;如果基因融合存在于生物样品中,则在允许扩增靶序列并产生包含靶序列的扩增产物的条件下加入聚合酶活性;和根据扩增产物存在与否,确定生物样品是否含有基因融合。另一方面涉及测定包含核酸的生物样品是否含有对应于其它基因或基因融合中不存在的外显子3*区域的mRNA或cDNA序列的方法,其中基因融合具有来自LSAMP基因的外显子3*的第一部分和来自LSAMP基因的外显子4的第二部分,所述方法包括:将含至少第一和第二多核苷酸引物的生物样品在杂交条件下混合,(a)其中第一多核苷酸引物包含与外显子3*的第一区域杂交的序列,第二多核苷酸引物包含与外显子3*的第二区域杂交的序列,其中第一和第二多核苷酸引物能够扩增来自对LSAMP基因的外显子3*是特有的外显子3*的靶序列;或(b)其中第一多核苷酸引物包含与来自外显子3*的基因融合的第一部分杂交的序列,第二多核苷酸引物包含与来自LSAMP基因的外显子4的基因融合的第二部分杂交的序列,其中第一和第二多核苷酸引物能够扩增来自跨越来自LSAMP基因的外显子3*的基因融合的第一部分和来自LSAMP基因的外显子4的基因融合的第二部分之间的连接处的基因融合的靶序列;如果基因融合存在于生物样品中,则在允许扩增靶序列并产生包含靶序列的扩增产物的条件下加入聚合酶活性;和根据扩增产物存在与否,测定生物样品是否含有对应于其它基因或基因融合中不存在的外显子3*区域的mRNA或cDNA序列。另一方面涉及测定包含核酸的生物样品是否含有对应于其它基因或基因融合中不存在的LSAMP基因的外显子0*的区域的mRNA或cDNA序列的方法,其中基因融合具有来自LSAMP基因的外显子0*的第一部分和来自LSAMP基因的外显子1的第二部分,所述方法包括:将含至少第一和第二多核苷酸引物的生物样品在杂交条件下混合,(a)其中第一多核苷酸引物包含与外显子0*的第一区域杂交的序列,第二多核苷酸引物包含与外显子0*的第二区域杂交的序列,其中第一和第二多核苷酸引物能够扩增来自对LSAMP基因的外显子0*是特有的外显子0*的靶序列;或(b)其中第一多核苷酸引物包含与来自外显子0*的基因融合的第一部分杂交的序列,第二多核苷酸引物包含与来自LSAMP基因的外显子1的基因融合的第二部分杂交的序列,其中第一和第二多核苷酸引物能够扩增来自跨越来自LSAMP基因的外显子0*的基因融合的第一部分和来自LSAMP基因的外显子1的基因融合的第二部分之间的连接处的基因融合的靶序列;如果基因融合存在于生物样品中,则在允许扩增靶序列并产生包含靶序列的扩增产物的条件下加入聚合酶活性;和根据扩增产物存在与否,测定生物样品是否含有对应于其它基因或基因融合中不存在的外显子0*的区域的mRNA或cDNA序列。备选或此外,ZBTB20/LSAMP基因组重排的表达水平可在蛋白质水平上测量,意味着当ZBTB20/LSAMP基因组重排导致截短的LSAMP蛋白或嵌合ZBTB20/LSAMP蛋白时,测量由ZBTB20/LSAMP基因组重排编码的这类蛋白质的水平。用于测定蛋白质的水平的若干方法和装置是众所周知的,包括例如描述于例如美国专利号6,143,576、6,113,855、6,019,944、5,985,579、5,947,124、5,939,272、5,922,615、5,885,527、5,851,776、5,824,799、5,679,526、5,525,524、5,458,852和5,480,792的免疫测定法,其每一个通过引用以其整体结合到本文中。这些测定法包括各种夹心、竞争性或非竞争性测定方式,以产生与目标蛋白质的存在或量有关的信号。可采用任何合适的免疫测定法,例如侧向流动、酶联免疫测定法(ELISA)、放射免疫测定法(RIA)、竞争结合测定法等。描述了用于抗体阵列的多种形式。所述阵列通常包括对待检测的不同蛋白质有特异性的不同抗体。例如,可使用至少100种不同的抗体来检测100种不同的蛋白质靶标,每种抗体对每个靶标有特异性。还可使用对特定蛋白质靶标有特异性的其它配体,例如公开于WO/2008/048970的合成抗体,其通过引用以其整体结合到本文中。可从肽或小分子的随机文库中选择具有所需结合特异性的其它化合物。美国专利号5,922,615(其通过引用以其整体结合到本文中)描述了使用膜上的固定化膜的多个分立区带以检测阵列中的多种靶抗原的装置。可使用微量滴定板或自动化以利于大量不同蛋白质的检测。免疫测定法的一个类型,称为核酸检测免疫测定法(NADIA),将免疫测定法的蛋白质抗原检测的特异性与聚合酶链式反应(PCR)的灵敏度和精确度结合。这种增强的DNA-免疫测定方法与酶免疫测定法的类似,包括抗体结合反应和中间洗涤步骤,只是酶标记被DNA的链置换,且采用扩增技术(例如PCR)通过扩增反应检验。示例性NADIA技术描述于美国专利号5,665,539和公开的美国申请2008/0131883,其两个通过引用以其整体结合到本文中。简单地说,NADIA使用对目标蛋白质有特异性和用试验特异性核酸标记的第一(报道)抗体。核酸的存在不干扰抗体的结合,抗体也不干扰核酸扩增和检测。通常,把对目标蛋白质上的不同表位有特异性的第二(俘获)抗体包被在固相(例如顺磁粒子)上。使报道抗体/核酸缀合物在微量滴定板中与样品反应,以与靶抗原形成第一免疫复合物。然后将免疫复合物俘获在用俘获抗体包被的固相粒子上,形成不溶性夹心免疫复合物。洗涤微粒子以除去过量的未结合的报道抗体/核酸缀合物。然后通过使悬浮粒子进行扩增反应(例如PCR)并监测扩增的核酸产物,来检测结合的核酸标记。虽然免疫测定法通常已被用于蛋白质的鉴定和量化,但是质谱学(MS)技术的最新发展导致灵敏的高通量MS蛋白质分析的研发。MS方法可用来检测复杂生物样品中的低浓度的蛋白质。例如可在MS分析之前使生物样品分级来进行定向MS。用于进行MS分析前的这类分级的常见技术包括二维电泳、液相色谱法和毛细管电泳[25],所述参考文献通过引用以其整体结合到本文中。还出现选择反应监测(SRM),亦称多反应监测(MRM),作为有用的高通量基于MS的技术用于定量测定复杂生物样品中的靶定蛋白质,包括由基因融合(例如TMPRSS2/ERG)编码的前列腺癌生物标志物[26,27],所述参考文献通过引用以其整体结合到本文中。样品本申请所述方法包括分析细胞(包括前列腺细胞)中的ZBTB20和LSAMP基因的基因组重排或PTEN基因。这些前列腺细胞存在于生物样品中,例如前列腺组织、血液、血清、血浆、尿液、唾液或前列腺液。可在检测基因表达之前,从细胞中分离核酸或多肽。在一个实施方案中,生物样品包含前列腺组织并通过活组织检查(例如直肠或经会阴活检)获得。在另一个实施方案中,生物样品是尿液。尿液样品可在直肠指检(DRE)或前列腺活检后收集。在另一个实施方案中,样品是血液、血清或血浆,并含有从原发性肿瘤剥离的循环肿瘤细胞。样品还可含有肿瘤来源的外来体。外来体是小的(通常30-100nm)膜结合颗粒,它从正常细胞、患病细胞和肿瘤细胞中释放,并存在于血液和其它体液中。本申请公开的方法可与从多种哺乳动物收集的样品,但优选与从人类受试者获得的样品一起使用。前列腺癌本申请公开了与前列腺癌有关的ZBTB20和LSAMP基因之间的某些染色体重排。生物样品ZBTB20/LSAMP基因组重排的检出可用于鉴定样品中癌细胞,例如前列腺癌细胞,或测量前列腺癌的严重程度或侵袭性,例如区分高度分化的前列腺(WD)癌和分化差的(PD)前列腺癌和/或鉴定在前列腺切除术后转移或复发或在前列腺切除术后更可能转移或复发的前列腺癌。本申请还公开了在高加索血统受试者中占优势地发生的肿瘤抑制物基因PTEN的缺失,即使不是专有的话。相反地,PTEN缺失是在非洲血统受试者(AD)前列腺癌中的稀有事件,特别是AD受试者Gleason6-7前列腺癌。值得注意的是,Gleason6-7(亦称主要模式3)CaP表示PSA筛查患者群中最常诊断的CaP形式。当在活检中发现前列腺癌时,通常进行分级以估计它将如何快地生长和扩散。最常用的前列腺癌分级系统,称为Gleason分级,根据其在显微镜下观察到的形式,以1-5的标度评价前列腺癌细胞。仍像健康前列腺细胞的癌细胞具有边界清晰可辨的均一形式,并被视为高度分化的(Gleason1和2级)。癌细胞越接近地类似于前列腺组织,细胞将越像正常前列腺组织运行,且癌越少侵袭性。Gleason3级,最常见的等级,显示中等分化的细胞,也就是说仍有些高度分化,但是边界不清晰可辨。分化差的癌细胞具有边界分辨不清的随机形式,并且不再像前列腺组织(Gleason4和5级),表明更大侵袭性的癌。前列腺癌常常具有不同等级的区域。通过将肿瘤内的2种最常见的癌细胞形式的等级相加求出综合Gleason评分。例如,如果最常见的形式是4级,次最常见的形式是3级,则综合Gleason评分为4+3=7。如果肿瘤内只有一种形式,则综合Gleason评分可低至1+1=2或高达5+5=10。2-4的综合评分被视为高度分化的,5-6的评分被视为中等分化的,7-10的评分被视为分化差。有高Gleason评分的癌症在发现时很可能已扩散到前列腺以外(已转移)。一般而言,Gleason评分越低,癌症侵袭性越小且预后越好(展望治愈或长期生存)。Gleason评分越高,癌症侵袭性越大且长期无转移生存的预后越差。患者治疗本申请描述了诊断和预测获自受试者的样品中的前列腺癌的方法,其中分析了前列腺细胞和/或组织中的基因表达。如果样品显示ZBTB20和LSAMP基因的基因组重排的表达,则受试者患有前列腺癌或前列腺癌的更晚期/攻击形式(例如PD前列腺癌)的可能性加大。如果是这样的结果的话,检测或预测前列腺癌的方法可包括下列步骤的一个或多个:知会患者他们可能患有前列腺癌或PD前列腺癌;进行前列腺组织的证实性组织学检查;和/或对受试者进行治疗。因此,在某些方面,如果检测步骤表明受试者的前列腺细胞具有ZBTB20和LSAMP基因的基因组重排,则该方法进一步包括从受试者中采取前列腺活检和在活检(例如组织学检查)中检查前列腺组织以证实患者是否患有前列腺癌或侵袭形式的前列腺癌的步骤。或者,检测或预测前列腺癌的方法可用来评价对治疗的需要或监测对治疗的反应(例如手术或其它疗法后的无病复发),并因此可包括治疗患有前列腺癌的受试者的额外步骤。前列腺癌治疗选择包括手术、放射疗法、激素疗法、化学疗法、生物疗法或高强度聚焦超声。获准用于前列腺癌的药物包括:醋酸阿比特龙、卡巴他赛(Cabazitaxel)、地加瑞克、Enzalutamide(XTANDI)、Jevtana(卡巴他赛)、泼尼松、Provenge(西普鲁塞T)、西普鲁塞T或多西他赛。因此在阳性结果之后,本申请所述方法可进一步包括手术、放射疗法、激素疗法、化学疗法、生物疗法或高强度聚焦超声的步骤。计算机执行模型按照本发明的所有方面和实施方案,所提供的方法可为计算机执行的。可在数字计算机中分析ZBTB20和LSAMP基因的基因组重排或PTEN基因的状况并与受试者的状况(例如前列腺癌的存在或疾病的严重程度(例如WD或PD前列腺癌))相关联。任选所述计算机接收与ZBTB20和LSAMP基因的基因组重排的表达或PTEN基因的缺失有关的实验测定信号的扫描仪等直接连接。或者,可通过其它方法输入表达水平。可对计算机编程以将原始信号转化成表达水平(绝对或相对),将测量表达水平与一个或多个参比表达水平或这类值的标度进行比较。还可根据与一个或多个参比表达水平的比较对计算机编程以以对表达水平赋值或指定其它标识,并汇集表达谱中多个基因的所述值或标识。还可对计算机编程以输出提供表示前列腺癌的存在或严重程度的值或其它标识以及用于确定所述值或标识的任何原始或中间数据。一台典型的计算机(参见美国专利号6,785,613;图4和5)包括使以下主要子系统互连的总线:例如中央处理器、系统存储器、输入/输出控制器、外部设备例如经由并行端口打印机、经由显示适配器的显示屏、串行端口、键盘、固定磁盘驱动器和有效接收外部存储设备的端口(例如USB端口)。可连接许多其它设置,例如经由I/O控制器的扫描仪、与串行端口或网络接口连接的鼠标。计算机含有保存允许计算机执行多种功能的代码的计算机可读介质。这些功能包括上述接收输入和发送输出的控制自动化设备。自动化设备可包括用于递送测定表达水平的试剂的机器臂以及小容器,例如用于进行表达分析的微量滴定板孔。一台典型的计算机系统106还可包括与在图3中给出且上文论述的操作系统控制下或与操作系统联合操作的随机存储器连接的一个或多个处理器110。在一个实施方案中,本发明的计算机执行方法的任一种可包括通过至少1个处理器获取反映生物样品是否含有ZBTB20和LSAMP基因的基因组重排或PTEN基因的缺失的信息的步骤。在一个实施方案中,生物样品获自非洲血统的受试者。在另一个实施方案中,生物样品获自受试者,其中受试者的前列腺癌不表达TMPRSS2/ERG基因融合。在另一个实施方案中,本发明的计算机执行方法的任一种可进一步包括通过至少1个处理器获得反映获自非洲血统患者的生物样品中下列人基因的至少2、3、4、5、6或7个的表达水平的信息的步骤:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1。在本发明的计算机执行方法的另一个实施方案中,该方法可进一步包括以用户可读格式输出在获取步骤中获得的信息的步骤。在本发明的计算机执行方法的另一个实施方案中,该方法可进一步包括以用户可读格式输出根据输出步骤传送的信息对受试者患有前列腺癌或分化差的前列腺癌的测定结果。组合物和试剂盒可在组合物或试剂盒中安排用于本申请所述方法的多核苷酸探针和/或引物或抗体或多肽探针。因此,一个实施方案涉及用于诊断或预测前列腺癌的组合物,其包含用于检出ZBTB20和LSAMP基因的基因组重排的多核苷酸探针。在某些实施方案中,在基因组重排包含ZBTB20和LSAMP基因之间的融合时,例如ZBTB20基因的外显子1(例如E1、E1A、E1B或E1C)和LSAMP基因的外显子3*或外显子4之间的融合,多核苷酸探针在高严格性条件下与嵌合核酸的连接处杂交,其中嵌合核酸包含来自ZBTB20基因的第一部分(例如外显子1的全部或一部分,这包括E1、E1A、E1B或E1C)和来自LSAMP基因的第二部分(例如外显子3*或外显子4的全部或一部分)。本文所述多核苷酸探针可以任选被标记。在一个实施方案中,用于诊断或预测前列腺癌的组合物包含多核苷酸探针,其中多核苷酸探针被设计来检测具有来自ZBTB20基因的第一部分(例如外显子1的全部或一部分,这包括E1、E1A、E1B或E1C)和来自LSAMP基因的第二部分(例如外显子3*或外显子4的全部或一部分)的基因组DNA的染色体重排。在一个实施方案中,多核苷酸探针在高严格性条件下与包含SEQIDNO:1的核苷酸序列的染色体重排杂交。在另一个实施方案中,用于诊断或预测前列腺癌的组合物包含多核苷酸探针,其中多核苷酸探针被设计来检测具有来自ZBTB20基因的第一核酸部分和来自LSAMP基因的第二核酸部分的嵌合mRNA或cDNA转录物。在一个实施方案中,嵌合mRNA或cDNA转录物包含ZBTB20基因的外显子1(例如E1、E1A、E1B或E1C)和LSAMP基因的外显子4之间的融合。例如,在某些实施方案中,多核苷酸探针在高严格性条件下与具有SEQIDNO:4、SEQIDNO:32、SEQIDNO:34、SEQIDNO:35、SEQIDNO:36、SEQIDNO:37、SEQIDNO:38或SEQIDNO:40的核苷酸序列的嵌合mRNA或cDNA转录物杂交。在另一个实施方案中,嵌合mRNA或cDNA转录物包含ZBTB20基因的外显子1(例如E1、E1A、E1B或E1C)和LSAMP基因的外显子3*之间的融合。例如,在某些实施方案中,多核苷酸探针在高严格性条件下与具有SEQIDNO:33、SEQIDNO:39或SEQIDNO:41的核苷酸序列的嵌合mRNA或cDNA转录物杂交。在另外又一个实施方案中,用于诊断或预测前列腺癌的组合物包含多核苷酸探针,其中多核苷酸探针被设计来检测染色体3q13区域的缺失,其中该缺失跨越ZBTB20和LSAMP基因。在其它实施方案中,多核苷酸探针在高严格性条件下与LSAMP基因的外显子3*(SEQIDNO:45)或嵌合核酸的连接处杂交,其中嵌合核酸包含来自LSAMP基因的外显子3*的第一部分和来自LSAMP基因的外显子4的第二部分。在另外其它的实施方案中,多核苷酸探针在高严格性条件下与LSAMP基因的外显子0*(SEQIDNO:47)或嵌合核酸的连接处杂交,其中嵌合核酸包含来自LSAMP基因的外显子0*的第一部分和来自LSAMP基因的外显子1的第二部分。用于诊断或预测前列腺癌的组合物还可包含引物。在一个实施方案中,其中基因组重排包含ZBTB20和LSAMP基因之间的基因融合,例如ZBTB20基因的外显子1(例如E1、E1A、E1B或E1C)和LSAMP基因的外显子3*或外显子4之间的融合,组合物包含用于扩增ZBTB20/LSAMP基因融合的引物,具体地说用于扩增来自跨越来自ZBTB20基因的基因融合的第一部分(例如外显子1的全部或一部分,这包括E1、E1A、E1B或E1C)和来自LSAMP基因的基因融合的第二部分(例如外显子3*或外显子4的全部或一部分)之间的连接处的基因融合的核苷酸序列的引物。这样,引物可用于特异性地鉴定ZBTB20和LSAMP基因之间的基因融合。在一个实施方案中,组合物含有包含在高严格性条件下与来自ZBTB20基因的基因融合的第一部分(例如外显子1的全部或一部分,这包括E1、E1A、E1B或E1C)杂交的序列的第一多核苷酸引物;和包含在高严格性条件下与来自LSAMP基因的基因融合的第二部分(例如外显子3*或外显子4的全部或一部分)杂交的序列的第二多核苷酸引物,其中第一和第二多核苷酸引物能够扩增来自跨越来自ZBTB20基因的基因融合的第一部分和来自LSAMP基因的基因融合的第二部分之间的连接处的基因融合的核苷酸序列。在某些实施方案中,组合物包含第一和第二多核苷酸引物用于扩增ZBTB20基因和LSAMP基因之间的基因融合,其中第一和第二多核苷酸引物能够扩增跨越来自ZBTB20基因的基因融合的第一部分和来自LSAMP基因的基因融合的第二部分之间的连接处的SEQIDNO:32-41或其部分的一个或多个。在一个实施方案中,来自LSAMP基因的基因融合的第一部分包含SEQIDNO:2的核苷酸序列,来自ZBTB20基因的基因融合的第二部分包含SEQIDNO:3的核苷酸序列。在另一个实施方案中,来自ZBTB20基因的基因融合的第一部分包含SEQIDNO:5的核苷酸序列,来自LSAMP基因的基因融合的第二部分包含SEQIDNO:6的核苷酸序列。在另外又一个实施方案中,第一多核苷酸引物包含SEQIDNO:7的核苷酸序列,第二多核苷酸引物包含SEQIDNO:8的核苷酸序列。外显子3*(SEQIDNO:45)来自之前未被诠释且现不存在于产生于ZBTB20和LSAMP基因的基因组重排的融合转录物中的LSAMP基因座的一个新的外显子序列。因此,扩增对应于其它基因中不存在的外显子3*的区域的mRNA/cDNA序列提供用于检测ZBTB20和LSAMP基因的基因组重排的又一个机制。在一个实施方案中,组合物含有包含在高严格性条件下与LSAMP的外显子3*的第一部分杂交的序列的第一多核苷酸引物和包含在高严格性条件下与同LSAMP的外显子3*的第二部分互补的序列杂交的序列的第二多核苷酸引物,其中LSAMP的外显子3*的第一和第二部分不重叠,且其中多核苷酸引物能够扩增对3*外显子是特有的且不存在于其它基因序列的LSAMP的外显子3*内的核苷酸序列,并因此可用于明确鉴定出来自LSAMP的外显子3*的扩增序列。在其它实施方案中,第二多核苷酸引物在高严格性条件下与同LSAMP的外显子4内的区域互补的序列杂交,使得引物对产生跨越LSAMP的外显子3*和外显子4之间的连接处的扩增产物。第二引物还可与同LSAMP的外显子5、6或7互补的序列杂交。外显子0*(SEQIDNO:47)表示之前未被诠释且在具有ZBTB20和LSAMP基因的基因组重排的患者的可变剪接的转录物中鉴定出的LSAMP基因座的一个新的外显子序列。因此,扩增对应于其它基因中不存在的外显子0*的区域的mRNA/cDNA序列提供用于检测ZBTB20和LSAMP基因的基因组重排的又一个机制。在一个实施方案中,组合物含有包含在高严格性条件下与LSAMP的外显子0*的第一部分杂交的序列的第一多核苷酸引物和包含在高严格性条件下与同LSAMP的外显子0*的第二部分互补的序列杂交的序列的第二多核苷酸引物,其中LSAMP的外显子0*的第一和第二部分不重叠,且其中多核苷酸引物能够扩增对0*外显子是特有的且不存在于其它基因序列中的LSAMP的外显子0*内的核苷酸序列,并因此可用于明确鉴定外显子0*的扩增序列为来自LSAMP。在其它实施方案中,第二多核苷酸引物在高严格性条件下与同与LSAMP的外显子1内的区域互补的序列杂交,使得引物对产生跨越LSAMP的外显子0*和外显子1之间的连接处的扩增产物。第二引物还可与同LSAMP的外显子2、3或4内的区域互补的序列杂交。在另一个实施方案中,其中基因组重排包含染色体3q13区域处的缺失,其中缺失跨越两个ZBTB20和LSAMP基因,用于诊断或预测前列腺癌的组合物包含用于扩增通过缺失产生的嵌合连接处的引物。因此,在一个实施方案中,组合物含有包含在高严格性条件下与接近缺失的5’端的第一核酸杂交的序列的第一多核苷酸引物和包含在高严格性条件下与接近缺失的3’端的第二核酸杂交的序列的第二多核苷酸引物,其中第一和第二多核苷酸引物能够扩增跨越通过缺失产生的嵌合连接处的核苷酸序列,其中缺失发生在染色体3q13区域并跨越ZBTB20和LSAMP基因。另一方面涉及双链寡核苷酸双链体,其中寡核苷酸双链体包含与第二核酸杂交的第一核酸,其中第一核酸包含与来自LSAMP基因的第二部分融合的来自ZBTB20基因的第一部分,且其中第二核酸是与来自ZBTB20基因的第一部分和来自LSAMP基因的第二部分之间的连接处杂交的多核苷酸探针。多核苷酸探针被任选标记。在另一个实施方案中,寡核苷酸双链体包含与第二核酸杂交的第一核酸,其中第一核酸包含与来自LSAMP基因的外显子4的第二部分融合的来自LSAMP基因的外显子3*的第一部分,且其中第二核酸是与来自LSAMP基因的外显子3*的第一部分和来自LSAMP基因的外显子4的第二部分之间的连接处杂交的多核苷酸探针。多核苷酸探针被任选标记。在另外又一个实施方案中,寡核苷酸双链体包含与第二核酸杂交的第一核酸,其中第一核酸包含与来自LSAMP基因的外显子1的第二部分融合的来自LSAMP基因的外显子0*的第一部分,且其中第二核酸是与来自LSAMP基因的外显子0*的第一部分和来自LSAMP基因的外显子4的第二部分之间的连接处杂交的多核苷酸探针。多核苷酸探针被任选标记。另一方面涉及用于诊断或预测前列腺癌的试剂盒。在一个实施方案中,用于诊断或预测前列腺癌的试剂盒含有包含用于检测上述ZBTB20/LSAMP基因组重排的一种或多种多核苷酸探针和/或引物的第一组合物和包含在高严格性条件下与选自COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1的基因杂交的多核苷酸探针的第二组合物。在另一个实施方案中,用于诊断或预测前列腺癌的试剂盒含有包含用于检测上述ZBTB20/LSAMP基因组重排的一种或多种多核苷酸探针和/或引物的第一组合物和包含在高严格性条件下与选自ERG、AMACR、PCA3和PSA的基因杂交的多核苷酸探针的第二组合物。用于诊断或预测前列腺癌的试剂盒还可包含抗体。因此,在一个实施方案中,用于诊断或预测前列腺癌的试剂盒包含与由ZBTB20/LSAMP基因融合编码的多肽结合的抗体。抗体可以任选被标记。在另一个实施方案中,试剂盒另包含一种或多种抗体用于检测由下列人基因编码的多肽的至少1、2、3、4、5、6或7种:COL10A1、HOXC4、ESPL1、MMP9、ABCA13、PCDHGA1和AGSK1。在另一个实施方案中,试剂盒另包含一种或多种抗体用于检测ERG、AMACR或PSA。在一个实施方案中,用于诊断或预测前列腺癌的试剂盒包括公开了在所公开的方法中使用试剂盒内容物的方法的说明资料。说明资料可以许多形式提供,包括但不限于书面形式(例如硬拷贝纸等)、以电子形式(例如计算机磁盘或光盘)提供或可以是可视的(例如可视文件)。试剂盒还可包括有利于试剂盒针对其而设计的特殊应用的部件。因此,例如,试剂盒可另外包括常规用于实施特定方法的其它试剂,包括但不限于缓冲液、酶(例如聚合酶)、标记化合物等。所述试剂盒和合适的内容物为本领域技术人员所熟知。试剂盒还可包括参比或对照样品。参比或对照样品可以是生物样品或数据库。描述于本申请的多核苷酸探针和抗体任选用可检测标记进行标记。可以使用与本领域普通技术人员已知的探针或抗体技术联用的任何可检测标记。在一个具体的实施方案中,探针用选自以下的可检测标记进行标记:荧光标记、化学发光标记、猝灭剂、放射性标记、生物素、质量标签和/或金。与嵌合ZBTB20/LSAMP融合蛋白结合的抗体本公开内容提供与由ZBTB20/LSAMP基因融合编码的蛋白质结合的抗体。抗体,亦称免疫球蛋白,通常是由各约25kDa的2条轻(L)链和各约50kDa的2条重(H)链组成的四聚糖基化蛋白质。2个类型的轻链,称为λ和κ,可存在于抗体中。根据重链的恒定结构域的氨基酸序列,免疫球蛋白可指定为5个主要类别:A、D、E、G和M,且这些中的几个可进一步分成亚类(同种型),例如IgG1、IgG2、IgG3、IgG4、IgA1和IgA2。各轻链包括N端可变(V)结构域(VL)和恒定(C)结构域(CL)。各重链包括N端V结构域(VH)、3个或4个C结构域(CH)和铰链区。最接近VH的CH结构域被命名为CH1。VH和VL结构域由相对保守序列的4区称为构架区(FR1、FR2、FR3和FR4)组成,其形成超变序列(互补决定区,CDR)的3个区域的支架。CDR含有负责抗体与抗原特异性相互作用的大部分残基。CDR被称为CDR1、CDR2和CDR3。因此,重链上的CDR组分被称为H1、H2和H3,而轻链上的CDR组分称为L1、L2和L3。构架和CDR残基的鉴定和编号描述于Chothia等,Structuraldeterminantsinthesequencesofimmunoglobulinvariabledomain(免疫球蛋白可变结构域序列中的结构决定簇),JMolBiol1998,278:457-79,其通过引用以其整体结合到本文中。在一个实施方案中,由ZBTB20/LSAMP基因融合编码的蛋白质是截短的LSAMP蛋白。在某些实施方案中,与截短的LSAMP蛋白结合的抗体与存在于截短的LSAMP蛋白中但不存在于野生型LSAMP蛋白中的表位结合。在一个实施方案中,抗体与由ZBTB20基因的外显子1和LSAMP基因的外显子4之间的基因融合编码的嵌合多肽结合。在某些实施方案中,与由ZBTB20基因的外显子1和LSAMP基因的外显子4之间的基因融合编码的嵌合多肽结合的抗体与由不存在于野生型ZBTB20或野生型LSAMP蛋白中的基因融合编码的多肽中存在的表位结合。在另一个实施方案中,基因融合包含SEQIDNO:4的核苷酸序列。制备抗体或其抗原结合片段和配制所述抗体或其抗原结合片段用于治疗性给予的方法是众所周知的,描述于例如PCT/US2010/032714,其通过引用以其整体结合到本文中。与由ZBTB20/LSAMP基因融合编码的多肽结合的本文所述抗体可用于多种研究和医学应用。一方面,本公开内容提供治疗受试者的前列腺癌的方法,所述方法包括给予所述受试者治疗有效量的在药学上可接受的载体中配制的与由ZBTB20/LSAMP基因融合编码的多肽结合的抗体。本公开内容还提供包含与由ZBTB20/LSAMP基因融合编码的多肽结合的抗体的组合物。在某些实施方案中,组合物适于药物应用和给予患者。这些组合物包含与由ZBTB20/LSAMP基因融合编码的多肽结合的抗体和药学上可接受的赋形剂。组合物还可含有提供补充、额外或加强的治疗功能的其它活性化合物。药物组合物还可与给药说明书一起包括在容器、包装或分配器中。在一个实施方案中,组合物包含与由ZBTB20/LSAMP基因融合编码的多肽结合的单克隆抗体用于治疗前列腺癌。将本发明的药物组合物配制成与其既定给药途径相容。实现给药的方法为本领域普通技术人员所知。这包括例如通过例如静脉内、血管内、动脉内、皮下、肌内、肿瘤内、腹膜内、心室内、硬膜内等胃肠外途径注射或其它途径以及口服、经鼻、经眼、直肠或局部。还明确考虑了通过例如长效注射剂或易蚀植入剂等方法的缓释给药。特别考虑了通过例如导管递送到一个或多个动脉(例如供应局部性肿瘤的肾动脉或血管)的方法的局部递送。可通过标准药学方法测定细胞培养物或实验动物中组合物的毒性和治疗功效,例如测定LD50(50%群体的致死剂量)和ED50(50%群体中的治疗有效剂量)。毒性和治疗作用间的剂量比为治疗指数,可表示为LD50/ED50比。显示大的治疗指数的抗体可为较少毒性和/或较大治疗有效性。除非另有定义,否则本文所用的全部技术和科学术语都具有本领域普通技术人员通常理解的相同含义。虽然类似或等同于本文所述的方法和材料可用于实施或测试本发明,但下面描述了合适的方法和材料。万一有抵触,则以本说明书(包括定义)为准。另外,材料、方法和实施例只是说明性的,并无意是限制性的。实施例实施例1.比较基因组DNA分析在7名AA和7名CACaP患者(28个样本)的组群中,使用原发性前列腺肿瘤和相应的正常组织(血液)进行了比较全基因组分析。根据以下标准选择组群:基本治疗根治性前列腺切除术、无新辅助治疗、Gleason6-7(即,主要模式3或Gleason3+3和3+4级,这表示在诊断/基本治疗时PSA筛查的CaP的大部分),具有80%或更高肿瘤细胞内容物的冷冻肿瘤组织,得到超过2μg高分子量基因组DNA的解剖肿瘤组织,相应血液基因组DNA的可得性和患者临床病理数据。将28个样品送往IlluminaInc.(UK)以测序。应用Illumina的ELAND比对算法,对来自肿瘤样品的序列与参比基因组作图。测序报告良好的覆盖度(平均37)。应用Strelka算法,同时进行调用单核苷酸多态性(SNP)、小插入与小缺失(InDel)、拷贝数变异(CNV)和结构变异(SV)的变体。在该组群中,以预期频率鉴定出所有已建立的CaP突变(TMPRSS2/ERG、SPOP、CHD1和PTEN)。表2提供14名患者的基因组序列覆盖度概要。表2在14名患者的至少2名中鉴定出具有SNP、CNV或InDel体细胞突变的31个基因(包括已知突变):AC091435.2、APC、ASMTL、ASMTL-AS1、CDC73、CHD1、CSF2RA、EYS、FRG1、FRG1B、HK2、IL3RA、KLLN、LIPF、LOC100293744、MT-ATP6、MT-BD4、MT-CO1、MT-CYB、MT-ND2、MT-ND3、MUC16、MUC6、NOX3、PDHA2、PTEN、SLC25A6、SLC9B1、SPOP、TRAV20和USH2A。表3给出14名患者的至少2人中存在的最前面的SV和CNV(最高的置信度):表3在表3中,“评分”是各患者对于SV最高5或对于CNV最高4的各评分的总和(14名患者的理论最高评分对于SV为14x5=70,或对于CNV为14x4=56)。对于SV指定1、4或5的评分(其中5意指SV受RNA数据支持,4意指含剪接读取(spliceread)的SV,如果无剪接读取或无法获得RNA数据则为1),而对于CNV指定2或4的评分(如果通过一种算法预测则为2,如果通过一个以上算法预测则为4)。某些SNV、CNV或InDel体细胞突变显示与特定人种群优选关联(AD(非洲血统)、CD(高加索血统)、AD>CD或CD>AD)。具体地说,未料到AA患者中不存在PTEN缺失。在7名CD的4个中检出PTEN缺失,表明CD病例中PTEN缺失的潜在排外性。实施例2.根据种族性的ZBTB20/LSAMP和PTEN遗传变异的分层在14名患者的这个组群中鉴定出已确立的前列腺癌基因组缺陷(TMPRSS2/ERG,PTEN)以及影响染色体3q13上的ZBTB20/LSAMP基因座的一种新的反复重排。通过RT-PCR或FISH分析证实了所有3个(ZBTB20/LSAMP(GP10)、TMPRSS2/ERG(GP1-14)和PTEN(GP1-14))。表4+表示存在;-表明不存在在AD患者的任一个中未观察到PTEN缺失(0/7)。只在CD患者中检出(4/7),表明PTEN丢失是AD男性CaP发生的稀有事件。与PTEN缺失有关的ERG(通过与雄激素调节基因(例如TMPRSS2)的基因融合)的激活在AD的CaP患者中也不太常见[20]。当与其它结构(SV)或拷贝数(CNV)变异相比时,所观察到的PTEN缺失与CD种族性的关联是明显的,并且只与ERG重排类似(参见上表3)。在7名AD和7名CD的整个封固前列腺的连续切片中在同一肿瘤病灶中通过FISH测定法独立证实了通过全基因组测序鉴定的PTEN缺失。图4A。为了证实这些研究结果,在表示Gleason6、Gleason7或Gleason8-10肿瘤的41个AD和58个CD病例的组织微阵列(TMA)中,通过FISH测定法评价了PTEN缺失。TMA中提供包括来自各病例的不同肿瘤病灶的多个样品。检查了TMA中的所有核心,当与CD(62.1)病例相比时,在AD(19.5%)中观察到显著较低的PTEN缺失的总频率(表5A)。与标明的来自ADCaP的Gleason6-7肿瘤的全基因组序列的PTEN缺失的低频率一致,在15个Gleason合计6的仅1人(6.7%)和15个Gleason合计7的AD病例中的仅4人(27%)中发现PTEN缺失(表5B)。这与在19个Gleason合计6的10个(52.6%)中和21个Gleason合计7CDCaP的14个(66.7%)中发现的PTEN缺失频率形成鲜明对比。总之,这些结果证实了PTEN丢失是AD的CaP中的稀有事件的研究结果。在CaP注意到的种族差异包括当与亚洲男性相比时,CD中PTEN缺失的较高基因组频率[34]。最新报告显示与CDCaP患者相比,ADCaP患者中PTEN缺失频率较低[35,36]。表5A表5B在患有ERG阴性CaP(即无ERG/TMPRSS2融合)的AD患者中,在4名患者的3人中观察到ZBTB20-LSAMP染色体基因座(染色体3q13)的基因组重排。受影响的基因座的详细分析和RNA-Seq数据显示3q13基因座的3种不同的基因组重排。受影响的基因座的详细分析和RNA-Seq数据表明在一个实例中,3q13基因座的基因组重排包括串联重复,产生ZBTB20的外显子1和LSAMP的外显子4之间的融合。图1和2。更准确地说,基因组重排产生以下之间的融合:(a)具有以下序列的LSAMP基因的一部分:AGAGTCTCTTCTTTGGGTCTCTTCCACATAGCTTGTTTGTAATCTCCAAGAAAGACTTCACATTACAGGCTGAAAAGAATCACCTACGGTTTCCATATTTTGAAAGAAATTTTTAAAAACCATGAAAACAAACAAACAAAAATCCTAGTTTCCTTTATAAAATAGCAAAGGAAAGTTCTCTCTCCTGTCACCAGGAATATGATTATGATCAGTTGGTTATTTAGGTCACATGTGAAAGAAATGAAAGAGGAGGCATGGGAATGTAAGGGAGAATAGTAGTCTGCCCTCAAGTCTGCAAACG(SEQIDNO:2);和(b)具有以下序列的ZBTB20基因的一部分:CTCGAGAACAGTGAGCAATAAATTTTTCTTTATACATTACCCAGTCTGTGGTATTCTGTTATGGCAACACAAAATAAACTAAGACAGTATTATGTATTTTTTCTTTTGTTTTACATTTTACTAAGTGCCGACTTATTCGAAAAGGTAATTAGCTTTGGTTAATTATCAAAGTTTTGTCTTTCCTTTCCTACTTTTGTCCCACTAAGCAAAAAACAAAACAATGAGCATTGACCTTTACCTTTCTCTGGTAAGGGAGTATGGAAGGTTTTCTACTACTTTGTAAAAATACTGCTACAGATGG(SEQIDNO:3)。当组合时,在这一个实例中,ZBTB20和LSAMP基因的基因组重排具有下列序列:CTCGAGAACAGTGAGCAATAAATTTTTCTTTATACATTACCCAGTCTGTGGTATTCTGTTATGGCAACACAAAATAAACTAAGACAGTATTATGTATTTTTTCTTTTGTTTTACATTTTACTAAGTGCCGACTTATTCGAAAAGGTAATTAGCTTTGGTTAATTATCAAAGTTTTGTCTTTCCTTTCCTACTTTTGTCCCACTAAGCAAAAAACAAAACAATGAGCATTGACCTTTACCTTTCTCTGGTAAGGGAGTATGGAAGGTTTTCTACTACTTTGTAAAAATACTGCTACAGATGGAGAGTCTCTTCTTTGGGTCTCTTCCACATAGCTTGTTTGTAATCTCCAAGAAAGACTTCACATTACAGGCTGAAAAGAATCACCTACGGTTTCCATATTTTGAAAGAAATTTTTAAAAACCATGAAAACAAACAAACAAAAATCCTAGTTTCCTTTATAAAATAGCAAAGGAAAGTTCTCTCTCCTGTCACCAGGAATATGATTATGATCAGTTGGTTATTTAGGTCACATGTGAAAGAAATGAAAGAGGAGGCATGGGAATGTAAGGGAGAATAGTAGTCTGCCCTCAAGTCTGCAAACG(SEQIDNO:1)产生于该基因组重排的ZBTB20/LSAMP基因融合的cDNA序列被鉴定为:CACAACATCAAGAGCAGGAAAATGGACTCATTAGGGAGGCAGGCAGTCATTACCACTCACACTGTACTTCCAGGGAGACACCGATTATAAGAAGAGAAACTCAGCGCTGGGGAAGAAGGAAGGGAATTTGAAGGAGAAGAAGAATATCTGGAGATCCTTGGCATCACCAGGGAGCAGTCAGGCAAATATGAGTGCAAAGCTGCCAACGAGGTCTCCTCGGCGGATGTCAAACAAGTCAAGGTCACTGTGAACTATCCTCCCACTATCACAGAATCCAAGAGCAATGAAGCCACCACAGGACGACAAGCTTCACTCAAATGTGAGGCCTCGGCAGTGCCTGCACCTGACTTTGAGTGGTACCGGGATGACACTAGGATAAATAGTGCCAATGGCCTTGAGATTAAGAGCACGGAGGGCCAGTCTTCCCTGACGGTGACCAACGTCACTGAGGAGCACTACGGCAACTACACCTGTGTGGCTGCCAACAAGCTGGGGGTCACCAATGCCAGCCTAGTCCTTTTCAGACCTGGGTCGGTGAGAGGAATAAATGGATCCATCAGTCTGGCCGTACCACTGTGGCTGCTGGCAGCATCTCTGCTCTGCCTTCTCAGCAAATGTTAA(SEQIDNO:4)。在这个ZBTB20/LSAMP基因融合中,下列序列来源于ZBTB20的外显子1:CACAACATCAAGAGCAGGAAAATGGACTCATTAGGGAGGCAGGCAGTCATTACCACTCACACTGTACTTCCAGGGAGACACCGATTATAAGAAGAGAAACTCAGCGCTGGGGAAGAAG(SEQIDNO:5),序列的其余部分(SEQIDNO:6)来源于LSAMP的外显子4-7。本领域普通技术人员可设计引物用于扩增这个ZBTB20/LSAMP基因融合。在一个实施方案中,正向引物包含SEQIDNO:5的核苷酸序列或与之互补的序列,反向引物包含SEQIDNO:6的核苷酸序列或与之互补的序列。下列示例性引物被设计成扩增这个具体的ZBTB20/LSAMP基因融合:ZBTB20/LSAMP正向引物:GCAGGCAGTCATTACCACTC(SEQIDNO:7)ZBTB20/LSAMP反向引物:TGACTTGTTTGACATCCGCC(SEQIDNO:8)在另一个实例中,3q13基因座的基因组重排包括缺失,产生ZBTB20基因和LSAMP基因之间的融合。图1。在第3个实例中,3q13基因座的基因组重排包括跨越ZBTB20和LSAMP基因两者的至少22.7Mb的大的缺失。图1。引人注目的是,具有染色体3q13重排的3名患者的2名发生了转移(在该组群中唯一的2个转移),且第3名具有CaP的生化复发(在该组群中1/3),表明了ZBTB20/LSAMP基因组重排与侵袭形式的前列腺癌或发生侵袭形式的前列腺癌的可能性加大有关。图1。为了证实ZBTB20/LSAMP融合转录物与AA种族性、阴性ERG状况和不利疾病结局的相关性,使用PCR和上述引物,对AA和CA血统的20个另外的CaP肿瘤和24个CaP肿瘤衍生细胞系进行了评价。所分析的20个AA肿瘤样品中,1个含有出现在GP10样本中的相同的ZBTB20/LSAMP融合转录物。该患者患有ERG阴性、侵袭形式的前列腺癌(分化差且向转移发展)。在34个正常样品(20个正常前列腺组织样本和来自7个CA和7个AA患者的组成型DNA)的mRNA或基因组DNA中未检出ZBTB20/LSAMP基因组重排。在1个CaP细胞系(CPDRRC92)中检出ZBTB20/LSAMP重排,该细胞系来源于患有分化差的前列腺癌的AD患者。在2个AD病例的肿瘤基因组中鉴定的ZBTB20-LSAMP缺失,用FISH测定法通过探测染色体3的基因组区(从ZBTB20启动子上游序列通过GAP43基因直到LSAMP基因座3’相邻区)得到证实,图4B。AD病例之一带有ZBTB20-LSAMP重复重排,预测了失活的ZBTB20的启动子和第一外显子与LSAMP编码序列之间的基因融合。通过界定5’融合配偶体、ZBTB20的第一外显子、融合连接处和3’融合配偶体、LSAMP外显子4的整个cDNA序列的5’-RACE方法,证实了该基因融合。融合转录物消除导致LSAMP蛋白的成熟前截短的LSAMP基因的天然翻译起始(ATG)。为了证实研究结果,在代表Gleason6、Gleason7或Gleason8-10肿瘤的23个AD和7个CD病例中,通过用于检测ZBTB20和LSAMP基因基因座之间的DNA区存在与否的FISH测定法,对组织微阵列(TMA)进行了评价。在26%(6/23)的AD病例中检出缺失,而在CD病例中仅在1/7(14%)中观察到缺失(表6A)。在具有缺失的7个病例中,6个具有生化复发(表6B)。只在具有ZBTB20-LSAMP区域中的缺失的7名患者的2名中发现ERG表达重排。只在7例中的1例中发现PTEN缺失(表6B)。具有ZBTB20-LSAMP区域的缺失的7名患者中6人遭受生化复发(BCR)或转移(Met)(表6B)。这些数据支持我们的最初发现,即在预后差(在前列腺切除术后生化复发或转移)的ADCaP患者中显示显著较高比例的ZBTB20-LSAMP区缺失的关联性。表6AADCD缺失6(26.09%)1(14.29%)无缺失17(73.91%)6(85.71%)合计237表6B种族性ERGPTENBCR/MetAD阴性wt是AD阴性wt否AD阴性wt是AD阳性wt是/是AD阴性wt是AD阳性wt是CD阴性缺失是受影响的3q13基因座和RNA-Seq数据的评价表明1个ADERG阴性病例中的基因组重排包括产生5’-ZBTB20-LSAMP-3’融合转录物的串联重复。还通过“cDNA末端快速扩增”(RapidAmplificationofcDNAEnd,RACE)方法,对来自该ADERG阴性病例的5’-ZBTB20-LSAMP-3’融合转录物进行了评价。将RACE产物的序列克隆在M13测序载体中,并通过各RACEcDNA产物的6个克隆的正向和反向DNA测序来证实。鉴定出10个原型CaP相关的融合cDNA,分别为ZBTB20-LSAMP融合1-10型和LSAMP基因座的1个可变剪接cDNA(LSAMP前列腺癌可变剪接形式1型:LPCS1)。图5。下面给出这些转录物的序列,用黑体文本表示来自ZBTB20基因的部分,下划线表示在RACE方法中用作反向引物的序列。通用5’RACE序列在这些扩增方法中用作正向引物。融合转录物的外显子和LPCS1在正常文本和斜体文本之间交替。1型:ZBTB20-E1-LSAMP-E4-E5-E6-E7AGAGTACATGCGGCGGGGGGAAGTTTAGGAGTTGAGGAAAGAAGATTAAAGAGCGCGAGGAGGAAGGGAATTTGAAGGAGAAGAAGAATATCTGGAGATCCTTGGCATCACCAGGGAGCAGTCAGGCAAATATGAGTGCAAAGCTGCCAACGAGGTCTCCTCGGCGGATGTCAAACAAGTCAAGGTCACTGTGAACTATCCTCCCACTATCACAGAATCCAAGAGCAATGAAGCCACCACAGGACGACAAGCTTCACTCAAATGTGAGGCCTCGGCAGTGCCTGCACCTGACTTTGAGTGGTACCGGGATGACACTAGGATAAATAGTGCCAATGGCCTTGAGATTAAGAGCACGGAGGGCCAGTCTTCCCTGACGGTGACCAACGTCACTGAGGAGCACTACGGCAACTACACCTGTGTGGCTGCCAACAAGCTGGGGGTCACCAATGCCAGCCTAGTCCTTTTCAGACCTGGGTCGGTGAGAGGAATAAATGGATCCATCAGTCTGGCCGTACCACTGTGGCTGCTGGCAGCATCTCTGCTCTGCCTTCTCAGCAAATGTTAATAGAATAAAAATTTAAAAATAATTTAAAAAACACACAAAAATGTGTCACACAGAATACAGAGAGAGAGAGACAGAGAGAGAGAGAGAGAGAGAGATGGGGGAGACCGTTTATTTCACAACTTTGTGTGTTTATACATGAAGGGGGAAATAAGAAAGTGAAGAAGAAAATNACAACATTTAAAACAATTTTACAGTCCATCATTAAAAATTTATGTATCATTCAGGATGGAGAAGGTTCTACTGGGATATGTTTATATCTACTAAGCAAATGTATGCTGTGTAAAGACTACACCACACTAAGGACATCTGGATGCTGTAAAAATAAGAGAAGAACCAGATGGATATTAAGCCCCCCAACACACACTTTATCCTTCCTTCCTTCATCTTTTTTCATCTGTGGGGAAGAAAATAAGGTCTTGCCTTTGGTGTTTATATTTCCATAACCTTTTAATTCTATTTTTCATTTGAGCTGACTTGTAGCCACTTCAGACTATCAATGGAATCTTATGTTGAGCCTTTCTCTGGCTTTCCTTCCTCCACTATCTCTCCAACTTTAGAGATCATCCCCTCTCCCTCCAGTGCGTTCTATCTCCCCCACACCCACCCAA(SEQIDNO:32)2型:ZBTB20-E1CLSAMPE3A-E4-E5-E6-E7ACATGGGGAGGTTGCAGTGTGTGTATATACACAACATCAAGAGCAGGAAAATGGACTCATTAGGGAGGCAGGCAGTCATTACCACTCACACTGTACTTCCAGGGAGACACCGATTATAAGAAGAGAAACTCAGCGCTGGGGAAGAAGATTAACTTACTCTTAATGATCTTCCAACACTTGAGAAGGTCAGTAGCCCTCCATCTGTCATTCTCCAAGTTCACCAACAGCTTATCCACCCATCAAAGGTGCTTTTGTAACAAAATCCATGCATAATGAAACCAAGAAAGGAAGGGAATTTGAAGGAGAAGAAGAATATCTGGAGATCCTTGGCATCACCAGGGAGCAGTCAGGCAAATATGAGTGCAAAGCTGCCAACGAGGTCTCCTCGGCGGATGTCAAACAAGTCAAGGTCACTGTGAACTATCCTCCCACTATCACAGAATCCAAGAGCAATGAAGCCACCACAGGACGACAAGCTTCACTCAAATGTGAGGCCTCGGCAGTGCCTGCACCTGACTTTGAGTGGTACCGGGATGACACTAGGATAAATAGTGCCAATGGCCTTGAGATTAAGAGCACGGAGGGCCAGTCTTCCCTGACGTGACCAACGTCACTGAGAGGNGAGCACTACGGCAACTACACCTGTGTGGCTGCCAACAAGCTGGGGGTCACAATGCCAGCCTAGTCCTTTTCAGACNTGGKYSGTGAGAGGAATAAATGGATCCATCAGTCTGGCCGTACCACTNGTGGCTGCTGGCAGCAATNNTCTCTGCTCTGCCGTCTCAGCAAATGTTAATAGAATAAAAATTTAAAAATAATTTAAAAAACACACAAAAATGCGTCACACAGAATACAGAGAGAGAGAGACAGAGAGAGAGAGAGAGAGAGAGATGGGGGAGACCGTTTATTTCACAACTTTGTGTGTTTATACATGAAGGGGGAAATAAGAAAGTGAAGAAGAAAATACAACATTTAAAACAATTTTACAGTCCATCATTAAAAATTTATGTATCATTCAGGATGGAGAAGGTTCTACTGGGATATGTTTATATCTACTAAGCAAATGTATGCTGTGTAAAGACTACACCACACTAAGGACATCTGGATGCTGTAAAAATAAGAGAAGAACCAGATGGATATTAAGCCCCCCAACACACACTTTATCCTTCCTTCCTTCATCTTTTTTCATCTGTGGGGAAGAAAATAAGGTCTTGCCTTTGGTGTTTATATTTCCATAACCTTTTAATTCTATTTTTCATTTGAGCTGACTTGTAGCCACTTCAGACTATCAATGGAATCTTATGTTGAGCCTTTCTCTGGCTTTCCTTCCTCCACTATCTCTCCAACTTTAGAGATCATCCCCTCTCCCTCCAGTGCGTTCTATCTCCCCCACACCCACCCAAGCTTGGCGTAATC(SEQIDNO:33)3型:ZBTB20-E1CLSAMP-E4-E5-E6-E7CAACGCAGAGTACATGGGACACAACATCAAGAGCAGGAAAATGGACTCATTAGGGAGGCAGGCAGTCATTACCACTCACACTGTACTTCCAGGGAGACACCGATTATAAGAAGAGAAACTCAGCGCTGGGGAAGAAGGAAGGGAATTTGAAGGAGAAGAAGAATATCTGGAGATCCTTGGCATCACCAGGGAGCAGTCAGGCAAATATGAGTGCAAAGCTGCCAACGAGGTCTCCTCGGCGGATGTCAAACAAGTCAAGGTCACTGTGAACTATCCTCCCACTATCACAGAATCCAAGAGCAATGAAGCCACCACAGGACGACAAGCTTCACTCAAATGTGAGGCCTCGGCAGTGCCTGCACCTGACTTTGAGTGGTACCGGGATGACACTAGGATAAATAGTGCCAATGGCCTTGAGATTAAGAGCACGGAGGGCCAGTCTTCCCTGACGGTGACCAACGTCACTGAGGAGCACTACGGCAACTACACCTGTGTGGCTGCCAACAAGCTGGGGGTCACCAATGCCAGCCTAGTCCTTTTCAGACCTGGGTCGGTGAGAGGAATAAATGGATCCATCAGTCTGGCCGTACCACTGTGGCTGCTGGCAGCATCTCTGCTCTGCCTTCTCAGCAAATGTTAATAGAATAAAAATTTAAAAATAATTTAAAAAACACACAAAAATGCGTCACACAGAATACAGAGAGAGAGACAGAGAGAGAGAGAGAGAGAGAGAGATGGGGGAGACCGTTTATTTCACAACTTTGTGTGTTTATACATGAAGGGGGAAATAAGAAAGTGAAGAAGAAAATACAACATTTAAAACAATTTTACAGTCCATCATTAAAAATTTATGTATCATTCAGGATGGAGAAGGTTCTACTGGGATATGTTTATATCTACTAAGCAAATGTATGCTGTGTAAAGACTACACCACACTAAGGACATCTGGATGCTGTAAAAATAAGAGAAGAACCAGATGGATATTAAGCCCCCCAACACACACTTTATCCTTCCTTCCTTCATCTTTTTTCATCTGTGGGGAAGAAAATAAGGTCTTGCCTTTGGTGTTTATATTTCCATAACCTTTTAATTCTATTTTTCATTTGAGCTGACTTGTAGCCACTTCAGACTATCAATGGAATCTTATGTTGAGCCTTTCTCTGGCTTTCCTTCCTCCACTATCTCTCCAACTTTAGAGATCATCCCCTCTCCCTCCAGTGCGTTCTATCTCCCCCACACCCACCCAAGCTTGGCGTAATC(SEQIDNO:34)4型:ZBTB20-E1-E1A-E1B-LSAMP-E4ACATGGGGAAGTTTAGGAGTTGAGGAAAGAAGATTAAAGAGCGCGAGGAGATTTTATAGACCAGTGGAATACAGCCCTTGTGCATATGAAGATCAGGTGACAAGTTTGSTGCCTACCAGCCTCCACAGCAATATGCCCTTTCACGAGTCCCTATCGCCCAGGCTGGAGTGCAGTGGCGTGATCTCTGCTCACTGCAACCTCCGCCTCCCGGGTTCAAGTGATTCTCTTGCCTCAGCCTCCCGAGTAGCTGGGATTACAGGAAGGGAATTTGAAGGAGAAGAAGAATATCTGGAGATCCTTGGCATCACCAGGGAGCAGTCAGGCAAAAGCTTGGCGTAATC(SEQIDNO:35)5型:ZBTB20-E1*A-LSAMP-E4ACATGGGGGAGGAAAGAAGATTAAAGAGCGCGAGGAGATTTTATAGACCAGTGGAATACAGGCCTTGTGCATATGAAGATCAGGTGACAAGTTTGCTGCCTACCAGCCTCCACAGCAATATGCCCTTTCACGGAAGGGAATTTGAAGGAGAAGAAGAATATCTGGAGATCCTTGGCATCACCAGGGAGCAGTCAGGCAAAAGCTTGGCGTAATC(SEQIDNO:36)6型:ZBTB20-E1-E1B-LSAMP-E4ACATGGGGGAGGAAAGAAGATTAAAGAGCGCGAGGAGACAGAGTCCCTATCGCCCAGGCTGGAGTGCAGTGGCGTGATCTCTGCTCACTGCAACCTCCGCCTCCCGGGTTCAAGTGATTCTCTTGCCTCAGCCTCCCGAGTAGCTGGGATTACAGGAAGGGAATTTGAAGGAGAAGAAGAATATCTGGAGATCCTTGGCATCACCAGGGAGCAGTCAGGCAAAAG(SEQIDNO:37)7型:ZBTB20-E1-LSAMP-E4ACATGGGGAAGTTTAGGAGTTGAGGAAAGAAGATTAAAGAGCGCGAGGAGGAAGGGAATTTGAAGGAGAAGAAGAATATCTGGAGATCCTTGGCATCACCAGGGAGCAGTCAGGCAAAAGCTTGGSGTAATC(SEQIDNO:38)8型:ZBTB20-E1-LSAMP-E3*-E4ACATGGGGGGGCGGGGGGAAGTTTAGGAGTTGAGGAAAGAAGATTAAAGAGCGCGAGGAGATTAACTTACTCTTAATGATCTTCCAACACTTGAGAAGGTCAGTAGCCCTCCATCTGTCATTCTCCAAGTTCACCAACAGCTTATCCACCCATCAAAGGTGCTTTTGTAACAAAATCCATGCATAATGAAACCAAGAAAGGAAGGGAATTTGAAGGAGAAGAAGAATATCTGGAGATCCTTGGCATCACCAGGGAGCAGTCAGGCAAAAGCTTGGCGTAATC(SEQIDNO:39)9型:ZBTB20-E1CLSAMP-E4ACATGGGGAAGAGCAGGAAAATGGACTCATTAGGGAGGCAGGCAGTCATTACCACTCACACTGTACTTCCAGGGAGACACCGATTATAAGAAGAGAAACTCAGCGCTGGGGAAGAAGGAAGGGAATTTGAAGGAGAAGAAGAATATCTGGAGATCCTTGGCATCACCAGGGAGCAGTCAGGCAAAAGCTTGGCGTAATC(SEQIDNO:40)10型:ZBTB20-E1C-LSAMP-E3*-E4ACATGGGGAGTACATGGGGATATACACAACATCAAGAGCAGGAAAATGGACTCATTAGGGAGGCAGGCAGTCATTACCACTCACACTGTACTTCCAGGGAGACACCGATTATAAGAAGAGAAACTCAGCGCTGGGGAAGAAGATTAACTTACTCTTAATGATCTTCCAACACTTGAGAAGGTCAGTAGCCCTCCATCTGTCATTCTCCAAGTTCACCAACAGCTTATCCACCCATCAAAGGTGCTTTTGTAACAAAATCCATGCATAATGAAACCAAGAAAGGAAGGGAATTTGAAGGAGAAGAAGAATATCTGGAGATCCTTGGCATCACCAGGGAGCAGTCAGGCAAAAGCTTGGCGTAATC(SEQIDNO:41)LPCS1:LSAMP-E0*-E1-E2-E3-E4GGAGGAGGATAGGAAGCAGGAAAGCGGGAGAGCTCGAGGGACAAGGGGGCTCGGTGTGTTTACACCAGGCACGGGCTACGAGCGTCCATCCCGGCCCCTGGCTTGCGCTCCCGAAGAGGAGAGCAAGGCTGTTCTGGGATCCGGCCGTCGTGCGGCAAGAGGCTTGTCTGTCCGGGTTGCCGGAACCAGGAGAACCCAGAGGGAAACCGAGGGAAAGGAGCGGCGCGTTTTACTAGAGAGAGCGCGAGCGGAAGAGGCGAGAGCAGGAGCGCGCGAGGGAGCATCGAGCGCAGCGGAGACATGAGGACCTACTGGCTGCACAGCGTCTGGGTGCTGGGCTTTTTCCTGTCCCTCTTCTCATTGCAAGGACTGCCTGTTCGCAGCGTGGATTTTAACCGAGGCACGGACAACATCACCGTGAGGCAGGGGGACACAGCCATCCTCAGGTGCGTTGTAGAAGACAAGAACTCAAAGGTGGCCTGGTTGAACCGTTCTGGCATCATTTTTGCTGGACATGACAAGTGGTCTCTGGACCCACGGGTTGAGCTGGAGAAACGCCATTCTCTGGAATACAGCCTCCGAATCCAGAAGGTGGATGTCTATGATGAGGGTTCCTACACTTGCTCAGTTCAGACACAGCATGAGCCCAAGACCTCCCAAGTTTACTTGATCGTACAAGTCCCACCAAAGATCTCCAATATCTCCTCGGATGTCACTGTGAATGAGGGCAGCAACGTGACTCTGGTCTGCATGGCCAATGGCCGTCCTGAACCTGTTATCACCTGGAGACACCTTACACCAACTGGAAGGGAATTTGAAGGAGAAGAAGAATATCTGGAGATCCTTGGCATCACCAGGGAGCAGTCAGGCAAAAGCTTGGCGTAATCC(SEQIDNO:46)所有cDNA序列均不同于野生型LSAMP和ZBTB20序列。10个融合转录物的7个包括ZBTB20的外显子1和LSAMP的外显子4之间的融合。图5。其余3个融合转录物包括ZBTB20的外显子1和LSAMP的外显子3*之间的融合。图5。外显子3*表示之前未被诠释且与ZBTB20和LSAMP基因的基因组重排有关的LSAMP基因座的一个新的外显子序列。外显子3*的核苷酸序列对应于SEQIDNO:45。可读框(ORF)搜索预测LSAMP蛋白的若干种N端截短或LSAMPcDNA中ORF不存在。本领域普通技术人员可设计引物用于扩增这些ZBTB20/LSAMP基因融合转录物。在一个实施方案中,正向引物被设计来与ZBTB20的外显子1的区域(例如E1、E1A、E1B或E1C)杂交,反向引物被设计来与LSAMP的外显子3*或外显子4杂交。在另一个实施方案中,引物被设计来扩增包含来自ZBTB20的外显子1的第1区(例如E1、E1A、E1B或E1C)和来自LSAMP的外显子3*或外显子4的第2区的扩增产物,其中检出扩增产物表明ZBTB20/LSAMP基因融合转录物存在。在某些实施方案中,正向引物在高严格性条件下与ZBTB20基因的E1内的区域杂交,反向引物(例如SEQIDNO:44)在高严格性条件下与LSAMP的外显子4内的区域杂交。LSAMP的外显子4对应于SEQIDNO:10的核苷酸1022-1156。这些引物可用来扩增例如图5中的1、4、5、6、7和8型融合转录物。在其它实施方案中,正向引物在严格条件下与ZBTB20基因的E1C内的区域杂交,反向引物(例如SEQIDNO:44)在严格条件下与LSAMP的外显子4内的区域杂交。这些引物可用来扩增例如图5中的2、3、9和10型融合转录物。在某些实施方案中,反向引物(例如SEQIDNO:42或SEQIDNO:43)与LSAMP的外显子7内的区域或与之互补的序列杂交。由于外显子3*是与ZBTB20和LSAMP基因的基因组重排有关的特有的LSAMP外显子,因此还可能设计引物以扩增外显子3*的特有区域或跨越LSAMP的外显子3*和外显子4之间的连接处的特有区域,并因此可用于特异性地鉴定ZBTB20和LSAMP基因的基因组重排。LPCS1(SEQIDNO:46)表示LSAMP基因座的可变剪接cDNA并包括外显子0*,所述外显子0*是来自之前未被诠释且与ZBTB20和LSAMP基因的基因组重排有关的LSAMP基因座的一种新的外显子序列。外显子0*的核苷酸序列对应于SEQIDNO:47。由于外显子0*是与ZBTB20和LSAMP基因的基因组重排有关的特有的LSAMP外显子,因此本领域技术人员可设计引物以扩增外显子0*的特有区域或跨越LSAMP的外显子0*和外显子1之间的连接处的特有区域,并因此可用于特异性地鉴定ZBTB20和LSAMP基因的基因组重排。本文引用的所有专利、专利申请和发表的参考文献均通过引用以其整体结合到本文中。虽然参照其优选的实施方案特别显示和描述了本发明,但本领域技术人员应了解,可在不偏离随附权利要求书所包括的本发明的范围的情况下在其中进行形式和详细资料的各种改变。参考文献下列参考文献被引用于本申请中,并且提供有关本发明领域的总体信息以及提供本申请中论述的测定法和其它详细资料。下列参考文献通过引用以其整体结合到本文中。1.Siegel,R.;Naishadham,D.;Jemal,A.Cancerstatistics(癌症统计学).CACancerJ.Clin.2013,63,11-30。2.Chornokur,G.;Dalton,K.;Borysova,M.E.;Kumar,N.B.Disparitiesatpresentation,diagnosis,treatment,andsurvivalinAfricanAmericanmenaffectedbyprostatecancer(患有前列腺癌的非裔美国男性的表现、诊断、治疗和生存期的差异).Prostate2011,71,985-997。3.Schwartz,K.;Powell,I.J.;Underwood,W.,3rd;George,J.;Yee,C.;Banerjee,M.Interplayofrace,socioeconomicstatus,andtreatmentonsurvivalofpatientswithprostatecancer(人种、社会经济地位和治疗对前列腺癌患者的生存的相互作用).Urology2009,74,1296-1302。4.Major,J.M.;Oliver,M.N.;Doubeni,C.A.;Hollenbeck,A.R.;Graubard,B.I.;Sinha,R.Socioeconomicstatus,healthcaredensity,andriskofprostatecanceramongAfricanAmericanandCaucasianmeninalargeprospectivestudy(大规模前瞻性研究中非裔美国男性和高加索美国男性间的社会经济地位、医疗保健密度和前列腺癌风险).CancerCausesControl2012,23,1185-1191。5.Sridhar,G.;Masho,S.W.;Adera,T.;Ramakrishnan,V.;Roberts,J.D.DoAfricanAmericanmenhavelowersurvivalfromprostatecancercomparedwithWhitemen(非裔美国男性因前列腺癌的生存比白种人男性的低吗)?Ameta-analysis.Am.JMens.Health2010,4,189-206。6.Cullen,J.;Brassell,S.;Chen,Y.;Porter,C.;L’Esperance,J.;Brand,T.;McLeod,D.G.Racial/ethnicpatternsinprostatecanceroutcomesinanactivesurveillancecohort(积极监督组群中前列腺癌结局的种族/人种模式).ProstateCancer2011,2011,doi:10.1155/2011/234519。7.Berger,A.D.;Satagopan,J.;Lee,P.;Taneja,S.S.;Osman,I.Differencesinclinicopathologicfeaturesofprostatecancerbetweenblackandwhitepatientstreatedinthe1990sand2000s(在1990和2000年代治疗的黑人和白人患者之间前列腺癌的临床病理学特征的差异).Urology2006,67,120-124。8.Kheirandish,P.;Chinegwundoh,F.Ethnicdifferencesinprostatecancer(前列腺癌的种族差异).Br.J.Cancer2011,105,481-485。9.Odedina,F.T.;Akinremi,T.O.;Chinegwundoh,F.;Roberts,R.;Yu,D.;Reams,R.R.;Freedman,M.L.;Rivers,B.;Green,B.L.;Kumar,N.ProstatecancerdisparitiesinblackmenofAfricandescent:AcomparativeliteraturereviewofprostatecancerburdenamongblackmenintheUnitedStates,Caribbean,UnitedKingdom,andWestAfrica(非洲血统黑人男性的前列腺癌差异:美国、加勒比海、联合王国和西非的黑人男性间前列腺癌负荷的比较文献综述).Infect.AgentsCancer2009,4,doi:10.1186/1750-9378-4S1-S2。10.Heath,E.I.;Kattan,M.W.;Powell,I.J.;Sakr,W.;Brand,T.C.;Rybicki,B.A.;Thompson,I.M.;Aronson,W.J.;Terris,M.K.;Kane,C.J.等,Theeffectofrace/ethnicityontheaccuracyofthe2001PartinTablesforpredictingpathologicstageoflocalizedprostatecancer(人种/种族性对用于预测局限性前列腺癌的病理分期的2001Partin表的准确性的影响).Urology2008,71,151-155。11.Moul,J.W.;Sesterhenn,I.A.;Connelly,R.R.;Douglas,T.;Srivastava,S.;Mostofi,F.K.;McLeod,D.G.Prostate-specificantigenvaluesatthetimeofprostatecancerdiagnosisinAfrican-Americanmen(非裔美国男性中在前列腺癌诊断时的前列腺特异性抗原值).JAMA1995,274,1277-1281。12.Tewari,A.;Horninger,W.;Badani,K.K.;Hasan,M.;Coon,S.;Crawford,E.D.;Gamito,E.J.;Wei,J.;Taub,D.;Montie,J.等,Racialdifferencesinserumprostate-specific(PSA)doublingtime,histopathologicalvariablesandlong-termPSArecurrencebetweenAfrican-AmericanandwhiteAmericanmenundergoingradicalprostatectomyforclinicallylocalizedprostatecancer(进行临床局限性前列腺癌的根治性前列腺切除术的非裔美国男性和美国白种男性间血清前列腺特异性(PSA)倍增时间、组织病理学变量和长期PSA复发的人种差异).BJUInt.2005,96,29-33。13.Wallace,T.A.;Prueitt,R.L.;Yi,M.;Howe,T.M.;Gillespie,J.W.;Yfantis,H.G.;Stephens,R.M.;Caporaso,N.E.;Loffredo,C.A.;Ambs,S.TumorimmunobiologicaldifferencesinprostatecancerbetweenAfrican-AmericanandCaucasian-Americanmen(非裔美国男性和高加索美国美国男性间前列腺癌的肿瘤免疫生物学差异).CancerRes.2008,68,927-936。14.Prensner,J.R.;Rubin,M.A.;Wei,J.T.;Chinnaiyan,A.M.BeyondPSA:Thenextgenerationofprostatecancerbiomarkers(超出PSA:下一代的前列腺癌生物标志物).Sci.Transl.Med.2012,4,doi:10.1126/scitranslmed.3003180。15.Rubin,M.A.;Maher,C.A.;Chinnaiyan,A.M.Commongenerearrangementsinprostatecancer(前列腺癌中的共同基因重排).J.Clin.Oncol.2011,29,3659-3668。16.Sreenath,T.L.;Dobi,A.;Petrovics,G.;Srivastava,S.OncogenicactivationofERG:Apredominantmechanisminprostatecancer(ERG的致癌基因激活:前列腺癌的主要机制).J.Carcinog.2011,11,10-21。17.Petrovics,G.;Liu,A.;Shaheduzzaman,S.;Furasato,B.;Sun,C.;Chen,Y.;Nau,M.Ravindranath,L.;Chen,Y.;Dobi,A.等,FrequentoverexpressionofETS-relatedgene-1(ERG1)inprostatecancertranscriptome(前列腺癌转录物组中ETS相关基因-1(ERG1)的频繁过量表达).Oncogene2005,24,3847-3852。18.Tomlins,S.A.;Rhodes,D.R.;Perner,S.;Dhanasekaran,S.M.;Mehra,R.;Sun,X.W.;Varambally,S.;Cao,X.;Tchinda,J.;Kuefer,R.等,RecurrentfusionofTMPRSS2andETStranscriptionfactorgenesinprostatecancer(前列腺癌中TMPRSS2和ETS转录因子基因的反复融合).Science2005,310,644-648。19.Magi-Galluzzi,C.;Tsusuki,T.;Elson,P.;Simmerman,K.;LaFarque,C.;Esqueva,R.;Klein,E.;Rubin,M.A.;Zhou,M.TMPRSS2-ERGgenefusionprevalenceandclassaresignificantlydifferentinprostatecancerofCaucasian,African-AmericanandJapanesepatients(TMPRSS2-ERG基因融合发生率和类别在高加索美国、非裔美国和日本患者的前列腺癌中显著不同).Prostate2011,71,489-497。20.Rosen,P.;Pfister,D.;Young,D.;Petrovics,G.;Chen,Y.;Cullen,J.;Bohm,D.;Perner,S.;Dobi,A.;McLeod,D.G.等,DifferencesinfrequencyofERGoncoproteinexpressionbetweenindextumorsofCaucasianandAfricanAmericanpatientswithprostatecancer(患有前列腺癌的高加索美国患者和非裔美国患者在指标肿瘤间ERG癌蛋白质表达的频率的差异).Urology2012,80,749-753。21.Hu,Y.;Dobi,A.;Sreenath,T.;Cook,C.;Tadase,A.Y.;Ravindranath,L.;Cullen,J.;Furusato,B.;Chen,Y.;Thanqapazham,R.L.等,DelineationofTMPRSS2-ERGsplicevariantsinprostatecancer(前列腺癌中TMPRSS2-ERG剪接变体的描述).Clin.CancerRes.2008,14,4719-4725。22.GaryKGeiss,等(2008)Directmultiplexedmeasurementofgeneexpressionwithcolor-codedprobepairs(用彩色编码探针对的基因表达的直接多路复用测量),NatureBiotechnology26:317-25。23.PaoloFortinaandSaulSurrey,(2008)DigitalmRNAProfiling(数字式mRNA概况分析),NatureBiotechnology26:317-25。FarrellJ,PetrovicsG,McLeodDG,SrivastavaS.:GeneticandmoleculardifferencesinprostatecarcinogenesisbetweenAfricanAmericanandCaucasianAmericanmen(非裔美国男性和高加索美国美国男性间前列腺癌病变的遗传和分子差异).InternationalJournalofMolecularSciences.2013;14(8):15510-31。25.Rodriquez-Suarez等,Urineasasourceforclinicalproteomeanalysis:Fromdiscoverytoclinicalapplication(作为临床蛋白质组分析来源的尿液:从发现到临床应用),BiochimicaetBiophysicaActa(2013)。26.Shi等,Antibody-free,targetedmass-spectrometricapproachforquantificationofproteinsatlowpicogrampermilliliterlevelsinhumanplasma/serum(定量测定人血浆/血清中微微克/毫升的低水平蛋白质的无抗体定向质谱方法),PNAS,109(38):15395-15400(2012)。27.Elentiboba-Johnson和Lim,Fusionpeptidesfromoncogenicchimericproteinsasspecificbiomarkersofcancer(来自作为癌症的特异性生物标志物的致癌嵌合蛋白的融合肽),MolCellProteomics,12:2714(2013)。28.Ras/Raf/MEK/ERKandPI3K/PTEN/Akt/mTORCascadeInhibitors:HowMutationsCanResultinTherapyResistanceandHowtoOvercomeResistance(Ras/Raf/MEK/ERK和PI3K/PTEN/Akt/mTOR级联抑制剂:突变如何导致疗法抗性和如何克服抗性),Oncotarget,3(10):1068-1111(2012)。29.Kuhn等,High-resolutiongenomicprofilingofadultandpediatriccore-bindingfactoracutemyeloidleukemiarevealsnewrecurrentgenomicalterations(成人和儿童核心结合因子急性髓系白血病的高分辨基因组概况分析显示新的反复基因组变化),Blood,119(10):e67(2012)。30.Pasic等,RecurrentFocalCopyNumberChangesandLossofHeterozygosityImplicateTwoNon-CodingRNAsandOneTumorSuppressorGeneatChromosome3q13.31inOsteosarcoma(反复病灶拷贝数变化和杂合性丢失涉及骨肉瘤中染色体3q13.31的2种非编码RNA和1种肿瘤抑制物基因),CancerResearch,70(1):160-71(2010)。31.Chen等,Thet(1;3)breakpoint-spanninggenesLSAMPandNORE1areinvolvedinclearcellrenalcellcarcinomas(t(1;3)断点跨越基因LSAMP和NORE1参与透明细胞肾细胞癌),CancerCell,4:405-413(2003)。32.Ntougkos等,ClinCancerRes,11:5764-5768(2005)。33.Huang等,EurJCancer49:3729-37(2013)。34.Mao等,CancerRes,70:5207-5212(2010)。35.Blattner等,Neoplasia16(1):14-20(2014)。36.Khani等,ClinCancerRes20(18):4925-34(2014)。当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1