一种米尔贝霉素正调控基因milR及其过表达基因工程菌、制备方法和应用与流程
本发明涉及一种米尔贝霉素正调控基因milR及其过表达基因工程菌、制备方法和应用
背景技术:
:
米尔贝霉素属于十六元大环内酯化合物,与阿维菌素具有相似的化学结构。米尔贝霉素首先被日本Sankyo公司1967年发现,由土壤放线菌Streptomyces hygroscopicus subsp.aureolacrimosus发酵液产生。米尔贝霉素组分A3和A4的混合物比目前使用最广泛的高效生物农药阿维菌素杀螨活性还要高,对大鼠的毒性比阿维菌素低40倍。因此,被美国环保署认定为危险性小的杀虫剂,荷兰批准成为“GNO”(作物生产中的天然产物),属生态友好型农药,适用于有机农业虫害综合防治,在发达国家中已成为一种受欢迎的杀虫杀螨剂,被认为是当今世界上最优良的杀螨剂,具有良好的应用价值和广阔的市场。现已在美国、日本等43个国家和地区登记,在苹果、柑橘、草莓、茶叶等24种植物上用于防治对阿维菌素、有机磷农药产生抗性的螨、斑潜蝇、蚜虫、粉虱。在米尔贝霉素系列药物中,由于米尔贝霉素A3和A4具有较强的杀虫活性,因此米尔贝霉素产品的商业化大多围绕着米尔贝霉素米尔贝霉素A3/A4而进行。除米尔贝霉素A3和A4的混合物被广泛用作杀螨外,米尔贝霉素A3/A4的肟化物米尔贝肟(Milbemycin oxime)是欧美评价最安全的抗寄生虫药物,在欧美市场抗寄生虫药物销售额排名第2位。此外,米尔贝霉素半合成乐平霉素(Lepimectin)在日本登记用于防治蔬菜、水果的鳞翅目、半翅类等害虫;半合成Latidectin也商品化用于抗动物寄生虫。
尽管米尔贝霉素系列产品有巨大的商业价值,世界各国也争相研发产米尔贝霉素菌株,但都没有成功,近半个世纪来全球现仅日本Sankyo公司垄断生产米尔贝霉素原药。1999年课题组筛选出了产米尔贝霉素新菌株(授权发明专利4项:CN101100651B、CN10046743C、CN100467472C、CN100490648C),新菌株不仅生物合成米尔贝霉素,而且生物合成12个未报道的米尔贝新化合物和1个新聚醚类化合物(日本用于产业化的米尔贝霉素菌株Streptomyces hygroscopicus subsp.aureolacrimosus未见报道生物合成聚醚类化合物)。通过对新菌株的形态特征、培养特征、生理生化特征、细胞壁化学成分及16S rDNA序列(DQ449953)分析鉴定,命名为冰城链霉菌(Streptomyces bingchenggensis)。这一菌株为我国工业化生产米尔贝霉素提供了前提基础。到目前为止,本实验室已经完成冰城链霉菌全基因组测序,确定了米尔贝霉素的生物合成基因簇,分析了米尔贝霉素生物合成途径(Wang X,Yan Y,Zhang B,An J,Wang J,Tian J,Jiang L,Chen Y,Huang S,Yin M et al.Genome sequence of the milbemycin-producing bacterium Streptomyces bingchenggensis.J Bacteriol.2010;192:4526-7)。通过基因敲除与互补、体外蛋白质表达与酶促分析、产物结构解析阐明了C5-O-甲基转移酶、C5-酮基还原酶、聚醚类化合物南昌霉素生物合成起始模块基因的功能,并构建了不产杂质5-甲氧基米尔贝霉素类和南昌霉素的优良、高产基因工程菌,70T发酵罐上单位产量达到2300±74g/ml,比野生菌株的27±12g/ml提高85倍(Zhang J,An J,Wang J,Yan Y,He H,Wang X,Xiang W.Genetic engineering of Streptomyces bingchenggensis to produce milbemycins A3/A4as main components and eliminate the biosynthesis of nanchangmycin.Appl Microbiol Biotechnol.2013;97:10091-101.)。进一步建立了冰城链霉菌基因组芯片分析技术,明确了冰城链霉菌在不同生长时期的基因表达情况(Wang X,Zhang B,Yan Y,An J,Zhang J,Liu C,Xiang W.Characterization and analysis of an industrial strain of Streptomyces bingchenggensis by genome sequencing and gene microarray.Genome.2013;56:677-689),为下一步研究冰城链霉菌米尔贝霉素生物合成代谢调控,开展冰城链霉菌的分子育种奠定了基础。
链霉菌中抗生素生物合成基因簇一般成簇存在,簇内通常具有数量不等的调控基因(正调控和负调控基因),而高表达簇内正调控基因是一种有效的提高抗生素产量的方法。在天蓝色链霉菌中,增加一个拷贝的正调控基因actII-orf4,可以使得放线紫红素产量提高30倍以上。在可可链霉菌中,增加一个拷贝的polR后,多氧霉素终产量提高2-3倍。而在龟裂链霉菌中,增加两个拷贝的otcR,并且用组成型启动子SF14进行启动,土霉素产量能够提高5-6倍。因此,在代谢工程育种中,在不影响细胞生长的前提下,最大量的进行正调控基因的过表达,被认为是一种有效的提高抗生素产量的方法。
本发明通过对米尔贝霉素生物合成基因簇中唯一正调控基因milR进行研究,阐明了其在米尔贝霉素生物合成中的功能,在此基础上,通过提高milR基因拷贝数,提高了米尔贝霉素产量,并因此构建了米尔贝霉素高产工程菌株。
技术实现要素:
本发明提供一种米尔贝霉素正调控基因milR、该基因的过表达基因工程菌、菌株的制备方法及应用该菌株生产米尔贝霉素的方法。
本发明提供的米尔贝霉素正调控基因milR,其序列如SEQ ID NO.1所示,该基因编码一个由964个氨基酸组成的蛋白质,其氨基酸序列如SEQ ID NO.2所示,正向调控米尔贝霉素产量。
本发明提供的高产米尔贝霉素基因工程菌过表达米尔贝霉素正调控基因milR,高产米尔贝霉素。所述米尔贝霉素正调控基因milR序列如SEQ ID NO.1所示,该基因编码一个由964个氨基酸组成的蛋白质,其氨基酸序列如SEQ ID NO.2所示,正向调控米尔贝霉素产量;该菌株表达体系中,载体为pSET152,宿主为冰城链霉菌BC-120-4。所述冰城链霉菌米尔贝霉素正调控基因milR来源于冰城链霉菌。所述冰城链霉菌为Streptomyces bingchengsis X-7、treptomyces bingchenggensis BC-109-6、Streptomyces bingchenggensis BC-101-4、Streptomyces bingchenggensis BC-102-26、Streptomyces bingchenggensis BC-103-46、Streptomyces bingchenggensis BC-104-28、Streptomyces bingchenggensis X-4、Streptomyces bingchenggensis BC-X-1、Streptomyces bingchenggensis BCJ13、Streptomyces bingchenggensis BCJ36、Streptomyces bingchenggensis BC-120-4、Streptomyces bingchenggensis BCJ60或Streptomyces bingchenggensis BCJ60/pCMF中的一种或二种以上。
本发明还提供上述高产米尔贝霉素基因工程菌的制备方法,包括以下步骤:
1)构建米尔贝霉素正调控基因milR过表达质粒,其含有完整的米尔贝霉素正调控基因milR编码序列和上游启动子序列;
2)将1)所得的米尔贝霉素正调控基因milR过表达质粒转化大肠杆菌宿主细胞,得到含有米尔贝霉素正调控基因milR过表达质粒的转化子;
3)以冰城链霉菌为受体,将步骤2)得到的转化子与冰城链霉菌进行接合转移,挑取具有安普霉素抗性的接合子,即得到目标菌株。
上述方法中所述米尔贝霉素正调控基因milR,其序列如SEQ ID NO.1所示,编码一个由964个氨基酸组成的蛋白质,其氨基酸序列如SEQ ID NO.2所示。
上述方法中所述步骤1)具体为:使用引物RCOMF(5’ACATATGCAACCTGTGGACCTCCGTGGCA 3’)和RCOMR(5’GCTCTAGACGAGACCGTCCCTATGTCCGATG 3’)从冰城链霉菌基因组上PCR扩增得到包含米尔贝霉素正调控基因milR完整编码区和自身启动子的片段。PCR反应体系:10×KOD Buffer 5μL,dNTPs(2mM)5μL,模板DNA(冰城链霉菌基因组)10pg-1000ng,上下游引物(RCOMF,RCOMR;10μM)各2μL,25mM MgSO4 2μL,KOD plus DNApolymerase(1U/μL)1μL,DMSO 3μL,ddH2O补足到50μL。反应条件:94℃4min,94℃1min,65℃30sec,68℃3min,31个循环,68℃10min。PCR产物切胶回收后,用XbaI进行酶切处理(酶切体系:milR片段1-2μg,10×M buffer 10μL,BSA 10μL,XbaI 3μL,ddH2O补足到100μL),然后连接入(连接体系:载体pSET152 10-20ng,片段milR 50-100ng,10×T4DNA ligase buffer 1μL,PEG 1μL,T4DNA ligase 0.8μL,ddH2O补足到10μL)经XbaI和EcoRV双酶切处理(酶切体系:质粒pSET152 1-2μg,10×M buffer 10μL,BSA 10μL,XbaI 3μL,EcoRV 3μL,ddH2O补足到100μL)的pSET152载体,将以上的连接产物在22℃水浴3h后转化到大肠杆菌DH5α中,37℃过夜培养后挑取单克隆,37℃小试管培养,提质粒后得到重组质粒pSET152::milR即为米尔贝霉素正调控基因milR过表达质粒;所述冰城链霉菌为Streptomyces bingchengsis X-7、Streptomyces bingchenggensis BC-109-6、Streptomyces bingchenggensis BC-101-4、Streptomyces bingchenggensis BC-102-26、Streptomyces bingchenggensis BC-103-46、Streptomyces bingchenggensis BC-104-28、Streptomyces bingchenggensis X-4、Streptomyces bingchenggensis BC-X-1、Streptomyces bingchenggensis BCJ13、Streptomyces bingchenggensis BCJ36、Streptomyces bingchenggensis BC-120-4、Streptomyces bingchenggensis BCJ60或Streptomyces bingchenggensis BCJ60/pCMF中的一种或二种以上。
本发明还提供利用上述高产米尔贝霉素基因工程菌发酵生产米尔贝霉素A3/A4的方法,包括培养产米尔贝霉素A3/A4的高产米尔贝霉素的基因工程菌株,从培养物中分离米尔贝霉素A3/A4。具体步骤如下:
1)构建米尔贝霉素正调控基因milR过表达质粒,其含有完整的米尔贝霉素正调控基因milR编码序列和上游启动子序列;
2)将1)所得的米尔贝霉素正调控基因milR过表达质粒转化大肠杆菌宿主细胞,得到含有米尔贝霉素正调控基因milR过表达质粒的转化子;
3)以冰城链霉菌为受体,将步骤2)得到的转化子与冰城链霉菌进行接合转移,挑取具有安普霉素抗性的接合子,即米尔贝霉素正调控基因milR过表达菌株;
4)以步骤3)得到的菌株发酵生产米尔贝霉素A3/A4。
上述步骤3)所述冰城链霉菌为Streptomyces bingchengsis X-7、Streptomyces bingchenggensis BC-109-6、Streptomyces bingchenggensis BC-101-4、Streptomyces bingchenggensis BC-102-26、Streptomyces bingchenggensis BC-103-46、StreptomyceS bingchenggensis BC-104-28、Streptomyces bingchenggensis X-4、Streptomyces bingchenggensis BC-X-1、Streptomyces bingchenggensis BCJ13、Streptomyces bingchenggensis BCJ36、Streptomyces bingchenggensis BC-120-4、Streptomyces bingchenggensis BCJ60或Streptomyces bingchenggensis BCJ60/pCMF中的一种或二种以上。
上述的米尔贝霉素正调控基因milR在冰城链霉菌基因组中对应于基因SBI_00734,对应的核苷酸序列为SEQ ID NO.1。MilR编码一个由964个氨基酸组成的蛋白质,其氨基酸序列为序列表中的SEQ ID NO.2。生物信息学分析显示其与已知的调控蛋白AveR(Genbank登录号BAA84600)一致性为49%,与PikD(Genbank登录号ALO10672)一致性为37%。通过基因功能研究表明milR破坏株失去了产米尔贝霉素的能力,而重新引入一个拷贝的milR可使米尔贝霉素重新产生,这表明该基因是一个正调控基因。同时将milR的一个拷贝导入出发菌株BC-120-4中,构建得到一个米尔贝霉素高产工程菌株,使得米尔贝霉素A3/A4产量由出发菌株BC-120-4产量的3833mg/l提高到4978mg/l,提高百分比约为30%。
本发明中涉及的Streptomyces bingchengsis X-7保藏编号为CGMCC1734,保藏日期为2006年6月12日,保藏机构为中国普通微生物菌种保藏管理中心(China General Microbiological Culture Collection Center);Streptomyces bingchenggensis,公开于Wang X-J,Yan Y-J,Zhang B,et al.Genome sequence of the milbemycin-producing bacterium Streptomyces bingchenggensis.Journal of Bacteriology 2010,192(17):4526-4527;Streptomyces bingchenggensis BC-109-6、Streptomyces bingchenggensis BC-101-4、Streptomyces bingchenggensis BC-102-26、Streptomyces bingchenggensis BC-103-46、Streptomyces bingchenggensis BC-104-28都公开于Wang X-J,Wang X-C,Xiang W-S.Improvement of milbemycin-producing Streptomyces bingchenggensis by rational screening of ultraviolet and chemically induced mutants.World J Microbiol Biotechnol 2009,25:1051-1056;Streptomyces bingchenggensis X-4公开于Zhang B-X,Zhang H,Wang X-J,et al.New milbemycins from mutant Streptomyces bingchenggensis X-4.J Antibiot 2011,64,753-756;Streptomyces bingchenggensis BC-X-1公开于Zhang B,Wang X,Xiang W.Optimization of fermentation medium for enhanced production of milbemycin by a mutant of Streptomyces bingchenggensis BC-X-1using response surface methodology.2011,10(37):7225-7235;Streptomyces bingchenggensis BCJ13、Streptomyces bingchenggensis BCJ36公开于Zhang J,An J,Wang J-J,et al.Genetic engineering of Streptomyces bingchenggensis to produce milbemycins A3/A4as main components and eliminate the biosynthesis of nanchangmycin.Appl Microbiol Biotechnol 2013,97:10091–10101;Streptomyces bingchenggensis BC-120-4、Streptomyces bingchenggensis BCJ60、Streptomyces bingchenggensis BCJ60/pCMF公开Wang H-Y,Zhang J,Zhang Y-J,et al.Combined application of plasma mutagenesis and gene engineering leads to 5-oxomilbemycins A3/A4as main components from Streptomyces bingchenggensis.Appl Microbiol Biotechnol 2014,98:9703–9712。
本发明的有益效果如下:
1.本发明通过同源重组双交换考察了milR的功能,并通过milR过表达提高了米尔贝霉素A3/A4的产量。
2.降低了生产成本,提高了经济效益。
附图说明
图1为卡那霉素抗性基因质粒pUC119::neo(a)、基因阻断用质粒pKC1139(b)和过表达用质粒pSET152(c)的物理谱图;
图2是milR阻断突变株构建示意图;
图3为milR基因过表达菌株构建示意图。
具体实施方式
下面用实施例来进一步说明本发明,但本发明并不受其限制。下列实施例中未注明具体条件的实验方法,通常按照常规条件,或按照制造厂商所建议的条件。
实施例1milR基因功能的研究
1、构建用于基因破坏的重组质粒
利用引物RD-LF(5’CGGAATTCCAGCGACACCATCACCGAGAACA3’,下划线为EcoRI位点)和RD-LR(5’CGGGGTACCGACTGAGCGAGTCGGAAATT3’,下划线为KpnI位点)从冰城链霉菌Streptomyces bingchengsis X-7基因组上PCR扩增得到milR基因同源重组左臂片段。PCR反应总体系为100μl,反应条件为:94℃,4min;94℃,1min,68℃,3min,31个循环;68℃,10min。PCR产物上样电泳后,回收长度为2.2kb的目的条带,然后回收片段再经EcoRI和KpnI酶切。
利用引物RD-RF(5’CGGGATCCCAGCAGTCGGCTCAGCAACG3’,下划线为BamHI位点)和RD-RR(5’GCTCTAGACTCGAAATCCTTCTGGGTCAGGT3’),下划线为XbaI位点)从冰城链霉菌Streptomyces bingchengsis X-7基因组上PCR扩增得到milR基因同源重组右臂片段。PCR反应总体系为100μl,反应条件为:94℃,4min;94℃,1min,68℃,3min,31个循环;68℃,10min。PCR产物上样电泳后,回收长度为2.2kb的目的条带,然后回收片段再经BamHI和XbaI酶切。
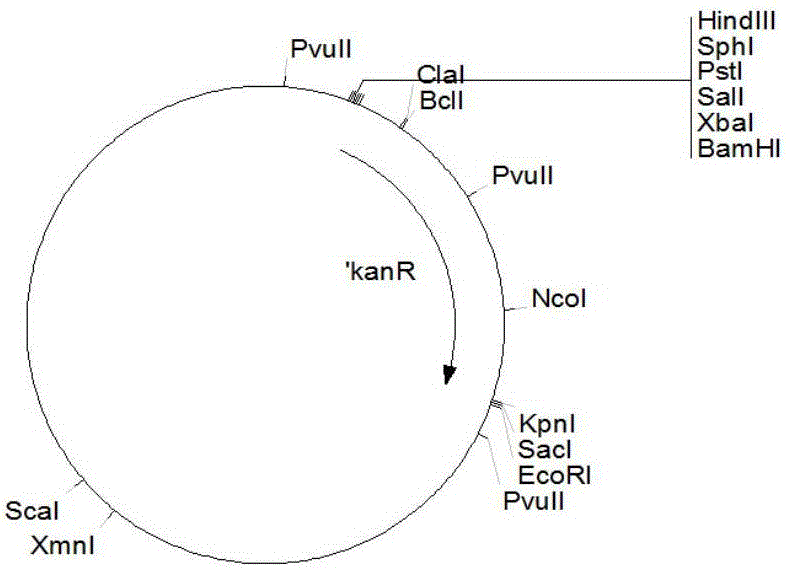
用BamHI和KpnI酶切质粒puc119::neo(该质粒的物理谱图如图1a),回收约1kb的卡那霉素抗性基因片段;将以上三个经过酶切的片段连接入经EcoRI和XbaI酶切的pKC1139(该质粒的物理谱图如图1b),得到milR敲除质粒pKC1139::milR::neo。
2、milR基因阻断突变株的构建
首先将质粒pKC1139::milR::neo转化E.coli ET12567/pUZ8002感受态细胞,进而通过接合转移转入冰城链霉菌BC-120-4。筛选具有安普霉素或卡那霉素抗性的接合转移子。将得到的接合子转接至含有卡那霉素和安普霉素的液体培养基SSPY中培养,经质粒提取和酶切验证的接合子挑取6个转接至含有卡那霉素的SSPY平板上,28℃培养7天,制备孢子悬液,以每个平皿约104个孢子的浓度涂布在含有卡那霉素的SSPY平板上,于37℃培养。pKC1139具有温度敏感型复制子,在高于34℃条件下培养不能自主复制,重组质粒只有与冰城链霉菌染色体DNA发生同源重组,才能在含有卡那霉素的MS培养基上生长。若发生双交换,则kanr正确地插入到靶基因内部,此时菌落表现为KanrAprs。将表现KanrAprs的菌落同时影印到SSPY/Kan和SSPY/Apr平板上,于28℃培养。随机挑选突变株,接种在没有任何选择压力的SSPY培养基上,28℃培养。转接3代后重新接种到分别含有卡那霉素和安普霉素的SSPY培养基上,结果仍然表现KanrAprs,说明通过双交换得到了遗传稳定的milR阻断突变株(图2)。
3、milR阻断突变株的验证
挑取6株以上的KanrAprs菌株,提取其基因组进行PCR验证。上游插入位置验证使用引物为conR1F(5’GCTCCACGGCTTCGATATGCG 3’)和conR1F(5’ATGCTCATTGACCGGAGCGCTCAC 3’),下游插入位置验证使用引物为conR1F(5‘AATGAATTCGAGCTCGGTACCCGAAC 3’)和conR1R(5’CGGCAATAGGTGATTCCAGGACAAC 3’)。PCR反应条件为94℃,4min;94℃,1min,64℃,35sec,68℃,3min,31个循环;68℃,10min。
发生双交换之后,上游插入位置验证用引物conR1F/R对扩增得到的理论大小的片段约为4.5kb,而野生型菌株大小应为5.3kb,下游插入位置验证用引物对conR2F/R扩增得到的理论大小片段约为3.5kb,而野生型则扩增不出条带。
4、米尔贝霉素的HPLC分析
将验证正确的milR阻断突变株和野生型分别接种在液体SSPY种子液中,28℃培养46h后,以6%的接种量在发酵培养基中进行发酵,培养9d后,发酵液和3倍体积甲醇混合浸泡12h,离心后取上清液进行HPLC分析。HPLC条件,流动相A:乙腈:水:甲醇=350:50:100,流动相B:甲醇,梯度洗脱:15min内流动相B的浓度从0升至100%,流速:1.0ml/min,柱温:25℃,检测波长:242nm,进样量:20μl,分析柱NOVA-PAKR C18(3.9×150mm,5μm,Waters)。结果表明milR阻断突变株中没有米尔贝霉素产生,说明milR基因是米尔贝霉素生物合成所必需的。
5、milR阻断突变株的互补分析
为了排除milR阻断突变株表型是由于染色体其它位置突变导致,进行了milR的遗传互补实验,使用引物RCOMF(5’ACATATGCAACCTGTGGACCTCCGTGGCA 3’)和RCOMR(5’GCTCTAGACGAGACCGTCCCTATGTCCGATG 3’)从冰城链霉菌Streptomyces bingchengsis X-7基因组上PCR扩增(PCR反应体系:10×KOD Buffer 5μL,dNTPs(2mM)5μL,模板DNA(冰城链霉菌Streptomyces bingchengsis X-7基因组)10pg-1000ng,上下游引物(RCOMF,RCOMR;10um)各2μL,25mM MgSO4 2μL,KOD plus DNA polymerase(1U/μL)1μL,DMSO 3μL,ddH2O补足到50μL。反应条件:94℃4min,94℃1min,65℃30sec,68℃3min,31个循环,68℃10min)得到包含milR完整编码区和自身启动子的3278bp片段。用XbaI进行酶切处理(酶切体系:milR片段1-2μg,10×M buffer 10μL,BSA 10μL,XbaI 3μL,ddH2O补足到100μL),然后连接(连接体系:载体pSET152 10-20ng,片段milR 50-100ng,10×T4DNA ligase buffer 1μL,PEG 1μL,T4DNA ligase 0.8μL,ddH2O补足到10μL)入经XbaI和EcoRV双酶切(酶切体系:质粒pSET152 1-2μg,10×M buffer 10μL,BSA 10μL,XbaI 3μL,EcoRV 3μL,ddH2O补足到100μL)处理的pSET152载体(pSET物理谱图见图1a)上(Bierman M,Logan R,O'Brien K,Seno ET,Rao RN,Schoner BE.Plasmid cloning vectors for the conjugal transfer of DNA from Escherichia coli to Streptomyces spp.Gene.1992;116:43-9.),(将以上的连接产物在22℃水浴3h后转化到大肠杆菌DH5α中,37℃过夜培养后挑取单克隆,37℃小试管培养,提质粒后)得到重组质粒pSET152::milR(图3)。
将pSET152::milR转化E.coli ET12567/pUZ8002感受态细胞,通过接合转移转入milR阻断突变株中。用安普霉素的抗性标记筛选转化子,pSET152为整合型载体,不能在链霉菌中自主复制,只能通过噬菌体ФC31attP位点与链霉菌基因组attB位点发生特异性重组而整合到染色体上。进而得到milR阻断突变株的互补株。随机筛选10株转化子,进行米尔贝霉素产素发酵,并进行HPLC分析,结果milR互补之后的突变株恢复了米尔贝霉素的产生能力。这说明milR是米尔贝霉素生物合成所必需的。
实施例2、米尔贝霉素高产工程菌株的构建
将实施例1中互补使用的重组载体pSET152::milR转化E.coli ET12567/pUZ8002感受态细胞,得到含有pSET152::milR的E.coli ET12567/pUZ8002单克隆,通过接合转移转入Streptomyces bingchenggensis BC-120-4菌株中,过程为挑取单克隆接种于加有相应抗性的液体LB培养基中(培养时添加100μg/ml安普霉素和25μg/ml氯霉素);37℃过夜培养;过夜培养物以1:100的比例,接种于加有相应抗生素的液体LB培养基中,37℃培养至OD600=0.4~0.6;离心,收集菌体,用等体积液体LB培养基洗涤菌体两次,用1/10体积的2YT悬浮;在洗涤大肠杆菌细胞的同时,用2×YT培养基收集链霉菌的孢子,每个接合转移约108个孢子/500μl,50℃热激10min,室温冷却;将500μl的大肠杆菌细胞与500μl链霉菌的孢子混合,3000rpm离心5min;弃上清,残留液重悬沉淀,涂布于含有10mM MgCl2的MS固体培养基上,28℃培养;培养6h后,每板加1ml无菌水稀释的萘啶酮酸(用0.15M NaOH配制)至25~30μg/ml和相应浓度抗生素(安普霉素至终浓度为4~6μg/ml),28℃继续培养3~5d。由此得到米尔贝霉素工程菌株。
从中筛选3株工程菌,进行发酵分析其米尔贝霉素产生能力,并与野生型菌株BC-120-4进行比较,结果表明3株工程菌米尔贝霉素A3/A4产量平均值为4978mg/l,出发菌株BC-120-4产量为3833mg/l,产量提高约30%。
实施例3
与实施例1的区别在于:将冰城链霉菌Streptomyces bingchengsis X-7替换为Streptomyces bingchenggensis BC-120-4。其他步骤同实施例1。
实施例4
与实施例2的区别在于:将实施例1中互补使用的重组载体pSET152::milR替换为实施例3中互补使用的重组载体pSET152::milR。其他步骤同实施例2,最终得到米尔贝霉素工程菌株。
- 还没有人留言评论。精彩留言会获得点赞!