一种与植物抗逆性相关蛋白GsNAC019及其编码基因与应用的制作方法
本发明属于生物
技术领域:
,具体涉及一种与植物抗逆性相关蛋白GsNAC019及其编码基因与应用。
背景技术:
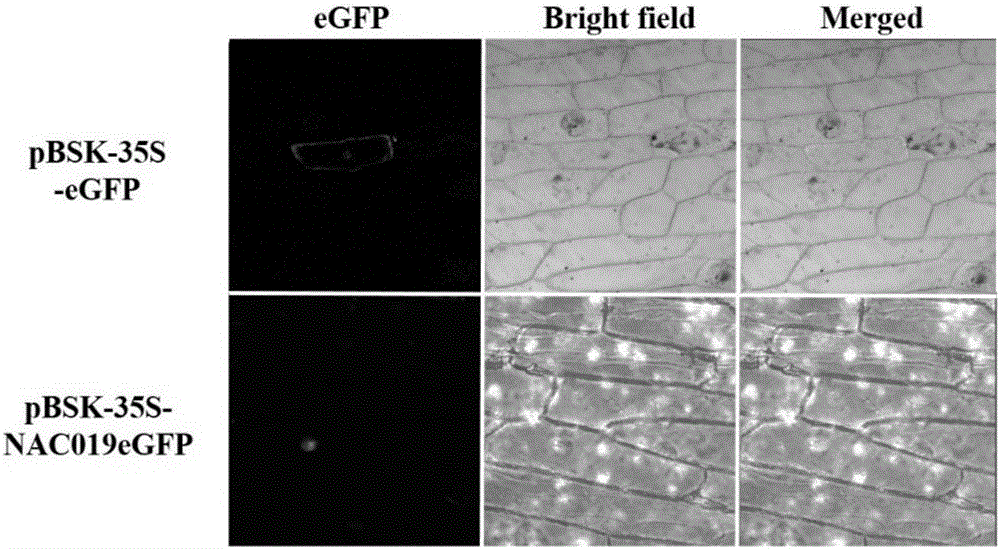
:盐碱逆境制约我国东北地区乃至全国的农业生产,其中,碱胁迫是影响植物生长、发育和地理分布的重要环境限制因素,严重影响作物的产量和品质。开发利用盐碱地,挖掘逆境农业生态区生产潜力,是保持我国农业持续高效发展、保证我国粮食安全亟待解决的重大问题。而迄今植物耐碱分子机制研究鲜有报道。大豆是我国尤其是黑龙江省的一种重要作物,既提供蛋白,又提供油料,其与固氮菌的共生关系使它成为轮作系统中的一种高利润作物。大豆在驯化过程中,栽培种丢失了很多与环境适应相关的重要基因,因此栽培大豆具有的遗传多样性比野生大豆低很多,比如很多栽培大豆对盐碱很敏感,而野生大豆则对盐碱有很强的适应性。所以将野生大豆中能够适应某一特定环境的基因重新引入到栽培大豆中,是大豆品种改良的快捷方法。这种将野生种优良性状基因转入到栽培种从而加速作物改良的策略已经在多种作物育种研究中获得成功。随着分子生物学的不断发展,利用日趋成熟的基因工程技术培育具有优良性状及良好耐逆功能的作物新品种已成为现代作物改良的重要手段之一。而转录因子在非生物胁迫信号传导中起到关键的调控作用,单基因的表达就可以启动信号传导网络,激活下游众多胁迫相关功能基因的转录和表达,从而达到更显著的改良作物抗逆性的效果。因此,挖掘抗逆转录因子基因,将为作物转基因育种提供功能更加显著的基因资源。技术实现要素:本发明所要解决的技术问题是如何调控植物抗逆性。为解决上述技术问题,本发明首先提供了与植物抗逆性相关蛋白,本发明所提供的与植物抗逆性相关蛋白的名称为GsNAC019蛋白质,GsNAC019蛋白质是如下a)或b)或c)的蛋白质:a)氨基酸序列是序列2所示的蛋白质;b)在序列2所示的蛋白质的N端和/或C端连接标签得到的融合蛋白质;c)将序列2所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加得到的具有相同功能的蛋白质。其中,序列2由343个氨基酸残基组成。为了使a)中的蛋白质便于纯化,可在序列表中序列2所示的蛋白质的氨基末端或羧基末端连接上如表1所示的标签。表1、标签的序列标签残基序列Poly-Arg5-6(通常为5个)RRRRRPoly-His2-10(通常为6个)HHHHHHFLAG8DYKDDDDKStrep-tagII8WSHPQFEKc-myc10EQKLISEEDL上述c)中的蛋白质,所述一个或几个氨基酸残基的取代和/或缺失和/或添加为不超过10个氨基酸残基的取代和/或缺失和/或添加。上述c)中的蛋白质可人工合成,也可先合成其编码基因,再进行生物表达得到。上述c)中的蛋白质的编码基因可通过将序列1第46-1077位所示的DNA序列中缺失一个或几个氨基酸残基的密码子,和/或进行一个或几个碱基对的错义突变,和/或在其5′端和/或3′端连上表1所示的标签的编码序列得到。为了解决上述技术问题,本发明还提供了与GsNAC019蛋白相关的生物材料。本发明提供的与GsNAC019蛋白相关的生物材料为下述A1)至A12)中的任一种:A1)编码GsNAC019蛋白质的核酸分子;A2)含有A1)所述核酸分子的表达盒;A3)含有A1)所述核酸分子的重组载体;A4)含有A2)所述表达盒的重组载体;A5)含有A1)所述核酸分子的重组微生物;A6)含有A2)所述表达盒的重组微生物;A7)含有A3)所述重组载体的重组微生物;A8)含有A4)所述重组载体的重组微生物;A9)含有A1)所述核酸分子的转基因植物细胞系;A10)含有A2)所述表达盒的转基因植物细胞系;A11)含有A3)所述重组载体的转基因植物细胞系;A12)含有A4)所述重组载体的转基因植物细胞系。上述相关生物材料中,A1)所述核酸分子为如下1)或2)或3)所示的基因:1)其编码序列是序列1第46-1077位所示的cDNA分子或基因组DNA分子;2)与1)限定的核苷酸序列具有75%或75%以上同一性,且编码GsNAC019蛋白质的cDNA分子或基因组DNA分子;3)在严格条件下与1)或2)限定的核苷酸序列杂交,且编码GsNAC019蛋白质的cDNA分子或基因组DNA分子。其中,所述核酸分子可以是DNA,如cDNA、基因组DNA或重组DNA;所述核酸分子也可以是RNA,如mRNA或hnRNA等。本领域普通技术人员可以很容易地采用已知的方法,例如定向进化和点突变的方法,对本发明的编码GsNAC019蛋白的核苷酸序列进行突变。那些经过人工修饰的,具有编码GsNAC019蛋白的核苷酸序列75%或者更高同一性的核苷酸,只要编码GsNAC019蛋白且具有相同功能,均是衍生于本发明的核苷酸序列并且等同于本发明的序列。这里使用的术语“同一性”指与天然核酸序列的序列相似性。“同一性”包括与本发明的编码序列2所示的氨基酸序列组成的蛋白质的核苷酸序列具有75%或更高,或85%或更高,或90%或更高,或95%或更高同一性的核苷酸序列。同一性可以用肉眼或计算机软件进行评价。使用计算机软件,两个或多个序列之间的同一性可以用百分比(%)表示,其可以用来评价相关序列之间的同一性。上述75%或75%以上同一性,可为80%、85%、90%或95%以上的同一性。上述生物材料中,所述严格条件是在2×SSC,0.1%SDS的溶液中,在68℃下杂交并洗膜2次,每次5min,又于0.5×SSC,0.1%SDS的溶液中,在68℃下杂交并洗膜2次,每次15min;或,0.1×SSPE(或0.1×SSC)、0.1%SDS的溶液中,65℃条件下杂交并洗膜。上述生物材料中,A2)所述的含有编码GsNAC019蛋白的核酸分子的表达盒(GsNAC019基因表达盒),是指能够在宿主细胞中表达GsNAC019蛋白的DNA,该DNA不但可包括启动GsNAC019转录的启动子,还可包括终止GsNAC019转录的终止子。进一步,所述表达盒还可包括增强子序列。可用于本发明的启动子包括但不限于:组成型启动子;组织、器官和发育特异的启动子及诱导型启动子。启动子的例子包括但不限于:花椰菜花叶病毒的组成型启动子35S:来自西红柿的创伤诱导型启动子,亮氨酸氨基肽酶("LAP",Chao等人(1999)PlantPhysiol120:979-992);来自烟草的化学诱导型启动子,发病机理相关1(PR1)(由水杨酸和BTH(苯并噻二唑-7-硫代羟酸S-甲酯)诱导);西红柿蛋白酶抑制剂II启动子(PIN2)或LAP启动子(均可用茉莉酮酸甲酯诱导);热休克启动子(美国专利5,187,267);四环素诱导型启动子(美国专利5,057,422);种子特异性启动子,如谷子种子特异性启动子pF128(CN101063139B(中国专利200710099169.7)),种子贮存蛋白质特异的启动子(例如,菜豆球蛋白、napin,oleosin和大豆betaconglycin的启动子(Beachy等人(1985)EMBOJ.4:3047-3053))。它们可单独使用或与其它的植物启动子结合使用。此处引用的所有参考文献均全文引用。合适的转录终止子包括但不限于:农杆菌胭脂碱合成酶终止子(NOS终止子)、花椰菜花叶病毒CaMV35S终止子、tml终止子、豌豆rbcSE9终止子和胭脂氨酸和章鱼氨酸合酶终止子(参见,例如:Odell等人(I985)Nature313:810;Rosenberg等人(1987)Gene,56:125;Guerineau等人(1991)Mol.Gen.Genet,262:141;Proudfoot(1991)Cell,64:671;Sanfacon等人GenesDev.,5:141;Mogen等人(1990)PlantCell,2:1261;Munroe等人(1990)Gene,91:151;Ballad等人(1989)NucleicAcidsRes.17:7891;Joshi等人(1987)NucleicAcidRes.,15:9627)。可用现有的表达载体构建含有所述GsNAC019基因表达盒的重组载体。所述植物表达载体包括双元农杆菌载体和可用于植物微弹轰击的载体等。如pAHC25、pBin438、pCAMBIA1302、pCAMBIA2301、pCAMBIA1301、pCAMBIA1300、pBI121、pCAMBIA1391-Xa或pCAMBIA1391-Xb(CAMBIA公司)等。所述植物表达载体还可包含外源基因的3′端非翻译区域,即包含聚腺苷酸信号和任何其它参与mRNA加工或基因表达的DNA片段。所述聚腺苷酸信号可引导聚腺苷酸加入到mRNA前体的3′端,如农杆菌冠瘿瘤诱导(Ti)质粒基因(如胭脂碱合成酶基因Nos)、植物基因(如大豆贮存蛋白基因)3′端转录的非翻译区均具有类似功能。使用本发明的基因构建植物表达载体时,还可使用增强子,包括翻译增强子或转录增强子,这些增强子区域可以是ATG起始密码子或邻接区域起始密码子等,但必需与编码序列的阅读框相同,以保证整个序列的正确翻译。所述翻译控制信号和起始密码子的来源是广泛的,可以是天然的,也可以是合成的。翻译起始区域可以来自转录起始区域或结构基因。为了便于对转基因植物细胞或植物进行鉴定及筛选,可对所用植物表达载体进行加工,如加入可在植物中表达的编码可产生颜色变化的酶或发光化合物的基因(GUS基因、萤光素酶基因等)、抗生素的标记基因(如赋予对卡那霉素和相关抗生素抗性的nptII基因,赋予对除草剂膦丝菌素抗性的bar基因,赋予对抗生素潮霉素抗性的hph基因,和赋予对氨甲喋呤抗性的dhfr基因,赋予对草甘磷抗性的EPSPS基因)或是抗化学试剂标记基因等(如抗除莠剂基因)、提供代谢甘露糖能力的甘露糖-6-磷酸异构酶基因。从转基因植物的安全性考虑,可不加任何选择性标记基因,直接以逆境筛选转化植株。上述生物材料中,所述载体可为质粒、黏粒、噬菌体或病毒载体。上述生物材料中,所述微生物可为酵母、细菌、藻或真菌,如农杆菌。上述生物材料中,所述转基因植物细胞系、转基因植物组织和转基因植物器官均不包括繁殖材料。为了解决上述技术问题,本发明还提供了GsNAC019蛋白或上述相关生物材料的新用途。本发明提供了GsNAC019蛋白或上述相关生物材料在调控植物抗逆性中的应用。本发明还提供了GsNAC019蛋白或上述相关生物材料在作为转录激活因子中的应用。本发明还提供了GsNAC019蛋白或上述相关生物材料在培育抗逆性转基因植物中的应用。上述应用中,所述调控为提高。上述应用中,所述抗逆性为抗碱胁迫;所述抗碱胁迫具体为抗NaHCO3胁迫,体现为在7mM或150mMNaHCO3胁迫的条件下:转基因植物的根长长于受体植物,或转基因植物的叶绿素含量高于受体植物,或转基因植物的存活率高于受体植物,或转基因植物的碱胁迫相关基因的表达量高于受体植物;所述碱胁迫相关基因具体为H+-ATPase基因和/或RD29A基因和/或KIN基因。为了解决上述技术问题,本发明最后提供了一种培育抗逆性提高的转基因植物的方法。本发明提供的培育抗逆性提高的转基因植物的方法包括在受体植物中过表达GsNAC019蛋白质,得到转基因植物的步骤,得到转基因植物的步骤;所述转基因植物的抗逆性高于所述受体植物。上述方法中,所述过表达的方法为将GsNAC019蛋白质的编码基因导入受体植物;所述GsNAC019蛋白质的编码基因的核苷酸序列是序列1所示的DNA分子。在本发明的实施例中,所述GsNAC019蛋白的编码基因(即序列表中序列1所示的DNA分子)是通过pCAMBIA330035Su载体导入受体植物,所述pCAMBIA330035Su载体为在pCAMBIA330035Su载体的PacⅠ和Nt.BbvCI两个酶切位点之间插入了如序列表中序列1所示的GsNAC019基因。pCAMBIA330035Su载体表达序列2所示的GsNAC019蛋白。上述方法中,所述抗逆性为抗碱胁迫。上述方法中,所述转基因植物的抗逆性高于所述受体植物体现在如下(1)-(4)中任一种:(1)转基因植物的根长长于受体植物;(2)转基因植物的叶绿素含量高于受体植物;(3)转基因植物的存活率高于受体植物;(4)转基因植物的碱胁迫相关基因的表达量高于受体植物;所述碱胁迫相关基因具体为H+-ATPase基因和/或RD29A基因和/或KIN基因。上述方法中,所述植物为单子叶植物或双子叶植物;所述双子叶植物具体可为豆科植物和/或十字花科植物和/或菊科植物;所述豆科植物可为大豆、百脉根、苜蓿或水黄皮;所述十字花科植物可为拟南芥或油菜;所述菊科植物可为向日葵;所述拟南芥可为拟南芥(哥伦比亚生态型col-0)。上述方法中,所述转基因植物理解为不仅包含将所述GsNAC019基因转化目的植物得到的第一代转基因植物,也包括其子代。对于转基因植物,可以在该物种中繁殖该基因,也可用常规育种技术将该基因转移进入相同物种的其它品种,特别包括商业品种中。所述转基因植物包括种子、愈伤组织、完整植株和细胞。扩增编码上述GsNAC019蛋白的核酸分子全长或其片段的引物对也属于本发明的保护范围。本发明从东北白城地区重度盐碱土地采集到了数百份东北野生大豆(GlycinesojaL.),其具有适应性广、抗逆性强等特点,其优质基因资源丰富,是抗逆基因克隆的理想材料,并从野生大豆中克隆到一种与植物抗逆性相关的GsNAC019基因。通过实验证明,将GsNAC019基因超表达于拟南芥中,得到的转GsNAC019拟南芥对碳酸盐胁迫的耐性明显高于野生型拟南芥,说明GsNAC019蛋白具有调控植物耐碱性的功能,可以为培育具有碳酸盐胁迫耐性的转基因植物的研究奠定基础。附图说明图1为野生大豆根和叶中的GsNAC019基因在50mMNaHCO3(pH8.5)和200mMNaCl胁迫处理下的表达模式。其中,图1a为野生大豆叶中的GsNAC019基因在50mMNaHCO3(pH8.5)胁迫处理下的表达模式;图1b为野生大豆根中的GsNAC019基因在50mMNaHCO3(pH8.5)胁迫处理下的表达模式;图1c为野生大豆根中的GsNAC019基因在200mMNaCl胁迫处理下的表达模式。图2为GsNAC019基因的亚细胞定位分析。图3为酵母细胞中GsNAC019基因的转录激活活性分析。图4为利用酵母单杂分析GsNAC019基因的结合元件。图5为转GsNAC019基因拟南芥在碱胁迫下的表型分析。图5a和图5b为转GsNAC019基因拟南芥在7mMNaHCO3处理下的幼苗期表型及根长测定;图5c、图5d和图5e为转GsNAC019基因拟南芥在150mMNaHCO3处理下的成苗期表型、叶绿素含量和存活率的统计分析。其中,6#、16#和22#均为T3代转GsNAC019基因拟南芥,WT为野生型拟南芥。图6为转GsNAC019基因拟南芥中各碱胁迫相关基因的相对表达量。具体实施方式下述实施例中所使用的实验方法如无特殊说明,均为常规方法。下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。下述实施例中的定量试验,均设置三次重复实验,结果取平均值。下述实施例中的野生大豆G07256种子在文献“MingzheSun,XiaoliSun,YangZhao,HuaCai,ChaoyueZhao,WeiJi,HuiziDuanMu,YangYu,YanmingZhu.EctopicexpressionofGsPPCK3andSCMRPinMedicagosativaenhancesplantalkalinestresstoleranceandmethioninecontent.PLOSONE2014,9(2):e89578”中公开过,公众可以从东北农业大学获得。下述实施例中的酿酒酵母(Saccharomycescerevisiae)AH109在文献“孙晓丽;段小红;才华;李勇;柏锡;纪巍;季佐军;朱延明利用酵母双杂交技术筛选与AtbZIP1相互作用的蛋白质。中国生物化学与分子生物学报,2010,26(11)1050-1058”中公开过,公众可以从东北农业大学获得。下述实施例中的酵母菌Y187在文献“LiangYang,WeiJi,YanmingZhu,PengGao,YongLi,HuaCai,XiBaiandDianjingGuo.GsCBRLK,acalcium/calmodulin-bindingreceptor-likekinase,isapositiveregulatorofplanttolerancetosaltandABAstress,journalofexperimentalbotany.201061(9)2519–2533,”中公开过,公众可以从东北农业大学获得。下述实施例中的野生型拟南芥(Arabidopsisthaliana)(Columbia-0亚型)在文献“KimH,HyunY,ParkJ,ParkM,KimM,KimH,LeeM,MoonJ,LeeI,KimJ.AgeneticlinkbetweencoldresponsesandfloweringtimethroughFVEinArabidopsisthaliana.NatureGenetics.2004,36:167-171”中公开过,公众可以从东北农业大学获得。下述实施例中的pGBKT7载体在文献“XiaoLuo,NaCui,YanmingZhu,LeiCao,HongZhai,HuaCai,WeiJi,XuedongWang,DanZhu,YongLi,XiBaiOver-expressionofGsZFP1,anABA-responsiveC2H2-typezincfingerproteinlackingaQALGGHmotif,reducesABAsensitivityanddecreasesstomatasize.JournalofPlantPhysiology169(12);1192-1202”中公开过,公众可以从东北农业大学获得。下述实施例中的pGBKT7-DREB载体在文献XiaoLuo,XiBai,XiaoliSun,DanZhu,BaohuiLiu,WeiJi,HuaCai,LeiCao,JingWu,MengranHu,XinLiu,LiliTangandYanmingZhu.ExpressionofwildsoybeanWRKY20inArabidopsisenhancesdroughttoleranceandregulatesABAsignaling.JournalofExperimentalBotany,2013,64(8).2155–2169”中公开过,公众可以从东北农业大学获得。下述实施例中的pGADT7载体在文献“LiangYang,WeiJi,YanmingZhu,PengGao,YongLi,HuaCai,XiBaiandDianjingGuo.GsCBRLK,acalcium/calmodulin-bindingreceptor-likekinase,isapositiveregulatorofplanttolerancetosaltandABAstress,journalofexperimentalbotany.201061(9)2519–2533,”中公开过,公众可以从东北农业大学获得。下述实施例中的pBSK-35S-eGFP载体在文献“XiaoliSun,WeiJi,XiaodongDing,XiBai,HuaCai,ShanshanYang,XueQian,MingzheSun,YanmingZhu.GsVAMP72,anovelGlycinesojaR-SNAREprotein,isinvolvedinregulatingplantsalttoleranceandABAsensitivity.PlantCellTissOrganCult2013,113:199–215”中公开过,公众可以从东北农业大学获得。下述实施例中的pCAMBIA330035Su载体在文献“XiaoliSun,WeiJi,XiaodongDing,XiBai,HuaCai,ShanshanYang,XueQian,MingzheSun,YanmingZhu.GsVAMP72,anovelGlycinesojaR-SNAREprotein,isinvolvedinregulatingplantsalttoleranceandABAsensitivity.PlantCellTissOrganCult2013,113:199–215”中公开过,公众可以从东北农业大学获得。下述实施例中的农杆菌LBA4404在文献“AilinLiu,YangYu,XiangboDuan,XiaoliSun,HuiziDuanmu,YanmingZhu.GsSKP21,aGlycinesojaSphasekinaseassociatedprotein,mediatestheregulationofplantalkalinetoleranceandABAsensitivity.PlantMolBiol(2015)87:111–124”中公开过,公众可以从东北农业大学获得。实施例1、大豆转录因子GsNAC019基因的克隆1、植物材料的处理挑选饱满的野生大豆G07256种子于浓H2SO4中处理10min以去除蜡质膜,倒净浓H2SO4,用无菌水冲洗3~4遍后放置于湿润的滤纸上,25℃暗培养3d催芽,待芽长到约1~2cm时,将其转移到盛有霍格兰培养液的钵中,用太空棉固定,使芽浸入培养液中,并将其放置于人工气候箱中培养。待幼苗长至3周龄,取其根部3cm放入EP管中,置于-80℃保存。2、RNA提取采用RNApreppure试剂盒(TRANSGENBIOTECH)提取上述步骤1获得的3周龄野生大豆幼苗的根的总RNA。3、cDNA的获得以上述步骤2获得的总RNA为模板,反转录得到cDNA。4、PCR扩增以上述步骤3或获得的cDNA为模板,采用Primer-KS和Primer-KAS引物进行PCR扩增,得到PCR扩增产物。引物序列如下:Primer-KS:5’-CAGTGGTTCTTTTCCTTCTGAGA-3’;Primer-KAS:5’-AAGAATCCCCACCTATTTACACC-3’。PCR扩增体系(50μl):cDNA4μl,10×PSbuffer(Mg2+)10μl,dNTPMixture(2.5mM)4μl,Primer-F1μl,Primer-R1μl,PrimeSTARDNAPolymerase(TaKaRa)0.5μl,ddH2O29.5μl。PCR扩增条件为:98℃8min;98℃10s,60℃10s,72℃1min,30个循环;72℃10min;4℃终止。将PCR扩增产物进行1.5%琼脂糖凝胶电泳检测,得到分子量略大于1Kb的条带,用琼脂糖凝胶回收试剂盒(TRANSGENBIOTECH)回收PCR扩增产物;将其与pEASY-BluntZero载体(TRANSGENBIOTECH)连接,得到重组质粒,将其命名为pEASY-BluntZero-GsNAC019,并将其转化大肠杆菌DH5α感受态细胞后送交测序。测序结果表明:PCR扩增得到大小为1145bp的扩增产物,其核苷酸序列如序列表中序列1所示,将其命名为GsNAC019基因,ORF为序列1的第46-1077位,GsNAC019基因编码的蛋白的氨基酸序列如序列表中序列2所示。实施例2、野生大豆转录因子GsNAC019基因的表达特性分析一、野生大豆根和叶中的GsNAC019基因在碱胁迫处理下不同时间的表达模式分析1、植物材料的处理挑选饱满的野生大豆G07256种子于浓H2SO4中处理10min以去除泥膜,倒净浓H2SO4,用无菌水冲洗3~4遍后放置于湿润的滤纸上,25℃暗培养3d催芽,待芽长到约1~2cm时,移出,用霍格兰液体培养基进行水培。待野生大豆幼苗长至3周龄,分别在50mMNaHCO3(pH8.5)和200mMNaCl条件下处理0h、1h、3h、6h、12h、24h后迅速剪取幼嫩的叶片和根,置于-80℃保存。2、总RNA的提取和cDNA的获得采用RNApreppure试剂盒(TRANSGENBIOTECH)分别提取上述步骤1获得的不同时间处理后的野生大豆幼苗的叶片和根的总RNA;并以获得总RNA为模板,反转录获得cDNA。3、Real-timePCR以上述步骤2获得的cDNA为模板,采用Primer-BS和Primer-BAS引物,通过Real-timePCR对GsNAC019基因进行表达量检测。引物序列如下所示:Primer-BS:5’-CTTCCCGAAAGAACACTGGC-3’;Primer-BAS:5’-CCGTTGCTGTATTGAGTGTGC-3’;Real-timePCR反应的条件:95℃7min→[95℃15s→60℃1min]×40→95℃1min→55℃1min→95℃30s。Real-timePCR采用比较CT法(ΔΔCT)计算基因表达量,以野生大豆GsGAPDH基因为内参基因,以未经处理的样品作为对照。目标基因表达差异通过经过处理的样本相对于每个时间点未经处理的样本的倍数来表示。每个样品包括3次生物学重复和3次技术重复,数据取3次生物学重复的平均值,如果有一个数值的偏差比较大则取两个数据的平均值。原始数据经标准化处理。标准化处理后的数据经T-test进行差异显著性分析。相对表达量计算方法:2-ΔΔCT=2-(ΔCT处理-ΔCT对照)=2-[(CT处理目的基因-CT处理内参基因)-(CT对照目的基因-CT对照内参基因)]。内参基因引物序列如下所示:GsGAPDH-S:5'-GACTGGTATGGCATTCCGTGT-3';GsGAPDH-AS:5'-GCCCTCTGATTCCTCCTTGA-3'。结果如图1所示:在未处理的野生大豆叶片中,GsNAC019基因表达量在12h上调,而在50mMNaHCO3处理下,GsNAC019基因在野生大豆叶中表达呈现先上调后下调再上调的趋势,并在1h和12h出现波峰,这说明碱胁迫处理使野生大豆提前表达该基因来响应碱胁迫,也说明碱胁迫中该基因的表达量呈现节律性变化。在碱胁迫处理的野生大豆根中GsNAC019基因表达量12h达到最大值,且各时间点相较于未处理组表达量较高,说明根中GsNAC019基因的表达明显受碱胁迫的诱导;而在200mMNaCl处理下,GsNAC019基因表达量表现出持续升高的趋势,这说明GsNAC019基因也响应盐胁迫,而且就同一基因而言,盐与碱胁迫对野生大豆的影响不尽相同。实施例3、基因枪轰击介导的GsNAC019基因瞬时表达与目的蛋白的亚细胞定位1、亚细胞定位载体的构建以野生大豆总cDNA为模板,采用GsNAC019-YS和GsNAC019-YAS引物进行PCR扩增,得到PCR扩增产物,即GsNAC019基因,引物序列如下所示(下划线标注引入的酶切位点序列,其左侧为保护碱基):GsNAC019-YS:5'-ACGCGTCGACATGGGAGTTCCAGAGAAAGACCC-3';GsNAC019-YAS:5'-AAAACTGCAGATTSCTGACCCGAACCCG-3';PCR反应的体系:20μL5×PrimeSTARTMHSPCR缓冲液,8μLdNTPmix(A、G、T、C、各2.5mM),2μL上下游引物(10μM),1μL稀释100倍(含目的基因的质粒)的通用模板,1μL高保真酶[PrimeSTARDNAPolymerase(TaKaRa)],无菌ddH2O补足体积(总体积100μL)。PCR反应条件:98℃8min;98℃10s,60℃10s,72℃1min,30个循环;72℃10m;4℃终止。用限制性内切酶SalI和PstI分别对GsNAC019基因和pBSK-35S-eGFP载体进行双酶切,连接,得到含GsNAC019基因的亚细胞定位载体。对含GsNAC019基因的亚细胞定位载体进行测序。测序结果表明:含GsNAC019基因的亚细胞定位载体为将pBSK-35S-eGFP载体的SalI和PstI酶切位点间的DNA片段替换为序列表中序列1所示的GsNAC019基因,保持pBSK-35S-eGFP载体的其他序列不变得到的载体。2、基因枪轰击介导的GsNAC019基因瞬时表达采用基因枪法将上述步骤1获得的含GsNAC019基因的亚细胞定位载体轰击洋葱表皮细胞(具体方法参见美国Bio-Rad伯乐Helios基因枪系统说明书),以pBSK-35S-eGFP空载体为阳性对照,剪取轰击过含GsNAC019基因的亚细胞定位载体和空载体的洋葱表皮细胞,装片,利用激光共聚焦显微镜下观察。结果如图2所示:GFP阳性对照在细胞内的细胞膜、细胞质和细胞核都有表达,整个细胞均能够检测到绿色荧光信号,而GsNAC019都主要定位在细胞核中。实施例4、野生大豆转录因子GsNAC019基因的转录激活活性分析1、含有GsNAC019基因或其结构域的重组载体的构建将序列表中序列1所示的GsNAC019基因替换pGBKT7载体的NdeⅠ和BamHⅠ酶切位点间的DNA片段,且保持pGBKT7载体的其他序列不变,得到pGBKT7-GsNAC019-1重组载体。将序列表中序列1的第1-39位所示的DNA分子替换pGBKT7载体的NdeⅠ和BamHⅠ酶切位点间的DNA片段,且保持pGBKT7载体的其他序列不变,得到pGBKT7-GsNAC019-2重组载体。将序列表中序列1的第40-1032位所示的DNA分子替换pGBKT7载体的NdeⅠ和BamHⅠ酶切位点间的DNA片段,且保持pGBKT7载体的其他序列不变,得到pGBKT7-GsNAC019-3重组载体。将序列表中序列1的第1-417位所示的DNA分子替换pGBKT7载体的NdeⅠ和BamHⅠ酶切位点间的DNA片段,且保持pGBKT7载体的其他序列不变,得到pGBKT7-GsNAC019-4重组载体。将序列表中序列1的第418-1032位所示的DNA分子替换pGBKT7载体的NdeⅠ和BamHⅠ酶切位点间的DNA片段,且保持pGBKT7载体的其他序列不变,得到pGBKT7-GsNAC019-5重组载体。2、重组菌的获得分别将步骤1获得的pGBKT7-GsNAC019-1重组载体,pGBKT7-GsNAC019-2重组载体、pGBKT7-GsNAC019-3重组载体、pGBKT7-GsNAC019-4重组载体、pGBKT7-GsNAC019-5重组载体转化酵母菌AH109,分别得到重组菌GsNAC019-1、重组菌GsNAC019-2、重组菌GsNAC019-3、重组菌GsNAC019-4和重组菌GsNAC019-5。将pGBKT7-DREB转化酵母菌AH109,得到阳性对照菌株;将pGBKT7空载体转化酵母菌AH109,得到阴性对照菌株。其中,酵母感受态细胞的制备(LiAc法)及小量LiAc/PEG法转化酵母感受态细胞的具体步骤参见《分子克隆实验指南》第三版及ClontechYeastProtocolsHandbook。3、β-半乳糖苷酶活性检测分别将重组菌GsNAC019-1、重组菌GsNAC019-2、重组菌GsNAC019-3、重组菌GsNAC019-4和重组菌GsNAC019-5用接种环接种在SD/-Trp固体培养基上划线,30℃培养3天后,将菌体影印到滤纸上,进行β-半乳糖苷酶活性检测。结果如图3所示:阳性对照菌株能使底物变蓝,而阴性对照菌株不能使底物变蓝。重组菌1和重组菌5均都能使底物变蓝,而重组菌GsNAC019-2、重组菌GsNAC019-3和重组菌GsNAC019-4均不能使底物变蓝。在SD/-Trp-Leu培养基上除阳性对照菌株、重组菌1和重组菌5外其他酵母菌均不生长,说明GsNAC019基因所表达蛋白和其全序列的C端具有自激活功能。推测其可能通过启动下游基因使植物响应碱胁迫。实施例5、GsNAC019与下游结合元件分析1、GsNAC019基因的获得以野生大豆总cDNA为模板,采用GsNAC019-JS和GsNAC019-JAS引物进行PCR扩增,得到大小为1032bp的PCR扩增产物,即为GsNAC019基因。GsNAC019-JS:5’-GGAATTCCATATGATGGGAGTTCCAGAGAAAGACCC-3’GsNAC019-JAS:5’-CGGGATCCTCAATTCTGACCCGAACCCG-3’2、pGADT7-GsNAC019的获得用限制性内切酶NdeⅠ和BamHⅠ分别对pGADT7载体和上述步骤1获得的PCR扩增产物进行双酶切,连接,得到pGADT7-GsNAC019重组载体,对pGADT7-GsNAC019重组载体进行测序验证。测序结果表明:pGADT7-GsNAC019重组载体为将pGADT7载体的NdeⅠ和BamHⅠ酶切位点间的DNA片段替换为序列表中序列1所示的GsNAC019基因,且保持pGADT7载体的其他序列不变得到的载体,表达GsNAC019蛋白。3、pHIS2-cis及pHIS2-mcis载体的获得将NAC-CIS-1结合元件的核苷酸序列替换pHIS2载体的EcoRⅠ和SacⅠ酶切位点间的DNA片段,且保持pHIS2载体的其他序列不变得到的载体,得到pHIS2-cis-1重组载体;将NAC-CIS-2结合元件的核苷酸序列替换pHIS2载体的EcoRⅠ和SacⅠ酶切位点间的DNA片段,且保持pHIS2载体的其他序列不变得到的载体,得到pHIS2-cis-2重组载体;将NAC-MCIS结合元件的核苷酸序列替换pHIS2载体的EcoRⅠ和SacⅠ酶切位点间的DNA片段,且保持pHIS2载体的其他序列不变得到的载体,得到pHIS2-cis-mcis重组载体;NAC-CIS-1结合元件的核苷酸序列为:CGTGTATACGTGTATACGTG;NAC-CIS-2结合元件的核苷酸序列为:CGTATTAACGTATTAACGTA;NAC-CIS-1结合元件的核苷酸序列为:GGGGTTAAGGGGTTAAGGGG。4、重组菌的获得将pGADT7-GsNAC019重组载体和pHIS2-cis-1重组载体共同转化酵母菌Y187,得到含质粒pGADT7-GsNAC019/pHIS2-cis-1的酵母菌Y187。将pGADT7-GsNAC019重组载体和pHIS2-cis-2重组载体共同转化酵母菌Y187,得到含质粒pGADT7-GsNAC019/pHIS2-cis-2的酵母菌Y187。将pGADT7-GsNAC019重组载体和pHIS2-cis-mcis重组载体共同转化酵母菌Y187,得到含质粒pGADT7-GsNAC019/pHIS2-cis-mcis的酵母菌Y187。将pGADT7载体和pHIS2载体共同转化酵母菌Y187,得到含质粒pGADT7/pHIS2的酵母菌Y187。5、结合活性检测分别用微量移液器吸取1ul的含质粒pGADT7-GsNAC019/pHIS2-cis-1的酵母菌Y187菌液、含质粒pGADT7-GsNAC019/pHIS2-cis-2的酵母菌Y187菌液、含质粒pGADT7-GsNAC019/pHIS2-cis-mcis的酵母菌Y187菌液和含质粒pGADT7/pHIS2的酵母菌Y187菌液在SD/-Trp/-Leu及SD/-Trp/-Leu/-His+50mM3-AT固体培养基上点点,以含质粒pGADT7/pHIS2的酵母菌Y187为阴性对照,若有转录因子与各元件结合并具有激活功能,则会表达HIS蛋白。30℃培养3天后,观察菌落是否生长。结果如图4所示:所有菌落均能在SD/-Trp/-Leu上生长,说明各组质粒均转化进入酵母菌Y187。阴性对照及含质粒pGADT7-GsNAC019/pHIS2-cis-1的酵母菌Y187不能在SD/-Trp/-Leu/-His+50mM3-AT的培养基上生长,说明GsNAC019不能与NAC-CIS-1结合元件结合。而含质粒pGADT7-GsNAC019/pHIS2-cis-2的酵母菌Y187能在SD/-Trp/-Leu/-His+50mM3-AT的培养基上生长,说明GsNAC019能与NAC-CIS-2结合元件结合。实施例6、转GsNAC019拟南芥植株的获得及其耐碱性分析一、转GsNAC019拟南芥植株的获得1、GsNAC019基因的获得以实施例1中的步骤4得到的pEASY-BluntZero-GsNAC019为模板,采用引物Primer-UA和Primer-UAS进行PCR扩增,得到PCR扩增产物,即GsNAC019基因。引物序列如下:Primer-UA:5’-GGCTTAAUCAGTGGTTCTTTTCCTTCTGAGA-3’;Primer-UAS:5’-GGTTTAAUAAGAATCCCCACCTATTTACACC-3’。PCR扩增体系(10μl):模板1μl,buffer1μl,dNTPMixture(2.5mM)0.4μl,Primer-UA0.5μl,Primer-UAS0.5μl,PfuCxDNAPolymerase(TaKaRa)0.2μl,ddH2O6.4μl。PCR扩增条件:95℃2min;95℃30s,55℃30s,72℃1min,30个循环;72℃10min;4℃终止反应。2、植物表达载体的获得用限制性内切酶PacⅠ和Nt.BbvCI对pCAMBIA330035Su载体进行酶切,将获得的载体酶切产物、USER酶(NEB,M5505S)和GsNAC019基因在37℃下孵育20min,利用USER酶对GsNAC019基因片段的尿嘧啶处进行切割,形成可与pCAMBIA330035Su载体双酶切后互补的粘性末端,得到的重组表达载体记作pCAMBIA330035Su-GsNAC019。将获得的重组表达载体pCAMBIA330035Su-GsNAC019在25℃下孵育20min,转化大肠杆菌感受态细胞DH5α(全式金,CD201-01),并送交测序。测序结果表明:在pCAMBIA330035Su载体的PacⅠ和Nt.BbvCI酶切位点之间插入了如序列表中序列1所示的GsNAC019基因。pCAMBIA330035Su载体表达序列2所示的GsNAC019蛋白。3、转化采用冻融法,将pCAMBIA330035Su-GsNAC019载体转化至根癌农杆菌LB4404,获得重组农杆菌pCAMBIA330035Su-GsNAC019/LB4404,将pCAMBIA330035Su转化至根癌农杆菌LB4404,获得重组农杆菌pCAMBIA330035Su/LB4404。经PCR鉴定得到阳性转化子(含有序列表中序列1所示的GsNAC019基因的转化子),用于侵染拟南芥植株。4、转GsNAC019拟南芥的获得将重组农杆菌pCAMBIA330035Su-GsNAC019/LB4404通过Floral-dip法侵染野生型拟南芥(哥伦比亚生态型col-0),将侵染过的拟南芥进行培养,得到T0代转GsNAC019拟南芥种子。将T0代转GsNAC019拟南芥种子表面消毒后,播种于含25mg/L固杀草(glufosinate-ammonium,Sigma,45520)的1/2MS培养基上筛选,得到T1代转GsNAC019拟南芥幼苗。如此重复,直至得到T3代转GsNAC019拟南芥纯合体株系。提取T3代转GsNAC019拟南芥幼苗的基因组RNA,进行RT-PCR鉴定。具体步骤如下:提取T3代转GsNAC019拟南芥植株的总RNA,反转录得到cDNA;以cDNA为模板,分别采用Primer-BS和Primer-BAS引物对、Actin2-S和Actin2-AS引物对,对GsNAC019基因进行表达量检测,得到PCR扩增产物。引物序列如下所示:Primer-BS:5’-CTTCCCGAAAGAACACTGGC-3’;Primer-BAS:5’-CCGTTGCTGTATTGAGTGTGC-3’;Actin2-S:5’-TTACCCGATGGGCAAGTC-3’;Actin2-AS:5’-GCTCATACGGTCAGCGATAC-3’。PCR反应的体系为:5μL2×EasyTaqDNAPolymerase,0.8μL上下游引物(10μM),1μL稀释100倍的cDNA,无菌ddH2O补足体积(总体积10μL)。PCR反应的条件:GsNAC019:94℃10min→[94℃30s→60℃30s→72℃90s]×30→72℃10min→4℃终止反应。Actin2:94℃10min→[94℃30s→60℃30s→72℃90s]×28→72℃10min→4℃终止反应。检测结果表明:野生型拟南芥植株的RT-PCR无扩增产物,而T3代转GsNAC019拟南芥纯合株系#6、T3代转GsNAC019拟南芥纯合株系#16和T3代转GsNAC019拟南芥纯合株系#22都可以扩增出大小为1032bp的目的条带,表明外源基因GsNAC019基因不但已顺利整合到拟南芥的基因组上,而且能够在转基因拟南芥中正常转录表达。选取T3代转GsNAC019拟南芥纯合株系6#、T3代转GsNAC019拟南芥纯合株系16#和T3代转GsNAC019拟南芥纯合株系22#用于下一步的表型分析。二、转GsNAC019拟南芥在碱胁迫下的表型分析1、转GsNAC019拟南芥在7mMNaHCO3胁迫下的幼苗期表型、根长选取饱满的野生型拟南芥(哥伦比亚生态型)、T3代转GsNAC019拟南芥纯合株系6#和T3代转GsNAC019拟南芥纯合株系16#种子,用5%次氯酸钠消毒液消毒10min后,于4℃春化3d,播种于1/2MS固体培养基上。1周后,挑选长势一致的转GsNAC019拟南芥和野生型拟南芥幼苗水平摆放于7mMNaHCO3胁迫培养基的平皿中,竖直生长,并以正常培养基培养的拟南芥幼苗作为对照。12d后观察碱胁迫处理组和对照组的拟南芥长势、根长,所有实验技术重复和生物学重复各3次。每次实验每个株系5株。结果如图5a和图5b所示:根据数据统计分析结果显示,在正常条件下,野生型平均根长为68.2mm,而T3代转GsNAC019拟南芥纯合株系6#的平均根长为64.8mm,T3代转GsNAC019拟南芥纯合株系16#的平均根长为67.9mm;在NaHCO3胁迫处理条件下,野生型的平均根长为34.0mm,T3代转GsNAC019拟南芥纯合株系6#的平均根长为44.0mm,T3代转GsNAC019拟南芥纯合株系16#的平均根长为47.6mm。上述结果表明:在正常条件下,T3代转GsNAC019拟南芥纯合株系6#和T3代转GsNAC019拟南芥纯合株系16#与野生型拟南芥的生长发育并没有显著差异,表明导入的GsNAC019基因并未在幼苗期对拟南芥的生长、发育造成影响;在NaHCO3胁迫处理条件下,转GsNAC019拟南芥与野生型拟南芥的生长都受到了抑制,但在7mMNaHCO3处理下,野生型生长抑制更明显,转GsNAC019拟南芥的根长明显长于野生型。因此超量表达GsNAC019基因提高了拟南芥在幼苗期对碱胁迫的耐性。2、转GsNAC019拟南芥在150mMNaHCO3处理下的成苗期表型、叶绿素含量及存活率选取饱满的野生型拟南芥(哥伦比亚生态型)、T3代转GsNAC019拟南芥纯合株系6#、T3代转GsNAC019拟南芥纯合株系16#和T3代转GsNAC019拟南芥纯合株系22#种子,4℃春化3d后,播种于营养钵中(营养土,君子兰土,蛭石按1:1:1混合),置于温室中培养(22℃,光照16h/d)。3周后,对于碱处理的苗每2~3d浇灌一次150mMNaHCO3溶液,并以未用NaHCO3溶液浇灌的正常条件下生长的拟南芥幼苗作为对照。15d后观察各碱处理组(150mMNaHCO3)和对照组的拟南芥长势,并测定各组叶绿素含量及存活率(检测方法参见文献“植物生理实验/郝再彬等主编哈尔滨工业大学出版社,2004.9”)。所有实验技术重复和生物学重复各3次。每次实验每个株系30株。150mMNaHCO3处理下转GsNAC019拟南芥与野生型拟南芥的长势情况如图5c所示:在正常条件下,T3代转GsNAC019拟南芥纯合株系6#、T3代转GsNAC019拟南芥纯合株系16#和T3代转GsNAC019拟南芥纯合株系22#与野生型拟南芥的成苗生长发育情况并没有显著差异,表明导入的GsNAC019基因并未在成苗期对拟南芥的生长、发育造成影响;对于浇灌150mMNaHCO3溶液的成苗,野生型大部分叶片发黄、卷曲最终死亡,但T3代转GsNAC019拟南芥纯合株系6#、T3代转GsNAC019拟南芥纯合株系16#和T3代转GsNAC019拟南芥纯合株系22#叶片发生轻度萎蔫,基本都完全存活并开始生殖生长。150mMNaHCO3处理下转GsNAC019拟南芥与野生型拟南芥的叶绿素含量和存活率如图5d和图5e所示:在正常条件下,T3代转GsNAC019拟南芥纯合株系6#、T3代转GsNAC019拟南芥纯合株系16#和T3代转GsNAC019拟南芥纯合株系22#与野生型拟南芥的叶绿素含量和存活率并没有显著差异,但在150mMNaHCO3条件下,T3代转GsNAC019拟南芥纯合株系6#、T3代转GsNAC019拟南芥纯合株系16#和T3代转GsNAC019拟南芥纯合株系22#与野生型拟南芥的叶绿素含量和存活率均明显高于野生型拟南芥。3、转GsNAC019拟南芥在50mMNaHCO3处理下的碱胁迫相关的marker基因的相对表达量分析将野生型拟南芥(哥伦比亚生态型)、T3代转GsNAC019拟南芥纯合株系6#、T3代转GsNAC019拟南芥纯合株系16#的种子播种于1/2MS固体培养基上,22℃光照培养10天,将其移出放入添加了50mMNaHCO3的1/2MS液体培养基中进行处理,在0h及6h采取处理及未处理的样品,提取全基因组RNA并进行反转录,得到各个时间点的cDNA,按照实施例6步骤一中的4的方法通过RT-PCR对碱胁迫相关的marker基因进行表达量检测。marker基因及内参基因ACTIN2引物如表3所示。表3、marker基因的引物序列Real-timePCR反应的条件:95℃7min→[95℃15s→60℃1min]×40→95℃1min→55℃1min→95℃30s。Real-timePCR采用比较CT法(ΔΔCT)计算基因表达量,以拟南芥ACTIN2基因为内参基因,以未经处理的样品作为对照。目标基因表达差异通过经过处理的样本相对于每个时间点未经处理的样本的倍数来表示。每个样品包括3次生物学重复和3次技术重复,数据取3次生物学重复的平均值,如果有一个数值的偏差比较大则取两个数据的平均值。原始数据经标准化处理。标准化处理后的数据经T-test进行差异显著性分析。相对表达量计算方法:2-ΔΔCT=2-(ΔCT处理-ΔCT对照)=2-[(CT处理目的基因-CT处理内参基因)-(CT对照目的基因-CT对照内参基因)]。A检测结果如图6所示。从图中可以看出:在转GsNAC019拟南芥中,超表达GsNAC019基因使耐碱相关基因H+-ATPase、RD29A和KIN发生上调表达而对NADP-ME、RD22和COR47基因的表达量影响不大。推测GsNAC019是通过上调H+-ATPase、RD29A和KIN等基因来提高植物耐碱性。当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1