水稻果胶裂解酶前体编码基因DEL1及其用途的制作方法
本发明属于植物基因工程领域。具体的说,本发明涉及一种利用图位克隆技术克隆水稻DEL1基因,以及利用转基因互补实验鉴定该基因的功能;同时还涉及利用该基因研究对水稻植株发育和叶片早衰的影响,为揭示水稻正常生长发育及调控水稻叶片的衰老形态提供了一定研究基础。
背景技术:
果胶是植物细胞壁的基本组成部分,是植物细胞壁功能和结构最为复杂的多糖。果胶是以原果胶、果胶、果胶酸的形态广泛分布于植物的根、茎、叶、果实中,能够与纤维素、半纤维素共同构成植物细胞壁的胞间层和初生细胞壁。果胶类多糖的结构十分复杂。根据其结构大致可以分为三个部分:同聚半乳糖醛酸、鼠李糖半乳糖醛酸聚糖I和取代型半乳糖醛酸聚糖。果胶裂解酶作为果胶降解酶之一,能够通过β-消除作用将去酯化的同聚半乳糖醛酸降解成一个短链的果胶分子和一个4,5-不饱和寡聚半乳糖醛酸。果胶裂解酶在微生物中的功能研究广泛,主要针对菊欧文式菌(Erwinia chrysanthemi)引起的软腐病。在植物中,自从1989年第一个果胶裂解酶基因在番茄中报道以来,相继又报道了许多果胶裂解酶基因的研究。从目前的研究来看,果胶裂解酶在植物中的功能包括以下几个方面:(1)花粉花药和雌蕊发育等方面。花粉的发育与细胞壁中果胶物质降解有一定关系。Wing等1989在番茄中首先发现果胶裂解酶基因影响花粉发育,番茄果胶裂解酶基因LAT56和LAT59在未成熟的花药中表达较低,在成熟的花药和花粉中表达水平最高,表明在花粉发育过程中需要果胶物质的降解。此后,在烟草、玉米、日本柳杉、日本雪松、苜蓿、鱼尾菊中相继发现果胶裂解酶影响花粉、花药和雌蕊方面的发育。(2)果实软化及成熟。有些果实在采摘后只能保存很短的时间,否则容易腐烂,主要是因为水果采摘后会经历一系列的变化,使其容易受病原菌入侵而导致腐烂。水果软化与其细胞壁的降解有很大关系,其中对细胞壁中果胶物质的修饰是水果软化和成熟的主要过程。对于果实成熟研究最开始也是在番茄中进行的,研究发现果皮中多聚半乳糖醛酸酶活性很高,人们一直认为该酶是果实软化和成熟的关键酶,然而对该酶过表达转基因植株研究表明,果实软化程度并没有提高。同时,在对草莓和香蕉软化研究中表明,多聚半乳糖醛酸酶的活性很低,因此认为该酶不是在果实软化过程中起作用。之后研究表明并确定果胶裂解酶在果实软化和成熟中起到非常重要的作用。(3)抗病方面。植物与病原菌互作是植物抗病研究中十分重要的组成部分。拟南芥抗白粉病途径主要有两条途径:一条是水杨酸途径,另一条是乙烯/茉莉酸途径。Vogel等2002从拟南芥中克隆了一个易感白粉病基因PMR6,该基因编码一个果胶裂解酶蛋白,当PMR6基因突变后导致了拟南芥增加了对白粉病的抗性,进一步研究表明PMR6基因突变后导致突变体中果胶含量升高,果胶的积累增强了细胞壁大分子间的结合密度,从而阻碍了病原菌的入侵,提高了对白粉病的抗性。pmr6的抗性机制与其它抗病基因有着很大的不同,它是通过对细胞壁结构的改变提高植物抗病性,是一种不依赖于SA或JA/ethylene的新防卫机制。
到目前为止,尽管许多植物中都报道了果胶裂解酶基因的功能,但在水稻中还鲜有报道。因此,挖掘果胶裂解酶基因突变体,将有助于我们阐明果胶裂解酶基因在植物发育过程中功能。
技术实现要素:
本发明要解决的技术问题是提供一种与水稻植株发育及叶片形态变异相关的蛋白质及其基因,以及由此获得的转基因植物细胞,和利用所述基因对水稻植株发育及叶片形态进行改造的方法。
为了解决上述技术问题,本发明提供一种水稻果胶裂解酶前体编码基因DEL1编码的蛋白质,该蛋白质为SEQ ID No:3所示的氨基酸序列。
作为本发明的水稻果胶裂解酶前体编码基因DEL1编码的蛋白质的改进,氨基酸序列还包括在SEQ ID No:3所示的氨基酸序列中添加、取代、插入或缺失一个或多个氨基酸或其他物种的同源序列而生成的氨基酸序列或衍生物。
本发明还同时提供了一种编码上述蛋白质的基因,该基因具有SEQ ID No:1、SEQ ID No:2所示的核苷酸序列。
备注说明:SEQ ID NO:1为cDNA全长,SEQ ID NO:2为gDNA全长。
作为本发明的基因的改进:所述核苷酸序列还包括在SEQ ID No:1、2所示的核苷酸序列中添加、取代,插入或缺失一个或多个核苷酸而生成的突变体、等位基因或衍生物。
本发明还同时提供了含有上述基因的质粒及含有上述基因的植物表达载体。
本发明还同时提供了一种宿主细胞,该宿主细胞含有上述基因序列。该宿主细胞为大肠秆菌细胞、农秆菌细胞或植物细胞。
本发明还同时提供了上述基因的用途,用于调控水稻植株正常生长发育及避免叶片的早衰。即,避免水稻出现矮秆和早衰。
作为本发明基因用途的改进:用于构建转基因水稻,所述转基因水稻的叶片早衰得以改良,从而用于高产品种的培育。
本发明还同时提供了一种改良水稻植株形态发育和叶片早衰的方法:包括用具有SEQ ID No:1、2所示的核苷酸序列的基因转化水稻细胞,再将转化后的水稻细胞培育成植株。
进一步具体说明:本发明的目的是提供一种从水稻矮秆早衰突变体中克隆的新基因DEL1,具有如SEQ ID No:1和SEQ ID No:2所示的DNA序列,也包括与SEQ ID No:1和SEQ ID No:2所示的DNA序列至少有70%同源性的基因序列。本发明中的SEQ ID No:3所示的蛋白质属于果胶裂解酶前体,其中进行一个或几个替换,插入或缺失所获得的功能类似物。另外,也包括在SEQ ID No:1和SEQ ID No:2中添加、取代、插入或缺失一个或多个核苷酸而生成的突变体、等位基因或衍生物,具有相同功能的序列也能达到本发明的目的。
本发明的另一个目的是提供一种用DEL1基因进行高效的植物转化的方法,具体地说,本发明提供了具有SEQ ID No:1和SEQ ID No:2所示的序列的基因或基因部分片段的载体,其中,如图5所示的pCAMBIA1300-DEL1,该载体可以表达有上述核苷酸序列编码的多肽或其同源类似物。
本发明还提供了一种利用植物表达载体转化植物细胞影响水稻植株发育和叶片早衰的方法。具体地是利用植物表达载体转化植物细胞以影响水稻植株发育和叶片早衰。
实现本发明的具体技术步骤如下:
一、水稻矮秆早衰突变体del1的分离和遗传分析:
本发明的水稻矮秆早衰突变体del1来自粳稻品种日本晴EMS(Ethyl Methyl Sulfonate)诱变产生的突变(图1,2)。del1突变体通过与野生型水稻的正、反交实验,证明该突变体受隐性单基因控制。
二、突变体del1活性氧水平增加
氮蓝四唑(NBT)和3,3'-二氨基联苯胺(DAB)染色是检测细胞中超氧阴离子自由基(O2·-)和过氧化氢(H2O2)含量的有效方法。从图3a我们看出,突变体del1叶片中蓝色明显增加,这说明突变体del1中超氧阴离子自由基在叶片中大量聚集,图3b中看出,突变体del1叶尖和叶边缘边缘出现褐色,说明叶尖和叶边缘过氧化氢发生积累。
三、DEL1基因的图位克隆:
(1)DEL1基因的初步定位:
为了分离DEL1基因,本发明首先组建了一个定位群体,由del1与籼稻品种TN1(Indica)杂交组配成F2定位群体,再通过图位克隆的方法,利用STS、SSR等分子标记对DEL1位点进行初步定位,将其初步定位在第10染色体的长臂端,并介于M1和M14两标记之间,见图4。
(2)DEL1基因的精细定位与预测:
通过对M1和M14两个标记之间的BAC序列分析,发展多态性标记将DEL1精确定位于BAC AC025905上M7与M8标记之间45-kb范围之内(图4),通过分析此区段开放阅读框(ORF)推测候选基因。
(3)DEL1基因的鉴定和功能分析:
为了验证候选基因功能,构建了如图5所示的互补载体,通过转基因技术,将互补载体转入到突变体del1中,结果表明本发明获得了使突变体恢复正常表型的转基因水稻(图6),证明本发明正确克隆了DEL1基因,氨基酸序列分析表明DEL1编码果胶裂解酶前体。我们还构建了如图7所示的亚细胞定位载体,通过水稻原生质体转化技术证明DEL1蛋白定位于细胞核、细胞质和细胞膜上(图8)。
综上所述,本发明利用水稻矮秆早衰突变体del1,通过图位克隆技术首次在水稻中克隆到了DEL1基因,该基因编码果胶裂解酶前体,在水稻中影响植株发育和叶片早衰。通过对DEL1基因的克隆和功能解读,进一步阐明了水稻植株发育和叶片早衰的遗传机制及其作用机理,为揭示水稻正常生长发育及调控水稻叶片的衰老形态提供了一定研究基础。
调控植株正常生长和叶片衰老是植物发育过程中十分重要的过程,尽管目前发现许多基因和途径参与其中,但是由果胶裂解酶基因突变导致植株发育异常和叶片早衰的现象还鲜有报道。本发明获得的矮秆早衰突变体,通过图位克隆技术本发明首次在水稻中克隆了相关基因DEL1,该基因编码果胶裂解酶前体基因,在水稻中影响植株正常发育及叶片的衰老。通过对DEL1基因的功能解读,将为深入研究植株形态调控和叶片早衰机制提供了一定研究基础。
附图说明
下面结合附图对本发明的具体实施方式作进一步详细说明。
图1为水稻野生型WT和矮秆早衰突变体del1各生长时期表型;
A:水稻野生型WT和矮秆早衰突变体del1苗期表型;
B:水稻野生型WT和矮秆早衰突变体del1成熟期表型;
C:水稻野生型WT和矮秆早衰突变体del1穗子表型;
D:水稻野生型WT和矮秆早衰突变体del1成熟期株高统计;
E:水稻野生型WT和矮秆早衰突变体del1各节间长度比较;
F:水稻野生型WT和矮秆早衰突变体del1各节间长度统计。
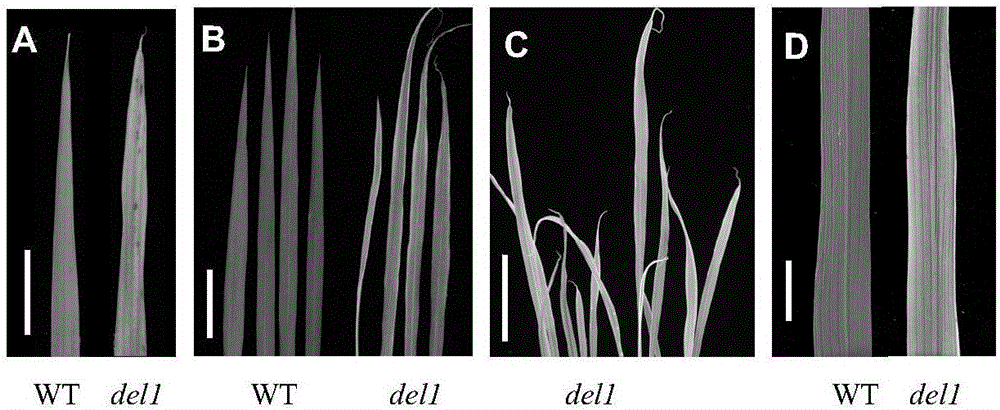
图2为水稻野生型WT和矮秆早衰突变体del1各时期叶片表型;
A:水稻野生型WT和矮秆早衰突变体del1苗期叶片表型;
B:水稻野生型WT和矮秆早衰突变体del1分蘖盛期叶片表型;
C:水稻矮秆早衰突变体del1分蘖盛期叶片表型;
D:水稻矮秆早衰突变体del1与野生型WT成熟期叶片表型。
图3为突变体del1及野生型WT叶片氮蓝四唑(NBT)和二氨基联苯胺(DAB)染色;
A:水稻矮秆早衰突变体del1与野生型WT叶片氮蓝四唑(NBT)染色;
B:水稻矮秆早衰突变体del1与野生型WT叶片二氨基联苯胺(DAB)染色。
图4为DEL1基因的定位图。
图5为pCAMBIA1300-DEL1载体图谱。
图6为功能互补实验转基因水稻的表型;
左图为:转基因功能互补载体恢复突变体植株高度;右图为:转基因功能互补载体恢复突变体叶片叶尖枯的早衰表型。
图7为DEL1与YFP融合表达载体示意图。
图8为荧光共聚焦显微镜观察转化DEL1亚细胞定位载体的水稻原生质体。
图9透射电镜分析突变体DEL1与对照日本晴叶绿体形态的变化。
具体实施方式
下面结合具体实施例对本发明进行进一步描述,但本发明的保护范围并不仅限于此。
实施例1
1、水稻材料:
水稻(Oryza sativa L.)突变体del1,原始野生材料为粳稻品种“日本晴”。
水稻矮秆早衰突变体del1来自日本晴EMS(Ethyl Methyl Sulfonate)诱变产生的突变(如图1,2所示),该突变体是在中国浙江省境内获得。
诱变方法为:
日本晴种子先在水中浸泡5小时,然后再浸渍到间断摇动的含有0.5%的化学诱变剂EMS的水溶液中8小时(p H调节至7),诱变处理结束后,再经流水冲洗种子12小时用于发芽。
突变体del1活性氧水平增加:
DAB染色:
(1)染色液的配置:称取适量DAB粉末,用蒸馏水稀释至1mg/ml。滴加浓盐酸至pH=3.8(此pH值下DNA溶解度最大)。4℃避光保存,使用前用1M NaOH溶液将pH调到5.8;
(2)染色:取同一部位野生型和突变体叶片叶片浸入DAB染色液中,抽真空10min,在28℃避光染色8h;
(3)脱色:将叶片从染色液中取出,转移到含有80%乙醇的玻璃试管中,沸水浴直至叶绿素完全脱去为止;
(4)拍照:将已脱色的材料转移到无水乙醇中,拍照或4℃保存。
NBT染色:
(1)染色液的配置:称取适量NBT粉末溶解于0.01M PBS(pH=7.6)缓冲液中至终浓度为0.5mg/ml;4℃避光保存;
(2)染色:取同一部位野生型和突变体叶片浸入NBT染色液中,抽真空10min,在28℃避光染色3h;
(3)脱色:将叶片从染色液中取出,转移到含有80%乙醇的玻璃试管中,沸水浴直至叶绿素完全脱去为止;
(4)拍照:将已脱色的材料转移到无水乙醇中,拍照或4℃保存。
上述氮蓝四唑(NBT)和3,3'-二氨基联苯胺(DAB)染色是检测细胞中超氧阴离子自由基(O2·-)和过氧化氢(H2O2)含量的有效方法。从图3a我们看出,突变体del1叶片中蓝色明显增加,这说明突变体del1中超氧阴离子自由基在叶片中大量聚集;图3b中看出,突变体del1叶尖和叶边缘出现褐色,说明叶尖和叶边缘过氧化氢发生积累;表明发生衰老。
2、分析和定位群体:
通过del1突变体与“TN1”的正反交实验,表明该突变体受隐性单基因控制。纯合的del1突变体和籼稻品种TN1进行杂交,F1代自交,并从F2群体中挑选出1081株同时具有矮秆和早衰表型的个体作为定位群体。每株取1克左右的嫩叶,用来提取总DNA。
说明:“早衰表型”是指叶片出现褐变黄化的现象表明叶片已经开始衰老,而比正常叶片出现褐变黄化早的植株,则表明其“衰老发生得早”,称之为“早衰”。
3、水稻基因组DNA提取
采用水稻微量DNA的快速提取方法从水稻叶片中提取用于基因定位的基因组DNA。取大约0.2g水稻叶片,经液氮冷冻,在直径5cm的小研钵中磨成粉状,转移到1.5ml离心管里提取DNA,获得的DNA沉淀溶解于150μl超纯水中。每一个PCR反应用2μl DNA样品。
4、DEL1基因的初步定位
从del1与TN1组合的F2群体中随机选取80个隐性个体,根据公布的粳稻和籼稻创建的分子遗传图谱,选取近似均匀分布于各条染色体上的SSR引物,根据已知的反应条件进行PCR扩增,经5%琼脂糖凝胶电泳分离和溴化乙啶(EB)染色,检测PCR产物的多态性,将DEL1初步定位在第10号染色体长臂端M1和M14两标记之间(如图4所示)。
5、DEL1基因的精细定位
选取del1与TN1组合的F2群体中共1081株隐性个体,在初定位的基础上进一步设计多态性标记,最终将DEL1精确定位于标记M7与M8之间,BAC号为AC025905上45-kb范围之内(图4)。引物序列如表1所示:
表1、DEL1基因的定位标记序列
6、基因预测和比较分析:
根据精细定位的结果,在45-kb范围内根据Rice Genome Annotation Project(http://rice.plantbiology.msu.edu/)的预测,发现在此区间内共有8个候选基因(图4)。根据两标记剩余的重组个体数,我们设计了各基因的测序引物,采用PCR方法分别从del1和野生型品种基因组中扩增出候选基因进行测序分析。发现del1突变体在LOC_Os10g31910的第3个外显子发生1个碱基的替换突变,由G突变为T,使对应的氨基酸由色氨酸突变为亮氨酸(图4)。将此用不同的突变单株及群体中突变表型的单株,各重复验证三次,突变位点稳定存在(测序引物序列见表2)。根据BAC克隆AC025905序列的基因注释信息(NCBI),推断此基因编码putative pectate lyase precursor(DEL1),该基因编码区全长5004bp,包含4个外显子和3个内含子。
该基因编码的蛋白质的氨基酸序列如序列表的SEQ ID No:3所示。该基因的cDNA全长如SEQ ID NO:1所示,gDNA全长如SEQ ID NO:2所示。
表2DEL基因的测序引物序列
实施例2
植物转化:
设计含有Kpn I及Kpn I和BamH I酶切位点两对引物Fw-DEL1-H1:GGTACCcgtcgctgtgccccagcagcatca,Re-DEL1-H1:GGATCCcacgttaaaattggaacgatgtga以及Fw-DEL1-H2:GGTACCtttcttatatttccatgtaact和Re-DEL1-H2:GGTACCtgccggactcggcgatgcaggt,以粳稻品种“日本晴”基因组DNA为模板,PCR分段扩增基因序列(SEQ ID No:2)、ATG前1998bp及TGA后841bp,共7843bp片段,并连接到pMD18-T上,经测序正确后,酶切、连接到pCAMBIA1300双元表达载体中,获得了互补载体pCAMBIA1300-DEL1。这个质粒通过电击的方法转入农秆菌(Agrobacterium tumefaciens)株系EHA105中转化水稻。我们利用突变体成熟种子诱导愈伤组织,经过诱导培养基培养3周后,挑选生长旺盛愈伤用作转化的受体。用含有双元质粒载体的EHA105菌株侵染水稻愈伤,在黑暗、25℃条件下共培养3天后,在含有300mg/L潮霉素的筛选培养基上培养。筛选抗性愈伤在含有250mg/L潮霉素预分化培养基上培养10天左右。将预分化的愈伤转至分化培养基上在光照条件下培养。一个月左右得到抗性转基因植株。对植株进行鉴定和连续的观察发现,与同时期的突变体比较,转基因植株表型与转入空载p1300的日本晴表型一致。
根据图6左图中的“del1”单株,出现叶尖枯萎、卷曲,即表明其已出现衰老特征。
因此,通过上述转基因技术,结果表明:本发明获得了使突变体恢复正常表型的转基因水稻(图6)。
说明:上文中所涉及的各种培养基的配方可参考Toki S.,Hara N.,Ono K.,Onodera H.,Tagiri A.,Oka S.,Tanaka H.(2006)Early infection of scutellum tissue with Agrobacterium allows high-speed transformation of rice.The Plant Journal 47:969-976。
实施例3
水稻原生质体的转化及DEL1黄色荧光融合蛋白的定位观察:
将DEL1基因cDNA序列克隆至pCAMBIA1301中构建YFP融合载体(图7)。
(1)生长15天左右的水稻幼苗用锋利的刀片在塑料培养皿板上切碎幼苗茎叶,移到200ml洗净的锥形瓶中(一般20ml酶解液每次60株左右;预先将酶解液倒入瓶中,根据瓶底大小合理分配酶解液),每瓶不宜装太多。放入28℃摇床,60-80rpm,4-6小时。
(2)在酶解时间完成之前,配置PEG4000 40%溶液,置于65℃水浴锅中或室温溶解,65℃溶解,中间取出来振荡一次。
(3)酶解完成后,先向酶解完成的瓶中加入约15ml左右的W5。用300目(或400目)的钢制滤网将碎叶片过滤掉,用干净的塑料培养皿收集酶解后的原生质体。然后将原生质体缓慢倒入(或用去枪尖的枪头吸取)50ml离心管中,天平称重平衡后,放入水平离心机,150g,5min,充分收集原生质体,离心完成后,缓慢吸走上清。
(4)向原生质体沉淀中加入1ml W5溶液,缓慢轻轻倾斜混匀重悬,然后用去枪尖的枪头吸移到2ml离心管中。此时50ml离心管中可能还有少量未重悬的原生质体,可再加入1ml W5溶解,吸移到之前的2ml离心管中,150g,3min,移除上清液。
(5)用MMG重悬,具体加多少根据原生质体的量和要转的质粒个数来定,一般最后每个质粒中加入100ul稀释的原生质体。或者用显微镜稍微观察下产量和酶解后完整个体的数目比例,然后根据原生质体的状态来确定MMG用量。
(6)准备转化用质粒(YFP融合载体)10-15ug左右或10ul,于2ml离心管中。
(7)加入100ul的重悬好的原生质体,然后加入PEG 40%110ul,混匀。
(8)28度避光静置15min。
(9)加入足量的W5稀释,混匀,然后离心150g,3min,缓慢移除上清,再用W5洗一次(中间会有损失,但不影响结果)。最后得到的沉淀,用W5重悬(用2ml的管子装满),轻轻混匀,移到细胞培养板中。用锡箔纸包裹,避光28度静置培养,14小时。
(10)培养时间完成后,将培养板各孔中沉淀的原生质体轻轻混匀,吸移到2ml管中,然后离心150g,3min,去除上清,保留100ul左右上清液,重悬原生质体。
(11)共聚焦显微镜观察拍照。
结果显示阳性对照在细胞各个部位均有表达,说明实验的表达系统工作正常,DEL1-GFP融合蛋白同样在细胞核质膜均有表达(图8);即,通过水稻原生质体转化技术证明DEL1蛋白定位于细胞核、细胞质和细胞膜上。
说明:上文中所涉及的各种溶液的配方可参考Zhang et al.A highly efficient rice green tissue protoplast system for transient gene expression and studying light chloroplast-related processes.Plant Methods 2011,7:30。
实施例4
叶绿体透射电镜的制备和观察:
(1)样品:取突变体苗期叶片和对照野生型苗期叶片,切成0.5~1mm3左右的小块;
(2)固定:将切好的样品块放入2ml离心管中,加入2.5%的戊二醛溶液(PH=7.2),在抽真空仪器中抽真空直到叶片完全下沉。0.1M磷酸漂洗三次,每15min一次,然后加入1%锇酸固定2~3小时,直至样品变黑;
(3)脱水:先用50%、70%、和90%的乙醇溶液依次脱水,每个浓度处理20分钟,再用乙醇和丙酮(1:1)溶液处理20分钟,以上均在4度冰箱内进行,最后将样品用纯丙酮室温处理20分钟;
(4)渗透:将样品在无水丙酮和包埋剂(3:1)混合液中作用4小时,再在无水丙酮和包埋剂(1:1)混合液处理3小时,最后在纯的包埋剂中作用12小时;
(5)包埋:将上述步骤中的样品挑的包埋盒内,37℃过夜,45℃处理12小时最后60℃处理24小时,获得包埋样品;
(6)切片、拍照:将包埋样品用超薄切片机切成60-70nm左右的超薄片,然后将切片用柠檬酸铅溶液染色10分钟,再有醋酸铀溶液染色30分钟,双蒸水清洗三次后晾干,用Hitachi H-7650型透射电镜观察并且选择清晰地倍数拍照。
结果显示野生型叶片的叶肉细胞中叶绿体数目较多,叶绿体结构完整,叶绿体中基粒片层层次丰富,排列紧密。突变体叶片出现叶绿体部分降解,类囊体排列紊乱,片层排列不规则等现象(图9)。观察结果表明目的基因的突变造成了叶绿体的发育异常。
最后,还需要注意的是,以上列举的仅是本发明的若干个具体实施例。显然,本发明不限于以上实施例,还可以有许多变形。本领域的普通技术人员能从本发明公开的内容直接导出或联想到的所有变形,均应认为是本发明的保护范围。
<110> 中国水稻研究所
<120> 水稻果胶裂解酶前体编码基因DEL1及其用途
<160> 3
<210> 1
<211> 1476
<212> DNA
<213> 水稻(Oryza sativa)cDNA全长
<400> 1
atggccgggc ccgtcgaatc cacccgcatt accctcctcc tcgtcgccgc caccctcctc 60
ctcctcccgc cgccgctcgc cgcctccctc aactcctccc tccccgaccc cgccgccgtc 120
gtcgccgact tccacagcaa ggtggcgacg tcgcggcggc ggatgcagaa ggccgacggc 180
ggcggcggcg gtggcggcgg ggggtgtttg acggggaacc ccatcgacca ctgctggcgg 240
tgcgccggga cggactggag gcaggaccgg cagcggctgg cggactgcgg catcgggttc 300
gggcgcaacg ccatgggcgg caagggcggg ccggtgtacg tcgtcaccga cccctccgac 360
ggcgacccgg tgaaccccgc gccggggacg ctccggtacg gcgccatcca ggagggcccc 420
ctctggatcg tgttcgccgg cgacatgacc atccgcctca acgaggagct cctcgtgaac 480
agctacaaga ccatcgacgg ccgcggcgcc aacgtccacg tcggcgccgg cggcgcgtgc 540
atcacgctcc agtacgtctc caacgtcatc atccacaaca tccacgtcca cgactgcgtc 600
cccgccggca acgccaacgt gcgcgcctcg ccgacgcact acgggtggcg cacccgctcc 660
gacggcgacg gcatctcgct ctactcggcg cgcgacgtgt gggtggacca ctgcgcgctg 720
tcgcgctgcg ccgacggcct catcgacgcc atcatggggt ccacggcgat caccgtgtcg 780
aacagctact tctcgcacca caacgaggtg atgctgctgg ggcacagcga cgggtacctg 840
ccggactcgg cgatgcaggt gaccatcgcg ttcaaccact tcgggatcca gctcgtgcag 900
cggatgcctc ggtgcaggag ggggtacttc cacatcgtca acaacgacta cacggcgtgg 960
gagatgtacg ccattggagg gagcgcgagc cccaccatca acagccaggg caaccgctac 1020
atcgcccccg ccgatcccaa cgccaaggag gtgacgaagc gggtggacac ggaggagggg 1080
cagtgggcgg ggtggaactg gaggacggag ggggacatga tggtcaacgg cgccttcttc 1140
gtcccctccg gcgagggcct cgaggccatc tacgacaagg cctccagcac cgaccccaag 1200
tcgtcggcgc tcgtcgacca gctcaccgcc ggcgccggcg tcctcggcgg ccccagggac 1260
aacggcgagg cggcggcgta cgccggcgtc aactacgccg gagttggaac cggcggcgga 1320
ggcggtggcg gcgccggtgc cggcgggatg gggtatggtt atctcgggat ggtctatggc 1380
agcggcggca attggagctg ccgagctgac ctgacactgc aattgacttc attgtttctt 1440
gcactctttg ctctcatatg cttgcaccca ttgtga 1476
<210> 2
<211> 5315
<212> DNA
<213> 水稻(Oryza sativa)gDNA全长
<400> 2
atggccgggc ccgtcgaatc cacccgcatt accctcctcc tcgtcgccgc caccctcctc 60
ctcctcccgc cgccgctcgc cgcctccctc aactcctccc tccccgaccc cgccgccgtc 120
gtcgccgact tccacaggta agcgtgcacc ccccgcaatg ccggtgcgat tttttcggaa 180
tcctttttct tgaggtttct tggctaatgg cagcaaggtg gcgacgtcgc ggcggcggat 240
gcagaaggcc gacggcggcg gcggcggtgg cggcgggggg tgtttgacgg ggaaccccat 300
cgaccactgc tggcggtgcg ccgggacgga ctggaggcag gaccggcagc ggctggcgga 360
ctgcggcatc gggttcgggc gcaacgccat gggcggcaag ggcgggccgg tgtacgtcgt 420
caccgacccc tccgacggcg acccggtgaa ccccgcgccg gggacgctcc ggtacggcgc 480
catccaggag ggccccctct ggatcgtgtt cgccggcgac atgaccatcc gcctcaacga 540
ggagctcctc gtgaacagct acaagaccat cgacggccgc ggcgccaacg tccacgtcgg 600
cgccggcggc gcgtgcatca cgctccagta cgtctccaac gtcatcatcc acaacatcca 660
cgtccacgac tgcgtccccg ccggcaacgc caacgtgcgc gcctcgccga cgcactacgg 720
gtggcgcacc cgctccgacg gcgacggcat ctcgctctac tcggcgcgcg acgtgtgggt 780
ggaccactgc gcgctgtcgc gctgcgccga cggcctcatc gacgccatca tggggtccac 840
ggcgatcacc gtgtcgaaca gctacttctc gcaccacaac gaggtgatgc tgctggggca 900
cagcgacggg tacctgccgg actcggcgat gcaggtgacc atcgcgttca accacttcgg 960
gatccagctc gtgcagcgga tgcctcggtg caggaggggg tacttccaca tcgtcaacaa 1020
cgactacacg gcgtgggaga tgtacgccat tggagggagc gcgagcccca ccatcaacag 1080
ccagggcaac cgctacatcg cccccgccga tcccaacgcc aaggaggtaa gcaagcgacg 1140
ccattgttgt ttgccgtgtt cttggcttct tgctctcttc gttgttgctt tgcttgtgag 1200
tcgtgagatg tgatagcaga aggaaaaaag aaaaatcttt ttgtgtgtgt gtgtgtgttg 1260
gattggtttt ggtctgtttg tttctcattt cggctcgaat tgctgatgct gtcggatgtt 1320
tgatgcccat ttttggatga ttttatttta ttgggtggtt atttcttgtt ctgttcattg 1380
gtttgcttat gaaaggaaaa aaaatgtctt ttatttagtt ggttttcctc cgtttgcttc 1440
tcattttggc tcaaattgct gaggttgtca cttgtcagct gtttgatgcc ccaatttggg 1500
atgaattcct ggtttaagtg gggtgtgaaa tttagagcaa tttgaccata tttagaaaaa 1560
ttcccggtta aagtggggtg tgaaatttag agcaatctga ccatatttag aaaaatttct 1620
caagtcgttt gacctgtttt taaggtgtga gtagcgctct atggtcatgt gaattgccac 1680
gcttttaggg cgaatgtttg gagcatcatc ataatagcgc ctcttcttta aagagcaaaa 1740
atttgggtga atgtttttcc cccttgccct cgtgatagca gcatccgagg caggcgcgtg 1800
caccgcacga tgcgatgcga tgagatgaga tgcaccctcc cccaagattc ggtgaggctg 1860
acatgtgggc cccacactgg gacaaaaaac gtgcaggtga cgaagcgggt ggacacggag 1920
gaggggcagt gggcggggtg gaactggagg acggaggggg acatgatggt caacggcgcc 1980
ttcttcgtcc cctccggcga gggcctcgag gccatctacg acaaggcctc cagcaccgac 2040
cccaagtcgt cggcgctcgt cgaccagctc accgccggcg ccggcgtcct cggcggcccc 2100
aggtacgtac ggcacccacc catccctcct acattgcatc gtcatcgcct cgccggctcg 2160
ccgctctcct tttgcacggt cgaatgaatc cgacatgtag cagcagcgtg ggtgtgtggg 2220
ccccactgcg tgaaatatcc gccggatccc tctcgctgac tggtggggcc cacgtgggca 2280
tttttttttc ttttgggttg ggccgtcttt tttttaatgc ggcagcggcg cctttaaact 2340
gccacctccg gtcgccggag gggcgccgct tgtcgagttg ttgtcggcgc cattgccgcg 2400
acctggatgg gtagagaggg atttgcggta gaacagctcg tcgatttggt aacattgagg 2460
tcccgttgga agcatgaaga ttaggaaagg gcacgtaaga cgagagttat tagcacacat 2520
taattgattt ttaatccctc catccaaaaa tataatggat tttgggtgga tgagtcgtag 2580
ggtctgtttg gtacagctcc aactcctaaa tttagaggag ttgggtctgg agtggatcta 2640
attcattttg tgagagctcc atccaactcc actcccagtt ctagtggagc tgaaactgtt 2700
tggccaagct ccagctctag aaggggtgga gctggagctg tgccaaacag gcccgtacta 2760
tagtactatg aatcttgaca acatggtact agaatatatc gcatccatcc aaaatccctt 2820
atattttagg acagatggag tagatattat aaacttaaaa ataaacttta tttgatacta 2880
gtttagatca acttctatat aaaaattttt cgcacgtaat atattattta acagtttaaa 2940
aaaagtgcta atgaaaatta ggatagaatc tagtagtatg atcttagtca gaaacaaacg 3000
ggcctgagag catgagagca gagcagtttg gctagctagc aacaagcgtg gcagtacagt 3060
aagaaatgca ggccggcgat gcaggtggac ttgtacaagc actctgactg gaactgagct 3120
aaccatgtcg gcgtcctacc gcgataatcc tacaattatt ttcctggcca aatcctcaat 3180
ttatagaccc ttagctggta aaaggcagga caggttagga gggccaaagt aaagtattct 3240
taacagatga ttactattac aaggctgagc actcaataga gagccgtacc actaacacta 3300
gcggctgact gacaaagtcg ggttgattaa acaccgggat aaaagtttat aaagcaactt 3360
tgtcttgtac taccagtagg cccagtactt gtattctggt acactgctag tgctagtatt 3420
ctttttagac agcgaactag actctgctcg tgtgggatgg gcgaaacgcg agacttgggg 3480
agtgggcagc acgggcaggc aaatgcactg ccgccgttgc attttcgttc tcccgttctt 3540
tcctctgaaa ccatggcaag acaagagcag actactcatg tcaagcttac aaaaaccttc 3600
caactatcat aaaataaaca aaacgatgtg acctagtatg aatctggata gatcgtattc 3660
gtaataagta tatgtttatt tttgacggat ggagtacatt tttacatgaa tgacttactc 3720
ccttcgtcct aaaaaaaatc aacttttaaa gaaaaatcta gacaaatgca tgaccggatt 3780
tatctttaga atttgatttt tttttttaaa gaacgaagag tgtacatcat tgcgttagac 3840
tttaatactc cattcgtttc taaaaaataa actaatacgg aaattagggt tgttttttat 3900
acgacgagga gggaatacat tgcaatgcta gtacgttggc acataaactc tcttcagtca 3960
atctgcctcc ggatgtccgg attatgaata aaaaataatg gaccagtgtt atcttttggt 4020
agccggtcat gtcgatgagc catcaactga atccacagat ggtggccggt cttttttcct 4080
cttcgctcga tgccaaaagt agccgggatc gaaaagatcg tcgcatgctc agcaacatgc 4140
gtgctatatc catcgattgc ataacccagg gatcacggta ggccggcaat cactcaccat 4200
gcattgcctt cttcttctcc acttgctcgc cactgacccc ttcgttttgt cgccgaatcc 4260
tgcaaaagat agccagaaaa gaaaaaaaag gctcaaaaga agctagtaca cacaaacaca 4320
aactggccca atgcattata gttgttgctg aattattcta gtactgcgag aagctgtgtt 4380
ctttgggcgt tttggatctc aaacagtgtc ctgtactgga gaccctacca tattgcaatg 4440
cgatgacctt ttcggatgga gccaagcaag aagagaacgt tgagggtcca tttgcaacga 4500
tccattagaa aatggcgtgc actttttgtc gttctgtcca tcactctgca ggagaagacc 4560
catgttcctc ggcatccaat tccttattct gcagcgtgca tatgcacttg tgacagacag 4620
cagtagtaga tctcacctta agtacagttt cacttgaaag tttttgctac aacttgtgcc 4680
agttttgcta atgacatact ggcagctact agctctctca tggtagtaca tgagaagaga 4740
atataagata gtttgctaat gtttttggag atttgatctg gcagggacaa cggcgaggcg 4800
gcggcgtacg ccggcgtcaa ctacgccgga gttggaaccg gcggcggagg cggtggcggc 4860
gccggtgccg gcgggatggg gtatggttat ctcgggatgg tctatggcag cggcggcaat 4920
tggagctgcc gagctgacct gacactgcaa ttgacttcat tgtttcttgc actctttgct 4980
ctcatatgct tgcacccatt gtgattgtaa gatgcttcat tatgattcaa ttggaaaggt 5040
taggaaaaag gggagaagaa aaaggaagca aaaggtgttg acagttgaca cttagggagt 5100
agtaggcaac agtgttacag tgagatgata tccaccatgt aatgagcaca tggatgggaa 5160
tattgcaggt ttagccaatg tagttgttgc aaaggggaat gggattattc agactaactt 5220
aagctattaa ctctaacctc tctgtaatct ctgtcgaagg gtaacgaatc tatggtttaa 5280
tggataaaca aagttgagca aatgctagaa agaaa 5315
<210> 3
<211> 491
<212> PRT
<213> 水稻(Oryza sativa)
<400> 3
Met Ala Gly Pro Val Glu Ser Thr Arg Ile Thr Leu Leu Leu Val Ala Ala Thr Leu Leu
1 5 10 15 20
Leu Leu Pro Pro Pro Leu Ala Ala Ser Leu Asn Ser Ser Leu Pro Asp Pro Ala Ala Val
25 30 35 40
Val Ala Asp Phe His Ser Lys Val Ala Thr Ser Arg Arg Arg Met Gln Lys Ala Asp Gly
45 50 55 60
Gly Gly Gly Gly Gly Gly Gly Gly Cys Leu Thr Gly Asn Pro Ile Asp His Cys Trp Arg
65 70 75 80
Cys Ala Gly Thr Asp Trp Arg Gln Asp Arg Gln Arg Leu Ala Asp Cys Gly Ile Gly Phe
85 90 95 100
Gly Arg Asn Ala Met Gly Gly Lys Gly Gly Pro Val Tyr Val Val Thr Asp Pro Ser Asp
105 110 115 120
Gly Asp Pro Val Asn Pro Ala Pro Gly Thr Leu Arg Tyr Gly Ala Ile Gln Glu Gly Pro
125 130 135 140
Leu Trp Ile Val Phe Ala Gly Asp Met Thr Ile Arg Leu Asn Glu Glu Leu Leu Val Asn
145 150 155 160
Ser Tyr Lys Thr Ile Asp Gly Arg Gly Ala Asn Val His Val Gly Ala Gly Gly Ala Cys
165 170 175 180
Ile Thr Leu Gln Tyr Val Ser Asn Val Ile Ile His Asn Ile His Val His Asp Cys Val
185 190 195 200
Pro Ala Gly Asn Ala Asn Val Arg Ala Ser Pro Thr His Tyr Gly Trp Arg Thr Arg Ser
205 210 215 220
Asp Gly Asp Gly Ile Ser Leu Tyr Ser Ala Arg Asp Val Trp Val Asp His Cys Ala Leu
225 230 235 240
Ser Arg Cys Ala Asp Gly Leu Ile Asp Ala Ile Met Gly Ser Thr Ala Ile Thr Val Ser
245 250 255 260
Asn Ser Tyr Phe Ser His His Asn Glu Val Met Leu Leu Gly His Ser Asp Gly Tyr Leu
265 270 275 280
Pro Asp Ser Ala Met Gln Val Thr Ile Ala Phe Asn His Phe Gly Ile Gln Leu Val Gln
285 290 295 300
Arg Met Pro Arg Cys Arg Arg Gly Tyr Phe His Ile Val Asn Asn Asp Tyr Thr Ala Trp
305 310 315 320
Glu Met Tyr Ala Ile Gly Gly Ser Ala Ser Pro Thr Ile Asn Ser Gln Gly Asn Arg Tyr
325 330 335 340
Ile Ala Pro Ala Asp Pro Asn Ala Lys Glu Val Thr Lys Arg Val Asp Thr Glu Glu Gly
345 350 355 360
Gln Trp Ala Gly Trp Asn Trp Arg Thr Glu Gly Asp Met Met Val Asn Gly Ala Phe Phe
365 370 375 380
Val Pro Ser Gly Glu Gly Leu Glu Ala Ile Tyr Asp Lys Ala Ser Ser Thr Asp Pro Lys
385 390 395 400
Ser Ser Ala Leu Val Asp Gln Leu Thr Ala Gly Ala Gly Val Leu Gly Gly Pro Arg Asp
405 410 415 420
Asn Gly Glu Ala Ala Ala Tyr Ala Gly Val Asn Tyr Ala Gly Val Gly Thr Gly Gly Gly
425 430 435 440
Gly Gly Gly Gly Ala Gly Ala Gly Gly Met Gly Tyr Gly Tyr Leu Gly Met Val Tyr Gly
445 450 455 460
Ser Gly Gly Asn Trp Ser Cys Arg Ala Asp Leu Thr Leu Gln Leu Thr Ser Leu Phe Leu
465 470 475 480
Ala Leu Phe Ala Leu Ile Cys Leu His Pro Leu ***
485 490 491
- 还没有人留言评论。精彩留言会获得点赞!