一种广谱检测致癌病原微生物的方法与流程
本发明涉及检测和鉴定病原微生物制剂领域,特别涉及一种广谱检测致癌病原微生物的方法。
背景技术:
2010年12月Patrick S.Moore(美国匹兹堡大学资深病毒学家,美国科学院院士)与夫人Yuan Chang教授作为卡波西肉瘤病毒(KSHV)以及默克尔细胞多瘤病毒(MCV)的共同发现人联名发表了“为什么病毒会引起癌症?人类肿瘤病毒学的第一个百年大事记”。自1911年弗朗西斯·佩顿·劳斯(Francis Peyton Rous)首次发现禽肉瘤病毒,划时代地提出“癌性肿瘤是病毒所致”成为“肿瘤病毒”第一人,并于1966年获诺贝尔生理学或医学奖。此后,病毒成为现代肿瘤研究的核心,1964年首次在Burkitt淋巴瘤细胞株中发现人类肿瘤病毒EB病毒;1965年发现肝炎由乙肝病毒(HBV)引起,1981年证实与肝癌相关,乙肝疫苗成为第一个抗癌疫苗上市;1983年德国科学家哈拉尔德·楚尔·豪森在宫颈癌病人样本中发现高危型人乳头瘤病毒(HPV),2006年疫苗获美国食品药品管理局(FDA)批准上市,2008年哈拉尔德·楚尔·豪森获诺贝尔生理学或医学奖;1989年美国科学家迈克尔·侯顿(Michael Houghton)和他的同事们发现并克隆丙肝病毒(HCV);1989年Varmus和Bishop因发现逆转录病毒癌基因的细胞学起源而获得诺贝尔医学奖;1994年Patrick S.Moore课题组在卡波济肉瘤中发现卡波济肉瘤病毒(KSHV),2008年在梅克尔细胞癌中发现致癌病毒梅克尔细胞多瘤病毒(MCV)。
2009年国际癌症研究机构(IARC)召集16个国家的36名专家,对已经归类为人类致癌的生物因子是否确有致癌性进行了重新评估,并对致癌的部位和癌症发生的机理作了进一步鉴定,并于2012年正式发表专论。明确提出:在全世界有17.8-26%的癌症是由下列一种人类病毒、细菌或寄生虫所引起,包括:7种病毒(EB病毒、乙肝病毒、丙肝病毒、卡波济肉瘤疱疹病毒、人类免疫缺陷病毒1(HIV-1)、人乳头状瘤病毒、嗜人类T淋巴细胞病毒I型)(HTLV-1),1种细菌(幽门螺旋菌),三种寄生虫(埃及血吸虫、中华肝吸虫和泰国肝吸虫)等共11种。到目前为止总共有16个型:HPV16和18型为IARC于1995年首次确认的主要高危致癌型,HIV-1和HIV-2型(2型为新出现的耐药型),HTLV-1,2,3,4型(后两个型为2008年3后发现的新型株)。
2014年美国国家卫生研究院(NIH)分子微生物学主任Dr.Kuan-Teh Jeang在“病毒与人类癌症:从基础科学到临床预防”一书中提出:此类微生物可导致相关脏器细胞的癌变,与恶性肿瘤的形成关系密切,如:EB病毒与鼻咽癌、淋巴瘤,乙肝病毒、丙肝病毒与肝癌,幽门螺杆菌与胃癌、胃淋巴瘤,人类乳头状瘤病毒与宫颈癌、阴户、阴道、阴茎、肛门、口腔、扁桃体癌、喉癌,嗜人类T淋巴细胞病毒I型HTLV-1与成人T細胞白血病/淋巴瘤,卡波济肉瘤疱疹病毒8与卡波济肉瘤、淋巴瘤,其中梅克尔细胞多瘤病毒(MCV)是2008年新发现的病毒与梅克尔细胞癌(MCC)有关等。本结论基于最近50年来大量实验研究和流行病学研究积累结果。鉴定出更多的癌症与生物致癌因子的关系,意味着更多的癌症是可以预防的。目前亟需一种全面涵盖所有生物致癌因子的检测方法,用于筛查那些目前尚未发现但可能与感染相关的癌症,如基因测序发现与自身免疫抗病毒蛋白基因突变相关的16种癌症,包括:急性淋巴細胞性白血病、膀胱癌、乳腺癌、宫颈癌、慢性淋巴性白血病、食道癌、头颈癌、乳头状肾细胞癌、肺腺癌、骨髓瘤、肺鳞癌、淋巴B细胞癌、胰腺癌、胃癌、甲状腺癌、子宫癌。
2011年12月瑞典卡罗林斯卡研究所John Inge Johnsen报道人巨细胞病毒(Cytomegalovirus HCMV_HHV5)在以下多种肿瘤中检出,包括:脑胶质瘤、髓母细胞瘤、神经母细胞瘤、乳腺癌、前列腺癌和结肠癌涎腺粘液表皮样肿瘤,因此可以利用这一分子靶向选择性杀伤该肿瘤细胞。
在癌症病因学研究中,一个最显著的成果就是确立了人类病毒的某些特定型别感染,是引起人类某些恶性肿瘤的主要原因。
2016年1月25日国家癌症中心赫捷院士和陈万青教授团队,在CA Cancer J Clin.发布“2015年中国癌症统计数据”。预计有429.2万例新发肿瘤病例。约22%的全球新发癌症出现在中国,27%的癌症死亡病例在中国。且国人的癌症谱与西方发达国家相差甚大,中国四种最常见肿瘤分别为肺癌、胃癌、肝癌和食管癌,占全国癌症病例的57%,而在美国这四种癌仅占18%。约60%的癌症死亡是可以通过减少可控危险因素暴露来预防的,减少国人癌症死亡的最可行途径就是控制慢性感染,29%的癌症死亡是与慢性感染相关,主要是胃癌(幽门螺杆菌Hp感染)、肝癌(肝炎病毒HBV和HCV感染)和宫颈癌(人乳头瘤病毒HPV感染)。大多数恶性肿瘤都有10至15年的潜伏时间来纠正,不要错过这个机会。
根据2012年国际肿瘤研究机构确定的11种共16个型生物致癌因子和国际权威杂志最新报道的癌症相关病毒MCV和HCMV,总共13种18个型,建立了一个完善的DNA+RNA生物致癌因子双基因探针(除外HTLV的四种型为四对单基因探针),高灵敏度、高特异性检测试剂盒。
DNA生物致癌因子(10种11个型)包括:
EB病毒(Epstein-Barr virus,HUMAN HERPES VIRUS疱疹病毒4)是一种疱疹病毒,1964年由Epstein和Barr首次从非洲儿童Burkitt淋巴瘤细胞中分离建株。该病毒被认为是多种恶性肿瘤(如鼻咽癌)的病因之一,它主要感染人类口咽部的上皮细胞和B淋巴细胞。在中国南方鼻咽癌患病人群中大多都检测到有EB病毒基因组存在。病毒携带者和病人是本病的传染源,经口密切接触为主要传播途径。EB病毒俗称为“接吻病毒”,原因是EB病毒的传播途径是唾液交换,接吻为最常见的传播方式。由于迄今还没有可消灭EB病毒的药物,市民要避免与陌生人接吻。除了鼻咽癌外,接吻还会引发一系列传染病,在台湾,经常会有20多岁的青年因接吻感染EB病毒,并引致严重咽喉扁桃体炎、喉咙痛、发烧、疲倦、颈部及全身淋巴腺肿大。在本检测中包括两个基因和一个重复序列非基因区,以提高检测灵敏度,
卡波济肉瘤疱疹病毒(KAPOSI SARCOMA HERPESVIRUS,HUMAN HERPES VIRUS 8,KSHV)是1872年匈牙利皮肤科专家Kaposi首先报道的一种罕见的肿瘤,在非洲多见,并可形成地方性流行,且多发于60岁以上的老人及免疫系统受损人群,如艾滋病患者等。近10年来,在欧美男性同性恋者中出现了暴发性流行,同时,卡波济肉瘤与艾滋病有一定的关系,它被认为是艾滋病的一种常见肿瘤。据统计,约30%同性恋的白种人中,卡波济肉瘤是艾滋病感染者的首要表现,且多最早出现。在我国新疆地区有报告,主要见于维吾尔族等少数民族。随着器官移植的广泛开展及免疫抑制剂的普遍应用,与移植物有关的卡波济肉瘤的发病有所增加。特别是近年来艾滋病的传播,艾滋病型卡波济肉瘤明显增多,而且进展快,治疗困难,死亡率高。
乙肝病毒(hepatitis B virus,HBV)的致癌性:现在已经肯定,HBV是致肝癌的重要因子,约80%—90%的肝癌都有HBV背景。有人观察发现,有20年HBV感染史者,约有5%—10%的发生癌变,癌变的原因是HBV的x基因整合到肝细胞基因上,发生了突变,导致肝癌。启动子(BCP)变异:基本核心蛋白的启动子(BCP)的变异是多位点和复合的,主要是1762(A→T)、l764(G→A)的双突变,BCP区是富含AT区域,具有肝脏富含因子的独立结合点,变异的BCP可改变这种结合,降低Prec区的RNA转录,最终使HBeAg表达减少,导致病情加重或为重型肝炎。BCP的变异还可使前基因组RNA转录增强,病毒的复制能力增加。目前,亦有文献报道该区域的突变有可能导致肝癌的发生。在本检测中包括两个基因:S和X基因,及一个BCP突变位点,仅见X基因阳性可能发生致癌整合,BCPl764(G→A)阳性,提示导致癌症的可能性增加。Jiang et al.,Genome Res.2012 22:593-601
人乳头状瘤病毒(human papillomavirus,HPV)可感染人体的表皮与黏膜组织,部分型别可引起性传播疾病。其中,高危型HPV16和18型明确为强高危致癌型别,可引起子宫颈、阴户、阴道、阴茎、肛门、口腔、口咽和扁桃腺癌。
幽门螺杆菌(Helicobacter pylori,Hp)首先由Barry J.Marshall和J.Robin Warren)二人发现,并因此获得2005年的诺贝尔生理学或医学奖。幽门螺杆菌感染是慢性活动性胃炎、消化性溃疡、胃黏膜相关淋巴组织(MALT)淋巴瘤和胃癌的主要致病因素。1994年世界卫生组织/国际癌症研究机构(WHO/IARC)将幽门螺杆菌定为Ⅰ类致癌原。
中华肝吸虫(C.sinensis)和泰国睾吸虫(O.viverrini)是一种可寄生在人类胆管和胆囊的吸虫,以胆汁为食。生食被肝吸虫污染的淡水鱼螺和腌制鱼类是感染的主要原因。中国有超过1200万人感染肝吸虫,其中大多数分布在东南、东北省份,这些地区群众喜欢吃生鱼片,淡水鱼很容易感染肝吸虫的幼虫。国际癌症研究机构将其归类为1类致癌物—O.viverrini是导致人类胆管癌的一个明确原因,而C.sinensis是一个可能原因。
埃及血吸虫(S.haematobium)1994年世界卫生组织国际癌症研究机构已将埃及血吸虫感染与麝猫后睾吸虫感染评定为确认人类致癌物(Group 1),将华支睾吸虫感染评定为对人很可能致癌(Group 2A),将日本血吸虫感染评定为对人可能致癌(Group 2B)。
梅克尔细胞多瘤病毒(MCPyV,MCV)是2008年匹兹堡大学癌症研究院分子病毒研究组的研究人员Patrick S.Moore与Yuan Chang等发现可引起梅克尔细胞癌,大约80%的梅克尔细胞癌与梅克尔细胞多瘤病毒感染相关,20%的梅克尔细胞癌未发现梅克尔细胞病毒感染,提示梅克尔细胞癌还有其他致病因素。在过去的20年间,MCC增长了三倍,每年出现1500个病例,这种癌症主要发生在免疫系统受到AIDS,或者移植相关免疫抑制药物破坏的个体身上,一半患有MCC的病人只能存活至多9个月,三分之二的病人在5年内会死亡。
人类巨细胞病毒(human cytomegalovirus,HCMV)属疱疹病毒科,又称为涎病毒,是人类疱疹病毒组中最大的一种病毒。这种病毒进入细胞内以后会导致细胞增大,所以叫做巨细胞病毒。在我国巨细胞病毒感染广泛流行,通常情况下,很多正常人身上都潜伏有巨细胞病毒,潜伏部位常在唾液腺、乳腺、肾脏、子宫颈、睾丸、白细胞以及其它腺体中,病毒可长期或间歇地自唾液、乳汁、尿液、精子及子宫颈分泌物中排出。但在大多数免疫正常个体呈无症状感染,而在免疫抑制个体、胎儿和婴幼儿如果感染则可出现明显病症。瑞典和美国等科学家最新报道,许多最新证据提示HCMV可能特定的与人类某些肿瘤如:胶质母细胞瘤、成神经管细胞瘤、前列腺癌、乳腺癌和结肠癌,以及涎腺粘液表皮样癌等有关。
生物致癌因子RNA组合(3种7型)包括:
丙肝病毒(Hepatitis C virus,HCV)是慢性肝炎的主要致病因子。在美国慢性肝病患者中,40~60%感染了HCV。全世界约有一亿七千万人感染了HCV,这个数字高于感染了HIV的人口。流行病学研究表明:约20%慢性肝炎患者要发展为肝纤维化和肝硬化,在这些肝硬化病人当中,20%要发展为肝癌。
人类T淋巴细胞病毒I型(HUMAN T-CELL LEUKEMIA VIRUS 1,HTLV-1)是80年代初第一个被明确为与引起人类癌症(成人T淋巴细胞白血病/淋巴瘤,ATLL)有关的逆转录酶病毒。此外,该病毒还可以引起炎症性疾病,如HTLV-1相关脊髓病(HAM)/热带痉挛性下肢轻瘫(TSP),眼色素层炎,传染性皮炎和肌炎。目前HTLV-1在全世界的感染人数大约有2千万人。HTLV-2,3,4型为2008年后在喀麦隆(非洲西部国家)发现的新型株。
人类免疫缺陷病毒I型(HUMAN IMMUNODEFICIENCEY VIRUS-1,HIV)属于逆转录病毒科慢病毒属中的人类慢病毒组,分为1型和2型。目前世界范围内主要流行HIV-1。HIV-1引起机体免疫缺陷,当与其它病毒共感染时,易发生各种肿瘤,如卡波济肉瘤,非霍奇金淋巴瘤,子宫颈、肛门、眼球结膜癌,以及阴户、阴道、阴茎、非黑色素瘤皮肤癌、肝细胞癌。HIV-2型为新出现的耐药型。HIV-2是经常用抗HIV-1感染的一些抗逆转录病毒药物治疗后产生天然抗性的新型株,治疗它需要选择其它确实有效的抗逆转录病毒药物治疗。
美国食品和药品管理局(FDA)目前批准的多种病原微生物检测方法有:呼吸道病毒检测试剂盒,例如:Biofire FilmArray RP20种呼吸道相关病原体检测,Genmark eSensor RVP 14种流感相关病毒和Luminex xTAG RVP fast multiplex assays 18种呼吸道病毒基因检测等。与致癌病原体相关的检测试剂盒均以单种病原体检测形式存在:如罗氏cobas人乳头瘤病毒(HPV)检测试剂盒可识别HPV 16和18基因型,能检测其它12种高风险基因型(31、33、35、39、45、51、52、56、58、59、66和68);AmpliPrep/COBAS TaqMan HBV检测;Abbott RealTime HBV检测,HIV-1检测,CT/NG衣原体/淋病检测等。
中国食品药品监督管理总局(CFDA)目前批准上市的多种病原微生物检测方法有:博奥生物的8种常见下呼吸道病原菌;中帜生物七项呼吸道病原体核酸检测试剂盒;肺炎支原体/肺炎衣原体核酸检测试剂盒(双扩增法);8种常见食源性细菌(沙门菌、志贺菌、金黄色葡萄球菌、霍乱弧菌、副溶血性弧菌、单增李斯特菌、奇异变形杆菌、大肠杆菌O157)核酸的快速检测,用于食源性疾病的辅助诊断。用于与致癌病原体相关的检测试剂盒不论是PCR方法还是ELISA免疫反应检测法,均为同类病原微生物的检测。如:凯普21种基因型HPV分型;厦门安普利乙型肝炎病毒基因分型检测试剂盒鉴别B、C、D基因型;赛默飞世尔合资公司立菲达安HBV耐药、HCV分型等。
目前国际、国内还没有一个商用试剂盒能够针对所有10多种生物致癌因子的基因检测试剂盒,也没有一个实验室具备同时对10多种生物致癌因子进行检测的能力。如果分别用实时PCR检测,需要抽十几管血,送到不同的实验室,用各种不同试剂和方法进行检测。不但其检测成本高,周期长,而且样本变质和被污染的可能性也大。对于器官移植和血液制品,以及日益高发的因生物致癌因子导致的感染性癌症,缺乏强有力的全面精准检测手段。
采用常规抗原与抗体的ELISA免疫反应检测法,其检测结果只能代表患者是否曾经感染过这些病原体。而大多数人在生活中均已感染过生物致癌因子,当人体自然免疫反应正常时,可以自身产生抗体清除病原体,使免疫反应呈现阳性。如同结核和乙肝,中国大多数人检测呈免疫反应阳性,但健康人体内的结核杆菌和乙肝病毒的DNA检测必须是阴性。一些癌症病人,由于机体缺乏免疫力,当EB病毒DNA强阳性时,EB病毒IgM抗体检测反而为阴性(本发明检测结果,尚未发表)。因此,只有检测生物致癌因子的DNA或RNA,才能真实反应病原体感染的活动状况。
质谱扫描检测生物致癌因子法,采用21世纪最先进的DNA质谱技术。通过数字化光电检测器,不仅能一次检测到所有10多种已知生物致癌因子,而且针对每种生物致癌因子均采用了2种以上靶向基因及致癌突变位点检测。相当于同时采用两种不同厂家的试剂盒进行平行检测,最大限度的避免了因DNA/RNA突变或降解造成的假阴性。其检测结果的灵敏度之高1-10copies/反应、特异性之强、重复性之好、检测动态范围之宽1-1.0X1010copies/反应,且费用相对低廉,出报告时间短,是目前其它方法所无法比拟的。
技术实现要素:
本发明提供了一种广谱检测致癌病原微生物的方法,其中所述的致癌病原微生物包括:2012年国际肿瘤研究机构确定的11种共16个型生物致癌因子,和国际权威杂志最新报道的与癌症相关的病毒MCV和HCMV总共13种18个型。所述的致癌病原微生物分为DNA和RNA两种,本发明的检测方法分为对DNA的检测和对RNA的检测。
本发明提供了一种广谱检测致癌病原微生物的方法,其中,方法一是对DNA生物致癌因子的检测,方法二是对RNA生物致癌因子的检测,以下分别描述:方法一、对DNA生物致癌因子的检测,即对11种DNA病原微生物进行检测,对于每一种每种DNA病原微生物需要两个或两个以上的探针,这些DNA病原微生物包括:
1、EB病毒_人类疱疹病毒4(EPSTEIN-BARRVIRUS,EBV_HHV-4)覆盖AB亚型;
2、卡波济肉瘤病毒_人类疱疹病毒8(KAPOSI SARCOMA HERPESVIRUS,KSHV_HHV-8);
3、人巨细胞病毒_人类疱疹病毒5(HUMAN CYTOMEGALOVIRUS,HCMV_HHV-5);
4、乙肝病毒(HEPATITIS B VIRUS,HBV):S基因、X基因及BCP致癌突变;
5、人乳头状瘤病毒16型(HUMAN PAPILLOMAVIRUSES,HPV-16)E6/L1基因;
6、人乳头状瘤病毒18型(HUMAN PAPILLOMAVIRUSES,HPV-18)E6/L1基因;
7、梅克尔细胞多瘤病毒(MERKEL CELL POLYOMAVIRUS,MCV);
8、幽门螺旋菌(Helicobacter pylori,Hp);
9、中华肝吸虫(CLONORCHIS SINENSIS,C-SINENSIS);
10、泰国肝吸虫(OPISTHORCHIS VIVERRINI,O-VIVERRINI);
11、埃及血吸虫(SCHISTOSOMA HAEMATOBIUM,S-HAEMATOBIUM)。
方法二、RNA生物致癌因子的检测,即对7种RNA病毒病原微生物进行检测,对于每一种每种RNA病毒病原微生物需要两个或两个以上的探针,这些RNA病毒病原微生物包括:
1、丙肝病毒(HEPATITIS C VIRUS,HCV);
2、人类免疫缺陷病毒(HUMAN IMMUNODEFICIENCEY VIRUS-1,HIV-1);
3、人类免疫缺陷病毒(HUMAN IMMUNODEFICIENCEY VIRUS-1,HIV-2);
4、嗜人类T淋巴细胞病毒I型(HUMAN T-CELL LEUKEMIA VIRUS 1,
5、HTLV-1)嗜人类T淋巴细胞病毒II型(HUMAN T-CELL LEUKEMIA VIRUS 2,HTLV-2);
6、嗜人类T淋巴细胞病毒III型(HUMAN T-CELL LEUKEMIA VIRUS 3,HTLV-3);
7、嗜人类T淋巴细胞病毒IV型(HUMAN T-CELL LEUKEMIA VIRUS 4,HTLV-4)。
本发明所述方法,包括以下步骤:
步骤1、引物设计:涵盖国际肿瘤研究机构IARC在2009年提出的全部生物致癌因子和国际权威机构新近发表的与癌症相关的生物致癌病毒,总共18种型。全部检测分为两部分,第一部分为DNA病原微生物,包括:EBV_HHV-4、KSHV_HHV-8、HCMV_HHV-5、HBV、HPV-16、HPV-18、MCV、Hp、C-SINENSIS、O-VIVERRINI和S-HAEMATOBIUM,总共11种型,分别采用了双基因探针包括致癌基因或突变位点检测。第二部分为RNA病毒,包括:HCV、HIV-1、HIV-2、HTLV-1、HTLV-2、HTLV-3和HTLV-4,总共7种型,分别采用了双基因探针(除外HTLV的四种型为四对单基因探针)。选择每个型别的特异性保守序列,设计一对PCR引物,在引物的5’端带有10个碱基(ACGTTGGATG)任意组合的标签序列,使总长度达到30个碱基以上,用以从分子量上将引物与探针区别开,避免干扰。在扩增区中的保守序列区设计一条单碱基延伸探针,探针的长度为14-28个碱基,在探针的3’末端,允许延伸一个经设计确定的碱基,作为该基因型特异性序列标记,单碱基延伸后总长度不超过29个碱基,每个探针的延伸产物之间分子量相差至少16Da。各种HPV扩增引物序列见表1,其对应的碱基延伸探针见表2。
步骤2、PCR反应:在PCR过程中引入尿嘧啶N糖基化酶(UNG酶)技术,在首次PCR反应体系中,以dUTP替代常规PCR的dTTP,使产物中掺入大量dU。在再次进行PCR扩增前,用UNG酶处理PCR混合液即可消除PCR产物的残留污染。由于UNG酶在PCR循环中一步便可被灭活,因此不会影响含dU的新的PCR反应及产物,彻底排除了PCR扩增产物污染引起的假阳性问题。通过第一轮PCR扩增,获得待测样本中靶序列扩增产物。
步骤3、虾碱性磷酸酶(SAP)处理:SAP可使未结合的剩余核酸(dNTPs)脱磷酸失活,避免干扰下一步碱基延伸反应。
步骤4、碱基延伸反应:在第二轮扩增中进行单碱基延伸探针的3’端,延伸一个序列特异性单核苷酸,作分子量标记,得到的延伸产物与所述延伸探针及各型别延伸产物之间的分子量差异不小于16Da,
步骤5、树脂脱盐:采用树脂对延伸产物进行纯化。
步骤6、质谱检测:将纯化后的产物采用微量点样仪点于芯片上,用基质辅助激光解析电离飞行时间质谱(MALDI-TOF-MS)系统进行分子量检测,根据分子量标记确定待测HPV基因型别,软件自动处理报告每个基因型的检测结果和可信程度。
优选的,本发明的检测方法,包括以下步骤:
步骤1,用一对PCR引物,在引物的5’端带有ACGTTGGATG碱基的任意组合的标签序列,使总长度达到30个碱基以上,以区别于探针;在扩增区中的保守序列设计一条单碱基延伸探针,探针的长度为14-28个碱基,在探针的3’末端,允许延伸一个经设计确定的碱基,作为该基因型特异性序列标记,单碱基延伸后总长度不超过29个碱基;
步骤2,第一次PCR扩增反应,将dUTP/dNTP混合物、UNG酶、Taq酶和多重PCR引物、水、待检样本一同加入PCR反应体系中,先进行dUTP的消化,降解PCR扩增产物,然后灭活UNG酶,随后作45个循环PCR扩增,获得待测样本中靶序列扩增产物;
步骤3,用虾碱性磷酸酶处理,使第一轮反应中剩余dNTP脱磷酸失活;
步骤4,第二次单碱基延伸扩增,将ddNTPs加入反应体系中,使之在单碱基延伸探针的3’端,延伸一个序列特异性单核苷酸,作分子量标记,扩增200个循环,每个探针的延伸产物之间分子量相差至少26Da;
步骤5,采用阳离子交换树脂吸附盐离子,纯化延伸反应产物;
步骤6,纯化后产物采用质谱技术进行分子量检测,根据分子量标记确定待测病原微生物的类型。
其中所述步骤(1)引物设计中每一种待测病原微生物型采用2个以上基因区及致癌突变位点检测。引物序列为SEQ ID NO.1至SEQ ID NO.70,探针序列为SEQ ID NO.75至SEQ ID NO.109,
所述步骤1中,优选的DNA病原微生物采用β球蛋白基因作为样品质量控制参照,引物序列为SEQ IDNO.71和SEQ ID NO.72,探针序列为SEQ ID NO.110。RNA病毒采用核糖核酸酶P基因作为样品质量控制参照,引物序列为SEQ IDNO.73和SEQ ID NO.74,探针序列为SEQ ID NO.111。
优选的在每次反应中加入阴性对照,所述阴性对照为去离子双蒸水。
本发明的方法,检测浓度低至1-10拷贝/反应。
本发明还包括,为便于应用本发明的检测方法而设计的检测试剂盒,所述试剂盒中包含表1和表2中的核苷酸。所述核苷酸为DNA/RNA致癌病原微生物的特异基因序列的正向和反向引物序列SEQ ID NO.1-SEQ ID NO.70;碱基延伸探针SEQ ID NO.75-SEQ ID NO.109;以及DNA/RNA质控引物序列为SEQ IDNO.71-SEQ ID NO.74,探针序列SEQ ID NO.110和SEQ ID NO.111。
本发明所述试剂盒,根据需要,所述试剂盒还可包括任何一种适合本发明方法的辅助试剂:如,缓冲液,溶液,酶,载体,试纸,试管,棉球等。
本发明的上述试剂盒的使用是以本发明方法为基础,通过定性或定量测定样本中的DNA,RNA,确定病原微生物存在与否,以达到诊断疾病的目的,其中所述样本包括血液,组织,体液,分泌物等。
本发明的试剂盒,各试剂分别包装,优选使用包装管,每个包装管中装入试剂的量以够一个样本使用量为基本量,可以扩大到10个,100个,1000个样本的使用量。
本发明的试剂盒的使用是根据上述检测方法进行的,使用时将试剂盒中的试剂根据需要配制成相应的浓度,按照所述方法测定即可。
本发明表1和表2中的引物和探针,是根据已知的致癌病原微生物的结构设计的,电子版本的形式见序列表SEQ ID NO.1-SEQ ID NO.111。
表1
表2.
与现有相关致癌病原微生物检测的其它技术相比,本发明技术方案最大限度地发挥了PCR-质谱联用平台检测病毒基因型的优势,将国际肿瘤研究机构IARC和国际权威机构及杂志所述的所有生物致癌因子全部囊括其中,每个型采用了双基因探针,确保在基因整合缺失或突变时,仍能够有效检出该基因型。本发明将所有18个致癌病原微生物分为DNA病毒、细菌和寄生虫和RNA病毒两个反应检测。临床可根据需求和样本来源分别使用或组合使用,最大程度地降低了使用成本。检测通量灵活一天可以检测几个至几千个样本。为致癌病原微生物感染的全面普及筛查提供了不可或缺的检测工具。
采用大于20重以上PCR反应产物,作一次性质谱检测,不需要荧光染料标记、不需要洗涤、反应在微量体系中进行,使试剂耗材成本大大降低。首轮PCR扩增区域为60-140bp的基因型特异性片段,第二轮PCR采用特异性探针末端单点法扩增,可以允许使用更多循环,最大限度地提高了检测灵敏度和特异性,使检测灵敏度达到1aM级单个拷贝水平,极大降低了检测漏诊造成的假阴性问题。同时采用UNG酶技术和基因型特异性片段扩增设计技术,彻底排除了PCR产物污染和同源序列探针错配引起的假阳性问题。为多种肿瘤病因学研究和相关疫苗的研制和应用提供了可靠的临床实验依据。
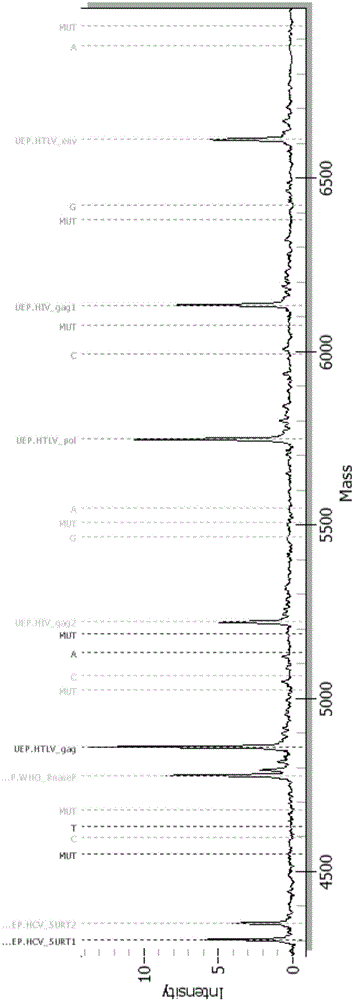
附图说明
图1、DNA生物致癌因子基因型碱基延伸探针质谱峰图和RNA生物致癌因子基因型碱基延伸探针质谱峰图(1-2)
具体实施方式
本发明包括同时检测18种已致癌病原微生物,每种基因型同时采用特异性基因作检测。此处所描述的具体实施方式仅用于解释本发明,并不用于限定本发明。
本发明实施方式提供的高精确度检测和/或鉴定致癌病原微生物基因型的方法,待检测致癌病原微生物基因型包括:EBV_HHV-4、KSHV_HHV-8、
HCMV_HHV-5、HBV、HPV-16、HPV-18、MCV、Hp、C-SINENSIS、O-VIVERRINI、S-HAEMATOBIUM,HCV、HIV-1、HIV-2、HTLV-1、HTLV-2、HTLV-3和HTLV-4,总共18种。每种基因型采用2个基因的特异片段检测。包括如下步骤:
(1)根据待测致癌病原微生物基因型全序列,获取特异性分型基因,选择每个型别的特异性保守序列,设计一对PCR引物,在引物的5’端带有10个碱基(ACGTTGGATG)任意组合的标签序列,使总长度达到30个碱基以上,用以从分子量上将引物与探针区别开。各种致癌病原微生物基因型的扩增引物序列见表1。在扩增区中的保守序列区设计一条单碱基延伸探针,探针的长度为14-28个碱基,在探针的3’末端,允许延伸一个经设计确定的碱基,作为该基因型特异性序列标记,单碱基延伸后总长度不超过29个碱基,每个探针的延伸产物之间分子量相差至少16Da。各种HPV扩增引物序列见表1,其对应的碱基延伸探针见表2。
(2)通过PCR扩增,获得待测样本中靶序列扩增产物。
PCR反应体系配制见表3.
表3
试剂最终浓度体积(1×)
Water,HPLC grade N/A 0.3μL
10X Buffer w/20mM MgCl2 2mM MgCl2 0.5μL
25mM MgCl2 2mM 0.4μL
25mM dUTP/dNTP Mix 500mM 0.1μL
UNG Enzyme(1U/ul)0.5U 0.5μL
0.5mM Primer mix 0.1mM 1.0μL
5U/ml PCR enzymea 1Unit 0.2μL
Template DNA(5-20ng/μL)2.0μL
Total 5.0μL
先用37℃-50℃2分钟,进行dUTP的消化,然后94℃4分钟灭活,(同时这一步
也达到预变性的效果),随后作PCR扩增。反应条件为95℃变性30秒,56℃退火30秒,72℃
延伸1分钟,共45个循环;最终72℃延伸5分钟,完成后恒温于4℃。
(3)通过虾碱性磷酸酶(SAP)处理,使未结合的剩余核酸(dNTPs)脱磷酸失活,预
防干扰下一步碱基延伸反应。SAP消化酶反应体系见表4。
表4
试剂体积/反应
Water,HPLC grade 1.53μL
10×SAP buffer 0.17μL
SAP enzyme(1.7U/ml)0.3μL
Total 2.0μL
反应条件为37℃孵育40分钟,去除剩余dNTPs;然后用85℃5分钟使SAP酶失活。
(4)通过碱基延伸反应,在单碱基延伸探针的3’端,延伸一个序列特异性单核苷酸,作分子量标记,得到的延伸产物与所述延伸探针及各型别延伸产物之间的分子量差异
不小于16Da,延伸反应体系见表5。
表5
试剂体积/反应
Water,HPLC grade 0.619μLc
10×iPLEX buffer plus 0.2μL
iPLEX terminator mix 0.2μL
Primer mix*0.94μL
iPLEX enzyme 0.041μL
Total 2.0μL
*延伸反应探针mix的浓度,按照各型分子量大小进行线性关系调节,总浓度约为
8-15M,最终浓度约为0.84-1.57M。
参考文献:
1.Moore PS,Chang Y:Why do viruses cause cancer?Highlights of the first century of human tumour virology.Nature Reviews Cancer.2010,10(12):878-89
2.Biological Agents Volume 100B A review of Human Carcinogens,2012eview
3.Mei Hwei Chang and Kuan-Teh Jeang Editors,Viruses and Human Cancer:From Basic Science to Clinical Prevention.Springer-Verlag Berlin
Heidelberg 2014
4.Alexandrov,LB,Nik-Zainal,S,Wedge,DC,and Aparicio,SA,et al.Signatures of mutational processes inhuman cancer.Nature 2013,500:415-421
5.John Inge Johnsen1,Ninib Baryawno1and Cecilia-Nauclér.Is human cytomegalovirus a target in cancer therapy?Oncotarget 2011;2:1329–1338
6.Wanqing Chen,Rongshou Zheng and Peter D.Baade,Cancer Statistics in China,2015.CA:A Cancer Journal for Clinicians 2016 Jan-Feb(1):5-29
SEQUENCE LISTING
<110> 北京万泉麟科技有限公司
<120> 一种广谱检测致癌病原微生物的方法
<130> 1
<160> 111
<170> PatentIn version 3.3
<210> 1
<211> 29
<212> DNA
<213> 人工序列
<400> 1
acgttggatg cgcacctctc tttacgcgg 29
<210> 2
<211> 29
<212> DNA
<213> 人工序列
<400> 2
acgttggatg ttcacggtgg trtccatgc 29
<210> 3
<211> 30
<212> DNA
<213> 人工序列
<400> 3
acgttggatg ctgcacgart kctgctcaag 30
<210> 4
<211> 30
<212> DNA
<213> 人工序列
<400> 4
acgttggatg ataggaatrt twcgaaagcc 30
<210> 5
<211> 30
<212> DNA
<213> 人工序列
<400> 5
acgttggatg taaagactgg gagyagttgg 30
<210> 6
<211> 30
<212> DNA
<213> 人工序列
<400> 6
acgttggatg gaccaattta tgrctacagc 30
<210> 7
<211> 30
<212> DNA
<213> 人工序列
<400> 7
acgttggatg tccgtgatga ayttaggagg 30
<210> 8
<211> 30
<212> DNA
<213> 人工序列
<400> 8
acgttggatg tacaacccct arcctaagag 30
<210> 9
<211> 30
<212> DNA
<213> 人工序列
<400> 9
acgttggatg tctatcttgc gtracatggg 30
<210> 10
<211> 30
<212> DNA
<213> 人工序列
<400> 10
acgttggatg tactgagagt gargggtttc 30
<210> 11
<211> 30
<212> DNA
<213> 人工序列
<400> 11
acgttggatg tctatcttgc yttacatggg 30
<210> 12
<211> 30
<212> DNA
<213> 人工序列
<400> 12
acgttggatg gcatagtata tagygatggg 30
<210> 13
<211> 30
<212> DNA
<213> 人工序列
<400> 13
acgttggatg gcatagtata tagygatggg 30
<210> 14
<211> 30
<212> DNA
<213> 人工序列
<400> 14
acgttggtag gttgtattgc tgttrtaatg 30
<210> 15
<211> 30
<212> DNA
<213> 人工序列
<400> 15
acgttggatg agcacagggc cycaataatg 30
<210> 16
<211> 30
<212> DNA
<213> 人工序列
<400> 16
acgttggatg tgaagtagat arggcagcac 30
<210> 17
<211> 30
<212> DNA
<213> 人工序列
<400> 17
acgttggatg cacttcactg caygacatag 30
<210> 18
<211> 30
<212> DNA
<213> 人工序列
<400> 18
acgttggatg ctgtctctat acacracaaa 30
<210> 19
<211> 30
<212> DNA
<213> 人工序列
<400> 19
acgttggatg tggcagctgt gtgrattctc 30
<210> 20
<211> 30
<212> DNA
<213> 人工序列
<400> 20
acgttggatg aaacaactgg yagtcagagg 30
<210> 21
<211> 30
<212> DNA
<213> 人工序列
<400> 21
acgttggatg ccaactcaag atrcagaaag 30
<210> 22
<211> 30
<212> DNA
<213> 人工序列
<400> 22
acgttggatg acccataccc agaygaagag 30
<210> 23
<211> 30
<212> DNA
<213> 人工序列
<400> 23
acgttggatg gaactgtygg agtrtgagag 30
<210> 24
<211> 29
<212> DNA
<213> 人工序列
<400> 24
acgttggatg gtatgytgtc ctytccttc 29
<210> 25
<211> 29
<212> DNA
<213> 人工序列
<400> 25
acgttggatg ttgtaacggg ttrggtcgg 29
<210> 26
<211> 30
<212> DNA
<213> 人工序列
<400> 26
acgttggtag tgtctgtttg tctytgcgcc 30
<210> 27
<211> 30
<212> DNA
<213> 人工序列
<400> 27
acgttggatg tcctagtgtg gargacctac 30
<210> 28
<211> 30
<212> DNA
<213> 人工序列
<400> 28
acgttggatg acaccagaga atragaggag 30
<210> 29
<211> 30
<212> DNA
<213> 人工序列
<400> 29
acgttggatg acacagtcac ttgrggtcag 30
<210> 30
<211> 30
<212> DNA
<213> 人工序列
<400> 30
acgttggatg gtttggtaga tgrcaaacgg 30
<210> 31
<211> 30
<212> DNA
<213> 人工序列
<400> 31
acgttggatg gacaaacgag tgytggtatc 30
<210> 32
<211> 30
<212> DNA
<213> 人工序列
<400> 32
acgttggatg aacggtgttt gtygcagttc 30
<210> 33
<211> 29
<212> DNA
<213> 人工序列
<400> 33
acgttggatg gggtatgcac ygttacgag 29
<210> 34
<211> 30
<212> DNA
<213> 人工序列
<400> 34
acgttggatg cctgatgatg tgarggatgg 30
<210> 35
<211> 30
<212> DNA
<213> 人工序列
<400> 35
acgttggatg accgcaacat ggcrgatttg 30
<210> 36
<211> 30
<212> DNA
<213> 人工序列
<400> 36
acgttggatg acacctcyca gttcygattg 30
<210> 37
<211> 30
<212> DNA
<213> 人工序列
<400> 37
acgttggatg cagagcagta atgrctgaag 30
<210> 38
<211> 30
<212> DNA
<213> 人工序列
<400> 38
acgttggatg aacagggayt agcccaacac 30
<210> 39
<211> 31
<212> DNA
<213> 人工序列
<400> 39
acgttggatg caattttcac aacgatacga c 31
<210> 40
<211> 30
<212> DNA
<213> 人工序列
<400> 40
acgttggatg attgttggtg gaagtgcctg 30
<210> 41
<211> 30
<212> DNA
<213> 人工序列
<400> 41
acgttggatg ttccattcat gggttgagtc 30
<210> 42
<211> 30
<212> DNA
<213> 人工序列
<400> 42
acgttggatg caaaatggtt gaaacacccc 30
<210> 43
<211> 30
<212> DNA
<213> 人工序列
<400> 43
acgttggatg taagcgacac gagaaggaac 30
<210> 44
<211> 30
<212> DNA
<213> 人工序列
<400> 44
acgttggatg gggtttgtcc tgtcttatgc 30
<210> 45
<211> 31
<212> DNA
<213> 人工序列
<400> 45
acgttggatg ggttatatac ttacggttgg g 31
<210> 46
<211> 30
<212> DNA
<213> 人工序列
<400> 46
acgttggatg tctctacgga catcatcacc 30
<210> 47
<211> 30
<212> DNA
<213> 人工序列
<400> 47
acgttggatg ctgggttttc ttgatacctg 30
<210> 48
<211> 30
<212> DNA
<213> 人工序列
<400> 48
acgttggatg gggccacgta atgagacaaa 30
<210> 49
<211> 30
<212> DNA
<213> 人工序列
<400> 49
acgttggatg tggcgttagt atgygtgtcg 30
<210> 50
<211> 30
<212> DNA
<213> 人工序列
<400> 50
acgttggatg ttgatccaag aaagyacccg 30
<210> 51
<211> 30
<212> DNA
<213> 人工序列
<400> 51
acgttggatg tgctagccga gtagrgttgg 30
<210> 52
<211> 29
<212> DNA
<213> 人工序列
<400> 52
acgttggatg tgcacggtct argagacct 29
<210> 53
<211> 30
<212> DNA
<213> 人工序列
<400> 53
acgttggatg gtaatacata cygacaatgg 30
<210> 54
<211> 30
<212> DNA
<213> 人工序列
<400> 54
acgttggatg actactccrt gactttgtgg 30
<210> 55
<211> 30
<212> DNA
<213> 人工序列
<400> 55
acgttggatg ccaatttgga aaygaccagc 30
<210> 56
<211> 30
<212> DNA
<213> 人工序列
<400> 56
acgttggatg ccacacaatc amcacctgcc 30
<210> 57
<211> 30
<212> DNA
<213> 人工序列
<400> 57
acgttggatg gtgaactatt gtygaaaggg 30
<210> 58
<211> 30
<212> DNA
<213> 人工序列
<400> 58
acgttggatg gcttttcttc tgggtaytac 30
<210> 59
<211> 31
<212> DNA
<213> 人工序列
<400> 59
acgttggatg gcttttctga asggytctaa g 31
<210> 60
<211> 30
<212> DNA
<213> 人工序列
<400> 60
acgttggatg gtaaacaatg cagyaccagg 30
<210> 61
<211> 30
<212> DNA
<213> 人工序列
<400> 61
acgttggatg aaaagaccag ragtcacagc 30
<210> 62
<211> 30
<212> DNA
<213> 人工序列
<400> 62
acgttggatg tatgaatccr cctatycccc 30
<210> 63
<211> 30
<212> DNA
<213> 人工序列
<400> 63
acgttggatg tgtacctccr ttgctattcg 30
<210> 64
<211> 30
<212> DNA
<213> 人工序列
<400> 64
acgttggatg ttaagaatyc cattagagcg 30
<210> 65
<211> 30
<212> DNA
<213> 人工序列
<400> 65
acgttggatg gcctggtcga yagraccaat 30
<210> 66
<211> 30
<212> DNA
<213> 人工序列
<400> 66
acgttggatg gggttcatga cytttagctg 30
<210> 67
<211> 30
<212> DNA
<213> 人工序列
<400> 67
acgttggatg ataaaatrcc sggtcttacc 30
<210> 68
<211> 30
<212> DNA
<213> 人工序列
<400> 68
acgttggatg cttccaccta crgggatgag 30
<210> 69
<211> 30
<212> DNA
<213> 人工序列
<400> 69
acgttggatg caggcaacaa rccagttttc 30
<210> 70
<211> 30
<212> DNA
<213> 人工序列
<400> 70
acgttggatg agtggtgatg gcattagtgg 30
<210> 71
<211> 30
<212> DNA
<213> 人工序列
<400> 71
acgttggatg actgtgcttg acctaggaac 30
<210> 72
<211> 30
<212> DNA
<213> 人工序列
<400> 72
acgttggatg aaagcagcac ttgactagag 30
<210> 73
<211> 30
<212> DNA
<213> 人工序列
<400> 73
acgttggatg cggtgtttgc agatttggac 30
<210> 74
<211> 30
<212> DNA
<213> 人工序列
<400> 74
acgttggatg tgaatagcca aggtgagcgg 30
<210> 75
<211> 15
<212> DNA
<213> 人工序列
<400> 75
ctcacttcgc ttcac 15
<210> 76
<211> 20
<212> DNA
<213> 人工序列
<400> 76
gatcccgcac ttgtattccc 20
<210> 77
<211> 24
<212> DNA
<213> 人工序列
<400> 77
gagcctacag cctcctagta caaa 24
<210> 78
<211> 15
<212> DNA
<213> 人工序列
<400> 78
gcgggagcca aagta 15
<210> 79
<211> 22
<212> DNA
<213> 人工序列
<400> 79
gacaccatat catctaattg tt 22
<210> 80
<211> 24
<212> DNA
<213> 人工序列
<400> 80
tgcctaggcc accttctcag tcca 24
<210> 81
<211> 20
<212> DNA
<213> 人工序列
<400> 81
tcctgctgtt ctaatgttgt 20
<210> 82
<211> 19
<212> DNA
<213> 人工序列
<400> 82
ctgacacgca gtacaaata 19
<210> 83
<211> 21
<212> DNA
<213> 人工序列
<400> 83
aagttgtgta tattgcaaga c 21
<210> 84
<211> 16
<212> DNA
<213> 人工序列
<400> 84
gtttctccct ctccaa 16
<210> 85
<211> 18
<212> DNA
<213> 人工序列
<400> 85
ctccgatcca gaaagcct 18
<210> 86
<211> 18
<212> DNA
<213> 人工序列
<400> 86
aggagagcct gtctgaat 18
<210> 87
<211> 16
<212> DNA
<213> 人工序列
<400> 87
tgtaatacgc gtccca 16
<210> 88
<211> 22
<212> DNA
<213> 人工序列
<400> 88
tgtagcctac actttggcca cc 22
<210> 89
<211> 23
<212> DNA
<213> 人工序列
<400> 89
ttcttacttt gtattgttct acc 23
<210> 90
<211> 21
<212> DNA
<213> 人工序列
<400> 90
gtaacacatc ccacccattt a 21
<210> 91
<211> 18
<212> DNA
<213> 人工序列
<400> 91
acccaacatc gcttcaat 18
<210> 92
<211> 18
<212> DNA
<213> 人工序列
<400> 92
cgattctgcg attactag 18
<210> 93
<211> 15
<212> DNA
<213> 人工序列
<400> 93
tgtctgaagc caacc 15
<210> 94
<211> 16
<212> DNA
<213> 人工序列
<400> 94
cgatacgacc aaccat 16
<210> 95
<211> 24
<212> DNA
<213> 人工序列
<400> 95
ggttttgatc tactcaaatt gatt 24
<210> 96
<211> 25
<212> DNA
<213> 人工序列
<400> 96
tcttaaaata accaaaactc aaacc 25
<210> 97
<211> 18
<212> DNA
<213> 人工序列
<400> 97
atcatcacca aagaagcc 18
<210> 98
<211> 25
<212> DNA
<213> 人工序列
<400> 98
gcaaaaatga acaacacata gatac 25
<210> 99
<211> 20
<212> DNA
<213> 人工序列
<400> 99
ctggggagag ccatagtggt 20
<210> 100
<211> 22
<212> DNA
<213> 人工序列
<400> 100
aagggttgtg gtactgcctg at 22
<210> 101
<211> 15
<212> DNA
<213> 人工序列
<400> 101
gtgggctgca tgttg 15
<210> 102
<211> 17
<212> DNA
<213> 人工序列
<400> 102
gggtctggaa aggtgaa 17
<210> 103
<211> 20
<212> DNA
<213> 人工序列
<400> 103
agggcagtca tcataaaggt 20
<210> 104
<211> 21
<212> DNA
<213> 人工序列
<400> 104
gattgctggg tgtattgaaa a 21
<210> 105
<211> 19
<212> DNA
<213> 人工序列
<400> 105
caatagtagg ctgacgact 19
<210> 106
<211> 16
<212> DNA
<213> 人工序列
<400> 106
tgagctggtt ggattg 16
<210> 107
<211> 21
<212> DNA
<213> 人工序列
<400> 107
cctatttgtg aatggcattg t 21
<210> 108
<211> 15
<212> DNA
<213> 人工序列
<400> 108
tcaaggtgga gtggg 15
<210> 109
<211> 19
<212> DNA
<213> 人工序列
<400> 109
tgaggtcatg gataaatcg 19
<210> 110
<211> 20
<212> DNA
<213> 人工序列
<400> 110
gcccaagtga gacattttag 20
<210> 111
<211> 17
<212> DNA
<213> 人工序列
<400> 111
agggttctga cctgaag 17
- 还没有人留言评论。精彩留言会获得点赞!