一种基于SSR分子标记构建甘薯核心种质资源库的方法与流程
本发明属于植物种质资源评价技术领域,具体涉及一种基于SSR分子标记构建甘薯核心种质资源库的方法。
背景技术:
丰富的遗传资源是新品种选育等各项研究工作的基础。但是,由于实际工作中真正利用的资源数量有限,而资源数量太大不利于对种质资源进行全面的保存与利用。为此,Frankle于1984年提出了核心种质的概念,即通过科学的方法,从整个种质资源中选出能以最小的资源数量和遗传重复最大限度保留整个资源遗传多样性的那部分资源,以此来深入评价和有效保护利用整体种质资源,并与Brown将其进一步发展,而原种质中除核心种质外的资源称为保留种质,被用作核心种质的候补资源。
核心种质的构建有利于对种质资源进行有效的研究、评价、利用与保护,被许多国际育种工作者所关注。到目前为止,国内外己对50多个物种进行了核心种质的构建。中国在核心种质构建方面的研究起步较晚,但已在如绿豆、苜蓿、大豆、小麦、水稻、板栗、野生板栗、野苹果、葡萄、李、桃、梨、果梅、普通杏、柚类、核桃、桃、柿、石榴和枣树等作物上进行了研究。目前,关于甘薯核心种质的构建仅有少量报道,且是基于表型性状数据研究构建方法。然而,甘薯农艺性状多为数量性状,其表现型值易于受环境条件影响而导致测量数据不准确,进而会影响核心种质构建结果的有效性。
简单重复序列(Simple Sequence Repeat,SSR),是一种基于PCR技术的分子标记,它能在分子水平上检测个体的核苷酸差异,其具有多个优点:
(1)在真核生物基因组中分布广、多态性高;
(2)SSR产物进行凝胶电泳分离时碱基分辨率高、共显性标记遗传信息量大;
(3)呈孟德尔式遗传,稳定性好,技术要求低,成本低廉,可重复性高。
基于上述优点,采用SSR标记构建甘薯核心种质,可克服田间农艺性状多态性低和易受环境影响的缺点。
截止目前,尚未见有基于SSR分子标记的甘薯核心种质构建方法的相关报道。
技术实现要素:
本发明目的是提供一种基于SSR分子标记的甘薯核心种质资源库的构建以及结合表型性状进行评价的方法,该方法能够高效、可靠的建立52份甘薯种质资源的核心种质,对甘薯种质资源的保存、评价及创新利用均具有重要意义。
为解决上述技术问题,本发明采用以下技术方案:
一种基于SSR分子标记构建甘薯核心种质资源库的方法,包括如下步骤:
(1)采用备份保存方式进行甘薯种质资源的繁育和保存;
(2)采用植物基因组试剂盒进行甘薯种质资源DNA的提取,和琼脂糖凝胶电泳检测DNA质量,并利用微量核酸蛋白检测仪检测DNA浓度;
(3)甘薯SSR引物的合成及多态性引物的筛选,得到14对多态性好的甘薯SSR引物;
(4)甘薯每份种质资源的PCR扩增及带型记录;
(5)依据SSR标记的带型数据,采用两种取样方法和三种遗传距离构建核心种质;
(6)采用多态性位点数(NPL)、多态性位点百分率(PPB)、观测等位基因数(Na)、有效等位基因数(Ne)、Nei’s基因多样性指数(H)和香农信息指数(I)来评价原种质、核心种质和保留种质的遗传多样性,分别对核心种质与原种质及保留种质的各指标进行t检验,来评价核心种质的代表性;
(7)利用表型数据采用主坐标分析法以及均值差异百分率(MD)、方差差异百分率(VD)、极差符合率(CR)和变异系数变化率(VR)对构建的甘薯核心种质进行进一步的验证。
作为优选,步骤(3)中所述的14对多态性好的甘薯SSR引物分别为引物对CB283-F和CB283-R、引物对CB223-F和CB223-R、引物对CB200-F和CB200-R、引物对CB144-F和CB144-R、引物对CB141-F和CB141-R、引物对CB940-F和CB940-R、引物对IB3-F和IB3-R、引物对IB8-F和IB8-R、引物对IB10-F和IB10-R、引物对IB12-F和IB12-R、引物对IB13-F和IB13-R、引物对IB16-F和IB16-R、引物对IB19-F和IB19-R、引物对IB21-F和IB21-R,其分别具有如SEQ ID No.1至SEQ ID No.28所示的序列。
作为优选,步骤(4)中PCR扩增的反应体系为10uL,其中:2×Es Taq MasterMix(Dye)为5uL,引物5‘10uM和引物3‘10uM分别为0.4uL,模板DNA 100ng/uL为0.2uL,ddH2O补足至10uL。
作为优选,所述的2×Es Taq MasterMix(Dye)的终浓度为1×,所述的引物5‘10uM和引物3‘10uM的终浓度均为0.4uM,所述的模板DNA 100ng/uL是终浓度为20ng。
作为优选,步骤(4)中PCR扩增的反应程序为:94℃预变性5min;94℃变性45s,45-62℃退火30s,72℃延伸45s,35个循环;72℃终延伸10min。
与现有技术相比,本发明具有以下有益效果:
(1)本发明首次公开了一种基于SSR分子标记的甘薯核心种质资源库的构建及评价方法。首先从保存的甘薯种质资源顶端嫩叶中提取DNA,然后通过筛选出的多态性SSR引物进行PCR扩增,根据扩增结果记录各个种质资源的带型数据。对获得的SSR标记“0”、“1”数据进行分析,依据整体聚类取样各组种质均能进入核心种质的原则,采用位点优先取样法结合Jaccard遗传距离构建甘薯核心种质,同时利用表型数据进行评价确认。
(2)本发明的方法中SSR标记扩增带型清晰稳定,重复性好。同时,与基于农艺性状表型数据构建核心种质比较而言,基于分子标记数据构建甘薯核心种质,具有省时省力、稳定性好等特点。
(3)本发明的方法采用位点优先取样策略和Jaccard遗传距离构建的52份核心种质,能够以最小的资源份数最大限度地代表原种质的遗传多样性,代表性强,对甘薯核心种质资源的保存、评价与创新利用都具有重要意义。
附图说明
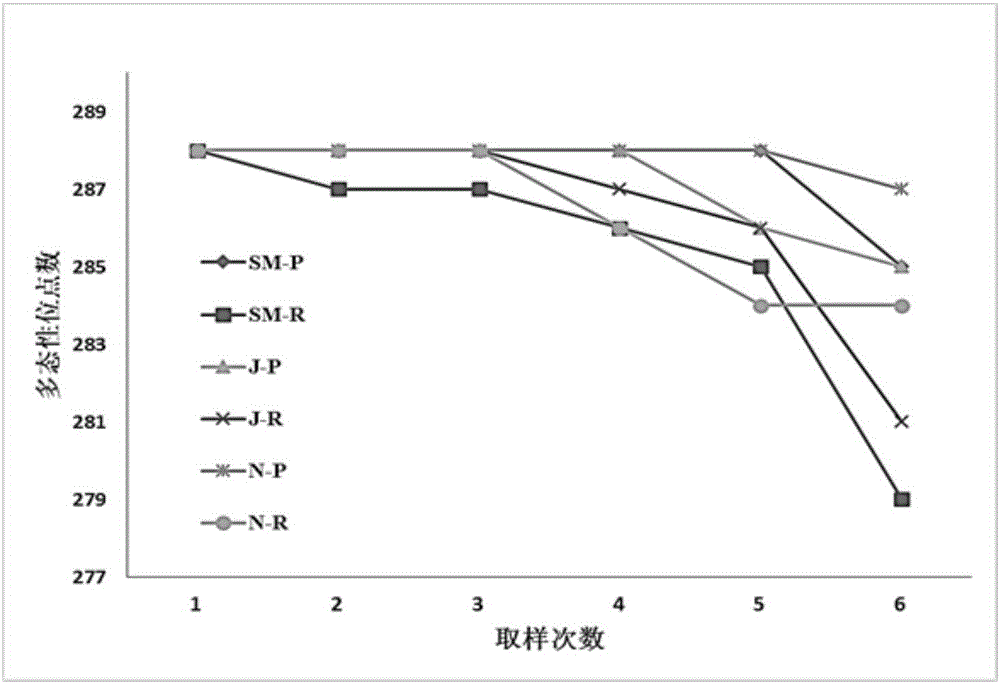
图1为不同取样条件下2种取样策略和3种遗传距离保留的多态性位点数;
其中:SM-SM遗传距离;J-Jaccard遗传距离;N-Nei&Li遗传距离;P-位点优先取样法;R-随机取样法。
图2为位点优先取样策略构建的核心种质与原种质的主坐标图;
其中,1-原种质,2-核心种质。
具体实施方式
下面结合具体实施例,对本发明作进一步详细的阐述,但本发明的实施方式并不局限于实施例表示的范围。这些实施例仅用于说明本发明,而非用于限制本发明的范围。此外,在阅读本发明的内容后,本领域的技术人员可以对本发明作各种修改,这些等价变化同样落于本发明所附权利要求书所限定的范围。
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。下述实施例中所使用的材料、试剂等,如无特殊说明,均可从商业途径得到。
实施例1:
1.材料与方法
1.1材料
378份甘薯种质资源分别来自于国内外甘薯种植区,分为4组,其中Ⅰ组来源于国内南方薯区(297份),Ⅱ组来源于国内长江中下游薯区(51份),Ⅲ组来源于国内北方薯区(23份),Ⅳ组来源于国外(7份),以上材料均种植于广西农业科学院明阳基地甘薯种质资源圃中。
1.2方法
1.2.1材料的繁育和保存
甘薯种质资源均通过大田和温室大棚两种方式双份保存于资源圃中。
1.2.2甘薯种质资源基因组DNA提取
在广西农业科学院明阳基地甘薯种质资源圃中,选取378份甘薯种质资源(表1)植株的顶部嫩叶,利用康为世纪的新型植物基因组DNA提取试剂盒提取DNA。提取后的DNA通过琼脂糖凝胶电泳检测质量后,利用Nanodrop ND-2000超微量核酸蛋白测定仪检测DNA浓度,并稀释到所需浓度。
1.2.3 SSR引物的合成及多态性引物的筛选
通过查阅文献找寻甘薯相关SSR引物序列,共查找到目标SSR引物72对,由生工生物工程(上海)股份有限公司合成。利用表型性状差异明显的两份种质资源对72份引物进行筛查,筛选出多态性好的14对引物用于所有种质资源材料的PCR扩增,多态性引物见(表3)。
1.2.4甘薯每份种质资源的PCR扩增及带型记录
SSR-PCR反应采用康为世纪的2×Es Taq MasterMix(Dye)试剂进行PCR反应,扩增在BIO-RAD S 1000PCR仪上进行,扩增产物采用8%聚丙烯酰胺凝胶电泳分离,采用硝酸银进行银染,并记录带型数据。具体PCR反应体系见表2和表3:
表1 SSR-PCR反应体系
表2 PCR反应程序
1.2.5依据SSR标记的带型数据,构建核心种质并进行构建方法筛选和评价
1.2.5.1遗传距离
采用NTSYSpc version 2.10e软件使用UPGMA聚类法分别对SM遗传距离、Jaccard遗传距离和Nei&Li遗传距离进行聚类。
1.2.5.2取样策略
采用位点优先取样策略和随机取样策略对比构建核心种质。位点优先取样策略是指在聚类图中,最先聚在一起的各组株系中,优先选择具有等位基因数最多的株系,若各组的两个株系稀有等位基因数目相等,则优先选择具有最小等位基因频率值的株系,若此数值仍然相同,那么进行随机选择,被选择的株系再次进行聚类取样;随机取样策略是指在聚类图中,最先聚在一起的各组株系中,随机保留某个株系,若某个组内只有一个株系,则直接被保留,被保留的株系再次进行聚类取样。
1.2.5.3取样原则
在进行整体聚类抽样时应保证每个采样点都有种质被抽取,如此进行逐步聚类直到其中某一组中再次聚类时无种质被抽取。
1.2.5.4核心种质的评价
原种质、核心种质和保留种质的遗传多样性采用多态性位点数(NPL)、多态性位点百分率(PPB)、观测等位基因数(Na)、有效等位基因数(Ne)、Nei’s基因多样性指数(H)和香农信息指数(I)来评价,以上参数采用POPGENE version 1.32软件进行计算,利用DPS 14.50软件分别对核心种质与原种质及保留种质的各指标进行t检验,来评价核心种质的代表性。
1.2.5.5核心种质的确认
核心种质的确认是利用表型数据及采用NTSYSpc version 2.10e软件通过主坐标轴分析法来实现的,对核心种质与原种质间表型性状的均值和方差分别进行F检验和t检验,用均值差异百分率(MD)、方差差异百分率(VD)、极差符合率(CR)和变异系数变化率(VR)来评价核心种质的代表性。
MD=(St/n)×100%
其中n为性状总数,St为原种质与核心种质进行t测验时在0.05水平上均值差异显著的性状个数;
VD=(SF/n)×100%
其中n为性状总数,SF为原种质与核心种质进行F测验时得到在0.05水平上方差差异显著的性状个数;
其中N为性状总数,RI(i)为原种质第i个性状的极差,RC(i)为核心种质第i个性状的极差;
其中n为性状总数,CVI(i)是原种质第i个性状的变异系数,CVC(i)为核心种质第i个性状的变异系数;
2.结果与分析
2.1 14对SSR引物的遗传多样性分析
表3 14对SSR引物的多态性分析
从表3可知,14对SSR引物扩增的条代数为12-32条,遗传多样性指数变化范围在0.2580-0.4164之间,香农信息指数变化范围在0.4075-0.6027之间。
2.2取样方法比较及候选核心种质的确认
从图1可以看出,与采用随机取样法相比较,随着整体聚类取样次数的增加,采用位点优先取样方法所构建的核心种质其多态性位点数减少的较少。
表4第5次取样后甘薯种质资源的遗传多样性比较
结合图1和表4的数据分析来看,SM遗传距离、Jaccard遗传距离和Nei&Li遗传距离的有效等位基因数、Nei’s基因多样性指数和香农信息指数均为位点优先取样策略高于随机取样策略。所以,位点优先取样策略要优于随机取样策略。同时考虑到各组取样数的问题,根据整体聚类取样原则,在第六次取样时,第Ⅳ组的甘薯种质未能进入核心种质,因此选取位点优先取样策略第五次取样后的3个种质作为候选核心种质进行进一步的比较。
2.3候选核心种质的评价
表5原种质与3个候选核心种质t检验结果
三个候选核心种质SM-P、J-P和N-P的取样数分别为52份、52份和53份。从表5遗传多样性各指标与原种质t检验结果可知,三个候选核心种质在观测等位基因数、有效等位基因数、Nei’s基因多样性指数和香农信息指数与原种质均无显著差异。但J-P核心种质的有效等位基因数、Nei’s基因多样性指数和香农信息指数与原种质的差值均数最大。
由此可见,采用位点优先取样策略和Jaccard遗传距离选取52份种质时,构建的核心种质遗传多样性最丰富,能够以最小的资源份数最大限度地代表原种质的遗传多样性,初步确定为甘薯种质构建核心种质的最优取样方法和取样量。
表6原种质、核心种质(J-P)和保留种质遗传多样性对比
从表6可知,核心种质保留了原种质13.76的种质,保留种质保留了原种质86.24%的种质,核心种质的观测等位基因数、多态性位点数、多态性位点百分率的保留率分别与保留种质持平,但核心种质的有效等位基因数、基因多样性指数、香农信息指数的保留率分别高于保留种质,可见核心种质能很好的代表原种质。
表7核心种质(J-P)与保留种质t检验结果
同时,对核心种质和保留种质的观测等位基因数、有效等位基因数、Nei’s基因多样性指数和香农信息指数分别作t测验发现(表7),核心种质与保留种质的差异均不显著,而有效等位基因数、Nei’s基因多样性指数和香农信息指数均略大于保留种质,所以应优先考虑核心种质。
2.4利用7对表型性状数据对核心种质进行确认
采用主坐标和7个表型性状对位点优先取样策略和Jaccard遗传距离构建的核心种质的代表性进行确认(图2),核心种质遍布于整个主坐标图,说明了核心种质的代表性。对核心种质和原种质的各表型性状指标分别作t测验(表8),除叶片宽外,核心种质其余各性状指标与原种质均无显著差异。从表9可以看出,核心种质的均值差异百分率小于1%,极差符合率和变异系数变化率分别为89.69%和100.08%,均大于80%,所以所选核心种质能够代表原种质的遗传多样性。研究结果从表型水平上进一步证明了位点优先取样策略和Jaccard遗传距离是一种较适宜的甘薯核心种质构建方法。
表8原种质与核心种质表型性状的遗传变异
注:t0.05=1.9600,t0.01=2.5758;F0.05=2.5700,F0.01=3.9400
表9原种质与核心种质性状差异百分率
3.结论
本发明首次将SSR分子标记技术用于甘薯核心种质的构建与评价。
表10 52份甘薯核心种质资源
结果如表10所示,采用位点优先取样策略和Jaccard遗传距离构建的52份核心种质,能够以最小的资源份数最大限度地代表原种质的遗传多样性,代表性强,对甘薯核心种质资源的保存、评价与创新利用都具有重要意义。
SEQUENCE LISTING
<110> 广西壮族自治区农业科学院玉米研究所
<120> 一种基于SSR分子标记构建甘薯核心种质资源库的方法
<130> ZYWS
<160> 28
<170> PatentIn version 3.3
<210> 1
<211> 22
<212> DNA
<213> 人工序列
<400> 1
ccaattatag gcttcactcc ag 22
<210> 2
<211> 19
<212> DNA
<213> 人工序列
<400> 2
ttaatgcagg caagcacgc 19
<210> 3
<211> 23
<212> DNA
<213> 人工序列
<400> 3
caatgctgag gctcctctga atc 23
<210> 4
<211> 23
<212> DNA
<213> 人工序列
<400> 4
aggaagttcc cacagttctg acc 23
<210> 5
<211> 20
<212> DNA
<213> 人工序列
<400> 5
aatccaaaga gaaaatggcg 20
<210> 6
<211> 21
<212> DNA
<213> 人工序列
<400> 6
gcagatcaga gatgaccgaa g 21
<210> 7
<211> 20
<212> DNA
<213> 人工序列
<400> 7
cgtcctgaat aaattgatcg 20
<210> 8
<211> 20
<212> DNA
<213> 人工序列
<400> 8
agcgagaggt ggttcttctg 20
<210> 9
<211> 18
<212> DNA
<213> 人工序列
<400> 9
agaagtgaag gaaagggc 18
<210> 10
<211> 20
<212> DNA
<213> 人工序列
<400> 10
gttgctgttc attgttgttc 20
<210> 11
<211> 18
<212> DNA
<213> 人工序列
<400> 11
atccctattt cagctcac 18
<210> 12
<211> 19
<212> DNA
<213> 人工序列
<400> 12
agaccttcac tgcctttcc 19
<210> 13
<211> 20
<212> DNA
<213> 人工序列
<400> 13
gtagagttga agagcgagca 20
<210> 14
<211> 20
<212> DNA
<213> 人工序列
<400> 14
ccatagaccc attgatgaag 20
<210> 15
<211> 18
<212> DNA
<213> 人工序列
<400> 15
ggcgacacct tagagtat 18
<210> 16
<211> 16
<212> DNA
<213> 人工序列
<400> 16
caccccctat tcacaa 16
<210> 17
<211> 19
<212> DNA
<213> 人工序列
<400> 17
ctacgatctc tcggtgacg 19
<210> 18
<211> 19
<212> DNA
<213> 人工序列
<400> 18
cagcttctcc actccctac 19
<210> 19
<211> 20
<212> DNA
<213> 人工序列
<400> 19
gatcgaggag aagctccaca 20
<210> 20
<211> 20
<212> DNA
<213> 人工序列
<400> 20
gccggcaaat taagtccatc 20
<210> 21
<211> 20
<212> DNA
<213> 人工序列
<400> 21
gtaccgagcc agacaggatg 20
<210> 22
<211> 20
<212> DNA
<213> 人工序列
<400> 22
cctttgggat tggaacacac 20
<210> 23
<211> 20
<212> DNA
<213> 人工序列
<400> 23
gacttccttg gtgtagttgc 20
<210> 24
<211> 18
<212> DNA
<213> 人工序列
<400> 24
agggttaagc gggagact 18
<210> 25
<211> 19
<212> DNA
<213> 人工序列
<400> 25
ggctagtgga gaaggtcaa 19
<210> 26
<211> 20
<212> DNA
<213> 人工序列
<400> 26
agaagtagaa ctccgtcacc 20
<210> 27
<211> 20
<212> DNA
<213> 人工序列
<400> 27
gacagtctcc ttctcccata 20
<210> 28
<211> 18
<212> DNA
<213> 人工序列
<400> 28
ctgaagctcg tcgtcaac 18
- 还没有人留言评论。精彩留言会获得点赞!