一种适配体组合文库、其构建方法及用途与流程
本发明涉及分子生物学、分析化学以及组合化学领域。具体而言,本发明涉及适配体组合文库及其构建方法,还涉及使用所述的适配体组合文库筛选评价所需适配体的方法以及由该方法筛选得到的适配体。本发明还涉及所述筛选得到的适配体的用途及含有所述筛选得到的适配体的试剂盒。利用本发明提供的适配体组合文库及筛选方法筛选得到的适配体亲和力更优,并且无冗余序列。
背景技术:
寡核苷酸适配体(Oligonucleotide aptamer),通常经指数富集的配基系统进化技术(Systematic Evolution of Ligands by Exponential Enrichment,SELEX)筛选得到(Nature 1990,346;Science 1990,249(4968):505-510),一般为一段短的单链寡核苷酸序列(ssDNA或RNA),通过折叠成一定的三维结构,经空间构型互补与靶分子形成高亲和力、高特异性结合,又称为“化学抗体”(Oncotarget 2016,7(12):13446-13463)。
对于通过SELEX技术筛选得到的适配体又称全长适配体,一般均包含引物序列和随机序列,长度一般在70~130碱基(nt)之间。全长适配体和靶分子的关键结合位点一般并不明确,这导致了全长适配体不适合作为功能性识别元件。许多经SELEX技术筛选出的适配体,其引物区与随机区产生碱基配对,某些引物区参与了特异性识别,或者随机区序列较长,存在冗余碱基序列。因此,对于全长适配体序列,需对其进行结构剪裁,以便于得到既保持结合活性、又仅有适当长度(10-40nt)的适配体,这样的适配体更适合作为功能性识别元件。为了得到这样的适配体,需要精确界定对识别功能产生贡献的适配体序列(或结构域)。
因此,适配体后筛选(post-SELEX)始终是研究热点之一,已发展了结构剪裁、化学修饰、多价组合、定点突变等方法,从中寻找亲和性更高、特异性更强、序列更短的适配体,但多数尚限于以试错法(J.Biol.Chem.2001,276(54):48644-48654)进行尝试。
试错法一般是针对末轮文库的富集序列,在共有的二级结构及计算所得较低自由能(ΔG)的基础上,依据一些核酸二级结构的经验性判断,对适配体序列进行较为随机的剪裁或定点突变,例如,上官等采用试错法,对其筛选得到的呈环1-茎1-环2-茎2-环3二级结构的肺癌细胞适配体Sgc8,进行结构剪裁,逐级截掉环1、茎1、环2等,最终得到茎2-环3的适配体序列Sgc8c,基本保留了原Sgc8的亲和力(Chem.Bio.Chem.2007,8:603-606)。又例如,发明人之前针对重组人促红细胞生成素-α筛选得到的全 长适配体828,经截掉全部随机序列后得到的828r,较原来828的亲和力稍优(CN102191250A;Bioorg.Med.Chem.2010,18,8016-8025)。
上述试错法效率较低,往往筛选得到许多无效序列。例如,Ferreira-Bravo等采用试错法对经SELEX技术筛选得到的HIV反转录蛋白的DNA适配体FA1进行碱基替换,仅将适配体的四条茎部互补碱基序列全部简单替换为G-C碱基对,结果发现该替换对亲和活性无明显影响;同时对于适配体的四个环部结构域,仅进行简单随机剪裁,亦未发现亲和活性优的剪裁序列(Nucleic Acids Res.2015,43(20):9587-9599)。又例如,Esposito等针对15nt长的凝血酶适配体TBA,创建了14条在不同位点极性反转(自5’-3’翻转为3’-5’)序列,虽可进行结构阐释,但并未发现活性更优的适配体(Nucleosides Nucleotides Nucleic Acids 2007,26(8-9):1145-1149)。上述方法很难在有限的序列范围内,得到亲和活性更优、特异性更强的优化适配体(Improved aptamer)序列。
还有Nonaka等学者将基于遗传算法的虚拟筛选引入post-SELEX领域,赋予下一代文库中高亲和力序列更高的出现几率,组合随机突变位点,从3代50多条等长突变序列里筛选到较之原VEGF适配体序列的亲和性高10倍的适配体(Anal.Chem.2013,85,1132-1137)。但仍无法回答适配体序列是否保守的问题,也无法确定那些是关键碱基。说明当前在post-SELEX中普遍沿用的试错法等进行结构剪裁或定点突变的方法,工作效率较低、难度较大,尚无系统的理论指导和较好的解决方案。
在筛选和优化适配体时,需要表征和评价适配体与靶分子间亲和力。目前,用于表征和评价适配体与靶分子间亲和力的方法,主要包括表面等离子体共振法(Surface Plasmon Resonance,SPR)、生物膜干涉法(Bio-Layer Interferometry,BLI)、等温滴定量热法(Isothermal Calorimetry,ITC)、凝胶电泳淌度变动分析法(Electrophoretic Mobility Shift Assay,EMSA)、微量热泳动法(Microscale Thermophoresis,MST)、后向散射干涉法(Backscattering Interferometry,BSI)以及荧光各向异性法(Fluorescence polarization,FP)等,依据结合作用前后的特征值变化来判定适配体与靶分子间亲和力。EMSA、FP、MST均需对靶分子或配体进行荧光或放射性标记;ITC属于免标记分析技术,但灵敏度较低、需用样品量较大;SPR、BLI、BSI和基于分子自发荧光的MST等则因其免标记、灵敏度高等备受关注。SPR和BLI除了能够提供亲和力数据外,还可以同时提供动力学数据,可实现高通量测定。SPR和BLI均需对靶分子进行固定,通过读取表面固定后的靶分子和溶液中的配体间作用后的表面等离子体共振信号强度的变化、或表面厚度的增加而实施测定。
在使用SPR技术时,需要对靶分子的固定方式、相互作用的溶液体系、相互作用后的再生条件等关键因素进行考察和优化,在建立合适、稳定、高灵敏的SPR方法体系后,方能进行后续的适配体序列筛选评价等工作。
为了筛选、优化亲和力更优、无冗余序列的最短适配体活性序列,需要找寻并开 发合适的裁剪方法对全长适配体(或待优化适配体序列)进行结构剪裁,还需要建立合适、稳定、高灵敏的表征和评价方法,从而能够高效地评价所剪裁序列和靶分子间的亲和力。
技术实现要素:
本发明的一个目的是提供一种构建适配体组合文库的方法。
本发明的另一个目的是提供一种基于上述构建适配体组合文库的方法构建的适配体组合文库。
本发明的又一个目的是提供一种通过上述构建适配体组合文库的方法所构建的适配体组合文库筛选适配体的方法。
本发明的又一个目的是提供通过上述筛选方法筛选的适配体。
本发明的又一个目的是提供通过上述筛选方法所筛选的适配体的用途。
本发明的再一个目的是提供含有通过上述筛选方法所筛选的适配体的试剂盒。
本发明的再一个目的是提供含有通过上述筛选方法所筛选的适配体的组合物。
本发明的再一个目的是提供含有通过上述筛选方法所筛选的适配体的芯片。
为实现上述目的,本发明提供一种构建适配体组合文库的方法,包括以下步骤:
提供具有N个碱基的待优化适配体,以待优化适配体作为初始序列;
构建第一分子群,其包含n1个分子,所述n1个分子各自为初始序列的5’末端截短体,并且每一个分子相对于初始序列具有不同的5’末端截短长度;
构建第二分子群,其包含n2个分子,所述n2个分子各自为初始序列的3’末端截短体,并且每一个分子相对于初始序列具有不同的3’末端截短长度;
构建第三分子群,其包含n3个分子,所述n3个分子各自为初始序列的5’和3’末端同时截短的截短体,在一个分子中5’和3’末端截短长度相同,并且每一个分子相对于初始序列具有不同的末端截短长度;
任选地,构建第四分子群,其包含n4个分子,
当初始序列能够形成茎环结构时,所述n4个分子各自为初始序列的环部序列延长体,所述环部序列延长体不包括初始序列的茎部序列,在5’末端包含1~n4个第一序列(例如由A和G组成的序列,例如AG),且在3’末端包含1~n4个第二序列,所述第二序列是第一序列的反向互补序列(例如由C和T组成的序列,例如CT),在一个分子中5’末端包含的第一序列和3’末端包含的第二序列数量相同,并且每一个分子相对于初始序列具有不同的末端延长长度;
当初始序列不能形成茎环结构时,所述n4个分子各自为初始序列的延长体,所述延长体的5’末端包含1~n4个第一序列(例如由A和G组成的序列,例如AG),且在3’末端包含1~n4个第二序列,所述第二序列为第一序列的反向互补序列(例如由C和T组成的序列,例如CT),在一个分子中5’末端包含的第一序列和3’末端包含的第二序列数量相同, 并且每一个分子相对于初始序列具有不同的末端延长长度,
所述第一序列的长度可以为1个或多个碱基,例如,1、2、3或4个碱基;
其中,N为大于零的整数(例如,30~130之间的整数),
n1、n2、n3、n4各自独立地为大于零且小于或等于N的整数(例如,5~80之间的整数)。
在另一个优选的实施方案中,本发明所述的构建适配体组合文库的方法,其中,
第一分子群的构建方法为:从5’末端对初始序列进行截短,共进行n1次,每次截短1个或多个(例如,1、2、3、4个)碱基,由此获得n1个5’末端截短的分子,构成第一分子群;
第二分子群的构建方法为:从3’末端对初始序列进行截短,共进行n2次,每次截短1个或多个(例如,1、2、3、4个)碱基,由此获得n2个3’末端截短的分子,构成第二分子群;
第三分子群的构建方法为:从5’和3’末端同时对初始序列进行截短,共进行n3次,每次对5’和3’末端同时截短1个或多个(例如,1、2、3、4个)碱基,由此获得n3个5’和3’末端同时截短的分子,构成第三分子群;
第四分子群的构建方法为:
当初始序列能够形成茎环结构时,保持初始序列的环部序列不变,将茎部序列去除,在环部序列的5’末端添加第一序列(例如由A和G组成的序列,例如AG),且在环部序列的3’末端添加第二序列,所述第二序列为第一序列的反向互补序列(例如由C和T组成的序列,例如CT),重复进行n4次,由此获得n4个5’和3’末端同时延长的分子,构成第四分子群;
当初始序列不能形成茎环结构时,在初始序列的5’末端添加第一序列(例如由A和G组成的序列,例如AG),且在3’末端添加第二序列,所述第二序列为第一序列的反向互补序列(例如由C和T组成的序列,例如CT),重复进行n4次,由此获得n4个5’和3’末端同时延长的分子,构成第四分子群。
在另一个优选的实施方案中,本发明所述的构建适配体组合文库的方法,其中第一分子群、第二分子群或第三分子群中长度最短的分子,其为具有10~40个碱基,例如,20~30个碱基。
在另一个优选的实施方案中,本发明所述的构建适配体组合文库的方法,其中所述待优化适配体为特异性结合EPO蛋白的适配体,例如,SEQ ID NO.1所示的全长适配体。
在另一个优选的实施方案中,本发明所述的构建适配体组合文库的方法,其中所述待优化适配体为特异性结合EPO蛋白的适配体,例如,SEQ ID NO.1所示的全长适配体,其中N=39,n1=9,n2=9,n3=9,n4=8,所述第一序列的长度为1个碱基、2个碱基或3个碱基。
本发明还提供一种适配体组合文库,包括:
第一分子群,其包含n1个分子,所述n1个分子各自为初始序列的5’末端截短体,并 且每一个分子相对于初始序列具有不同的5’末端截短长度;
第二分子群,其包含n2个分子,所述n2个分子各自为初始序列的3’末端截短体,并且每一个分子相对于初始序列具有不同的3’末端截短长度;
第三分子群,其包含n3个分子,所述n3个分子各自为初始序列的5’和3’末端同时截短的截短体,在一个分子中5’和3’末端截短长度相同,并且每一个分子相对于初始序列具有不同的末端截短长度;以及
任选地,第四分子群,其包含n4个分子,
当初始序列能够形成茎环结构时,所述n4个分子各自为初始序列的环部序列延长体,所述环部序列延长体不包括初始序列的茎部序列,在5’末端包含1~n4个第一序列(例如由A和G组成的序列,例如AG),且在3’末端包含1~n4个第二序列,所述第二序列是第一序列的反向互补序列(例如由C和T组成的序列,例如CT),在一个分子中5’末端包含的第一序列和3’末端包含的第二序列数量相同,并且每一个分子相对于初始序列具有不同的末端延长长度;
当初始序列不能形成茎环结构时,所述n4个分子各自为初始序列的延长体,所述延长体的5’末端包含1~n4个第一序列(例如由A和G组成的序列,例如AG),且在3’末端包含1~n4个第二序列,所述第二序列为第一序列的反向互补序列(例如由C和T组成的序列,例如CT),在一个分子中5’末端包含的第一序列和3’末端包含的第二序列数量相同,并且每一个分子相对于初始序列具有不同的末端延长长度,
所述第一序列的长度可以为1个或多个碱基,例如,1、2、3、4个碱基;
其中,N为大于零的整数(例如,30~130之间的整数),
n1、n2、n3、n4各自独立地为大于零且小于或等于N的整数(例如,5~80之间的整数),
所述初始序列为待优化适配体。
在一个优选的实施方案中,本发明所述的适配体组合文库,其中所述待优化适配体为特异性结合EPO蛋白的适配体,例如,SEQ ID NO.1所示的全长适配体。
在另一个优选的实施方案中,本发明所述的适配体组合文库,由SEQ ID NO.2~SEQ ID NO.36所示序列构成。
本发明还提供一种筛选适配体的方法,该方法使用前述本发明任意项所述的适配体组合文库筛选所需适配体,例如,能够特异性结合EPO蛋白的适配体。
在一个优选的实施方案中,本发明所述的适配体的筛选方法,其包括:
(1)提供适配体组合文库;
(2)将适配体组合文库中的每一个分子与靶蛋白(例如EPO蛋白)接触;
(3)检测适配体组合文库中每一个分子与靶蛋白之间的亲和力。
在一个优选的实施方案中,本发明所述的适配体的筛选方法,该方法采用表面等离子体共振法(SPR法)检测每一个分子与靶蛋白之间的亲和力。
在另一个优选的实施方案中,本发明所述的适配体筛选方法,包括以下步骤:
1)将靶蛋白固定于芯片(例如SA芯片)表面;
2)进行SPR动力学分析,进样,使所述适配体组合文库中的分子与固定在芯片表面的靶蛋白结合,生成复合物,进样结束,注入空白缓冲溶液,芯片上的复合物发生解离,得到各序列单点RU值变化曲线,根据响应值(RU值)判断待筛选适配体是否具有活性,RU值大于0的适配体具有活性;
3)对于具有活性的分子,进行多循环动力学分析(MCK分析),得到解离常数KD,根据KD数值的大小判断每个分子与靶蛋白之间的亲和力,KD数值越小,亲和力越强。
在另一个优选的实施方案中,本发明所述的适配体的筛选方法或任意上文适用的实施方式,其中,步骤1)是将所述靶蛋白偶联生物素后固定在芯片表面,例如,所述靶蛋白在氨基酸残基的氨基末端、羧基末端或糖基的唾液酸末端与生物素共价偶联,优选靶蛋白与生物素共价偶联的化学计量比为1:1。
在另一个优选的实施方案中,本发明所述的适配体的筛选方法或任意上文适用的实施方式,其中,步骤2)所述适配体组合文库中的分子于90~98℃(例如95℃)变性5~10min(例如5min),自然冷却至室温,再进样,进样温度为20~30℃(例如25℃),进样结合时间为60~180秒(例如120秒),解离时间为120~600秒(例如300秒),流速为20~50μL/min(例如30μL/min),所用缓冲液为Tris-T缓冲液。
在另一个优选的实施方案中,本发明所述的适配体的筛选方法或任意上文适用的实施方式,本发明所述的适配体筛选方法,其中,步骤3)中所述MCK分析,将所述适配体组合文库中的分子于90~98℃(例如95℃)变性5~10min(例如5min),自然冷却至室温,再进样,进样温度为20~30℃(例如25℃),进样结合时间为60~180秒(例如120秒),解离时间为120~600秒(例如300秒),流速为20~50μL/min(例如30μL/min),MCK分析过程中所用缓冲液为Tris-T缓冲液。
在另一个优选的实施方案中,本发明所述的适配体的筛选方法或任意上文适用的实施方式,其中所述的Tris-T缓冲液的配方为:20mM Tris,140mM NaCl,5mM MgCl2,5mM KCl,Tween 20,以HCl调至pH为7.4-7.6。其中,Tween 20浓度范围为0.005%~0.1%,例如为0.05%。
在另一个优选的实施方案中,本发明所述的适配体的筛选方法或任意上文适用的实施方式,其中,芯片的再生条件可以为高离子强度的盐,例如1~3M NaCl,例如3M NaCl;也可以为酸性条件,例如pH为1.5~2.5的10mM甘氨酸,例如pH为2.5的10mM甘氨酸;还可以为碱性条件,例如1-15mM NaOH,例如5mM NaOH。优选的再生条件为5mM NaOH,再生时间为15秒。
本发明还提供一种能够特异性结合EPO蛋白的适配体,其选自SEQ ID NO.4~9所示的序列,以及如式I至式VII所示的序列,
N-AGGT-Y-TGTTTTTGGGGTTGGTTTGGG(式I)
AGAGAGAGAGAGAGA-N-AGGT-Y-TGTTTTTGGGGTTGG-TTTGGG-TCTCTC TCTCTCTCT(式II)
GAGAGAGAGAGA-N-AGGT-Y-TGTTTTTGGGGTTGGTTT-GGG-TCTCTCTCTCTC(式III)
GAGAGAGAGA-N-AGGT-Y-TGTTTTTGGGGTTGGTTT-GGG-TCTCTCTCTC(式IV)
AGAGAGA-N-AGGT-Y-TGTTTTTGGGGTTGGTTTGGG-TCTCTCT(式V)
GAGAGA-N-AGGT-Y-TGTTTTTGGGGTTGGTTTGGG-TCTCTC(式VI)
AGAGA-N-AGGT-Y-TGTTTTTGGGGTTGGTTTGGG-TCTCT(式VII)
其中,N为G、A、C或T;Y为C或T。
本发明还提供所述能够特异性结合EPO蛋白的适配体用于检测EPO蛋白的用途。
本发明还提供用于检测EPO蛋白的组合物、试剂盒或芯片,其中含有上述本发明所述的能够特异性结合EPO蛋白的适配体。
本发明还提供一种检测EPO蛋白的方法,包括:
(1)提供待测样品;
(2)使权利要求16所述的适配体与待测样品接触;
(3)检测适配体与EPO之间的结合。
本发明中,所述的待优化适配体,可以是任何与靶分子形成高亲和力或高特异性结合的适配体,例如能够特异性结合EPO蛋白的适配体。
本发明中,所述的EPO蛋白可以是天然EPO蛋白、rHuEPO-α蛋白或rHuEP O-β蛋白,也可以是去糖基化的上述EPO蛋白。
发明详述
发明人选择重组人促红细胞生成素-α(recombinant human Erythropoietin-α,rHuEPO-α,简称EPO-α)的单链DNA适配体828r作为初始序列或模式分子(参见CN102191250A;Bioorg.Med.Chem.2010,18,8016-8025),具体说明本发明的所述构建适配体组合文库的方法,基于上述构建适配体组合文库的方法构建的适配体组合文库,以及通过所构建的适配体组合文库筛选所需适配体的方法。
该EPO-α的单链DNA适配体的如SEQ ID NO:1所示,其序列长度为39nt,在本发明中将其命名为828r-39nt(简称39nt)。39nt的二级结构呈茎环状,其与EPO-α间的解离常数KD通过EMSA法测定为KD(39nt/EPO-α)=39±27nM,其与EPO-α间的具体作用位点尚不明确。
发明人则巧妙地借助了组合化学思路,提出“序列中相邻碱基位点极可能通过碱基堆积影响空间相互作用”的假说,发明设计了四组步进型序列,构成步进型适配体组合文库。该文库中的四组序列分别命名为向左收缩(Left contraction)分子群、向右收缩(Right contraction)分子群、两端收缩(Inner contraction)分子群、和两端扩展(又称茎部延长,Stem-nbp)分子群。
其中,向左收缩分子群是指在初始序列39nt的基础上,从其5’末端逐级剪裁掉相邻的两个碱基,构成从Left contraction 37、35、33、……、一直到Left contraction 21(简称L37、L35、L33、……、L21)等9条序列。向右收缩分子群则正好相反,是在初始序列807-39nt的基础上,从其3’末端逐级剪裁掉相邻的两个碱基,构成从Right contraction 37、35、33、……、一直到Right contraction 21(简称R37、R35、R33、……、R21)等9条序列。两端收缩分子群是指在初始序列39nt的基础上,从其5’和3’末端两侧各逐级剪裁掉单个碱基,构成从Inner contraction 37、35、33、……、一直到Inner contraction 21(简称In37、In35、In33、……、In21)等9条序列。如此三组各9条序列,共得到27条序列(参见图1)。茎部延长分子群则指在保持适配体环部序列不变的情况下,其茎部序列改为第一序列(例如包含G和A的序列,例如GA、AGA、AGAGA、GAGAGA、AGAGAGA、GAGAGAGAGA、GAGAGAGAGAGA、AGAGAGAGAGAGAGA、)……第二序列(例如包含C和T的序列,例如TC、TCT、TCTCT、TCTCTC、TCTCTCT、TCTCTCTCTC、TCTCTCTCTCTC、TCTCTCTCTCTCTCT)互补碱基,构成从Stem-2bp、3bp、5bp、6bp、7bp、10bp、12bp到Stem-15bp等8条序列。如此构成的适配体步进型组合文库,仅用了35条序列,即涵盖了能产生特异亲和作用的大多数可能范围,同时能够明确特异性适配体中关键碱基位点的作用。这就为得到最短适配体,及探讨可能的结合机制奠定了基础。
本发明人使用上述所构建的步进型适配体组合文库筛选亲和活性高的特异性结合EPO-α蛋白的适配体。筛选时,采用SPR法表征和评价适配体与靶分子间亲和力。具体地,所使用的仪器可以是Biacore T100、T200(美国GE公司)、ProteOn XPR36(美国伯乐公司)、Reichert4SPR(美国Reichert公司)等。
在测定亲和活性时,采用链霉亲和素包被的葡聚糖芯片(简称SA芯片)固定靶蛋白EPO-α。将EPO-α与生物素共价偶联后,固定在SA芯片表面。
EPO-α与生物素共价偶联后的产物称为生物素化EPO-α。EPO-α可以通过多种方式与生物素共价偶联,共价偶联的方式取决于EPO-α分子结构中的末端基团类型。例如,所述EPO-α可以通过其氨基酸残基的末端氨基偶联生物素,或是其氨基酸残基的末端羧基偶联生物素,或是可以通过其糖基的末端唾液酸基团偶联生物素。
偶联试剂的结构中一般分为生物素基团、反应基团以及连接臂部分。适当的连接臂长度可以有效克服固定时的空间位阻。可以根据EPO-α末端基团的类型选择不同的偶联试剂。
例如,当EPO-α的氨基酸残基末端为氨基基团时,选择的生物素偶联试剂可以为不同形式的琥珀酰亚胺化生物素,例如Sulfo-NHS-LC-Biotin(Sulfosuccinimidyl-6-[biotin-amido]hexanoate,6-[生物素酰胺基]己酸磺酸基-N-羟基琥珀酰亚胺酯)、Sulfo-NHS-LC-LC-Biotin(Sulfosuccinimidyl-6-[biotin-amido]-6-hexanamido hexanoate,6-[生物素酰胺基]-6-己酰氨基己酸-磺酸基-N-羟基琥珀酰亚胺酯)、NHS-LC-Biotin(Succinimidyl 6-(biotinamido)hexanoate,6-[生物素酰胺基]己酸-N-羟基琥 珀酰亚胺酯)、NHS-LC-LC-Biotin(Succinimidyl-6-(biotin-amido)-6-hexanamido hexanoate,6-[生物素酰胺基]-6-己酰氨基己酸-N-羟基琥珀酰亚胺酯)、NHS-PEG4-Biotin(生物素四聚乙二醇N-羟基琥珀酰亚胺酯)、NHS-PEG12-Biotin(生物素十二聚乙二醇N-羟基琥珀酰亚胺酯)等。
当EPO-α的氨基酸残基末端为羧基基团时,选择的生物素偶联试剂可以为末端氨基化生物素,例如,Amine-PEG3-Biotin(生物素三聚乙二醇胺)、Biocytin Hydrazide(生物素酰肼)等,偶联时需同时加入碳二亚胺(EDC)。
当EPO-α的糖基末端为唾液酸时,选择的生物素偶联试剂可以为生物素化的肼,例如Biocytin Hydrazide(生物素酰肼)、Biotin-LC-Hydrazide(6-(biotinamido)hexanehydrazide,6-[生物素酰胺]-己酰肼)、Biotin-PEG4-Hydrazide(生物素四聚乙二醇酰肼),偶联时需先将唾液酸基团以高碘酸钠氧化成醛。
在一个优选的实施方案中,EPO-α与偶联试剂以1:1摩尔比共价偶联,称为单分子生物素化EPO-α蛋白,简称Bio-EPO-α。
在一个优选的实施方案中,EPO-α的氨基酸残基末端为氨基基团时,选取偶联试剂Sulfo-NHS-LC-Biotin与EPO-α共价偶联,得到单分子生物素化的NH2-Bio-EPO-α。EPO-α的氨基酸残基末端为羧基基团时,选取偶联试剂Biotin Hydrazide与EPO-α共价偶联,得到单分子生物素化的COOH-Bio-EPO-α。EPO-α的糖基末端为唾液酸基团时,选取偶联试剂Biocytin Hydrazide与EPO-α共价偶联,得到单分子生物素化的Sialyl-Bio-EPO-α。
在芯片表面固定靶蛋白时,根据不同的实验目的,可以选择不同水平的靶蛋白浓度。在进行多循环动力学(MCK)分析时,选择适宜浓度,使目标分析物的最大结合量(Rmax)≧30响应单位(RU)即可。而若进行稳态分析,则靶蛋白浓度可以更高,以使固定量达到饱和。Rmax=配体固定量×(目标分析物的分子量/配体的分子量)×结合计量比。
在芯片表面固定靶蛋白时,还需要考察并优化固定时间、流速等条件。在本发明的一个实施例中,首先以50mM NaOH、1M NaCl溶液进行通道初始化,进样3次,流速为20μL/min,每次进样的时间为60秒,然后用70%甘油水溶液对芯片进行归一化处理。然后在将1μM的单分子生物素化的Sialyl-Bio-EPO或NH2-Bio-EPO-α,在流速10μL/min,进样时间60秒的条件下,固定于SA芯片上。再生条件为5mM NaOH,流速30μL/min,再生时间为15秒。
在一个优选的实施方案中,本发明利用单点进样方式进行SPR动力学分析实验,即单点筛选法,筛选出各适配体分子群中与EPO-α具有亲和力的序列(又称活性序列),具体表现为结合-解离曲线(即进样后,响应值(RU值)的变化曲线)具有正响应。单点进样,也就是单一浓度进样,能够直观地表示出样品是否与靶蛋白存在结合活性的响应(即RU值>0))。
SPR动力学分析能够确定在一个给定的时间跨度内,适配体与固定在芯片上的靶蛋白 形成的复合物是否能够结合和完全解离,进样后,适配体和靶蛋白结合形成复合物,响应值(RU值)增加;然后结束进样,注入空白缓冲溶液,复合物解离,RU值下降。
采用单点法筛选具有活性的适配体序列时,进样后,所述适配体组合文库中的待筛选适配体与固定在芯片表面的靶蛋白结合,生成复合物,进样结束,注入空白缓冲溶液,芯片上的复合物发生解离,得到各序列单点RU值变化曲线,根据响应值(RU值)判断待筛选适配体是否具有活性,RU值大于0的适配体具有活性。
对于具有活性的适配体序列,再进行多循环动力学分析(MCK分析),得到亲和力参数解离常数KD,以及动力学参数,包括结合速率常数Ka和解离速率常数Kd。其中KD=Kd/Ka。根据KD数值的大小判断待筛选适配体与靶蛋白之间的亲和力,KD数值越小,亲和力越强,从而筛选出亲和力强的适配体序列。
在进行单点筛选或者进行MCK分析时,所述适配体组合文库中的待筛选适配体在进样前,均先于90~98℃(例如95℃)变性5~10min(例如5min),自然冷却至室温,然后再进样。进样温度为20~30℃(例如25℃),进样结合时间为60~180秒(例如120秒),解离时间为120~600秒(例如300秒),流速为20~50μL/min(例如30μL/min),所用缓冲液为Tris-T缓冲液。再生条件为5mM NaOH,流速30μL/min,再生时间为15秒。
其中所述的Tris-T缓冲液的配方为:20mM Tris,140mM NaCl,5mM MgCl2,5mM KCl,Tween 20,以HCl调至pH7.4-7.6。其中,Tween 20浓度范围为0.005%~0.1%,优选为0.05%。
在进行单点筛选时,所述步进型适配体组合文库中的各序列进样浓度可以为200~1000nM,例如500nM。
在一个具体的实施方案中,单点筛选可以通过以下具体步骤得以实现:
1)各适配体分子群的各序列进样浓度为500nM。各序列首先于95℃,5min变性后,自然冷却至室温。缓冲液Tris-T缓冲液的配方为:20mM Tris,140mM NaCl,5mM MgCl2,5mM KCl,0.05%Tween 20,以HCl调至pH 7.4-7.6;
2)进行SPR动力学分析实验(n≧2),依据每条适配体序列的RU值筛选出具有活性的适配体序列,RU值>0的适配体序列具有活性。实验时,进样温度选择25℃,进样结合时间120s,解离时间300s,流速30μL/min;
3)再生条件可以为高离子强度的盐,例如1~3M NaCl;也可以为酸性条件,例如10mM甘氨酸,pH 1.5~2.5;还可以为碱性条件,例如1-15mM NaOH。优选再生条件为5mM NaOH,再生时间设为15秒。
在进行MCK分析时,可以根据待分析适配体序列与EPO-α的单点结合的RU值,选择合适的浓度梯度范围进行MCK分析。例如500nM的Stem-3bp,其RU值较低,仅为3,所选择的浓度梯度则酌情选择为较高浓度范围,例如125nM~2000nM(各点间为倍增关系);而500nM的Stem-7bp,其RU值较高,为80RU,选择的浓度梯度为12.5nM ~400nM。
在一个具体的实施方案中,MCK分析通过以下具体步骤得以实现:
1)各序列首先于95℃,5min变性后,自然冷却至室温。结合缓冲液为Tris-T缓冲液。
2)进行MCK分析,进样结合时间设为120s,解离时间300s,流速30μL/min;再生条件为5mM NaOH,15秒。
3)对MCK结果进行非线性拟合,得出Ka,Kd和KD值。
在进行MCK分析时,还需从拟合结果符合度判断该非线性拟合结果是否正常。拟合结果符合度由U值和Chi2值决定。U值一般需≦15,说明拟合符合度较高;U值越低,符合度越高,U值最低为1;Chi2一般需≦0.1Rmax,说明拟合符合度较高。
采用本发明所述的适配体的筛选方法对所构建的步进型适配体组合文库进行筛选,结果表明:
1)向左收缩分子群,仅L37、L35、L33为活性序列,其KD值分别为320±30nM、360±110nM和390±30nM,呈现从低到高逐渐增加的特征;
2)向右收缩分子群,仅R37、R35、R33、R31为活性序列,其KD值分别为560±170nM、1300±150nM和140±40nM、130±30nM,基本呈现从高到低逐渐降低的特征;
3)两端收缩分子群,In37、In35、In33、In31、In29、In27、In25有活性,但活性差别较大。其KD值分别为310±120nM(In37)、390±40nM(In35)、750±30nM(In33)、390±50nM(In31)、200±1nM(In29)、52±4nM(In27)和2060±125nM(In25),其KD值从In37到In33逐渐升高,然后至In27又逐渐降低,In27的KD值降低最为明显,而后In25的KD值又急剧升高,In23、In21的亲和活性基本丧失;In 27是所有剪裁序列中亲和活性最高的序列;
4)茎部延长分子群,所有8条序列均有活性,其KD值分别为390±50nM(Stem-2bp)、440±40nM(Stem-3bp)、80±10nM(Stem-5bp)、40±2nM(Stem-6bp)、30±5nM(Stem-7bp)、40±20nM(Stem-10bp)、40±0.1nM(Stem-12bp)、50±20nM(Stem-15bp)。其中仅Stem-2bp、Stem-3bp的亲和活性较低,其KD值从2bp一直到7bp逐渐降低,从7bp到15bp,其KD值基本一致。亲和活性最高的为Stem-7bp。同时,Stem-7bp、Stem-6bp、Stem-10bp、Stem-12bp、Stem-15bp的亲和活性均优于39nt。
可以看出,通过对本发明提供的步进型适配体组合文库进行亲和力及动力学评价,可迅速筛选得到亲和力更强、序列更短的适配体序列In 27。
另外,根据上述4组适配体分子群的亲和力及动力学特征,也可以判断适配体序列中是否存在关键识别位点。向左收缩分子群中,仅有能够形成高级结构G-四聚体结构(GQ)的L37、L35、L33存在亲和识别能力,并且随着茎部结构的缩短,亲和活性也逐渐下降。
向右收缩分子群中,亦只有能够形成高级结构GQ的R37、R35、R33、R31具有亲和活性,特别地,该分子群中,7A8A碱基是否充分暴露对亲和活性有较大的影响。7A8A的 完全暴露(R33)较之被掩蔽(R35、R37)时,适配体的活性基本恢复。而7A8A去除前后(R33、R31),活性基本相同。
而两端收缩分子群中,随着茎部的缩短,亲和活性逐渐降低,而保留了整个环部的In27亲和力完全恢复,提示可能形成了新的高级结构。
本发明还针对所得到的最短适配体In27,构建了其定点突变分子群,以明确具体碱基位点对结合的贡献。In27的二级序列特征为,仅保留了39nt的环部序列;其一级结构序列特征为,--GG---------GGGG--GG--GGG;结合其圆二色谱结果,推测In27可能形成了以GQ为骨架的三级结构。
将最短适配体In27的碱基序列划分为GQ骨架结构和除GQ骨架外的5个结构域(Domain)。从而构建了各定点突变分子群,共7组,分别命名为D1、D2、D3&4、D5、GQ和简并序列定点突变分子群,共31条序列。其中:
D1定点突变分子群包括:1A碱基改为C/T/G/X、2A碱基改为T/G、以及1A2A碱基同时改为T碱基等7条序列;
D2定点突变分子群包括:6C碱基改为T/A/G/X、8G碱基改为T碱基、6C8G同时改为T碱基、11T碱基改为X(X代表该位点的碱基缺失),以及10T11T碱基同时改为X等8条序列;
D3&4定点突变分子群包括:18T碱基改为A碱基、以及23T碱基改为A碱基等2条序列;
D5定点突变分子群包括:27G碱基改为A/T/X等3条序列;
GQ定点突变分子群包括:25G改为T/X(X代表该位点的碱基缺失),即25T、25X;在6位的C改为T的前提下,14G、15G、16G、17G中的G碱基分别改为T碱基,即为14T、15T、16T、17T,以及14G15G、15G16G、16G17G、14G17G中的两个G碱基均改为T碱基,即14T15T、15T16T、16T17T、14T17T等10条序列;
简并序列分子群包括1A6C碱基同时改为T碱基,即1T6T、1A6C27G碱基分别改为1T16T27A碱基、以及1A27G碱基改为1T27A碱基等3条序列。
利用单点筛选法,对所述定点突变分子群进行其亲和活性的筛选和评价。
结果表明,GQ骨架对识别非常重要,25G参与了GQ中GG骨架的形成,14-17位的G碱基均可能与GQ骨架相关;D2结构域是最短适配体In27与EPO-α发生相互作用的主要结构域;D2、D3、D4环上的序列保守性较高,除6C外,其他碱基不能任意改动。该结果表明了In27的高度保守性。
从而可得到其简并序列为:
1NAGGT6YTGT10TTTTGGGGTT20GGTTTGGG
其中,N代表A、G、T或C,Y代表C或T。
本发明的有益技术效果包括以下一个或多个方面:
1)本发明提供的构建适配体组合文库的方法,采用高效的结构剪裁方式构建内含较少序列的组合文库,所构建的适配体组合文库涵盖了能产生特异亲和作用的大多数可能范围,为得到最短适配体,及探讨可能的结合机制奠定了基础。
2)本发明提供的适配体筛选方法,使用本发明提供的适配体组合文库筛选适配体,可筛选出亲和力更优、无冗余序列的最短适配体活性序列,并能够明确特异性适配体中关键碱基位点的作用。
3)应用本发明提供的适配体筛选方法,筛选得到了亲和力更优的序列:Stem-7bp、Stem-6bp、Stem-10bp、Stem-12bp、Stem-15bp、In27,其中In27为无冗余序列的最短适配体活性序列。这些序列可以用于检测EPO蛋白。
4)对于筛选得到的该最短适配体序列In27,结合该序列的特征,利用定点碱基突变,阐明了关键碱基对结合的贡献。
5)得到最短适配体In27的简并序列。In27及其简并序列均具备和EPO-α间良好的亲和活性,适于用作亲和传感功能元件。
附图说明
图1.步进型适配体组合文库的序列构成图;
图2.各定点突变分子群序列构成图;
图3A.EPO-α的氨基酸残基末端为氨基基团时,与生物素共价偶联的反应流程图;
图3B.EPO-α的氨基酸残基末端为羧基基团时,与生物素共价偶联的反应流程图;
图3C.EPO-α的糖基末端为唾液酸基团时,与生物素共价偶联的反应流程图;
图4.在SA芯片上固定Bio-EPO-α(1μM)后的适配体序列(500nM,39nt)的结合响应,其中:
(A)为在SA芯片上固定Bio-NH2-EPO-α时的RU值变化曲线,RUstability=2342,固定条件:进样流速为10μL/min,进样时间为60秒;
(B)为500nM 39nt作为分析物评价Bio-NH2-EPO的固定效果时的RU值变化曲线;
(C)为在SA芯片上固定Bio-Sialyl-EPO-α时的RU值变化曲线,RUstability=2342,固定条件:进样流速为10μL/min,进样时间为60秒;
(D)为500nM 39nt作为分析物评价Sialyl-NH2-EPO的固定效果时的RU值变化曲线;
图5A.SA芯片上固定Bio-NH2-EPO-α时,39nt的结合解离谱图;
图5B.SA芯片上固定Sialyl-Bio-EPO-α时,39nt的的结合解离谱图;
图6.向左收缩适配体分子群各序列的单点RU值变化曲线;
图7.向右收缩适配体分子群各序列的单点RU值变化曲线;
图8.两端收缩适配体分子群各序列的单点RU值变化曲线;
图9.两端延展适配体分子群各序列的单点RU值变化曲线;
图10为In27在含不同浓度K+的Tris-HCl缓冲液中的圆二色谱图;
图11A.GQ定点突变适配体分子群中,14-17位的G碱基的定点突变单点筛选结果,参比序列为In27,每条序列浓度为500nM;
图11B.图11A中方框部分的局部放大图,可以看到25位G碱基的定点突变单点筛选结果;
图12.D1定点突变适配体分子群各序列的单点筛选结果,参比序列为In27,每条序列浓度为500nM;
图13.D2定点突变适配体分子群各序列的单点筛选结果,参比序列为In27,每条序列浓度为500nM;
图14.D3&4定点突变适配体分子群各序列的单点筛选结果,参比序列为In27,每条序列浓度为500nM;
图15.D5定点突变适配体分子群各序列的单点筛选结果,参比序列为In27,每条序列浓度为500nM;
图16.简并序列适配体分子群各序列的单点筛选结果,参比序列为In27,每条序列浓度为500nM;
图17A.在SA芯片上固定3’-Biotin-39nt时,EPO-α的MCK谱图,每条序列浓度为500nM;
图17B.在SA芯片上固定3’-Biotin-12T-In27时,EPO-α的MCK谱图,每条序列浓度为500nM。
具体实施方式
下面将结合实施例对本发明的实施方案进行详细叙述,但是本领域技术人员将会理解,下列实施例仅用于说明本发明,而不应视为限定本发明的范围。实施例中未注明具体条件者,按照常规条件和制造商建议的条件进行,所用试剂或仪器未注明生产厂商者,均为可以通过市购获得的产品。
本发明的实施例中采用SPR法表征和评价适配体与靶分子间亲和力。具体地,所使用的仪器是BIAcore T100生物大分子相互作用仪,购自美国GE公司。
实验时,样品以恒定的流速和浓度流过芯片表面,样品中的待分析物(例如本发明所构建的适配体步进型组合文库中的适配体)和固定在芯片表面的分子(例如NH2-Bio-EPO-α或Sialyl-Bio-EPO-α)发生结合形成复合物,芯片表面物质的质量发生改变,仪器记录下对应的响应值(Response Unit,以下简称RU值)的变化。结束进样后,使空白缓冲液流过芯片表面,复合物发生解离,芯片表面物质的质量再次发生改变,仪器记录对应的RU值的变化。
该技术能够跟踪记录样品中的待分析物与芯片表面的分子结合、解离整个过程的变化,记录RU值的变化,生成结合-解离曲线。通过多循环动力学(Multiple Cycle Kinetics,MCK)分析,还能提供动力学数据(例如,结合速率常数Ka、解离速率常数Kd)以及亲和力数据(例如,解离常数KD,KD=Kd/Ka)。
待分析的适配体序列与偶联在芯片表面的分子结合后,RU值>0的适配体序列具有活性。对于具有活性的适配体序列,进行MCK分析,可以得到各条适配体序列与EPO-α间的亲和力及动力学参数。亲和力以解离常数KD表征,KD越低,则亲和力越高。动力学参数中,结合速率常数Ka表示分析物与配体分子间形成复合物的快慢,其数值相当于1M的分析物与配体混合时形成的复合物的数量,单位为M-1s-1;解离速率常数Kd表示复合物解离的快慢,其数值相当于复合物每秒解离的比例,单位为s-1)。依活性不同,各条适配体与EPO-α的KD值一般在nM~μM范围。
本发明实施例中所用的缓冲液如下:
1)5×PBS缓冲液,其配方为:0.75M NaCl,0.1M Na3PO4,pH值为7.2;
2)1×PBS缓冲液,其配方为:0.15M NaCl,0.02M Na3PO4,pH值为7.2;
3)5×Tris-HCl缓冲液,其配方为100mM Tris,700mM NaCl,25mM MgCl2,25mM KCl,pH值为7.4-7.6;
4)Tris-T缓冲液,其配方为20mM Tris,140mM NaCl,5mM MgCl2,5mM KCl,0.05wt%Tween 20,以HCl调至pH 7.4-7.6。
实施例1适配体步进型组合文库的构建
如图1所示,选择SEQ ID NO:1所示的EPO-α的单链DNA适配体作为初始序列,分别构建向左收缩分子群、向右收缩分子群、两端收缩分子群和茎部延长分子群,由这四个分子群共同构成适配体步进型组合文库。
1)向左收缩分子群的构建
在初始序列39nt的基础上,从其5’末端逐级剪裁掉相邻的两个碱基,得到如表1中所示的Left contraction 37、35、33、……、一直到Left contraction 21(分别简称L37、L35、L33、……、L21)等9条序列。
2)向右收缩分子群的构建
向右收缩分子群则正好相反,在初始序列39nt的基础上,从其3’末端逐级剪裁掉相邻的两个碱基,得到如表1中所示的Right contraction 37、35、33、……、一直到Right contraction 21(分别简称R37、R35、R33、……、R21)等9条序列。
3)两端收缩分子群的构建
两端收缩分子群是在初始序列39nt的基础上,从其5’和3’末端两侧分别逐级剪裁掉一个碱基,得到如表1中所示的Inner contraction 37、35、33、……、一直到Inner contraction21(分别简称In37、In35、In33、……、In21)等9条序列。
4)茎部延长分子群的构建
茎部延长分子群则是在保持适配体环部序列不变的情况下,其茎部序列改为包含G和A的第一序列……包含C和T的第二序列互补碱基,其中第一序列分别为:GA、AGA、AGAGA、GAGAGA、AGAGAGA、GAGAGAGAGA、GAGAGAGAGAGA、AGAGAGAGAGAGAGA,第二序列分别为:TC、TCT、TCTCT、TCTCTC、TCTCTCT、TCTCTCTCTC、TCTCTCTCTCTC、TCTCTCTCTCTCTCT。得到如表1中所示的Stem-2bp、Stem-3bp、Stem-5bp、Stem-6bp、Stem-7bp、Stem-10bp、Stem-12bp、Stem-15bp(分别简称为S2bp、S3bp、S5bp、S6bp、S7bp、S10bp、S12bp、S15bp)等8条序列。
本实施例所构建的适配体步进型组合文库,仅用了35条序列,即涵盖了能产生特异亲和作用的大多数可能范围。使用上述步进型组合文库筛选适配体,可以确定特异性适配体中关键碱基的位点,从而得到亲和活性更优、特异性更强的优化适配体序列。
上述适配体步进型组合文库由上海生工生物工程公司合成。
表1步进型组合文库的序列表
实施例2EPO-α的氨基酸残基末端为氨基基团时,与生物素的共价偶联
EPO-α的氨基酸残基末端为氨基基团时,与生物素共价偶联的反应流程如图3A所示。
称取偶联试剂(Sulfo-NHS-LC-Biotin,购自美国Thermo公司)0.35mg,252μL 5×PBS缓冲液,1008μL灭菌超纯水,配制成1260μL的0.5mM Sulfo-NHS-LC-Biotin溶液,该溶液现用现配,并且避光操作。取15.5μL的EPO-α蛋白原液(3.25g/L,深圳赛保尔生物药业公司赠送),加入20μL 5×PBS和64.5μL灭菌超纯水,配制成100μL的0.5g/L EPO-α溶液。向该100μL 0.5mg/mL的EPO-α溶液中,加入6.25μL的0.5mM Sulfo-NHS-LC-Biotin溶液,混匀后,于室温振荡温育30min。温育反应完成后,进行纯化,将反应液加入到脱盐小柱(如Zeba Spin脱盐小柱,购自Thermo公司)中,除去剩余的未反应分子。重复纯化2次。
将纯化后的溶液离心浓缩至50μL,并利用紫外可见分光光度法,以HABA/Avidin法表征,得到生物素的标记个数为1,即得到了单分子生物素化的NH2-Bio-EPO-α。用紫外可见分光光度法对该NH2-Bio-EPO-α的浓度进行测定,浓度计算公式为:蛋白浓度(mg/mL)=1.55A280-0.76A260),计算得到的浓度为1.04g/L,回收率为103%。
实施例3EPO-α的氨基酸残基末端羧基基团时,与生物素的共价偶联
EPO-α的氨基酸残基末端为羧基基团时,与生物素共价偶联的反应流程如图3B所示。
称取偶联试剂(Biotin Hydrazide,购自美国Sigma Aldrich公司)0.34mg,229μL灭菌超纯水,配制成4mM Biotin Hydrazide溶液,该溶液现用现配,并且避光操作。取4mM Biotin Hydrazide溶液50μL,加入100μL的0.5M 2-(N-吗啡啉)-乙磺酸溶液(MES溶液,pH=5.0)和350μL灭菌超纯水,配制成0.4mM Biotin Hydrazide溶液,缓冲体系为0.1M MES(pH 5.2)。称取碳二亚胺(EDC)5.57mg,加入1166μL灭菌超纯水配制成25mM EDC溶液。取40μL的25mM EDC溶液,加入200μL的0.5M MES溶液(pH=5.0),760μL灭菌超纯水配制成1mM EDC溶液,缓冲体系为0.1M MES(pH 5.2)。 取15.5μL的EPO-α蛋白原液(3.25g/L),加入20μL的0.5M MES缓冲液和64.5μL灭菌超纯水配制为100μL的0.5g/L EPO-α溶液。向该100μL 0.5g/L的EPO-α溶液中,加入7.5μL的0.4mM Biotin Hydrazide溶液和15μL的1mM EDC溶液混匀后,于室温振荡温育2h。温育反应完成后,进行纯化,将反应液加入到脱盐小柱(如Zeba Spin脱盐小柱,购自Thermo公司)中,除去剩余的未反应分子,并把反应缓冲体系换成PBS缓冲液。重复纯化2次,得到纯化后的溶液。
将纯化后的溶液离心浓缩至50μL,并利用紫外可见分光光度法,以HABA/Avidin法表征,得到生物素的标记个数为1,即得到了单分子生物素化的COOH-Bio-EPO-α。用紫外可见分光光度法对该COOH-Bio-EPO-α的浓度进行测定,浓度计算公式同实施例1,计算得到的浓度为1.07g/L,回收率为109%。
实施例4EPO-α的糖基末端为唾液酸基团时,与生物素的共价偶联
EPO-α的糖基末端为唾液酸基团时,与生物素共价偶联的反应流程如图3C所示。
避光称取8.96mg高碘酸钠(NaIO4),加入873.8μL灭菌超纯水,得到终浓度为50mM的NaIO4储备液,取4μL的50mM NaIO4储备液加入40μL的0.5M醋酸钠(NaAc)溶液(pH=5.5),156μL灭菌超纯水配制成1mM NaIO4溶液,缓冲体系为0.1M NaAc(pH5.5)。取15.5μL的EPO-α蛋白原液(3.25g/L),加入20μL 0.5M NaAc溶液和64.5μL灭菌超纯水配制成100μL的0.5g/L EPO-α溶液。向该100μL的0.5g/L EPO-α溶液中,加入2.35μL的1mM NaIO4溶液混匀,于4℃避光振荡温育30min。温育反应完成后,进行纯化,将反应液加入到脱盐小柱(如Zeba Spin脱盐小柱,购自Thermo公司)中,除去剩余的未反应分子,并把反应缓冲体系换成PBS缓冲液。重复纯化2次,得到纯化后的EPO-α反应液。
避光称取0.31mg偶联试剂Biocytin Hydrazide,加入333.8μL的PBS缓冲液,得到终浓度为2.5mM的Biocytin Hydrazide溶液。向纯化后的EPO-α反应液中,加入4.7μL的2.5mM Biocytin Hydrazide溶液,混匀,于室温下避光振荡温育2小时。温育反应完成后,进行纯化,将反应液加入到脱盐小柱(如Zeba Spin脱盐小柱,购自Thermo公司)中,除去剩余的未反应分子。重复纯化2次,得到纯化后的溶液。
将纯化后的溶液离心浓缩至50μL。并利用紫外可见分光光度法,以HABA/Avidin法表征,得到生物素的标记个数为1,即得到了单分子生物素化的Sialyl-Bio-EPO-α。用紫外可见分光光度法对该Sialyl-Bio-EPO-α的浓度进行测定,浓度计算公式同实施例1,计算得到的浓度为1.38mg/mL,回收率为137%。
实施例5SA芯片上NH2-Bio-EPO-α的固定
取2.7μL的NH2-Bio-EPO-α储备液(该储备液用超纯水配置,浓度为36.5μM),加入20μL的5×Tris-HCl缓冲液和77.3μL纯化水,即得到1μM的NH2-Bio-EPO-α溶液。
取用一块新的SA芯片,首先以50mM NaOH,1M NaCl溶液进行通道初始化。进样3次,流速为20μL/min,每次进样的时间为60秒。然后用70%甘油水溶液对SA芯片进行归一化处理。
在流速为10μL/min的条件下进样,进样时间为60秒,将1μM的NH2-Bio-EPO-α(制备方法见实施例2)固定于SA芯片的通道2上。进样过程中,Bio-NH2-EPO-α与SA芯片形成生物素-链霉亲和素强非共价结合,RU值上升。进样结束时,RU值上升至最大值。然后再以空白缓冲液冲洗SA芯片表面,除去附着的Bio-NH2-EPO-α,RU值会略有下降。RU值变化曲线参见图4中的(A),上升后的最大RU值减去下降后的RU值,即得到响应稳定性(stability)RU值为2342,即,RUstability=2342。
以通道1为参比通道,使用500nM的39nt,对通道2上固定的Bio-NH2-EPO-α蛋白的活性进行评价。进样的流速为30μL/min,进样时间为120秒。进样后,39nt与Bio-NH2-EPO-α蛋白形成非共价结合,RU值上升。进样结束时,RU值上升至最大值。然后再以空白缓冲液冲洗SA芯片表面,非共价结合物解离,RU值下降。RU值变化曲线(即结合-解离曲线)参见图4中的(B),39nt与Bio-NH2-EPO-α蛋白结合的过程中,RU最大值减去RU最小值即为相应稳定性ΔRU,2-1通道的响应稳定性ΔRU为30。说明该固定方式下,EPO-α和39nt间保留着亲和活性。
再生条件为5mM NaOH,流速30μL/min,再生时间为15秒。
实施例6SA芯片上Sialyl-Bio-EPO-α的固定
取1.8μL的Sialyl-Bio-EPO-α储备液(该储备液用超纯水配置,浓度为54.3μM),加入20μL的5×Tris-HCl缓冲液和78.2μL纯化水,即得到1μM Sialyl-Bio-EPO溶液。
取用一块新的SA芯片,首先以50mM NaOH、1M NaCl溶液进行通道初始化。进样3次,流速为20μL/min,每次进样的时间为60秒。然后用70%甘油水溶液对芯片进行归一化处理。
在流速为10μL/min的条件下进样,进样时间为60秒,将1μM的Sialyl-Bio-EPO-α(制备方法见实施例4)固定于SA芯片通道4上。RU值变化曲线参见图4中的(C),响应稳定性(stability)RU值上升了1159,即,RUstability=1159。
以通道3为参比通道,使用500nM的39nt,对通道4上固定的Sialyl-Bio-EPO-α蛋白的活性进行评价。进样的流速为30μL/min,进样时间为120秒。RU值变化曲线参见图4中的(D),4-3通道的响应稳定性ΔRU为50。说明该固定方式下,EPO-α和39nt间仍然保留着亲和活性。
再生条件为5mM NaOH,流速30μL/min,再生时间为15秒。
实施例7 39nt的MCK分析
取2μL的100μM 39nt储备液(该储备液用超纯水配置),加入4μL的5×Tris-HCl 缓冲液,2μL的1wt%Tween 20,以及12μL灭菌超纯水,得到10μM的39nt溶液。取该39nt溶液20μL,加入380μL的Tris-T缓冲液,即得到500nM的39nt溶液。采用Tris-T缓冲液倍比稀释的方法,制备得到浓度分别为25、50、100、200、400nM的39nt溶液,于95℃下变性5分钟,再自然冷却至室温。
在SA芯片上,使用NH2-Bio-EPO-α(通道2)和Sialyl-Bio-EPO-α(通道4),进行EPO-α和39nt间的亲和力和动力学分析。结合时间为120秒,解离时间为300秒,流速为30μL/min。再生条件为5mM NaOH,进样时间为15秒,流速为30μL/min。包含以空白溶剂为零点在内的39nt六个浓度的进样顺序为0、25nM、50nM、100nM、200nM、400nM,得到结合解离谱图,如图5A和图5B所示,其中图5A为通道2(固定NH2-Bio-EPO-α)得到的结合解离谱图,图5B为通道4(固定Sialyl-Bio-EPO-α)得到的结合解离谱图。
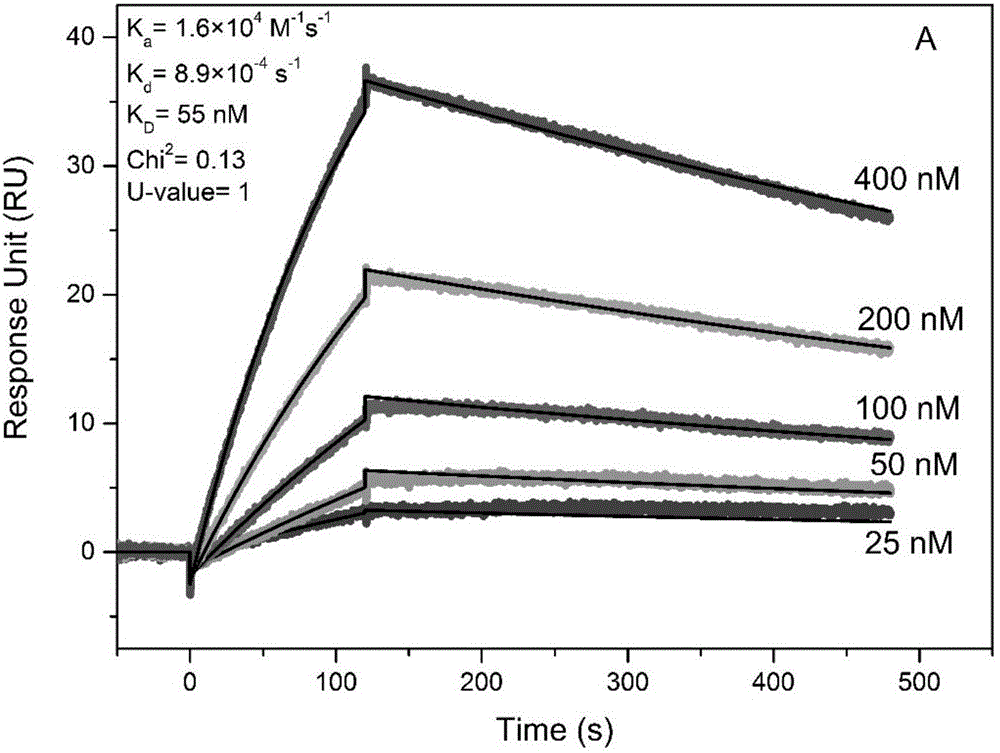
采用Biacore数据分析软件对图5A和图5B所示的结合解离谱图进行非线性拟合。结果表明,利用通道2-1数据得到39nt与EPO-α相互作用的解离常数KD=55nM,结合速率常数Ka=1.6×104M-1s-1,解离速率常数Kd=8.9×10-4s-1(U-value=1,Chi2=0.13)。利用通道4-3数据得到39nt与EPO-α相互作用的解离常数KD=52nM,结合速率常数Ka=1.8×104M-1s-1,解离速率常数Kd=9.3×10-4s-1(U-value=1,Chi2=0.07)。上述结果表明以NH2-Bio-EPO-α和Sialyl-Bio-EPO-α为固定方式时,两者符合度较好,也说明了SPR方法已经成功建立。
较之Sialyl-Bio-EPO-α,NH2-Bio-EPO-α对5mM NaOH为再生条件的耐受性更高。
实施例8向左收缩适配体分子群在SA芯片上的单点活性评价及活性序列的MCK分析
待评价的适配体序列为:向左收缩适配体分子群中的所有序列,包括L21、L23、L25、L27、L29、L31、L33、L35、L37,以及参比序列39nt,共10条序列。分别量取2μL上述10条序列的储备液(该储备液用超纯水配置,浓度为100μM),加入4μL的5×Tris-HCl缓冲液,2μL的1wt%Tween 20,12μL灭菌超纯水,得到10μM的各序列中间储备液。取20μL的各序列中间储备液(10μM),加入380μL的Tris-T缓冲液,得到500nM的各序列工作溶液,于95℃下变性5min,自然冷却至室温。
使用固定了NH2-Bio-EPO-α的SA芯片,以所述500nM的向左收缩适配体分子群中的各序列以及参比序列39nt作为分析物,采用单点筛选法进行SPR动力学分析。具体实验条件同实施例7。进样后,待分析物与SA芯片上的EPO-α结合,生成复合物,进样结束,注入空白缓冲溶液,芯片上的复合物发生解离,各序列单点RU值变化曲线图如图6所示。
结果表明,仅有能够形成高级结构G-四聚体(以下简称GQ)结构的L37、L35、L33存在亲和识别能力,并且随着茎部结构的缩短,响应值也逐渐下降。
针对L37、L35、L33这三条活性序列,分别选取浓度为31.25-1000nM(L37);62.5-2000 nM(L35);62.5-2000nM(L33),进行MCK分析。具体SPR条件同实施例7。得到亲和力和动力学参数,如表2所示。结果表明,其KD值分别为320±30nM、360±110nM和390±30nM,呈现从低到高逐渐增加的特征。
表2向左收缩适配体分子群的亲和力和动力学参数
实例9向右收缩适配体分子群在SA芯片上的单点活性评价及活性序列的MCK分析
待评价的适配体序列为:向右收缩适配体分子群中的所有序列,包括R21、R23、R25、R27、R29、R31、R33、R35、R37,以及参比序列39nt,共10条序列。分别量取2μL上述10条序列的储备液(该储备液用超纯水配置,浓度为100μM),加入4μL的5×Tris-HCl缓冲液,2μL的1wt%Tween 20,12μL灭菌超纯水,得到10μM的各序列中间储备液。取20μL的各序列中间储备液(10μM),加入380μL的Tris-T缓冲液,得到500nM的各序列工作溶液,于95℃下变性5min,自然冷却至室温。
使用固定了NH2-Bio-EPO-α的SA芯片,以所述500nM的向右收缩适配体分子群中的各序列以及参比序列39nt为分析物,采用单点筛选法进行SPR动力学分析。具体实验条件同实施例7。进样后,待分析物与SA芯片上的EPO-α结合,生成复合物,进样结束,注入空白缓冲溶液,芯片上的复合物发生解离,各序列单点RU值变化曲线图如图7所示。
结果表明,只有能够形成高级结构GQ的R37、R35、R33、R31具有亲和活性,特别地,该分子群中,7A8A位点的碱基是否充分暴露对响应值有较大的影响。7A8A位点的的碱基完全暴露(R33)较之被掩蔽(R35、R37)时,适配体的活性基本恢复。而7A8A位点的碱基去除前(R33)和去除后(R31),响应值基本相同。
针对R37、R35、R33、R31这三条活性序列,分别选取浓度为31.25-1000nM(R37);125-4000nM(R35);31.25-1000nM(R33);31.25-1000nM(R31),进行MCK分析。具体SPR条件同实施例7。得到亲和力和动力学参数,如表3所示。结果表明,其KD值分别为560±170nM、1300±150nM、140±40nM和130±30nM,呈现从高到低逐渐降低的特征。
表3向右收缩适配体分子群的亲和力和动力学参数
实施例10两端延展适配体分子群在SA芯片上的单点活性评价及活性序列的MCK分析
待评价的适配体序列为:两端延展适配体分子群中的所有序列,包括Stem-2bp、Stem-3bp、Stem-5bp、Stem-6bp、Stem-7bp、Stem-10bp、Stem-12bp、Stem-15bp,以及参比序列39nt,共10条序列。分别量取2μL上述10条序列的储备液(储备液的浓度为100μM),加入4μL的5×Tris-HCl缓冲液,2μL的1wt%Tween 20,12μL灭菌超纯水,得到10μM的各序列储备液。取20μL的各序列储备液(10μM),加入380μL的Tris-T缓冲液,得到500nM的各序列工作溶液,于95℃下变性5min,自然冷却至室温。
使用固定了NH2-Bio-EPO-α的SA芯片,以所述500nM的两端延展适配体分子群中的各序列以及参比序列39nt为分析物,采用单点筛选法进行SPR动力学分析。具体实验条件同实施例7。进样后,待分析物与SA芯片上的EPO-α结合,生成复合物,进样结束,注入空白缓冲溶液,芯片上的复合物发生解离,各序列单点RU值变化曲线图如图8所示。
结果表明,从Stem-2bp到Stem-7bp,随着茎部的延长,响应值逐渐升高。而从Stem-7bp一直到Stem-15bp,响应值则逐渐下降。除Stem-5bp外,其它序列的响应值均高于阳性序列39nt。而亲和活性的最大增强出现于Stem-7bp。
针对两端延展适配体分子群中的所有序列,分别选取浓度为31.25-1000nM(2bp)、125-4000nM(3bp)、31.25-500nM(5bp)、31.25-500nM(6bp)、31.25-500nM(7bp)、31.25-500nM(10bp)、31.25-500nM(12bp)、31.25-500nM(15bp),进行MCK分析。具体SPR条件同实施例7。得到亲和力和动力学参数,如表4所示。结果表明,KD值从Stem-2bp一直到Stem-7bp逐渐降低,然从Stem-7bp到Stem-15bp,其KD值基本趋平。亲和活性最高的为Stem-7bp。同时,Stem-7bp、Stem-6bp的亲和活性均优于39nt。
表4两端延展适配体分子群的亲和力和动力学参数
实施例11两端收缩适配体分子群在SA芯片上的单点活性评价及活性序列的MCK分析
待评价的适配体序列为:两端收缩适配体分子群中的所有序列,包括In21、In23、In25、In27、In29、In31、In33、In35、In37,以及参比序列39nt,共10条序列。分别量取2μL上述10条序列的储备液(该储备液用超纯水配置,浓度为100μM),加入4μL的5×Tris-HCl缓冲液,2μL的1wt%Tween 20,12μL灭菌超纯水,得到10μM的各序列中 间储备液。取20μL的各序列中间储备液(10μM),加入380μL的Tris-T缓冲液,得到500nM的各序列工作溶液,于95℃下变性5min,自然冷却至室温。
使用固定了NH2-Bio-EPO-α的SA芯片,以所述500nM的两端收缩适配体分子群中的各序列以及参比序列39nt为分析物,采用单点筛选法进行SPR动力学分析。具体实验条件同实施例7。进样后,待分析物与SA芯片上的EPO-α结合,生成复合物,进样结束,注入空白缓冲溶液,芯片上的复合物发生解离,各序列单点RU值变化曲线图如图9所示。
实验结果表明,随着茎部的缩短,响应值逐渐降低,而保留了整个环部的In27亲和力完全恢复,提示可能形成了新的高级结构。
针对In37、In35、In33、In31、In29、In27、In25这七条活性序列,分别选取浓度为31.25-1000nM(In37、In31)、125-4000nM(In35、In33、In25)、62.5-2000nM(In29)、12.5-400nM(In27),进行MCK分析。具体SPR条件同实施例7。得到亲和力和动力学参数,如表5所示。结果表明,KD值从In37到In33逐渐升高,然后又逐渐降低,In27的KD值降低最为明显,而后In25的KD值又急剧升高,In 27是所有剪裁序列中亲和活性最高的序列。
表5两端收缩适配体分子群的亲和力和动力学参数
实施例12采用圆二色谱测定In27可能形成的高级结构
分别取0.6OD(0.6OD相当于0.6*33μg=19.8μg)的In27冻干粉溶解于含有不同浓度K+的三种Tris-HCl(20mM Tris,140mM NaCl,5mM MgCl2,以HCl调至pH 7.4-7.6)缓冲液中,其中KCl分别为0、5、100mM。设定温度为25℃,进行圆二色谱测定,得到如图10所示的圆二色谱图。所用仪器为MOS-450多功能圆二色光谱仪,法国BioLogic Science Instruments公司,比色池光程1cm。
从图10所示的圆二色谱图可以看出,在不同浓度K+的Tris-HCl缓冲液中,In27均在260nm处形成了负峰,在290nm处形成了正峰,符合反平行G-四聚体(GQ)的高级构象特征。说明In27在Tris-HCl缓冲液中形成了反平行G四聚体结构。K+具有诱导促进G-四聚体高级结构形成的作用,但该圆二色谱图中可以看出,K+的浓度为0和5mM时,所形成的谱图几乎重合,说明In27自身便可形成G-四聚体,且该高级结构较为稳定,K+存在与否对In27的高级结构影响不显著。
实施例13构建In27的定点突变适配体分子群
针对最短适配体In27,构建其定点突变分子群,以明确In27与EPO-α特异性结合时,具体碱基位点的贡献。
In27的二级序列特征为:仅保留了39nt的环部序列,其一级结构序列特征为,--GG---------GGGG--GG--GGG;结合其圆二色谱结果,推测In27可能形成了以GQ为骨架的三级结构。
除In27上的GG骨架外,其它序列按-GG(G)-为间隔,划分为D1~5个结构域(Domain),进行定点突变。如图2所示,构建了各定点突变分子群,共6组,分别命名为GQ、D1、D2、D3&4、D5和简并序列定点突变分子群,共31条序列。突变遵循的大原则是:基于经验的试错法,其目的在于找到简并序列,使该优化适配体能够有更广泛的适用性。考虑到G-C配对,不引入C碱基;考虑到G四聚体高级结构的连接子一般为Tn或T+A等有序结构,将非T碱基换为T;对于有序结构如从第9位T开始的T5G4T2G2T3G3(此处“T5G4T2G2T3G3”是指连续的5个T、4个G等等),仅采取连续T截短、T突变为A等处理;对于前8位中的非G碱基,基本上采用穷举法。
如表6中所示,
D1定点突变适配体分子群包括:1A碱基改为C/T/G/X、2A碱基改为T/G、以及1A2A碱基同时改为T碱基等7条序列;
D2定点突变适配体分子群包括:6C碱基改为T/A/G/X、8G碱基改为T碱基、6C8G同时改为T碱基、11T碱基改为X,以及10T11T碱基同时改为X等8条序列;
D3&4定点突变适配体分子群包括:18T碱基改为A碱基、以及23T碱基改为A碱基等2条序列;
D5定点突变适配体分子群包括:27G碱基改为A/T/X等3条序列;
GQ定点突变适配体分子群包括:25G改为T/X(X代表无),即25T、25X;在6位的C改为T的前提下,14G、15G、16G、17G中的G碱基分别改为T碱基,即为14T、15T、16T、17T,以及14G15G、15G16G、16G17G、14G17G中的两个G碱基均改为T碱基,即14T15T、15T16T、16T17T、14T17T等10条序列;
简并序列适配体分子群包括1A6C碱基同时改为T碱基,即1T6T;1A6C27G碱基分别改为1T6T27A碱基;以及1A27G碱基改为1T27A碱基等3条序列。
上述适配体步进型组合文库由上海生工生物工程公司合成。
表6In27的定点突变适配体分子群
注:表6中,“~”表示该位置的碱基缺失。
实施例14GQ定点突变分子群在SA芯片上的单点活性评价
GQ定点突变适配体分子群中的所有序列,包括如SEQ ID NO.57~SEQ ID NO.66所示的In27-6T14T、In27-6T15T、In27-6T16T、In27-6T17T、In27-6T1415TT、In27-6T1516TT、In27-6T1617TT、In27-6T1417T、In27-6T25X、In27-6T25T,以及参比序列In27(如SEQ ID NO.33所示),共11条序列,分别量取2μL上述11条序列的储备液(该储备液用超纯水配置,浓度为100μM),加入4μL的5×Tris-HCl缓冲液,2μL的1wt%Tween 20,12μL灭菌超纯水,得到10μM的各序列中间储备液。取20μL的各序列中间储备液(10μM),加入380μL的Tris-T缓冲液,得到500nM的各序列工作溶液,于95℃下变性5min,自 然冷却至室温。
使用固定了NH2-Bio-EPO-α的SA芯片,以所述500nM的GQ定点突变适配体分子群中的各序列以及参比序列In27为分析物,采用单点筛选法进行SPR动力学分析。具体实验条件同实施例7。进样后,待分析物与SA芯片上的EPO-α结合,生成复合物,进样结束,注入空白缓冲溶液,芯片上的复合物发生解离,各序列单点RU值变化曲线如图11A和图11B所示。
结果表明,与In27相比,对14到17位上的G碱基进行突变,其响应值都大大降低,说明在第三对GG骨架的形成可能具有一定的流动性。而In27-25X和In27-25T活性也基本丧失,说明25G可能参与了第四对GG骨架形成。
实施例15D1定点突变适配体分子群在SA芯片上的单点活性评价
D1定点突变适配体分子群中的所有序列,包括如SEQ ID NO.37~SEQ ID NO.43所示的In27-1X、In27-1T、In27-1G、In27-1C、In27-2G、In27-2T、In27-1T2T,以及参比序列In27、39nt,共9条序列,分别量取2μL上述9条序列的储备液(该储备液用超纯水配置,浓度为100μM),加入4μL的5×Tris-HCl缓冲液,2μL的1wt%Tween 20,12μL灭菌超纯水,得到10μM的各序列中间储备液。取20μL的各序列中间储备液(10μM),加入380μL的Tris-T缓冲液,得到500nM的各序列工作溶液,于95℃下变性5min,自然冷却至室温。
使用固定了NH2-Bio-EPO-α的SA芯片,以所述500nM的D1定点突变适配体分子群中的各序列以及参比序列In27、39nt为分析物,采用单点筛选法进行SPR动力学分析。具体实验条件同实施例7。各序列单点RU值变化曲线如图12所示。
结果表明,与In27相比,In27-1T的响应值略有升高,In27-1G与In27的响应值基本持平,In27-1C的响应值与39nt基本持平。而In27-2G、In27-2T和In27-1T2T的响应值下降明显。说明在D1结构域中1A能改为其它三种碱基而不影响活性,而2A碱基具有特异性。
实施例16D2定点突变适配体分子群在SA芯片上的单点活性评价
D2定点突变适配体分子群中的所有序列,包括如SEQ ID NO.44~SEQ ID NO.51所示的In27-6X、In27-6T、In27-6A、In27-6G、In27-8T、In27-6T8T、In27-11X、In27-10X11X,以及参比序列In27,共9条序列,分别量取2μL上述9条序列的储备液(100μM),加入4μL的5×Tris-HCl缓冲液,2μL的1wt%Tween 20,12μL灭菌超纯水,得到10μM的各序列中间储备液。取20μL的各序列中间储备液(10μM),加入380μL的Tris-T缓冲液,得到500nM的各序列工作溶液,于95℃下变性5min,自然冷却至室温。
使用固定了NH2-Bio-EPO-α的SA芯片,以所述500nM的D2定点突变适配体分子群中各序列以及参比序列In27为分析物,采用单点筛选法进行SPR动力学分析。具体实 验条件同实施例7。各序列单点RU值变化曲线如图13所示。
结果表明,与In27相比,In27-6T(RU 60)的亲和活性略高,而In27-6A(RU 30)和In27-6G(RU 29)的响应值有所下降。In27-6X(RU 15)、In27-11X(RU 3)和In27-10X11X(RU 5)的响应值下降明显,且与其它序列的动力学特征不同,这三条序列结合和解离的速率都非常迅速。从而说明D2结构域是重要的识别位点,其11个碱基形成的结构保证了识别活性的发挥。
实施例17D3&D4定点突变适配体分子群在SA芯片上的单点活性评价
D3&D4定点突变适配体分子群中的所有序列,包括如SEQ ID NO.52~SEQ ID NO.53所示的In27-18A、In27-23A,以及参比序列In27,共3条序列,分别量取2μL上述3条序列的储备液(该储备液用超纯水配置,浓度为100μM),加入4μL的5×Tris-HCl缓冲液,2μL的1wt%Tween 20,12μL灭菌超纯水,得到10μM的各序列中间储备液。取20μL的各序列中间储备液(10μM),加入380μL的Tris-T缓冲液,得到500nM的各序列工作溶液,于95℃下变性5min,自然冷却至室温。
使用固定了NH2-Bio-EPO-α的SA芯片,以所述500nM的D3&D4定点突变适配体分子群中的各序列以及参比序列In27为分析物,采用单点筛选法进行SPR动力学分析。具体实验条件同实施例7。各序列单点RU值变化曲线如图14所示。
结果表明,与In27相比,In27-18A(RU 8)以及In27-23A(RU 8)的响应值下降明显。说明D3和D4结构域在识别过程中发挥着一定的作用。
实施例18D5定点突变适配体活性簇分子群在SA芯片上的单点活性评价
D5定点突变适配体分子群中的所有序列,包括如SEQ ID NO.54~SEQ ID NO.56所示的In27-27X、In27-27A、In27-27T,以及参比序列In27,共4条序列,分别量取2μL上述4条序列的储备液(该储备液用超纯水配置,浓度为100μM),加入4μL的5×Tris-HCl缓冲液,2μL的1wt%Tween 20,12μL灭菌超纯水,得到10μM的各序列中间储备液。取20μL的各序列中间储备液(10μM),加入380μL的Tris-T缓冲液,得到500nM的各序列工作溶液,于95℃下变性5min,自然冷却至室温。
使用固定了NH2-Bio-EPO-α的SA芯片,以所述500nM的D5定点突变适配体分子群中的各序列以及参比序列In27为分析物,采用单点筛选法进行SPR动力学分析。具体实验条件同实施例7。各序列单点RU值变化曲线如图15所示。
结果表明,与In27相比,In27-27X(RU 6)、In27-27T(RU 5)和In27-27A(RU 3)的响应值下降明显。说明27G碱基具有特异性。
实施例19简并序列适配体分子群在SA芯片上的单点活性评价
简并序列适配体分子群中的所有序列,包括如SEQ ID NO.67~SEQ ID NO.69所示的In27-1T6T、In27-1T27A、In27-1T6T27A,以及参比序列In27,共4条序列,分别量取2μL上述4条序列的储备液(该储备液用超纯水配置,浓度为100μM),加入4μL的5×Tris-HCl缓冲液,2μL的1%Tween 20,12μL灭菌超纯水,得到10μM的各序列中间储备液。取20μL的各序列中间储备液(10μM),加入380μL的Tris-T缓冲液,得到500nM的各序列工作溶液,于95℃下变性5min,自然冷却至室温。
使用固定了NH2-Bio-EPO-α的SA芯片,以所述500nM的简并序列适配体分子群中的各序列以及参比序列In27为分析物,采用单点筛选法进行SPR动力学分析。具体实验条件同实施例7。各序列单点RU值变化曲线如图16所示。
结果表明,与In27相比,1T6T的响应值明显升高,同时1T6T的响应值介于1T和6T之间。1T27A和1T27A6T响应值下降明显,再次说明27位碱基(G)在识别过程中发挥了重要的作用,而与两端1T27A的配对无关。
实施例20以In27为识别元件,与EPO-α进行亲和识别检测
取2μL的3’-dT12-Biotin-In27(由上海生工生物工程有限公司合成,在In27的3’端修饰上一个Biotin分子)储备液(该储备液用超纯水配置,浓度为100μM),加入5×Tris-HCl缓冲液8μL,4μL的1wt%Tween 20,26μL灭菌超纯水,得到5μM的3’-dT12-Biotin-In27中间储备液。取4μL的中间储备液(5μM),加入396μL的Tris-T缓冲液,得到50nM的固定溶液,于95℃下变性5min,4℃放置。
依上述方法,配置400μL的50nM的3’-Biotin-39nt(由上海生工生物工程有限公司合成,在39nt的3’端修饰上一个Biotin分子)的固定溶液。
取用一块新的SA芯片,固定前处理同实施例5。将50nM的3’-Biotin-39nt固定溶液,在流速10μL/min,进样时间300秒的条件下,固定于SA芯片通道2上,此时响应稳定性(stability)RU值上升了1550。将50nM的3’-dT12-Biotin-In27固定溶液,在流速10μL/min,进样时间300秒的条件下,固定于SA芯片通道4上,此时响应稳定性(stability)RU值上升了1800。
在SA芯片上,使用3’-Biotin-39nt(通道2)和3’-dT12-Biotin-In27(通道4),进行EPO-α和39nt及In27间的亲和力和动力学分析。结合时间为120秒,解离时间为300秒,流速为30μL/min。再生条件为5mM NaOH,进样时间15秒,流速为30μL/min。包含以空白溶剂为零点在内的EPO-α六个浓度的进样顺序为0、0、6.25、12.5、25、50、100、200nM,得到结合解离谱图,如图17A和图17B所示。
非线性拟合结果表明,39nt与EPO-α相互作用的解离常数KD=55nM,结合速率常数Ka=2.7×104M-1s-1,解离速率常数Kd=1.4×10-4s-1(U-value=1,Chi2=0.23);In27与EPO-α相互作用的解离常数KD=78nM,结合速率常数Ka=2.2×104M-1s-1,解离速率常数Kd=1.7×10-4s-1(U-value=1,Chi2=0.06)。说明In 27作为识别元件,很好地实现了对EPO-α蛋白的亲和识别与检测。
SEQUENCE LISTING
<110> 中国人民解放军军事医学科学院毒物药物研究所
<120> 一种适配体组合文库、其构建方法及用途
<130> IDC160136
<160> 69
<170> PatentIn version 3.5
<210> 1
<211> 39
<212> DNA
<213> 适配体
<400> 1
gattgaaagg tctgtttttg gggttggttt gggtcaata 39
<210> 2
<211> 31
<212> DNA
<213> 适配体
<400> 2
gaaaggtctg tttttggggt tggtttgggt c 31
<210> 3
<211> 33
<212> DNA
<213> 适配体
<400> 3
agaaaggtct gtttttgggg ttggtttggg tct 33
<210> 4
<211> 37
<212> DNA
<213> 适配体
<400> 4
agagaaaggt ctgtttttgg ggttggtttg ggtctct 37
<210> 5
<211> 39
<212> DNA
<213> 适配体
<400> 5
gagagaaagg tctgtttttg gggttggttt gggtctctc 39
<210> 6
<211> 41
<212> DNA
<213> 适配体
<400> 6
agagagaaag gtctgttttt ggggttggtt tgggtctctc t 41
<210> 7
<211> 47
<212> DNA
<213> 适配体
<400> 7
gagagagaga aaggtctgtt tttggggttg gtttgggtct ctctctc 47
<210> 8
<211> 51
<212> DNA
<213> 适配体
<400> 8
gagagagaga gaaaggtctg tttttggggt tggtttgggt ctctctctct c 51
<210> 9
<211> 57
<212> DNA
<213> 适配体
<400> 9
agagagagag agagaaaggt ctgtttttgg ggttggtttg ggtctctctc tctctct 57
<210> 10
<211> 37
<212> DNA
<213> 适配体
<400> 10
gattgaaagg tctgtttttg gggttggttt gggtcaa 37
<210> 11
<211> 35
<212> DNA
<213> 适配体
<400> 11
gattgaaagg tctgtttttg gggttggttt gggtc 35
<210> 12
<211> 33
<212> DNA
<213> 适配体
<400> 12
gattgaaagg tctgtttttg gggttggttt ggg 33
<210> 13
<211> 31
<212> DNA
<213> 适配体
<400> 13
gattgaaagg tctgtttttg gggttggttt g 31
<210> 14
<211> 29
<212> DNA
<213> 适配体
<400> 14
gattgaaagg tctgtttttg gggttggtt 29
<210> 15
<211> 27
<212> DNA
<213> 适配体
<400> 15
gattgaaagg tctgtttttg gggttgg 27
<210> 16
<211> 25
<212> DNA
<213> 适配体
<400> 16
gattgaaagg tctgtttttg gggtt 25
<210> 17
<211> 23
<212> DNA
<213> 适配体
<400> 17
gattgaaagg tctgtttttg ggg 23
<210> 18
<211> 21
<212> DNA
<213> 适配体
<400> 18
gattgaaagg tctgtttttg g 21
<210> 19
<211> 37
<212> DNA
<213> 适配体
<400> 19
ttgaaaggtc tgtttttggg gttggtttgg gtcaata 37
<210> 20
<211> 35
<212> DNA
<213> 适配体
<400> 20
gaaaggtctg tttttggggt tggtttgggt caata 35
<210> 21
<211> 33
<212> DNA
<213> 适配体
<400> 21
aaggtctgtt tttggggttg gtttgggtca ata 33
<210> 22
<211> 31
<212> DNA
<213> 适配体
<400> 22
ggtctgtttt tggggttggt ttgggtcaat a 31
<210> 23
<211> 29
<212> DNA
<213> 适配体
<400> 23
tctgtttttg gggttggttt gggtcaata 29
<210> 24
<211> 27
<212> DNA
<213> 适配体
<400> 24
tgtttttggg gttggtttgg gtcaata 27
<210> 25
<211> 25
<212> DNA
<213> 适配体
<400> 25
tttttggggt tggtttgggt caata 25
<210> 26
<211> 23
<212> DNA
<213> 适配体
<400> 26
tttggggttg gtttgggtca ata 23
<210> 27
<211> 21
<212> DNA
<213> 适配体
<400> 27
tggggttggt ttgggtcaat a 21
<210> 28
<211> 37
<212> DNA
<213> 适配体
<400> 28
attgaaaggt ctgtttttgg ggttggtttg ggtcaat 37
<210> 29
<211> 35
<212> DNA
<213> 适配体
<400> 29
ttgaaaggtc tgtttttggg gttggtttgg gtcaa 35
<210> 30
<211> 33
<212> DNA
<213> 适配体
<400> 30
tgaaaggtct gtttttgggg ttggtttggg tca 33
<210> 31
<211> 31
<212> DNA
<213> 适配体
<400> 31
gaaaggtctg tttttggggt tggtttgggt c 31
<210> 32
<211> 29
<212> DNA
<213> 适配体
<400> 32
aaaggtctgt ttttggggtt ggtttgggt 29
<210> 33
<211> 27
<212> DNA
<213> 适配体
<400> 33
aaggtctgtt tttggggttg gtttggg 27
<210> 34
<211> 25
<212> DNA
<213> 适配体
<400> 34
aggtctgttt ttggggttgg tttgg 25
<210> 35
<211> 23
<212> DNA
<213> 适配体
<400> 35
ggtctgtttt tggggttggt ttg 23
<210> 36
<211> 21
<212> DNA
<213> 适配体
<400> 36
gtctgttttt ggggttggtt t 21
<210> 37
<211> 26
<212> DNA
<213> 适配体
<400> 37
aggtctgttt ttggggttgg tttggg 26
<210> 38
<211> 27
<212> DNA
<213> 适配体
<400> 38
taggtctgtt tttggggttg gtttggg 27
<210> 39
<211> 27
<212> DNA
<213> 适配体
<400> 39
gaggtctgtt tttggggttg gtttggg 27
<210> 40
<211> 27
<212> DNA
<213> 适配体
<400> 40
caggtctgtt tttggggttg gtttggg 27
<210> 41
<211> 27
<212> DNA
<213> 适配体
<400> 41
agggtctgtt tttggggttg gtttggg 27
<210> 42
<211> 27
<212> DNA
<213> 适配体
<400> 42
atggtctgtt tttggggttg gtttggg 27
<210> 43
<211> 27
<212> DNA
<213> 适配体
<400> 43
ttggtctgtt tttggggttg gtttggg 27
<210> 44
<211> 26
<212> DNA
<213> 适配体
<400> 44
aaggttgttt ttggggttgg tttggg 26
<210> 45
<211> 27
<212> DNA
<213> 适配体
<400> 45
aaggtttgtt tttggggttg gtttggg 27
<210> 46
<211> 27
<212> DNA
<213> 适配体
<400> 46
aaggtatgtt tttggggttg gtttggg 27
<210> 47
<211> 27
<212> DNA
<213> 适配体
<400> 47
aaggtgtgtt tttggggttg gtttggg 27
<210> 48
<211> 27
<212> DNA
<213> 适配体
<400> 48
aaggtctttt tttggggttg gtttggg 27
<210> 49
<211> 27
<212> DNA
<213> 适配体
<400> 49
aaggtttttt tttggggttg gtttggg 27
<210> 50
<211> 26
<212> DNA
<213> 适配体
<400> 50
aaggtctgtt ttggggttgg tttggg 26
<210> 51
<211> 25
<212> DNA
<213> 适配体
<400> 51
aaggtctgtt tggggttggt ttggg 25
<210> 52
<211> 27
<212> DNA
<213> 适配体
<400> 52
aaggtctgtt tttggggatg gtttggg 27
<210> 53
<211> 27
<212> DNA
<213> 适配体
<400> 53
aaggtctgtt tttggggttg gtatggg 27
<210> 54
<211> 26
<212> DNA
<213> 适配体
<400> 54
aaggtctgtt tttggggttg gtttgg 26
<210> 55
<211> 27
<212> DNA
<213> 适配体
<400> 55
aaggtctgtt tttggggttg gtttgga 27
<210> 56
<211> 27
<212> DNA
<213> 适配体
<400> 56
aaggtctgtt tttggggttg gtttggt 27
<210> 57
<211> 27
<212> DNA
<213> 适配体
<400> 57
aaggtttgtt ttttgggttg gtttggg 27
<210> 58
<211> 27
<212> DNA
<213> 适配体
<400> 58
aaggtttgtt tttgtggttg gtttggg 27
<210> 59
<211> 27
<212> DNA
<213> 适配体
<400> 59
aaggtttgtt tttggtgttg gtttggg 27
<210> 60
<211> 27
<212> DNA
<213> 适配体
<400> 60
aaggtttgtt tttgggtttg gtttggg 27
<210> 61
<211> 27
<212> DNA
<213> 适配体
<400> 61
aaggtttgtt tttttggttg gtttggg 27
<210> 62
<211> 27
<212> DNA
<213> 适配体
<400> 62
aaggtttgtt tttgttgttg gtttggg 27
<210> 63
<211> 27
<212> DNA
<213> 适配体
<400> 63
aaggtttgtt tttggttttg gtttggg 27
<210> 64
<211> 27
<212> DNA
<213> 适配体
<400> 64
aaggtttgtt ttttggtttg gtttggg 27
<210> 65
<211> 26
<212> DNA
<213> 适配体
<400> 65
aaggtttgtt tttggggttg gtttgg 26
<210> 66
<211> 27
<212> DNA
<213> 适配体
<400> 66
aaggtttgtt tttggggttg gttttgg 27
<210> 67
<211> 27
<212> DNA
<213> 适配体
<400> 67
taggtttgtt tttggggttg gtttggg 27
<210> 68
<211> 27
<212> DNA
<213> 适配体
<400> 68
taggtctgtt tttggggttg gtttgga 27
<210> 69
<211> 27
<212> DNA
<213> 适配体
<400> 69
taggtttgtt tttggggttg gtttgga 27
- 还没有人留言评论。精彩留言会获得点赞!
- 一种舌癌细胞的核酸适配体及试剂盒的制造方法与工艺
- 金黄色葡萄球菌肠毒素C2的核酸适配体C203及其筛选方法和应用与制造工艺
- 胰岛素核酸适体INS5及其制备方法与制造工艺
- 一种高亲和力兴奋剂核酸适体EPO4及其制备方法与制造工艺
- 一种邻苯二甲酸酯类增塑剂单链DNA核酸适配体及其筛选与表征方法及电化学传感器与制造工艺
- 基于高特异性核酸适体荧光探针INS1胰岛素试剂盒及其检测方法与制造工艺
- 运用核酸适配体与溶菌酶特异性识别并形成G四链体进行荧光显现潜指纹的方法与制造工艺
- 基于快速识别核酸适体荧光探针INS2胰岛素试剂盒及其检测方法与制造工艺
- 一种用于检测沙门氏菌的增敏型核酸适配体试纸条及其制备方法
- 一组特异性结合结核分枝杆菌的核酸适配体及其应用